Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Virus pseudotypés en tant qu’outil moléculaire pour surveiller les réponses immunitaires humorales contre le SRAS-CoV-2 via un test de neutralisation
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les virus pseudotypés (PV) sont des virions défectueux de réplication qui sont utilisés pour étudier les interactions hôte-virus dans des conditions plus sûres que la manipulation de virus authentiques. Nous présentons ici un protocole détaillé qui montre comment les PV du SRAS-CoV-2 peuvent être utilisés pour tester la capacité de neutralisation du sérum des patients après la vaccination contre la COVID-19.
Résumé
Les virus pseudotypés (PV) sont des outils moléculaires qui peuvent être utilisés pour étudier les interactions hôte-virus et pour tester la capacité neutralisante d’échantillons de sérum, en plus de leur utilisation plus connue en thérapie génique pour l’administration d’un gène d’intérêt. Les PV sont défectueux dans la réplication car le génome viral est divisé en différents plasmides qui ne sont pas incorporés dans les PV. Ce système sûr et polyvalent permet l’utilisation de PV dans des laboratoires de niveau de biosécurité 2. Nous présentons ici une méthodologie générale pour produire des PV lentiviraux à partir de trois plasmides mentionnés ici : (1) le plasmide de squelette portant le gène rapporteur nécessaire au suivi de l’infection ; (2) le plasmide d’emballage portant les gènes de toutes les protéines structurelles nécessaires à la génération des PV ; (3) le plasmide d’expression de la glycoprotéine de surface de l’enveloppe qui détermine le tropisme du virus et intervient dans l’entrée virale dans la cellule hôte. Dans ce travail, la pointe du SRAS-CoV-2 est la glycoprotéine d’enveloppe utilisée pour la production de lentivirus pseudotypés non réplicatifs du SRAS-CoV-2.
Brièvement, les cellules d’emballage (HEK293T) ont été co-transfectées avec les trois plasmides différents à l’aide de méthodes standard. Après 48 h, le surnageant contenant les PV a été récolté, filtré et stocké à -80 °C. L’infectiosité des PV du SRAS-CoV-2 a été testée en étudiant l’expression du gène rapporteur (luciférase) dans une lignée cellulaire cible 48 h après l’infection. Plus la valeur des unités de luminescence relative (RLU) est élevée, plus le taux d’infection/transduction est élevé. De plus, les PV infectieux ont été ajoutés aux échantillons de sérum dilués en série pour étudier le processus de neutralisation de l’entrée des pseudovirus dans les cellules cibles, mesuré par la réduction de l’intensité du RLU : des valeurs inférieures correspondant à une activité neutralisante élevée.
Introduction
Les virus pseudotypés (PV) sont des outils moléculaires utilisés en microbiologie pour étudier les interactions hôte-virus et pathogène-pathogène 1,2,3,4. Les PV sont constitués d’une partie interne, le noyau viral qui protège le génome viral, et d’une partie externe, les glycoprotéines d’enveloppe à la surface du virus qui définissent le tropisme5. Un pseudovirus est incompétent en matière de réplication dans la cellule cible car il ne contient pas toute l’information génétique nécessaire pour générer de nouvelles particules virales. Cette combinaison de caractéristiques particulières fait des PV une alternative sûre à un virus de type sauvage. Les virus de type sauvage, en revanche, sont hautement pathogènes et ne peuvent pas être utilisés dans les laboratoires BSL 2 pour l’analyse6.
L’infectiosité des PV peut être surveillée par un gène rapporteur, généralement codant pour une protéine fluorescente (GFP, RFP, YFP) ou une enzyme qui produit des produits chimiluminescents (luciférase). Celui-ci est contenu dans l’un des plasmides utilisés pour la production de PV et incorporé dans le génome du pseudovirus7.
Il existe actuellement plusieurs types de cœurs photovoltaïques, notamment des particules dérivées de lentiviraux basées sur le génome du VIH-1. Le grand avantage des PV à base de VIH-1 par rapport aux autres plateformes est leur processus d’intégration intrinsèque dans le génome de la cellule cible8. Bien que le VIH-1 soit un virus très contagieux et qu’il soit l’agent causal du sida, ces vecteurs lentiviraux peuvent être utilisés en toute sécurité en raison des nombreuses étapes d’optimisation au fil des ans. Des conditions de sécurité optimales ont été obtenues avec l’introduction de vecteurs lentiviraux de 2e génération, dans lesquels les gènes viraux ont été appauvris sans influencer les capacités de transduction9. Les 3eet 4egénérations ont contribué à accroître la sécurité de la manipulation des vecteurs lentiviraux avec la division du génome viral en plasmides distincts10,11. Les dernières générations de PV sont généralement utilisées pour produire des vecteurs lentiviraux pour la thérapie génique.
Les PV peuvent être utilisés pour étudier les interactions entre les virus et les cellules hôtes, tant pendant les phases de production que d’infection. Les PV sont particulièrement utilisés dans les essais de neutralisation des pseudovirus (PVNA). Les PVNA sont largement validés pour évaluer le potentiel de neutralisation du sérum ou du plasma en ciblant la glycoprotéine virale sur l’enveloppe du PV12,13. L’activité de neutralisation, exprimée par la concentration inhibitrice 50 (CI50), est définie comme la dilution du sérum/plasma qui bloque 50 % de l’entrée des particules virales14. Dans ce protocole, nous avons décrit la mise en place d’un PVNA pour tester l’activité des anticorps contre le syndrome respiratoire aigu sévère - Coronavirus 2 (SARS-CoV-2) dans des sérums prélevés avant et après avoir reçu une dose de rappel du vaccin.
Protocole
Le présent protocole a été approuvé par le Comité d’éthique de l’Université de Vérone (protocole d’approbation numéro 1538) et suit les directives de celui-ci. Le consentement écrit éclairé des sujets humains participant à l’étude a été obtenu. Des échantillons de sang total ont été prélevés sur des volontaires du personnel de santé qui étaient en train de recevoir des vaccins contre le SRAS-CoV-2. Ces échantillons ont été prélevés dans des tubes en plastique contenant des anticoagulants pour l’isolement ultérieur du sérum15.
Tous les processus suivants doivent être effectués dans une hotte biologique de classe 2, travaillant dans des conditions stériles. La manipulation des virus doit être effectuée avec précaution et tous les déchets doivent être neutralisés dans une solution d’eau de Javel diluée. Une vue d’ensemble du protocole est présentée à la figure 1.
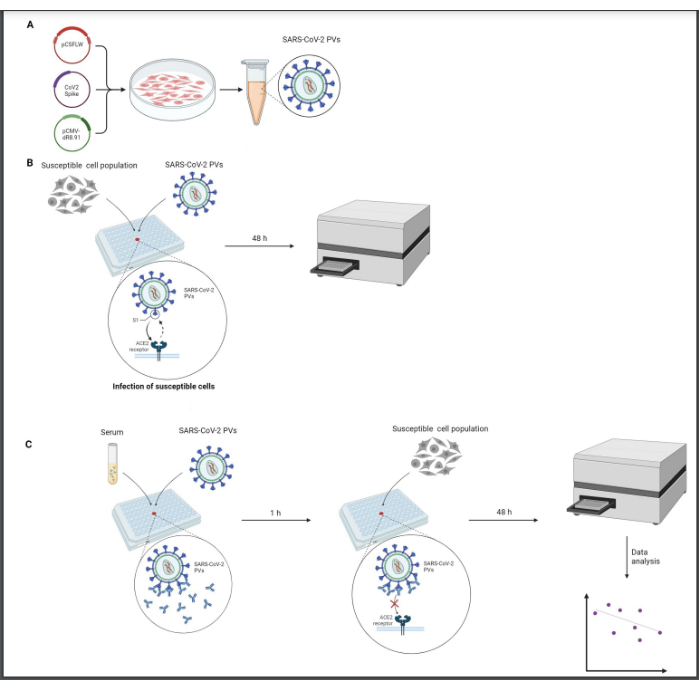
Figure 1 : Représentation graphique d’un essai de neutralisation. (A) Production PV, (B) titrage PV et (C) Essai de neutralisation. Toutes les procédures sont réalisées dans une hotte biologique de classe 2 dans des conditions stériles. L’étape de titrage (B) doit être effectuée pour normaliser les niveaux d’infectiosité des PV avant de les utiliser dans l’essai de neutralisation (C). Cette figurine a été créée avec BioRender. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Production de PV du SARS-CoV-2 et test d’infectiosité
- Semez 5 x 10cellules de 5 HEK293T dans le milieu Eagle modifié complet de Dulbecco (DMEM, haute teneur en glucose, 10 % de sérum de veau fœtal (FBS), 1 % de L-glutamine, 1 % de pénicilline/streptomycine) dans une plaque à 6 puits (6WP) pour atteindre une densité cellulaire appropriée compatible avec le réactif de transfection utilisé. Dans le cas d’une transfection avec de la polyehtylénimine (PEI) (préparer le réactif en suivant les instructions du fabricant), s’assurer que les cellules atteignent une densité de 40 à 60 % le jour de la transfection (étape 1.3). Conservez les cellules dans un incubateur humidifié à 37 °C et 5 % de CO2.
- Avant la transfection, remplacer le milieu cellulaire épuisé par un milieu frais sans antibiotiques (DMEM, hyperglycémie, 10 % FBS, 1 % de L-glutamine) pour obtenir une efficacité de transfection plus élevée.
REMARQUE : Le lendemain de l’ensemencement, HEK293T cellules sont prêtes à être transfectées. - Transfectez les cellules adhérentes HEK293T avec un réactif de transfection approprié selon les instructions du fabricant. Si vous utilisez PEI, préparez deux mélanges et suivez les étapes ci-dessous.
- Pour préparer le mélange A, ajouter 500 ng de plasmide d’emballage pCMV-dR8.9116, 750 ng de plasmide rapporteurpCSFLW 16 et 450 ng de plasmide exprimant Spike du SRAS-CoV-2 dans 100 μL de milieu sérique réduit.
- Pour préparer le mélange B, ajouter 17,5 μL de PEI (concentration : 1 mg/mL) à 100 μL de milieu sérique réduit.
- Laisser les deux mélanges incuber à température ambiante (RT) pendant 5 min. Ensuite, mélangez le contenu des deux tubes en ajoutant le mélange PEI B au mélange d’ADN A.
- Incubez le tube pendant 20 à 30 min à RT. Effleurez doucement le tube toutes les 3-4 min pour améliorer le mélange. Enfin, ajoutez le mélange aux HEK293T cellules.
- 16 à 20 h après la transfection, remplacer le milieu de culture par du DMEM frais et complet. Incuber à 37 °C et 5 % de CO2, pour permettre la production de PV par des cellules transfectées.
- 72 h après la transfection, récolter le surnageant contenant des PV. Ensuite, centrifugez à 1600 x g pendant 7 min à température ambiante pour éliminer les débris cellulaires et les cellules mortes et filtrez-les à travers un filtre en acétate de cellulose de 0,45 μm.
- ÉTAPE FACULTATIVE : Pour augmenter le rendement final du titre PV, effectuez plusieurs transfections, mettez en commun le milieu cellulaire contenant des PV et concentrez-le à l’aide de tubes de concentration.
- Passer directement aux étapes suivantes (« Titrage PV », section 2) ou aliquoter le milieu contenant PV dans des tubes appropriés à stocker à -80 °C jusqu’à utilisation. Préparer une aliquote supplémentaire (400-500 μL) à utiliser pour le titrage.
REMARQUE : La fabrication de plusieurs aliquotes garantira la reproductibilité entre les expériences en évitant les cycles excessifs de décongélation-congélation.
2. Titrage des PV
- Utiliser le milieu frais contenant du PV pour les étapes suivantes ou décongeler l’aliquote d’essai (étape 1.7) pour effectuer le titrage de la nouvelle souche virale. La congélation d’aliquotes d’un même stock PV garantira la reproductibilité.
- Ajouter 50 μL de DMEM complet (ou milieu complet compatible avec la lignée cellulaire cible utilisée) dans tous les puits d’une plaque à 96 puits (96WP) nécessaire pour tester en double le stock PV, en laissant la ligne « A » vide. Ajouter 100 μL de stock de PV à la rangée « A ». En fonction du nombre de préparations à tester, laissez une colonne sans virus comme témoin « cellulaire seulement » (figure 2).
- Pipeter 50 μL de la rangée A à la rangée B et répéter ce processus jusqu’à la rangée G pour obtenir des dilutions en série de l’étalon initial. Jetez l’excédent de volume de la dernière rangée.
- Détacher les cellules à l’aide de trypsine/acide éthylènediaminetétraacétique 1x (EDTA) dans le sérum physiologique tampon phosphate 1x (DPBS 1x) de Dulbecco, après avoir retiré le milieu usé et lavé les cellules avec DPBS 1x deux fois. Préparer les cellules à une densité de 4 x 105 cellules/mL.
REMARQUE : Dans ce protocole, l’infection par les PV a été testée sur la lignée cellulaire sensible HEK293T/ACE2 ; ces cellules ont été dérivées de HEK293T, transduites à l’aide d’un vecteur lentiviral pour exprimer le récepteur ACE2. - Ajouter 50 μL de suspension cellulaire dans chaque puits pour assurer un nombre de cellules de 2 x 104 cellules par puits.
- Incuber à 37 °C et 5% de CO2, pendant 48 h.
- Après l’incubation, effectuez le test de la luciférase pour obtenir la lecture selon les instructions du fabricant. Ajouter 100 μL de réactif de luciférase dans les puits et incuber dans l’obscurité à RT pendant 2 min. Déplacez le contenu de chaque puits sur une plaque noire à 96 puits (compatible avec le lecteur de plaques disponible) et lisez les plaques dans un lecteur de plaques à 96 puits.
REMARQUE : Le luminomètre utilisé pour la lecture de la luciférase produira un fichier de feuille de calcul avec les données brutes non traitées qui seront utilisées pour l’analyse en aval (dans ce cas, un fichier Excel). L’infectiosité du virus sera exprimée en unités de luminescence relative (RLU) (décrites au paragraphe 4.1).
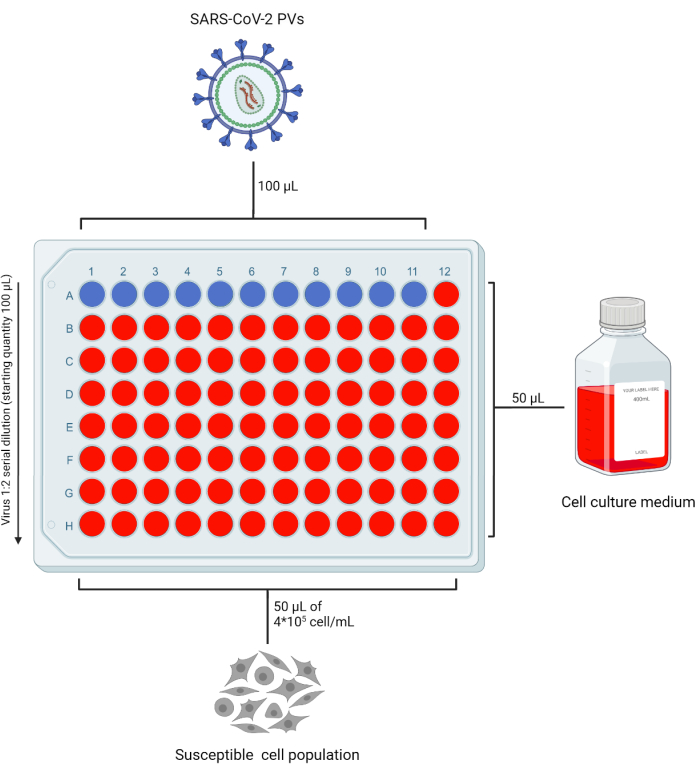
Figure 2 : Disposition représentative d’une plaque à 96 puits pour le titrage des PV. Un volume fixe de surnageant contenant du PV est ajouté à la rangée A, colonnes 1 à 11, et dilué en série. La dernière colonne est laissée en tant que contrôle « cellule uniquement ». Cette figurine a été créée avec BioRender. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Essai de neutralisation
- Décongeler les sérums des patients sur de la glace. Inactiver les échantillons de sérum en les incubant à 56 °C pendant 30 min.
- Dans une plaque à 96 puits, ajouter 50 μL de DMEM frais et complet (ou milieu complet compatible avec la lignée cellulaire cible utilisée) dans chacun des puits suivants : de la rangée B (colonnes 1 à 10) à la rangée H (colonnes 1 à 10). Mettez 95 μL de DMEM frais et complet dans la rangée A (colonnes 1 à 10). Ajouter 50 μL et 100 μL de DMEM complet dans les puits des colonnes 11 et 12, respectivement. Il s’agira respectivement des témoins infectés (contrôle du virus, ou VC) et non infectés (cellule seule, ou CC) (Figure 3).
- Ajouter 5 μL d’échantillons de sérum/plasma inactivés par la chaleur dans la rangée A (colonnes 1 à 10). Chaque échantillon sera en double. À l’aide d’une pipette multicanaux, mélanger les échantillons de la première rangée et déplacer 50 μL de milieu contenant du sérum de la rangée A à la rangée B. Répétez ce processus jusqu’à la dernière rangée (Figure 3). Jeter les 50 μL restants.
- Décongeler le nombre nécessaire d’aliquotes de PV et diluer à ≥ 104 RLU/mL. Ajouter 50 μL du milieu contenant du PV dilué dans chaque puits (de la colonne 1 à la colonne 11) à l’aide d’une pipette multicanal pour obtenir une dilution de 1 :1 du sérum/plasma inactivé par la chaleur en virus. Incuber à 37 °C et 5 % de CO2 pendant 1 h pour permettre aux anticorps contenus dans les échantillons de sérum de se lier à la protéine de pointe du SRAS-CoV-2 sur les PV.
- Préparer au moins 5 mL de suspension de cellules sensibles (HEK293T/ACE2) à une densité cellulaire de 4 x 105 cellules/mL. Ajouter 50 μL de la suspension cellulaire dans chaque puits et incuber à 37 °C et 5 % de CO2, pendant 48 h.
- Après l’incubation, effectuez la lecture du dosage de la luciférase conformément aux instructions du fabricant, comme décrit à l’étape 2.7.
REMARQUE : Le luminomètre utilisé pour la lecture de la luciférase produira un fichier de feuille de calcul (dans ce cas, .xlsx) avec les données brutes non traitées qui seront utilisées pour l’analyse en aval (le fichier de dosage de la luciférase).
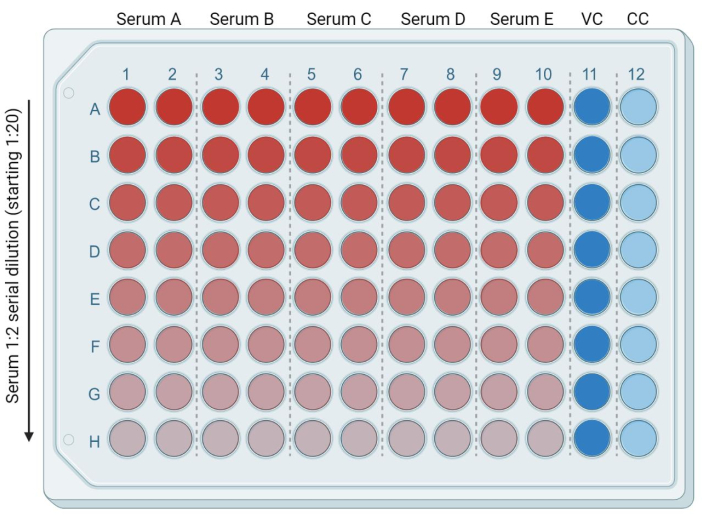
Figure 3 : Représentation de la plaque basée sur la dilution du sérum. Le rouge vif correspond à une plus grande quantité de sérum, et le couloir bleu vif (colonne 11) correspond au contrôle des cellules infectées (VC, contrôle du virus). La voie bleu clair (colonne 12) correspond aux cellules non infectées (CC, cellule témoin). Cette figurine a été créée avec BioRender. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Analyse de titrage
- Dans le fichier de dosage de la luciférase, attribuez les noms/titres aux échantillons correspondants.
- Multipliez la mesure du RLU par les facteurs de dilution (du haut vers le bas de la grille : 20x, 40x, 80x, 160x, 320x, 640x, 1,280x, 2,560x) pour obtenir le RLU/mL. Si différents facteurs de dilution sont utilisés, modifiez les facteurs de multiplication en conséquence.
- Calculer le RLU/mL moyen pour chaque préparation PV.
5. Analyse du test de neutralisation des PV
- Dans le fichier de feuille de calcul du test de la luciférase (dans ce cas, .xlsx), attribuez les titres correspondants aux échantillons testés. Entrez le facteur de dilution de l’échantillon (40s, 80x, 160x, 320x, 640x, 1,280x, 2,560x, 5,120x). Calculer le Log10 des facteurs de dilution.
- Calculer le RLU moyen des témoins non infectés et infectés (figure 3, colonnes 11 et 12, respectivement). Ces valeurs seront utiles pour la normalisation à l’étape 5.5.
- Ouvrez un nouveau document pour l’analyse des données. Sélectionnez l’analyse X/Y, saisissez X sous forme de nombres et Y sous le nom d’Entrez 2 valeurs répliquées dans des sous-colonnes côte à côte.
- Entrez les valeurs Log10 (dilution) sous forme de nombres X. Saisissez le RLU dupliqué des échantillons.
- Accédez à Analyser > Normaliser > Marquer tous les échantillons sur la même feuille. Saisissez les valeurs moyennes de VC et de CC dans Comment le 0 % est-il défini ? et Comment 100 % est-il défini ?, respectivement. Cliquez sur OK.
- Dans la feuille de données normalisée, reportez-vous à Analyser > analyses XY > Analyses non linéaires (ajustement de courbe). Marquez tous les échantillons et cliquez sur OK. Pour la fonction Dose-réponse - Inhibition, sélectionnez log(inhibiteur) vs réponse normalisée - pente variable.
- Sous Contraindre, remplacez HillSlope par Must least than 0.
- Sous Output(Sortie), sélectionnez Create summary table and graph (Créer un tableau récapitulatif et un graphique). Cliquez sur OK pour obtenir les analyses finales. Une feuille de travail avec un modèle pour l’analyse est fournie dans le fichier supplémentaire 1.
Résultats
Ce protocole décrit la production de PV du SARS-CoV-2 et une application en aval de ces PV pour analyser l’activité de neutralisation du sérum/plasma des sujets recevant un vaccin anti-COVID-1917. De plus, ce protocole peut être appliqué pour produire des pseudotypes de chaque variant préoccupant du SRAS-CoV-2 (COV) afin de tester l’évolution de la réponse neutralisante. Bien que ce protocole facilite l’étude de la réponse immunitaire humorale après la vaccination contre la COVID-...
Discussion
Bien que l’utilisation d’un virus de type sauvage simule l’infection réelle, les PV lentiviraux sont une option plus sûre pour étudier les mécanismes associés à l’entrée virale et à l’infection sans les exigences de sécurité strictes nécessaires pour travailler avec des virus pathogènes 4,20,21. Les PV sont composés d’un noyau viral défectueux en réplication entouré de la glycoprotéine d’enveloppe ...
Déclarations de divulgation
Les auteurs déclarent n’avoir aucun conflit d’intérêts.
Remerciements
Nous reconnaissons la contribution des travailleurs de la santé bénévoles. Ce projet a été soutenu par le Département d’Excellence 2023/2027, MUR, Italie. L’AR et la DZ ont été soutenues par PRIN2022 (financements de l’UE ; NextGenerationEU)
matériels
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | SARSTEDT | 83 1826 | |
| 6-well plate | SARSTEDT | 83 3920 | |
| 96-well plate | SARSTEDT | 8,33,924 | |
| Amicon Ultra-15 Centrifugal Filter Units | Merck | 10403892 | |
| Black Opaque 96-well Microplate | Perkin Elmer | 60005270 | |
| Dulbecco's Modified Eagle Medium | SIGMA-ALDRICH | D6546 - 500ML | |
| Dulbecco's phosphate buffered saline (PBS 1x) | AUROGENE | AU-L0615-500 | |
| Foetal Bovine Serum | AUROGENE | AU-S1810-500 | |
| Graphpad Prism version 7 | graphpad dotmatics | NA | In the manuscript, we replace the commercial name with 'data analysis program' |
| HEK293T cells | ATCC | CRL-3216 | |
| HEK293T/ACE2 cells | ATCC | CRL-3216 | HEK293T has been transduced to overexpress ACE2 with a lentiviral vector. |
| L-glutamine | AUROGENE | AU-X0550-100 | |
| Luminometer - Victor3 | Perkin Elmer | HH35000500 | In the manuscript, we replace the commercial name with 'luminometer' |
| Opti-MEM | Thermo Fisher Scientific | 11058021 | In the manuscript, we replace the commercial name with 'reduced serum medium' |
| p8.91 packaging plasmid | Di Genova et al., 2021 | A kind gift from Prof. Nigel Temperton (ref 16.) | |
| pCSFLW reporter plasmid | Di Genova et al., 2021 | A kind gift from Prof. Nigel Temperton (ref 16.) | |
| Penicillin/streptomycin | AUROGENE | AU-L0022-100 | |
| Polyethylenimine, branched (PEI) (25 kDa) | SIGMA-ALDRICH | 408727 | |
| RRL.sin.cPPT.SFFV/Ace2.IRES-puro.WPRE (MT126) | Addgene | 145839 | This plasmid was used to generate HEK293Tcells/ACE2 |
| SARS-CoV-2 Spike expressing plasmid | Addgene | pGBW-m4137382 | |
| steadylite plus Reporter Gene Assay System | Perkin Elmer | 6066759 | In the manuscript, we replaced the commercial name with 'luciferase reading reagent' |
| Trypsin EDTA 1x | AUROGENE | AU-L0949-100 |
Références
- Ozaki, D. A., et al. International technology transfer of a GCLP-compliant HIV-1 neutralizing antibody assay for human clinical trials. Plos One. 7 (1), e30963 (2012).
- Pouget, M., et al. Generation of liposomes to study the effect of Mycobacterium tuberculosis lipids on HIV-1 cis- and trans-infections. International Journal of Molecular Sciences. 22 (4), 1945 (2021).
- McKay, L. G. A., et al. The HCV envelope glycoprotein down-modulates NF-κB signalling and associates with stimulation of the host endoplasmic reticulum stress pathway. Frontiers in Immunology. 13, 831695 (2022).
- Xiang, Q., Li, L., Wu, J., Tian, M., Fu, Y. Application of pseudovirus system in the development of vaccine, antiviral-drugs, and neutralizing antibodies. Microbiological Research. 258, 126993 (2022).
- Li, Q., Liu, Q., Huang, W., Li, X., Wang, Y. Current status on the development of pseudoviruses for enveloped viruses. Reviews in Medical Virology. 28, e1963 (2018).
- D'Apice, L., et al. Comparative analysis of the neutralizing activity against SARS-CoV-2 Wuhan-Hu-1 strain and variants of concern: Performance evaluation of a pseudovirus-based neutralization assay. Frontiers in Immunology. 13, 981693 (2022).
- Falzarano, D., Groseth, A., Hoenen, T. Development and application of reporter-expressing mononegaviruses: current challenges and perspectives. Antiviral Research. 103, 78-87 (2014).
- Gutierrez-Guerrero, A., Cosset, F. -. L., Verhoeyen, E. Lentiviral vector pseudotypes: Precious tools to improve gene modification of hematopoietic cells for research and gene therapy. Viruses. 12, 1016 (2020).
- Zufferey, R., Nagy, D., Mandel, R. J., Naldini, L., Trono, D. Multiply attenuated lentiviral vector achieves efficient gene delivery in vivo. Nature Biotechnology. 15 (9), 871-875 (1997).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. Journal of Virology. 72 (11), 8463-8471 (1998).
- Berkhout, B. A Fourth generation lentiviral Vector: Simplifying genomic gymnastics. Molecular Therapy. 25 (8), 1741-1743 (2017).
- Wu, X. Development and evaluation of a pseudovirus-luciferase assay for rapid and quantitative detection of neutralizing antibodies against Enterovirus 71. Plos One. 8 (6), e64116 (2013).
- Ferrara, F., et al. Development of lentiviral vectors pseudotyped with Influenza B hemagglutinins: application in vaccine immunogenicity, mAb potency, and sero-surveillance studies. Frontiers in Immunology. 12, 661379 (2021).
- Hu, J., et al. Development of cell-based pseudovirus entry assay to identify potential viral entry inhibitors and neutralizing antibodies against SARS-CoV-2. Genes & Diseases. 7 (4), 551-557 (2020).
- Dalle Carbonare, L., et al. Serology study after BTN162b2 vaccination in participants previously infected with SARS-CoV-2 in two different waves versus naïve. Communications Medicine. 1 (1), 38 (2021).
- Di Genova, C., et al. Production, titration, neutralisation, storage and lyophilisation of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) lentiviral pseudotypes. Bio-protocol. 11 (21), e4236 (2021).
- Chmielewska, A. M., Czarnota, A., Bieńkowska-Szewczyk, K., Grzyb, K. Immune response against SARS-CoV-2 variants: The role of neutralization assays. NPJ Vaccines. 6 (1), 1-8 (2021).
- Chen, Q., et al. Development and optimization of a sensitive pseudovirus-based assay for HIV-1 neutralizing antibodies detection using A3R5 cells. Human Vaccines & Immunotherapeutics. 14 (1), 199-208 (2018).
- Gauger, P. C., Vincent, A. L. Serum virus neutralization assay for detection and quantitation of serum neutralizing antibodies to influenza A virus in swine. Methods in Molecular Biology (Clifton, N.J). 2123, 321-333 (2020).
- Miglietta, R., Pastori, C., Venuti, A., Ochsenbauer, C., Lopalco, L. Synergy in monoclonal antibody neutralization of HIV-1 pseudoviruses and infectious molecular clones. Journal of Translational Medicine. 12 (1), 346 (2014).
- Chen, M., Zhang, X. -. E. Construction and applications of SARS-CoV-2 pseudoviruses: A mini review. International Journal of Biological Sciences. 17 (6), 1574-1580 (2021).
- Zipeto, D., et al. Induction of human immunodeficiency virus neutralizing antibodies using fusion complexes. Microbes and Infection. 8 (6), 1424-1433 (2006).
- WHO Coronavirus (COVID-19) Dashboard. Available from: https://covid19.who.int (2022)
- Zhou, P. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 579 (7798), 270-273 (2020).
- Chen, X., Huang, H., Ju, J., Sun, R., Zhang, J. Impact of vaccination on the COVID-19 pandemic in U.S. states. Scientific Reports. 12 (1), 1554 (2022).
- Stefani, C., Fantoni, T., Bissoli, M., Thomas, J., Ruggiero, A. HIV and SARS-CoV-2 Co-Infection: From Population Study Evidence to In Vitro Studies. Life. 12 (12), 2089 (2022).
- Watson, O. J., et al. Global impact of the first year of COVID-19 vaccination: a mathematical modelling study. The Lancet Infectious Diseases. 22 (9), 1293-1302 (2022).
- Cantoni, D. Analysis of antibody neutralisation activity against SARS-CoV-2 variants and seasonal human coronaviruses NL63, HKU1, and 229E induced by three different COVID-19 vaccine olatforms. Vaccines. 11 (1), 58 (2023).
- Siracusano, G., et al. Different decay of antibody response and VOC sensitivity in naïve and previously infected subjects at 15 weeks following vaccination with BNT162b2. Journal of Translational Medicine. 20 (1), 22 (2022).
- Ruggiero, A. SARS-CoV-2 vaccination elicits unconventional IgM specific responses in naïve and previously COVID-19-infected individuals. eBioMedicine. 77, (2022).
- Piubelli, C. Subjects who developed SARS-CoV-2 specific IgM after vaccination show a longer humoral immunity and a lower frequency of infection. eBioMedicine. 89, 104471 (2023).
- Zhang, G. F. Infectivity of pseudotyped SARS-CoV-2 variants of concern in different human cell types and inhibitory effects of recombinant spike protein and entry-related cellular factors. Journal of Medical Virology. 95 (1), e28437 (2023).
- da Costa, K. A. S. Influenza A (N1-N9) and Influenza B (B/Victoria and B/Yamagata) neuraminidase pseudotypes as tools for pandemic preparedness and improved influenza vaccine design. Vaccines. 10 (9), 1520 (2022).
- Condor Capcha, J. M. Generation of SARS-CoV-2 spike pseudotyped virus for viral entry and neutralization assays: a 1-week protocol. Frontiers in Cardiovascular Medicine. 7, 618651 (2021).
- Diomede, L., et al. Doxycycline inhibition of a pseudotyped virus transduction does not translate to inhibition of SARS-CoV-2 infectivity. Viruses. 13 (9), 1745 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon