Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La separación de una sola hebra de ADN, el ADN de doble cadena y el ARN de una comunidad ambiental viral mediante cromatografía de hidroxiapatita
En este artículo
Resumen
Se describe un método eficiente para separar ADN de cadena sencilla, doble cadena de ADN y las moléculas de ARN viral de las comunidades del medio ambiente. Los ácidos nucleicos son fraccionados utilizando cromatografía de hidroxiapatita con una creciente concentración de fosfato que contienen buffers. Este método permite el aislamiento de todos los tipos de ácido nucleico viral de las muestras ambientales.
Resumen
Los virus, particularmente los bacteriófagos (fagos), son las entidades biológicas más numerosas en 1,2 la Tierra. Los virus modulan la abundancia de la célula huésped y la diversidad, contribuir a los ciclos de los nutrientes, alterar anfitrión fenotipo de las células, y la influencia de la evolución de la célula huésped y de las comunidades viral a través de la transferencia lateral de genes 3. Numerosos estudios han puesto de relieve la asombrosa diversidad genética de los virus y su potencial funcional en una variedad de ambientes naturales.
Técnicas de metagenómica se han utilizado para estudiar la diversidad taxonómica y el potencial funcional de los complejos conjuntos viral cuyos miembros contienen ADN de cadena sencilla (ssDNA), el ADN de doble cadena (dsDNA) y los genotipos de ARN 4-9. Los protocolos actuales de construcción de la biblioteca para estudiar el medio ambiente que contienen ADN o ARN que contienen los virus requieren un tratamiento inicial de la nucleasa con el fin de quitar las plantillas no focalizados a 10. Sin embargo, una comprensión global de la dotación genética colectiva de la comunidad de virus y la diversidad del virus requiere el conocimiento de todos los miembros, independientemente de la composición del genoma. Fraccionamiento de ácidos nucleicos purificados subtipos proporciona un mecanismo eficaz para que el estudio conjuntos viral sin tener que sacrificar una parte de la firma genética de la comunidad.
Hidroxiapatita, una forma cristalina de fosfato de calcio, se ha empleado en la separación de los ácidos nucleicos, así como las proteínas y los microbios, desde 1960 11. Mediante la explotación de la interacción de cargas entre la carga positiva iones Ca 2 + de la hidroxiapatita y el esqueleto de fosfato con carga negativa de los subtipos de ácidos nucleicos, es posible eluir preferentemente cada subtipo de ácidos nucleicos independiente de los demás. Recientemente hemos empleado esta estrategia de forma independiente a fraccionar los genomas de ssDNA, dsDNA y los virus que contienen ARN en la preparación de la secuenciación del ADN 12. A continuación, presentamos un método para el fraccionamiento y la recuperación de ssDNA, dsDNA y el ARN viral ácidos nucleicos de los conjuntos mixtos viral mediante cromatografía de hidroxiapatita.
Protocolo
1. Preparación de las soluciones
Antes de realizar la cromatografía de hidroxiapatita, tampones de fosfato debe estar preparado y la hidroxiapatita debe estar correctamente hidratado.
- Solución de fosfato 1 M, pH 6,8: En un matraz de 1 litro se disuelven 119.98g de fosfato de sodio monobásico en 1 litro de agua estéril, tratada con DEPC H 2 O. Prepare un fosfato de sodio dibásico 1M solución en un frasco de 1 litro de disolución 141.96g de fosfato de sodio dibásico en 1 litro de agua estéril, tratada con DEPC H 2 O. Combine los mono-y di-solución en una proporción de 1:1. Lugar matraz en una placa de agitación y mezcla con una varilla de agitación magnética. Ajustar el pH a 6,8 mediante la adición de hidróxido de sodio (para aumentar el pH) o el ácido fosfórico (para disminuir el pH).
- Tampón fosfato 0.12M: En un matraz de 500 ml, 30 ml de combinar la solución de fosfato 1 M, pH 6,8, 2,5 ml de sodio al 10% dodecilsulfato (SDS), y 5 ml de 0,5 M EDTA. Añadir estéril tratada con DEPC H2O hasta un volumen de 225 ml. Ajustar el pH a 6.8. Añadir estéril tratada con DEPC H2O a 250 ml. Autoclave.
- Tampón fosfato 0.18M: En un matraz de 500 ml, 45 ml de combinar la solución de fosfato 1 M, pH 6,8, 2,5 ml de sodio al 10% dodecilsulfato (SDS), y 5 ml de 0,5 M EDTA. Añadir estéril tratada con DEPC H 2 O a un volumen de 225 ml. Ajustar el pH a 6.8. Añadir estéril tratada con DEPC H 2 O a 250 ml. Autoclave.
- Tampón fosfato 0,20 M: En un matraz de 500 ml, 50 ml de combinar la solución de fosfato 1 M, pH 6,8, 2,5 ml de sodio al 10% dodecilsulfato (SDS), y 5 ml de 0,5 M EDTA. Añadir estéril tratada con DEPC H 2 O a un volumen de 225 ml. Ajustar el pH a 6.8. Añadir estéril tratada con DEPC H 2 O a 250 ml. Autoclave.
- 0,40 M tampón fosfato: en un frasco de 500 ml, 100 ml de combinar la solución de fosfato 1 M, pH 6,8, 2,5 ml de sodio al 10% dodecilsulfato (SDS), y 5 ml de 0,5 M EDTA. Añadir estéril tratada con DEPC H 2 O a un volumen de 225 ml. Ajustar el pH a 6.8. Añadir estéril tratada con DEPC H 2 O a 250 ml. Autoclave.
- "Tampón fosfato 1.00M" (concentración real de fosfato en esta solución es 0,91 M): En un matraz de 500 ml, 30 ml de combinar la solución de fosfato 1 M, pH6.8, 2,5 ml de sodio al 10% dodecilsulfato (SDS), y 5 ml de 0,5 M EDTA. Añadir estéril tratada con DEPC H2O hasta un volumen de 225 ml. Ajustar el pH a 6.8. Añadir estéril tratada con DEPC H 2 O a 250 ml. Autoclave.
- Hidratación de hidroxiapatita: Peso de 1 g de hidroxiapatita y añadir a un tubo de 50 ml. Agregar 6 ml de tampón fosfato 0.12M a la hidroxiapatita e invierta para mezclar. Antes de su uso llevar la temperatura a 60 ° C y mezclar bien. Almacenar por largos períodos de tiempo a 4 ° C.
- Antes de su uso, se equilibran todos los tampones de fosfato de hidroxiapatita y la hidratación a 60 ° C.
2. Preparación de la columna Econo-
- Cerrar llave de paso. Enjuague varias veces la columna H 2 O desionizada invirtiendo varias veces por la columna y decantación.
- Retire la llave de paso de la Econo-columna y la columna de autoclave para esterilizar.
- Vuelva a colocar y cerrar llave de paso. Añadir 1 ml de Sigmacote a la estéril Econo-columna y cubra todas las superficies de vidrio. Abrir la llave y se decanta.
- Coloque la columna a un baño de agua circulante y ajustar la temperatura a 60 ° C. Para einsure la menor cantidad de pérdida de calor a través de la columna de envolver la columna en un agente aislante como papel de aluminio o aislamiento de tuberías de espuma se recomienda.
- Enjuague la columna Econo-dos veces con agua estéril, libre de RNasa H 2 O y luego dos veces con agua estéril, libre de RNasa tampón fosfato 0.12M.
3. Hydroxypaptite Cromatografía
- Asegurarse de que la llave está cerrada, poco a poco añadir 2 ml de preparado de antemano, la hidroxiapatita se resuspendieron en la parte inferior de la columna Econo-ayuda de una pipeta serológica de 2 ml.
- Deje que la hidroxiapatita de resolver por la fuerza de gravedad durante 30 minutos.
- Drenaje de amortiguación de la columna y la llave de cierre. Combine los ácidos nucleicos con tampón fosfato 0.12M en un volumen final de 500 l, y se incuba a 60 ° C durante 10 minutos en un bloque de calor o baño de agua.
- Aplicar rápidamente los ácidos nucleicos preparados en tampón fosfato 0.12M a la hidroxiapatita con una pipeta serológica de 2 ml, asegurándose de no molestar a la columna.
- Permita que los ácidos nucleicos que se unen a la hidroxiapatita durante 30 minutos.
- Colocar un tubo de 15 ml en la columna, abra la llave de paso y recoger la muestra inicial que contiene ADN de cadena simple. Cerrar llave de paso.
- Agregar 6 ml de tampón fosfato 0.12M a la hidroxiapatita, asegurándose de no molestar a la columna, abra la llave de paso, y cobrar de una sola cadena de ADN en el tubo de 15 ml de la etapa anterior. Cerrar llave de paso.
- Añadir un volumen de un fenol: cloroformo: alcohol isoamílico (25:25:1 v / v / v) en el tubo, mezclar vigorosamente y se centrifuga a 3500 xg durante 15 minutos. La transferencia de ADN de cadena simple que contiene sobrenadante a un tubo de 15 ml nuevos.
- Repita 3,76 y 3,87 con 6 ml de solución amortiguadora de fosfato 0,20 M de elaúd y purificar el ARN de la columna y asegúrese de usar un tubo de 15 ml nueva colección.
- Repita 3,76 y 3,87 con 6 ml de solución amortiguadora de fosfato 0,40 m para eluir y purificar DNA de doble cadena de la columna y asegúrese de usar un tubo de 15 ml nueva colección.
- Repita 3,76 y 3,87 con 6 ml de tampón fosfato 1.00M para despojar a la columna de cualquier dsDNA residual asegurándose de usar un tubo de 15 ml nueva colección.
4. Desalinización de muestras de ácidos nucleicos
- Transferencia de 4 de 5 ml de muestra a un Amicon Ultra-4 dispositivo centrífugo de filtro equipado con un peso molecular 30.000 cortar la membrana Ultracel.
- Concentrar la muestra a <500μl al girar a 6.000 xg, 30 ° C, a 5 minutos (o hasta que el volumen de concentrado se logra) en un rotor de ángulo fijo. Deseche el flujo a través.
- Agregue el resto de la muestra (s) a la Amicon Ultra-4 dispositivo de filtro centrífugo y llevar el volumen hasta 4 5 ml con RNasa libre Buffer 1X TE.
- Concentrar la muestra a <500μl al girar a 6.000 xg, 30 ° C, a 5 minutos (o hasta que el volumen de concentrado se logra) en un rotor de ángulo fijo. Deseche el flujo a través.
- Agregar 4 5 ml de RNasa libre Buffer 1X TE a la Amicon Ultra-4 dispositivo de filtro centrífugo. Concentrar la muestra a <500μl por centrifugación a 6.000 xg, 30 ° C, a 5 minutos (o hasta que el volumen de concentrado se logra) en un rotor de ángulo fijo. Deseche el flujo a través.
- Repita el paso 4.5 un adicional de 5 veces para desalar por completo muestra de ácido nucleico (s).
- La transferencia de la muestra restante (s) a un tubo estéril eppendorf 1.7ml. Añadir un volumen de acetato de sodio 3M 1/10th, pH 7,0, dos volúmenes de etanol al 100%, y 1μl de Glycoblue. Mezclar bien por agitación.
- Centrifugar a 28.000 xg, 4 º C, 60 minutos. Decantar con cuidado de no perturbar el sedimento. Agregar 300μl de etanol al 70%. Giran a 28.000 xg temperatura ambiente, durante 10 minutos. Se decanta, seco y resuspender cada fracción en un volumen adecuado de RNasa libre de buffer TE.
5. Los resultados representativos:
La Figura 1 resume los métodos presentados para el uso de cromatografía de hidroxiapatita para fraccionar ssDNA, dsDNA y los ácidos nucleicos ARN viral a partir de un conjunto mixto. Este método explota la interacción de cargas entre el esqueleto de fosfato con carga negativa de los ácidos nucleicos y la carga positiva iones Ca 2 + presentes en la hidroxiapatita y permite el fraccionamiento eficiente de los subtipos de ácido nucleico (ssDNA, dsDNA y ARN) con el aumento de las concentraciones de tampón fosfato 12.
El fraccionamiento de una hidroxiapatita in vitro "comunidad" de los más conocidos ssDNA (M13mp18), ADN de doble cadena (lambda) y ARN (MS2 y phi6) genomas virales se ilustra en la Figura 2. Los ácidos nucleicos se combinaron en concentraciones iguales, aplicada a la columna de hidroxiapatita y se eluyeron con un aumento de la concentración de tampón fosfato. Cada subtipo de ácido nucleico eluye independiente de los demás con menos del 9% acumulado de excedentes de una fracción a otra.
La aplicación de esta técnica a los ácidos nucleicos aislados de una comunidad viral recogidos en la bahía de Chesapeake se muestra en la Figura 3. El rendimiento total de ácidos nucleicos aislados de virus purificado se aplicó a la columna de hidroxiapatita. Material genómico que consta de ssDNA, el ARN y el ADN de doble cadena se eluyó de forma independiente con 0.12M, y las concentraciones de 0,20 M de tampón fosfato 0.40M/1.00M respectivamente. Doble hebra de ADN se eluye en las concentraciones de fosfato de 0,40 m y 1.00M y en este caso, domina esta comunidad viral en comparación con los genotipos ssDNA y el ARN. Esta observación es consistente con la composición de la comunidad espera viral de ambientes marinos y estuarinos donde la mayoría de los ácidos nucleicos son recuperables dsDNA 13.

Figura 1. Diagrama de flujo del método de cromatografía de hidroxiapatita para el fraccionamiento de los ácidos nucleicos. Una mezcla de ssDNA, dsDNA y ARN preparado en tampón fosfato 0.12M se calienta a 60 ° C y se aplica a una columna de hidroxiapatita mantenido a una temperatura constante de 60 ° C con un baño de agua circulante. Los ácidos nucleicos (ADN de cadena simple, ARN y ADN de doble cadena) se eluyen de la hidroxiapatita con concentraciones crecientes de un tampón fosfato que contienen. Esta cifra ha sido reproducido con el permiso de la Sociedad Americana de Microbiología y fue presentado originalmente en Andrews-Pfannkoch et al. 2010 12.

Figura 2. Separación de los más conocidos ácidos nucleicos virales usando la cromatografía de hidroxiapatita. Concentraciones iguales de M13mp18 ssDNA, ssRNA MS2, dsRNA phi6 y lambda ADN de doble cadena (carriles 2-5, respectivamente) se combinaron (calle 6) y se aplicó a una columna de hidroxiapatita. Single-ADN de cadena (M13mp18), el ARN (MS2/phi6) y ADN de doble cadena (lambda) se eluyeron con independencia de la columna de hidroxiapatita con 0.12M (carril 8), 0.18M (línea 9) y 0.40M/1.00M (calle 10) . Una escalera de 1kb (líneas 1, 7) se utilizó para confirmar tamaño del genoma. Esta cifra ha sido reproducido con el permiso de la Sociedad Americana de Microbiología y fue presentado originalmente en Andrews-Pfannkoch et al. 2010 12.
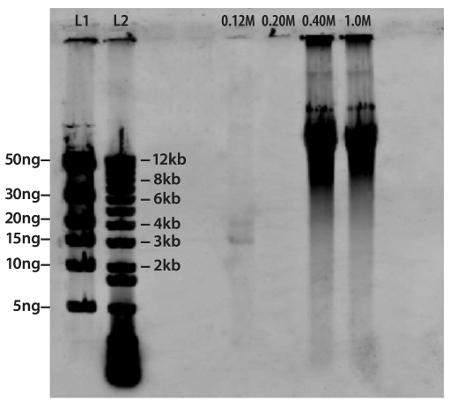
Figura 3. Separación de ácidos nucleicos virales aisladas de una comunidad dentro de la bahía de Chesapeake. Los ácidos nucleicos fueron aislados y se aplicó a una columna de hidroxiapatita. ssDNA (carril 0.12M), el ARN (carril de 0,20 M) y ADN de doble cadena (carriles 0.40M/1.0M) se eluye de forma independiente mediante la concentración de tampón fosfato de 0.12M, 0,20 M y 0.40M/1.00M, respectivamente. Misa de alta escalera del ADN (carril L1) y Ladder 1kb (carril L2) fueron utilizados para confirmar visualmente peso molecular aproximado y la masa de los ácidos nucleicos. Esta cifra ha sido reproducido con el permiso de la Sociedad Americana de Microbiología y fue presentado originalmente en Andrews-Pfannkoch et al. 2010 12.
Discusión
La metodología de la cromatografía de hidroxiapatita que aquí se presenta es una herramienta muy eficiente y robusto para el fraccionamiento de los ácidos nucleicos de los conjuntos mixtos viral, cuando el objetivo es el estudio de la composición total de ácidos nucleicos de la comunidad. Por lo general, ssDNA, el ARN y ADN de doble cadena se eluyen de la columna de las concentraciones de pho tampón fosfato mayor que 0 y 0,40 m aproximadamente ~ respectivamente. Sin embargo, cada preparación de hidroxia...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Esta investigación fue apoyada por la Oficina de Ciencia (BER), EE.UU. Departamento de Energía, del Acuerdo de Cooperación no. De-FC02-02ER63453 Microbiana de la Fundación Nacional de Ciencias del Programa de la secuenciación del genoma (los números 0626826 y 0731916 premio). Agradecemos a John Glass por su experiencia y asesoramiento técnico y K. Eric Wommack por su ayuda con la recogida de muestras ambientales.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del Material | Empresa | Número de catálogo | Número de catálogo |
|---|---|---|---|
| Econo-Columna | Bio-Rad | 737-0717 | 0.7cm paquete de ID / 2 |
| Hidroxiapatita | Bio-Rad | 130-0520 | Grado de ADN de Bio-Gel Gel HTP, 100g/td> |
| El fosfato de sodio, monobásico | VWR | VW1497-01 | Monohidrato, cristal 500g |
| Fosfato sódico, dibásico | VWR | VW1496-01 | Anhidro, en polvo de 500 g |
| El agua tratada con DEPC | Invitrogen | AM9922 | 1L |
| 10% SDS solución | Invitrogen | 24730-020 | UltraPure, 1L |
| 0,5 M EDTA | Invitrogen | AM9262 | pH 8,0, 1 litro |
| Sigmacote | Sigma-Aldrich | SL2-25ML | |
| 2 ml serológicas pippette | VWR | 89130-884 | Poliestireno, estéril |
| BD Falcon Tubos de centrífuga | VWR | 21008-936 | 15 ml, estéril |
| Fenol: cloroformo: alcohol isoamílico (25:24:1 v / v / v) | Invitrogen | 15593-031 | UltraPure, 100ml |
| Amicon Ultra-4 dispositivos centrífugos | Millipore | UFC803024 | Ultracel-30 de membrana |
| 20X buffer TE, RNasa libre | Invitrogen | T11493 | 100ml |
| Glycoblue | Invitrogen | AM9516 | 15mg/ml |
Referencias
- Whitman, W. B., Coleman, D. C., Wiebe, W. J. Prokaryotes: The Unseen Majority. Proceedings of the National Academy of Sciences of the United States of America. 95, 6578-65 (1998).
- Hendrix, R. W. Bacteriophages: evolution of the majority. Theor Popul Biol. 61, 471-471 (2002).
- Weinbauer, M. G. Bacteriophages: Evolution of the Majority. FEMS Microbiol Rev. 28, 127-127 (2004).
- Dinsdale, E. A., Edwards, R. A., Hall, D. Functional Metabolic Profiling of Nine Biomes. Nature. 455, 830-830 (2008).
- McDaniel, L., Breitbart, M., Mobberley, J. Metagenomic Analysis of Lysogeny in Tampa Bay: Implications for Prophage Gene Expression. PLoS ONE. 3, e3263-e3263 (2008).
- Williamson, S. J., Rusch, D. B., Yooseph, S. The Sorcerer II Global Ocean Sampling Expedition: Metagenomic Characterization of Viruses within Aquatic Microbial Samples. PLoS ONE. 3, e1456-e1456 (2008).
- Lang, A. S., Rise, M. L., Culley, A. I. RNA Viruses in the Sea. FEMS Microbiol Rev. 33, 295-295 (2009).
- Ng, T. F., Manire, C., Borrowman, K. Discovery of a Novel Single-Stranded DNA Virus from a Sea Turtle Fibropapilloma by using Viral Metagenomics. J. Virol. 83, 2500-2500 (2009).
- Rosario, K., Duffy, S., Breitbart, M. Diverse Circovirus-like Genome Architectures Revealed by Environmental Metagenomics. J Gen Virol. 90, 2418-2418 (2009).
- Culley, A. I., Lang, A. S., Suttle, C. A. Metagenomic Analysis of Coastal RNA Virus Communities. Science. 312, 1795-1795 (2006).
- Bernardi, G. Chromotography of Nucleic Acids on Hydroxyapatite. Nature. 209, 779-779 (1965).
- Andrews-Pfannkoch, C., Fadrosh, D. W., Thorpe, J. Hydroxyapatite-Mediated Separation of Double-Stranded DNA, Single-Stranded DNA and RNA Genomes from Natural Viral Assemblages. Applied and environmental microbiology. 76, 5039-5039 (2010).
- Wommack, K. E., Colwell, R. R. Virioplankton: Viruses in Aquatic Ecosystems. Microbiol Mol Biol Rev. 64, 69-69 (2000).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados