Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Trennung von Einzelstrang-DNA, doppelsträngige DNA und RNA aus ökologischer Viral Gemeinschaft mit Hydroxyapatit-Chromatographie
In diesem Artikel
Zusammenfassung
Wir beschreiben eine effiziente Methode, um Einzelstrang-DNA, doppelsträngige DNA-und RNA-Molekülen aus Umwelt-virale Gemeinden zu trennen. Nukleinsäuren sind fraktionierte mit Hydroxyapatit-Chromatographie mit steigenden Konzentrationen von Phosphat-haltigen Puffer. Diese Methode erlaubt die Isolierung aller viralen Nukleinsäure-Typen aus Umweltproben.
Zusammenfassung
Viren, vor allem Bakteriophagen (Phagen), sind die zahlreichen biologischen Einheiten auf der Erde 1,2. Viren modulieren Wirtszelle Fülle und Vielfalt, um den Kreislauf der Nährstoffe beitragen, zu verändern Wirtszelle Phänotyp, und Einfluss auf die Entwicklung der beiden Wirtszelle und Virus-Communities durch die seitlichen Transfer von Genen 3. Zahlreiche Studien haben die erstaunliche genetische Vielfalt der Viren und ihre funktionelle Potenzial in einer Vielzahl von natürlichen Umgebungen hervorgehoben.
Metagenom Techniken wurden verwendet, um die taxonomische Diversität und funktionale Potenzial von komplexen viralen Assemblagen, deren Mitglieder enthalten Einzelstrang-DNA (ssDNA), doppelsträngige DNA (dsDNA) und RNA-Genotypen 4-9 studieren. Aktuelle Bibliothek Bau-Protokolle verwendet werden, um Umwelt-DNA-haltigen oder RNA-Viren enthalten Studie erfordern eine erste Nuklease Behandlung zur Entfernung nontargeted Templates 10. Allerdings erfordert ein umfassendes Verständnis des kollektiven Gen Ergänzung des Virus-Community und Viren Vielfalt Wissen aller Mitglieder, unabhängig von Genom Zusammensetzung. Die Fraktionierung des gereinigten Nukleinsäure-Subtypen bietet einen wirksamen Mechanismus, durch die virale Assemblagen, ohne dabei eine Teilmenge der Gemeinde genetische Signatur zu studieren.
Hydroxyapatit, eine kristalline Form von Calciumphosphat, hat in der Trennung von Nukleinsäuren, sowie Proteine und Mikroben eingesetzt worden, seit den 1960er Jahren 11. Durch die Ausnutzung der Ladungs-Wechselwirkung zwischen den positiv geladenen Ca 2 +-Ionen des Hydroxylapatit und der negativ geladenen Phosphat-Rückgrat der Nukleinsäure-Subtypen, ist es möglich, bevorzugt eluieren jede Nukleinsäure-Subtyp unabhängig von den anderen. Vor kurzem haben wir diese Strategie, unabhängig fraktionieren die Genome von ssDNA, dsDNA und RNA-Viren enthalten in der Vorbereitung der DNA-Sequenzierung 12 verwendet. Hier stellen wir ein Verfahren zur Fraktionierung und Rückgewinnung von ssDNA, dsDNA und RNA viralen Nukleinsäuren aus gemischten virale Assemblagen mit Hydroxyapatit-Chromatographie.
Protokoll
1. Herstellung von Lösungen
Vor der Durchführung Hydroxyapatit-Chromatographie, muss Phosphatpuffer hergestellt werden und die Hydroxyapatit muss richtig hydratisiert.
- 1M Phosphat-Lösung, pH 6,8: In einem 1L Flasche auflösen 119.98g Natriumphosphat einbasigen in 1L steril, DEPC-behandeltem H 2 O. Bereiten Sie eine 1M Natriumdihydrogenphosphat-Lösung in einem 1L Flasche durch Auflösen von 141.96g von Natriumdihydrogenphosphat in 1L steril, DEPC-behandeltem H 2 O. Kombinieren Sie die Mono-und Di-Lösungen mit einem Verhältnis von 1:1. Küvette auf einer Rührplatte und mischen mit einem magnetischen Rührstab. Der pH-Wert auf 6,8 durch Zugabe von Natronlauge (zur pH-Erhöhung) oder Phosphorsäure (zur pH-Abnahme).
- 0,12 M Phosphatpuffer: In einem 500 ml-Kolben, kombinieren 30ml 1M Phosphat-Lösung, pH 6,8, 2,5 ml 10% Natriumdodecylsulfat (SDS) und 5 ml 0,5 M EDTA. Add sterile, DEPC-behandeltem H2O auf ein Volumen von 225ml. Der pH-Wert auf 6,8. Add sterile, DEPC-behandeltem H2O auf 250ml. Autoclave.
- 0,18 M Phosphatpuffer: In einem 500 ml-Kolben, kombinieren 45ml 1M Phosphat-Lösung, pH 6,8, 2,5 ml 10% Natriumdodecylsulfat (SDS) und 5 ml 0,5 M EDTA. Add sterile, DEPC-behandeltem H 2 O auf ein Volumen von 225ml. Der pH-Wert auf 6,8. Add sterile, DEPC-behandeltem H 2 O auf 250ml. Autoclave.
- 0,20 M Phosphatpuffer: In einem 500 ml-Kolben, kombinieren 50ml 1M Phosphat-Lösung, pH 6,8, 2,5 ml 10% Natriumdodecylsulfat (SDS) und 5 ml 0,5 M EDTA. Add sterile, DEPC-behandeltem H 2 O auf ein Volumen von 225ml. Der pH-Wert auf 6,8. Add sterile, DEPC-behandeltem H 2 O auf 250ml. Autoclave.
- 0.40 M Phosphatpuffer: in einer 500ml Flasche, kombinieren 100ml 1M Phosphat-Lösung, pH 6,8, 2,5 ml 10% Natriumdodecylsulfat (SDS) und 5 ml 0,5 M EDTA. Add sterile, DEPC-behandeltem H 2 O auf ein Volumen von 225ml. Der pH-Wert auf 6,8. Add sterile, DEPC-behandeltem H 2 O auf 250ml. Autoclave.
- "1,00 M Phosphat-Puffer" (tatsächliche Konzentration von Phosphat in dieser Lösung ist 0,91 M): In einem 500 ml-Kolben, kombinieren 30ml 1M Phosphat-Lösung, pH 6,8, 2,5 ml 10% Natriumdodecylsulfat (SDS) und 5 ml 0,5 M EDTA. Add sterile, DEPC-behandeltem H2O auf ein Volumen von 225ml. Der pH-Wert auf 6,8. Add sterile, DEPC-behandeltem H 2 O auf 250ml. Autoclave.
- Hydroxyapatit Hydratation: Gewicht aus 1g aus Hydroxylapatit und hinzufügen, um eine 50ml Tube. Add 6ml von 0,12 M Phosphat-Puffer auf die Hydroxyapatit und umkehren zu mischen. Vor dem Einsatz bringen Temperatur auf 60 ° C und gründlich mischen. Shop für längere Zeit bei 4 ° C.
- Vor dem Gebrauch Gleichgewicht aller Phosphat-Puffer und hydratisiert Hydroxyapatit bei 60 ° C.
2. Vorbereitung von Econo-Column
- Absperrhahn schließen. Spülen Spalte mehrmals mit deionisiertem H 2 O Durch wiederholtes Umdrehen der Säule und Dekantieren.
- Entfernen Sie den Hahn aus der Econo-Säule und der Säule Autoklaven zu sterilisieren.
- Ersetzen und schließen Hahn. 1ml von Sigmacote die sterile Econo-Säule und Mantel aller Glasflächen. Öffnen Sie den Wasserhahn und dekantieren.
- Befestigen Sie die Spalte, um einen zirkulierenden Wasserbad und eingestellte Temperatur auf 60 ° C. Um einsure den geringsten Wärmeverlust über die Säule das Einwickeln der Spalte in einer isolierenden Mittel wie Aluminiumfolie oder Schaum Rohrisolierung wird empfohlen.
- Spülen Sie die Econo-Säule zweimal mit sterilem, RNase-freien H 2 O dann zweimal mit sterilem, RNase-frei 0,12 M Phosphatpuffer.
3. Hydroxypaptite Chromatographie
- Sicherstellen, dass der Hahn geschlossen ist, langsam zugeben 2ml vorbereiteten, resuspendiert Hydroxyapatit an der Unterseite des Econo-Säule mit einer sterilen 2 ml serologischen Pipette.
- Lassen Sie die Hydroxylapatit unter der Schwerkraft für 30 Minuten absetzen.
- Abfluss-Puffer aus der Säule und in der Nähe Hahn. Kombinieren Nukleinsäuren mit 0,12 M Phosphat-Puffer in einem Endvolumen von 500 ul und Inkubieren bei 60 ° C für 10 Minuten in einem Heizblock oder Wasserbad.
- Schnell gelten Nukleinsäuren in 0,12 M Phosphat-Puffer auf die Hydroxyapatit mit einem 2ml serologischen Pipette darf dabei nicht um die Spalte zu stören vorbereitet.
- Lassen Sie die Nukleinsäuren an das Hydroxyapatit für 30 Minuten zu binden.
- Legen Sie eine 15ml Tube unter der Spalte, öffnen Sie den Hahn und sammelt die ersten Probe mit ssDNA. Absperrhahn schließen.
- Add 6ml von 0,12 M Phosphat-Puffer auf die Hydroxyapatit darf dabei nicht um die Spalte zu stören, öffnen Sie den Hahn, und sammeln Einzelstrang-DNA in der 15ml Tube aus dem vorherigen Schritt. Absperrhahn schließen.
- Add 1 Volumen Phenol: Chloroform: Isoamylalkohol (25:25:1 v / v / v) Lösung für das Rohr, kräftig mischen und zentrifugieren bei 3500 xg für 15 Minuten. Überstand mit ssDNA zu einem neuen 15ml Tube.
- Wiederholen 3,76 und 3,87 mit 6ml von 0,20 M Phosphat-Puffer, um eLaute und reinigen RNA aus der Säule achten Sie darauf, ein neues 15ml Tube für die Sammlung verwenden.
- Wiederholen 3,76 und 3,87 mit 6ml von 0,40 M Phosphat-Puffer zu eluieren und zu reinigen dsDNA aus der Spalte achten Sie darauf, ein neues 15ml Tube für die Sammlung verwenden.
- Wiederholen 3,76 und 3,87 mit 6ml von 1,00 M Phosphat-Puffer auf die Säule der verbleibenden dsDNA achten Sie darauf, ein neues 15ml Tube für die Sammlung verwenden Streifen.
4. Entsalzung Nukleinsäure-Proben
- Transfer-4-5 ml Probe an einen Amicon Ultra-4 Zentrifugalfilter Gerät mit einer 30.000 Molekulargewicht off Ultracel Membran geschnitten ausgestattet.
- Konzentrieren Probe <500μl durch Spinnen bei 6.000 xg, 30 ° C, 5 Minuten (oder bis konzentrierten Volumen erreicht ist) in einem Festwinkelrotor. Entsorgen Sie durchströmen.
- Fügen Sie den Rest der Probe (n) der Amicon Ultra-4 Zentrifugalfilter Gerät und bringen die Lautstärke von bis zu 4-5ml mit RNase frei 1X TE-Puffer.
- Konzentrieren Probe <500μl durch Spinnen bei 6.000 xg, 30 ° C, 5 Minuten (oder bis konzentrierten Volumen erreicht ist) in einem Festwinkelrotor. Entsorgen Sie durchströmen.
- Add 4-5 ml RNase frei 1X TE-Puffer auf die Amicon Ultra-4 Zentrifugalfilter Gerät. Konzentrieren Probe <500μl durch Zentrifugation bei 6.000 xg, 30 ° C, 5 Minuten (oder bis konzentrierten Volumen erreicht ist) in einem Festwinkelrotor. Entsorgen Sie durchströmen.
- Wiederholen Sie Schritt 4.5 eine zusätzliche 5-mal vollständig entsalzt Nukleinsäureprobe (s).
- Restlichen Probe (n) in ein steriles 1,7 ml Eppendorf-Röhrchen. Add 1:10 Volumen 3M Natriumacetat, pH 7,0, zwei Bände mit 100% Ethanol und 1μl der Glycoblue. Gut mischen durch Vortexen.
- Zentrifuge bei 28.000 xg, 4 ° C, 60 Minuten. Dekantieren darf dabei nicht das Pellet stören. Fügen Sie 300μl 70% Ethanol. Spin bei 28.000 xg, Raumtemperatur für 10 Minuten. Dekantieren, trocken und resuspendieren jeder Fraktion in einem geeigneten Volumen RNase-freiem TE-Puffer.
5. Repräsentative Ergebnisse:
Abbildung 1 fasst die Methoden für den Einsatz Hydroxyapatit-Chromatographie zur Fraktionierung ssDNA, dsDNA und RNA Nukleinsäuren aus einer gemischten virale Assemblage vorgestellt. Diese Methode nutzt die Ladungs-Wechselwirkung zwischen den negativ geladenen Phosphat-Rückgrat der Nukleinsäuren und die positiv geladenen Ca 2 +-Ionen in der Hydroxylapatit und ermöglicht die effiziente Fraktionierung von Nukleinsäure-Subtypen (ssDNA, dsDNA und RNA) mit steigender Phosphat-Puffer-Konzentrationen 12.
Die Hydroxyapatit Fraktionierung von einem in vitro-"Community" bekannt ssDNA (M13mp18), dsDNA (Lambda) und RNA (MS2 und phi6) viralen Genomen ist in Abbildung 2 dargestellt. Die Nukleinsäuren wurden in gleichen Konzentrationen kombiniert, angewandt auf die Hydroxyapatit-Säule und eluiert mit einer zunehmenden Konzentration von Phosphat-Puffer. Jeder Nukleinsäure-Subtyp eluiert unabhängig von den anderen mit weniger als 9% von einem Bruchteil-over zum nächsten zu befördern.
Die Anwendung dieser Technik, um Nukleinsäuren aus einer viralen Gemeinde aus der Chesapeake Bay gesammelt isoliert ist in Abbildung 3 dargestellt. Die gesamte Nukleinsäure-Ausbeute von gereinigten Viren isoliert, um die Hydroxyapatit-Säule aufgetragen wurde. Genomic Material, bestehend aus ssDNA, RNA und dsDNA wurde unabhängig mit 0,12 M, 0,20 M und 0.40M/1.00M Konzentrationen von Phosphat-Puffer bzw. eluiert. Doppelsträngige DNA ist bei Phosphat-Konzentrationen von 0,40 M und 1,00 M eluiert und in diesem Fall dominiert diese virale Community im Vergleich zu ssDNA und RNA-Genotypen. Diese Beobachtung steht im Einklang mit der erwarteten virale Community Zusammensetzung der Meeres-und Mündungs-Umgebungen, in denen die Mehrheit der erzielbare Nukleinsäuren dsDNA 13.

Abbildung 1. Flussdiagramm des Hydroxyapatit-Chromatographie-Verfahren zur Fraktionierung von Nukleinsäuren. Eine Mischung aus ssDNA, dsDNA und RNA in 0,12 M Phosphat-Puffer bereit ist, 60 ° C erhitzt und auf eine Hydroxyapatit-Säule bei konstant 60 ° C gehalten mit einem zirkulierenden Wasserbad. Nukleinsäuren (ssDNA, RNA und dsDNA) sind vom Hydroxyapatit mit steigenden Konzentrationen von Phosphat-Puffer eluiert. Diese Zahl ist mit freundlicher Genehmigung von American Society of Microbiology reproduziert worden und war ursprünglich in Andrews-Pfannkoch et al featured. 2010 12.

Abbildung 2. Trennung von bekannten viralen Nukleinsäuren mittels Hydroxyapatit-Chromatographie. Equal Konzentrationen von M13mp18 ssDNA, MS2 ssRNA, phi6 dsRNA und Lambda-dsDNA (Spuren 2-5, jeweils) wurden vereinigt (Spur 6) und auf eine Hydroxyapatit-Säule. Single-DNA (M13mp18), RNA (MS2/phi6) und dsDNA (Lambda) waren unabhängig von der Hydroxyapatit-Säule mit 0,12 M (Spur 8), 0,18 M (Spur 9) und 0.40M/1.00M (Spur 10) eluiert . Ein 1kb ladder (Bahnen 1, 7) wurde verwendet, um Genomgrößen bestätigen. Diese Zahl ist mit freundlicher Genehmigung von American Society of Microbiology reproduziert worden und war ursprünglich in Andrews-Pfannkoch et al featured. 2010 12.
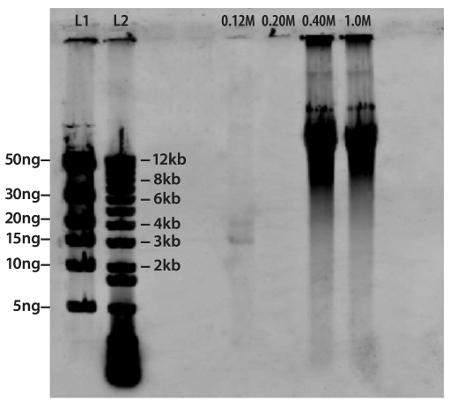
Abbildung 3. Separation von viralen Nukleinsäuren aus einer Gemeinde innerhalb der Chesapeake Bay getrennt. Nukleinsäuren wurden isoliert und auf eine Hydroxyapatit-Säule. ssDNA (Spur 0,12 M), RNA (Spur 0,20 M) und dsDNA (Spuren 0.40M/1.0M) wurden unabhängig voneinander eluiert mit Phosphatpuffer Konzentrationen von 0,12 M, 0,20 M und 0.40M/1.00M jeweils. Hallo Messe DNA Ladder (Spur L1) und 1kb Ladder (Spur L2) wurden verwendet, um visuell bestätigen ungefähre Molekulargewicht und die Masse der Nukleinsäuren. Diese Zahl ist mit freundlicher Genehmigung von American Society of Microbiology reproduziert worden und war ursprünglich in Andrews-Pfannkoch et al featured. 2010 12.
Diskussion
Die Hydroxyapatit-Chromatographie-Methodik präsentiert hier ist eine hocheffiziente und robustes Werkzeug für die Fraktionierung von Nukleinsäuren aus gemischten virale Assemblagen, wenn das Ziel, die gesamte Nukleinsäure Zusammensetzung der Gemeinde-Studie ist. Im Allgemeinen wird ssDNA, RNA und dsDNA aus der Spalte pho-Phosphat-Puffer-Konzentrationen von mehr als etwa ~ 0und 0,40 M bzw. eluieren. Allerdings kann jeder Zubereitung von Hydroxyapatit haben eine etwas andere Zusammensetzung und Elutionsprofil...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde vom Office of Science (BER), US Department of Energy, Cooperative Agreement nicht unterstützt. De-FC02-02ER63453, die National Science Foundation Mikrobielle Genome Sequencing Program (Auszeichnung Zahlen 0626826 und 0731916). Wir danken John Glass für seine technische Kompetenz und Beratung und K. Eric Wommack für seine Unterstützung bei der Umwelt-Probenentnahme.
Materialien
| Name | Company | Catalog Number | Comments |
| Material Name | Firma | Katalog-Nummer | Katalog-Nummer |
|---|---|---|---|
| Econo-Column | Bio-Rad | 737-0717 | 0.7cm ID-Paket / 2 |
| Hydroxyapatit | Bio-Rad | 130-0520 | DNA Grade Bio-Gel HTP Gel, 100g/td> |
| Sodium Phosphate, Einbasisches | VWR | VW1497-01 | Monohydrate, Crystal 500g |
| Sodium Phosphate, Dibasic | VWR | VW1496-01 | Wasserfrei, Pulver 500g |
| DEPC-behandeltem Wasser | Invitrogen | AM9922 | 1L |
| 10% SDS-Lösung | Invitrogen | 24730-020 | UltraPure, 1L |
| 0,5 M EDTA | Invitrogen | AM9262 | pH 8,0, 1L |
| Sigmacote | Sigma-Aldrich | SL2-25ML | |
| 2ml serologische pippette | VWR | 89130-884 | Polystyrol, steril |
| BD Falcon Zentrifugenröhrchen | VWR | 21008-936 | 15ml, steril |
| Phenol: Chloroform: Isoamylalkohol (25:24:1 v / v / v) | Invitrogen | 15593-031 | UltraPure, 100ml |
| Amicon Ultra-4 Kreiselpumpen Geräte | Millipore | UFC803024 | Ultracel-30-Membran |
| 20X TE-Puffer, RNase-frei | Invitrogen | T11493 | 100ml |
| Glycoblue | Invitrogen | AM9516 | 15mg/ml |
Referenzen
- Whitman, W. B., Coleman, D. C., Wiebe, W. J. Prokaryotes: The Unseen Majority. Proceedings of the National Academy of Sciences of the United States of America. 95, 6578-65 (1998).
- Hendrix, R. W. Bacteriophages: evolution of the majority. Theor Popul Biol. 61, 471-471 (2002).
- Weinbauer, M. G. Bacteriophages: Evolution of the Majority. FEMS Microbiol Rev. 28, 127-127 (2004).
- Dinsdale, E. A., Edwards, R. A., Hall, D. Functional Metabolic Profiling of Nine Biomes. Nature. 455, 830-830 (2008).
- McDaniel, L., Breitbart, M., Mobberley, J. Metagenomic Analysis of Lysogeny in Tampa Bay: Implications for Prophage Gene Expression. PLoS ONE. 3, e3263-e3263 (2008).
- Williamson, S. J., Rusch, D. B., Yooseph, S. The Sorcerer II Global Ocean Sampling Expedition: Metagenomic Characterization of Viruses within Aquatic Microbial Samples. PLoS ONE. 3, e1456-e1456 (2008).
- Lang, A. S., Rise, M. L., Culley, A. I. RNA Viruses in the Sea. FEMS Microbiol Rev. 33, 295-295 (2009).
- Ng, T. F., Manire, C., Borrowman, K. Discovery of a Novel Single-Stranded DNA Virus from a Sea Turtle Fibropapilloma by using Viral Metagenomics. J. Virol. 83, 2500-2500 (2009).
- Rosario, K., Duffy, S., Breitbart, M. Diverse Circovirus-like Genome Architectures Revealed by Environmental Metagenomics. J Gen Virol. 90, 2418-2418 (2009).
- Culley, A. I., Lang, A. S., Suttle, C. A. Metagenomic Analysis of Coastal RNA Virus Communities. Science. 312, 1795-1795 (2006).
- Bernardi, G. Chromotography of Nucleic Acids on Hydroxyapatite. Nature. 209, 779-779 (1965).
- Andrews-Pfannkoch, C., Fadrosh, D. W., Thorpe, J. Hydroxyapatite-Mediated Separation of Double-Stranded DNA, Single-Stranded DNA and RNA Genomes from Natural Viral Assemblages. Applied and environmental microbiology. 76, 5039-5039 (2010).
- Wommack, K. E., Colwell, R. R. Virioplankton: Viruses in Aquatic Ecosystems. Microbiol Mol Biol Rev. 64, 69-69 (2000).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten