Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Selección y aislamiento de las colonias de humanos células madre pluripotentes inducidas a partir de fibroblastos adultos Reprogramado
En este artículo
Resumen
Se presenta un protocolo para la reprogramación eficiente de las células somáticas humanas en células madre pluripotentes inducidas (hiPSC) utilizando vectores retrovirales que codifican Oct3 / 4, Sox2, Klf4 y c-myc (OSKM) y la identificación de hiPSC correctamente reprogramado por tinción en vivo con Tra- 1-81 anticuerpo.
Resumen
El presente artículo presenta un protocolo de reprogramar los fibroblastos humanos adultos en las células madre pluripotentes inducidas (hiPSC) utilizando vectores retrovirales que codifican Oct3 / 4, Sox2, Klf4 y c-myc (OSKM) en presencia de sodio 1.3 butirato. Hemos utilizado este método para reprogramar el paso tardío (> p10) fibroblastos humanos adultas derivadas de los pacientes la ataxia de Friedreich (GM03665, Coriell repositorio). El enfoque de reprogramación incluye protocolo de transducción de alta eficiencia utilizando centrifugación repetitiva de los fibroblastos en la presencia de virus que contienen medios de comunicación. Las colonias hiPSC reprogramadas se identificaron utilizando inmunotinción vivo para Tra-1-81, un marcador de superficie de las células pluripotentes, separado de los fibroblastos no reprogramadas y manualmente passaged 4,5. Estos hiPSC fueron trasladados a las placas de Matrigel y crecido en condiciones libres de alimentador, directamente desde la placa de la reprogramación. A partir de el primer paso, las colonias hiPSC demuestran características HES-lmorfología de Ike. Utilizando este protocolo más del 70% de las colonias seleccionadas pueden ser ampliado con éxito y se estableció en líneas celulares. Las líneas establecidas hiPSC muestra características marcadores de pluripotencia, incluyendo los marcadores de superficie TRA-1-60-4 y SSEA, así como los marcadores nucleares Oct3 / 4, Sox2 y Nanog. El protocolo que aquí se presenta se ha establecido y probado usando fibroblastos adultos obtenidos de pacientes con AF e individuos de control 6, fibroblastos humanos recién nacidos, así como los queratinocitos humanos.
Protocolo
1. Virus de la producción y la transducción
- Placas Phoenix anfo células a una densidad de ~ 7-8x10 6 por placa de 10 cm en 10 ml de medio DMEM (glucosa DMEM alto, 10% de FBS inactivado calor, 2 mM de L-glutamina, sin antibióticos). Lugar en la incubadora y la cultura durante toda la noche a 37 ° C, 5% de CO 2.
- Al día siguiente, transfección de células de Phoenix con 12 mg de un vector de codificación sea Oct3 / 4, Sox2, Klf4, c-myc, o gen GFP (Addgene plásmidos 17217, 17218, 17219, 17220) y 35 l Fugene 6. Prepare la mezcla de transfección en 500 l de medio DMEM (glucosa DMEM alto, 2 mM L-glutamina, sin SFB y antibióticos). Incubar 20 minutos. Pipetear con cuidado los complejos de ADN en las placas de 10 cm que contienen 70-80% confluentes células de Phoenix.
- Vuelva a colocar los medios de comunicación 6 a 8 horas después de la transfección con medio DMEM (glucosa alta DMEM, 10% al calor de SFB inactivado, 2 mM L-glutamina), que contiene la penicilina y la estreptomicina.
- Posteriormente, recoger el virus que contiene los medios 4 veces en 12 hintervalos, se combinan todas las partes y se filtra utilizando un filtro de 0,45 micras para eliminar las células desprendidas y escombros (los medios de comunicación que contienen partículas virales se pueden mantener en el refrigerador por 2 semanas sin perder la actividad infecciosa, sin embargo, no se recomienda la congelación).
- Para retrovirales transducción, los fibroblastos humanos adultos de la placa (paso 10, Coriell Laboratories) de un stock congelado 24 horas anteriores al último día de recolección de los medios de comunicación retroviral. Semillas de las celdas de 6-así gelatina placas cubiertas a la densidad 1x10 5 células por pocillo en DMEM glucosa alta, 10% de FBS, 2 mM de L-glutamina, penicilina, estreptomicina y no esenciales aminoácidos.
- El sustituto día siguiente medio DMEM con medios que contienen partículas virales obtenidos a partir de células de Phoenix. Añadir los medios de comunicación viral (1 ml de cada Oct4, Sox2, Klf4 y c-myc los medios de comunicación, 4 ml en total) suplementado con 6 mg / ml de polibreno en cada pocillo de la placa de 6 pocillos de los cultivos de fibroblastos y centrifugar a 1600 g de 1 hora a 20 ° C. Aproximadamente 12 horas after de la transducción, sustituir a los medios de comunicación virales con medio de fibroblastos cultura. Realizar la infección viral y centrifugación 3 veces en intervalos de 24 horas.
- Fibroblastos de cultivo en medio DMEM durante 48 horas después de la última infección. Controlar la eficacia de la infección por transducciones paralelas con un medio de retrovirales que expresan GFP. La Figura 1 ilustra la eficacia de centrifugación-facilitado infección retroviral de fibroblastos humanos.
2. Reprogramación
- Preparar placas de 6 pocillos con γ-irradiados capas alimentadoras MEF sembrando células MEF a una densidad de ~ 2x10 5 células por pocillo de la placa de gelatina tratada 6 pocillos, en medio de crecimiento de fibroblastos. El día siguiente dividir fibroblastos humanos infectados con 0,05% de tripsina / EDTA y semillas ellos en la densidad de 1.2x10 ~ 4 por pocillo en el medio de crecimiento de fibroblastos.
- Al día siguiente, reemplazar a los medios de comunicación con HES medios de comunicación (DMEM/F12, la sustitución del 20% de suero golpe de gracia, no aminoácidos esenciales, penicillin / estreptomicina, 2 mM L-glutamina, 0,1 mM β-mercaptoetanol, 20 ng / ml de factor de crecimiento de fibroblastos basal (bFGF), suplementado con 0,5 mM butirato sódico durante los primeros 7 días de la reprogramación. Cambiar los medios de comunicación todos los días.
- Monitorear los cambios morfológicos en las células transducidas todos los días. Las colonias de las caderas-como las células comenzarán a surgir aproximadamente 5 - 10 días después de la transferencia de los fibroblastos en células transducidas de alimentación. Las colonias hiPSC están listos para ser aislado aproximadamente 14 - 28 días después de placas ellos en las células alimentadoras MEF.
3. El aislamiento de colonias hiPSC
- Un día antes de recoger las colonias hiPSC preparar placas de 24 pocillos con la γ-células irradiadas MEF alimentador (aproximadamente 4x10 4 células por pocillo) en medio de crecimiento de fibroblastos. En esta etapa colonias hiPSC también pueden ser transferidos a la cultura alimentador gratis con Matrigel y medio mTeSR1.
- Antes de aislamiento de colonias hiPSC preparar placas MEF mediante la eliminación de los fibroblastos crecimiento medium y aclarado MEFs con PBS para eliminar las trazas de FBS. Posteriormente añadir 0,5 ml hES medio (o mTeSR1 medio en el caso de Matrigel pocillos recubiertos) con 10 mM inhibidor ROCA Y27632 a cada pocillo 7,8.
- Si es necesario, eliminar los fibroblastos que rodean las colonias hiPSC con una aguja de calibre 21 bajo un microscopio montado en una campana de flujo laminar (Figura 2A - C). Lavar las placas con PBS y añadir frescos medios hES que contienen el anticuerpo específico Tra-1-81 StainAlive (1:200, Stemgent). Después de 30 min., Reemplazar medios que contienen el anticuerpo con frescos medios hES suplementado con 10 mM inhibidor ROCA. Examinar las placas bajo el microscopio de fluorescencia y la marca de TRA-1-81 colonias positivas utilizando un marcador objetivo (Figura 2D).
- Cortar los Tra-1-81 colonias positivas hiPSC bajo un microscopio en una campana laminar, en varias piezas pequeñas con una aguja de calibre 21 como se muestra en la Figura 2E y F.
- Usando una pipeta automática (P200) de transferencia de fragmentos de colonias en hiPSC individual pozos de la placa de 24 pocillos con MEFs o Matrigel. Evitar la transferencia de las células que no son las caderas. Coloque las placas en un 37 ° C, 5% de CO 2 incubadora y permitir que las colonias hiPSC para conectar de 24-36 h.
- Cambio hES o mTeSR1 (para las colonias cultivadas en Matrigel) los medios de comunicación todos los días. Las colonias con hiPSC correcta HES-como la morfología será visible 48 horas después de la transferencia inicial sobre una placa de 24 pocillos.
- Manual de paso hiPSC colonias cada 6 - 8 días sobre un 12-así y, posteriormente, en una placa de 6 pocillos.
- Después de la expansión clonal y establecer las líneas hiPSC, analizar la expresión de marcadores de pluripotencia TRA-1-60, SSEA-4, Oct3 / 4, Sox2 y Nanog utilizando inmunocitoquímica como se muestra en la Figura 3. Evaluar la integridad genómica y potencial de diferenciación de las líneas obtenidas mediante cariotipo y análisis de la formación de teratomas 6,9.
4. Los resultados representativos
Transducción eficiente con retrovirus que contiene los medios de comunicación is crítico para el éxito reprogramación. Se recomienda llevar a cabo toda la transfección / infección procedimiento utilizando un GFP expresando virus cada experimento individual reprogramación para supervisar la eficiencia, como se muestra en la Figura 1. El título del virus GFP expresar determinado, como se describe en 10, a través de la transducción de fibroblastos humanos utilizando no concentrados medios virales era típicamente en el intervalo de 0,5 a 5 x 10 7 partículas virales por ml (VP / ml).
Los fibroblastos cambiar la morfología como a los 2 días después de la última infección. Trypsinized fibroblastos humanos deben ser cuidadosamente contarán antes de la siembra de ellos en las células alimentadoras del MEF. Se recomienda a las células de semillas a 3 densidades diferentes (6x10 3, 4 1.2x10, 2.5x10 4 por un solo pocillo de una placa de 6 pocillos), ya que cada línea celular se muestra característica de crecimiento diferente y densidad de siembra es crítico para la eficiencia de reprogramación. Butirato sódico utilizado para elinicial 7 - 14 días de reprogramación aumenta la eficiencia de la formación de hiPSC aproximadamente 5 veces. Con frecuencia, especialmente cuando los fibroblastos infectados se sembraron a una densidad más alta, las células similares a fibroblastos pueden recubrir una placa de cultivo y cubrir colonias hiPSC como se muestra en la Figura 2A. En este caso, la capa de fibroblastos puede ser cuidadosamente levantado y retirado para descubrir colonias hiPSC (Figura 2B). Posteriormente, las colonias hiPSC debe enjuagarse con HES medios y se tiñeron con Tra-1-81 anticuerpo. Dependiendo de las células de fibroblasto, aproximadamente 20 a 40% de las colonias que demuestran con CMPi-como morfología no se tiñen con Tra-1-81 anticuerpo. Identificadas colonias hiPSC puede ser transferido de una placa dentro de la próxima 12 - 24h. Incubación prolongada dará lugar a la diferenciación rápida de hiPSCs. Las colonias pueden ser manualmente pasajes en cualquiera de las células alimentadoras MEF o placas Matrigel recubiertos. Después de la expansión establecida clones hiPSC deben ser evaluados con inmunocitoquímica (CPI) para la expresión de pluripotenCY marcadores como se demuestra en la Figura 3. Además, la caracterización molecular detallada de las líneas de células iPS generadas debe incluir: análisis de la expresión génica pluripotencia mediante RT-PCR, la demostración de la desmetilación del ADN a los promotores de los genes de pluripotencia y análisis de los transgenes de silenciamiento 9.
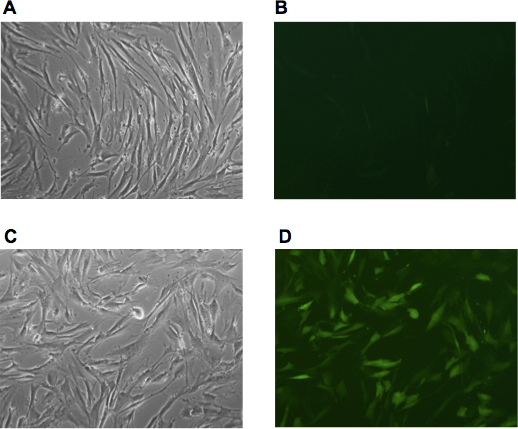
Figura 1. La eficiencia de la transducción viral determinó a través de los retrovirus que expresa GFP. (A, B) fibroblastos humanos adultas derivadas de los pacientes la ataxia de Friedreich (GM03665, Coriell repositorio) se visualizaron después de dos consecutivos con los medios de comunicación las infecciones retrovirales las buenas prácticas agrarias. (C, D) fibroblastos humanos fueron infectados con el mismo lote de los medios de GFP retrovirales seguido por centrifugación de las células directamente sobre las placas 6-y durante 1 hora a 1600g. Las imágenes fueron capturados 48h después de la infección.

Figura 2. Identificación y aislamiento de colonias hiPSC. (A) de la imagen de contraste de fase de una placa que contiene hiPSCs rodeadas por los fibroblastos. Las células se cultivaron durante 21 días en hES medios. (B, C) La misma colonia hiPSC después de la eliminación de la capa de fibroblastos circundante. (D) correctamente reprogramadas colonias hiPSC se identifican mediante tinción vivo con anticuerpos marcador Tra-1-81 superficie, cortar utilizando una aguja estéril (E, F) y se transfirieron a pocillos separados de una placa de 24 pocillos.
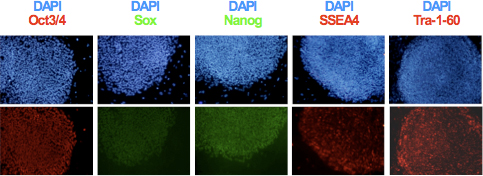
Figura 3. La expresión de los marcadores de pluripotencia específicos Oct3 / 4, Nanog, Sox2, SSEA4 y Tra-1-60 en hiPSCs se determinó por inmunocitoquímica.
Discusión
El estudio de enfermedades humanas, especialmente neurológicas y neurodegenerativas, ha sido particularmente difícil debido a la inaccesibilidad de los adecuados modelos celulares humanos. La posibilidad de reprogramar células somáticas que se obtienen fácilmente en células madre pluripotentes inducidas y el potencial para diferenciarse en diversos tipos de células se abrió la posibilidad de crear modelos celulares de enfermedades genéticas. Además, iPSCs celebrar una gran promesa para el futuro de la medicina...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la Ataxia de Friedreich Research Alliance y una donación de la familia de Arnold piloto de la Fundación y el Centro de Células Madre y Biología del Desarrollo en el MD Anderson Cancer Center.
Materiales
| Name | Company | Catalog Number | Comments |
| Reactivo | Empresa | Número de catálogo | |
| DMEM | Invitrogen | 11965 | |
| DMEM/F12 | Invitrogen | 11330 | |
| KSR | Invitrogen | 10828 | |
| Aminoácidos no esenciales | Invitrogen | 11140 | |
| Butirato sódico | Sigma | B5887 | |
| Y27632 | Stemgent | 04-0012 | |
| bFGF | Stemgent | 03-0002 | |
| Tra-1-81 anticuerpo | Stemgent | 09-0069 | |
| Oct3 / 4 de anticuerpos | Santa Cruz | SC-8628 | |
| Nanog anticuerpos | Señalización Celular Tecnología | 4903S | |
| Tra-1-60 anticuerpo | Millipore | MAB4360 | |
| Sox2 anticuerpos | Señalización Celular Tecnología | 3579S | |
| SSEA4 | Millipore | MAB4304 | |
| CF1 MEFs | Globalstem | GSC-6201G | |
| Marcador de objetivo | Nikon | MBW10010 | |
| Matrigel | BD Biosciences | 354277 | |
| mTeSR1 | StemCell Tecnologías | 05850 | |
| β-mercaptoetanol | Sigma | M7522 | |
| Fugene 6 | Roche | 11814443001 | |
| polibreno | Sigma | H9268 | |
| Objeto marcador | Nikon | MBW10010 |
Referencias
- Takahashi, K., Tanabe, K., Ohnuki, M., Narita, M., Ichisaka, T., Tomoda, K., Yamanaka, S. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131, 861-872 (2007).
- Mali, P., Chou, B. K., Yen, J., Ye, Z., Zou, J., Dowey, S., Brodsky, R. A., Ohm, J. E., Yu, W., Baylin, S. B., Yusa, K., Bradley, A., Meyers, D. J., Mukherjee, C., Cole, P. A., Cheng, L. Butyrate greatly enhances derivation of human induced pluripotent stem cells by promoting epigenetic remodeling and the expression of pluripotency-associated genes. Stem Cells. 28, 713-720 (2011).
- Wernig, M., Meissner, A., Foreman, R., Brambrink, T., Ku, M., Hochedlinger, K., Bernstein, B. E., Jaenisch, R. In vitro reprogramming of fibroblasts into a pluripotent ES-cell-like state. Nature. 448, 318-324 (2007).
- Lowry, W. E., Richter, L., Yachechko, R., Pyle, A. D., Tchieu, J., Sridharan, R., Clark, A. T., Plath, K. Generation of human induced pluripotent stem cells from dermal fibroblasts. Proc. Natl. Acad. Sci. U.S.A. 105, 2883-2888 (2008).
- Dick, E., Matsa, E., Bispham, J., Reza, M., Guglieri, M., Staniforth, A., Watson, S., Kumari, R., Lochmuller, H., Young, L., Darling, D., Denning, C. Two new protocols to enhance the production and isolation of human induced pluripotent stem cell lines. Stem Cell Res. 6, 158-167 (2011).
- Ku, S., Soragni, E., Campau, E., Thomas, E. A., Altun, G., Laurent, L. C., Loring, J. F., Napierala, M., Gottesfeld, J. M. Friedreich's ataxia induced pluripotent stem cells model intergenerational GAATTC triplet repeat instability. Cell Stem Cell. 7, 631-637 (2010).
- Emre, N., Vidal, J. G., Elia, J., O'Connor, E. D., Paramban, R. I., Hefferan, M. P., Navarro, R., Goldberg, D. S., Varki, N. M., Marsala, M., Carson, C. T. The ROCK inhibitor Y-27632 improves recovery of human embryonic stem cells after fluorescence-activated cell sorting with multiple cell surface markers. PLoS One. 5, e12148-e12148 (2011).
- Watanabe, K., Ueno, M., Kamiya, D., Nishiyama, A., Matsumura, M., Wataya, T., Takahashi, J. B., Nishikawa, S., Muguruma, K., Sasai, Y. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nat. Biotechnol. 25, 681-686 (2007).
- Maherali, N., Hochedlinger, K. Guidelines and techniques for the generation of induced pluripotent stem cells. Cell Stem Cell. 3, 595-605 (2008).
- Yu, J., Thomson, J. A., Lanza, R. Induced Pluripotent Stem Cell Derivation. Essentials of Stem Cell Biology. , 331-337 (2009).
- Yu, J., Hu, K., Smuga-Otto, K., Tian, S., Stewart, R., Slukvin, I. I., Thomson, J. A. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324, 797-801 (2009).
- Woltjen, K., Michael, I. P., Mohseni, P., Desai, R., Mileikovsky, M., Hamalainen, R., Cowling, R., Wang, W., Liu, P., Gertsenstein, M., Kaji, K., Sung, H. K., Nagy, A. piggyBac transposition reprograms fibroblasts to induced pluripotent stem cells. Nature. 458, 766-770 (2009).
- Zhou, W., Freed, C. R. Adenoviral gene delivery can reprogram human fibroblasts to induced pluripotent stem cells. Stem Cells. 27, 2667-2674 (2009).
- Warren, L., Manos, P. D., Ahfeldt, T., Loh, Y. H., Li, H., Lau, F., Ebina, W., Mandal, P. K., Smith, Z. D., Meissner, A., Daley, G. Q., Brack, A. S., Collins, J. J., Cowan, C., Schlaeger, T. M., Rossi, D. J. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell Stem Cell. 7, 618-630 (2010).
- Kim, D., Kim, C. H., Moon, J. I., Chung, Y. G., Chang, M. Y., Han, B. S., Ko, S., Yang, E., Cha, K. Y., Lanza, R., Kim, K. S. Generation of human induced pluripotent stem cells by direct delivery of reprogramming proteins. Cell Stem Cell. 4, 472-476 (2009).
- Han, S. S., Williams, L. A., Eggan, K. C. Constructing and deconstructing stem cell models of neurological disease. Neuron. 70, 626-644 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados