Method Article
Método para el aislamiento e identificación de mRNAs, microRNAs y componentes proteicos de ribonucleoprotein complejos de extractos celulares usando RIP-Chip
En este artículo
Resumen
Un paso a paso el protocolo para aislar e identificar los complejos de ARN asociados a través de RIP-Chip.
Resumen
Como resultado del desarrollo de la secuenciación de alto rendimiento y análisis de microarrays eficiente, el análisis global de la expresión génica se ha convertido en una forma fácil y fácilmente disponible de recogida de datos. En la investigación de muchos modelos de enfermedad y sin embargo, los niveles de estado estacionario de ARNm del gen diana no siempre se correlacionan directamente con equilibrio los niveles estatales de proteínas. Regulación post-transcripcional de genes es una probable explicación de la divergencia entre los dos. Impulsada por la unión de las proteínas de unión a ARN (RBP), post-transcripcional regulación afecta a la localización del mRNA, la estabilidad y la traducción mediante la formación de una ribonucleoproteína (RNP) con complejo ARNm diana. La identificación de estos desconocidos objetivos de mRNA de novo a partir de extractos celulares en el complejo de RNP es fundamental para comprender los mecanismos y funciones de la RBP y su efecto resultante en la producción de proteínas. Este protocolo describe un método denominado RNP inmunoprecipitación-microarray (RIP-Chip), que permite la identificación de smRNAs ESPECÍFICAS asociado en el complejo ribonucleoprotein, bajo las cambiantes condiciones experimentales, junto con las opciones para optimizar aún más un experimento para el investigador individual. Con esta herramienta experimental importante, los investigadores pueden explorar los intrincados mecanismos asociados con la regulación post-transcripcional de genes, así como otras interacciones ribonucleoprotein.
Protocolo
Experimento preparación
Antes de comenzar el experimento, es crítico tener todos los reactivos, recipientes y utensilios libre de RNasa. Tratar cristalería con inhibidor de RNasa (RNaseZAP, Ambion) seguido de aclarado con agua tratada con DEPC. Asegúrese de que todos los reactivos se confirman como RNasa libre.
1. Preparar mRNP Lysate
- Cultivar y cosechar las células del tejido que crecen exponencialmente a producir entre 2-5 mg de proteína total de cada RIP.
- Dos P150 placas de cultivo suelen ser suficientes.
- Para cada RBP está investigando, el total de las cantidades celulares número y proteína debe ser optimizada para maximizar ARNm diana apropiada y las interacciones PER.
- RIP para QRT-PCR análisis puede requerir lisado celular menos (aproximadamente 400 g de proteína total), debido a su amplificación y método de detección especialmente para las prácticas comerciales restrictivas alta abundancia tales como HuR, AUF1, TIAR.
- Precipitar las células mediante centrifugación a 200 xg durante 8-10 min a 4 ° C, luego lavar tres veces con PBS enfriado con hielo.
- Después del lavado final, dé golpecitos en la parte inferior del tubo y se resuspende el sedimento celular en volúmenes iguales de tampón de lisis preparada polisomas (PLB) con inhibidores de proteasa y RNasa.
- Se recomienda medir el volumen exacto del sedimento celular.
- Suavemente pipeta de mezcla (no vórtex) para separar las matas de células lisado y se incuba en hielo durante 5 min.
- Se centrifuga a 13.000 xg durante 20 min a 4 ° C para eliminar el lisado de los desechos.
- Transferir inmediatamente aclaró sobrenadante a un tubo de microcentrífuga pre-enfriado.
- Mantener en hielo y se almacena a -80 ° C.
- Inmediatamente la congelación completa la lisis apropiado de las células y previene la unión no deseada. Proceda con deshielo RIP muestra inmediatamente siguiente y mantener las muestras en hielo para evitar la degradación del ARN.
- Este lisado se puede almacenar durante hasta seis meses a -80 ° C. Evitar ciclos repetidos de congelación y descongelación ya que esto puede dar lugar a proteínas y / o degradación del ARNm.
- Cuantificar la concentración de proteínas en el lisado utilizando estándar de ensayo de proteínas de Bradford.
2. Proteína de la cubierta A Sepharose bolas con anticuerpos y Wash
- Pre-oleaje de proteína A Sepharose (PAS) las perlas durante la noche en tampón NT2 (volúmenes 3-4) con 5% de BSA. Almacenar a 4 ° C.
- El almacenamiento a largo plazo de hasta varios meses a 4 ° C es posible cuando se suplementa con azida sódica al 0,1%.
- Perlas de Sepharose proteína debe ser elegido sobre la base de isotipo de anticuerpo diana RBP. Las proteínas A y G, y A / G todos tienen objetivos específicos de isotipo y variar en la afinidad de destino. Proteína A Sepharose perlas se utilizaron basa en la especificidad de isotipo y la afinidad por anticuerpo de proteína HuR.
- Antes de uso, eliminar el tampón NT2 exceso, por loque las cuentas finales al ratio de amortiguación es de 1:1.
- El uso de 1,5 ml RNasa libre de tubos de microcentrífuga, eliminar el 100 l de suspensión de PAS y añadir 30 g de anticuerpo para cada reacción IP individual (es decir, específica y las prácticas comerciales restrictivas de control de isotipo).
- Añadir 100-200 l de tampón NT2 para anticuerpo mezcla de microesferas.
- Mezcla puede conservarse durante varias semanas a 4 ° C cuando se suplementa con azida sódica al 0,1%.
- Un anticuerpo isotipo o suero normal de las mismas especies conjunto se debe utilizar en paralelo como un control de anticuerpo contra ARN fondo.
- Añadir anticuerpo apropiado para mezcla de microesferas y se incuba durante la noche, dando tumbos extremo sobre extremo a 4 ° C.
- Optimizar el título de anticuerpos para la proteína específica que se está investigando (g 1, 5, 10 o 30 de anticuerpo es generalmente suficiente).
- Preparar perlas recubiertas con anticuerpo inmediatamente antes de su uso mediante lavado con 1 ml deenfriado con hielo NT2 tampón 5 veces.
- Lavar mezcla de perlas por centrifugación a 13.000 xg durante 1-2 min a 4 ° C.
- Retire con cuidado la cantidad máxima de sobrenadante con pipeta o aspirador de mano, pero tenga cuidado de no perturbar pellet.
- Lavar ayuda a eliminar el anticuerpo no unido, así como contaminantes de RNasa mezcla de anticuerpos.
- Una vez que el lavado final se ha completado, resuspender las perlas en 700 l de tampón enfriado con hielo NT2 seguido de tratamiento con diversos inhibidores de RNasa para proteger a los ARNm diana, incluyendo 10 l de RNasa a cabo a 40 U / l, 10 l de 100 mM DTT y 15 l de EDTA (15 mM). Se ajusta el volumen a 1.000 ml con tampón NT2.
- Complejos de vanadilo ribonucleósidos no se utilizan debido a un efecto inhibidor de EDTA.
3. Inmunoprecipitación y precipitación del ARN
- Paso preclaro Opcional:
- Para reducir el fondo, las perlas y el anticuerpo de control puede ser usado para pre-lisado claro. Esto puede reducir la señal en la salida.
- Este paso puede ser necesario para reducir el fondo cuando se hace IP seguido por microarray. Generalmente, no es necesario que IP seguida por QRT-PCR.
- Preclear con 15 g de isotipo de control durante 30 min / 4 º C final cayendo sobre el extremo.
- Añadir 50 l de perlas preswollen PAS no recubiertas con anticuerpos del paso 2.1.
- Incubar 30 min / 4 º C con rotación sobre el extremo final
- Centrifugar a 10.000 xg a 4 ° C. Guardar el sobrenadante por IP.
- Añadir 100 l de lisado aclarado aislado (aproximadamente 2-5 mg) a la mezcla de anticuerpos preparada.
- La dilución de lisado ayudará a reducir el fondo y la unión no específica.
- Cantidad de entrada de lisado puede variar dependiendo del método de detección y la abundancia de RBP or eficiencia de anticuerpo RBP como se indica en el paso 1.1.c.
- (Opcional) Añadir inmediatamente tubo suavemente agitando varias veces seguidas por breve centrifugación a 10.000 xg a 4 ° C con perlas de pellets y retirar inmediatamente 100 l de sobrenadante como una representación de entrada total de ARNm para el análisis de qPCR utilizando técnicas estándar de aislamiento de ARN.
- Este paso es confirmar que la entrada de lisado de ARN es óptimo para IP y sólo debe realizarse como un paso de verificación o como un paso de solución de problemas siguiendo RIP con pobres resultados de ARN.
- Wrap tubo en parafilm para asegurar el cierre hermético y se incuba a 4 ° C durante 2 a 4 horas, cayendo de punta a punta.
- Timing en incubación debe ser optimizado basado en la abundancia de destino y debe reducirse al mínimo para evitar reordenamiento complejo o degradación. Para algunas prácticas comerciales restrictivas incubaciones más cortos pueden ser más óptima.
- Pellet cuentas a 5.000 XGFo 5 min a 4 ° C y guardar el sobrenadante para el análisis del potencial por Western blot. Tienda de alícuotas a -80 ° C.
- Las alícuotas de sobrenadante con altas cantidades de proteína diana residual puede indicar un fallo de la proteína a ser precipitada por perlas de sefarosa.
- Como se ha descrito anteriormente, lavar perlas de 5 veces con 1 ml de tampón enfriado con hielo NT2 y centrifugación (5.000 xg durante 5 min a 4 ° C) y luego eliminar el sobrenadante con pipeta de mano o un aspirador.
- Métodos de lavado más rigurosas pueden ser utilizados con el fin de reducir el fondo, completándolo NT2 tampón con desoxicolato de sodio, urea o SDS.
- Mantener las muestras en hielo tanto como sea posible y trabajar con rapidez para minimizar la degradación de ARNm diana.
- Opcional: Guardar una pequeña alícuota (10%) de mezcla de perlas o ejecutar una muestra adicional en paralelo para comprobar la eficacia de IP proteína diana usando análisis de transferencia Western.
4. DNasa y Tratamientos proteinasa K
- Después del lavado, se resuspenden las perlas con 100 l de tampón NT2 suplementado con 5 l de RNasa libre de DNasa 1 (2 U / l).
- Mantener a 37 ° C durante 5-10 minutos. Añadir 1 ml de tampón NT2 y centrifugar a 5.000 xg durante 1 min a temperatura ambiente.
- Un pequeño (~ 10%) alícuota de perlas se puede utilizar para comprobar la eficiencia de IP por análisis de SDS-PAGE.
- Resuspender el pellet en 100 PAS l tampón NT2, 5 l de proteinasa K (10 mg / ml), y 1 l 10% de SDS.
- Incubar la mezcla resuspendida de cuentas durante 30 min a 55 ° C en un baño de agua, agitando suavemente en cada 10 min.
- Proteinasa K ayudará en la liberación de los componentes RNP.
- Pellets perlas a 5.000 xg durante 5 min a temperatura ambiente (RT) y recoger el sobrenadante (~ 100 l).
- Añadir 200 l NT2 tampón a las perlas, centrifuge 5.000 xg durante 2 min a TA, recoger el líquido sobrenadante (~ 200 l), y perlas de descarte.
- Combine sobrenadantes (100 l y l 200) y añadir 300 l capa inferior de ácido fenol-CHCl 3.
- Vortex, 1 min RT (o 37 ° C en agitador) y se centrifuga a 16.000 xg a TA durante 1 min.
- Tomar 250 l de capa superior (NO interrumpir interface), se añaden 25 l de acetato de sodio a pH 5,2, 625 l 100% ETOH y 5 glycoblue l, y mezclar bien.
- Almacenar durante la noche a -20 ° C.
- Para asegurar la recuperación adecuada de ARN, la adición de glycoblue aumentará la visibilidad de la ARN precipitado al actuar como una molécula portadora.
- El día siguiente, los tubos se mezcla por inversión 3-5 veces, centrifugado a 12.000 xg a 4 ° C durante 30 min y descartar el sobrenadante.
- Añadir 1 ml de EtOH al 70% a la pastilla azul y mezclar por inversión o agitación con vórtex.
- Centrifugar 12.000 xg a 4 ° C durante 2 min.
- Discard sobrenadante y centrifugar 12.000 xg durante 1 min a 4 ° C.
- Eliminar cualquier ETOH residual 70% con una pipeta y el aire de gránulo seco a temperatura ambiente durante 5 min.
- Resuspender en 20-40 l de RNasa / DNasa libre de agua o de otro volumen apropiado según sea necesario.
- Muestra está ahora lista para otras aplicaciones posteriores tales como QRT-PCR o microarrays.
- Espectrofotometría Nanodrop se puede utilizar en la evaluación de la concentración de la muestra, sin embargo, con preciosas muestras, puede ser beneficioso para evitar muestras nanodropping como puede ser un desperdicio y relativamente inexacta. Sugerimos a normalizar las muestras transcripciones de genes de limpieza para evaluar la pureza y la eficiencia de rendimiento IP para muestras preciosas o baja.
5. Los resultados representativos
Si el procedimiento se optimiza y se realiza correctamente, la inmunoprecipitación debe producir enriquecimiento significativo de mRNA objetivos. Típicamente, Dependiendo de la RBP y su ARNm objetivo (s), vemos enriquecimiento de aproximadamente 10 - a 50-veces cuando se evaluó mediante qRT-PCR. Muchos objetivos de las prácticas comerciales restrictivas pueden ser descubiertos de forma masiva mediante el análisis de microarrays. Sin embargo, este método es más sensible a la degradación en comparación con QRT-PCR. Dependiendo de la RBP, el número de objetivos y la eficiencia de la reacción, microarray puede revelar cientos de nuevas dianas, o sólo puede descubrir unos pocos, si los hay. Por ejemplo, uno de los mejor caracterizados ARN proteínas de unión HuR, post-transcriptionally regula la expresión y traducción de muchos genes fisiológicos importantes 1, 2. Aislamiento de la HuR-ribonucleo-complejo a través de RIP-Chip mama en las líneas celulares de cáncer, por ejemplo, reveló el enriquecimiento de varios objetivos importantes Hur conocidos, incluyendo β-actina utilizando QRT-PCR como se muestra en la figura 1. En ambas líneas celulares de cáncer de β-actina se enriquece 12 - a 15-veces. Por lo general, cuando se realiza correctamente, vemos un significativo enrichment de β-actina en una variedad de líneas celulares. Sin embargo, si el RIP no revela ningún enriquecimiento significativo para la β-actina esto indica un problema con el PIR y el procedimiento puede ser necesario repetir. Por otra parte, el análisis de microarrays de muestras inmunoprecipitadas de estas líneas celulares reveló subconjuntos distintos de expresión de los objetivos de Hur en el receptor de estrógeno (ER diferente) de células positivas MCF-7 de cáncer en comparación con ER negativos MB-231 líneas celulares de cáncer de mama como se muestra en la Figura 2 1. Estos objetivos se dividen en varias categorías: objetivos Hur conocidos y desconocidos que estaban asociados ya sea o no relacionado con el cáncer. Por ejemplo, CALM2 y CD9 son ambos genes del cáncer que no habían sido identificados como objetivos Hur. Uso del microarray y confirmar con QRT-PCR, CALM2 y CD9 se encontró que 5 - a 180-veces enriquecido en el sedimento HuR indicando una interacción entre la proteína prominente HuR y estos genes objetivo.

Figura 1. Inmunoprecipitación y RIP en MB-231 (ER-) y MCF-7 (ER +) células de cáncer de mama. Inmunoprecipitaciones se realizaron a partir de MB-231 o MCF-7 lisados celulares usando anticuerpos anti-Hur anticuerpo monoclonal (3A2) y de control de isotipo IgG1. A. propiedad intelectual occidental de Hur reveló banda esperaba tamaño detectado por 3A2. Panel de la derecha revela cantidades de HuR en lisados de entrada utilizado de ambas líneas celulares, con β-tubulina como control de carga. B. Verificación por cuantitativos RT-PCR mostró quince y once veces enriquecimiento de β-actina, un objetivo Hur conocido, en los proyectos de investigación 3A2 de MB-231 y MCF-7, respectivamente. Todos los valores se normalizaron a ΔΔCT GAPDH. Los experimentos se realizaron por duplicado (n = 2).
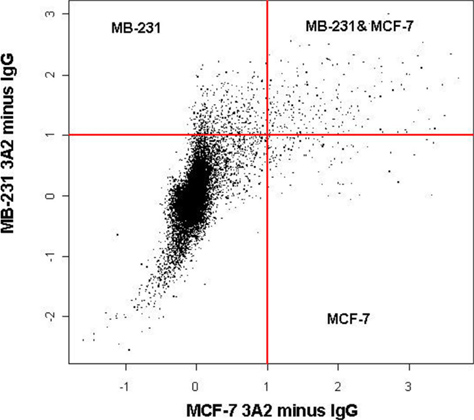
Figura 2. Hur RIP-CHIP identifica distintos perfiles genéticos en ER + y ER-células de cáncer de mama.Hur inmunoprecipitaciones se realizaron a partir de MB-231 o MCF-7 de células lisados utilizando anticuerpo HuR y control de isotipo IgG1 hibridó a Illumina matrices Sentrix (47.000 genes). Las señales de control se restaron. Los resultados representan los datos acumulados de 12 conjuntos diferentes. Los experimentos se realizaron por triplicado (n = 3) para cada línea celular con los controles correspondientes. Las escalas son log2.
Discusión
Debido a la naturaleza de este experimento, la optimización y la experiencia serán las únicas maneras garantizadas para adquirir satisfactoriamente los resultados previstos. En muchas etapas de este procedimiento, la temperatura y el manejo eficiente de los reactivos y los productos son de importancia crítica. La correcta planificación y ejecución de la técnica ayudará a asegurar que el experimento se llevó a cabo en un plazo adecuado a las temperaturas óptimas recomendadas. Un problema importante con los experimentos de aislamiento de ARN es la sensibilidad del ARN a la degradación por RNasas. Todos los reactivos deben estar libre de RNasa y almacenarse o usarse en RNasa libre de contenedores. Este es un paso crítico para asegurar la integridad de la muestra de ARNm. Incluso cuando el experimento se lleva a cabo correctamente, sin embargo, el resultado deseado no se puede lograr debido a la naturaleza de la interacción entre el RBP y sus mRNAs diana.
Un problema potencial es que tiene una baja o incluso ninguna señal desde el ARN aislado por RIP-Chip.Aunque puede haber señal partir de ARN total, esto puede ser el resultado de la inadecuada de proteínas de unión está empujado hacia abajo por las perlas. La etapa de resolución de problemas primero es para confirmar que el lisado celular que se utiliza tiene una expresión adecuada de la RBP específico. Tras la confirmación, la proteína puede aislarse después de la última NT2 lavado y se resuspendieron en tampón de Laemmli u otro tampón apropiado de desnaturalización y se calentó a 95 º C durante 5 min. El análisis Western blot se pueden utilizar en estas muestras en coordinación con lisado de entrada, así como controles negativos para asegurar suficiente tirar hacia abajo de la proteína asociada.
Además, debido a lisis de la célula es necesaria para acceder a estos componentes, el potencial para las interacciones anormales y no deseados entre las proteínas normalmente separados y el ARNm se puede introducir. Estas interacciones podrían unirse y "disfrutar" de sus mRNAs diana o proteínas de unión a través de interacciones no específicas. Además, las proteínas en estos co variandonditions puede doblar en múltiples variaciones y sus motivos de unión puede llegar a ser inaccesible para sus mRNAs diana, prevenir sus interacciones. Ambos reforzar la importancia de trabajar de manera eficiente así como la utilización de las temperaturas óptimas enumerados para limitar estas interacciones no deseadas. Adicionalmente, la optimización de las condiciones de lavado para cada proteína diana específica será crítico para maximizar la pureza de la interacción. Condiciones de lavado más rigurosas pueden ser necesarios. Por ejemplo, el tampón de lavado puede ser suplementado con SDS o una cantidad apropiada de urea para reducir las interacciones no específicas y de fondo en la señal de salida. Esto será completamente dependiente de RBP objetivo del experimentador, así como el ARNm diana en sus condiciones fisiológicas únicas. Algunas condiciones no será adecuado para ciertas herramientas de análisis de ARNm, que deben tenerse en cuenta en la preparación de muestras.
Por último, aunque RIP tiene éxito en el enriquecimiento de ARN-RBP interacciones, un problema bien conocido con RIP (CHIP) método es la incapacidad para identificar los dominios de unión específica del RBP sobre los objetivos de mRNA transitorios. Varios enlazan transversal técnicas se pueden utilizar seguido de RIP para aislar objetivos únicos de secuencia, sin embargo, el uso de UV de onda corta tiende a conducir a daño por ácido nucleico. Un nuevo método conocido como PAR-CLIP, o ribonucleósido reticulación fotoactivable y immuoprecipitation, emplea UV de onda larga para incorporar tiouridina en ARN naciente que permiten la identificación de los únicos sitios de unión de las interacciones ARN tanto estables y transitorios.
En general, RIP-Chip se ha establecido como una excelente herramienta para aislar y estudiar las interacciones entre proteínas de unión al ARN y sus objetivos de mRNA por nuestro grupo, así como muchos otros grupos de investigación. Aunque de naturaleza sensible y práctica, la correcta ejecución de este procedimiento producirá el aislamiento de estos complejos RNP, que hasta hace poco, han sido INACCessible para el descubrimiento y el análisis.
Divulgaciones
Los autores declaran que no tienen intereses en conflicto.
Agradecimientos
Departamento de Defensa (Premio Idea W81XWH-07-0406) - Para Ulus Atasoy
NIH RO1 A1080870 - Para Ulus Atasoy
NIH R21 A1079341 - Para Ulus Atasoy
Universidad de Missouri Fondos Institucionales - Para Ulus Atasoy
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre de Reactivo | Empresa | Número de Catálogo | Comentarios |
| 1 M de ditiotreitol | Pescador | BP172-5 | TDT |
| DNasa 1 | Ambion | 2235 | RNasa libre |
| Ácido etilendiamina Tetraccetic | Pescador | BP118-500 | EDTA |
| El glucógeno | Ambion | 9516 | |
| 1 HEPES | Sigma | H3375-100G | pH 7,0 |
| Igepal Nonidet P-40 | USB | 78641 | NP40 |
| 1 M KCl | Pescador | BP366-500 | |
| 1 M MgCl 2 | Pescador | BP214-500 | |
| NT2 Buffer | * Ver más abajo | ||
| Tampón de Lisis polisomas | * Ver más abajo | ||
| Proteasa comprimidos cóctel inhibidor | Roche | 11873580001 | |
| Proteína A Sefarosa | Sigma | P3391 | |
| Proteinasa K | Pescador | BP1700-100 | |
| Fuera RNasa inhibidor de RNasa | Invitrogen | 10777-019 | 40 U / l |
| 1 M NaCl | Pescador | BP358-212 | |
| Dodecil sulfato de sodio | Pescador | BP166-500 | SDS |
| 1 M Tris-HCl | Pescador | BP153-500 | pH 7,4 |
| Trizol | Invitrogen | 15596-026 | |
| Complejos ribonucleósido vanadilo | New England Labs | S1402S | VRC |
| Reactivo Workup |
| Preparar los reactivos en RNasa / DNasa libre, tratada con DEPC cristalería |
| Tampón de lisis polisomas |
| 100 mM KCl |
| 5 mM MgCl 2 |
| 10 mM de HEPES (pH 7,0) |
| 0,5% de NP40 |
| 1 mM DTT |
| 100 unidades / ml de RNasa Salida |
| 400 mM VRC |
| Inhibidor de proteasa cóctel comprimidos |
| 5 ml de tampón de lisis polisomas |
| Añadir 50 l de 1 M de HEPES (pH 7,0) |
| 500 l de 1 M KCL |
| 25 l de 1 M de MgCl 2 |
| 25 l de NP40 |
| 4,7 ml de RNasa libre de DNasa H 2 O |
| 50 l de 1 M DTT |
| 12,5 l de 100 U / ml de RNasa Salida |
| 200 l cóctel inhibidor de la proteasa (disuelto según fabricante) |
| 10 l 200 mM VRC (en el momento de uso) |
| NT2 Buffer |
| 50 mM Tris-HCl (pH 7,4) |
| 150 mM NaCl |
| 1 mM MgCl 2 |
| 0,05% de NP40 |
| 1 L de tampón NT2 |
| 50 ml de Tris (pH 7,4) |
| 30 ml de NaCl 5 M |
| 1 ml 1 M MgCl 2 |
| 500 l NP40 |
| 820 ml de RNasa libre de DNasa H 2 O |
Referencias
- Calaluce, R., Gubin, M. M., Davis, J. W., Magee, J. D., Chen, J., Kuwano, Y., Gorospe, M., Atasoy, U. The RNA binding protein HuR differentially regulates unique subsets of mRNAs in estrogen receptor negative and estrogen receptor positive breast cancer. BMC Cancer. 10, 126-140 (2010).
- Gubin, M. M., Calaluce, R., Davis, J. W., Magee, J. D., Strouse, C. S., Shaw, D. P., Ma, L., Brown, A., Hoffman, T., Rold, T. L., Ulus Atasoy, U. Overexpression of the RNA binding protein HuR impairs tumor growth in triple negative breast cancer associated with deficient angiogenesis. Cell Cycle. 9, 3337-3346 (2010).
- Keene, J. D., Komisarow, J. M., Friedersdorf, M. B. RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts. Nature Protocols. 1, 302-307 (2006).
- Tenenbaum, S. A., Lager, P. J., Carson, C. C., Keene, J. D. Ribonomics: identifying mRNA subsets in mRNP complexes using antibodies to RNA-binding proteins and genomic arrays. Methods. 26, 191-198 (2002).
- Tenenbaum, S. A., Carson, C. C., Atasoy, U., Keene, J. D. Genome-wide regulatory analysis using en masse nuclear run-ons and ribonomic profiling with autoimmune sera. Gene. 317, 79-87 (2003).
- Tenenbaum, S. A., Carson, C. C., Lager, P. J., Keene, J. D. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Natl. Acad. Sci. U.S.A. 97, 14085-14090 (2000).
- Baroni, T. E., Chittur, S. V., George, A. D., Tennebaum, S. A. Advances in RIP-Chip Analysis. Methods in Molecular Biology. 419, 93-108 (2008).
- de Silanes Lopez, I., Zhan, M., Lal, A., Yang, X., Gorospe, M. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Natl. Acad. Sci. U.S.A. 101, 2987-2992 (2004).
- de Silanes Lopez, I., Lal, A., Gorospe, M. HuR: post-transcriptional paths to malignancy. RNA Biol. 2, 11-13 (2005).
- Grigull, J., Mnaimneh, S., Pootoolal, J., Robinson, M. D., Hughes, T. R. Genome-wide analysis of mRNA stability using transcription inhibitors and microarrays reveals posttranscriptional control of ribosome biogenesis factors. Mol. Cell Biol. 24, 5534-5547 (2004).
- Hieronymus, H., Silver, P. A. Genome-wide analysis of RNA-protein interactions illustrates specificity of the mRNA export machinery. Nat. Genet. 33, 155-161 (2003).
- Hieronymus, H., Yu, M. C., Silver, P. A. Genome-wide mRNA surveillance is coupled to mRNA export. Genes Dev. 18, 2652-2662 (2004).
- Mukherjee, N., Corcoran, D. L., Nusbaum, J. D. Integrative Regulatory Mapping Indicates that the RNA-Binding Protein HuR Couples Pre-mRNA Processing and mRNA Stability. Molecular Cell. 43, 1-13 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados