Method Article
Método para o isolamento e identificação de mRNAs, microRNAs e componentes proteicos de complexos de ribonucleoproteínas de extratos celulares utilizando RIP-Chip
Neste Artigo
Resumo
Um passo a passo para protocolo de isolamento e identificação de RNA complexos associados através de RIP-Chip.
Resumo
Como resultado do desenvolvimento de alto rendimento de sequenciação e análise de microarray eficiente, a análise de expressão génica global, tornou-se uma forma fácil e prontamente disponível de recolha de dados. Em muitas pesquisas e modelos de doenças no entanto, níveis estáveis de mRNA do gene alvo nem sempre directamente correlacionadas com os níveis de proteína em estado estacionário. Pós-transcricional regulação gênica é a provável explicação para a divergência entre os dois. Impulsionado pela ligação de proteínas de ligação de ARN (RBP), regulação pós-transcricional afecta localização do mRNA, a estabilidade e a tradução através da formação de uma ribonucleoproteína (RNP) complexo com mRNAs alvo. A identificação dessas desconhecidos DE alvos de ARNm de novo a partir de extractos celulares do complexo RNP é fundamental para compreensão dos mecanismos e funções do RBP e o seu efeito sobre a produção de proteína resultante. Este protocolo descreve um método denominado RNP-imunoprecipitação de microarray (RIP-Chip), o que permite a identificação de smRNAs ESPECÍFICAS associado no complexo ribonucleoproteína, sob mudança condições experimentais, juntamente com opções para otimizar ainda mais uma experiência para o pesquisador individual. Com esta importante ferramenta experimental, os pesquisadores podem explorar os intrincados mecanismos associados com pós-transcricional regulação gênica, bem como interacções ribonucleoproteínas outros.
Protocolo
Preparação experiência
Antes de iniciar o experimento, é fundamental ter todos os reagentes, recipientes e utensílios RNAse. Tratar vidro com RNase inibidor (RNaseZAP, Ambion) seguido de lavagem com água tratada com DEPC. Garantir que todos os reagentes são confirmados como RNase livre.
1. Prepare mRNP Lysate
- Cultivar e colher as células de tecido em crescimento exponencial para produzir entre 2-5 mg de proteína total, para cada RIP.
- Dois P150 placas de cultura geral, são suficientes.
- Para cada RBP sendo investigado, o total de celulares quantidades numéricas e proteína deve ser otimizado para maximizar mRNA alvo apropriado e interações da RBP.
- RIP por qRT-PCR pode exigir lisado celular inferior (cerca de 400 ug de proteína total), devido à sua amplificação e método de detecção, especialmente para RBPs abundância elevados, tais como Hur, AUF1, TIAR.
- Células de pelotas através de centrifugação a 200 xg, durante 8-10 min a 4 ° C, em seguida, lavar três vezes com PBS gelado.
- Após a lavagem final, suavemente flick o fundo do tubo e ressuspender pellet celular em volumes iguais de tampão de lise preparado polissoma (PLB), com inibidores de RNase e protease.
- É recomendado para medir o volume exato de pellet celular.
- Mistura suavemente pipeta (não vórtice) para quebrar aglomerados de células e incubar lisado em gelo durante 5 min.
- Centrifugar a 13.000 xg durante 20 min a 4 ° C, para limpar o lisado de detritos.
- Transferir imediatamente apagado sobrenadante para pré-refrigerada tubo de microcentrífuga.
- Manter em gelo e armazenar a -80 ° C.
- Congelamento imediato completa a lise eficaz das células e impede a ligação indesejada. Prossiga com RIP degelo amostra imediatamente a seguir e manter amostras de gelo para evitar a degradação do RNA.
- Este lisado pode ser armazenado durante até seis meses a -80 ° C. Evitar ciclos repetidos de congelamento e descongelamento, pois isso pode levar a proteína e / ou a degradação do mRNA.
- Quantificar a concentração de proteína de lisado usando o padrão de ensaio de proteína de Bradford.
2. Proteína um casaco de Contas Sepharose com anticorpo e Wash
- Pré-inchamento proteína A-Sepharose (PAS) grânulos durante a noite em tampão de NT2 (3-4 volumes) com BSA a 5%. Armazenar a 4 ° C.
- Armazenamento a longo prazo de vários meses a 4 ° C é possível quando suplementado com azida de sódio a 0,1%.
- Pérolas de Sepharose de proteína devem ser escolhidos com base no isotipo do anticorpo RBP alvo. Proteínas A, G e A / G tem todos os alvos específicos de isotipos e variam em afinidade alvo. Pérolas de proteína A-Sepharose foram utilizados com base na especificidade e afinidade para o isotipo do anticorpo a proteína Hur.
- Antes da utilização, remover o tampão de NT2 em excesso, de modoque as contas finais para tampão que é de 1:1.
- Utilizando os tubos de 1,5 ml de RNAse-livre de microcentrífuga, remover 100 ul de suspensão de PAS e adicionar 30 mg de anticorpo para cada reacção de IP individual (ou seja, a RBP específico e de controlo de isotipo).
- Adicione 100-200 ul de tampão de NT2 mistura do anticorpo do grânulo.
- Mistura pode ser conservada durante várias semanas a 4 ° C quando suplementado com azida de sódio a 0,1%.
- Um anticorpo isotipo-combinado ou soro normal de todo mesmas espécies devem ser utilizados em paralelo como um controlo de anticorpo contra ARN de fundo.
- Adicionar anticorpo apropriado para mistura de esferas e incubar durante a noite, caindo sobre a extremidade final a 4 ° C.
- Optimizar a título de anticorpos para a proteína específica que está sendo pesquisado (1, 5, 10 ou 30 | ig de anticorpo é geralmente suficiente).
- Preparar pérolas revestidas com anticorpo imediatamente antes da utilização, por lavagem com 1 ml degelada tampão NT2 5 vezes.
- Lavar mistura de esferas por meio de centrifugação a 13.000 xg, durante 1-2 min a 4 ° C.
- Remova cuidadosamente a quantidade máxima de sobrenadante com pipeta de mão ou aspirador, mas tome cuidado para evitar a perturbação da pelota.
- Lavagem ajuda a remover o anticorpo não ligado, assim como contaminantes de RNase mistura de anticorpos.
- Uma vez que a lavagem final ter sido concluída, voltar a suspender as pérolas em 700 ul de tampão gelado NT2 seguido por tratamento com vários inibidores de RNase para proteger os mRNAs alvo, incluindo 10 uL de RNase Out, 40 U / ul, 10 ul de 100 mM DTT e 15 ul de EDTA (15 mM). Levar o volume a 1000 uL com tampão de NT2.
- Complexos de ribonucleósido de vanadilo não são usados devido ao efeito inibitório do EDTA.
3. Imunoprecipitação e RNA Precipitação
- Passo Preclear opcional:
- Para reduzir o fundo, contas e do anticorpo de controlo pode ser utilizado para pré-claro lisado. Isto pode reduzir o sinal na saída.
- Este passo pode ser necessário para reduzir o fundo quando se faz IP seguido por microarranjo. Não é geralmente necessário para IP seguido por qRT-PCR.
- Preclear com 15 ug de controlo de isotipo para 30 min / 4 ° C extremidade caindo sobre a extremidade.
- Adicionar 50 ul de pérolas de preswollen PAS não-revestidos com anticorpo a partir do passo 2.1.
- Incubar 30 min / 4 ° C com rotação sobre a extremidade final
- Centrifugar a 10.000 xg a 4 ° C. Guarde o sobrenadante para IP.
- Adicionar 100 ul de ligado clarificado isolado (aproximadamente 2-5 mg) à mistura de anticorpo preparada.
- Diluindo lisado vai ajudar a reduzir o fundo e ligação não específica.
- Quantidade de entrada de lisado pode variar dependendo do método de detecção e a abundância de RBP or a eficiência do anticorpo RBP como observado no passo 1.1.c.
- (Opcional) Imediatamente misturar tubo suavemente flicking várias vezes seguidas por centrifugação breve a 10.000 xg a 4 ° C para grânulos aglomerados e remover imediatamente 100 pi de sobrenadante como uma representação total de entrada de mRNA por qPCR análise usando técnicas de isolamento convencionais de ARN.
- Este passo é confirmar que a entrada lisado RNA é ideal para IP e só deve ser realizada como uma etapa de seleção ou como um passo de solução de problemas seguindo RIP com maus resultados de RNA.
- Tubo envoltório em parafilme para assegurar uma vedação estanque e incubar a 4 ° C durante 2 a 4 horas, caindo sobre a extremidade final.
- Temporização em incubação deve ser optimizada com base na abundância alvo e deve ser minimizada para evitar o rearranjo complexo ou degradação. Para algumas RBPs incubações mais curtos pode ser mais optimizada.
- Grânulos de pelotas em 5000 xgfou 5 min a 4 ° C e guarde o sobrenadante para análise por Western blot potencial. Alíquotas de armazenar a -80 ° C.
- Alíquotas de sobrenadante com quantidades elevadas de proteína alvo residual pode indicar um defeito da proteína a ser precipitado por pérolas de sefarose.
- Como descrito anteriormente, lavar grânulos 5 vezes com 1 ml de tampão arrefecido com gelo e centrifugação NT2 (5000 xg durante 5 min a 4 ° C), em seguida, remover o sobrenadante com pipetador de mão ou um aspirador.
- Métodos de lavagem mais severas podem ser usados a fim de reduzir o fundo, completando NT2 tampão com desoxicolato de sódio, ureia ou SDS.
- Manter as amostras sobre gelo tanto quanto possível e trabalhar rapidamente para minimizar a degradação do mRNA alvo.
- Opcional: Guardar numa pequena alíquota (10%) da mistura de esferas ou de executar uma amostra adicional, em paralelo para verificar a eficiência de IP da proteína alvo, utilizando análise de Western blot.
4. DNase e Tratamentos proteinase K
- Após a lavagem, os grânulos Ressuspenda com 100 ul de tampão NT2 suplementado com 5 uL de RNase-free DNase 1 (2 U / ul).
- Manter a 37 ° C durante 5-10 min. Adicionar 1 ml de tampão NT2 e centrifugar a 5000 g durante 1 min à temperatura ambiente.
- Uma alíquota (~ 10%) de pequenas esferas pode ser utilizado para verificar a eficiência de IP por análise SDS-PAGE.
- Ressuspender o sedimento em 100 PAS tampão NT2 ul, 5 jil de proteinase K (10 mg / ml), e 1 ul de SDS a 10%.
- Incubar a mistura de grânulo ressuspenso durante 30 min a 55 ° C num banho de água, suavemente sacudindo a cada 10 min.
- Proteinase K vai ajudar na liberação dos componentes da RNP.
- Grânulos de pelotas a 5000 xg durante 5 min à temperatura ambiente (RT) e recolhe-se o sobrenadante (~ 100 uL).
- Adicionar 200 ul de tampão NT2 a esferas, centrifuge 5000 xg durante 2 min a RT, recolher o sobrenadante (~ pi 200), e os grânulos de descarte.
- Combinar os sobrenadantes (100 uL e 200 uL) e adicionar 300 uL da camada inferior de ácido fenol-CHCl 3.
- Vortex, 1 min RT (ou 37 ° C, em agitador) e centrifugação a 16.000 xg à temperatura ambiente durante 1 min.
- Recolher 250 uL da camada superior (não perturbem interface), adicionar 25 ul de acetato de sódio a pH 5,2, 625 uL de ETOH a 100% e 5 ul glycoblue, e misturar bem.
- Armazenar durante a noite a -20 ° C.
- Para assegurar a recuperação adequada de RNA, a adição de glycoblue irá aumentar a visibilidade do sedimento de ARN, agindo como uma molécula transportadora.
- No dia seguinte, os tubos de centrifugação misturar por inversão 3-5 vezes, a 12000 xg a 4 ° C durante 30 min e desprezar o sobrenadante.
- Adicionar 1 ml de ETOH 70% para a pelota azul e misturar por inversão ou vortex.
- Centrifugar 12.000 xg a 4 ° C durante 2 min.
- Discard sobrenadante e centrifugue 12.000 xg durante 1 min a 4 ° C.
- Remover qualquer ETOH residual de 70%, com uma pipeta e ar seco sedimento à temperatura ambiente durante 5 min.
- Ressuspender em 20-40 ul de RNase / DNase-livre de água ou de outro volume adequado, conforme necessário.
- A amostra está agora pronta para aplicações a jusante, tais como qRT-PCR ou microarray.
- Nanodrop espectrofotometria pode ser utilizado para avaliar a concentração da amostra, no entanto, com amostras preciosas, pode ser benéfico para evitar nanodropping amostras, uma vez que pode ser um desperdício e relativamente impreciso. Sugerimos amostras de normalização para transcritos do gene de limpeza para avaliar a pureza e eficiência IP para amostras preciosas ou de rendimento baixo.
5. Resultados representativos
Se o processo é optimizado e realizada correctamente, a imunoprecipitação deve produzir um enriquecimento significativo de alvos de RNAm. Tipicamente, Dependendo da RBP e seu alvo mRNA (s), vemos enriquecimento de cerca de 10 - a 50 vezes maior quando avaliada por qRT-PCR. Muitos alvos de RBPs pode ser descoberto em massa usando análise de microarray. No entanto, este método é mais sensível à degradação em comparação com qRT-PCR. Dependendo da RBP, o número de alvos e a eficiência da reacção, o microarray pode revelar centenas de novos alvos, ou pode apenas revelar algumas, se houver. Por exemplo, um dos mais bem caracterizadas proteínas de ligação de ARN Hur, pós-transcricionalmente regula a expressão e tradução de muitos genes importantes fisiológicas 1, 2. O isolamento do-Hur ribonucleo complexo via RIP-Chip em linhas celulares de cancro de mama, por exemplo, revelou enriquecimento de vários alvos importantes Hur conhecidos, incluindo β-actina utilizando qRT-PCR, como mostrado na Figura 1. Em ambas as linhas celulares de cancro β-actina é enriquecido 12 - a 15 vezes. Normalmente, quando realizada corretamente, vemos um significativo enrichment de β-actina em uma variedade de linhas celulares. No entanto, se o RIP não revela qualquer enriquecimento significativo para β-actina isto indica um problema com a RIP e o procedimento pode necessitar de ser repetido. Além disso, a análise de amostras de microarray imunoprecipitadas a partir destas linhas celulares revelou subconjuntos distintos de expressão alvos Hur em receptores de estrogénio (ER diferente) positivos células MCF-7 de cancro contra ER negativos MB-231 linhas celulares de cancro da mama, como demonstrado na Figura 2 1. Essas metas cair em várias categorias: metas Hur conhecidos e desconhecidos que foram associada ou não associada ao câncer. Por exemplo, CALM2 CD9 e são ambos genes do cancro, que não foram previamente identificadas como alvos Hur. Usando o microarray e confirmar com qRT-PCR, e CALM2 CD9 foram considerados 5 - a 180 vezes enriquecida no pellet Hur indicando uma interacção entre a proteína proeminente Hur e estes genes alvo.

Figura 1. Imunoprecipitação e RIP em MB-231 (ER-) e MCF-7 (ER +) células de cancro da mama. Imunoprecipitações foram realizadas a partir de MB-231 ou de células MCF-7 lisados utilizando anti-Hur anticorpo monoclonal (3A2) e IgG1 de controlo de isotipo. IP A. ocidental de Hur revelou banda tamanho esperado detectada por 3A2. Painel direito revela em quantidades de Hur em lisados de entrada usado a partir de ambas as linhas celulares, com β-tubulina como controlo de carregamento. B. Verificação por RT-PCR quantitativa mostrou-15 e 11 vezes enriquecimentos de β-actina, um alvo conhecido Hur, nas IPs 3A2 de MB-231 e MCF-7, respectivamente. Todos os valores foram normalizados para ΔΔCT GAPDH. Experimentos foram realizados em duplicata (n = 2).
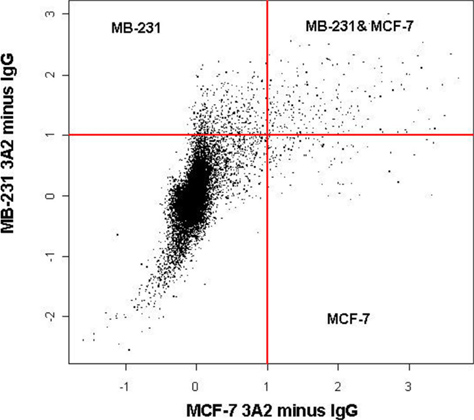
Figura 2. Hur RIP-CHIP identifica diferentes perfis genéticos em ER + e as células de câncer de mama ER-.Hur imunoprecipitações foram realizadas a partir de MB-231 ou de células MCF-7 lisados utilizando o anticorpo Hur e controlo de isotipo IgG1 hibridado com matrizes Illumina Sentrix (47.000 genes). Os sinais de controlo foram subtraídos. Os resultados representam os dados cumulativos de 12 matrizes diferentes. As experiências foram realizadas em triplicado (n = 3) para cada linha de células com os controlos correspondentes. As escalas são log2.
Discussão
Devido à natureza desta experiência, optimização e experiência serão os únicos meios com sucesso garantidos para adquirir os resultados pretendidos. Em muitas etapas deste processo, a temperatura e a manipulação eficiente dos reagentes e produtos são extremamente importantes. Bom planejamento e execução da técnica irá ajudar a garantir que o experimento foi realizado em um prazo adequado para as temperaturas ideais recomendadas. Um grande problema com experiências de isolamento de ARN é a sensibilidade dos RNAs de degradação por RNases. Todos os reagentes devem ser RNase livre e armazenados ou usados de RNase livre de recipientes. Este é um passo fundamental para garantir a integridade de sua amostra de mRNA. Mesmo quando a experiência for realizada correctamente, no entanto, o resultado desejado pode não ser conseguido, devido à natureza da interacção entre o RBP e mRNAs-alvo.
Um problema potencial é ter baixo ou mesmo nenhum sinal da RNA isolado por RIP-Chip.Embora possa haver sinal a partir de ARN total, isto pode ser o resultado de a proteína de ligação inadequada sendo puxado para baixo por os grânulos. O passo de resolução de problemas é primeiro para confirmar que o lisado celular a ser utilizado tem a expressão adequada da RBP específico. Após a confirmação, a proteína pode ser isolado após a lavagem final, NT2 e ressuspensas em tampão de Laemmli ou outro tampão adequado de desnaturação e à temperatura de 95 ° C durante 5 min. A análise por Western blot pode ser utilizada com as amostras em coordenação com lisado de entrada, bem como controlos negativos para assegurar suficiente puxar para baixo de proteína associada.
Além disso, porque a lise da célula é necessário para aceder a estes componentes, o potencial para interacções anormais entre proteínas e indesejáveis normalmente separados e de mRNA podem ser introduzidos. Estas interações poderiam ligar e "absorver" mRNAs seu alvo ou proteínas de ligação através de interações inespecíficas. Além disso, as proteínas em co variando estesnditions pode dobrar em múltiplas variações e seus motivos de ligação pode se tornar inacessível para mRNAs-alvo, impedindo suas interações. Ambos reforçar a importância de se trabalhar eficientemente bem como utilizando as temperaturas óptimas listados para limitar estas interacções indesejáveis. Além disso, a optimização de condições de lavagem para cada proteína alvo específico serão críticos para maximizar a pureza da interacção. Condições de lavagem mais rigorosas podem ser necessários. Por exemplo, o tampão de lavagem pode ser completado com SDS ou de uma quantidade adequada de ureia para reduzir as interacções não específicas e de fundo na sua saída de sinal. Este será completamente dependente de RBP do experimentador alvo, bem como o ARNm alvo em suas únicas condições fisiológicas. Algumas condições não será adequado para ferramentas de mRNA certas análises, que devem ser observados na preparação de amostras.
Finalmente, embora o RIP é bem sucedido no enriquecimento de RNA-RBP interacções, um problema bem conhecido com RIP método (CHIP) é a incapacidade de identificar os domínios de ligação específicos da RBP nos alvos de ARNm de transientes. Várias técnicas de ligação cruzada podem ser usados seguindo-RIP para isolar alvos sequências únicas, no entanto, a utilização de ondas curtas UV tende a levar a danos de ácido nucleico. Um novo método denominado PAR-CLIP, ou de reticulação e ribonucleósido fotoactivável immuoprecipitation, emprega UV de onda longa para incorporar tiouridina em ARN nascentes que permitem a identificação de sítios de ligação únicos de interacções tanto estável e transitória de RNA.
No geral, RIP-Chip foi estabelecida como uma excelente ferramenta usada para isolar e estudar as interações entre proteínas de ligação de RNA e das respectivas metas de mRNA por nosso grupo, assim como muitos outros grupos de pesquisa. Embora sensível por natureza e prática, boa execução deste procedimento irá produzir o isolamento desses complexos da RNP, que até recentemente, têm sido inaccessible para a descoberta e análise.
Divulgações
Os autores declaram que não têm interesses conflitantes.
Agradecimentos
Departamento de Defesa (Prêmio Idea W81XWH-07-0406) - Para Ulus Atasoy
NIH RO1 A1080870 - Para Ulus Atasoy
NIH R21 A1079341 - Para Ulus Atasoy
Universidade de Missouri Fundos Institucionais - Para Ulus Atasoy
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| 1 M de ditiotreitol | Pescador | BP172-5 | TDT |
| DNase 1 | Ambion | 2235 | RNase livre |
| Ácido etilenodiaminotetracético Tetraccetic | Pescador | BP118-500 | EDTA |
| Glicogênio | Ambion | 9516 | |
| 1 HEPES | Sigma | H3375-100G | pH 7,0 |
| Igepal Nonidet P-40 | USB | 78641 | NP40 |
| 1 M KCl | Pescador | BP366-500 | |
| 1 M de MgCl2 | Pescador | BP214-500 | |
| NT2 Tampão | * Veja Abaixo | ||
| Tampão de Lise polissoma | * Veja Abaixo | ||
| Protease comprimidos de cocktail inibidor | Roche | 11873580001 | |
| Proteína A Sepharose Beads | Sigma | P3391 | |
| Proteinase K | Pescador | BP1700-100 | |
| RNase Out inibidor RNase | Invitrogen | 10777-019 | 40 U / ul |
| NaCl 1 M | Pescador | BP358-212 | |
| Dodecil sulfato de sódio | Pescador | BP166-500 | SDS |
| 1 M de Tris-HCl | Pescador | BP153-500 | pH 7,4 |
| Trizol | Invitrogen | 15596-026 | |
| Complexos de vanádio ribonucloesídeos | New England Labs | S1402S | VRC |
| A manipulação de reagente |
| Preparar reagentes em RNase / DNase-livre, tratada com DEPC vidraria |
| Tampão de lise polissoma |
| 100 mM de KCl |
| 5 mM de MgCl2 |
| HEPES 10 mM (pH 7,0) |
| 0,5% NP40 |
| 1 mM DTT |
| 100 unidades / ml de RNase Out |
| 400 uM VRC |
| Protease comprimido cocktail inibidor |
| 5 ml de tampão de lise polissoma |
| Adicionar 50 ul de HEPES 1 M (pH 7,0) |
| 500 ul de 1 M KCl |
| 25 uL de 1 M de MgCl2 |
| 25 ul de NP40 |
| 4,7 ml de RNase DNase-livre H2O |
| 50 uL de 1 M DTT |
| 12,5 uL de 100 U / ml de RNase Out |
| 200 ul de mistura de inibidores de protease (dissolvido de acordo com o fabricante) |
| 10 ul de 200 mM VRC (no momento da utilização) |
| NT2 Tampão |
| 50 mM Tris-HCl (pH 7,4) |
| 150 mM de NaCl |
| 1 mM de MgCl2 |
| 0,05% de NP40 |
| 1 L de tampão de NT2 |
| 50 ml de Tris (pH 7,4) |
| 30 ml de NaCl 5 M |
| 1 ml 1 M MgCl 2 |
| 500 NP40 ul |
| 820 ml de RNase DNase-livre H2O |
Referências
- Calaluce, R., Gubin, M. M., Davis, J. W., Magee, J. D., Chen, J., Kuwano, Y., Gorospe, M., Atasoy, U. The RNA binding protein HuR differentially regulates unique subsets of mRNAs in estrogen receptor negative and estrogen receptor positive breast cancer. BMC Cancer. 10, 126-140 (2010).
- Gubin, M. M., Calaluce, R., Davis, J. W., Magee, J. D., Strouse, C. S., Shaw, D. P., Ma, L., Brown, A., Hoffman, T., Rold, T. L., Ulus Atasoy, U. Overexpression of the RNA binding protein HuR impairs tumor growth in triple negative breast cancer associated with deficient angiogenesis. Cell Cycle. 9, 3337-3346 (2010).
- Keene, J. D., Komisarow, J. M., Friedersdorf, M. B. RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts. Nature Protocols. 1, 302-307 (2006).
- Tenenbaum, S. A., Lager, P. J., Carson, C. C., Keene, J. D. Ribonomics: identifying mRNA subsets in mRNP complexes using antibodies to RNA-binding proteins and genomic arrays. Methods. 26, 191-198 (2002).
- Tenenbaum, S. A., Carson, C. C., Atasoy, U., Keene, J. D. Genome-wide regulatory analysis using en masse nuclear run-ons and ribonomic profiling with autoimmune sera. Gene. 317, 79-87 (2003).
- Tenenbaum, S. A., Carson, C. C., Lager, P. J., Keene, J. D. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Natl. Acad. Sci. U.S.A. 97, 14085-14090 (2000).
- Baroni, T. E., Chittur, S. V., George, A. D., Tennebaum, S. A. Advances in RIP-Chip Analysis. Methods in Molecular Biology. 419, 93-108 (2008).
- de Silanes Lopez, I., Zhan, M., Lal, A., Yang, X., Gorospe, M. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Natl. Acad. Sci. U.S.A. 101, 2987-2992 (2004).
- de Silanes Lopez, I., Lal, A., Gorospe, M. HuR: post-transcriptional paths to malignancy. RNA Biol. 2, 11-13 (2005).
- Grigull, J., Mnaimneh, S., Pootoolal, J., Robinson, M. D., Hughes, T. R. Genome-wide analysis of mRNA stability using transcription inhibitors and microarrays reveals posttranscriptional control of ribosome biogenesis factors. Mol. Cell Biol. 24, 5534-5547 (2004).
- Hieronymus, H., Silver, P. A. Genome-wide analysis of RNA-protein interactions illustrates specificity of the mRNA export machinery. Nat. Genet. 33, 155-161 (2003).
- Hieronymus, H., Yu, M. C., Silver, P. A. Genome-wide mRNA surveillance is coupled to mRNA export. Genes Dev. 18, 2652-2662 (2004).
- Mukherjee, N., Corcoran, D. L., Nusbaum, J. D. Integrative Regulatory Mapping Indicates that the RNA-Binding Protein HuR Couples Pre-mRNA Processing and mRNA Stability. Molecular Cell. 43, 1-13 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados