Method Article
Méthode pour l'isolement et l'identification des ARNm, les microARN et composants protéiques des complexes ribonucléoprotéiques partir d'extraits cellulaires utilisant RIP-Chip
Dans cet article
Résumé
Une étape par étape pour le protocole isolement et l'identification des complexes ARN associés via RIP-Chip.
Résumé
À la suite de la mise au point de séquençage à haut débit et l'analyse par microréseau efficace, l'analyse d'expression génique globale est devenue une forme simple et facilement disponible de collecte de données. Dans la recherche de nombreux modèles de la maladie cependant, l'état d'équilibre de l'ARNm du gène cible ne sont pas toujours directement en corrélation avec les taux de protéines stables de l'État. Régulation post-transcriptionnelle est une explication probable de la divergence entre les deux. Poussé par la liaison de protéines liant l'ARN (RBP), régulation post-transcriptionnelle affecte localisation des ARNm, la stabilité et la traduction en formant une ribonucléoprotéine (RNP) complexe avec des ARNm cibles. L'identification de ces cibles inconnues de novo d'ARNm à partir d'extraits cellulaires dans le complexe RNP est essentielle à la compréhension des mécanismes et des fonctions de la RBP et leur effet résultant sur la production de protéines. Ce protocole décrit une méthode appelée RNP immunoprécipitation de microréseau (RIP-Chip), qui permet l'identification de sARNm SPECIFIQUE associé dans le complexe de ribonucléoprotéine, sous l'évolution des conditions expérimentales, avec des options pour optimiser une expérience pour le chercheur individuel. Avec cet outil expérimental important, les chercheurs peuvent étudier les mécanismes complexes associés à la régulation post-transcriptionnelle, ainsi que les interactions ribonucléoprotéiques autres.
Protocole
Préparation Expérience
Avant de commencer l'expérience, il est essentiel d'avoir tous les réactifs, les contenants et les ustensiles RNase libres. Traiter la verrerie avec inhibiteur de RNase (RNaseZAP, Ambion) suivie d'un rinçage à l'eau traitée au DEPC. Assurez-vous que tous les réactifs sont confirmés comme RNase.
1. Préparer mRNP Lysate
- Cultiver et récolter les cellules des tissus en croissance exponentielle pour produire entre 2-5 mg de protéines totales pour chaque RIP.
- Deux P150 boîtes de culture sont généralement suffisants.
- Pour chaque RBP objet d'une enquête, le total des quantités cellulaires numériques et les protéines doivent être optimisées afin de maximiser l'ARNm cible appropriée et les interactions pratiques commerciales restrictives.
- RIP pour qRT-PCR peut exiger lysat cellulaire moins (environ 400 ug de protéine totale) grâce à son amplification et la méthode de détection en particulier pour les pratiques commerciales restrictives abondance élevés tels que HuR, AUF1, TIAR.
- Sédimenter les cellules par centrifugation à 200 xg pendant 8-10 min à 4 ° C, puis laver trois fois avec PBS glacé.
- Après lavage final, tapotez doucement la partie inférieure du tube et resuspendre le culot cellulaire dans des volumes égaux de tampon de lyse préparée polysome (PLB) avec les inhibiteurs de la protéase et la RNase.
- Il est recommandé de mesurer le volume exact de culot cellulaire.
- Mélange pipette doucement (ne pas vortexer) pour briser les amas de cellules et incuber lysat sur glace pendant 5 min.
- Centrifugeuse à 13.000 xg pendant 20 min à 4 ° C pour effacer le lysat de débris.
- Transférer immédiatement effacé surnageant dans le tube de centrifugeuse pré-réfrigérés.
- Garder sur la glace et conserver à -80 ° C.
- Congélation immédiate complète la lyse des cellules correcte et empêche la liaison indésirable. Passez par le dégel RIP échantillon immédiatement suivant et conserver les échantillons sur la glace pour éviter la dégradation des ARN.
- Ce lysat peut être stocké pendant jusqu'à six mois à -80 ° C. Eviter de multiplier les cycles de congélation-décongélation, car cela peut conduire à des protéines et / ou la dégradation de l'ARNm.
- Quantifier la concentration de protéines dans le lysat en utilisant un dosage standard de protéines Bradford.
2. Protéine d'enveloppe A billes de sépharose avec des anticorps et Wash
- Pré-houle protéine A-Sepharose (PAS) des perles pendant la nuit dans NT2 tampon (volumes 3-4) avec 5% de SAB. Stocker à 4 ° C.
- Le stockage à long terme de plusieurs mois à 4 ° C est possible lorsque complété avec de l'azide de sodium à 0,1%.
- Billes de Sepharose protéines doit être choisie en fonction de l'isotype de l'anticorps RBP cible. Un protéines G, et A / G ont toutes des cibles spécifiques d'isotype et de modification de l'affinité cible. Un protéines perles de sépharose ont été utilisés en fonction de la spécificité et de l'affinité isotype des anticorps anti-protéines HuR.
- Avant toute utilisation, enlever l'excès de tampon NT2, de sorteque les perles finales au rapport du tampon est de 1:1.
- En utilisant des tubes de 1,5 ml microtubes RNase, retirez PAS 100 pl de suspension et ajouter 30 pg d'anticorps pour chaque réaction IP individuelle (c.-à-spécifique RBP et contrôle isotypique).
- Ajouter 100-200 ul de tampon NT2 à l'anticorps talon mix.
- Mélange peut être conservé pendant plusieurs semaines à 4 ° C quand il est complété avec de l'azide de sodium à 0,1%.
- Un anticorps d'isotype apparié ou toute sérums normaux de même espèce doit être utilisé en parallèle comme un contrôle de l'anticorps contre l'ARN de fond.
- Ajouter anticorps approprié mélange de billes et incuber pendant la nuit, tumbling fin sur l'extrémité à 4 ° C.
- Optimiser le titre d'anticorps de la protéine spécifique à l'étude (pg 1, 5, 10 ou 30 de l'anticorps est généralement suffisant).
- Préparer anticorps billes revêtues immédiatement avant l'utilisation d'un lavage avec 1 ml deglacée NT2 tampon de 5 fois.
- Laver mélange de billes par centrifugation à 13.000 xg pendant 1-2 min à 4 ° C.
- Retirez délicatement le maximum de surnageant avec pipette à la main ou un aspirateur mais attention à ne pas perturber le culot.
- Lavage aide à éliminer les anticorps non liés ainsi que les contaminants RNase partir d'un mélange d'anticorps.
- Une fois que le dernier lavage est terminé, remettre en suspension les billes dans 700 ul d'glacé NT2 tampon suivi d'un traitement avec des inhibiteurs de RNase différents pour protéger les ARNm cibles, y compris 10 pl de RNase Rupture à 40 U / ul, 10 ul de 100 mM TNT et 15 ul d'EDTA (15 mM). Amener le volume à 1000 pi avec NT2 tampon.
- Complexes de vanadyle ribonucléoside ne sont pas utilisés en raison de l'effet inhibiteur de l'EDTA.
3. Immunoprécipitation et de l'ARN Précipitations
- Étape facultative Préclair:
- Pour réduire le fond, des perles et des anticorps de contrôle peut être utilisé pour pré-clair lysat. Cela peut réduire de signal à la sortie.
- Cette étape peut être nécessaire de réduire le fond lorsque vous faites IP suivie par microarray. Il n'est généralement pas nécessaire pour IP suivie par qRT-PCR.
- Préclair avec 15 pg de contrôle isotypique pour 30 min / 4 ° C fin tombant sur la fin.
- Ajouter 50 ul de billes PAS pré-gonflée non enrobées avec l'anticorps de l'étape 2.1.
- Incuber 30 min / 4 ° C avec rotation sur l'extrémité fin
- Centrifuger à 10000 xg à 4 ° C. Conserver le surnageant pour la propriété intellectuelle.
- Ajouter 100 ul de lysat clarifié isolé (environ 2-5 mg) de mélange d'anticorps préparée.
- Dilution lysat aidera à réduire le fond et la liaison non spécifique.
- Montant de la contribution lysat peut varier en fonction de la méthode de détection et l'abondance des RBP ol'efficacité de l'anticorps r RBP comme indiqué dans 1.1.C. étape
- (Optionnel) Mélanger immédiatement tube en effleurant doucement plusieurs fois suivi par une brève centrifugation à 10.000 xg à 4 ° C à billes de granulés et de retirer immédiatement 100 pl de surnageant comme une représentation ARNm total des intrants pour l'analyse qPCR en utilisant des techniques d'isolement d'ARN.
- Cette étape consiste à confirmer que l'entrée lysat ARN est optimale pour la propriété intellectuelle et ne doivent être effectuées que d'une étape de contrôle ou comme une étape de dépannage suivant RIP avec des résultats ARN pauvres.
- Envelopper dans un tube de parafilm afin d'assurer une fermeture hermétique et laisser incuber à 4 ° C pendant 2 à 4 heures, tumbling fin sur l'extrémité.
- Timing sur incubation doit être optimisée en fonction de l'abondance cible et doit être minimisée pour éviter réarrangement complexe ou de dégradation. Pour certaines pratiques commerciales restrictives courtes incubations peuvent être plus optimale.
- Perles pellets à 5.000 XGFou 5 min à 4 ° C et conserver le surnageant pour l'analyse par Western blot potentiel. Boutique aliquotes à -80 ° C.
- Des aliquotes de surnageant avec de grandes quantités de protéine cible résiduel peut indiquer une défaillance de la protéine à être précipité par billes de sépharose.
- Comme décrit précédemment, laver perles 5 fois avec 1 ml de tampon glacé NT2 et centrifugation (5000 xg pendant 5 min à 4 ° C), puis enlever le surnageant avec pipette à la main ou un aspirateur.
- Méthodes de lavage plus rigoureuses peuvent être utilisés pour réduire le fond en le complétant NT2 tampon avec le désoxycholate de sodium, l'urée ou le SDS.
- Conserver les échantillons sur la glace autant que possible et de travailler rapidement pour minimiser la dégradation de l'ARNm cible.
- En option: Enregistrer une petite aliquote (10%) du mélange de billes ou d'exécuter un échantillon supplémentaire en parallèle pour vérifier l'efficacité de la protéine cible IP en utilisant une analyse par Western blot.
4. DNase et la protéinase K Traitements
- Après le lavage, les billes remettre les avec 100 ul de tampon NT2 supplémenté avec 5 ul de RNase-free DNase 1 (2 U / pl).
- Conserver à 37 ° C pendant 5-10 min. Ajouter 1 ml de tampon NT2 et centrifuger à 5000 xg pendant 1 min à température ambiante.
- Un petit (~ 10%) aliquote de billes peuvent être utilisés pour vérifier l'efficacité IP par analyse SDS-PAGE.
- Resuspendre PAS concentré dans 100 ul de tampon NT2, 5 pi protéinase K (10 mg / ml), et 1 pi SDS à 10%.
- Incuber le mélange remis en suspension de billes pendant 30 min à 55 ° C dans un bain d'eau, doucement effleurant à toutes les 10 min.
- Protéinase K facilitera la libération des composants RNP.
- Perles granulés dans 5000 xg pendant 5 min à température ambiante (RT) et recueillir le surnageant (~ 100 pi).
- Ajouter 200 ul de tampon NT2 à billes, centrifuge 5000 xg pendant 2 min à température ambiante, ramasser le surnageant (~ 200 pi), et des perles de rejet.
- Combinez surnageants (100 pi et 200 pi) et ajouter 300 ul couche inférieure de l'acide phénol-CHCl 3.
- Vortex, 1 min RT (ou 37 ° C dans un shaker) et centrifuger à 16000 xg à température ambiante pendant 1 min.
- Recueillir 250 ul de la couche supérieure (NE PAS perturber l'interface), ajouter 25 ul d'acétate de sodium à pH 5,2, 625 ul EtOH à 100% et 5 glycoblue pi, et bien mélanger.
- Rangez la nuit à -20 ° C.
- Pour assurer une récupération correcte de l'ARN, l'addition de glycoblue va augmenter la visibilité de la culot d'ARN, en agissant comme une molécule porteuse.
- Le lendemain, les tubes de mélange par inversion 3-5 fois, tournent à 12.000 xg à 4 ° C pendant 30 min et éliminer le surnageant.
- Ajouter 1 ml de EtOH à 70% à la pastille bleue et mélanger par inversion ou vortex.
- Centrifuger 12.000 xg à 4 ° C pendant 2 min.
- Discard surnageant et centrifuger 12.000 xg pendant 1 min à 4 ° C.
- Retirez tout ETOH résiduelle de 70% avec une pipette et l'air sec de bouletage à TA pendant 5 min.
- Remettre en suspension dans ul 20-40 du RNase / DNase eau ou un autre volume approprié selon les besoins.
- L'échantillon est maintenant prêt pour d'autres applications en aval tels que qRT-PCR ou puces à ADN.
- Spectrophotométrie Nanodrop peut être utilisée pour évaluer la concentration d'échantillon, mais de précieux échantillons, il peut être avantageux d'éviter échantillons nanodropping car il peut être inutile et relativement imprécis. Nous vous proposons des échantillons de normalisation à transcrits du gène d'entretien ménager pour évaluer la pureté et l'efficacité IP pour les échantillons précieux de rendement ou faible.
5. Les résultats représentatifs
Si la procédure est optimisé et réalisé correctement, l'immunoprécipitation devrait donner enrichissement significatif des ARNm cibles. Typiquement, En fonction de la RBP et son ARNm cible (s), nous voyons enrichissement d'environ 10 - à 50 fois lorsqu'elle est évaluée par qRT-PCR. De nombreux objectifs de pratiques commerciales restrictives peuvent être découverts en masse en utilisant l'analyse des microréseaux. Cependant, cette méthode est plus sensible à la dégradation par rapport à qRT-PCR. En fonction de la RBP, le nombre de cibles et de l'efficacité de la réaction, puces à ADN peuvent révéler des centaines de nouvelles cibles, ou il ne peut découvrir quelques-uns, le cas échéant. Par exemple, l'un des mieux caractérisés ARN protéines HuR obligatoire, post-transcriptionnel régule l'expression et la traduction de nombreux gènes importants physiologiques 1, 2. L'isolement du HuR-ribonucleo complexe via le protocole RIP-Chip dans les lignes cellulaires du cancer du sein, par exemple, a révélé l'enrichissement de plusieurs objectifs importants Hur connus, y compris β-actine en utilisant qRT-PCR comme le montre la figure 1. Dans les deux lignées de cellules cancéreuses β-actine est enrichie 12 - à 15 fois. En règle générale, lorsqu'il est effectué correctement, nous voyons une importante enrichment de β-actine dans différentes lignées cellulaires. Toutefois, si le RIP ne révèle aucun enrichissement significatif pour β-actine cela indique un problème avec le RIP et la procédure peut être répétée. Par ailleurs, l'analyse des microréseaux d'échantillons immunoprécipités provenant de ces lignées cellulaires ont révélé des sous-ensembles distincts d'expression de cibles Hur en récepteurs aux œstrogènes différents (ER) de cellules positives MCF-7 de cancer par rapport aux ER négatives lignes du sein MB-231 de cellules cancéreuses comme le montre la figure 2 1. Ces objectifs se répartissent en plusieurs catégories: les objectifs Hur connus et inconnus qui étaient soit associés ou non associés au cancer. Par exemple, CALM2 et CD9 sont à la fois les gènes du cancer qui n'ont pas été précédemment identifiées comme cibles Hur. Utilisation de la puce à ADN et en confirmant avec qRT-PCR, CALM2 et CD9 sont révélés être de 5 - à 180 fois enrichi dans le culot HuR indiquant une interaction importante entre la protéine et HuR ces gènes cibles.

Figure 1. Immunoprécipitation et RIP MB-231 (ER-) et MCF-7 (ER +) des cellules cancéreuses du sein. Immunoprécipitations ont été réalisées à partir de MB-231 ou MCF-7 lysats cellulaires en utilisant anti-HuR anticorps monoclonal (3A2) et IgG1 isotype contrôle. IP A. ouest de HuR révélé bande de taille attendue détectée par 3A2. Groupe d'experts sur le droit révèle quantités de HuR en entrée lysats occasion à partir de deux lignées cellulaires, avec β-tubuline comme un contrôle du chargement. B. Vérification par RT-PCR quantitative a montré quinze et onze fois enrichissements de β-actine, une cible HuR connu, dans les PI 3A2 du MB-231 et MCF-7, respectivement. Toutes les valeurs ont été normalisées à ΔΔCT GAPDH. Des expériences ont été faites en double exemplaire (n = 2).
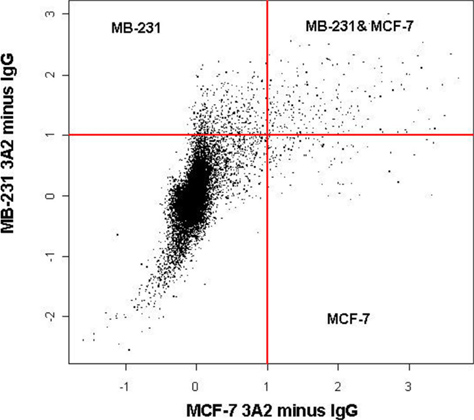
Figure 2. HuR RIP-CHIP identifie différents profils génétiques dans ER + et les cellules cancéreuses du sein ER-.Immunoprécipitations ont été réalisées à partir de Hur MB-231 ou MCF-7 lysats cellulaires en utilisant un anticorps IgG1 et HuR contrôle isotypique hybridé à Illumina tableaux Sentrix (47.000 gènes). Les signaux de commande ont été soustraites. Les résultats représentent les données cumulatives de 12 tableaux différents. Des expériences ont été réalisées en triple exemplaire (n = 3) pour chaque lignée cellulaire avec des contrôles correspondants. Les échelles sont log2.
Discussion
En raison de la nature de cette expérience, l'optimisation et l'expérience seront les seuls moyens garantis pour réussir à acquérir les résultats escomptés. Dans de nombreuses étapes de cette procédure, la température et un traitement efficace des réactifs et les produits sont d'une importance capitale. Une bonne planification et l'exécution de la technique aidera à assurer que l'expérience a été réalisée dans un délai approprié aux températures optimales recommandées. Un problème majeur avec les expériences d'isolement d'ARN est la sensibilité des ARN à la dégradation par les RNases. Tous les réactifs doivent être RNase et stocké ou utilisé dans des récipients sans RNase. Il s'agit d'une étape essentielle pour assurer l'intégrité de votre échantillon d'ARNm. Même lorsque l'expérience est réalisée correctement, cependant, le résultat souhaité ne peut être atteint en raison de la nature de l'interaction entre le RBP et ses ARNm cibles.
Un problème potentiel est faible, voire avoir aucun signal de l'ARN isolé par RIP-Chip.Bien qu'il y ait peut être un signal à partir d'ARN total, cela peut être le résultat d'une protéine de liaison insuffisante étant tiré vers le bas par les billes. La première étape de dépannage consiste à confirmer que le lysat cellulaire a utilisé l'expression adéquate de la RBP spécifique. Lors de la confirmation, la protéine peut être isolée après le dernier lavage NT2 et remises en suspension dans du tampon de Laemmli ou un autre tampon approprié de dénaturation et on a chauffé à 95 ° C pendant 5 min. Analyse par Western blot peut être utilisé sur ces échantillons en coordination avec lysat d'entrée ainsi que les contrôles négatifs pour assurer suffisamment de tirer vers le bas de la protéine associée.
En outre, parce que la lyse de la cellule est nécessaire pour accéder à ces composants, le risque d'interactions anormales ou indésirables entre les protéines normalement séparés et de l'ARNm peut être mis en place. Ces interactions pourraient se lier et "absorber" vos ARNm cibles ou des protéines de liaison à travers des interactions non spécifiques. De plus, les protéines de ces co variantnditions peuvent se replier en de multiples variations et leurs motifs de liaison peut devenir inaccessible à leurs ARNm cibles, empêchant leurs interactions. Ces deux renforcer l'importance de travailler de manière efficace ainsi que l'utilisation des températures optimales énumérées à limiter ces interactions indésirables. De plus, l'optimisation des conditions de lavage pour chaque protéine cible spécifique sera critique pour optimiser la pureté de l'interaction. Les conditions de lavage plus strictes peuvent être nécessaires. Par exemple, le tampon de lavage peuvent être complétés avec du SDS ou d'une quantité appropriée d'urée pour réduire les interactions non spécifiques et de fond dans votre signal. Ce sera totalement dépendante de RBP cible de l'expérimentateur, ainsi que l'ARNm cible dans leurs propres conditions physiologiques. Certaines conditions ne conviennent pas à certains outils d'analyse d'ARNm, qui doivent être notées dans la préparation des échantillons.
Enfin, bien que RIP est couronnée de succès dans l'enrichissement de l'ARNRBP-interactions, un problème bien connu avec RIP (CHIP) méthode est l'incapacité à identifier les domaines spécifiques de liaison de la RBP sur les ARNm cibles transitoires. Plusieurs techniques de réticulation peut être utilisé, suivi par RIP pour isoler les cibles de séquence uniques, mais l'utilisation des ondes courtes UV tend à conduire à des dégâts d'acide nucléique. Une nouvelle méthode connue sous le nom de PAR-CLIP, ou la réticulation photoactivable ribonucléoside et immuoprecipitation, emploie longueur d'onde UV pour intégrer thiouridine en ARN naissants permettant l'identification des sites de liaison spécifiques d'interactions ARN à la fois stables et transitoires.
Dans l'ensemble, RIP-Chip a été établi comme un excellent outil pour isoler et d'étudier les interactions entre les protéines liant l'ARN et leurs cibles ARNm par notre groupe ainsi que de nombreux autres groupes de recherche. Bien que de nature délicate et la pratique, l'exécution correcte de cette procédure donnera l'isolement de ces complexes RNP, qui, jusqu'à récemment, ont été inaccessible de découverte et d'analyse.
Déclarations de divulgation
Les auteurs déclarent n'avoir aucun conflit d'intérêts.
Remerciements
Department of Defense (Prix Idée W81XWH-07-0406) - Pour Ulus Atasoy
NIH RO1 A1080870 - Pour Ulus Atasoy
NIH R21 A1079341 - Pour Ulus Atasoy
Université de Missouri fonds institutionnels - Pour Ulus Atasoy
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| 1 M de dithiothréitol | Pêcheur | BP172-5 | TNT |
| DNase 1 | Ambion | 2235 | RNase |
| Acide éthylènediamine Tetraccetic | Pêcheur | BP118-500 | EDTA |
| Le glycogène | Ambion | 9516 | |
| HEPES 1 | Sigma | H3375-100G | pH 7,0 |
| Igepal Nonidet P-40 | USB | 78641 | NP40 |
| 1 M KCl | Pêcheur | BP366-500 | |
| 1 M de MgCl 2 | Pêcheur | BP214-500 | |
| NT2 tampon | * Voir ci-dessous | ||
| Tampon de lyse polysome | * Voir ci-dessous | ||
| Comprimés inhibiteurs de la protéase cocktail | Roche | 11873580001 | |
| Protéines Perles A Sepharose | Sigma | P3391 | |
| Protéinase K | Pêcheur | BP1700-100 | |
| RNase Out inhibiteur de RNase | Invitrogen | 10777-019 | 40 U / ul |
| 1 M de NaCl | Pêcheur | BP358-212 | |
| Dodécyl sulfate de sodium | Pêcheur | BP166-500 | SDS |
| 1 M de Tris-HCl | Pêcheur | BP153-500 | pH 7,4 |
| Trizol | Invitrogen | 15596-026 | |
| Complexes ribonucléoside vanadyle | Nouvelle-Angleterre Labs | S1402S | VRC |
| Préparation en vue de réactif |
| Préparer les réactifs dans RNase / DNase, traitée au DEPC verrerie |
| Tampon de lyse polysome |
| 100 mM KCl |
| MgCl2 5 mM |
| HEPES 10 mM (pH 7,0) |
| 0,5% NP40 |
| 1 mM DTT |
| 100 unités / ml de RNase Out |
| 400 VRC uM |
| Tablette cocktail inhibiteur de la protéase |
| 5 ml de tampon de lyse polysome |
| Ajouter 50 ul d'HEPES 1 M (pH 7,0) |
| 500 pi de 1 M KCL |
| 25 pl de 1 M MgCl 2 |
| 25 ul de NP40 |
| 4,7 ml de RNase DNase H 2 O |
| 50 pl de 1 M DTT |
| 12,5 ul de 100 U / ml de RNase Out |
| 200 ul de cocktail d'inhibiteurs de protéase (dissous selon le fabricant) |
| 10 pl 200 mM VRC (au moment de l'utilisation) |
| NT2 tampon |
| 50 mM Tris-HCl (pH 7,4) |
| NaCl 150 mM |
| MgCl 2 1 mM |
| 0,05% de NP40 |
| 1 L de tampon NT2 |
| 50 ml de Tris (pH 7,4) |
| 30 ml de NaCl 5 M |
| 1 ml 1 M MgCl 2 |
| 500 ul NP40 |
| 820 ml de RNase DNase H 2 O |
Références
- Calaluce, R., Gubin, M. M., Davis, J. W., Magee, J. D., Chen, J., Kuwano, Y., Gorospe, M., Atasoy, U. The RNA binding protein HuR differentially regulates unique subsets of mRNAs in estrogen receptor negative and estrogen receptor positive breast cancer. BMC Cancer. 10, 126-140 (2010).
- Gubin, M. M., Calaluce, R., Davis, J. W., Magee, J. D., Strouse, C. S., Shaw, D. P., Ma, L., Brown, A., Hoffman, T., Rold, T. L., Ulus Atasoy, U. Overexpression of the RNA binding protein HuR impairs tumor growth in triple negative breast cancer associated with deficient angiogenesis. Cell Cycle. 9, 3337-3346 (2010).
- Keene, J. D., Komisarow, J. M., Friedersdorf, M. B. RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts. Nature Protocols. 1, 302-307 (2006).
- Tenenbaum, S. A., Lager, P. J., Carson, C. C., Keene, J. D. Ribonomics: identifying mRNA subsets in mRNP complexes using antibodies to RNA-binding proteins and genomic arrays. Methods. 26, 191-198 (2002).
- Tenenbaum, S. A., Carson, C. C., Atasoy, U., Keene, J. D. Genome-wide regulatory analysis using en masse nuclear run-ons and ribonomic profiling with autoimmune sera. Gene. 317, 79-87 (2003).
- Tenenbaum, S. A., Carson, C. C., Lager, P. J., Keene, J. D. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Natl. Acad. Sci. U.S.A. 97, 14085-14090 (2000).
- Baroni, T. E., Chittur, S. V., George, A. D., Tennebaum, S. A. Advances in RIP-Chip Analysis. Methods in Molecular Biology. 419, 93-108 (2008).
- de Silanes Lopez, I., Zhan, M., Lal, A., Yang, X., Gorospe, M. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Natl. Acad. Sci. U.S.A. 101, 2987-2992 (2004).
- de Silanes Lopez, I., Lal, A., Gorospe, M. HuR: post-transcriptional paths to malignancy. RNA Biol. 2, 11-13 (2005).
- Grigull, J., Mnaimneh, S., Pootoolal, J., Robinson, M. D., Hughes, T. R. Genome-wide analysis of mRNA stability using transcription inhibitors and microarrays reveals posttranscriptional control of ribosome biogenesis factors. Mol. Cell Biol. 24, 5534-5547 (2004).
- Hieronymus, H., Silver, P. A. Genome-wide analysis of RNA-protein interactions illustrates specificity of the mRNA export machinery. Nat. Genet. 33, 155-161 (2003).
- Hieronymus, H., Yu, M. C., Silver, P. A. Genome-wide mRNA surveillance is coupled to mRNA export. Genes Dev. 18, 2652-2662 (2004).
- Mukherjee, N., Corcoran, D. L., Nusbaum, J. D. Integrative Regulatory Mapping Indicates that the RNA-Binding Protein HuR Couples Pre-mRNA Processing and mRNA Stability. Molecular Cell. 43, 1-13 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon