Method Article
Metodo per l'isolamento e l'identificazione di mRNA, microRNA e componenti proteiche dei complessi Ribonucleoproteina da estratti cellulari con RIP-Chip
In questo articolo
Riepilogo
Un passo dal protocollo passo per isolare e identificare complessi RNA associati attraverso RIP-Chip.
Abstract
Come risultato dello sviluppo di alta sequenziamento e analisi microarray efficiente, globale analisi di espressione genica è diventata una forma semplice e facilmente disponibile di raccolta dati. In numerose ricerche e modelli di malattia tuttavia, i livelli allo stato stazionario di mRNA target gene non sempre direttamente correlata con i livelli stabili di proteine di Stato. Regolazione post-trascrizionale del gene è una probabile spiegazione della divergenza tra i due. Spinto dal legame di RNA binding proteins (RBP), regolazione post-trascrizionale riguarda la localizzazione mRNA, la stabilità e la traduzione formando un Ribonucleoproteina (RNP) complesso con mRNA bersaglio. Identificare questi obiettivi sconosciuti de novo da mRNA estratti cellulari nel complesso RNP è fondamentale per la comprensione dei meccanismi e delle funzioni del RBP e la loro conseguente effetto sulla produzione di proteine. Questo protocollo descrive un metodo denominato RNP immunoprecipitazione-microarray (RIP-Chip), che consente l'identificazione di smRNA SPECIFICHE associato nel complesso ribonucleoproteina, alle mutevoli condizioni sperimentali, insieme con la possibilità di ottimizzare ulteriormente un esperimento per il singolo ricercatore. Con questo importante strumento sperimentale, i ricercatori possono esplorare i complessi meccanismi connessi con regolazione post-trascrizionale del gene, nonché le interazioni ribonucleoproteina altri.
Protocollo
Esperimento di preparazione
Prima di iniziare la sperimentazione, è fondamentale disporre di tutti i reagenti, contenitori e utensili RNasi libero. Trattare vetreria con RNasi inibitore (RNaseZAP, Ambion) seguito da risciacquo con acqua trattata con DEPC. Assicurarsi che tutti i reagenti sono confermati come RNasi libero.
1. Preparare mRNP lisato
- Coltivare e raccogliere le cellule del tessuto in crescita esponenziale a produrre tra 2-5 mg di proteina totale per ogni RIP.
- Due piastre di coltura P150 sono in genere sufficienti.
- Per ogni RBP in fase di studio, il numero totale ammonta cellulari e proteine deve essere ottimizzata per massimizzare l'mRNA di destinazione appropriata e le interazioni RBP.
- RIP per qRT-PCR può richiedere meno lisato cellulare (circa 400 mg di proteine totali) a causa della sua amplificazione e metodo di rilevazione soprattutto per RBP abbondanza elevati, come HuR, AUF1, TIAR.
- Cellule pellet tramite centrifugazione a 200 xg per 8-10 min a 4 ° C, quindi lavare tre volte con PBS ghiacciato.
- Dopo il lavaggio finale, picchietti leggermente la parte inferiore del tubo e risospendere il pellet cellulare in volumi uguali di preparato tampone di lisi polisomi (PLB) con inibitori della proteasi e RNasi.
- Si consiglia di misurare il volume esatto di pellet.
- Miscela delicatamente pipetta (non vortex) a spezzare grumi di cellule e incubare lisato in ghiaccio per 5 min.
- Centrifugare a 13.000 xg per 20 min a 4 ° C per cancellare il lisato di detriti.
- Trasferire immediatamente eliminato sopranatante in pre-raffreddata provetta per microcentrifuga.
- Tenere su ghiaccio e conservare a -80 ° C.
- Congelamento immediato completa la lisi adeguata delle cellule e previene il legame indesiderato. Procedere con disgelo campione RIP immediatamente dopo e mantenere i campioni in ghiaccio per evitare la degradazione dell'RNA.
- Questo lisato può essere conservato per un massimo di sei mesi a -80 ° C. Evitare ripetuti cicli di gelo-disgelo in quanto ciò può portare a proteine e / o degradazione mRNA.
- Quantificare la concentrazione proteica nel lisato mediante saggio standard Protein Bradford.
2. Proteina di rivestimento A Perle Sepharose con anticorpi e Wash
- Pre-swell proteina A Sepharose (PAS) in perline overnight NT2 tampone (3-4 volumi) con 5% BSA. Conservare a 4 ° C.
- Conservazione a lungo termine fino a diversi mesi a 4 ° C è possibile quando è integrato con sodio azide allo 0,1%.
- Perline Sepharose proteine devono essere scelti sulla base di isotipo di anticorpo bersaglio RBP. Proteine A, G e A / G tutti hanno obiettivi isotipo specifici e variano in affinità di destinazione. Proteine perline A Sepharose sono stati utilizzati sulla base di specificità isotipo e affinità per gli anticorpi della proteina Hur.
- Prima dell'uso, rimuovere l'eccesso di tampone NT2, cosìche perle finali al rapporto del buffer è di 1:1.
- Con 1,5 ml RNasi-free microprovette, rimuovere 100 pl di PAS liquami e aggiungere 30 mg di anticorpo per ogni reazione IP individuale (ad esempio, specifica RBP e controllo isotipo).
- Aggiungere 100-200 ml di tampone per NT2 anticorpo miscela di sferette.
- Miscela può essere conservata per diverse settimane a 4 ° C quando è integrato con 0,1% di sodio azide.
- Un isotipo-abbinato anticorpo o tutta sieri normali da una stessa specie devono essere utilizzati in parallelo come un controllo anticorpo contro RNA sfondo.
- Aggiungi anticorpo opportuno miscela di sferette ed incubare una notte, cadendo fine sull'estremità a 4 ° C.
- Ottimizzate il titolo anticorpale per proteina specifica in fase di studio (mg 1, 5, 10 o 30 di anticorpo è di solito sufficiente).
- Preparare anticorpi perle rivestite immediatamente prima dell'uso mediante lavaggio con 1 ml dighiacciato NT2 buffer di 5 volte.
- Lavare miscela di sferette per centrifugazione a 13.000 xg per 1-2 minuti a 4 ° C.
- Rimuovere con attenzione la quantità massima di supernantant con pipetta mano o aspiratore, ma stare attenti a non interrompere pellet.
- Lavaggio aiuta a rimuovere l'anticorpo non legato e contaminanti RNasi da miscela di anticorpi.
- Dopo l'ultimo lavaggio è stato completato, risospendere le sfere in 700 pl di tampone ghiacciato NT2 seguita da trattamento con vari inibitori della RNasi per proteggere gli mRNA bersaglio, di cui 10 ml di RNasi a 40 U / pl, 10 pl di 100 mM DTT e 15 microlitri EDTA (15 mM). Portare il volume a 1000 ml con tampone NT2.
- Complessi ribonucleosidi Vanadil non sono utilizzati a causa dell'effetto inibitorio di EDTA.
3. Immunoprecipitazione e RNA Precipitazione
- Opzionale Passo preclear:
- Per ridurre sfondo, perline e anticorpo di controllo può essere utilizzato per pre-clear lisato. Questo può ridurre il segnale in uscita.
- Questo passaggio può essere necessario ridurre il fondo quando si fa IP seguita da microarray. Non è generalmente necessario per IP seguita da RT-PCR.
- Preclear con 15 microgrammi di controllo isotipo per 30 min / 4 ° C fine oscillando negli fine.
- Aggiungere 50 ml di perline preswollen PAS non rivestiti con anticorpo dal punto 2.1.
- Incubare 30 min / 4 ° C con l'estremità di rotazione rispetto a fine
- Centrifugare a 10.000 xg a 4 ° C. Salva surnatante per IP.
- Aggiungere 100 ml di lisato isolato eliminato (circa 2-5 mg) al mix di anticorpi preparati.
- Diluizione lisato contribuirà a ridurre il fondo e legame non specifico.
- Quantità di input lisato può variare a seconda del metodo di rilevazione e abbondanza di RBP oefficienza r di anticorpi RBP come indicato nel passo 1.1.c.
- (Facoltativo) Miscelare immediatamente tubo delicatamente muovendo più volte, centrifugando breve a 10.000 xg a 4 ° C per perline pellet e rimuovere immediatamente 100 pl di surnatante come rappresentazione totale mRNA input per analisi qPCR utilizzando le normali tecniche di isolamento dell'RNA.
- Questo passo è quello di confermare che l'input lisato RNA è ottimale per IP e deve essere eseguita solo come un passo di controllo o come un passo di risoluzione dei problemi in seguito PIR con scarsi risultati RNA.
- Tube Wrap in parafilm per assicurare la tenuta stretta e incubare a 4 ° C per 2 a 4 ore, cadendo fine su fine.
- Tempi di incubazione dovrebbe essere ottimizzato basato sull'abbondanza bersaglio e deve essere minimizzato per evitare complesso riarrangiamento o degradazione. Per alcuni RBPs brevi incubazioni possono essere più ottimale.
- Perline pellet a 5.000 XGFo 5 min a 4 ° C il surnatante e potenziale per analisi mediante Western blot. Aliquote Conservare a -80 ° C.
- Aliquote di supernatante con elevate quantità di proteine bersaglio residua può indicare un guasto della proteina per essere precipitato da perline sefarosio.
- Come descritto in precedenza, lavare perline 5 volte con 1 ml di tampone ghiacciato e NT2 centrifugazione (5000 xg per 5 minuti a 4 ° C), quindi rimuovere il surnatante con pipettatore mano o un aspiratore.
- Metodi di lavaggio più rigorose possono essere utilizzati per ridurre sfondo completandolo NT2 tampone con sodio desossicolato, urea o SDS.
- Tenere campioni in ghiaccio per quanto possibile e lavorare rapidamente per minimizzare la degradazione di mRNA bersaglio.
- Opzionale: Salva una piccola aliquota (10%) di miscela di sferette o eseguire un campione supplementare in parallelo per il controllo della efficienza IP proteina bersaglio mediante analisi Western blot.
4. DNasi e proteinasi K Trattamenti
- Dopo il lavaggio, risospendere le perline con 100 pl di tampone NT2 supplementato con 5 ml di RNase-free DNase 1 (2 U / pl).
- Mantenere a 37 ° C per 5-10 min. Aggiungere 1 ml di tampone NT2 e centrifugare a 5000 xg per 1 minuto a temperatura ambiente.
- Una piccola (~ 10%) aliquota di perle possono essere utilizzati per verificare l'efficienza IP mediante SDS-PAGE.
- Risospendere PAS pellet in 100 microlitri tampone NT2, 5 pl proteinasi K (10 mg / ml), e 1 pl SDS 10%.
- Incubare la miscela risospeso tallone per 30 min a 55 ° C in un bagno d'acqua, delicatamente sfogliando ogni 10 min.
- Proteinasi K sarà di aiuto nel rilascio dei componenti RNP.
- Perline pellet a 5000 xg per 5 minuti a temperatura ambiente (RT) e raccogliere il surnatante (~ 100 pl).
- Aggiungere 200 microlitri NT2 buffer perline, centrifugheFuge 5000 xg per 2 minuti a temperatura ambiente, raccogliere il supernatante (~ 200 pl), e perline rigetti.
- Combinare supernatanti (100 microlitri e 200 microlitri) e aggiungere strato inferiore 300 ul di acido fenolo-CHCl 3.
- Vortice, 1 min RT (o 37 ° C in shaker) e centrifugare a 16.000 xg a temperatura ambiente per 1 min.
- Raccogliere 250 ml di strato superiore (NON interrompere l'interfaccia), aggiungere 25 microlitri di acetato di sodio a pH 5,2, 625 microlitri ETOH 100% e 5 glycoblue microlitri, e mescolare bene.
- Memorizzare una notte a -20 ° C.
- Per garantire il corretto recupero di RNA, l'aggiunta di glycoblue aumenterà la visibilità del pellet di RNA agendo come una molecola di trasporto.
- Il giorno seguente, i tubi Mescolare capovolgendo 3-5 volte, girare a 12.000 xg a 4 ° C per 30 minuti e scartare il surnatante.
- Aggiungere 1 ml di 70% ETOH per il pellet blu e mescolare per inversione o vortex.
- Centrifugare 12.000 xg a 4 ° C per 2 min.
- Discard surnatante e centrifugare 12.000 xg per 1 min a 4 ° C.
- Rimuovere qualsiasi residuo di ETOH 70% con una pipetta e pellet aria secca a temperatura ambiente per 5 min.
- Risospendere in 20-40 microlitri RNasi / DNasi-free acqua o un altro volume appropriato in base alle esigenze.
- Il campione è ora pronto per ulteriori applicazioni a valle, come qRT-PCR o microarray.
- Spettrofotometria Nanodrop può essere utilizzato per valutare la concentrazione del campione, tuttavia, con campioni preziosi, può essere utile per evitare campioni nanodropping come può essere uno spreco e relativamente imprecisa. Si consiglia di campioni per la normalizzazione trascritti del gene housekeeping per valutare la purezza e l'efficienza IP per campioni di rendimento preziosi o bassa.
5. Risultati rappresentativi
Se la procedura è ottimizzato ed eseguito correttamente, il immunoprecipitazione dovrebbe produrre un arricchimento significativo di mRNA target. Tipicamente, A seconda della sua destinazione e RBP mRNA (s), vediamo arricchimento di circa 10 - a 50 volte quando valutata mediante RT-PCR. Molti obiettivi RBP può essere scoperto in massa usando l'analisi microarray. Tuttavia, questo metodo è più sensibile alla degradazione rispetto qRT-PCR. A seconda della RBP, il numero di obiettivi e l'efficienza della reazione, microarray può rivelare centinaia di nuovi bersagli, o può scoprire solo pochi, se presente. Ad esempio, uno dei meglio caratterizzato RNA binding proteins HuR, post-trascrizionalmente regola l'espressione e la traduzione di molti geni importanti fisiologiche 1, 2. Isolamento del HuR-ribonucleo-complesso tramite RIP-Chip in linee cellulari di tumore mammario, per esempio, ha rivelato arricchimento di alcuni importanti obiettivi Hur note, comprese β-actina mediante RT-PCR, come mostrato in Figura 1. Nelle linee di cellule tumorali sia β-actina è arricchito 12 - a 15 volte. In genere, quando eseguita correttamente vediamo un significativo Enrichment di β-actina in una varietà di linee cellulari. Tuttavia, se il RIP non rivela alcuna arricchimento significativo per la β-actina questo indica un problema con il RIP e la procedura può essere necessario ripetere. Inoltre, l'analisi di campioni microarray immunoprecipitate da queste linee cellulari di espressione rivelato sottoinsiemi distinti di obiettivi in Hur recettore degli estrogeni (ER diversa) positive cellule MCF-7 cancro rispetto negativi per ER-MB 231 linee cellulari di tumore mammario come evidenziato in Figura 2 1. Tali obiettivi rientrano in diverse categorie: obiettivi Hur conosciuti e sconosciuti che erano o associati o non associati con il cancro. Ad esempio, CALM2 e CD9 sono entrambi geni del cancro che non sono stati precedentemente identificati come obiettivi Hur. Utilizzando il microarray e confermando con RT-PCR, e CALM2 CD9 sono risultati 5 - a 180 volte arricchito in pellet HuR indica una interazione tra la proteina prominente HuR e questi geni bersaglio.

Figura 1. Immunoprecipitazione e RIP in MB-231 (ER-) e MCF-7 (ER +), le cellule di cancro al seno. Immunoprecipitazione sono stati eseguiti da MB-231 o MCF-7 lisati cellulari utilizzando anticorpi anti-Hur anticorpo monoclonale (3A2) e di controllo isotipo IgG1. A. IP occidentale HuR rivelato banda dimensioni previste, come rilevato da 3A2. Pannello a destra rivela quantità di HuR in ingresso lisati utilizzato da entrambe le linee cellulari, con β-tubulina come controllo di caricamento. B. Verifica da RT-PCR ha mostrato quindici e undici volte arricchimenti di β-actina, un bersaglio HuR noto, nei PI 3A2 da MB-231 e MCF-7, rispettivamente. Tutti i valori sono stati normalizzati per ΔΔCT GAPDH. Gli esperimenti sono stati eseguiti in duplicato (n = 2).
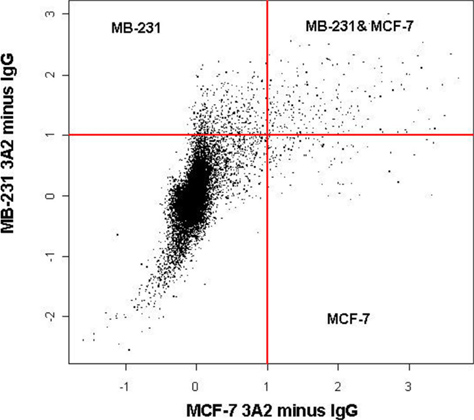
Figura 2. HuR RIP-CHIP identifica distinti profili genetici in ER + e ER-cellule di cancro al seno.Immunoprecipitazione Hur sono state eseguite da MB-231 o MCF-7 lisati cellulari con anticorpi IgG1 e HuR controllo isotipo ibridato Illumina array Sentrix (47.000 geni). Segnali di controllo sono stati sottratti. I risultati rappresentano dati cumulativi da 12 matrici differenti. Esperimenti sono stati fatti in triplicato (n = 3) per ciascuna linea cellulare con controlli corrispondenti. Le scale sono log2.
Discussione
A causa della natura di questa ottimizzazione esperimento, e l'esperienza saranno gli unici modi garantiti per acquisire con successo i risultati attesi. In molti passi di questo procedimento, la temperatura e la gestione efficiente di reagenti e prodotti sono criticamente importanti. Una corretta pianificazione e l'esecuzione della tecnica aiuterà ad assicurare che l'esperimento è stato eseguito in un periodo di tempo adeguato alle temperature ottimali consigliate. Una questione importante con gli esperimenti di isolamento di RNA è la sensibilità di RNA alla degradazione da RNasi. Tutti i reagenti devono essere RNasi libero e immagazzinati o utilizzati in contenitori privi di RNasi. Questo è un passo fondamentale per garantire l'integrità del campione di mRNA. Anche quando l'esperimento viene eseguito correttamente, tuttavia, il risultato desiderato non può essere raggiunto a causa della natura della interazione tra l'RBP e suoi mRNA bersaglio.
Un potenziale problema è avere basso o addirittura nessun segnale dal RNA isolato da RIP-Chip.Anche se ci può essere il segnale di RNA totale, questo può essere il risultato di insufficiente legame proteico essere tirato giù dalle perle. Il primo passo di risoluzione dei problemi è confermare che il lisato cellulare utilizzato ha espressione adeguata del RBP specifico. Alla conferma, le proteine possono essere isolati dopo il lavaggio finale NT2 e risospese in tampone Laemmli o altro tampone appropriato denaturazione e riscaldata a 95 ° C per 5 min. Analisi Western blot può essere utilizzato su questi campioni in coordinamento con lisato ingresso nonché controlli negativi per garantire sufficiente tirare verso il basso di proteine associate.
Inoltre, poiché la lisi cellulare è necessario per accedere a questi componenti, il potenziale di interazioni anormali e indesiderabili tra proteine normalmente separati e mRNA possono essere introdotti. Queste interazioni potrebbero legare e "assorbire" le mRNA bersaglio o proteine di legame attraverso interazioni non specifiche. Inoltre, in queste proteine co variandonditions può piegare in più varianti e dei loro motivi di legame può diventare inaccessibile ai loro mRNA bersaglio, impedendo loro interazioni. Entrambi rafforzare l'importanza di lavorare efficacemente, nonché utilizzando le temperature ottimali elencati limitare queste interazioni indesiderate. Inoltre, l'ottimizzazione delle condizioni di lavaggio per ogni proteina bersaglio specifico sarà fondamentale per massimizzare la purezza dell'interazione. Condizioni di lavaggio più severi può essere necessaria. Per esempio, il tampone di lavaggio può essere integrato con SDS o una quantità appropriata di urea per ridurre le interazioni non specifiche e sfondo nel segnale di uscita. Questo sarà completamente dipendente RBP bersaglio dello sperimentatore nonché l'mRNA bersaglio nelle loro condizioni fisiologiche uniche. Alcune condizioni non sarà adatto a determinati strumenti di analisi di mRNA, che devono essere conosciute per la preparazione dei campioni.
Infine, anche se è riuscito a RIP l'arricchimento di RNA-RBP interazioni, un problema ben noto con RIP (CHIP) metodo è l'incapacità di identificare i domini di legame specifico del RBP sui bersagli mRNA transitori. Diverse tecniche di reticolazione può essere utilizzata seguita da RIP isolare obiettivi sequenza univoci, tuttavia, l'uso di onde corte UV tende a portare a danni acido nucleico. Un nuovo metodo noto come PAR-CLIP, o reticolazione ribonucleoside fotoattivabile e immuoprecipitation, impiega onda lunga UV per incorporare thiouridine in RNA nascenti che consentono l'identificazione di siti di legame unici sia da stabili e transitori interazioni RNA.
Nel complesso, RIP-Chip è stato stabilito come un ottimo strumento utilizzato per isolare e studiare le interazioni tra proteine legano l'RNA e ai loro obiettivi di mRNA dal nostro gruppo, così come molti altri gruppi di ricerca. Anche se di natura sensibile e pratica, la corretta esecuzione di questa procedura produrrà l'isolamento di questi complessi RNP, che fino a poco tempo fa, sono stati inaccessible per la scoperta e l'analisi.
Divulgazioni
Gli autori dichiarano di non avere interessi in gioco.
Riconoscimenti
Dipartimento della Difesa (Premio Idea W81XWH-07-0406) - Per Ulus Atasoy
NIH RO1 A1080870 - Per Ulus Atasoy
NIH R21 A1079341 - Per Ulus Atasoy
University of Missouri fondi istituzionali - Per Ulus Atasoy
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
| 1 M ditiotreitolo | Pescatore | BP172-5 | DTT |
| DNasi 1 | Ambion | 2235 | RNasi libero |
| Etilendiammina Acid Tetraccetic | Pescatore | BP118-500 | EDTA |
| Glicogeno | Ambion | 9516 | |
| 1 HEPES | Sigma | H3375-100G | pH 7,0 |
| Igepal Nonidet P-40 | USB | 78641 | NP40 |
| 1 M KCl | Pescatore | BP366-500 | |
| 1 M MgCl 2 | Pescatore | BP214-500 | |
| NT2 Buffer | * Vedere di seguito | ||
| Lysis Buffer polisomi | * Vedere di seguito | ||
| Inibitori della proteasi cocktail compresse | Roche | 11873580001 | |
| Proteina A Sepharose Beads | Sigma | P3391 | |
| Proteinasi K | Pescatore | BP1700-100 | |
| RNasi Out inibitore della RNasi | Invitrogen | 10777-019 | 40 U / pl |
| 1 M NaCl | Pescatore | BP358-212 | |
| Sodio dodecil solfato | Pescatore | BP166-500 | SDS |
| 1 M Tris-HCl | Pescatore | BP153-500 | pH 7,4 |
| Trizol | Invitrogen | 15596-026 | |
| Complessi ribonucleosidi Vanadil | New England Labs | S1402S | VRC |
| Reagente Workup |
| Preparare i reagenti in RNasi / DNasi-free, trattata con DEPC vetro |
| Lisi buffer polisomi |
| 100 mM KCl |
| 5 mM MgCl 2 |
| HEPES 10 mM (pH 7,0) |
| 0,5% NP40 |
| 1 mM DTT |
| 100 unità / ml RNasi Out |
| 400 pM VRC |
| Inibitore della proteasi tablet cocktail |
| 5 ml di tampone di lisi polisomi |
| Aggiungere 50 ml di 1 M HEPES (pH 7,0) |
| 500 microlitri di 1 M KCl |
| 25 ml di 1 M MgCl 2 |
| 25 pl di NP40 |
| 4,7 ml DNasi RNasi-free H 2 O |
| 50 ml di 1 M DTT |
| 12,5 microlitri di 100 U / ml RNasi Out |
| 200 pl cocktail di inibitori delle proteasi (dissolto secondo costruttore) |
| 10 pl 200 mM VRC (al momento dell'uso) |
| NT2 Buffer |
| 50 mM Tris-HCl (pH 7,4) |
| 150 mM NaCl |
| 1 mM MgCl 2 |
| 0,05% NP40 |
| 1 L di tampone NT2 |
| 50 ml di Tris (pH 7,4) |
| 30 ml di NaCl 5 M |
| 1 ml 1 M MgCl 2 |
| 500 NP40 pl |
| 820 ml DNasi RNasi-free H 2 O |
Riferimenti
- Calaluce, R., Gubin, M. M., Davis, J. W., Magee, J. D., Chen, J., Kuwano, Y., Gorospe, M., Atasoy, U. The RNA binding protein HuR differentially regulates unique subsets of mRNAs in estrogen receptor negative and estrogen receptor positive breast cancer. BMC Cancer. 10, 126-140 (2010).
- Gubin, M. M., Calaluce, R., Davis, J. W., Magee, J. D., Strouse, C. S., Shaw, D. P., Ma, L., Brown, A., Hoffman, T., Rold, T. L., Ulus Atasoy, U. Overexpression of the RNA binding protein HuR impairs tumor growth in triple negative breast cancer associated with deficient angiogenesis. Cell Cycle. 9, 3337-3346 (2010).
- Keene, J. D., Komisarow, J. M., Friedersdorf, M. B. RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts. Nature Protocols. 1, 302-307 (2006).
- Tenenbaum, S. A., Lager, P. J., Carson, C. C., Keene, J. D. Ribonomics: identifying mRNA subsets in mRNP complexes using antibodies to RNA-binding proteins and genomic arrays. Methods. 26, 191-198 (2002).
- Tenenbaum, S. A., Carson, C. C., Atasoy, U., Keene, J. D. Genome-wide regulatory analysis using en masse nuclear run-ons and ribonomic profiling with autoimmune sera. Gene. 317, 79-87 (2003).
- Tenenbaum, S. A., Carson, C. C., Lager, P. J., Keene, J. D. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Natl. Acad. Sci. U.S.A. 97, 14085-14090 (2000).
- Baroni, T. E., Chittur, S. V., George, A. D., Tennebaum, S. A. Advances in RIP-Chip Analysis. Methods in Molecular Biology. 419, 93-108 (2008).
- de Silanes Lopez, I., Zhan, M., Lal, A., Yang, X., Gorospe, M. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Natl. Acad. Sci. U.S.A. 101, 2987-2992 (2004).
- de Silanes Lopez, I., Lal, A., Gorospe, M. HuR: post-transcriptional paths to malignancy. RNA Biol. 2, 11-13 (2005).
- Grigull, J., Mnaimneh, S., Pootoolal, J., Robinson, M. D., Hughes, T. R. Genome-wide analysis of mRNA stability using transcription inhibitors and microarrays reveals posttranscriptional control of ribosome biogenesis factors. Mol. Cell Biol. 24, 5534-5547 (2004).
- Hieronymus, H., Silver, P. A. Genome-wide analysis of RNA-protein interactions illustrates specificity of the mRNA export machinery. Nat. Genet. 33, 155-161 (2003).
- Hieronymus, H., Yu, M. C., Silver, P. A. Genome-wide mRNA surveillance is coupled to mRNA export. Genes Dev. 18, 2652-2662 (2004).
- Mukherjee, N., Corcoran, D. L., Nusbaum, J. D. Integrative Regulatory Mapping Indicates that the RNA-Binding Protein HuR Couples Pre-mRNA Processing and mRNA Stability. Molecular Cell. 43, 1-13 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon