Method Article
Pintura de cromosomas 2D y 3D en mosquitos de la malaria
En este artículo
Resumen
La pintura del cromosoma es un método útil para estudiar la organización del núcleo de la célula y la evolución del karyotype. Aquí, demostramos un acercamiento para aislar y para amplificar regiones específicas del interés de los cromosomas solos del politeno que se utilizan posteriormente para el hibridación in situ fluorescente de dos y tridimensional (PESCADO).
Resumen
El hibridación in situ fluorescente (PESCADO) de las puntas de prueba enteras del cromosoma del brazo es una técnica robusta para trazar regiones genomic del interés, detectar cambios cromosómicos, y estudiar la organización tridimensional (3D) de cromosomas en el núcleo de la célula. La llegada de la microdisección por captura láser (LCM) y la amplificación del genoma completo (WGA) permite obtener grandes cantidades de ADN de células individuales. La mayor sensibilidad de los kits wga nos llevó a desarrollar pinturas cromosómicas y a utilizarlas para explorar la organización cromosómica y la evolución en organismos no modelo. Aquí, presentamos un método simple para aislar y amplificar los segmentos eucromáticos de los brazos de cromosomas de politeno único de las células de la enfermera ovárica del mosquito africano de la malaria Anopheles gambiae. Este procedimiento proporciona una plataforma eficiente para la obtención de pinturas cromosómicas, al tiempo que reduce el riesgo general de introducir ADN extraño en la muestra. El uso de WGA permite varias rondas de reamplización, lo que resulta en altas cantidades de ADN que se pueden utilizar para múltiples experimentos, incluidos 2D y 3D FISH. Demostramos que las pinturas desarrolladas del cromosoma se pueden utilizar con éxito para establecer la correspondencia entre las porciones eucromáticas de politeno y los brazos mitotic del cromosoma en Gambiae del An. En general, la unión de LCM y WGA de un solo cromosoma proporciona una herramienta eficiente para crear cantidades significativas de ADN diana para futuros estudios citogenéticos y genómicos.
Introducción
La pintura cromosómica es una técnica útil para estudiar la evolución de los cariotipos 1-5 y para visualizar anomalías citogenéticas mediante hibridación del ADN cromosómico marcado fluorescentemente 1,6-8. Este método también se aplica al estudio de la dinámica 3D de los territorios cromosómicos en el desarrollo 9 y la patología 10. Las pinturas cromosómicas etiquetadas diferencialmente se utilizan para la visualización de los cromosomas mitóticos individuales11, 12, meióticos13, 14,o interfase no politeno 15,16 y politeno 9,11,17. Un protocolo simple y robusto para obtener pinturas cromosómicas sería muy útil para expandir esta técnica a organismos no modelo, como los mosquitos. El complejo Anopheles gambiae es un grupo de siete mosquitos morfológicamente indistinguibles que varían en comportamiento y adaptación, incluyendo la capacidad de transmitir la malaria. Estos mosquitos sirven como un excelente modelo para comprender mejor la evolución de especies estrechamente relacionadas que difieren mucho en su capacidad vectorial. La mayoría de los estudios citogenéticos en mosquitos de la malaria se han realizado utilizando cromosomas bien desarrollados y altamente politenizados (revisados en 18,19). Los patrones de bandas legibles de los cromosomas politenos permitieron a los investigadores demostrar la asociación entre las inversiones polimórficas y las adaptaciones ecológicas en Anopheles gambiae 20. También, 21 tejido-específicos y 22 características especie-específicas de la organización del cromosoma del politenio 3D se han caracterizado en el complejo de los macullipennis del An. Sin embargo, el estudio de los cromosomas mitóticos podría proporcionar información adicional importante. Por ejemplo, un mayor nivel de polimorfismo de heterocromatina observado en cromosomas mitóticos se ha correlacionado con una menor actividad de apareamiento y fertilidad en An. gambiae 23. La correspondencia entre los segmentos eucromáticos del politeno y los brazos mitotic del cromosoma podía ser difícil de establecer solamente comparando sus longitudes relativas. Esto se debe a que la heterocromatina constituye una porción significativa de los cromosomas mitóticos, pero está subrepresentada en los cromosomas politeno 24. La disponibilidad de pinturas cromosómicas para los mosquitos de la malaria permitiría a los investigadores ampliar significativamente los estudios citogenéticos mediante la inclusión de especies adicionales, al tiempo que reduciría sustancialmente el costo y el tiempo en los análisis de la evolución cariotípica y la dinámica 3D de los cromosomas en este grupo de insectos epidemiológicamente importantes.
Con el fin de obtener grandes extensiones de cromosomas, la microdisección se ha convertido en una técnica integral para manipular y aislar regiones específicas de interés del complemento cromosómico. Cuando se combina con la amplificación del genoma completo (WGA), la microdisección da lugar a potentes aplicaciones aguas abajo, incluyendo FISH 11,12 y la secuenciación del genoma de próxima generación 25-27. Anteriormente, la técnica requería el uso de agujas de microdisección especializadas que debían ser controladas manualmente por un usuario experimentado 28. El avance de la microdisección de captura láser (LCM) ha dado como resultado una herramienta simplificada más adecuada para aislar células individuales 29,30 o cromosomas individuales 31-33 con un menor riesgo de contaminación. Este enfoque permite al usuario estudiar las heterogeneidades genéticas y anomalías cromosómicas que ocurren en células individuales, en lugar de un paisaje de consenso que resulta de la agrupación de múltiples células entre sí 34-36. Se han utilizado múltiples métodos para amplificar el ADN producido a partir de la microdisección. Dop-PCR, una técnica útil para la amplificación de secuencias altamente repetitivas, se ha utilizado para amplificar cromosomas microdissecados de especies como el saltamontes 37,la anguila espinosa 38,y la tilapia del Nilo 39. Más recientemente, el kit de una sola célula GenomePlex WGA4 basado en PCR y el kit de una sola célula Repli-G basado en amplificación de desplazamiento múltiple (MDA) se han convertido en herramientas valiosas para experimentos que implican el análisis genético de células humanas individuales, así como cromosomas40-42,incluidos los sistemas de cromosoma B en saltamontes 37 y langostas 43. Estos dos kits tienen ventajas y desventajas, pero se ha demostrado su aparente superioridad sobre otros sistemas de amplificación disponibles 44.
La cantidad de ADN que se puede obtener de un solo cromosoma o un segmento cromosómico es significativamente menor que la de un núcleo entero. Por lo tanto, la microdisección, la amplificación y el posterior análisis de un solo cromosoma son mucho más desafiantes, especialmente en organismos con genomas pequeños como en Drosophila o Anopheles. Aunque las pinturas se han desarrollado a partir de cromosomas microdissecados individuales de un ser humano 33 y una avispa 45,un experimento exitoso de FISH requirió múltiples (al menos 10-15) cromosomas mitóticos microdissecados de una mosca de la fruta 11. Sin embargo, la capacidad de microdiseccionar y amplificar un solo cromosoma sería importante para (i) reducir la posibilidad de contaminación con material de un cromosoma diferente, (ii) minimizar el número de preparaciones cromosómicas necesarias para la microdisección, (iii) bajar el nucleótido y el polimorfismo estructural de la muestra microdissecada tanto en FISH como en secuenciar aplicaciones posteriores. Los cromosomas politensos que se encuentran en muchas especies de dípteros ofrecen una oportunidad única para adquirir una cantidad inicial mucho mayor de ADN. También proporcionan una resolución y una estructura cromosómica más altas que no se pueden lograr mediante el uso de cromosomas mitóticos. Esta resolución adicional puede ser crítica en la visualización de reordenamientos cromosómicos, estructura de cromatina y segmentos cromosómicos para ser microdissecados 28,46.
Aquí presentamos un procedimiento para aislar eficientemente un segmento eucromático de un solo brazo del cromosoma del politeno, amplificar la DNA, y utilizarlo en usos rio abajo de los PESCADOS en mosquitos de la malaria. En primer lugar, aplicamos LCM para aislar y extraer un solo brazo cromosómico de portaobjetos de membrana especialmente preparados. En segundo lugar, WGA se utiliza para amplificar el ADN del material microdissecado. En tercer lugar, hibridamos el ADN amplificado en experimentos FISH para preparar preparaciones de calabaza politeno 47,metafase y diapositivas cromosómicas interfase 48,así como muestras de montaje entero ovárico 3D. Este procedimiento se ha hecho para pintar con éxito una mayoría de la eucromatina en brazos cromosómicos de An. gambiae.
Protocolo
1) Preparación de diapositivas cromosómicas de politeno para la microdisección de captura láser
- Diseccione las hembras medio-grávidas de Anopheles en la alimentación de la poste-sangre de 25 horas. Fije los ovarios de aproximadamente cinco hembras en 500 μl de solución fresca modificada de Carnoy (100% metanol: ácido acético glacial, 3:1) a temperatura ambiente durante 24 horas. Transferir ovarios a -20 °C para un almacenamiento a largo plazo.
- Prepare la solución de Carnoy (100% etanol: ácido acético glacial, 3:1) y ácido propiónico al 50% justo antes de hacer diapositivas cromosómicas.
- Coloque un par de ovarios en una gota de la solución de Carnoy en un portaobjetos de membrana zeiss 1.0 PET. Dependiendo del tamaño, divida los ovarios en aproximadamente 2-4 secciones con agujas de disección y colóquelos en una gota de ácido propiónico al 50% en portaobjetos limpios bajo un microscopio de disección.
- Separe los folículos y retire el tejido restante con toalla de papel debajo de un estereomicroscopio de disección. Agregue una nueva gota de ácido propiónico al 50% a los folículos y déjenlos reposar durante 3-5 minutos a temperatura ambiente.
- Coloque un cubrebocas siliconado en la parte superior de la gota. Deje reposar la diapositiva durante aproximadamente 1 min.
- Cubra la diapositiva con un material absorbente (el papel de filtro se utiliza para este método), y mientras usa el lado del borrador de un lápiz, aplique una generosa cantidad de presión al cubrebocas tocándolo repetidamente con el borrador.
- Caliente la diapositiva a 60 °C en un sistema de desnaturalización/hibridación de la diapositiva durante 15-20 minutos para ayudar a aplanar los cromosomas del politeno. Coloque los portaobjetos en una cámara húmeda a 4 °C durante la noche para permitir que el ácido aplane aún más los cromosomas.
- Colocar portaobjetos en frío al 50% etanol durante 10 min. Retire suavemente el cubrebocas y reemplácelo en frío al 50% de etanol durante 10 min más.
- Deshidrate los portaobjetos en 70%, 90%, 100% etanol durante 5 min cada uno. Toboganes secos al aire.
- Prepare una solución de solución tampón GURR agregando una sola tableta tampón a 1 L de agua destilada. autoclave.
- Prepare la solución de Giemsa añadiendo 1 ml de solución de tinción de Giemsa a 50 ml de tampón GURR.
- Coloque los portaobjetos secados al aire en la solución de Giemsa durante 10 min y lave tres veces en 1X PBS. Se desliza al aire de nuevo en un clima estéril controlado para evitar la contaminación.
2) Microdisección de captura láser de un solo brazo cromosómico politeno
Esta sección detalla el uso del software PALMRobo, que viene con el sistema de microdisección láser PALM MicroBeam.
- Limpie el microscopio con etanol al 100%. Esterilice guantes y tubos con una luz UV en un reticulculo UV.
- Encienda el microscopio de microdisección láser PALM MicroBeam y encienda el láser. Abra la suite de disección láser, PALMRobo, y configure los ajustes "Power" y "Focus" según sea necesario.
- Búsqueda de brazo de cromosoma politeno de interés.
- Con la herramienta "Lápiz", delinee la región seleccionada.
- Abra la "ventana elementos" desde la barra de menús.
- Seleccione el "Elemento dibujado", asegúrese de que ha seleccionado "Cortar".
- Instale el tubo adhesivo de la tapa en el soporte y colóquelo encima de la corredera, dejando un pequeño espacio <1 mm de tamaño, e inicie el corte con láser.
- Coloque la "selección de catapulta" dentro del sitio de corte, dejando espacio entre el borde y el cromosoma.
- Seleccione "LPC" en la opción desplegable y comience a catapultar.
- Compruebe para asegurarse de que la muestra fue catapultada en la tapa pulsando el icono "Ojo".
3) Purificación del ADN de un solo brazo cromosómico politético microdissecado
Siga las instrucciones del QIAamp DNA Micro Kit para liberar y purificar el ADN recolectado. El paso 3.1 se modificó para acomodar el tubo invertido.
- Añadir 15 μl tampón ATL y 10 μl de proteinasa K al tubo invertido (dentro de la tapa) e incubar a 56 °C durante 3 horas.
- Añadir 25 μl tampón ATL, 50 μl tampón AL y 1 μl de ARN portador; mezclar. Añadir 50 μl 100% EtOH; mezclar.
- Transferir el lisato a la columna QIAamp; centrífuga. Lavar añadiendo 500 μl de tampón AW1; centrífuga. Coloque la columna en el nuevo tubo de recolección, agregue el tampón de 500 μl AW2; centrífuga. Coloque la columna en un tubo nuevo; centrífuga para eliminar el exceso de líquido.
- Coloque la columna en un tubo de microcentrífuga de 1,5 μl y agregue 20 μl de agua para eluir; centrífuga.
- Evaporar el ADN recién eludido hasta un volumen final de 9 μl usando un vacuumfuge.
4) Amplificación del ADN de un solo brazo cromosómico polisecado microdissecado
Dos diversos protocolos fueron utilizados para el WGA de un solo brazo del cromosoma.
- Amplificación del ADN y preparación de la sonda a través de GenomePlex WGA
- Siga el protocolo GenomePlex Single Cell WGA4 Kit para producir el primer lote de ADN amplificado:
- Añadir la solución de proteinasa K recién preparada a la muestra de 9 μl; mezclar. Incubar el ADN a 50 °C durante 1 hora, luego calentar a 99 °C durante 4 min. Manténgase en el hielo.
- Agregue 2 μl 1X Single Cell Library Preparation Buffer y 1 μl de Library Stabilization Solution; mezcla. Calentar la muestra a 95 °C durante 2 min. Enfriar sobre hielo y centrífuga.
- Añadir 1 μl de enzima de preparación de biblioteca; mezcla y centrífuga. Incubar de la siguiente manera:
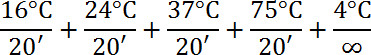
- Añadir 7,5 μl 10X Amplificación Master Mix, 48,5 μl de agua. 5,0 μl WGA ADN polimerasa; mezcla y centrífuga.
- Termociclo de la siguiente manera:
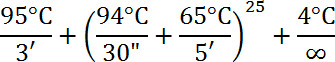
- Purificar el ADN utilizando el kit genomic DNA Clean &Concentrator. El protocolo es el siguiente:
- Añadir 5:1 Tampón de unión al ADN:Muestra de ADN (específicamente para ADN genómico de menos de 2 kb. Si el ADN de la muestra es mayor que 2 kb, utilice una relación 2:1) y transfiera a la columna de espín proporcionada. centrífuga.
- Añadir 200 μl de Tampón de lavado de ADN y centrífuga. Repita el paso de lavado. Luego, agregue 50 μl de agua y elua el ADN en un nuevo tubo de 1,5 ml.
- Vuelva a amplificar el ADN de la muestra utilizando el kit de reamplificación GenomePlex WGA3 de la siguiente manera:
- Agregue 10 μl de ADN al tubo de PCR (el kit recomienda 10 ng total de ADN) con 49.5 μl de agua, 7.5 μl 10x Amplification Master Mix, 3.0 μl 10 mM DNTP mix, y 5.0 μl WGA DNA Polymerase. Mezcla y centrífuga.
- Utilice el siguiente perfil para la reacción:
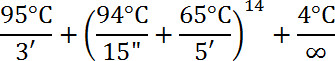
- Almacene el ADN a -20 °C.
- Etiquete el ADN para FISH usando el Kit de Reamplificación GenomePlex WGA3 de la siguiente manera:
- Cree una mezcla maestra a partir del kit de reamplificación GenomePlex WGA3 agregando 10 μl de ADN al tubo de PCR con 49,5 μl de agua, 7,5 μl de mezcla maestra de amplificación 10X, mezcla dNTP de 3,0 μl 1 mM (1 mM de dATP, dCTP, dGTP, 0,3 μl 1 mM dTTP - si se usa dUTP etiquetado), 1 μl de 25 nM etiquetado dUTP , y 5,0 μl wga ADN polimerasa.
- Utilice el siguiente perfil para la reacción:
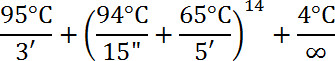
- El etanol precipita la sonda etiquetada añadiendo 1/10 el volumen de reacción final (7,5 μl para la reacción de 75 μl) de acetato de sodio de 3 M pH 5,2 y 2-3 volúmenes de etanol al 100%. Enfríe la muestra de ADN a -80 °C durante al menos 30 min.
- Muestra de centrífuga a 4 °C durante 10 min para crear pellets etiquetados y eliminar el sobrenadante y el pellet seco al aire.
- Cree el búfer de hibridación de la siguiente manera:
0,2 g Sulfato de dextrano
1200 μl Formamida desionizada
580 μl H2O
120 μl 20X SSC - Añadir 40 μl de tampón de hibridación al pellet secado al aire.
- Siga el protocolo GenomePlex Single Cell WGA4 Kit para producir el primer lote de ADN amplificado:
- Amplificación del ADN y preparación de la sonda a través de REPLI-G Single Cell WGA seguido de nick-translation
- Siga el protocolo REPLI-g Single Cell WGA Kit para producir el ADN amplificado:
- Preparar Buffer D2 (3 μl de 1 M DTT + 33 μl Buffer DLB).
- Mezclar 4 μl de material microdissecado purificado con 3 μl buffer D2. Tubo de flick para mezclar. Incubar durante 10 min a 65 °C. Añadir 3 μl de solución Stop; mezclar.
- Agregue 9 μl H2O, 29 μl REPLI-g Reaction Buffer y 2 μl de REPLI-g ADN Polimerasa a la muestra. Incubar a 30 °C durante 8 hr. Inactivar la ADN polimerasa calentando a 65 °C durante 3 min. Almacene el ADN a -20 °C.
- Siga el protocolo de traducción de nick para etiquetar el ADN amplificado REPLI-g:
- Prepare la siguiente mezcla de etiquetado:
1 μg de ADN amplificado
5 μl 10X Tampón de ADN polimerasa
5 μl 10X dNTP
5 μl 1X BSA
1 μl 1 mM etiquetado dNTP
4 μl 1 U/μl DNasa I
1 μl 10 U/μl ADN Polimerasa I
H2O a 50 μl - Incubar a 15 °C durante 2 horas. Añadir 2 μl de EDTA de 0,5 M para detener la reacción. Compruebe el tamaño del fragmento de ADN funcionando con gel.
- Siga los pasos de 4.1.4.3-4.1.4.6 para precipitar y solubilizar el pellet.
- Prepare la siguiente mezcla de etiquetado:
- Siga el protocolo REPLI-g Single Cell WGA Kit para producir el ADN amplificado:
5) PESCADO 2D en preparaciones del calabaza del politeno y mitotic del cromosoma
Consulte los protocolos detallados para FISH sobre preparaciones de calabazas de politenos 47 y diapositivas cromosómicas mitóticas 48. Aquí proporcionamos protocolos breves.
- FISH en preparaciones de calabaza cromosómica politeno
- Sumerja las diapositivas en 1X PBS durante 20 min en RT. Fijar diapositivas en 4% Paraformaldehído en 1X PBS durante 1 min.
- Deshidrates slides through ethanol washes: 50%, 70%, 90%, 100% for 5 min each at RT. Air-dry slides.
- Preguerra la sonda en tampón de hibridación a 37 °C. Agregue 10-20 μl de sonda para deslizarse. Cubierta con 22 X 22 mm cubrebocas. Presione las burbujas de aire con una punta de pipeta.
- Desnaturalizar los cromosomas y sondear a 90 °C durante 10 min. Selle los bordes de la cubierta con cemento de goma. Transferir el portaobjetos a la cámara húmeda e incubar a 39 °C durante la noche.
- Lave los portaobjetos con 1X SSC a 39 °C durante 20 min. Lave las guías con 1X SSC en RT durante 20 min. Enjuague los portaobjetos en 1X PBS, luego agregue Prolong anti-fade con DAPI. Cubra con cubrebocas y guárdelo en la caja deslizante a 4 °C durante al menos una hora antes de la visualización.
- PESCADO EN PREPARACIONES MITÓTICAS DE LA CALABAZA DEL CROMOSOMA
- Extraer cromosomas mitóticos de discos imaginales de larva de4º estadio de An. gambiae.
- Prepare las diapositivas del cromosoma convenientes para los PESCADOS.
- Agregue 2-3 μl de sonda de ADN etiquetada al tampón de hibridación en un tubo y mezcle suavemente pipeteando.
- Aplique 10 μl de la mezcla de la sonda a la corredera y cubra con una tapa de 22 X 22 mm. Presione las burbujas de aire con una punta de pipeta.
- Para contratending y detección, aplique Prolong anti-fade with DAPI a la preparación y manténgalo en la oscuridad durante al menos una hora antes de la visualización.
6) FISH 3D en el tejido ovárico de montaje completo
- Prepare la siguiente mezcla de búfer A:
KCl de 60 mM
NaCl de 15 mM
0,5 mM espermidina
0,15 mM Espermina
EDTA de 2 mM
0,5 mM EGTA
TUBERÍAS DE 15 mM - Prepare diapositivas para la visualización nuclear agregando una capa de nailpolish en un patrón cuadrado para que coincida con el tamaño de las cubiertas. Esto crea una superficie elevada para evitar el aplastamiento de los núcleos al colocar en la cubierta en el futuro.
- Diseccione los ovarios frescos de las hembras en etapa 3 de Christopher y manténgase en una solución de 150-250 μl Buffer A con digitonina al 0,5%. Ejecute una aguja de disección más grande sobre los folículos (en tubo con tampón A con digitonina al 0,5%) para destruir la membrana folicular.
- Vórtice durante 5-10 min para perturbar aún más los folículos. Raspe cualquier pieza folicular grande y tubo de centrífuga durante 30 segundos en el ajuste más bajo de ~ 500 revoluciones por minuto (RPM). Transferir sobrenadante a un nuevo tubo eppendorf de 2 ml y añadir 100 μl de tampón A. Repetir el paso 6.3 entre 5-7 veces, hasta que el tejido visible se rompa en partículas pequeñas.
- Gire ambos tubos durante 10 minutos a 2.000 RPM. Deseche el sobrenadante en ambos tubos. Nota: Ambos tubos se utilizarán para hacer diapositivas de visualización nuclear finales. El tubo con sobrenadante recogido debe contener principalmente núcleos extraídos, mientras que el tubo original con tejido contendrá una mezcla de tejido y núcleos incrustados en células nodrizas. Añadir 200 μl de Tampón A – 0.1% Tritón e incubar durante la noche a 4 °C. Centrífuga 5 min a 10.000 RPM (10.621 x G) y retire el sobrenadante.
- Añadir 200 μl 4% de Paraformaldehído en PBS. Incubar en termomezclador durante 30 min, mezclando a 450 RPM. Centrífuga 5 min a 5.000 RPM (2.655 x G) y retire el sobrenadante. Lavar con Buffer A con Tritón al 0,1% durante 5 min, mezclando a 450 RPM en termomezclador. Centrífuga 5 min a 5.000 RPM (2.655 G) y retire el sobrenadante.
- Agregue la sonda precalentada a 37 °C etiquetada al tubo. Desnaturalizar a 95 °C en termomezclador, mezclando a 450 RPM, durante 10 min. Continuar la desnaturalización a 80 °C durante 15 min con mezcla continua. Incubar a 37 °C en termomezclador, con mezcla de 450 RPM durante la noche. Centrífuga 5 min a 5.000 RPM (2.655 x G). Retire el sobrenadante.
- Lavar con Buffer A con Tritón al 0,1% durante 5 min. Centrífuga 5 min a 5.000 RPM (2.655 x G). Repetir 2 veces. Aplique drop de Prolong Anti-fade con DAPI.
- Pipetee los núcleos / solución DAPI con cuidado (evitando burbujas), aplique sobre la diapositiva y cubra con cubrebocas.
Resultados
La figura 1 describe el flujo general del protocolo descrito en este artículo. El usuario comienza inicialmente por microdissecting muestras de ADN cromosómico de diapositivas de membrana. El material microdissecado se extrae y purifica. El ADN purificado se amplifica, se vuelve a amplificar, se etiqueta y luego se usa para que FISH etiquete las diseminaciones cromosómicas.
El protocolo LCM se puede dividir en tres pasos generales: 1) Encontrar cromosomas de interés y preparar la región para el corte(Figura 2A),2) Cortar y catapultar la región cromosómica de interés vía láser(Figura 2B), y 3)Comprobar para determinar si la muestra está realmente catapultada en tapa adhesiva(Figura 2C). El proceso de LCM utilizado en los cromosomas del politeno de la tensión de An. gambiae Sua se muestra en el vídeo 1. El video detalla todo el proceso desde el inicio del programa, incluidas las funciones y consejos importantes del software.
Los kits WGA unicelulares GenomePlex y REPLI-g utilizados en este protocolo difieren en gran medida en el tamaño del producto resultante, así como en el rendimiento general. La Figura 3 muestra los resultados de la electroforesis en gel para los kits GenomePlex y REPLI-g, así como la cuantificación de las muestras por Nanodrop.
El material microdissecado se ha utilizado para crear sondas FISH que se dirigen a las regiones eucromáticas de un cromosoma. La Figura 4 muestra FISH de cinco sondas generadas a partir de material microdissecado en cromosomas politeno de An. gambiae. Cuatro brazos autosómicos fueron etiquetados con tres fluoróforos usando el kit WGA3: el cromosoma 3R está etiquetado en verde (fluoresceína), el cromosoma 3L está en una mezcla de rojo (Cy-3) y amarillo (Cy-5), el cromosoma 2R está en amarillo (Cy-5), y el cromosoma 2L está en rojo (Cy-3). El cromosoma de X fue etiquetado en la naranja (Cy-3) usando la mella-traducción del material de REPLI-g en un experimento separado. Para establecer la correspondencia entre las porciones eucromáticas de politeno y los brazos cromosómicos mitóticos, se han hibridado pinturas cromosómicas para interfase, profase, prometafase y metafase cromosomas de An. gambiae (Figura 5). Para visualizar la organización 3D de un solo brazo del cromosoma del politeno en el núcleo de la célula, los PESCADOS enteros del montaje fueron realizados en las células ováricas de la enfermera de la tensión de An. gambiae Sua (vídeo 2). Los distintos territorios del brazo cromosómico se ven claramente en núcleos con cromosomas interfase(Figura 5D)y politeno (Video 2).
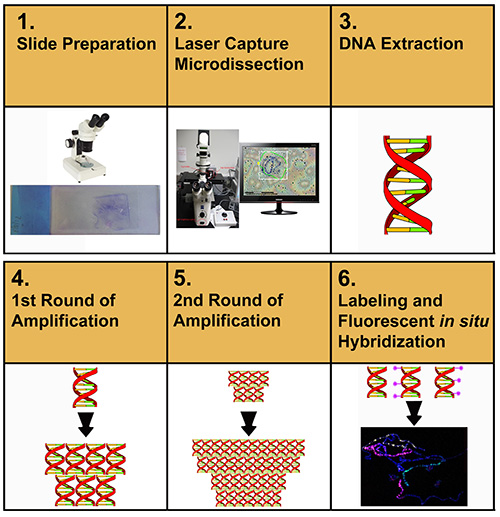
Figura 1. Representación esquemática de los procedimientos experimentales hacia la preparación de pinturas cromosómicas. Haga clic aquí para ver la imagen más grande.
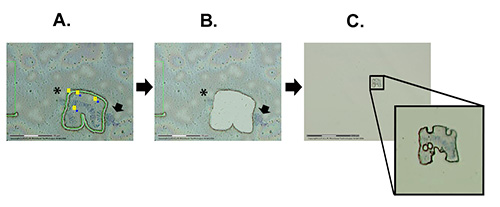
Figura 2. Los pasos principales en la microdisección cromosómica. A) Corte asistido por láser de la región cromosómica de interés a través de la membrana. B) Se realiza la membrana con un orificio después de la catapulta. C) La vista de la pieza catapultada de la membrana con un segmento cromosómico en ella unido a la tapa adhesiva. La flecha indica la heterocromatina del cromosoma X que permaneció en la diapositiva. El asterisco muestra un pedazo de otro cromosoma que permaneció en la diapositiva. Haga clic aquí para ver la imagen más grande.

Figura 3. Imágenes de gel de agarosa que muestran ADN después de WGA. A) ADN de bajo peso molecular (200-500 pb) del brazo 3R después de usar los kits WGA4 y WGA3 GenomePlex. B) ADN de alto peso molecular (10-20 kb) del brazo 2L después de la amplificación de REPLI-g. La escalera de 100 pb se muestra en los carriles izquierdos. Las siguientes tablas de imágenes de gel muestran concentraciones de ADN medidas por Nanodrop. Haga clic aquí para ver la imagen más grande.
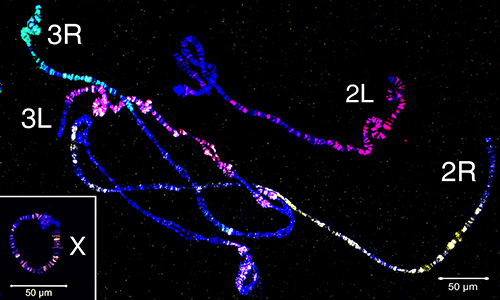
Figura 4. Pintura de los cromosomas del politeno de las células ováricas de la enfermera del Gambiae del An. usando cuatro puntas de prueba generadas del material microdissected. El cromosoma X está etiquetado en naranja (Cy-3) por la nick-traducción del material REPLI-g. El brazo 2R está etiquetado en amarillo (Cy-5); el brazo de 2L está en rojo (Cy-3); el brazo 3R está etiquetado en verde (fluoresceína); el brazo de 3L está etiquetado en una mezcla de rojo (Cy-3) y amarillo (Cy-5). Los autosomas están etiquetados con el kit de amplificación WGA3. La cromatina se tiñe de azul (DAPI). Los nombres de los cromosomas se colocan cerca de las regiones teloméricas. Haga clic aquí para ver la imagen más grande.
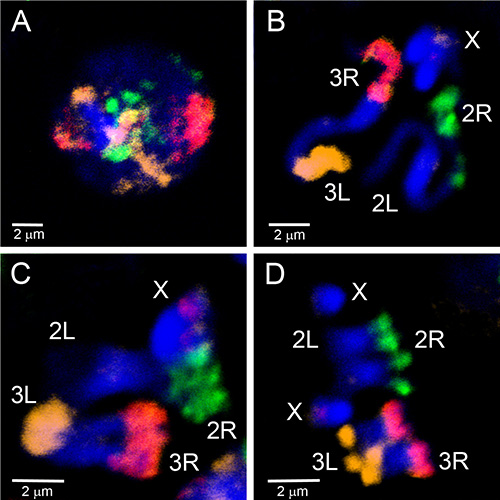
Figura 5. Pintura de los cromosomas de interfase (A), profase (B), prometaphase (C) y metaphase (D) de los discos imaginales larvales de la tensión de An. gambiae Mopti usando tres puntas de prueba generadas del material microdissected etiquetado por WGA3. El brazo 2R está etiquetado en verde (fluoresceína); el brazo de 2L no está etiquetado; el brazo 3R es en rosa, una mezcla de rojo (Cy-3) y naranja (Cy-5); el brazo de 3L está etiquetado en naranja (Cy-5). El cromosoma X tiene una etiqueta roja correspondiente a la sonda de ADNr 18S. La cromatina se tiñe de azul (DAPI). Las regiones brillantemente manchadas de cromosomas corresponden a la heterocromatina. Haga clic aquí para ver la imagen más grande.
Vídeo 1. The process of LCM of the An. gambiae polytene chromosomes. Haga clic aquí para ver el video 1.
Vídeo 2. El pescado 3D entero del montaje se realizó en las células ováricas de la enfermera de An. gambiae. La sonda está etiquetada en Cy-3 (representada en azul) y fue hecha de un brazo microdisecado del cromosoma 2R. La cromatina fue teñida con DAPI y es representada por el pseudo-colorante cian (azul claro). Haga clic aquí para ver el video 2.
Discusión
Hay múltiples pasos críticos para amplificar con éxito el ADN de muestras microdissecadas de cromosomas politeno. El protocolo emplea el uso de LCM, un método que aumenta la eficiencia general y reduce la exposición al ADN extraño al eliminar la interacción de las herramientas físicas con la muestra. Sin embargo, la amplificación de ADN extraño sigue siendo el mayor escollo potencial de este experimento. Por lo tanto, durante todo el proceso, es esencial mantener las muestras protegidas de la contaminación. A lo largo de las fases de preparación de diapositivas y microdisección, es esencial para el etanol lavar agujas de disección, portaobjetos, resbalones de cubierta, así como el espacio de trabajo. También se recomienda tratar con UV todos los equipos aplicables (agujas, portaobjetos, cubrebocas) antes de su uso.
La membrana en los portaobjetos de disección hace que la propagación de los cromosomas sea muy difícil. Es importante usar más del ovario (de la mitad a un par completo de ovarios) al hacer estos portaobjetos, para proporcionar una mayor probabilidad de encontrar un núcleo bien extendido. También se recomienda usar tejido fresco al hacer diapositivas, ya que las tasas de amplificación parecen disminuir a medida que los tejidos envejecen 49 y la propagación de cromosomas se vuelven más desafiantes. La preparación de diapositivas para la microdisección es el paso que más tiempo consume en este protocolo. Los cromosomas deben estar bien diseminados para evitar la adquisición accidental de material no deseado. La tinción de Giemsa permite al usuario comprobar la calidad de la propagación con un microscopio de contraste de fase antes de utilizar el sistema de microdisección.
La combinación de microdisección y amplificación proporciona la oportunidad de extraer y analizar fragmentos cromosómicos que varían en tamaño desde pequeñas regiones de interés hasta la mayoría de los brazos. Este protocolo permite al usuario obtener ADN de regiones morfológicamente distintas como inversiones, bandas eucromáticas específicas e interbandas, heterocromatina telomérica, centromérica e intercalaria. El usuario puede aplicar las sondas de pintura generadas para examinar cromosomas aberrantes, estudiar la homología entre especies en loci particulares o caracterizar la organización espacial de los cromosomas en un núcleo 3D intacto. Para desarrollar pinturas cromosómicas, seleccionamos segmentos eucromáticos para evitar la hibridación del ADN repetitivo con múltiples regiones cromosómicas. Como resultado, obtuvimos pintura específica del brazo sin usar un competidor, como el ADN genómico total ola fracciónde ADN C 0 t-1.
Elegimos utilizar los kits GenomePlex WGA y REPLI-G basados en revisiones que compararon la eficiencia y la tasa de abandono de múltiples kits de amplificación 40,44. Ambos kits tuvieron el mejor desempeño entre los métodos disponibles en ambas tasas de abandono (GenomePlex tuvo una tasa de 12.5% en comparación con el 37.5% de REPLI-g%) y porcentaje de marcadores amplificados (GenomePlex tuvo una tasa de amplificación del 45,24% frente al 30,0% de REPLI-g) 40. El kit GenomePlex también proporcionó una mayor cantidad de ADN, y por lo tanto lo convirtió en un mejor candidato para múltiples técnicas aguas abajo. Un kit de re-amplificación para el sistema GenomePlex también está disponible, lo que permite una mayor amplificación del ADN. Sin embargo, es importante tener en cuenta que la amplificación no es perfecta. La posibilidad sigue siendo que la amplificación acertada pueda introducir errores o tener un prejuicio hacia los loci específicos en la DNA de la blanco. Es importante considerar el tamaño final del fragmento de los métodos de amplificación del genoma disponibles. La fragmentación de GenomePlex da como resultado una biblioteca con fragmentos que van desde 200-500 bp, mientras que el kit REPLI-g produce fragmentos de aproximadamente 10-20 kb de tamaño. La aplicación posterior prevista de este protocolo es FISH, lo que hace que el kit GenomePlex sea una opción más factible, ya que proporciona el tamaño de fragmento deseado y la capacidad de etiquetar fragmentos de ADN directamente a través de WGA. Las moléculas de ADN largas producidas por la amplificación REPLI-g deben fragmentarse en la reacción de etiquetado de nick-translation aguas abajo.
Este protocolo se ha adaptado para amplificar con éxito la DNA de un solo brazo del cromosoma del politeno. Otros protocolos requieren la agrupación de muchos (típicamente 10-30) fragmentos cromosómicos para amplificar con éxito la muestra 7,11,28,40. Aunque la agrupación de cromosomas múltiples es posible utilizando nuestro método, la mayor probabilidad de contaminación de la muestra enfatiza la importancia de comenzar el experimento con el menor número posible de cromosomas. Si los cromosomas del politeno no están disponibles, nuestro protocolo se podría adaptar para el uso en cromosomas mitotic. Puede ser necesario, sin embargo, agrupar 10-15 cromosomas mitóticos antes de la amplificación para los PESCADOS acertados 11. El sesgo de amplificación es menor con altas cantidades de ADN de plantilla inicial 50. Los cromosomas politeno proporcionan aproximadamente 512 copias de una sola secuencia de ADN y 1024 copias de dos secuencias de ADN homólogas. Por lo tanto, la agrupación de cromosomas mitóticos ayudará a aumentar la calidad general del producto de ADN después de la amplificación.
Este procedimiento tiene muchos usos potenciales en estudios citogenéticos y genómicos. Aquí, utilizamos pinturas cromosómicas para establecer la correspondencia entre los segmentos eucromáticos de politeno y los brazos cromosómicos mitóticos en An. gambiae. Las mismas puntas de prueba de la pintura se pueden aplicar a la caracterización citogenética de las variedades de células de An. gambiae. Los cambios genómicos extensos y los cambios en números del cromosoma son características comunes de variedades de células 51,52. Se han detectado reordenamientos aneuploides, poliploides y cromosómicos como translocaciones, inversiones, deleciones y cromosomas en anillo en diferentes líneas celulares de mosquitos 53,54. Las observaciones citogenéticas clásicas 55 y el mapeo físico 56 han demostrado la aparición de translocaciones cromosómicas de brazo entero en la evolución de los mosquitos de la malaria. Por lo tanto, el material microdissected se puede utilizar conjuntamente con experimentos de los PESCADOS para comparar la homología de regiones cromosómicas entre las especies. Realizamos con éxito PESCADOS 3D con la punta de prueba de la 2R-pintura en cromosomas del politeno en células ováricas enteras de la enfermera del montaje. Este método facilitará el trazado de las trayectorias cromosómicas y el estudio de la arquitectura nuclear en Anopheles. Técnicas como el genotipado del ADN 57,58,la secuenciación del genoma de próxima generación 26,27,el desarrollo de mapas físicos y de enlace, y la generación de sondas para la pintura cromosómica 33,59, todas pueden beneficiarse de la microdisección específica del brazo y la región. Mediante la combinación de la tecnología LCM y el avance de WGA unicelular, demostramos un método eficiente y práctico para producir cantidades razonables de ADN a partir de un solo brazo cromosómico politeno.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la subvención de los Institutos Nacionales de Salud 1R21AI094289 a Igor V. Sharakhov
Materiales
| Name | Company | Catalog Number | Comments |
| Acetic acid | Fisher Scientific | A491-212 | |
| Methanol | Fisher Scientific | A412-4 | |
| Propionic acid | Sigma-Aldrich | 402907 | |
| Membrane slides 1.0 PET | Zeiss | 415190-9051-000 | |
| Buffer tablets “GURR” | Life Technologies | 10582-013 | |
| KaryoMAX Giemsa Stain | Life Technologies | 10092-013 | |
| ThermoBrite Slide Denaturation/Hybridization System | Abbott Molecular | 30-144110 | |
| Vacufuge vacuum concentrator | Eppendorf | 22820001 | |
| Spectroline Microprocessor-Controlled UV Crosslinker XL-1000 | Fisher Scientific | 11-992-89 | |
| Thermo Scientific NanoDrop | Fisher Scientific | ND-2000 | |
| REPLI-g Single Cell Kit | Qiagen | 150343 | |
| GenomePlex Single Cell Kit (WGA4) | Sigma-Aldrich | WGA4-10RXN | |
| GenomePlex WGA Reamplification Kit (WGA3) | Sigma-Aldrich | WGA3-50RXN | |
| MZ6 Leica stereomicroscope | Leica | VA-OM-E194-354 | A different stereomicroscope can be used |
| Olympus CX41 Phase Microscope | Olympus | CX41RF-5 | A different phase microscope can be used |
| PALM MicroBeam Laser Microdissection Microscope | Zeiss | ||
| Thermomixer | Eppendorf | 22670000 | |
| 10x PBS | Invitrogen | P5493 | |
| 50x Denhardt’s solution | Sigma-Aldrich | D2532 | |
| 99% Formamide | Fisher Scientific | BP227500 | |
| Dextran sulfate sodium salt | Sigma | D8906 | |
| 20x SSC buffer | Invitrogen | AM9765 | |
| ProLong Gold antifade reagent with DAPI | Invitrogen | P-36931 | |
| dATP, dCTP, dGTP, dTTP | Fermentas | R0141, R0151, R0161, R0171 | |
| Cy3-dUTP, Cy5-dUTP | GE Healthcare | PA53022, PA55022 | |
| BSA | Sigma-Aldrich | A3294 | |
| DNA polymerase I | Fermentas | EP0041 | |
| DNase I | Fermentas | EN0521 | |
| Spermine | Sigma-Aldrich | S3256-1G | |
| Spermidine | Sigma-Aldrich | S0266-1G | |
| Potassium Chloride (KCl) | Fisher Scientific | BP366-500 | |
| Sodium Chloride (NaCl) | Fisher Scientific | BP3581 | |
| EGTA | Sigma-Aldrich | E0396-25G | |
| PIPES | Sigma-Aldrich | P6757-25G | |
| EDTA | Fisher Scientific | S311-500 | |
| Digitonin | Sigma-Aldrich | D141-100MG | |
| Triton-X100 | Fisher Scientific | BP151-100 | |
| AdhesiveCap 500 clear | Zeiss | 415190-9211-000 | |
| QIAamp DNA Micro Kit (50) | Qiagen | 56304 | |
| Genomic DNA Clean and Concentrator Kit | Zymo Research | D4010 | |
| 37% Paraformaldehyde | Fisher Scientific | F79-500 |
Referencias
- Ried, T., Schrock, E., Ning, Y., Wienberg, J. Chromosome painting: a useful art. Human molecular genetics 7. , 1619-1626 (1998).
- Yang, F., et al. Reciprocal chromosome painting illuminates the history of genome evolution of the domestic cat, dog and human. Chromosome Res. 8, 393-404 (2000).
- Badenhorst, D., Dobigny, G., Robinson, T. J. Karyotypic evolution of hapalomys inferred from chromosome painting: a detailed characterization contributing new insights into the ancestral murinae karyotype. Cytogenet Genome Res 136. , 83-88 (2012).
- Trifonov, V. A., et al. Chromosomal evolution in Gekkonidae. I. Chromosome painting between Gekko and Hemidactylus species reveals phylogenetic relationships within the group. Chromosome Res. 19, 843-855 (2011).
- Nie, W., et al. Chromosomal rearrangements and karyotype evolution in carnivores revealed by chromosome painting. Heredity (Edinb. , 108-1017 (2012).
- Guan, X. Y., et al. Detection of chromosome 6 abnormalities in melanoma cell lines by chromosome arm painting probes). Cancer Genet Cytogenet. 107, 89-92 (1998).
- Guan, X. Y., et al. Characterization of a complex chromosome rearrangement involving 6q in a melanoma cell line by chromosome microdissection. Cancer genetics and cytogenetics 134. , 65-70 (2002).
- Breen, M., et al. Detection of equine X chromosome abnormalities in equids using a horse X whole chromosome paint probe (WCPP). Vet J. 153, 235-238 (1997).
- Kokhanenko, A. A., Anan'ina, T. V., Stegniy, V. N. The changes in chromosome 6 spatial organization during chromatin polytenization in the Calliphora erythrocephala Mg. (Diptera: Calliphoridae) nurse cells. Protoplasma 250. , 141-149 (2013).
- Timme, S., et al. Nuclear position and shape deformation of chromosome 8 territories in pancreatic ductal adenocarcinoma. Anal Cell Pathol (Amst. 34, 21-33 (2011).
- Drosopoulou, E., et al. Sex chromosomes and associated rDNA form a heterochromatic network in the polytene nuclei of Bactrocera oleae (Diptera: Tephritidae). Genetica. 140, 169-180 (2012).
- Pazian, M. F., Shimabukuro-Dias, C. K., Pansonato-Alves, J. C., Oliveira, C., Foresti, F. Chromosome painting of Z and W sex chromosomes in Characidium (Characiformes). Crenuchidae). Genetica. 141, 1-9 (2013).
- Howe, E. S., Murphy, S. P., Bass, H. W. Three-dimensional acrylamide fluorescence in situ hybridization for plant cells. Methods Mol Biol 990. , 53-66 (2013).
- Lysak, M. A., Mandakova, T. Analysis of plant meiotic chromosomes by chromosome painting. Methods Mol Biol 990. , 13-24 (2013).
- Ji, Z., Zhang, L. Chromosomics: detection of numerical and structural alterations in all 24 human chromosomes simultaneously using a novel OctoChrome FISH assay. J Vis Exp. 10, (2012).
- Idziak, D., et al. Painting the chromosomes of Brachypodium: current status and future prospects. Chromosoma. 120, 469-479 (2011).
- Fuchs, J., Kuhfittig, S., Reuter, G., Schubert, I. Chromosome painting in Drosophila. Chromosome Res. 6, 335-336 (1998).
- Zhimulev, I. F. . Morphology and structure of polytene chromosomes. 34, 1-490 (1996).
- Sharakhov, I. V., Sharakhova, M. V. in Chromosome Mapping Research Developments eds. J.F. Verrity & L.E. Abbington) (Nova Science. , (2008).
- Coluzzi, M., Sabatini, A., Torre, d. e. l. l. a., Di Deco, A., A, M., Petrarca, V. A polytene chromosome analysis of the Anopheles gambiae species complex). Science. 298, 1415-1418 (2002).
- Stegnii, V. N. Systemic reorganization of the architectonics of polytene chromosomes in the onto- and phylogenesis of malaria mosquitoes. Genetika. 23, 821-827 (1987).
- Stegnii, V. N. Systemic reorganization of the architectonics of polytene chromosomes in the onto- and phylogenesis of malarial mosquitoes. II. Species specificity in the pattern of chromosome relations with the nuclear envelope of nutrient ovarian cells. Genetika. 23, 1194-1199 (1987).
- Bonaccorsi, S., Santini, G., Gatti, M., Pimpinelli, S., Colluzzi, M. Intraspecific polymorphism of sex chromosome heterochromatin in two species of the Anopheles gambiae complex. Chromosoma. 76, 57-64 (1980).
- Zhimulev, I. F. Polytene chromosomes, heterochromatin, and position effect variegation. Adv Genet. 37, 1-566 (1998).
- Seifertova, E., et al. Efficient high-throughput sequencing of a laser microdissected chromosome arm. BMC Genomics. 14, 1471-2164 (2013).
- Weise, A., et al. High-throughput sequencing of microdissected chromosomal regions. European journal of human genetics. EJHG. 18, 457-462 (2010).
- Murphy, S. J., et al. Mate pair sequencing of whole-genome-amplified DNA following laser capture microdissection of prostate cancer. DNA research : an international journal for rapid publication of reports on genes and genomes 19. , 395-406 (2012).
- Moshkin, Y. M., et al. Microdissection and sequence analysis of pericentric heterochromatin from the Drosophila melanogastermutant Suppressor of Underreplication. Chromosoma. 111, 114-125 (2002).
- Decarlo, K., Emley, A., Dadzie, O. E., Laser Mahalingam, M. capture microdissection: methods and applications. Methods Mol Biol 755. , 1-15 (2011).
- Iyer, E. P., Cox, D. N. Laser capture microdissection of Drosophila peripheral neurons. J Vis Exp. 10, (2010).
- Kubickova, S., Cernohorska, H., Musilova, P., Rubes, J. The use of laser microdissection for the preparation of chromosome-specific painting probes in farm animals. Chromosome Res. 10, 571-577 (2002).
- Fukova, I., et al. Probing the W chromosome of the codling moth, Cydia pomonella, with sequences from microdissected sex chromatin. Chromosoma. 116, 135-145 (2007).
- Thalhammer, S., Langer, S., Speicher, M. R., Heckl, W. M., Geigl, J. B. Generation of chromosome painting probes from single chromosomes by laser microdissection and linker-adaptor PCR. Chromosome Res. 12, 337-343 (2004).
- Sims, C. E., Allbritton, N. L. Analysis of single mammalian cells on-chip. Lab on a chip 7. , 423-440 (2007).
- Hutchison, C. A., 3rd, J. C., Venter, Single-cell genomics. Nature biotechnology. 24, 657-658 (2006).
- Lasken, R. S., Egholm, M. Whole genome amplification: abundant supplies of DNA from precious samples or clinical specimens. Trends in biotechnology 21. , 531-535 (1016).
- Teruel, M., et al. Microdissection and chromosome painting of X and B chromosomes in the grasshopper Eyprepocnemis plorans. Cytogenet Genome Res. 125, 286-291 (2009).
- Liu, J. D., et al. Sex chromosomes in the spiny eel (Mastacembelus aculeatus) revealed by mitotic and meiotic analysis. Cytogenet Genome Res. 98, 291-297 (2002).
- Harvey, S. C., et al. Molecular-cytogenetic analysis reveals sequence differences between the sex chromosomes of Oreochromis niloticus: evidence for an early stage of sex-chromosome differentiation. Cytogenet Genome Res. 97, 76-80 (2002).
- Hockner, M., Erdel, M., Spreiz, A., Utermann, G., Kotzot, D. Whole genome amplification from microdissected chromosomes. Cytogenet Genome Res. 125, 98-102 (2009).
- Kitada, K., Taima, A., Ogasawara, K., Metsugi, S., Aikawa, S. Chromosome-specific segmentation revealed by structural analysis of individually isolated chromosomes. Genes Chromosomes Cancer. 50, 217-227 (2011).
- Ma, L., et al. Direct determination of molecular haplotypes by chromosome microdissection. Nature methods. 7, 299-301 (2010).
- Teruel, M., et al. Microdissection and chromosome painting of X and B chromosomes in Locusta migratoria. Chromosome Res. 17, 11-18 (2009).
- Treff, N. R., Su, J., Tao, X., Northrop, L. E., Scott, R. T. Single-cell whole-genome amplification technique impacts the accuracy of SNP microarray-based genotyping and copy number analyses. Molecular human reproduction 17. , 335-343 (2011).
- Rutten, K. B., et al. Chromosomal anchoring of linkage groups and identification of wing size QTL using markers and FISH probes derived from microdissected chromosomes. in Nasonia(Pteromalidae : Hymenoptera). Cytogenetic and Genome Research 105. , 126-133 (2004).
- Post, R. J., Kruger, A., Somiari, S. B. Laser-assisted microdissection of polytene chromosomes from Diptera for the development of molecular markers. Molecular Ecology Notes. 6, 634-637 (2006).
- George, P., Sharakhova, M. V., Sharakhov, I. V. High-throughput physical mapping of chromosomes using automated in situ hybridization. Journal of visualized experiments : JoVE. 10, (2012).
- Timoshevskiy, V. A., Sharma, A., Sharakhov, I. V., Sharakhova, M. V. Fluorescent in situ Hybridization on Mitotic Chromosomes of Mosquitoes. J Vis Exp. 10, (2012).
- Frumkin, D., et al. Amplification of multiple genomic loci from single cells isolated by laser micro-dissection of tissues. BMC biotechnology. 8, 10-1186 (2008).
- Raghunathan, A., et al. Genomic DNA amplification from a single bacterium. Applied and environmental microbiology 71. , 3342-3347 (2005).
- Cassio, D. Long term culture of MDCK strains alters chromosome content. BMC Res Notes. 6, 162-1610 (2013).
- Landry, J. J., et al. The Genomic and Transcriptomic Landscape of a HeLa Cell Line. G3. , (1534).
- Steiniger, G. E., Mukherjee, A. B. Insect chromosome banding: technique for G- and Q-banding pattern in the mosquito Aedes albopictus. Can J Genet Cytol. 17, 241-244 (1975).
- Brown, S. E., et al. Toward a physical map of Aedes aegypti. Insect Mol Biol. 4, 161-167 (1995).
- Green, C., Hunt, R. Interpretation of variation in ovarian polytene chromosomes of Anopheles funestus Giles. A. parensis Gillies, and A. aruni? Genetica. 51, 187-195 (1980).
- Sharakhova, M. V., Xia, A., Leman, S. C., Sharakhov, I. V. Arm-specific dynamics of chromosome evolution in malaria mosquitoes. BMC evolutionary biology. 11, 10-1186 (2011).
- Gu, L. H., et al. DNA genotyping of oral epithelial cells by laser capture microdissection]. Fa yi xue za zhi 22. , 196-197 (2006).
- Rook, M. S., Delach, S. M., Deyneko, G., Worlock, A., Wolfe, J. L. Whole genome amplification of DNA from laser capture-microdissected tissue for high-throughput single nucleotide polymorphism and short tandem repeat genotyping. The American journal of pathology 164. , 23-33 (2004).
- Houen, A., Field, B. L., Saunders, V. A. Microdissection and chromosome painting of plant B chromosomes. Methods in cell science : an official journal of the Society for In. Vitro Biology. 23, 115-124 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados