Method Article
マラリア蚊の2Dと3D染色体絵画
要約
染色体の絵画は、細胞核の組織化や核型の進化を研究するのに役立つ方法です。ここでは、その場での2次元蛍光化および3次元蛍光(FISH)に使用される単一ポリテン染色体から特定の領域を分離および増幅するアプローチ を 示す。
要約
腕染色体プローブ全体の 蛍 光蛍光体は、対象となるゲノム領域をマッピングし、染色体の再配置を検出し、細胞核における染色体の3次元(3D)組織を研究するための堅牢な技術です。レーザー捕捉マイクロディシス(LCM)と全ゲノム増幅(WGA)の出現により、単一細胞から大量のDNAを得ることができます。WGAキットの感度の向上により、染色体塗料を開発し、非モデル生物の染色体組織や進化を探求するためにそれらを使用するようになりました。ここでは、アフリカマラリア蚊 アノフェレスガンビアの卵巣看護師細胞から単一ポリテーン染色体アームの発腸セグメントを単一および増幅するための簡単な方法を提示する。この手順は、染色体塗料を得るための効率的なプラットフォームを提供し、サンプルに外来DNAを導入する全体的なリスクを低減します。WGAを使用することで、数回の再増幅が可能になり、2Dおよび3D FISHを含む複数の実験に使用できる大量のDNAが得られます。我々は、開発された染色体塗料が 、アンガンビアのポリテーンと有糸状染色体アームのユークロマチック部分との間の対応を確立するためにうまく使用できることを実証した。全体として、LCMと単染色体WGAの組合は、将来の細胞遺伝学的およびゲノム研究のためにかなりの量の標的DNAを作成するための効率的なツールを提供する。
概要
染色体絵画は、1-5のカルギタイプの進化を研究し、蛍光標識染色体DNA1,6-8のハイブリダイゼーションを介して細胞遺伝学的異常を可視化するのに有用な技術である。この方法は、開発9および病理10における染色体領域の3Dダイナミクスの研究にも適用される。微分標識染色体塗料は、個々の有糸11、12、及び、及び、異相非ポリテン15、16及びポリテン9、11、17染色体の可視化に使用される。染色体塗料を得るためのシンプルで堅牢なプロトコルは、この技術を蚊などの非モデル生物に拡大するのに非常に役立ちます。アノフェレスガンビア複合体は、マラリアを伝染させる能力を含む行動と適応が異なる7つの形態学的に区別できない蚊のグループです。 これらの蚊は、そのベクター容量が大きく異なる密接に関連する種の進化をよりよく理解するための優れたモデルとして機能します。 マラリア蚊の細胞遺伝学的研究のほとんどは、よく発達した高度に多性化された染色体を使用して行われてきた(18,19でレビュー)。多種染色体の読み取り可能なバンディングパターンは、研究者がアノフェレスガンビア20における多形反転と生態学的適応との関連を実証することを可能にした。また、3Dポリテーン染色体組織の組織特異的21および種特異的22の特徴は、An.マクリペニス複合体において特徴付けられている。しかし、有糸染色体を研究することは、追加の重要な情報を提供する可能性があります。例えば、有糸分裂性染色体において観察されるヘテロクロマチン多型の高レベルは、アンガンビア23における交配活性および生殖能力の低下と相関している。ポリテーンと有糸状染色体の腕の水色セグメント間の対応は、相対的な長さを比較することによってのみ確立することが困難である可能性があります。これは、ヘテロクロマチンが有糸分裂性染色体のかなりの部分を構成するが、ポリテネ染色体24において過小評価されているからである。マラリア蚊の染色体塗料の入手可能性は、研究者が追加の種を含めることによって細胞遺伝学的研究を大幅に拡大することを可能にする一方で、この疫学的に重要な昆虫群における核型進化と染色体の3Dダイナミクスの分析におけるコストと時間を大幅に削減する。
染色体の大きなストレッチを得るために、マイクロ解剖は染色体補体の関心のある特定の領域を操作し、分離するための不可欠な技術となっている。 全ゲノム増幅(WGA)と結合すると、微細解剖はFISH 11、12および次世代ゲノムシーケンシング25-27を含む強力な下流アプリケーションをもたらす。以前は、この技術は、経験豊富なユーザー 28によって手動で制御されなければならなかった特殊なマイクロ解剖針の使用を必要としました。 レーザーキャプチャマイクロディスセクション(LCM)の進歩により、単一細胞29、30または個々の染色体31-33を汚染のリスクを低減するに適した簡素化されたツールが得られました。このアプローチは、ユーザーが34-36を一緒に複数の細胞をプールすることから生じるコンセンサスの風景の代わりに、単一細胞で発生する遺伝的不均一性および染色体異常を研究することを可能にする。マイクロディスセクションから生成されたDNAを増幅するために、複数の方法が使用されている。 DOP-PCRは、反復性の高い配列の増幅に有用な技術であり、バッタ37、スピニーウナギ38、ナイルティラピア39などの種からマイクロ分解染色体を増幅するために使用されてきた。 最近では、PCRベースのゲノムプレックスWGA4単一細胞キットと複数変位増幅ベース(MDA)Repli-Gシングルセルキットは、バッタ37およびイナゴ43のB染色体系を含む、単一ヒト細胞の遺伝子解析と染色体40-42を含む実験のための貴重なツールとなっています。これら2つのキットはそれぞれ長所と短所を持っていますが、他の利用可能な増幅システムよりも明らかな優位性が実証されています44.
単一の染色体または染色体セグメントから得られるDNAの量は、核全体の量より有意に少ない。したがって、単一の染色体の微細解剖、増幅、およびその後の分析は、特にショウジョウバエやアノフェレスのような小さなゲノムを持つ生物において、はるかに困難である。 塗料は、ヒト33およびスズメバチ45の単一のマイクロ分解染色体から開発されているが、成功したFISH実験では、フルーツフライ11の複数(少なくとも10〜15)の微小分裂した有糸状染色体が必要であった。しかし、(i)異なる染色体からの材料で汚染される可能性を減らし、(ii)微小解剖に必要な染色体製剤の数を最小限に抑えるため、(iii)FISHと下流の両方の両方で微小化したサンプルのヌクレオチドおよび構造多型を低下させるため、単一の染色体をマイクロ分解および増幅する能力が重要である。多くのディプテラン種に見られるポリテーン染色体は、DNAのはるかに高い開始量を取得するユニークな機会を提供します。また、染色体を使用しても達成できない高分解能と染色体構造を提供します。この追加された解像度は、染色体再配置、クロマチン構造、および染色体セグメントを28,46にマイクロセックする視覚化において重要であり得る。
ここでは、単一のポリテーン染色体アームからユークロマティックセグメントを効率的に分離し、DNAを増幅し、マラリア蚊の下流のFISHアプリケーションで使用する手順を提示します。 まず、LCMを適用して、特別に調製した膜スライドから単一の染色体アームを分離して抽出します。第二に、WGAは、マイクロディスセクテッド材料からDNAを増幅するために使用される。第3に、FISH実験において増幅DNAをポリテーンスカッシュ製剤47、メタフェーズおよび相間染色体スライド48、ならびに3D卵巣全体実装サンプルにハイブリダイズする。この手順は、アンガンビアの染色体の腕にエウクロマチンの大部分を正常にペイントするために行われています.
プロトコル
1) レーザー捕捉マイクロ解剖のためのポリテーン染色体スライド調製
- 25時間のポスト・フィードで半グラビッド ・アノフェレス の女性を解剖する。約5人の女性の卵巣を、室温で500μlの新鮮な変性カルノイ溶液(メタノール100%:氷河酢酸、3:1)に24時間固定します。長期保存のために-20°Cに卵巣を移す。
- カルノイ溶液(100%エタノール:氷酢酸、3:1)と50%プロピオン酸を染色体スライドの直前に調製します。
- ツァイス1.0 PET膜スライド上のカルノイ溶液の1滴に卵巣の1組を置きます。サイズに応じて、卵巣を解剖針で約2〜4個のセクションに分割し、解剖顕微鏡下のきれいなスライドに50%のプロピオン酸の低下に置きます。
- 毛包を分離し、解剖実体顕微鏡下でペーパータオルを使用して残りの組織を除去する。卵胞に50%プロピオン酸の新しい滴を加え、室温で3〜5分間座るようにします。
- シリコン化カバースリップを液滴の上に置きます。スライドを約1分間放置します。
- スライドを吸収材で覆い(この方法ではフィルターペーパーを使用)、鉛筆の消しゴム側を使用しながら、消しゴムで繰り返しタップしてカバースリップに寛大な圧力を加えます。
- スライド変性/ハイブリダイゼーションシステムでスライドを60°Cに15〜20分間加熱し、ポリテーン染色体の平坦化を支援します。一晩4°Cの湿気の多いチャンバーにスライドを入れ、酸が染色体をさらに平らにできるようにします。
- 冷たい50%エタノールにスライドを10分間入れる。カバースリップをそっと取り出し、冷たい50%エタノールで10分交換します。
- 脱水スライドを70%、90%、100%エタノールずつ5分間乾燥させる。空気乾燥スライド。
- 1Lの蒸留水に単一の緩衝剤を添加してGURR緩衝液の溶液を調製する。オートクレーブ。
- 50 ml の GURR バッファーに 1 ml の Giemsa 染色液を加えて Giemsa 溶液を調製します。
- 10分間、ギームサ溶液に空気乾燥スライドを入れ、1X PBSで3回洗浄します。空気乾燥は汚染を避けるために制御された無菌の気候で再び滑る。
2)単一ポリテーン染色体アームのレーザー捕捉マイクロディセクション
このセクションでは、PALMマイクロビームレーザーマイクロディセクショクシステムに付属のPALMRoboソフトウェアの使用について詳しく説明します。
- 100%エタノールで顕微鏡を洗浄してください。UVクロスリンカーでUV光で手袋やチューブを殺菌します。
- PALMマイクロビームレーザーマイクロディション顕微鏡の電源を入れ、レーザーをオンにします。レーザー解剖スイート、PALMRoboを開き、必要に応じて「パワー」と「フォーカス」の設定を構成します。
- 関心のあるポリテン染色体アームを検索します。
- 「鉛筆」ツールを使用して、選択した領域の輪郭を描きます。
- メニューバーから「要素」ウィンドウを開きます。
- 「描画要素」を選択し、「カット」を選択したことを確認します。
- 粘着キャップチューブをホルダーに取り付け、スライドの上に小さな隙間<1mmを残して、レーザーカットを開始します。
- カット部位内に「カタパルト選択」を配置し、縁と染色体の間にスペースを残します。
- ドロップダウンオプションから「LPC」を選択し、カタパルトを開始します。
- 「目」アイコンを押して、サンプルがキャップにカタパルトされたことを確認します。
3) 単一のマイクロ分解されたポリテーン染色体アームからのDNAの精製
QIAamp DNAマイクロキットの指示に従って、収集したDNAを放出して精製します。ステップ3.1は、反転管を収容するように修正されました。
- 15 μl バッファー ATL と 10 μl プロテイナーゼ K を反転チューブ(キャップの内側)に加え、56 °Cで3時間インキュベートします。
- 25 μl バッファー ATL、50 μl バッファー AL、および 1 μl キャリア RNA を追加します。ミックス. 50 μl 100% EtOH を追加します。混ぜる。
- リセートをQIAampカラムに移す;遠心機。500 μl バッファー AW1 を追加して洗浄します。遠心機。新しいコレクションチューブにカラムを配置し、500 μlバッファAW2を追加します。遠心機。新しいチューブに柱を配置します。余分な液体を除去するために遠心分離機。
- カラムを1.5μlマイクロ遠心チューブに入れ、20μlの水を溶出させます。遠心機。
- 真空化したDNAを真空化した状態で9μlの最終体積まで蒸発させる。
4) 単一のマイクロ分解されたポリテーン染色体アームからのDNAの増幅
2つの異なるプロトコルが、単一の染色体アームのWGAに使用された。
- ゲノムプレックスWGAによるDNA増幅とプローブ調製
- GenomePlex シングルセル WGA4 キット プロトコルに従って、増幅 DNA の最初のバッチを生成します。
- 9 μl サンプルに作りたてのプロテイナーゼ K溶液を加えます。混合.50°CでDNAを1時間インキュベートし、99°Cに加熱して4分間加熱します。氷の上に置いておきなさい。
- 2 μl 1X シングルセルライブラリ調製バッファーと 1 μl のライブラリ安定化ソリューションを追加します。混ぜる. 95 °Cに2分間加熱します。氷と遠心分離機で冷やします。
- ライブラリ調製酵素の1 μlを追加します。ミックスと遠心分離機。次のようにインキュベートします。
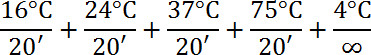
- 7.5 μl 10X 増幅マスターミックス、48.5 μl水を加えます。5.0 μl WGA DNAポリメラーゼ;ミックスと遠心分離機。
- 以下のようなサーモサイクル:
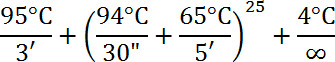
- ゲノムDNAクリーン&コンセントレータキットを使用してDNAを精製します。プロトコルは次のとおりです。
- 5:1 DNA結合バッファー:DNAサンプルを追加します(特に2kb未満のゲノムDNAに対して。サンプルDNAが2kbを超える場合は、2:1比を使用し、提供されたスピンカラムに転送します。遠心機。
- 200 μl の DNA 洗浄バッファーと遠心分離機を追加します。洗浄工程を繰り返します。その後、50 μlの水を加え、DNAを新しい1.5 mlチューブに溶出させます。
- GenomePlex WGA3 再増幅キットを使用してサンプル DNA を次のように再増幅します。
- PCRチューブに10 μl DNA(キットは合計10 ngのDNAを推奨)、49.5 μlの10x増幅マスターミックス、3.0 μl 10 mM DNTPミックス、5.0 μl WGA DNAポリメラーゼを加えます。ミックスと遠心分離機。
- 反応には以下のプロファイルを使用します。
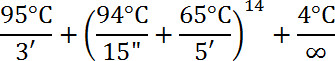
- DNAを-20°Cに保存する。
- ゲノムプレックス WGA3 再増幅キットを使用して FISH の DNA を次のようにラベル付けします。
- 49.5 μlの水を含むPCRチューブに10 μl DNAを加えることで、ゲノムプレックス WGA3 再増幅キットからマスターミックスを作成し、 7.5 μl 10X 増幅マスターミックス、3.0 μl 1 mM dNTP ミックス (1 mM dATP、dCTP、dGTP、0.3 μl 1 mM dTTP - ラベル dUTP を使用する場合)、25 nM ラベル dUTP の 1 μl、および5.0 μl WGA DNAポリメラーゼ。
- 反応には以下のプロファイルを使用します。
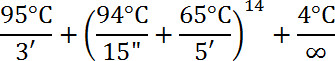
- エタノールは、3 M酢酸ナトリウムpH 5.2および100%エタノールの2〜3容量の最終反応容積(75μl反応に対して7.5 μl)を1/10に添加することにより標識プローブを沈殿させます。DNAサンプルを-80°Cで少なくとも30分間冷やします。
- 遠心分離機サンプルを4°Cで10分間、ラベル付きペレットを作成し、上清および空気乾燥ペレットを除去します。
- 次のように、ハイブリダイゼーション バッファを作成します。
0.2 g デキストラン硫酸
1200 μl 脱イオン化フォームアミド
580 μl H2O
120 μl 20X SSC - 空気乾燥ペレットに40 μlのハイブリダイゼーションバッファーを加えます。
- GenomePlex シングルセル WGA4 キット プロトコルに従って、増幅 DNA の最初のバッチを生成します。
- REPLI-GシングルセルWGAによるDNA増幅とプローブ調製、続いてニック翻訳
- REPLI-g シングルセル WGA キットプロトコルに従って、増幅された DNA を生成します。
- バッファー D2 (3 μl の 1 M DTT + 33 μl バッファー DLB) を準備します。
- 精製されたマイクロディスセクテッド材料の4 μlを3 μlのバッファーD2と混合します。ミックスするフリックチューブ。65°Cで10分間インキュベートします。 ストップソリューションを3 μl追加します。混ぜる。
- 9 μl H2O、29 μl の REPLI-g反応バッファー、および 2 μl の REPLI-g DNA ポリメラーゼをサンプルに加えます。30°Cで8時間インキュベートします。65°Cに加熱して3分間加熱することによりDNAポリメラーゼを不活性化する。DNAを-20°Cに保存する。
- NICK 変換プロトコルに従って、REPLI-g 増幅 DNA のラベルを付けます。
- 次のラベリングミックスを準備します。
増幅DNA 1 μg
5 μl 10X DNA ポリメラーゼバッファー
5 μl 10X dNTP
5 μl 1X BSA
1 μl 1 mM ラベル付き dNTP
4 μl 1 U/μl DNase I
1 μl 10 U/μl DNAポリメラーゼ I
H2O~50 μl - 15°Cで2時間インキュベートします。0.5 M EDTAの2 μlを加え、反応を止めてください。ゲルで動作させることでDNAフラグメントサイズを確認します。
- ペレットを沈殿させ、可溶化するために 4.1.4.3-4.1.4.6 からのステップに従ってください。
- 次のラベリングミックスを準備します。
- REPLI-g シングルセル WGA キットプロトコルに従って、増幅された DNA を生成します。
5)ポリテーンおよびミトティック染色体スカッシュ製剤上の2D FISH
ポリテーンスカッシュ 47 および有糸分裂性染色体スライド 48の準備については、FISHの詳細なプロトコルを参照してください。ここでは、簡単なプロトコルを提供します。
- ポリテーン染色体スカッシュ製剤上のFISH
- RTで20分間1X PBSにスライドを浸し、1X PBSで4%パラホルムアルデヒドのスライドを1分間修正します。
- エタノールの流れによって脱水スライド:50%、70%、90%、100%RTで5分のための。空気乾燥スライド。
- プローブをハイブリダイゼーションバッファーで37°Cで予熱する。 スライドするプローブを10~20μl追加します。カバーは22×22mmのカバースリップです。ピペットチップを使用して気泡を押し出します。
- 染色体を変性させ、90°Cで10分間プローブを変性する。カバースリップのシールエッジにゴムセメントを付けます。滑り込み気湿ったチャンバーに移し、一晩で39°Cでインキュベートします。
- スライドを39°Cで1X SSCで20分間洗います。RTで1X SSCでスライドを20分間洗います。1X PBSでスライドをすすい、DAPIで延長アンチフェードを追加します。カバースリップで覆い、視覚化の前に少なくとも1時間は4 °Cのスライドボックスに保管してください。
- 有糸分裂性染色体スカッシュ製剤上のFISH
- アンガンビアの4番目のインスター幼虫の想像力の円盤から有糸分裂性染色体を抽出する。
- FISHに適した染色体スライドを準備します。
- 2~3 μlの標識DNAプローブをチューブ内のハイブリダイゼーションバッファに加え、ピペット処理で軽く混合します。
- 10 μlのプローブ混合物をスライドに塗布し、22 X 22 mmのカバースリップでカバーします。ピペットチップを使用して気泡を押し出します。
- 対抗および検出の場合は、DAPI によるアンチフェードを延長して準備を行い、可視化の前に少なくとも 1 時間は暗闇の中に保管してください。
6)全体マウント卵巣組織上の3D FISH
- 次のバッファ A ミックスを準備します。
60 mM KCl
15 mM ナクル
0.5 mM スペルミジン
0.15 mM スペルミン
2 mM EDTA
0.5 mM EGTA
15 mMパイプ - カバーリップのサイズに合わせて正方形のパターンでマニキュアの層を追加することにより、核の視覚化のためのスライドを準備します。これにより、将来カバースリップに置くときに核の押しつぶしを防ぐために、表面を隆起させます。
- クリストファーのステージ3メスから新鮮な卵巣を解剖し、0.5%デジコニンを有する150-250 μl緩衝液Aの溶液に保つ。卵胞膜を破壊するために卵胞(0.5%デジトニンを持つ緩衝管で)の上に大きな解剖針を実行します。
- 5〜10分間の渦をさらに邪魔する。1分間に500回転(RPM)の最低設定で、大きな濾胞片と遠心管 を 30秒間削り取ります。上清を新しい2mlエペンドルフチューブに移し、100 μlのバッファAを加え、目に見える組織が小さな粒子に割れるまで、ステップ6.3を5~7回繰り返します。
- 2,000 RPMで10分間、両方のチューブを回転させます。両方のチューブに上清を捨てます。 注: 両チューブは、最終的な核可視化スライドの作成に使用されます。収集された上清を有するチューブは主に抽出された核を含み、組織を持つ元のチューブには看護師細胞に埋め込まれた組織と核の混合物が含まれるべきである。 バッファーA – 0.1%トリトンの200 μlを加え、4°Cで一晩インキュベートします。 遠心分離機 10,000 RPM (10,621 x G) で 5 分、上清を除去します。
- PBSに200μl 4%パラホルムアルデヒドを加えます。450 RPMで混合し、30分間、サーモミキサーでインキュベート。5,000 RPM(2,655 x G)で5分の遠心分離機を使用し、上清を除去します。0.1%トリトンで5分間バッファAで洗浄し、サーモミキサーで450 RPMで混合します。5,000 RPM (2,655 G) で 5 分遠心分離機を使用し、上清を除去します。
- 37°C標識プローブで予温してチューブに加えます。サーモミキサーで95°Cで変性し、450RPMで10分間混合します。混合を続けて80°Cで15分間変性を続けます。サーモミキサーで37°Cでインキュベートし、一晩450 RPM混合します。5,000 RPMで5分の遠心分離機(2,655 x G)。上清を除去します。
- 0.1%トリトンで5分間バッファAで洗います。5,000 RPMで5分の遠心分離機(2,655 x G)。2回繰り返します。DAPIで延長アンチフェードのドロップを適用します。
- 核/DAPI溶液を慎重にパイプアウト(気泡を避ける)、スライドに適用し、カバースリップで覆います。
結果
図 1 では、この記事で説明するプロトコルの全体的なフロースルーについて説明します。 ユーザーは最初に膜スライドから染色体DNAサンプルをマイクロディスセプティングすることによって開始する。 マイクロディセクテッド材料を抽出し、精製します。精製されたDNAは、次いで増幅され、再増幅され、標識され、そしてFISHが染色体の広がりを標識するために使用される。
LCMプロトコルは、3つの全体的なステップに分けることができます:1)目的の染色体を見つけ、切断のための領域を準備する(図2A)、2)レーザーを介して目的の染色体領域を切断してカタパルトする(図2B)、および3)サンプルが実際に接着剤キャップにカタパルトされているかどうかを判断する(図2C)。An. ガンビアスア株ポリテーン染色体で使用される LCM のプロセスは、ビデオ 1 に示されています。ビデオでは、重要なソフトウェア機能やヒントを含む、プログラムの起動からプロセス全体を詳しく説明します。
このプロトコルで使用されるゲノムプレックスとREPLI-g単一細胞WGAキットは、結果として生じる製品サイズと全体的な収率において大きく異なります。 図3 は、ゲノムプレックスおよびREPLI-gキットに対して実行されたゲル電気泳動の結果と、Nanodropによるサンプルの定量化を示しています。
マイクロ分解された材料は、染色体のユークロマティック領域を標的とするFISHプローブを作成するために使用されてきました。 図4 は 、An. gambiaeのポリテーン染色体上のマイクロ分解された物質から生成された5つのプローブのFISHを示しています。 4本の常染色体腕に3つのフルオロフォアがWGA3キットを使用して標識された:3R染色体は緑色(フルオレセイン)で標識され、3L染色体は赤(Cy-3)と黄色(Cy-5)の混合物であり、2R染色体は黄色(Cy-5)、2L染色体は赤(Cy-3)である。X染色体は、別の実験でREPLI-g材料のニック翻訳を用いてオレンジ色(Cy-3)で標識した。ポリテーンと有糸状染色体の腸内部分と有糸状染色体の間の対応を確立するために、染色体塗料は、 ガンビアの 交相、プロフェーズ、プロメタ相および形相染色体にハイブリダイズされている(図5)。 細胞核内の単一ポリテーン染色体アームの3D組織を可視化するために、全体のマウントFISHを アンガンビア スア株卵巣看護師細胞上で行った(ビデオ2)。 異なる染色体の腕の領域は、相間(図5D)とポリテーン(ビデオ2)染色体を有する核ではっきりと見られる。
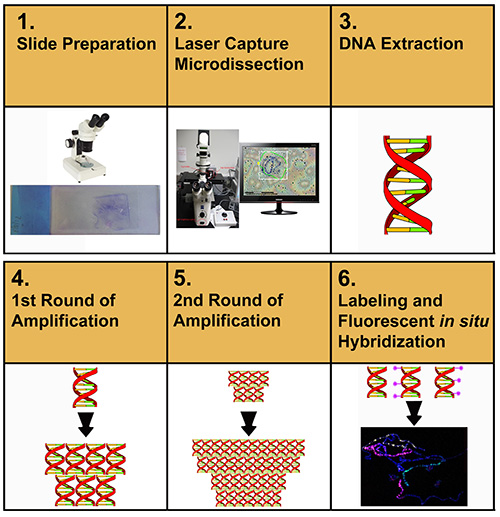
図 1. 染色体塗料の調製に向けた実験手順の概略表現。 ここをクリックすると、より大きな画像が表示されます。
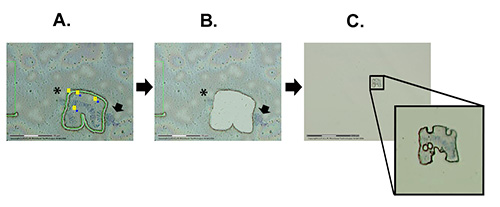
図 2. 染色体微小解剖における主要なステップ。A) 膜を通して目的の染色体領域のレーザー支援切断.B)カタパルト後に穴を開けた膜が行われる。C)粘着キャップに染色体セグメントが付いている膜のカタプル部分のビュー。矢印は、スライド上に残ったX染色体のヘテロクロマチンを示す。アスタリスクは、スライドに残った別の染色体の一部を示しています。 ここをクリックすると、より大きな画像が表示されます。

図 3. WGA後のDNAを示すアガロースゲル画像。A) WGA4およびWGA3ゲノムプレックスキットを使用した後のアーム3Rからの低分子量(200-500 bp)DNA。B) REPLI-g増幅後のアーム2Lからの高分子量(10〜20kb)DNA。100 bpの梯子は左車線に示されている。下の表は、ナノドロップで測定したDNA濃度を示しています。 ここをクリックすると、より大きな画像が表示されます。
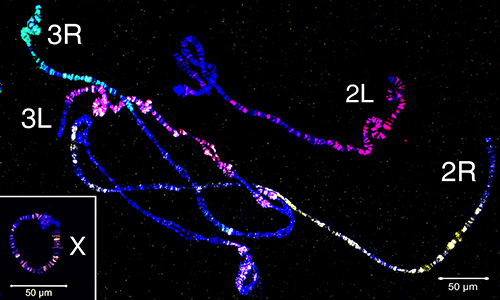
図 4. 微小分解材料から生成された4つのプローブを用いた アンガンビア の卵巣看護師細胞からのポリテーン染色体の塗装。X染色体は、REPLI-g材料のニック翻訳によりオレンジ色(Cy-3)でラベル付けされます。2Rアームは黄色(Cy-5)でラベル付けされています。2Lアームは赤(Cy-3)です。3Rアームは緑色(フルオレセイン)でラベル付けされています。3Lアームは、赤(Cy-3)と黄色(Cy-5)の混合物で標識されています。オートソームにはWGA3増幅キットでラベルが付いています。クロマチンは青色(DAPI)で染色される。染色体名はテロメリック領域の近くに置かれます。 ここをクリックすると、より大きな画像が表示されます。
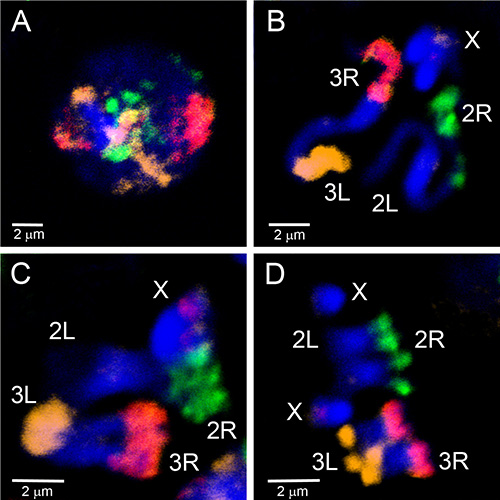
図 5. WGA3によって標識されたマイクロディセプト材料から生成された3つのプローブを用いて、 アンガンビア ・モプティ株の幼虫イマジナルディスクからの相間(A)、前相(B)、前転移(C)およびメタフェーズ(D)染色体の塗装。2Rアームは緑色(フルオレセイン)でラベル付けされています。2Lアームはラベル付けされていない。3Rアームはピンク色で、赤(Cy-3)とオレンジ(Cy-5)の混合物です。3Lアームはオレンジ(Cy-5)でラベル付けされています。X染色体は、18S rDNAプローブに対応する赤色のラベルを有する。クロマチンは青色(DAPI)で染色される。染色された領域はヘテロクロマチンに相当する。 ここをクリックすると、より大きな画像が表示されます。
ビデオ 1.アンガンビアポリテーン染色体のLCMのプロセス。ビデオ 1 を表示するには、ここをクリックします。
ビデオ 2.アンガンビア卵巣看護師細胞上で行われた全体のマウント3D FISH。プローブはCy-3(青色で描かれている)で標識され、マイクロ分解された2R染色体アームから作られました。 クロマチンはDAPIで染色され、シアン擬色(水色)で描かれています。ビデオ2を見るには、ここをクリックしてください。
ディスカッション
マイクロ分解されたポリテン染色体サンプルからDNAを正常に増幅するために重要な複数のステップがあります。このプロトコルは、LCMの使用を採用しています, 両方の全体的な効率を向上させ、サンプルとの物理的なツールの相互作用を除去することにより、外来DNAへの暴露を低減する方法.しかし、外来DNAの増幅は、この実験の最大の潜在的な落とし穴です。したがって、プロセス全体の間に、汚染からサンプルを保護することが不可欠です。スライド調製およびマイクロ解剖段階を通して、それはエタノール洗浄解剖針、スライド、カバースリップ、ならびにワークスペースに不可欠である。 また、UVは、使用する前に、すべての適用可能な機器(針、スライド、カバーリップ)を治療することをお勧めします。
解剖スライド上の膜は染色体の広がりに非常に困難になります。 これらのスライドを作るときに卵巣の多くを使用することが重要です (卵巣の完全なペアに半分) , よく広がる核を見つけるより大きなチャンスを提供します. また 、49 歳の組織や染色体の広がりが難しくなるにつれて増幅率が低下するように見えるため、スライドを作るときに新鮮な組織を使用することをお勧めします。 マイクロディシスセクションのスライドの準備は、このプロトコルで最も時間のかかるステップです。 染色体は、不要な材料の偶発的な取得を避けるためによく広がっている必要があります。 Giemsa染色により、マイクロディスセクションシステムを使用する前に位相コントラスト顕微鏡で広がり品質を確認できます。
マイクロディションと増幅の組み合わせは、対象となる小さな領域から大部分の腕に至るまでの染色体断片を抽出して分析する機会を提供します。 このプロトコルは、ユーザーが反転、特定の発泡バンドおよびインターバンド、テロメリック、中心および中基性ヘテロクロマチンなどの形態学的に異なる領域からDNAを得ることを可能にする。生成された絵画プローブを、異常な染色体の調べ、特定の遺伝子座における種間相同性学の研究、または無傷の3D核における染色体の空間組織の特徴付けに適用することができる。染色体塗料の開発では、複数の染色体領域を持つ反復DNAのハイブリダイゼーションを避けるために、ユークロマティックセグメントを選択しました。その結果、総ゲノムDNAやC0t-1 DNA画分など、競合者を用いずに腕特異的な絵画を得た。
複数の増幅キット40,44の効率とドロップアウト率を比較したレビューに基づいて、ゲノムプレックスWGAキットとREPLI-Gキットを使用することを選択しました。 両方のキットは、両方の中退率で利用可能な方法の中で最高のパフォーマンスを発揮しました(GenomePlexはREPLI-gの37.5%と比較して12.5%の割合を持っていました増幅マーカーの割合(GenomePlexは45.24%の増幅率を持ち、REPLI-gの30.0%)40.GenomePlexキットは、より多くのDNAを提供し、したがって、複数の下流技術のためのより良い候補にしました。 ゲノムプレックスシステムの再増幅キットも利用可能で、DNAのさらなる増幅が可能です。しかし、増幅は完璧ではないことに注意することが重要です。 増幅が成功すると、エラーが発生したり、標的DNAの特定の遺伝子座に偏りが生じる可能性が残っています。利用可能なゲノム増幅法の最終的なフラグメントサイズを考慮することが重要である。 GenomePlex の断片化により、200 ~ 500 bp の断片を含むライブラリが作成され、REPLI-g キットは約 10 ~ 20 kb のサイズのフラグメントを生成します。このプロトコルの下流の用途はFISHであるため、望ましいフラグメントサイズとWGAを介してDNA断片を直接ラベル付けする機能を提供するため、GenomePlexキットはより実行可能な選択肢となっています。REPLI-g増幅によって生成される長いDNA分子は、下流のニック翻訳標識反応で断片化されなければならない。
このプロトコルは、単一のポリテン染色体アームからDNAを正常に増幅するために適応されています。 他のプロトコルは、サンプル7、11、28、40を正常に増幅するために、多くの(典型的には10〜30)染色体断片のプーリングを必要とする。 我々の方法を用いて複数の染色体のプールは可能であるが、サンプル汚染の可能性が高まっていることは、できるだけ少ない染色体で実験を開始することの重要性を強調している。ポリテーン染色体が利用できない場合、我々のプロトコルは、有糸染色体での使用に適応することができる。しかし、FISH11を正常に増幅する前に10-15ミトティック染色体をプールする必要があるかもしれません。増幅バイアスは、大量の開始テンプレートDNA 50と低い。ポリテン染色体は、1つのDNA配列の約512コピーと2つの相同DNA配列の1024コピーを提供します。 したがって、有糸分裂性染色体をプールすることは、増幅後のDNA産物の全体的な品質を高めるのに役立ちます。
この手順は、細胞遺伝学的およびゲノム研究における多くの潜在的な用途を有する。ここでは、染色体塗料を使用して、アンガンビアのポリテーンと有糸状染色体アームのユークロマティックセグメントの対応関係を確立しました。同じ絵画プローブを、ガンビア細胞株の細胞遺伝学的特徴化に適用することができる。広範囲なゲノム再構成と染色体数の変化は、細胞株51,52の一般的な特徴である。異なる蚊細胞株53,54において、異なる蚊細胞株中で異数プロイド、ポリプロイド、および染色体の再配置、転位、反転、欠失、および環染色体などの再調整が検出された。古典的な細胞遺伝学的観測55および物理マッピング56は、マラリア蚊の進化における全腕染色体転座の発生を実証した。そのため、微生物分解物質は、種間の染色体領域の相同性を比較するためにFISH実験と組み合わせて使用することができる。卵巣看護師細胞全体のポリテーン染色体に対する2Rペイントプローブで3D FISHを正常に行いました。この方法は、染色体軌道のトレースとアノフェレスの核建築を研究しやすくします。DNAジェノタイピング57、58、次世代ゲノムシーケンシング26、27、物理およびリンケージマップの開発、染色体絵画33、59のプローブの生成などの技術は、すべて腕および領域特異的な微小解剖の恩恵を受けることができます。 LCM技術を組み合わせ、単一細胞WGAを進めることで、単一ポリテーン染色体アームから妥当な量のDNAを生成する効率的で実用的な方法を実証しています。
開示事項
著者らは開示するものは何もない。
謝辞
この研究は、国立衛生研究所1R21AI094289からイゴールV.シャラホフへの助成金によって支えられた
資料
| Name | Company | Catalog Number | Comments |
| Acetic acid | Fisher Scientific | A491-212 | |
| Methanol | Fisher Scientific | A412-4 | |
| Propionic acid | Sigma-Aldrich | 402907 | |
| Membrane slides 1.0 PET | Zeiss | 415190-9051-000 | |
| Buffer tablets “GURR” | Life Technologies | 10582-013 | |
| KaryoMAX Giemsa Stain | Life Technologies | 10092-013 | |
| ThermoBrite Slide Denaturation/Hybridization System | Abbott Molecular | 30-144110 | |
| Vacufuge vacuum concentrator | Eppendorf | 22820001 | |
| Spectroline Microprocessor-Controlled UV Crosslinker XL-1000 | Fisher Scientific | 11-992-89 | |
| Thermo Scientific NanoDrop | Fisher Scientific | ND-2000 | |
| REPLI-g Single Cell Kit | Qiagen | 150343 | |
| GenomePlex Single Cell Kit (WGA4) | Sigma-Aldrich | WGA4-10RXN | |
| GenomePlex WGA Reamplification Kit (WGA3) | Sigma-Aldrich | WGA3-50RXN | |
| MZ6 Leica stereomicroscope | Leica | VA-OM-E194-354 | A different stereomicroscope can be used |
| Olympus CX41 Phase Microscope | Olympus | CX41RF-5 | A different phase microscope can be used |
| PALM MicroBeam Laser Microdissection Microscope | Zeiss | ||
| Thermomixer | Eppendorf | 22670000 | |
| 10x PBS | Invitrogen | P5493 | |
| 50x Denhardt’s solution | Sigma-Aldrich | D2532 | |
| 99% Formamide | Fisher Scientific | BP227500 | |
| Dextran sulfate sodium salt | Sigma | D8906 | |
| 20x SSC buffer | Invitrogen | AM9765 | |
| ProLong Gold antifade reagent with DAPI | Invitrogen | P-36931 | |
| dATP, dCTP, dGTP, dTTP | Fermentas | R0141, R0151, R0161, R0171 | |
| Cy3-dUTP, Cy5-dUTP | GE Healthcare | PA53022, PA55022 | |
| BSA | Sigma-Aldrich | A3294 | |
| DNA polymerase I | Fermentas | EP0041 | |
| DNase I | Fermentas | EN0521 | |
| Spermine | Sigma-Aldrich | S3256-1G | |
| Spermidine | Sigma-Aldrich | S0266-1G | |
| Potassium Chloride (KCl) | Fisher Scientific | BP366-500 | |
| Sodium Chloride (NaCl) | Fisher Scientific | BP3581 | |
| EGTA | Sigma-Aldrich | E0396-25G | |
| PIPES | Sigma-Aldrich | P6757-25G | |
| EDTA | Fisher Scientific | S311-500 | |
| Digitonin | Sigma-Aldrich | D141-100MG | |
| Triton-X100 | Fisher Scientific | BP151-100 | |
| AdhesiveCap 500 clear | Zeiss | 415190-9211-000 | |
| QIAamp DNA Micro Kit (50) | Qiagen | 56304 | |
| Genomic DNA Clean and Concentrator Kit | Zymo Research | D4010 | |
| 37% Paraformaldehyde | Fisher Scientific | F79-500 |
参考文献
- Ried, T., Schrock, E., Ning, Y., Wienberg, J. Chromosome painting: a useful art. Human molecular genetics 7. , 1619-1626 (1998).
- Yang, F., et al. Reciprocal chromosome painting illuminates the history of genome evolution of the domestic cat, dog and human. Chromosome Res. 8, 393-404 (2000).
- Badenhorst, D., Dobigny, G., Robinson, T. J. Karyotypic evolution of hapalomys inferred from chromosome painting: a detailed characterization contributing new insights into the ancestral murinae karyotype. Cytogenet Genome Res 136. , 83-88 (2012).
- Trifonov, V. A., et al. Chromosomal evolution in Gekkonidae. I. Chromosome painting between Gekko and Hemidactylus species reveals phylogenetic relationships within the group. Chromosome Res. 19, 843-855 (2011).
- Nie, W., et al. Chromosomal rearrangements and karyotype evolution in carnivores revealed by chromosome painting. Heredity (Edinb. , 108-1017 (2012).
- Guan, X. Y., et al. Detection of chromosome 6 abnormalities in melanoma cell lines by chromosome arm painting probes). Cancer Genet Cytogenet. 107, 89-92 (1998).
- Guan, X. Y., et al. Characterization of a complex chromosome rearrangement involving 6q in a melanoma cell line by chromosome microdissection. Cancer genetics and cytogenetics 134. , 65-70 (2002).
- Breen, M., et al. Detection of equine X chromosome abnormalities in equids using a horse X whole chromosome paint probe (WCPP). Vet J. 153, 235-238 (1997).
- Kokhanenko, A. A., Anan'ina, T. V., Stegniy, V. N. The changes in chromosome 6 spatial organization during chromatin polytenization in the Calliphora erythrocephala Mg. (Diptera: Calliphoridae) nurse cells. Protoplasma 250. , 141-149 (2013).
- Timme, S., et al. Nuclear position and shape deformation of chromosome 8 territories in pancreatic ductal adenocarcinoma. Anal Cell Pathol (Amst. 34, 21-33 (2011).
- Drosopoulou, E., et al. Sex chromosomes and associated rDNA form a heterochromatic network in the polytene nuclei of Bactrocera oleae (Diptera: Tephritidae). Genetica. 140, 169-180 (2012).
- Pazian, M. F., Shimabukuro-Dias, C. K., Pansonato-Alves, J. C., Oliveira, C., Foresti, F. Chromosome painting of Z and W sex chromosomes in Characidium (Characiformes). Crenuchidae). Genetica. 141, 1-9 (2013).
- Howe, E. S., Murphy, S. P., Bass, H. W. Three-dimensional acrylamide fluorescence in situ hybridization for plant cells. Methods Mol Biol 990. , 53-66 (2013).
- Lysak, M. A., Mandakova, T. Analysis of plant meiotic chromosomes by chromosome painting. Methods Mol Biol 990. , 13-24 (2013).
- Ji, Z., Zhang, L. Chromosomics: detection of numerical and structural alterations in all 24 human chromosomes simultaneously using a novel OctoChrome FISH assay. J Vis Exp. 10, (2012).
- Idziak, D., et al. Painting the chromosomes of Brachypodium: current status and future prospects. Chromosoma. 120, 469-479 (2011).
- Fuchs, J., Kuhfittig, S., Reuter, G., Schubert, I. Chromosome painting in Drosophila. Chromosome Res. 6, 335-336 (1998).
- Zhimulev, I. F. . Morphology and structure of polytene chromosomes. 34, 1-490 (1996).
- Sharakhov, I. V., Sharakhova, M. V. in Chromosome Mapping Research Developments eds. J.F. Verrity & L.E. Abbington) (Nova Science. , (2008).
- Coluzzi, M., Sabatini, A., Torre, d. e. l. l. a., Di Deco, A., A, M., Petrarca, V. A polytene chromosome analysis of the Anopheles gambiae species complex). Science. 298, 1415-1418 (2002).
- Stegnii, V. N. Systemic reorganization of the architectonics of polytene chromosomes in the onto- and phylogenesis of malaria mosquitoes. Genetika. 23, 821-827 (1987).
- Stegnii, V. N. Systemic reorganization of the architectonics of polytene chromosomes in the onto- and phylogenesis of malarial mosquitoes. II. Species specificity in the pattern of chromosome relations with the nuclear envelope of nutrient ovarian cells. Genetika. 23, 1194-1199 (1987).
- Bonaccorsi, S., Santini, G., Gatti, M., Pimpinelli, S., Colluzzi, M. Intraspecific polymorphism of sex chromosome heterochromatin in two species of the Anopheles gambiae complex. Chromosoma. 76, 57-64 (1980).
- Zhimulev, I. F. Polytene chromosomes, heterochromatin, and position effect variegation. Adv Genet. 37, 1-566 (1998).
- Seifertova, E., et al. Efficient high-throughput sequencing of a laser microdissected chromosome arm. BMC Genomics. 14, 1471-2164 (2013).
- Weise, A., et al. High-throughput sequencing of microdissected chromosomal regions. European journal of human genetics. EJHG. 18, 457-462 (2010).
- Murphy, S. J., et al. Mate pair sequencing of whole-genome-amplified DNA following laser capture microdissection of prostate cancer. DNA research : an international journal for rapid publication of reports on genes and genomes 19. , 395-406 (2012).
- Moshkin, Y. M., et al. Microdissection and sequence analysis of pericentric heterochromatin from the Drosophila melanogastermutant Suppressor of Underreplication. Chromosoma. 111, 114-125 (2002).
- Decarlo, K., Emley, A., Dadzie, O. E., Laser Mahalingam, M. capture microdissection: methods and applications. Methods Mol Biol 755. , 1-15 (2011).
- Iyer, E. P., Cox, D. N. Laser capture microdissection of Drosophila peripheral neurons. J Vis Exp. 10, (2010).
- Kubickova, S., Cernohorska, H., Musilova, P., Rubes, J. The use of laser microdissection for the preparation of chromosome-specific painting probes in farm animals. Chromosome Res. 10, 571-577 (2002).
- Fukova, I., et al. Probing the W chromosome of the codling moth, Cydia pomonella, with sequences from microdissected sex chromatin. Chromosoma. 116, 135-145 (2007).
- Thalhammer, S., Langer, S., Speicher, M. R., Heckl, W. M., Geigl, J. B. Generation of chromosome painting probes from single chromosomes by laser microdissection and linker-adaptor PCR. Chromosome Res. 12, 337-343 (2004).
- Sims, C. E., Allbritton, N. L. Analysis of single mammalian cells on-chip. Lab on a chip 7. , 423-440 (2007).
- Hutchison, C. A., 3rd, J. C., Venter, Single-cell genomics. Nature biotechnology. 24, 657-658 (2006).
- Lasken, R. S., Egholm, M. Whole genome amplification: abundant supplies of DNA from precious samples or clinical specimens. Trends in biotechnology 21. , 531-535 (1016).
- Teruel, M., et al. Microdissection and chromosome painting of X and B chromosomes in the grasshopper Eyprepocnemis plorans. Cytogenet Genome Res. 125, 286-291 (2009).
- Liu, J. D., et al. Sex chromosomes in the spiny eel (Mastacembelus aculeatus) revealed by mitotic and meiotic analysis. Cytogenet Genome Res. 98, 291-297 (2002).
- Harvey, S. C., et al. Molecular-cytogenetic analysis reveals sequence differences between the sex chromosomes of Oreochromis niloticus: evidence for an early stage of sex-chromosome differentiation. Cytogenet Genome Res. 97, 76-80 (2002).
- Hockner, M., Erdel, M., Spreiz, A., Utermann, G., Kotzot, D. Whole genome amplification from microdissected chromosomes. Cytogenet Genome Res. 125, 98-102 (2009).
- Kitada, K., Taima, A., Ogasawara, K., Metsugi, S., Aikawa, S. Chromosome-specific segmentation revealed by structural analysis of individually isolated chromosomes. Genes Chromosomes Cancer. 50, 217-227 (2011).
- Ma, L., et al. Direct determination of molecular haplotypes by chromosome microdissection. Nature methods. 7, 299-301 (2010).
- Teruel, M., et al. Microdissection and chromosome painting of X and B chromosomes in Locusta migratoria. Chromosome Res. 17, 11-18 (2009).
- Treff, N. R., Su, J., Tao, X., Northrop, L. E., Scott, R. T. Single-cell whole-genome amplification technique impacts the accuracy of SNP microarray-based genotyping and copy number analyses. Molecular human reproduction 17. , 335-343 (2011).
- Rutten, K. B., et al. Chromosomal anchoring of linkage groups and identification of wing size QTL using markers and FISH probes derived from microdissected chromosomes. in Nasonia(Pteromalidae : Hymenoptera). Cytogenetic and Genome Research 105. , 126-133 (2004).
- Post, R. J., Kruger, A., Somiari, S. B. Laser-assisted microdissection of polytene chromosomes from Diptera for the development of molecular markers. Molecular Ecology Notes. 6, 634-637 (2006).
- George, P., Sharakhova, M. V., Sharakhov, I. V. High-throughput physical mapping of chromosomes using automated in situ hybridization. Journal of visualized experiments : JoVE. 10, (2012).
- Timoshevskiy, V. A., Sharma, A., Sharakhov, I. V., Sharakhova, M. V. Fluorescent in situ Hybridization on Mitotic Chromosomes of Mosquitoes. J Vis Exp. 10, (2012).
- Frumkin, D., et al. Amplification of multiple genomic loci from single cells isolated by laser micro-dissection of tissues. BMC biotechnology. 8, 10-1186 (2008).
- Raghunathan, A., et al. Genomic DNA amplification from a single bacterium. Applied and environmental microbiology 71. , 3342-3347 (2005).
- Cassio, D. Long term culture of MDCK strains alters chromosome content. BMC Res Notes. 6, 162-1610 (2013).
- Landry, J. J., et al. The Genomic and Transcriptomic Landscape of a HeLa Cell Line. G3. , (1534).
- Steiniger, G. E., Mukherjee, A. B. Insect chromosome banding: technique for G- and Q-banding pattern in the mosquito Aedes albopictus. Can J Genet Cytol. 17, 241-244 (1975).
- Brown, S. E., et al. Toward a physical map of Aedes aegypti. Insect Mol Biol. 4, 161-167 (1995).
- Green, C., Hunt, R. Interpretation of variation in ovarian polytene chromosomes of Anopheles funestus Giles. A. parensis Gillies, and A. aruni? Genetica. 51, 187-195 (1980).
- Sharakhova, M. V., Xia, A., Leman, S. C., Sharakhov, I. V. Arm-specific dynamics of chromosome evolution in malaria mosquitoes. BMC evolutionary biology. 11, 10-1186 (2011).
- Gu, L. H., et al. DNA genotyping of oral epithelial cells by laser capture microdissection]. Fa yi xue za zhi 22. , 196-197 (2006).
- Rook, M. S., Delach, S. M., Deyneko, G., Worlock, A., Wolfe, J. L. Whole genome amplification of DNA from laser capture-microdissected tissue for high-throughput single nucleotide polymorphism and short tandem repeat genotyping. The American journal of pathology 164. , 23-33 (2004).
- Houen, A., Field, B. L., Saunders, V. A. Microdissection and chromosome painting of plant B chromosomes. Methods in cell science : an official journal of the Society for In. Vitro Biology. 23, 115-124 (2001).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved