Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes de fluorescencia con un nanómetros Precisión (FIONA)
En este artículo
Resumen
Fluoróforos individuales pueden ser localizados con precisión nanométrica utilizando FIONA. Aquí se reporta un resumen de la técnica de FIONA, y la forma de llevar a cabo experimentos de FIONA se describe.
Resumen
Imágenes de fluorescencia con una precisión de un nanómetro (FIONA) es una técnica sencilla pero útil para la localización de fluoróforos individuales con precisión nanométrica en el plano xy. Aquí un resumen de la técnica FIONA se informa y ejemplos de investigaciones que se han realizado utilizando FIONA se describen brevemente. En primer lugar, cómo configurar el equipo necesario para los experimentos de FIONA, es decir, un microscopio de fluorescencia de reflexión interna total (TIRFM), con detalles sobre la alineación de la óptica, se describe. Entonces cómo llevar a cabo un sencillo experimento FIONA en la localización de moléculas individuales de ADN-Cy3 inmovilizada usando protocolos apropiados, seguido por el uso de FIONA para medir el tamaño de paso de 36 nm de un solo miosina Va motor truncada marcada con un punto cuántico, se ilustra. Por último, se informó reciente esfuerzo para extender la aplicación de FIONA para muestras gruesas. Se muestra que, utilizando un objetivo de inmersión en agua y puntos cuánticos empapado profundamente en sol-geles oculares y córneas de conejo (>200 micras), la localización de precisión de 2-3 nm se puede lograr.
Introducción
Alrededor de 1882, Ernst Abbe encontró que la resolución de un microscopio de luz visible es de ~ λ / 2NA, o ~ 200 nm (donde λ es la longitud de onda y NA es la apertura numérica) 1,2. Por lo tanto cualquier objeto más pequeña que esta dimensión aparecería como un punto de difracción limitada en un microscopio óptico. Sin embargo, es posible determinar el centro de la mancha, es decir, la ubicación del objeto, con una precisión mucho más alta 3. Imágenes de fluorescencia con una precisión de un nanómetro (FIONA) es una técnica sencilla pero útil para la localización de fluoróforos individuales con precisión nanométrica en el plano xy 4. La precisión de la localización, σ μ (es decir, el error estándar de la media), depende del número total de fotones recogidos, 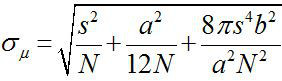 , Donde N es el recuento de fotones, s es la desviación estándar de la mancha fluorescente, a esel tamaño de píxel del detector de formación de imágenes, y b es la desviación estándar de los antecedentes 3,4. Para un fluoróforo que emite fotones ~ 10.000, Fiona puede alcanzar ~ 1 nm de precisión 4.
, Donde N es el recuento de fotones, s es la desviación estándar de la mancha fluorescente, a esel tamaño de píxel del detector de formación de imágenes, y b es la desviación estándar de los antecedentes 3,4. Para un fluoróforo que emite fotones ~ 10.000, Fiona puede alcanzar ~ 1 nm de precisión 4.
FIONA se puede utilizar para determinar con precisión la posición de un emisor fijo, o uno móvil (suponiendo que las imágenes pueden tomarse lo suficientemente rápido). FIONA se puede aplicar secuencialmente a las tramas de la película y por lo tanto seguir el movimiento de la molécula única de 4 8. Reactivos fotoprotectores pueden ser necesarias para asegurar que la muestra no fotodegrada. Además, el objeto fluorescente en sí puede ser de cualquier tamaño, menor o mayor que la de difracción LIMIT- por ejemplo, puede consistir de un orgánulo (~ 1 m) con muchas proteínas fluorescentes dispersos en su membrana. Usando FIONA todavía puede producir un (nanómetros) promedio muy precisa de su centro de masa media. La gran mejora en la precisión de localización por FIONA permite resolver nanomemovimientos ter-escala en el tiempo. Esto ha empujado a la microscopía en la escala de longitud molecular 4 8.
Desde su invención, se han desarrollado variantes de FIONA. Por ejemplo, las imágenes de campo claro con exactitud de un nanómetro (bFIONA) 9, una ligera variante de FIONA, imágenes y localiza objetos densos como melanosomas in vivo (objetos oscuros que contienen el pigmento melanina) con luz transmitida. Además, Fiona se ha empleado para resolver múltiples colorantes. Por ejemplo, una sola molécula de alta resolución de imagen con fotoblanqueo (camarones) 10,11 o de una sola molécula de alta resolución colocalización (SHREC) 12 se han desarrollado para resolver dos colorantes dentro de aproximadamente 10 nm. (Tenga en cuenta que esta es la resolución, es decir, con qué precisión se puede saber colorantes idénticos aparte.) Más recientemente, el análisis FIONA ha contribuido al proceso de localización de cierta super-resolución de microscopía óptica como reco estocásticomicroscopía nstruction (TORMENTA) 13 y 15 y la localización microscopía foto-activado (PALMA) 16, en el que fluoróforos oscuros temporales están entusiasmados, y luego la fluorescencia se localiza. Por repetidamente emocionante bastante baja densidad de tintes (menos de uno por difracción limitado punto), y luego recoger la fluorescencia, el análisis de cada uno de ellos por Fiona, se puede construir un mapa de alta resolución. La resolución está a continuación, sólo limitado por el número de fotones cada colorante pone a cabo, así como cosas como mantener estacionaria la muestra (incluyendo, por ejemplo, la platina del microscopio) durante la adquisición.
En este trabajo, un resumen de la técnica FIONA y describa brevemente ejemplos de investigaciones que se han realizado utilizando FIONA se informó. En primer lugar, cómo configurar el equipo necesario para los experimentos de FIONA, es decir, un microscopio de fluorescencia de reflexión interna total (TIRFM), con detalles sobre la alineación de la óptica, se describe. Entonces, ¿cómollevar a cabo un sencillo experimento FIONA sobre la localización de las moléculas individuales de ADN-Cy3 inmovilizada utilizando protocolos apropiados, se ilustra. Después de eso, el uso de FIONA para medir el tamaño de paso de 36 nm de un solo miosina Va motor truncada marcada con un punto cuántico se presenta. La miosina Va es una proteína motora processive esencial que transporta carga celular mientras que a lo largo de los filamentos de actina translocación. Aquí una miosina Va construir truncado se utiliza para eliminar dominios irrelevantes para el tamaño del paso, y con una etiqueta FLAG añadió a la C-terminal para permitir la facilidad de etiquetado con puntos cuánticos funcionalizados con anticuerpos anti-FLAG. Este experimento se realiza en condiciones de baja ATP para ralentizar la miosina y permitir el uso de tiempos de exposición suficientemente largo para conseguir un buen recuento de fotones en cada fotograma. Cualquier marcador fluorescente suficientemente brillante podría ser sustituido en el siguiente protocolo. Por último, se informó reciente esfuerzo de extender la aplicación de FIONA para muestras gruesas. Como prueba de principio-, se empaparon los puntos cuánticosen sol-geles y las córneas de los ojos de conejo y luego fotografiada y localizada usando FIONA. Para imágenes, un objetivo de inmersión 60X agua con NA = 1,2 fue utilizado porque este objetivo tiene una distancia de trabajo de más de 100X objetivo de inmersión en aceite utilizado anteriormente. Para compensar la pérdida en la escala de reproducción en el objetivo, un objetivo extra-ampliación (3.3X o 4,0X) fue colocado en la trayectoria de emisiones. Además, epi-fluorescencia (no TIR) necesita microscopía para ser utilizado para acceder a regiones profundas en las muestras gruesas. Se demuestra que los puntos cuánticos empapados profundo de sol-gel y en las córneas de los ojos de conejo (Z> 200 m) se pueden localizar con 2-3 nm precisión.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Declaración de Ética: El tejido de la córnea de conejos se recogió de acuerdo con la Universidad de Illinois Institucional Cuidado de Animales y el uso de directrices.
Configuración 1. TIRFM
NOTA: Use gafas láser de seguridad todo el tiempo.
- Asegúrese de que todos los componentes ópticos necesarios que figuran en la Lista de Materiales están disponibles y listos para la alineación. Si es necesario, use sustitutos con funciones equivalentes de otras compañías. Asegúrese de que los espejos y las lentes deben tener (AR) recubrimientos antirreflectantes que coinciden con el láser en uso.
- Establecer las alturas de todos los componentes ópticos a la altura del centro del microscopio de nuevo puerto.
- Montar el láser, disparo de láser y el filtro ND (s). Uso de filtros ND para atenuar la potencia del láser hacia abajo tan bajo como sea posible mientras se mantiene el haz visible. Apriete los tornillos con llaves hexagonales apropiadas.
- Planee una ruta de viga y marcarlo con cinta o marcador en la mesa óptica (puntoted líneas azules en la Figura 1). Para simplificar, mantener caminos rectos a lo largo de las líneas de agujeros en la mesa óptica.
- Lugar espejo M1 (Figura 1) en el primer giro en ángulo recto. Coloque los dos iris lo largo de la segunda sección recta de la trayectoria del haz planeado. Ajustar la posición y la inclinación de M1 tal que el láser pasa a través de los iris.
- Lugar espejo M2 (Figura 1) en el segundo giro en ángulo recto. Coloque el expansor de haz de 10X (L1 y L2, la Figura 1) a lo largo de la tercera sección recta de la trayectoria. Ajustar su inclinación tal que el expansor de haz es paralelo tanto a la mesa óptica y la trayectoria del haz planeado.
- Ajuste M1 y M2 iterativamente de manera que el láser va recto a través de los centros de ambas lentes del expansor de haz. Repita este paso hasta que el perfil de la viga de la viga ampliado no es recortado de Gauss.
- Ajuste M1 para centrar el haz en L1 (Fifigura 1) y M2 para centrar el haz sobre L2 (Figura 1) de manera iterativa. Un perfil del haz mala por lo general significa que el láser se recorta; usar un pedazo de papel blanco para bloquear el haz después del expansor de haz para comprobar el perfil del haz con los ojos. Para el análisis de alta precisión, utilizar un perfilador haz óptico.
- Ajustar la distancia entre L1 y L2 de tal manera que el haz se colima. Repita el paso 1.8 y 1.9 si es necesario.
NOTA: Cuando el tamaño del haz no cambia con la distancia, el haz colimado es suficiente para una configuración TIRFM. Para mejorar aún más la colimación del haz, se podrían utilizar herramientas tales como un interferómetro de cizalladura. Un típico tamaño del haz después de la expansión es de ~ 20 mm. - Obturador del láser. Aflojar el objetivo del microscopio y el tornillo en un objetivo de alineación fluorescente. Lugar espejos M3 y M4 (Figura 1) para dirigir el haz expandido en el puerto de microscopio y sobre el espejo dicroico dentro de la torreta. Asegúrese de que el rayo láser rebota en tél dicroico y hacia el techo.
- Ajuste M3 para centrar la parte más brillante del haz sobre el blanco fluorescente, y M4 para ajustar la inclinación del haz de ser vertical.
- Obturador del láser y atornillar el objetivo de nuevo. Si la alineación en el paso anterior se hace bien debe haber un punto simétrico de salir del objetivo. Perfeccionar las inclinaciones de M3 y M4 para optimizar la potencia del láser y perfil del haz fuera del objetivo.
- Montar una cámara EMCCD al microscopio y conecte la cámara a un ordenador. Inicie el software de la cámara.
- Montar una muestra fluorescente (solución de fluoróforos) en el microscopio. Mira la mancha fluorescente brillante en la cámara. Compruebe que el punto no se desplaza en la pantalla a medida que cambia el enfoque.
- Coloque la lente TIR (L3, Figura 1) en la etapa de traducción XYZ a una distancia desde el plano focal posterior del objetivo que es igual a la longitud focal de L3 (~ 30 cm). Ajuste la posición de L3de tal manera que el láser pasa a través del centro de la lente.
- Traducir L3 a lo largo de la trayectoria del haz para ajustar la colimación del haz. Asegúrese de que el haz se sigue centrada en el monitor y simétrica en forma.
NOTA: El área de iluminación en el plano de la muestra aumenta con la disminución de la lente TIR longitud focal. En general, utilice la longitud focal más pequeño que podría caber en el set-up. - Traducir L3 perpendicular a la trayectoria del haz para inclinar el haz fuera del objetivo. Mantener la traducción de la lente TIR TIR de tal manera que se consigue. Observar una muestra de perlas fluorescentes a través de la cámara EMCCD y L3 ajustar para obtener una buena SNR.
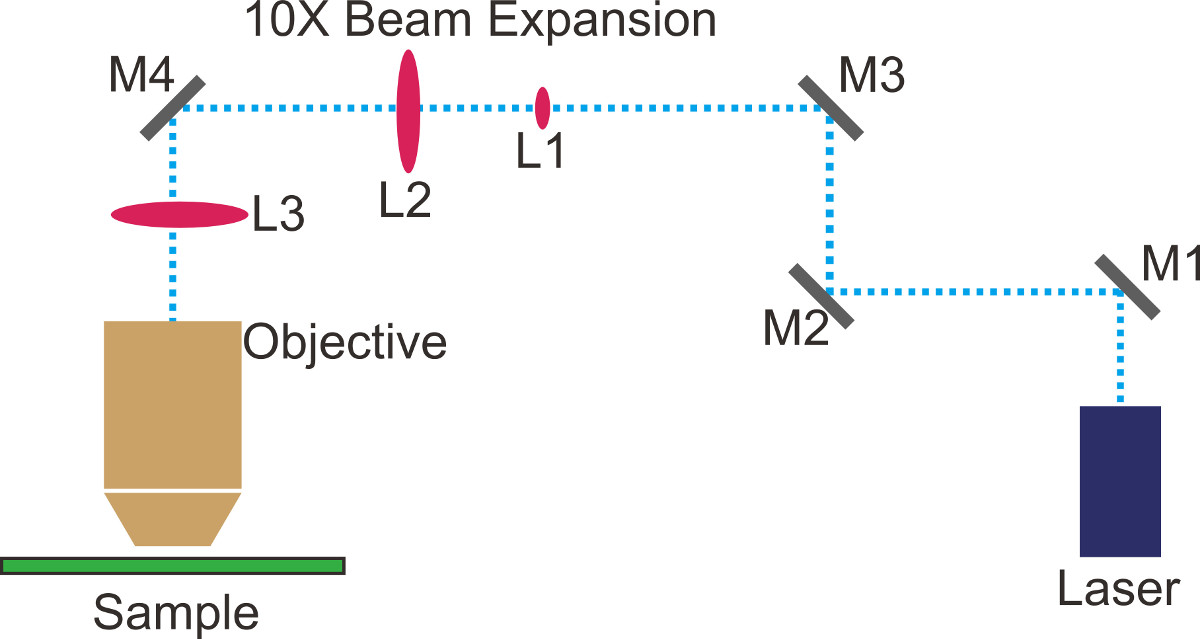
Figura 1. configuración óptica para la microscopía de reflexión interna total de fluorescencia (TIRFM).
2. FIONA en Cy3-ADN
- Cdiapositivas de microscopio y cubreobjetos magras: Limpiar los portaobjetos y cubreobjetos con ddH2O e isopropanol y secarlos con nitrógeno gaseoso; Colocar los portaobjetos y cubreobjetos en el limpiador de plasma durante 5 min bajo argón plasma.
- Construir cámaras de muestra (como esbozado en la Figura 2).
- Coloque un pañuelo en el banco y luego poner un pedazo de papel de la lente en la parte superior del tejido. Posicionar el cursor en el papel de la lente. Asegúrese de que el lado limpio de la diapositiva es hacia arriba.
- Aplicar dos tiras de cinta de doble cara sobre el portaobjetos a lo largo de los bordes largos, dejando un espacio de 3-5 mm en el centro. Coloque el cubreobjetos limpiado en la parte superior de la corredera. Asegúrese de que el lado limpio de la cubreobjetos se enfrenta a la diapositiva.
- Utilice una punta de pipeta para presionar hacia abajo sobre la cinta de doble cara. Utilice una maquinilla de afeitar para eliminar el exceso de cinta de la diapositiva para que la cinta sigue siendo sólo bajo el cubreobjetos.
NOTA: Los extremos abiertos de la cámara se dejan abiertas y sirven como entrada y salidadejar. El volumen de la cámara es de varios microlitros.

Figura 2 Esquema de una cámara de muestra típica (a) Vista superior.; (B) Vista lateral desde la derecha; (C) La vista lateral de la parte delantera.
- Inmovilizar Cy3-ADN en las superficies interiores de las cámaras de muestra.
- Preparar T-50 tampón (10 mM Tris-HCl pH 8,0, NaCl 50 mM). Preparar BSA-biotina en T-50 a una concentración final de 1 mg / ml. Preparar tampón T50-BSA por disolución de BSA en T-50 a una concentración final de 10 mg / ml.
- Preparar 0,5 mg / ml en tampón de neutravidina T50-BSA. Preparar biotinilado Cy3 ADN marcado (Cy3-DNA) en T50-BSA a una concentración final de 5-10 pM.
- Pipetear 10 l de BSA-biotina (1 mg / ml) en la cámara de muestra. Espere 5 min.
- Lave la cámara con 40 l T50-BSA. Pipetear 10 l neutravidina (0,5 mg / ml) en la cámara de muestra. Incubar durante 5 min.
- Lave la cámara con 40 l T50-BSA. Colocar 20 l Cy3-ADN en la cámara de muestras. Incubar durante 5 min y luego lavar la cámara con 80 l T50-BSA.
- Imagen Cy3-ADN moléculas individuales bajo TIRFM.
- Preparar buffer de imágenes (100 l) mediante la mezcla de 1 l protocatecuato-3,4-dioxigenasa (PCD, 5 M), 4 l de ácido protocatéquico (PCA, 62,5 mM), 50 l 6-hidroxi-2,5,7,8- tetramethylchromane-2-carboxílico (Trolox, 2 mM en T-50), y 45 l T50-BSA.
- Pipetear en buffer de imágenes 30 l y esperar 8-10 min.
- Montar la muestra para formación de imágenes en un TIRFM que está equipado con un láser verde (532 nm), un objetivo de inmersión en aceite 100X (1,45 NA), y una cámara de EMCCD.
- Ajuste el tiempo de exposición a 100 a 500 ms y EM ganancia para 25 a 100 Adquirir una película de la muestra de 1,000 marcos.
- Realizar análisis de datos FIONA en las imágenes grabadas de Cy3-ADN.
- Determinar el tamaño efectivo de píxel (es decir, factor de conversión de píxeles a nanómetros) dividiendo el tamaño de píxel físico (leída de la hoja de especificaciones de la cámara EMCCD) por el aumento total (el aumento del objetivo de microscopio multiplicado por cualquier aumentos adicionales).
- Determinar el factor de conversión de intensidades de los píxeles de fotones números dividiendo sensibilidad CCD (es decir, electrones por conteo A / D, leer la hoja de especificaciones de la cámara CCD) por el multiplicador de electrones (EM) ganancia utilizada durante la adquisición de imágenes.
- Compila y ejecuta FIONA.pro para el análisis FIONA. Utilice este programa IDL para importar la imagen adquirida, para introducir el tamaño de los píxeles efectivos (del Paso 2.5.1) y el factor de conversión de intensidad para el número de fotones (de la etapa 2.5.2), y elegir los puntos para el análisis de FIONA.
NOTA: Al final, el program voluntad de salida de los resultados apropiados con funciones gaussianas 2D, así como el número total de fotones y la precisión de la localización. Un resultado típico se muestra en la sección de los resultados y las figuras representativas 4c-4d. - Compila y ejecuta phcount.pro caracterizar recuento de fotones. Utilice este programa IDL para medir el número medio de fotones emitidos por un fluoróforo antes de photobleaching, para importar la imagen adquirida y para introducir el factor de conversión de intensidad a número de fotones (de la etapa 2.5.2).
NOTA: El programa detectará manchas fluorescentes automáticamente (selección manual es una opción), el cálculo de recuentos de fotones en función del número de cuadro, y la salida de los restos de recuentos de fotones.- Deseche malas trazas y especificar rangos marco después de photobleaching para la corrección de la línea base. Al final, el programa de salida de una lista de números totales de fotones para todos los puntos no se descarta. Se representa la distribución de los números de fotones y adaptarse a la distribuciónción con un decaimiento exponencial para obtener el número medio de fotones. Un resultado típico se muestra en la sección de resultados representativos y las figuras 4e-4f.
3. FIONA Aplicada para cuantificar Motor (por ejemplo, la miosina en la actina) Dinámica a escala del nanómetro
- Polimerizar la actina (es decir, preparar a la actina F) un día antes de FIONA experimento.
- Reconstituir G-actina (monómero) y biotina G-actina (monómero) a 10 mg / ml con tampón de actina general. Revuelva para asegurarse de que ambos se disuelve por completo, y mantener tanto en el hielo.
- Mezclar 10 l G-actina (monómero) con 1,7 l de biotina G-actina en un tubo de 1.5 ml. Añadir 100 l de tampón de hielo frío polimerización de la actina.
- Deje la mezcla durante la noche a 4 ° C (F-actina formado) y luego añadir ddH2O hasta un volumen total de 1 ml.
- Guarde los filamentos de actina (F-actina) a 4 ° C para su uso posterior en los experimentos.
NOTA: Filamentos se desintegrará y acortar el tiempo, pero se puede utilizar durante al menos dos semanas.
- Preparar la muestra para la imagen.
- Hacer una cámara de muestra (como se describe en el Protocolo 2.1 y 2.2). Pipetear en 20 l de BSA biotinilada a 1 mg / ml en ddH2O Incubar durante 10 min. Enjuagar con 30 l ddH 2 O.
NOTA: Esto bloquea la superficie de cristal y establece biotina para la unión de los filamentos de actina. Biotinilado poli-L-lisina - polietilenglicol (PEG-PLL) podría servir la misma función. - Pipetear en 0,5 mg / ml neutravidina. Incubar durante 2 minutos y luego lavar la cámara con tampón M5 30 l.
- Pipetear en el F-actina preparado diluyó 25 veces en tampón de actina en general, a una concentración final ~ 0,004 mg / ml. Espere 10 minutos, enjuague la cámara con 30 l de tampón.
- Diluir la miosina Va con las etiquetas FLAG 30 veces en tampón de M5 (HEPES 20 mM (pH 7,6), 2 mM de MgCl2, 25 mM KCl, 1 mM EGTA) a una concentración final of 250 nM. Mezclar 1 l miosina con 1 l anti-FLAG-Qdot705 (~ 1 M, conjugado de anticuerpos anti-FLAG y Qdot705 utilizando la conjugación Kit Qdot705 anticuerpo de acuerdo con el manual de instrucciones del fabricante). Añadir en 8 l M5 para llenar a 10 l. Pipetear hacia arriba y abajo para mezclar bien. Incubar durante 10 min en hielo.
NOTA: Esto produce una mezcla de 1 motor a 4 puntos cuánticos, en ~ 25 nM concentración de miosina.
- Hacer una cámara de muestra (como se describe en el Protocolo 2.1 y 2.2). Pipetear en 20 l de BSA biotinilada a 1 mg / ml en ddH2O Incubar durante 10 min. Enjuagar con 30 l ddH 2 O.
- Obtención de imágenes de la miosina caminando sobre la actina.
- Preparar buffer de imágenes (100 l) mediante la mezcla de 84 l M5-BSA (tampón M5 con 1 mg / ml de BSA), 1 l ATP (50 mM en ddH 2 O), 2 l de DTT (500 mM en ddH 2 O), 1 l CK (500 U / ml), 5 l CP (200 mM), 1 l PCD, 4 l PCA, 1 l de miosina-Qdot diluyó después otra 10 veces a 2,5 nM concentración de miosina, y 1 l BME.
- Pipetear en buffer de imágenes 20 l para probar la cámara y se incuba durante 8-10 min.
- Imagen de la muestra de TMicroscopio IRF a 30 ms de exposición. Adquirir un mínimo de 1.000 marcos. Ajuste el volumen de la miosina-Qdot en el Paso 3.3.1 si es necesario.
- Realizar el análisis de datos y encontrar el tamaño de paso de la miosina a pie.
- Abra el archivo de vídeo en ImageJ 17 y recortar el vídeo en torno a un punto móvil. Recortar un área lo suficientemente grande que el lugar nunca llega dentro de 20 píxeles del borde, y asegúrese de que no hay otros lugares en el video. Asegúrese de que este punto se mueve en una trayectoria lineal.
- Realizar el seguimiento del punto a través del video para generar coordenadas xey a través del tiempo, en píxeles, mediante la aplicación de análisis de FIONA (Paso 2.5.3) para cada fotograma del vídeo.
- Convertir píxeles a nanómetros, como se describe en la sección anterior.
- Calcular el desplazamiento desde la posición inicial como una función del tiempo.
- Ejecutar la prueba t en el desplazamiento para obtener los pasos de la miosina a pie.
NOTA: El programa previsto t-test (step_t_test.zip) se codifica en IDL y en contraconsiste de 14 subprogramas de la carpeta.- Abra todas las subrutinas en IDL y compilar todo dos veces. A continuación, ejecute mtltyanalysis_ttest.pro y elija un archivo de texto que contiene sólo los datos de distancia en una sola columna. Un archivo de salida de Excel se generará, que contiene los datos en bruto, la forma, y el tamaño del paso.
- Elimine todos los valores cero de la columna de paso grande. Trazar la distribución de los tamaños de paso utilizando Origen o MATLAB. Montar un Gaussian al histograma.
NOTA: los tamaños de paso de cero valores necesitan ser suprimido porque un paso de cero significa que no hay paso se toma de la trama anterior. El accesorio da un pico alrededor de 36 nm (como se muestra en la Figura 5).
4. gruesa Preparación de muestras para FIONA
- Preparar los puntos cuánticos encapsulados en sol-gel.
- Mezclar 4,5 ml de TMOS, 1 ml ddH 2 O y 100 l de HCl (120 mM). Sonicar la mezcla en hielo durante 30 min en el limpiador ultrasónico specified en la Lista de Materiales (frecuencia = 40 kHz, calentador = off). Mezcle la solución cada 10 min.
- Diluir 1,5 l Qdot605 en 1,5 ml de HEPES 50 mM (pH 7,2). Mezclar la solución con 1,5 ml de TMOS de la etapa anterior.
- Vierta la mezcla en un plato con fondo de cristal. Selle el plato de fondo de vidrio con Parafilm y se almacena a 4 º C durante 1, 5 horas.
- Añadir 2 ml de 1% en PBS BME al plato de la muestra y se incuba a temperatura ambiente durante 30 min antes de la formación de imágenes.
- Preparar la muestra córnea manchada con puntos cuánticos.
NOTA: Conejo-ojos eran regalos de Dr. Marina Marjanovic.- Se separa la córnea de los ojos y lo dividirá en 3 mm x 3 mm pedazos.
- Diluir 1 l Qdot 605-estreptavidina en 1 ml de PBS. Incubar el tejido de la córnea con solución 1 nM a 4 qdots º C durante 1 hora. Lave el tejido con PBS.
- Tome un portaobjetos de vidrio limpio y un cubreobjetos # 1.5. Ponga 4 capas de cinta de doble cara en el portaobjetos de vidrio a lo largo del lado más largo, unnd introducir otras 4 capas de cinta de doble cara paralela a la cinta anterior y dejar un canal de aproximadamente 1 cm en el medio. Coloque el tejido de la etapa anterior en el centro del canal, y se cubre con un cubreobjetos.
- Presione suavemente en los lados del cubreobjetos para hacer que se pegue a las cintas. Moje el canal con 50 l de PBS antes de exponer.
- Puntos cuánticos de imagen en sol-gel y la córnea.
- Para FIONA imágenes en muestras gruesas, utilice un objetivo 60X de inmersión en agua con una distancia de trabajo de 0,27 mm, o un objetivo 60X de inmersión en agua con una distancia de trabajo de 0,28 mm.
- Montar la muestra en el microscopio. Ajuste de la lente TIRF tal que alcanza el modo de epi-fluorescencia (es decir, el haz de láser sale del objetivo con un ángulo contra el cubreobjetos). Inserte una lente de aumento adicional (3.3X o 4,0X).
- Mueva el plano focal del objetivo a una posición z deseada (por ejemplo,> 200 m). Graba imágenes de inmóvilpuntos cuánticos en la muestra.
- Realizar análisis FIONA como se describe en la sección 2.5 de este protocolo.
Access restricted. Please log in or start a trial to view this content.
Resultados
Una configuración típica TIRFM tipo objetivo se muestra en la Figura 3. En primer lugar, la muestra Cy3-ADN inmovilizado en la superficie fue fotografiada. Una imagen típica se muestra en la Figura 4a. La imagen fue tomada con el tiempo de exposición de 0,5 segundos, con el aumento de EM = 50 y la sensibilidad CCD = 12,13 para la cámara. La propagación--función de punto (PSF) de una sola molécula de Cy3-DNA se muestra en la Figura 4b (desde el punto indicado por...
Access restricted. Please log in or start a trial to view this content.
Discusión
FIONA es una técnica para localizar la posición de un emisor fluorescente (fluoróforo orgánico o punto cuántico) con una precisión nanométrica y resolución temporal a 1 mseg 4 8. Cuando se recojan suficientes fotones, esta técnica permite determinar la posición de un emisor fluorescente mucha más precisión que el límite de difracción (~ 200 nm) y por lo tanto esta técnica abre un camino para observar lo que no se ha visto con el microscopio óptico convencional / tradicional 4 - 8. D...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Este trabajo fue apoyado por el NIH subvenciones 068.625, becas NSF 1063188 y Centro de Física de Células Vivas 0822613. Un agradecimiento especial al Dr. Marina Marjanovic en Beckman Instituto de Ciencia y Tecnología Avanzada para el regalo de los ojos del conejo.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Double-sided tape | 3M | ~75 µm thick | |
| EMCCD camera | Andor Technology | DU-897E-CS0-#BV | |
| Ultrasonic cleaner | Branson | 2510 | |
| Fluorescence filter set | Chroma | 49016 | |
| Actin polymerization buffer | Cytoskeleton | BSA02 | |
| Biotin G-actin | Cytoskeleton | AB07 | |
| G-actin | Cytoskeleton | AKL95 | |
| General actin buffer | Cytoskeleton | BSA01 | |
| Laser shutter (with driver) | Electro-Optical Products Corp. | SH-10-MP | |
| IDL | Exelis Visual Information Solutions | ||
| Neutravidin | Fisher Scientific | PI-31000 | |
| Coverslip | Fisherbrand | 22X30-1.5 | 0.16-0.19 mm thick |
| Microscope slide | Gold Seal Microslides | 30103X1 | 0.93-1.05 mm thick |
| Plasma cleaner | Harrick Plasma | PDC-001 | |
| Glass bottom dish | In Vitro Scientific | D35-20-1.5-N | |
| Cy3-DNA oligos | Integrated DNA Technologies | 5'-Cy3/GCCTCGCTGCCGTCGCCA-3'Bio | |
| Fluorescent beads | Invitrogen | T-7280 | |
| Qdot 605-streptavidin | Invitrogen | Q10101MP | |
| Qdot605 | Invitrogen | Q21301MP | |
| Qdot705 | Invitrogen | Q22021MP | |
| Qdot705 Antibody Conjugation Kit | Invitrogen | Q22061MP | |
| MATLAB | MathWorks | ||
| Optical table | Newport Corp | RS4000 Series | |
| 60X Objective | Nikon | Plan Apo VC 60x WI | |
| 100X Objective | Olympus | PlanApo 100X/1.45 Oil ∞/0.17 | |
| 60X Objective | Olympus | UPlanApo 60X/1.20W | |
| Inverted microscope | Olympus | IX71/IX70/IX81 | |
| Origin | OriginLab | ||
| Anti-FLAG antibody | Sigma Aldrich | F7425-.2MG | |
| ATP | Sigma Aldrich | A7699 | |
| BME | Sigma Aldrich | 63689-25ML-F | |
| BSA | Sigma Aldrich | A7906 | |
| BSA-biotin | Sigma Aldrich | A8549-10MG | |
| CK | Sigma Aldrich | C3755 | Creatine Phosphokinase from rabbit muscle |
| CP | Sigma Aldrich | P1937 | Phosphocreatine di(tris) salt |
| DTT | Sigma Aldrich | 43815 | DL-Dithiothreitol |
| EGTA | Sigma Aldrich | E3889 | Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid |
| HCl | Sigma Aldrich | 93363-500G | |
| HEPES | Sigma Aldrich | H0887 | |
| KCl | Sigma Aldrich | P9333 | |
| MgCl2 | Sigma Aldrich | M1028 | |
| NaCl | Sigma Aldrich | S7653 | |
| PCA | Sigma Aldrich | 03930590 | Protocatechuic acid |
| PCD | Sigma Aldrich | P8279 | Protocatechuate-3,4-dioxygenase |
| TMOS | Sigma Aldrich | 341436-25G | Tetramethyl orthosilicate |
| Tris-HCl | Sigma Aldrich | 93363 | |
| Trolox | Sigma Aldrich | 238813 | 6-hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid |
| 1” diameter broadband dielectric mirrors with mounts | Thorlabs | BB1-E02, KM100 | Quantity: 2 |
| ½” diameter posts | Thorlabs | TR4 | Quantity ≥ 6 |
| 10X beam expander | Thorlabs | BE10M-A | |
| 2” diameter f = 300 mm lens with mount | Thorlabs | LA1256-A, LMR2 | TIR lens |
| Fluorescent alignment target | Thorlabs | VRC2SM1 | |
| Laser safety goggles | Thorlabs | LG3 | |
| ND filter(s) | Thorlabs | FW1AND | |
| Optical beam profiler | Thorlabs | BP209-VIS | |
| Post-mounted iris diaphragm | Thorlabs | ID25 | Quantity: 2 |
| Shearing interferometer | Thorlabs | SI100 | |
| XYZ translation stage, ½” travel | Thorlabs | T12XYZ | |
| Laser | World Star Technologies | TECGL-30 | 532 nm, 30 mW |
Referencias
- Abbe, E. The Relation of Aperture and Power in the Microscope. Journal of the Royal Microscopical Society. 2 (3), 300-309 (1882).
- Abbe, E. The Relation of Aperture and Power in the Microscope (continued). Journal of the Royal Microscopical Society. 2 (4), 460-473 (1882).
- Thompson, R. Precise Nanometer Localization Analysis for Individual Fluorescent Probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Yildiz, A., et al. Myosin V Walks Hand-Over-Hand: Single Fluorophore Imaging with 1.5-nm Localization. Science. 300 (5628), 2061-2065 (2003).
- Yildiz, A., Tomishige, M., Vale, R. D., Selvin, P. R. Kinesin Walks Hand-Over-Hand. Science. 303 (5658), 676-678 (2004).
- Yildiz, A., et al. Myosin VI Steps via a Hand-over-Hand Mechanism with Its Lever Arm Undergoing Fluctuations when Attached to Actin. Journal of Biological Chemistry. 279 (36), 37223-37226 (2004).
- Yildiz, A., Selvin, P. R. Fluorescence Imaging with One Nanometer Accuracy: Application to Molecular Motors. Accounts of Chemical Research. 38 (7), 574-582 (2005).
- Toprak, E., Yildiz, A., Hoffman, M. T., Rosenfeld, S. S., Selvin, P. R. Why kinesin is so processive. Proceedings of the National Academy of Sciences. 106 (31), (2009).
- Kural, C., et al. Tracking melanosomes inside a cell to study molecular motors and their interaction. Proceedings of the National Academy of Sciences. 104 (13), 5378-5382 (2007).
- Gordon, M. P., Ha, T., Selvin, P. R. Single-molecule high-resolution imaging with photobleaching. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6462-6465 (2004).
- Qu, X., Wu, D., Mets, L., Scherer, N. F. Nanometer-localized multiple single-molecule fluorescence microscopy. Proceedings of the National Academy of Sciences of the United States of America. 101 (31), 11298-11303 (2004).
- Churchman, L. S., Ökten, Z., Rock, R. S., Dawson, J. F., Spudich, J. A. Single molecule high-resolution colocalization of Cy3 and Cy5 attached to macromolecules measures intramolecular distances through time. Proceedings of the National Academy of Sciences of the United States of America. 102 (5), 1419-1423 (2005).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-Dimensional Super-Resolution Imaging by Stochastic Optical Reconstruction Microscopy. Science. 319 (5864), 810-813 (2008).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3 (10), 793-796 (2006).
- Bates, M., Huang, B., Dempsey, G. T., Zhuang, X. Multicolor Super-Resolution Imaging with Photo-Switchable Fluorescent Probes. Science. 317 (5845), 1749-1753 (2007).
- Betzig, E., et al. Imaging Intracellular Fluorescent Proteins at Nanometer Resolution. Science. 313 (5793), 1642-1645 (2006).
- Abramoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics international. 11 (7), 36-42 (2004).
- Enderlein, J., Toprak, E., Selvin, P. R. Polarization effect on position accuracy of fluorophore localization. Optics Express. 14 (18), 8111-8120 (2006).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Rasnik, I., McKinney, S. A., Ha, T. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3 (11), 891-893 (2006).
- Zhuang, X., et al. A Single-Molecule Study of RNA Catalysis and Folding. Science. 288 (5473), 2048-2051 (2000).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados