Method Article
La automatización de experimentos-chip siguientes para generar perfiles epigenéticos en 10.000 células HeLa
En este artículo
Resumen
Methods for mapping in vivo protein-DNA interactions are becoming crucial for every aspect of genomic research but they are laborious, costly, and time consuming. Here a commercially available robotic liquid handling system that automates chromatin immunoprecipitation for mapping in vivo protein-DNA interactions with limited amounts of cells is presented.
Resumen
Chromatin immunoprecipitation followed by next generation sequencing (ChIP-seq) is a technique of choice for studying protein-DNA interactions. ChIP-seq has been used for mapping protein-DNA interactions and allocating histones modifications. The procedure is tedious and time consuming, and one of the major limitations is the requirement for high amounts of starting material, usually millions of cells. Automation of chromatin immunoprecipitation assays is possible when the procedure is based on the use of magnetic beads. Successful automated protocols of chromatin immunoprecipitation and library preparation have been specifically designed on a commercially available robotic liquid handling system dedicated mainly to automate epigenetic assays. First, validation of automated ChIP-seq assays using antibodies directed against various histone modifications was shown, followed by optimization of the automated protocols to perform chromatin immunoprecipitation and library preparation starting with low cell numbers. The goal of these experiments is to provide a valuable tool for future epigenetic analysis of specific cell types, sub-populations, and biopsy samples.
Introducción
Como Next-Generation Sequencing (NGS) tecnologías se han generalizado y más accesible, el principal método para la cartografía de todo el genoma de la proteína-DNA ahora cromatina inmunoprecipitación seguido por la detección NGS (chip-ss), que permite el descubrimiento del factor de transcripción sitios o los tipos de modificaciones de las histonas vinculante. ChIP-seq es ventajosa para proporcionar alto rendimiento de datos de todo el genoma que puede ser utilizado para el análisis cuantitativo y cualitativo de las interacciones proteína-DNA mediante la medición de los fragmentos de ADN enriquecidos. Sin embargo, hay algunas desventajas en experimentos-Chip SEQ estándar, tales como la dificultad de obtener material suficiente para crear una biblioteca de secuenciación.
Chip experimentos se dividen en seis pasos básicos que incluyen regiones de unión 1) reticulación de proteínas de ADN 2) la preparación de muestras que incluye la lisis celular y la esquila de la cromatina mediante ultrasonidos, 3) formación de los inmunocomplejos,4) la precipitación de los inmunocomplejos, 5) lavado de los inmunocomplejos, y 6) la elución del material enriquecido y el análisis de qPCR y NGS.
El éxito de un chip de ensayo depende de tres factores principales: una buena preparación de la cromatina, la cantidad de antígeno en la muestra original, y la especificidad y afinidad del anticuerpo por su antígeno afín. Una limitación importante es el requisito de altas cantidades de comenzar el número de células con el fin de obtener suficiente ADN enriquecido para crear una biblioteca de secuenciación. Para los científicos que trabajan con cantidades de muestra limitados, tales como muestras de biopsia o subpoblaciones celulares, los experimentos-chip siguientes son muy difíciles. Estudios recientes han demostrado que los ensayos de chip siguientes se pueden realizar cuando se trabaja con una baja cantidad de células 1, 2. Diagenode ha desarrollado un sistema de tratamiento de líquidos robótico que puede automatizar completamente experimentos-chip siguientes cuando se parte de un número limitado de células.
Automation proporcionamuchas ventajas sobre la preparación manual de las muestras-chip siguientes, ya que disminuye el error humano, reduce la variabilidad, y reduce el costo experimental. Protocolos semiautomáticos para inmunoprecipitación de la cromatina y la preparación de la biblioteca se han reportado, pero ninguno de estos estudios ha mostrado datos cuando se utiliza el número de células bajo 3, 4, 5, 6.
En este trabajo un flujo de trabajo automatizado completa se describe, por tanto inmunoprecipitación y ensayos de preparación de la biblioteca de la cromatina en un sistema de manejo de líquidos robótico que utiliza tecnología basada en perlas magnéticas y que puede abordar múltiples parámetros en la optimización de protocolos. Aquí, los experimentos-chip siguientes automatizados se realizaron con éxito en un número limitado de células con el objetivo de simplificar, estandarizar y proporcionar una solución fiable para estudiar los perfiles epigenéticos en las poblaciones de células pequeñas. El chip protocolo automatizado descrito en este documento se ha optimizado en células HeLa utilizando anticuerpos específicos de las histonas y reactivos but el flujo de trabajo se puede adaptar a otras líneas celulares y anticuerpos con sus correspondientes optimización experimental.
Protocolo
1. Los experimentos estándar de chip
- Recogida de células y la reticulación de ADN-proteína.
- Se cultivan las células HeLa-S3 a una confluencia de 80% -90%. Retire medio de cultivo, lavar el plato dos veces con 10 ml de 1x tampón fosfato salino (PBS), y añadir tripsina-EDTA (1x) a la placa de cultivo. Incubar durante un máximo de 2 min para separar las células de la placa. Recoger las células y lavar dos veces con 10 ml de PBS.
NOTA: El aumento de los tiempos de incubación dará lugar a daño celular. - Centrifugar las células durante 5 min a 500 xg y resuspender las células en 20 ml de PBS. Procederán al recuento de las células.
- Centrifugar las células durante 5 min a 500 xg, descartar el sobrenadante y añadir 500 l de PBS. El número óptimo de células para la etapa de fijación es de 10 millones de células por 500 l de PBS.
- Añadir 13,5 l de 37% de formaldehído fresca en cada alícuota de 500 l de suspensión celular. Fijar las células durante 8 min a TA.
- Añadir 57 l de solut 1,25 M glicinaion para detener la fijación. Incubar durante 5 min a temperatura ambiente con mezcla constante por vórtice suave. Trabajar en el hielo desde este punto en adelante.
- Centrifugar las células a 500 xg durante 5 min a 4 ° C y descartar el sobrenadante sin alterar el sedimento celular.
- Lavar las células dos veces con 1 ml de PBS. Desechar el sobrenadante con cuidado y mantener el sedimento de células en hielo.
- Se cultivan las células HeLa-S3 a una confluencia de 80% -90%. Retire medio de cultivo, lavar el plato dos veces con 10 ml de 1x tampón fosfato salino (PBS), y añadir tripsina-EDTA (1x) a la placa de cultivo. Incubar durante un máximo de 2 min para separar las células de la placa. Recoger las células y lavar dos veces con 10 ml de PBS.
- La lisis celular y esquila de la cromatina
- Añadir 10 ml de iL1 tampón de lisis de hielo frío al sedimento celular (1 ml de tampón de lisis por 1 millón de células es la proporción óptima). Pipeta hacia arriba y abajo varias veces e incubar durante 10 minutos a 4 ° C con una mezcla suave.
- Se centrifuga el lisado durante 5 min a 500 xg y 4 ° C. Eliminar el sobrenadante.
- Añadir 10 ml de tampón de lisis de hielo frío iL2 a los lisados y mezclar suavemente pipeteando arriba y abajo. Incubar los lisados durante 10 min a 4 ° C.
- Centrifugar durante 5 min a 500 xg y 4 ° C y descartar el sobrenadante.
- Prepare el tampón de corte completa añadiendo el cóctel inhibidor de la proteasa 200x (PIC) a la memoria intermedia de cizallamiento IS1. Mantenga el tampón en hielo durante 5 min y trabajar en hielo después. Añadir 1 ml de la memoria intermedia completa de cizallamiento IS1 a cada uno de 10 millones de células de pellets y mezclar suavemente pipeteando arriba y abajo. Antes de la sonicación Incubar las muestras en hielo durante 10 min para reducir la viscosidad de la muestra.
- Distorsión de 300 ml de alícuotas de la cromatina mediante ultrasonidos utilizando un aparato de ultrasonidos baño de agua durante 2 a 3 series de 10 ciclos cada uno. Un ciclo consta de 30 seg "ON" y 30 segundos "OFF" en un entorno de alta potencia. También puede utilizar un dispositivo de pico-sonicación con un tiempo de sonicación más corta de 5 a 10 ciclos de 30 segundos "ON", 30 seg "OFF". Vórtice brevemente y girar los tubos entre las carreras. Si se utilizan otros tipos de sonicadores, siga las instrucciones del fabricante correspondiente para la esquila de la cromatina.
- Centrifugar la cromatina esquilada a 16.000 xg durante 10 min y collect el sobrenadante se utiliza inmediatamente en la etapa de IP. Alternativamente, guarde la cromatina a -80 ° C durante un máximo de 2 meses para su uso futuro.
- Analizar la eficacia de esquila cromatina antes de la etapa de inmunoprecipitación utilizando 1-1,5% geles de agarosa TAE o bionalyzer. Tamaños de los fragmentos de cromatina óptimas oscilan entre 100 a 600 pb.
2. Experimentos Bajos chip Cell
- Recogida de células y la reticulación de ADN-proteína
- Se cultivan las células HeLa-S3 a una confluencia de 80% -90%. Retire medio de cultivo, lavar el plato dos veces con 10 ml de 1x tampón fosfato salino (PBS), y añadir 1x Tripsina-EDTA a la placa de cultivo. Incubar durante un máximo de 2 min para separar las células de la placa.
NOTA: El aumento de los tiempos de incubación dará lugar a daño celular. - Recoger las células mediante la adición de medio de cultivo que contiene 1 ml de suero en un tubo de centrifugación de 1 ml. Contar las células.
- Centrifugar las células durante 5 min a 500 x g. Traiga el número de célulasa 10.000 células por ml de medio de cultivo para la fijación.
- Añadir 27 l de 36,5% de formaldehído fresco preparado en cada tubo para la fijación. Invertir el tubo dos o tres veces e incubar 10 min a TA.
- Añadir 115 l de solución de glicina 1,25 M a la muestra, invertir el tubo de dos o tres veces y se incuba 5 min a TA. Trabajar en el hielo desde este punto en adelante.
- Centrifugar las células a 300 xg durante 10 min a 4 ° C. Descartar el sobrenadante lentamente.
- Lavar las células con 1 ml de hielo frío HBSS con PIC (200x, concentración final 1x). Invertir el tubo dos o tres veces para resuspender las células y centrifugar a 300 xg durante 10 min a 4 ° C. Desechar el sobrenadante con cuidado y mantener el sedimento de células en hielo.
- Se cultivan las células HeLa-S3 a una confluencia de 80% -90%. Retire medio de cultivo, lavar el plato dos veces con 10 ml de 1x tampón fosfato salino (PBS), y añadir 1x Tripsina-EDTA a la placa de cultivo. Incubar durante un máximo de 2 min para separar las células de la placa.
- La lisis celular y esquila de la cromatina
- Añadir 25 l de tampón de lisis completa de TL1 (Tampón de Lisis TL1 + PIC) por 10.000 células y agitar manualmente el fondo del tubo para resuspender las células. Incubar en hielo durante 5 min.
- Añadir 75 l de HBSS completa (HBSS + PIC) de tampón en cada alícuota que contiene 10.000 células.
- Distorsión de 100 ml de alícuotas de 10.000 células por sonicación durante 5 series de 5 ciclo de cada uno. Un ciclo consta de 30 seg "ON" y 30 segundos "OFF" en el ajuste de alta potencia. También puede utilizar un dispositivo de pico-sonicación se utiliza con un tiempo de sonicación más corto de 5 ciclos de 30 segundos "ON", 30 seg "OFF". Tamaños de los fragmentos de cromatina óptimas oscilan entre 100 a 600 pb. Tenga en cuenta que las preparaciones de la cromatina, y diferentes tipos de células sonicadores requieren experimentos de optimización de corte separados.
- Centrifugar la cromatina esquilada a 14.000 xg durante 10 min para descartar el material insoluble y recoger el sobrenadante a utilizarse inmediatamente en el paso IP. Alternativamente, guarde la cromatina a -80 ° C durante un máximo de 2 meses para su uso futuro.
- Analizar la eficacia de esquila cromatina antes de la etapa de inmunoprecipitación utilizando 1-1,5% TAE geles de agarosa ola bionalyzer. Tratar muestras con RNasa antes de agarosa análisis en gel con el fin de mejorar la evaluación visual de cizallamiento. Tamaños de los fragmentos de cromatina óptimas oscilan entre 100 a 600 pb.
3. La cromatina inmunoprecipitación y Biblioteca Prep
- Para experimentos chip estándar automatizado
- Añadir 120 l de chip Buffer H (CHIP Buffer H + PIC) a 100 l cromatina cortados. Utilice 200 l para la IP y mantener 2 l 20 l como muestra de entrada.
- Seleccione el chip 200 l protocolo automatizado en el instrumento de automatización. El rendimiento del protocolo es de 1 a 16 muestras por corrida.
- Ejecutar un chip experimento automatizado utilizando, cromatina correspondiente al 1-2000000 células, 2.1 g de anti-H3K79me3, grado -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 y H3K9me3 chip-ss anticuerpos policlonales de conejo. Cantidades óptimas de anticuerpos varían dependiendo de la modificación de las histonas y la afinidad y specificity del anticuerpo correspondiente.
- Use cantidades iguales de IgG de conejo no inmune como un anticuerpo de control de isotipo. Como alternativa, utilice los granos no revestidos o anticuerpo bloqueado específica como controles de chip. Añadir 20 l de una proteína de perlas magnéticas recubiertas para cada reacción.
- Utilice las histonas automatizado reactivos del kit-chip siguientes para llevar a cabo experimentos automatizados con chip -H3K4me2 anticuerpos policlonales anti-H3K79me3 y. Utilizar los reactivos ideales kit-chip siguientes para llevar a cabo experimentos con chip anti-H3K27me3, -H3K4me3, -H3K9ac, -H3K9 / 14ac, -H3K36me3 y -H3K9me3.
- Seleccione el chip protocolo automatizado siguiendo las instrucciones del software implementado en el dispositivo de automatización. Establezca los parámetros experimentales chip a 4 horas para la etapa de revestimiento de anticuerpos y 15 horas para la etapa de inmunoprecipitación. La etapa de reticulación inverso tiene lugar en el instrumento automático a 65 ° C durante 4 h.
- Se purifica el ADN reticulado inversa en el sistema automatizado. Seleccione autprotocolos omated para purificación de ADN con un protocolo o un kit de purificación de ADN basada en el uso de cuentas magnéticas. Eluir el ADN en 25 l de agua.
- Cuantificar el ADN inmunoprecipitado mediante la extracción de 10% del ADN inmunoprecipitado. El rendimiento de ADN inmunoprecipitado depende de la calidad de la cromatina y el anticuerpo, tipo de célula y la modificación de las histonas objetivo. Cuantificar el ADN utilizando un kit de ensayo de acuerdo con las instrucciones del fabricante.
- Analizar la calidad del ADN inmunoprecipitado por PCR cuantitativa usando cebadores de por lo menos 1 de control positivo y 1 negativo regiones genómicas. No utilice más de 10% del ADN inmunoprecipitado total para la evaluación de enriquecimientos chip.
- Prepare las reacciones de qPCR. Añadir 10 l de una mezcla maestra 2x SyberGreen qPCR, 1 l de mezcla de cebadores, 5/1 l de ADN immunoprecipitated o entrada y agua estéril hasta 20 l de volumen de reacción final. El programa qPCR incluye un paso de desnaturalización a 95 ° Cdurante 5-10 minutos dependiendo del proveedor de la Taq polimerasa y las temperaturas de recocido se deben establecer de acuerdo con los cebadores seleccionados.
- Use automatizado protocolos compatibles con los reactivos de preparación biblioteca Illumina chip-ss disponibles en el mercado para construir las bibliotecas que utilizan tanto chip de ADN, así como la entrada de ADN salvado de la misma preparación de cromatina biblioteca de preparación. Utilice 10-20 ng de ADN inmunoprecipitada a partir de cada anticuerpo para la preparación de la biblioteca. Prepara hasta 16 bibliotecas automatizadas por corrida.
- Secuenciar las bibliotecas y generar grupos de acuerdo a las instrucciones del fabricante Illumina. Realizar análisis primario bioinformática (filtrado cluster, llamado base, etc.) siguiendo el estándar Illumina tubería, filtro y alinear las lecturas a la última asamblea del genoma humano (versión actual es GRCh38) con el alineador ELAND. Utilice SICER 7 u 8 MACS para el pico de llamadas y realizar los análisis de aguas abajo de los picos wITH Homero 9, BEDTools 10 o software preferido.
- Por número automatizado bajo celulares chip experimentos
- Añadir 120 l de tampón chip completa tC1 (CHIP Buffer tC1 + PIC) a 100 l cromatina cortados. Utilice 200 l para la IP y mantener 20 l como entrada.
- Seleccione automatizado chip 200 l protocolo en el sistema de automatización. El rendimiento del protocolo es de 1 a 16 muestras por corrida.
- Ejecutar un experimento-chip siguientes automatizada usando reactivos chip automatizados y anticuerpos grado chip optimizado para trabajar en bajas cantidades de cromatina. Utilice la cromatina que corresponde a 10.000 células y 100.000 células, 0,5 g de anti-H3K27me3, 0,25 g -H3K4me3, 0,1 mg -H3K27ac, 0,25 g -H3K9me3 grado anticuerpos policlonales de conejo-chip siguientes conejo premium. Cantidades óptimas de anticuerpos varían dependiendo de la modificación de las histonas y la afinidad y especificidad del anticuerpo correspondiente.
- Utilice la misma cantidad de rab no inmunebit IgG como un anticuerpo de control de isotipo. Como alternativa, utilice los granos no revestidos o anticuerpo bloqueado específica como controles de chip. Añadir 10 l de una proteína de perlas magnéticas recubiertas para cada reacción.
- Seleccione el chip protocolo automatizado siguiendo las instrucciones del software implementado en el dispositivo de automatización. Establezca los parámetros experimentales chip a 4 horas para la etapa de revestimiento de anticuerpos y 15 horas para la etapa de inmunoprecipitación. La etapa de reticulación inverso tiene lugar en el instrumento automático a 65 ° C durante 4 h.
- Se purifica el ADN reticulado inversa utilizando columnas de centrifugación siguiendo las instrucciones del fabricante y se eluye en el volumen de 6 l a 25 l de agua.
- Cuantificar el ADN utilizando un kit de ensayo comercial. Analizar los resultados por qPCR utilizando cebadores para las regiones positivas y negativas de control para evaluar la calidad de las astillas.
- Utilice un kit de preparación de biblioteca con biblioteca optimizado reactivos de preparación para preparar bibliotecas con baja quan ADNtidades. Utilice 30 pg y 300 pg de chip de ADN (que corresponde a 10.000 y 100.000 células experimentos, respectivamente) para la preparación de la biblioteca. Preparar las bibliotecas que utilizan el protocolo automatizado compatible con reactivos de preparación de la biblioteca. La preparación rendimiento biblioteca automatizada es 1 a 48 bibliotecas por corrida.
- Después de la reparación final de las plantillas de ADN de doble cadena, ligar los adaptadores de tallo-bucle escindibles que contienen los sitios de los cebadores de secuenciación. Después de la etapa de extensión de ADN, amplificar la muestra con el método de amplificación de alta fidelidad se describe en el protocolo del kit de preparación de la biblioteca.
- Después de la amplificación de la biblioteca, cuantificar y purificar las bibliotecas siguiendo las directrices del kit de preparación de la biblioteca. Tenga en cuenta que la selección de tamaño después de la purificación no es necesario.
- Secuenciar las bibliotecas y generar grupos de acuerdo a las instrucciones del fabricante. Realizar análisis primario bioinformática (filtrado cluster, llamado base, etc.) siguiendo el soportetubería fabricante ard, filtro, y alinear las lecturas a la última asamblea del genoma humano (versión actual es GRCh38) con el alineador ELAND 7. Utilice SICER 8 o 9 MACS para el pico de llamadas y realizar los análisis de aguas abajo de los picos con Homero, BEDTools 10, o cualquier software preferido.

Figura 1. Imágenes del software que muestra cómo configurar experimentos automatizados chip en el IP-Star el acuerdo. El software proporciona la flexibilidad para seleccionar la cantidad de muestras por corrida, así como para cambiar los parámetros experimentales clave (anticuerpo de recubrimiento, IP y lavados ) de acuerdo con las necesidades del investigador. El procedimiento automatizado permite probar diferentes condiciones en paralelo (es decir, diferentes tipos y cantidades de anticuerpos, diferentes tipos y cantidades de las células e incluso diferente tipos y cantidades de perlas magnéticas en el mismo plazo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Imágenes del software que muestra cómo configurar la preparación automatizada de biblioteca para la secuenciación de próxima generación utilizando el kit de biblioteca en el sistema de automatización. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Optimización experimentos-chip siguientes automatizados durante ocho marcadores histona diferentes
Con el fin de desarrollar y validar los protocolos de chip automatizados, anticuerpos grado-chip siguientes que fueron validados previamente en experimentos-chip Sec manuales éxito (datos no mostrados) fueron seleccionados. Los siguientes anticuerpos grado-chip siguientes fueron elegidos para este estudio: anti-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 y -H3K9me3. La especificidad de todos los anticuerpos de grado-Chip SEQ fue confirmado previamente por dot blot, las matrices de péptidos y experimentos de transferencia Western (datos no presentados). Experimentos piloto chip-qPCR con cantidades crecientes de anticuerpos se realizaron para determinar la sensibilidad de los anticuerpos (Figura 3). qPCR con al menos dos positivos y dos blancos de control negativo se analizaron y perfiles con enriquecimientos de positivo sobre el objetivo negativo mayores de cinco veces están calificados para exper secuenciación experi-. Es importante realizar chip y chip-ss experimentos con una alta calidad de la cromatina esquilada. Todos los experimentos chip que se muestran en esta publicación se realizaron con la cromatina fresco. También es posible congelar las células fijadas a -80 ° C y proceder a la preparación de la cromatina y cizallamiento en un día diferente. Sin embargo, la cromatina preparado a partir de células fijas congeladas pueden comportarse de manera diferente de la cromatina recién preparados y, por tanto, pueden necesitar ser optimizado para cada preparación cromatina condiciones de sonicación. Cuando se trabaja con diferentes tipos de células, se pueden utilizar tampones de cizallamiento con diferentes composiciones detergentes (SDS). Los tipos de células tales como líneas de células primarias o células cultivadas en suspensión son células difíciles de cizallamiento y requieren de altas concentraciones de SDS (1%), mientras que las líneas celulares que son fáciles de cizallamiento, tal como HeLa requerirá bajas concentraciones de SDS (0,1%) en el tampones cizallamiento.
res.jpg "/>
Figura 3. Validación de anticuerpos chip-grado utilizando el sistema de automatización. Chip se realizó con anti-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, H3K9 / 14ac, -H3K36me3 y -H3K9me3 anticuerpos policlonales de conejo en la cromatina esquilada de 1 millón de células HeLa-S3 en función de las modificaciones de las histonas. Protocolos de chip automatizada con volúmenes de trabajo 200 l se utilizaron en el instrumento de automatización para experimentos de titulación de anticuerpos. Cantidades de anticuerpo de 1, 2, 5 y 10 mg fueron probados por experimento chip y 2 mg de IgG se utilizaron como control negativo en cada experimento. Enriquecimientos se evaluaron mediante qPCR. Los resultados se muestran como% de entrada (la cantidad relativa de ADN immunoprecipitated en comparación con el ADN de entrada después de un análisis qPCR). Por favor, haga clic aquí para ver una versión más grande de esta figura.
Después de validar y determinar los óptimos l cantidades de anticuerpos chip-grado para ser utilizados en el sistema de automatización, los experimentos-chip siguientes automatizados se realizaron con el fin de generar perfiles de secuenciación para cada modificación de las histonas (Figura 4).

Figura 4. histonas perfiles-chip ss generadas por experimentos-chip siguientes automatizados. La figura muestra los perfiles-chip ss en diferentes regiones genómicas para H3K4me3, H3K9ac y H3K9 / 14acH3K4me2 H3K79me3 y H3K36me3. 4A muestra la distribución de pico largo de la completa X -chromosome y 4B la distribución en una región de 75 kb que rodea el gen GAPDH. 4C muestra los perfiles de H3K27me3, H3K36me3 y H3K4me3 en una región de 500 kb que rodea el gen Myt1 y 4D muestra la distribución de H3K9me3 en una región de 200 kb ZNF12 circundante.large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
Perfiles epigenéticos histonas durante seis diferentes modificaciones de las histonas asociadas con la expresión de genes se generaron (H3K4me3, H3K9ac, H3K9 / 14ac, H3K4me2, H3K79me3 y H3K36me3). Figura 4A muestra perfiles chip-ss a lo largo del cromosoma X de los diferentes marcadores de histonas. La correlación altamente pico observado entre los 6 perfiles diferentes de histonas indica las capacidades de sistema automatizado para generar datos precisos y fiables. Figura 4B, 4C y 4D muestran la distribución de picos para las diferentes modificaciones de las histonas en las regiones genómicas específicas.
Automatizados experimentos de inmunoprecipitación de la cromatina abajo a 200 células
La cantidad mínima de células que puede ser utilizado en experimentos de ChIP depende de la calidad de la cromatina, la specificitY y la sensibilidad del anticuerpo y la abundancia de la modificación de las histonas o proteína estudiada. Selección de buenas anticuerpos grado-chip ss es importante cuando se trabaja con cantidades limitadas de la muestra y la selección de los reactivos óptimos y los diferentes operadores mejora la eficiencia de la recuperación del ADN y contribuir al éxito del experimento de chip. Para determinar la cantidad mínima de células que el chip protocolo automatizado puede procesar, diferentes cantidades de la cromatina, anticuerpo, y perlas magnéticas fueron probados en el sistema automatizado de IP-Star usando reactivos chip optimizado específicamente para trabajar con bajas cantidades de la cromatina.
En primer lugar, la cromatina de 10.000 células se sometió a ultrasonidos como se describe en el protocolo. Los resultados de chip se confirmaron por qPCR (Figura 5A), que muestra enriquecimientos significativas con anticuerpo H3K4me3 en las regiones de control positivo y la señal insignificante en las regiones de control negativo. Para la comparación y la prueba de consistencia, additional datos obtenidos con H3K27ac, H3K9me3 y anticuerpos H3K27me3, utilizando 10.000 células se proporciona.
Experimentos automatizados chip se realizaron a continuación, para demostrar las capacidades del sistema automatizado para trabajar con bajas cantidades de células usando el mismo anticuerpo H3K4me3. El chip automatizado tuvo un buen desempeño, demostrado por una serie de diez reacciones IP que eran reproducibles y altamente comparable con los resultados de chip manuales (Figura 5B). Experimentos manuales y automatizados se realizaron y se observaron beneficios de los protocolos automáticos en la reducción de experimento a experimento variabilidad (Figura 5C).
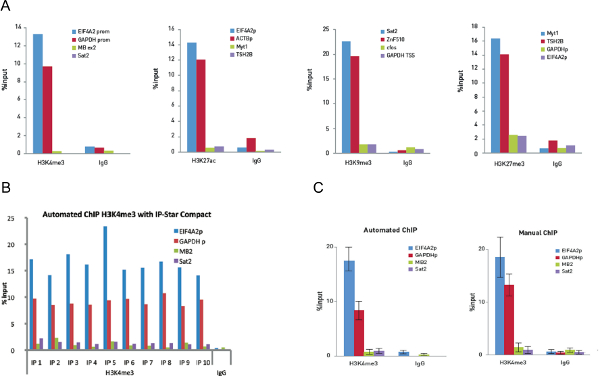
Figura 5. Optimización de chip y Auto chip experimentos con 10.000 células se realizaron experimentos chip Manual de 10.000 células y usando 0,25 g de H3K4me3, 0,1 g de H3K27ac, 0,5 g de H3K9me3 y 0,25 g de anticuerpos H3K27me3. Cantidades idénticas de IgG de conejo se utilizaron como control. El qPCR se realizó con cebadores para dos loci positivo y dos loci negativo para cada chip de ensayo. La Figura 5A muestra la recuperación, expresada como un porcentaje de la entrada (la cantidad relativa de ADN inmunoprecipitado en comparación con ADN de entrada después de un análisis qPCR). La Figura 5B muestra 10 reacciones de chip se ejecutan en el IP-Star compacto utilizando 0,25 g H3K4me3 anticuerpo policlonal y 0,25 g de IgG anticuerpo de conejo como control negativo. Entonces análisis qPCR se realizó con cebadores para el promotor positivo EIF4A2 loci y GAPDH TSS y el negativo exon2 mioglobina loci y SAT2. La figura muestra la recuperación, expresada como un porcentaje de entrada (cantidad relativa de ADN inmunoprecipitado en comparación con el ADN de entrada después del análisis de qPCR). La Figura 5C muestra los datos H3K4me3 chip de 10 chip experimentos manuales en comparación con 10 experimentos chip automatizados. Las barras de error representan s desviaciones stándar de cada una de las diez repeticiones. Haga clic aquí para ver una versión más grande de esta figura.
A fin de comprender la sensibilidad de los protocolos de chip automatizados, los experimentos se realizaron con una cantidad de células que oscila entre 100.000 abajo para 200 células por IP. Se utilizó el anticuerpo anti-H3K27me3 ya que es una modificación muy común de histonas. El uso de otros anticuerpos histonas o no histonas puede requerir células más o menos, dependiendo de la abundancia del epítopo y la calidad del anticuerpo. Los experimentos fueron validados por PCR cuantitativa y se observó que mediante la reducción de las cantidades de granos y de fondo de anticuerpos en los experimentos se reduce permitiendo exitosos resultados chip-qPCR con tan poco como 200 células de anticuerpos (Figura 6).
iles / ftp_upload / 52150 / 52150fig6highres.jpg "/>
Figura 6. automatizado ensayos de chip de 200 células. Células HeLa-S3 y anticuerpo dirigido contra H3K27me3. La cromatina se esquilada de 1 millón de células y diluciones seriadas de esta cromatina (de 100.000 a 200 células equivalente) fueron utilizados por reacción chip. 1 g de H3K27me3 y 10 l de perlas magnéticas recubiertas de proteína A se usaron en 100.000 células experimento, 0,5 g de H3K27me3 y 10 l de perlas en 10.000 y 1.000 células, y 0,25 g de H3K27me3 y 5 l de perlas con 500 y 200 las células. 1 g y 0,5 g de de IgG de conejo se usaron como anticuerpo de control negativo cuando se realizan experimentos con 100.000 células y 1000 células, respectivamente. 6A muestra la ocupación de genes TSH2B y GAPDH en% sobre la entrada. 6B muestra la ocupación relativa de TSH2B versus control GAPDH negativo región genómica.
Análisis de aguas abajo de los resultados de chip-ss sobre10.000 células
Con el fin de evaluar la calidad global de los experimentos automatizado chip-ss con números iniciales bajos de células, automatizados se realizaron ensayos de-chip ss con 0,25 mg de anticuerpo H3K4me3 en 10.000 células HeLa y chip experimentos se utilizaron en 100.000 células HeLa-S3 como control positivo para el experimento. Las bibliotecas automatizadas se prepararon utilizando la biblioteca Microplex reactivos del kit de preparación adaptados a las bibliotecas preparadas con cantidades de ADN de baja. Tenga en cuenta que a pesar de que es posible la realización de chip-experimentos automatizados exitosos con menos de 10.000 células, las cantidades de ADN derribado no serán suficientes para preparar bibliotecas utilizando los reactivos del kit. Generación de clústeres y la secuenciación se realizaron de acuerdo con las instrucciones del fabricante. La bioinformática analiza después los secuenciación mostrar resultados destacados de las muestras de chip de bajo número de células. El conjunto de datos 30 pg (correspondiente a 10.000 células de material de partida ) Contienen bajo nivel de ruido de fondo y los picos de enriquecimiento altamente fiables, que son confirmados por tanto el conjunto de datos pg 300 (correspondiente a 100.000 células del material de partida) y el conjunto de datos H3K4me3 generada por el Instituto Broad para el proyecto ENCODE que se utilizó como una referencia externa. Es importante tener en cuenta la relación de los datos Top 40 superposición, que se refiere a un método estándar utilizado en el proyecto ENCODE 11 en el que el chip-ss se considera reproducible si se comparan dos conjuntos de datos hay al menos un solapamiento del 80% de los mejores 40% de los picos clasificados según su puntuación importancia. El conjunto de datos de 30 pg cumple estos criterios en comparación tanto al conjunto de datos de 300 pg (teniendo en cuenta la totalidad de sus picos, no sólo el mejor 40%) y los datos del Instituto Broad (Tabla 1). El conjunto de datos 300 pg muestra picos casi idénticos a los datos del Instituto Broad con una tapa relación de solapamiento 98% 40 (Figura 7).
ighres.jpg "/>
Figura 7. ensayos de chip y la generación de bibliotecas de 10.000 células experimentos chip-ss se generaron en 10.000 y 100.000 células HeLa-S3 utilizando anticuerpos H3K4me3 (0,25 mg / l). Las etiquetas de 35 bp fueron asignadas al genoma humano con el alineador ELAND. Durante el pico posterior llamando SICER podría identificar de forma fiable los enriquecimientos de números bajos de células, así como de millones de células. Se analizaron y se compararon entre sí y a los datos de referencia generados por el Instituto Broad Los conjuntos de datos. Las muestras de células bajas son coherentes y tienen muy alta similitud. La muestra de 30 pg cumple los criterios de ENCODE 11 (min. 80% de la parte superior del 40% de los picos debe superponerse). Haga clic aquí para ver una versión más grande de esta figura.
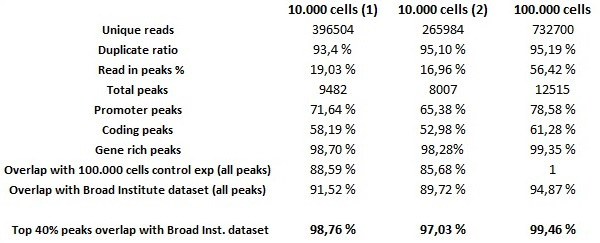
Tabla 1.
Discusión
Inmunoprecipitación de la cromatina seguida de la secuenciación es ahora un procedimiento estándar. Aquí un protocolo-chip siguientes automatizado que puede generar perfiles epigenéticos cromatina con tan sólo 10.000 células de material de partida se presenta.
La automatización de los ensayos de chip y de preparación de la biblioteca permite estandarizar el procedimiento de optimización chip y la reducción de la variabilidad experimental. El sistema de tratamiento de líquidos que se presenta aquí elimina muchos de los procedimientos manuales asociados con chip reduciendo las manos en vez de sólo 30 minutos, minimiza la pérdida de muestra y permite precisa chip-ss con unos pocos picogramos de entrada de la biblioteca. Para lograr experimentos-chip siguientes automatizados éxito, también es crucial utilizar preparaciones cromatina cortadas de alta calidad y anticuerpos grado-chip siguientes en cada experimento El sistema utiliza tecnología basada en la perla magnética y ofrece flexibilidad para cambiar principales parámetros experimentales, como la incubación tiempo para la capa de anticuerpoción y medidas de inmunoprecipitación o modificación de las condiciones de lavado que permiten al investigador para llevar a cabo todos los experimentos necesarios para la optimización-chip ss. El sistema automatizado es una plataforma "abierta", que también permite la comparación de múltiples reactivos en paralelo para la optimización de las condiciones experimentales para cada línea celular individual y de anticuerpo y permite la comparación directa de diferentes tipos y concentraciones de la cromatina, diferentes anticuerpos e incluso diferentes tipos de magnética perlas.
Una de las limitaciones del sistema automatizado es la necesidad de automatizar todos los protocolos en los volúmenes que van desde los 5 l de 200 l. Sin embargo, la miniaturización de los experimentos en esta plataforma automatizada también permite el ahorro de costes en reactivos.
Además de los protocolos descritos en este estudio, el sistema es adaptable y también automatiza una variedad de otras aplicaciones basadas en perlas magnéticas tales como inmunoprecipitación unand captura de ADN metilado (tecnologías MeDIP y MethylCap), inmunoprecipitación de ADN hydroxylmethylated (hMEDIP), inmunoprecipitación de la cromatina secuencial (RECHIP), inmunoprecipitación de ARN (ARN-IP), la conversión de bisulfito, y ensayos de purificación de ADN.
Divulgaciones
The authors of this article, at the time of its writing, are employed by Diagenode S.A. and Diagenode, Inc., the manufacturer of the automated system described.
Agradecimientos
This work was supported by the BLUERPINT EU grant (BLUEPRINT – A BLUEPRINT of Haematopoietic Epigenomes). We also thank the Walloon Region (DG06) for its financial support.
Materiales
| Name | Company | Catalog Number | Comments |
| PBS | Life technologies | 14190-094 | |
| Trypsin-EDTA | Sigma | T3924-100ML | |
| Formaldehyde 37% | Sigma | F8775-25 | |
| 1.25 M Glycine Solution | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL2 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Shearing Buffer iS1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Protease Inhibitors Mix (200x) | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| HBSS (no calcium, no magnesium, no phenol red) | Life technologies | 14175-053 | |
| Lysis Buffer tL1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ChIP Buffer tC1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ideal ChIP-seq kit | Diagneode | C01010051 | |
| ChIP-Buffer H | Diagenode | C01010020 | Component of the Auto Histone ChIP-seq kit |
| Auto Histone ChIP-seq kit | Diagenode | C01010020 | |
| Auto True Micro ChIP kit | Diagenode | C01010130 | |
| H3K79me3 polyclonal antibody-Classic | Diagenode | C15310068 | |
| H3K27me3 polyclonal antibody-Classic | Diagenode | C15410069 | |
| H3K4me3 polyclonal antibody-Classic | Diagenode | C15410030 | |
| H3K4me2 polyclonal antibody-Classic | Diagenode | C15410035 | |
| H3K9ac polyclonal antibody-Premium | Diagenode | C15410004 | |
| H3K9/14ac polyclonal antibody-Premium | Diagenode | C15410200 | |
| H3K36me3 polyclonal antibody-Premium | Diagenode | C15410058 | |
| H3K9me3 polyclonal antibody-Premium | Diagenode | C15410193 | |
| Rabbit IgG | Diagenode | C15410206 | |
| Protein-A coated paramagnetic beads | Diagenode | C01010020 | |
| Auto IPure | Diagenode | C03010010 | |
| MicroChIP DiaPure columns | Diagenode | C03040001 | |
| Universal SyberGreenMaster Mix 1.25 ml | Diagenode | DMMLD2D100 | |
| Quant-IT dsDNA | Invitrogen | Q32854 | |
| Illumina Sample Preparation kit fro Genomic DNA | Illumina | FC-121-3001 | |
| Illumina True-seq kit ChIP library Prep kit | Illumina | IP-202-1012 | |
| MicroPlex Library Preparation Kit | Diagenode | C05010010 | |
| Agencourt AMPure XP beads | Beckman Coulter | A63881 | |
| Illumina Library prep Quantification kit | Kapa Biosystems | KK4844 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | |
| Bioruptor Plus | Diagenode | B01020001 | |
| Bioruptor Pico | Diagenode | B01060001 | |
| Qubit system | Invitrogen | Q32857 | |
| Illumina Hiseq systems | Illumina |
Referencias
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Adli, M., Bernstein, B. E. Whole-genome chromatin profiling from limited numbers of cells using nano-ChIP-seq. Nature Protocols. 6, 1656-1668 (2011).
- Blecher-Gonen, R., et al. High-throughput chromatin immunoprecipitation for genome-wide mapping of in vivo protein-DNA interactions and epigenomic states. Nature Protocols. 8, 539-554 (2013).
- Aldridge, S., et al. AHT-ChIP-seq: a completely automated robotic protocol for high-throughput chromatin immunoprecipitation. Genome Biology. 14, 124 (2013).
- Farias-Hesson, E., et al. Semi-automated library preparation for high-throughput DNA sequencing platforms. Journal of Biomedicin., & Biotechnology. 2010, (2010).
- Callejas, S., Alvarez, R., Benguria, A., Dopazo, A. AG-NGS: a powerful and user-friendly computing application for the semi-automated preparation of next-generation sequencing libraries using open liquid handling platforms. BioTechniques. 56, 28-35 (2014).
- Zang, C., et al. A clustering approach for identification of enriched domains from histone modification ChIP-Seq data. Bioinformatics. 25, 1952-1958 (2009).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9, 137 (2008).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Molecular Cell. 38, 576-589 (2010).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics (Oxford, England). 26, 841-842 (2010).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados