Method Article
Автоматизация чип-след эксперименты для генерации эпигенетические профили на 10000 клеток HeLa
В этой статье
Резюме
Methods for mapping in vivo protein-DNA interactions are becoming crucial for every aspect of genomic research but they are laborious, costly, and time consuming. Here a commercially available robotic liquid handling system that automates chromatin immunoprecipitation for mapping in vivo protein-DNA interactions with limited amounts of cells is presented.
Аннотация
Chromatin immunoprecipitation followed by next generation sequencing (ChIP-seq) is a technique of choice for studying protein-DNA interactions. ChIP-seq has been used for mapping protein-DNA interactions and allocating histones modifications. The procedure is tedious and time consuming, and one of the major limitations is the requirement for high amounts of starting material, usually millions of cells. Automation of chromatin immunoprecipitation assays is possible when the procedure is based on the use of magnetic beads. Successful automated protocols of chromatin immunoprecipitation and library preparation have been specifically designed on a commercially available robotic liquid handling system dedicated mainly to automate epigenetic assays. First, validation of automated ChIP-seq assays using antibodies directed against various histone modifications was shown, followed by optimization of the automated protocols to perform chromatin immunoprecipitation and library preparation starting with low cell numbers. The goal of these experiments is to provide a valuable tool for future epigenetic analysis of specific cell types, sub-populations, and biopsy samples.
Введение
Как Next-Generation Секвенирование (NGS) технологии получили широкое распространение и более доступными, основным методом генома отображения взаимодействий белок-ДНК в настоящее время иммунопреципитации хроматина сопровождается обнаружением NGS (чип-след), которая позволяет открытие фактора транскрипции сайты связывания или узоры модификаций гистонов. Чип и SEQ имеет преимущество в обеспечении высокой пропускной данные целого генома, который может быть использован для количественного и качественного анализа взаимодействий белок-ДНК путем измерения обогащенных фрагментов ДНК. Тем не менее, есть некоторые недостатки в стандартный чип-след экспериментов, таких как сложность в получении достаточно материала, чтобы создать секвенирования библиотеку.
ChIP эксперименты делятся на шесть основных шагов, включая участками связывания 1) сшивания белок-ДНК 2) пробоподготовки, которая включает лизис клеток и сдвига хроматина с помощью ультразвука, 3) формирование иммунных комплексов,4) осаждение иммунных, 5) промывка иммунных и 6) элюирование обогащенного материала и анализа количественной ПЦР и NGS.
Успех чип анализа зависит от трех основных факторов: хорошая хроматина подготовки, количество антигена в исходном образце, и специфичности и аффинности антитела для его антигеном. Основным недостатком является необходимость больших количеств исходного количества клеток, чтобы получить достаточное количество обогащенного ДНК, чтобы создать библиотеку секвенирования. Для ученых, которые работают с ограниченным количеством образцов, таких как образцы биопсии или клеточных субпопуляций, чип-след эксперименты очень сложной задачей. Недавние исследования показали, что чип-последующие анализы могут быть выполнены при работе с низким количеством клеток 1, 2. Diagenode разработала роботизированную систему обработки жидкости, которое может полностью автоматизировать чип-последующие эксперименты при запуске с ограниченным числом клеток.
Автоматизация обеспечиваетмного преимуществ по сравнению с ручной подготовки чип-след образцов, как это унижает человеческое ошибку, уменьшает изменчивость, а также снижает экспериментальный стоимость. Полуавтоматическая протоколы иммунопреципитации хроматина и подготовки библиотеки были зарегистрированы, но ни один из этих исследований не показал данные при использовании число клеток низкие 3, 4, 5, 6.
В этой статье полная автоматизированный рабочий процесс описывается как для иммунопреципитации хроматина и подготовка библиотека анализов в роботизированной жидкой системе обработки, которая использует технологию магнитной шарик на основе и что может обратиться нескольких параметров в оптимизации протокола. Здесь автоматизированные чип-след эксперименты были успешно выполнены на ограниченном числе клеток с целью упрощения, стандартизации и обеспечения надежного решения для изучения эпигенетических профили в малых популяциях клеток. Автоматизированный протокол чип описано в этой статье была оптимизирована на HeLa клеток с использованием специфических гистонов антитела и реагенты бут рабочий процесс может быть приспособлен к другим клеточных линий и антител с соответствующими экспериментальными оптимизации.
протокол
1. стандартный чип Эксперименты
- Сбора клеток и ДНК-белок сшивающего.
- Расти HeLa-S3 клетки до впадения 80% -90%. Удалить культуральной среды, промыть блюдо дважды 10 мл 1х забуференного фосфатом физиологического раствора (PBS), и добавить трипсин-ЭДТА (1x) в культивируемых пластины. Выдержите в течение максимум 2 мин до отделения клеток от чашки. Сбор клеток и дважды промывали 10 мл PBS.
ПРИМЕЧАНИЕ: Длительное время инкубации приведет к повреждению клеток. - Центрифуга клетки в течение 5 мин при 500 х г и ресуспендирования клеток в 20 мл PBS. Приступают к подсчету клеток.
- Центрифуга клетки в течение 5 мин при 500 х г, отбросить супернатант и добавить 500 мкл PBS. Оптимальное количество клеток на стадии фиксации является 10000000 клеток в 500 мкл PBS.
- Добавить 13,5 мкл свежего 37% формальдегида в каждой аликвоты 500 мкл клеточной суспензии. Сосредоточение клеток в течение 8 минут при комнатной температуре.
- Добавить 57 мкл 1,25 М глицин Solutиона, чтобы остановить фиксацию. Инкубировать в течение 5 минут при комнатной температуре при постоянном перемешивании, осторожно вихря. Работа на льду с этого момента.
- Центрифуга клетки при 500 х г в течение 5 мин при 4 ° С и отбросить супернатант, не нарушая клеточный осадок.
- Вымойте клетки в два раза с 1 мл PBS. Отменить мягко супернатант и сохранить осадок клеток на льду.
- Расти HeLa-S3 клетки до впадения 80% -90%. Удалить культуральной среды, промыть блюдо дважды 10 мл 1х забуференного фосфатом физиологического раствора (PBS), и добавить трипсин-ЭДТА (1x) в культивируемых пластины. Выдержите в течение максимум 2 мин до отделения клеток от чашки. Сбор клеток и дважды промывали 10 мл PBS.
- Клеточный лизис и хроматина стрижки
- Добавить 10 мл охлажденного льдом буфера для лизиса IL1 к осадку клеток (1 мл буфера для лизиса на 1 млн клеток является оптимальное соотношение). Пипетка вверх и вниз несколько раз, и инкубируют в течение 10 мин при 4 ° С при осторожном перемешивании.
- Центрифуга лизата в течение 5 мин при 500 х г и 4 ° С. Удалите супернатант.
- Добавить 10 мл ледяной буфера для лизиса iL2 к лизатов и аккуратно перемешать с помощью пипетки вверх и вниз. Инкубируйте лизаты в течение 10 мин при 4 ° С.
- Центрифуга в течение 5 мин при 500 х г и 4 ° С и отбросить супернатант.
- ПреRe полного буфера сдвига, добавляющий 200x коктейль ингибиторов протеазы (ПОС) в сдвига буфера Is1. Хранить буфер на льду в течение 5 мин и работа на льду после этого. Добавить 1 мл полного Is1 сдвига буфера каждые 10 миллионов клеток на пеллетах и аккуратно перемешать с помощью пипетки вверх и вниз. Перед обработкой ультразвуком инкубировать образцы на льду в течение 10 мин, чтобы уменьшить вязкость образца.
- Прочность на сдвиг 300 мкл аликвоты хроматина с помощью ультразвука с использованием ванны ультразвукового воды от 2 до 3 подхода по 10 циклов каждый. Цикл состоит из 30 сек "ON" и 30 сек "OFF" на высоком установленной мощности. Кроме того, использовать пико-обработки ультразвуком устройство с более коротким временем обработки ультразвуком от 5 до 10 циклов 30 сек "ON", 30, ТРЦ "OFF". Кратко вихрь и спин трубы между запусками. При использовании других типов sonicators, следуйте соответствующие указания изготовителя по хроматина стрижки.
- Центрифуга стриженой хроматина при 16000 х г в течение 10 мин и сСобрать супернатант, который будет использоваться сразу же на стадии IP. Кроме того, хранить хроматина при -80 ° С в течение 2 месяцев для использования в будущем.
- Анализ хроматина эффективности сдвига до иммунопреципитации шагом, используя 1-1,5% гели TAE агарозном или bionalyzer. Оптимальные хроматина размеры фрагментов в диапазоне от 100-600 б.п..
2. Низкая экспериментах с клеточными ChIP
- Сбора клеток и ДНК-белок сшивания
- Расти HeLa-S3 клетки до впадения 80% -90%. Удалить культуральной среды, промыть блюдо дважды 10 мл 1х забуференного фосфатом физиологического раствора (PBS), и добавить 1x трипсин-ЭДТА в культивируемых пластины. Выдержите в течение максимум 2 мин до отделения клеток от чашки.
ПРИМЕЧАНИЕ: Длительное время инкубации приведет к повреждению клеток. - Сбор клеток путем добавления 1 мл культуральной среды, содержащей сыворотку в 1 мл центрифужную пробирку. Граф клеток.
- Центрифуга клетки в течение 5 мин при 500 х г. Принесите мобильный номер10000 клеток на мл культуральной среды для фиксации.
- Добавить 27 мкл 36,5% свежеприготовленным формальдегида в каждую пробирку для фиксации. Переверните пробирку два или три раза и инкубировать 10 мин при комнатной температуре.
- Добавить 115 мкл 1,25 М раствора глицина к образцу, инвертировать трубку два или три раза и инкубировали 5 мин при комнатной температуре. Работа на льду с этого момента.
- Центрифуга клетки при 300 х г в течение 10 мин при 4 ° С. Удалите супернатант медленно.
- Вымойте клеток с 1 мл ледяной HBSS с фото (200x, конечная концентрация 1x). Инвертировать трубку два или три раза для ресуспендирования клеток и центрифуге при 300 х г в течение 10 мин при 4 ° С. Отменить мягко супернатант и сохранить осадок клеток на льду.
- Расти HeLa-S3 клетки до впадения 80% -90%. Удалить культуральной среды, промыть блюдо дважды 10 мл 1х забуференного фосфатом физиологического раствора (PBS), и добавить 1x трипсин-ЭДТА в культивируемых пластины. Выдержите в течение максимум 2 мин до отделения клеток от чашки.
- Клеточный лизис и хроматина стрижки
- Добавить 25 мкл полного буфера для лизиса (TL1 лизирующего буфера tL1 + PIC) на 10000 клеток и перемешивают вручную в нижней части трубки для ресуспендирования клеток. Инкубируют на льду в течение 5 мин.
- Добавить 75 мкл полной ССРХ (HBSS + ПОС) буфера в каждую аликвоту, содержащую 10000 клеток.
- На сдвиг 100 мкл аликвоты 10000 клеток с помощью ультразвука в течение 5 наборов 5 цикла каждого. Цикл состоит из 30 сек "ON" и 30 сек "OFF" при настройке высокой мощности. Кроме того, использовать пико-обработки ультразвуком устройство, используемое с более коротким временем обработки ультразвуком 5 циклов 30 сек "ON", 30, ТРЦ "OFF". Оптимальные хроматина размеры фрагментов в диапазоне от 100-600 б.п.. Обратите внимание, что хроматина препараты, клеточные типы и различные sonicators требуют отдельных экспериментов по оптимизации сдвига.
- Центрифуга сдвигу хроматина при 14000 х г в течение 10 мин, чтобы отбросить нерастворимого материала и сбор супернатанта быть использованы сразу же на стадии IP. Кроме того, хранить хроматина при -80 ° С в течение 2 месяцев для использования в будущем.
- Анализ хроматина эффективности сдвига до иммунопреципитации шагом, используя 1-1,5% TAE агарозном геле илиbionalyzer. Лечить образцы с РНКазы до агарозном анализ геля для того, чтобы улучшить визуальную оценку стрижки. Оптимальные хроматина размеры фрагментов в диапазоне от 100-600 б.п..
3. иммунопреципитации хроматина и библиотека Prep
- Для стандартных автоматизированная ChIP экспериментов
- Добавить 120 мкл чипа буфера H (чип буфера H + ПОС) в 100 мкл стригли хроматина. Используйте 200 мкл для IP и держать 2 мкл до 20 мкл, входного образца.
- Выберите автоматизированной чип 200 протокол мкл в приборе автоматизации. Пропускная способность протокола 1 к 16 проб в перспективе.
- Запуск автоматизированной ChIP эксперимент с использованием хроматина, соответствующей 1-2 миллионов клеток, 1-2 мкг анти-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 и H3K9me3 чип-след класса кроличьи поликлональные антитела. Оптимальные количества антител варьируются в зависимости от модификации гистонов и сродства и спецификацииificity соответствующего антитела.
- Используйте равное количество неиммунной кролика IgG в качестве контроля изотипа. Кроме того, использование непокрытых бусы или конкретные заблокированы антитела, управления чипом. Добавить 20 мкл белка с покрытием магнитных шариков для каждой реакции.
- Используйте автоматизированной гистонов чип-след реагенты для выполнения автоматизированных экспериментов чип с анти-H3K79me3 и -H3K4me2 поликлональных антител. Используйте идеальные чип-след реагенты для выполнения ChIP эксперименты с анти-H3K27me3, -H3K4me3, -H3K9ac, -H3K9 / 14ac, -H3K36me3 и -H3K9me3.
- Выберите протокол автоматического ChIP, следуя инструкциям программного обеспечения, осуществляемых в устройстве автоматизации. Установка чипа экспериментальные параметры 4 ч на стадии нанесения покрытия антитела и 15 ч для стадии иммунопреципитации. Сшивающий шаг обратного происходит в автоматическом приборе при 65 ° С в течение 4 ч.
- Очищают обратной сшитую ДНК по автоматизированной системе. Выберите автomated протоколы для очистки ДНК с протоколом или набор с помощью магнитно-шарик на основе очистки ДНК. Элюции ДНК в 25 мкл воды.
- Количественная Иммунопреципитированные ДНК путем извлечения 10% от иммунопреципитации ДНК. Выход иммунопреципитации ДНК зависит от качества хроматина и антитела, типа клеток и модификации целевой гистона. Количественная ДНК с использованием набора для анализа в соответствии с инструкциями изготовителя.
- Анализ качества по иммунопреципитации ДНК с помощью количественной ПЦР с использованием праймеров, по крайней мере, один положительный и один отрицательный контроль геномных областей. Не следует использовать более чем 10% от общего иммунопреципитации ДНК для оценки ChIP обогащение.
- Подготовьте реакции КПЦР. Добавить 10 мкл смеси мастер-микс 2x SyberGreen КПЦР, 1 мкл праймера смеси, 1-5 мкл Иммунопреципитированные или ввода ДНК и стерильной водой до 20 мкл конечного объема реакции. Программа включает в себя КПЦР начальной стадии денатурации при 95 ° Св течение 5-10 мин в зависимости от поставщика к Taq-полимеразы, а температура отжига должна быть установлена в соответствии с праймеров, выбранных.
- Использовать автоматизированные протоколы, совместимые с коммерчески доступных Illumina Чип-SEQ подготовки библиотека реагентов для построения библиотеки, используя как ДНК-чип, а также сохраненный входной ДНК из одной и той же хроматина подготовки библиотека преп. Используйте 10-20 нг Иммунопреципитированные ДНК из каждого антитела для библиотеки подготовки. Подготовка до 16 автоматизированных библиотек в перспективе.
- Последовательность библиотеки и создавать кластеры в соответствии с инструкциями Illumina производителя. Выполнить анализ первичной биоинформатики (фильтрация кластера, базовый призвание, и т.д.) в соответствии со стандартным Illumina трубопроводов, фильтра и совместите читает до последней сборки генома человека (текущая версия GRCh38) с выравнивателя Эланд. Используйте SICER 7 или MACS 8 для обеспечения максимальной призвания и выполнять вниз по течению анализ пиков WIth Гомер 9, BEDTools 10 или предпочтительным программного обеспечения.
- Для низких номеру сотового автоматизированная ChIP экспериментов
- Добавить 120 мкл полной буфера чип tc1 (чип буфера tC1 + ПОС) в 100 мкл стригли хроматина. Используйте 200 мкл для IP и держать 20 мкл в качестве входных данных.
- Выберите Автоматическое чип 200 протокол мкл в системе автоматизации. Пропускная способность протокола 1 к 16 проб в перспективе.
- Запуск автоматизированной чип-след эксперимент с использованием автоматизированных ChIP реагенты и чип класса антител оптимизирован для работы на низких объемах хроматина. Используйте хроматина, соответствующей 10000 клеток и 100 000 клеток, 0,5 мкг анти-H3K27me3, 0,25 мкг -H3K4me3, 0,1 мкг -H3K27ac, 0,25 мкг -H3K9me3 кролик Премиум чип-след класса кроличьих поликлональных антител. Оптимальные количества антител варьируются в зависимости от модификации гистонов и аффинностью и специфичностью соответствующего антитела.
- Используйте равное количество неиммунной Рабнемного IgG в качестве контроля изотипа. Кроме того, использование непокрытых бусы или конкретные заблокированы антитела, управления чипом. Добавить 10 мкл белка с покрытием магнитных шариков для каждой реакции.
- Выберите протокол автоматического ChIP, следуя инструкциям программного обеспечения, осуществляемых в устройстве автоматизации. Установка чипа экспериментальные параметры 4 ч на стадии нанесения покрытия антитела и 15 ч для стадии иммунопреципитации. Сшивающий шаг обратного происходит в автоматическом приборе при 65 ° С в течение 4 ч.
- Очищают обратной сшитую ДНК с использованием спиновых столбцы в соответствии с инструкциями производителя и элюируют в объемах от 6 мкл до 25 мкл воды.
- Количественная ДНК с использованием коммерческого набора для анализа. Проанализируйте результаты КПЦР с использованием праймеров для положительных и отрицательных регионах контроля для оценки качества щепы.
- Используйте библиотеку подготовка комплекта с оптимизированной библиотеки подготовки реагентов для получения библиотеки с низким цюань ДНКчины. Используйте 30 стр и 300 мкг ДНК-чип (в соответствии с 10000 до 100000 клеток экспериментов соответственно) для библиотеки подготовки. Подготовка библиотеки, используя автоматизированную протокол, совместимый с библиотекой реагентов подготовки. Автоматизированной библиотечной подготовительные пропускная способность от 1 до 48 библиотек в перспективе.
- После окончания ремонта шаблонов двухцепочечной ДНК, лигируют с расщепляемые Шпилька адаптеры, содержащие сайты секвенирования праймеров. После дополнительного этапа ДНК, амплификации образца с помощью метода амплификации высокой точностью, описанной в библиотеке протокола Подготовка комплекта.
- После усиления библиотека, количественно и очистить библиотеки следующие библиотеки руководящих принципов подготовки набора. Обратите внимание, что выбор размера после очистки не требуется.
- Последовательность библиотеки и создавать кластеры в соответствии с инструкциями изготовителя. Выполнить анализ первичной биоинформатики (фильтрация кластера, базовый призвание, и т.д.) после стендаARD производитель трубопроводов, фильтров, и выровнять читает до последней сборки генома человека (текущая версия GRCh38) с ELAND 7 выравнивателя. Используйте SICER 8 или MACS 9 для достижения максимальной призвания и выполнять вниз по течению анализ пиков с Гомером, BEDTools 10, или любой предпочтительный программного обеспечения.

Рисунок 1. Скриншоты программного обеспечения, показывающий, как создать автоматизированные эксперименты чипа в IP-Star Compact. Программное обеспечение обеспечивает гибкость, чтобы выбрать количество образцов в перспективе, а также изменить ключевые экспериментальные параметры (антитела покрытия, IP и моет ) в соответствии с потребностями исследователя. Автоматизированная процедура позволяет протестировать различные условия параллельно (т.е. различные типы и количество антител, различные типы и количество клеток и даже разные тypes и количества магнитных шариков в одной и той же перспективе. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.

Рисунок 2. Скриншоты программного обеспечения, показывающий, как создать автоматизированную подготовку к библиотеке для следующего поколения секвенирования с использованием библиотеки набора в системе автоматизации. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
Результаты
Оптимизация автоматизированных чип-след эксперименты для восьми различных маркеров гистонов
Для того, чтобы успешно разработать и утвердить автоматизированные протоколы Чип, Чип-сл класса антител, которые ранее были подтверждены в ручном чип-Seq экспериментов (данные не показаны), были отобраны. Следующие чип-след класса антитела были выбраны для этого исследования: анти-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 и -H3K9me3. Специфичность всех чип-SEQ класса антител был ранее подтвержден дот-блот, пептидные массивов и Вестерн-блот-экспериментов (данные не показаны). Пилот чип-КПЦР эксперименты с увеличением антител количествах, чтобы определить чувствительность антител (рисунок 3). КПЦР, по крайней мере, два положительных и два отрицательных показателей в области борьбы были проанализированы и профили с обогащением положительный над негативными цели выше, чем в пять раз квалифицированы для секвенирования Exper опыты могут. Важно, чтобы выполнить чипа и чип-след эксперименты с высоким качеством стриженой хроматина. Все эксперименты ChIP показанные в этой публикации были выполнены с использованием свежих хроматин. Кроме того, можно заморозить фиксированные клетки при -80 ° С и приступить к подготовке и хроматина сдвига на другой день. Тем не менее, хроматина получены из замороженных фиксированных клеток может вести себя по-разному из хроматина свежеприготовленный и, следовательно, условия обработки ультразвуком возможно, должны быть оптимизированы для каждого хроматина подготовки. При работе с различными типами клеток, ножницы буферы с различными составами моющих (SDS) могут быть использованы. Типы клеток, такие как клеточные линии первичной или клетки выращивают в суспензии сложные клетки стричь и потребует высоких концентраций SDS (1%), тогда как клеточные линии, которые легко стричь такие как HeLa потребует низкие концентрации SDS (0,1%) в ножницы буферы.
res.jpg "/>
Рисунок 3. Проверка чип-класса антител с использованием системы автоматизации. Чип производится с анти-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, H3K9 / 14ac, -H3K36me3 и -H3K9me3 кроличьих поликлональных антител на стриженой хроматина от 1000000 HeLa-S3 клетки в зависимости от модификации гистонов. Автоматизированные протоколов чип с рабочими объемами 200 мкл были использованы в автоматизации инструмента антитела титрования экспериментов. Антитела количества 1, 2, 5 и 10 мкг были испытаны на чип эксперимента и 2 мкг IgG, были использованы в качестве отрицательного контроля в каждом эксперименте. Обогащения были оценены кПЦР. Результаты представлены в виде% от входа (относительного количества иммунопреципитации ДНК по сравнению с входным ДНК после анализа КПЦР). Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
После проверки и определения оптимума л количество чип-класса антител, которые будут использоваться в системе автоматизации, автоматизированных чип-след эксперименты проводились с целью получения секвенирования профили для каждой модификации гистонов (рис 4).

Рисунок 4. Гистоновые чип-след профили генерируются с помощью автоматизированных чип-след экспериментов. Рисунок показывает чип-след профили в различных геномных регионов для H3K4me3, H3K9ac и H3K9 / 14acH3K4me2 H3K79me3 и H3K36me3. 4А показывает пик распределения вдоль всей X -chromosome и 4В распределение в регионе 75 кб, окружающей ген GAPDH. 4C показаны профили H3K27me3, H3K36me3 и H3K4me3 в области 500 кб, окружающей ген MYT1 и 4D показывает распределение H3K9me3 в 200 кб область, окружающая ZNF12.large.jpg "TARGET =" _ пустое "> Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
Гистоновые эпигенетические профили для шести различных модификаций гистонов, связанных с генной экспрессии были получены (H3K4me3, H3K9ac, H3K9 / 14ac, H3K4me2, H3K79me3 и H3K36me3). На рисунке 4а показаны чип-SEQ профили вдоль хромосомы X для различных маркеров гистонов. Очень пик корреляции наблюдается между 6 различных профилей гистонов показывает возможности автоматизированной системы для получения точных и достоверных данных. 4В, 4С и 4D показывают распределение пиков для различных модификаций гистонов в определенных геномных областей.
Автоматизированные эксперименты иммунопреципитации хроматина вплоть до 200 клеток
Минимальное количество клеток, которые могут быть использованы в микросхему экспериментов зависит от качества хроматина, specificitу и чувствительность антитела и обилие модификации гистонов или белка учился. Выбор хорошей степенью антитела чип-след очень важно при работе с ограниченным количеством образца и выбора оптимальных реагентов и различных носителей повышает эффективность восстановления ДНК и внести свой вклад в успех эксперимента чип. Чтобы определить минимальное количество клеток, которые могут обрабатывать автоматизированный протокол Чип, различным количеством хроматина, антитела, и магнитных шариков были испытаны в автоматизированной системе IP-Star используя обломок реагенты оптимизированные специально для работы с малым количеством хроматина.
Во-первых, хроматин от 10000 клеток ультразвуком, как описано в протоколе. Результаты ChIP были подтверждены КПЦР (рис 5А), который показывает значительные обогащение с H3K4me3 антитела в положительных регионах контроля и незначительной сигнала в отрицательных областях управления. Для сравнения и доказательства непротиворечивости, additionaл данные, полученные с H3K27ac, H3K9me3 и H3K27me3 антитела, используя 10000 клеток обеспечивается.
Автоматизированные эксперименты проводились ChIP затем, чтобы продемонстрировать возможности автоматизированной системы для работы с низким количеством клеток с использованием той же H3K4me3 антитела. Автоматизированный ЧИП показали хорошие результаты, свидетельствует серии десять IP реакций, которые были воспроизводимыми и очень сопоставим с результатами ручного кристалле (фиг.5В). Ручные и автоматические эксперименты проводились и преимущества автоматизированных протоколов были замечены в сокращении эксперимента к эксперименту изменчивость (рис 5в).
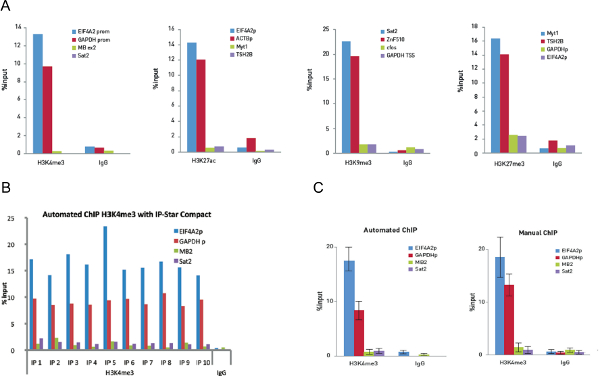
Рисунок 5. Оптимизация чипа и Auto ChIP экспериментов по 10000 клеток Руководство эксперименты чипе, выполненных на 10000 клеток и с использованием 0,25 мкг H3K4me3, 0,1 мкг H3K27ac, 0,5 мкг H3K9me3 и 0,25 мкг H3K27me3 антител. Идентичные количества IgG кролика, использовали в качестве контроля. КПЦР проводили с использованием праймеров для двух положительных локусов и два отрицательных локусов для каждого чипа анализа. 5А показывает восстановление, выраженное в процентах от входа (относительное количество иммунопреципитации ДНК по сравнению с входным ДНК после анализа КПЦР). 5В показана 10 реакции чипу работать на IP-Star Compact с использованием 0,25 мкг H3K4me3 поликлональные антитела и 0,25 мкг кролика IgG в качестве отрицательного контрольного антитела. Тогда анализ КПЦР проводили с использованием праймеров для положительного промотора локуса EIF4A2 и GAPDH TSS и отрицательным локусы Myoglobin exon2 и Sat2. Рисунок показывает восстановление, выраженное в процентах от входного (относительное количество иммунопреципитации ДНК по сравнению с входным ДНК после анализа КПЦР). Рисунок 5C показывает H3K4me3 чипом данных о 10 ручных экспериментов чип в сравнении с 10 автоматизированных экспериментов чип. Усы представляют S tandard отклонения каждого из десяти повторов. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
Для того, чтобы понять чувствительность автоматизированных протоколов Чип, были проведены эксперименты, используя количество клеток, которые варьировались от 100 000 до 200 клеток на IP. Анти-H3K27me3 использовали антитела, как это очень распространенная модификация гистонов. Использование других гистонов или негистоновых антител может потребовать больше или меньше клеток, в зависимости от избытка эпитопа и качества антитела. Эксперименты были подтверждены с помощью количественной ПЦР и было отмечено, что за счет снижения количества шариков и фона антител в экспериментах снижается позволяет успешных результатов чип-КПЦР с всего лишь 200 клеток антитела (рисунок 6).
Иль / ftp_upload / 52150 / 52150fig6highres.jpg "/>
Рисунок 6. Автоматизированная ChIP анализов на 200 клеток. Hela S3-клетки и антитела, направленные против H3K27me3. Хроматина был стрижки 1000000 клеток и серийных разведений этого хроматина (от 100000 до 200 клеток эквивалента) были использованы в реакции чипа. 1 мкг H3K27me3 и 10 мкл белка А-покрытием магнитных шариков были использованы на 100000 клеток эксперимента, 0,5 мкг H3K27me3 и 10 мкл шариков на 10000 и 1000 клеток, и 0,25 мкг H3K27me3 и 5 мкл шариков с 500 и 200 клетки. 1 мкг и 0,5 мкг на IgG кролика, использовали в качестве отрицательного контрольного антитела при выполнении экспериментов с клетками 100000 и 1000 клеток соответственно. 6A показывает загруженность TSH2B и GAPDH генов в% ввода. 6B показывает относительную загруженность TSH2B против отрицательного контроля GAPDH область генома.
Вниз по течению анализ результатов чип-Seq на10000 клеток
Для оценки глобального качества автоматизированной чип-последующие эксперименты с низкой начальной числа клеток, автоматизированный проводили чип-последующие анализы с 0,25 мкг этого антитела по H3K4me3 10000 HeLa клеток и чип экспериментов были использованы на 100000 HeLa-S3 клетки В качестве положительного контроля для эксперимента. Автоматизированные библиотеки были подготовлены с использованием библиотеки MicroPlex подготовка реагенты, адаптированные к подготовленных библиотек с низким количеством ДНК. Обратите внимание, что, хотя это можно успешно вести автоматизированные чип-эксперименты с менее чем 10 000 клеток, количества снесены ДНК не будет достаточно, чтобы подготовиться библиотеки с использованием набора реагентов. Кластер поколения и последовательность были выполнены в соответствии с инструкциями изготовителя. Биоинформатика анализы после секвенирования шоу выдающихся результатов проб чип с малым количеством клеток. 30 пг набор данных (соответствует 10000 клеток исходного материала ) Содержат низкий фоновый шум и высокую надежность пики по обогащению, которые подтверждены как пг набора данных 300 (что соответствует 100 000 клеток исходного материала) и набора данных H3K4me3 порожденной Broad Institute в рамках проекта ENCODE, который был использован в качестве внешней ссылки. Важно отметить отношение данных 40 перекрытия, которое ссылается на стандартный метод, используемый в проекте КОДИРОВАНИЯ 11, в котором чип-след считается воспроизводимым в случае сравнения двух наборов данных Существует по крайней мере 80% перекрытие из лучших 40% пиков, ранжированных по счету значимости. 30 стр набор данных выполняет эти критерии, по сравнению как с пг набора данных 300 (с учетом всех его вершин, не только лучше 40%), и данные Broad Institute (таблица 1). 300 стр набор данных показывает почти одинаковые пики в данных Broad Institute с 98% Топ 40 соотношении перекрытия (рис 7).
ighres.jpg "/>
Рисунок 7. ChIP анализы и библиотека поколение на 10000 клеток чип-Seq экспериментов были получены на 10000 и 100000 HeLa-S3 клетки с использованием H3K4me3 антитела (0,25 мкг / мкл). В 35 б.п. теги были нанесены на карту генома человека с выравнивателя Эланд. Во время последующего пика вызывающего SICER может надежно определить обогащение от низких числа клеток, а также из миллионов клеток. Наборы данных были проанализированы и сравнены друг с другом и с базовыми данными, порожденных Broad Institute. Образцы низкие клеток соответствуют и имеют очень высокое сходство. Образец 30 пг выполняет критерии кодировать 11 (мин. 80% из топ 40% пиков должны перекрываться). Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
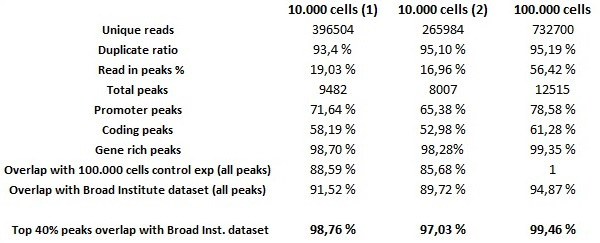
Таблица 1.
Обсуждение
Иммунопреципитации хроматина с последующим секвенирования теперь стандартная процедура. Здесь автоматизированная чип-след протокол, который может генерировать хроматина эпигенетические профили с всего лишь 10 000 клеток исходного материала представлена.
Автоматизация чипа и подготовки библиотека анализов позволяет стандартизировать процедуру оптимизации чипа и обработки экспериментальных изменчивости. Жидкостная система обработки представлены здесь устраняет многие из ручных процедур, связанных с чипом уменьшая руки на время, чтобы просто 30 минут, сводит к минимуму потери пробы и дает возможность точной чип-SEQ с помощью всего лишь нескольких пикограмм библиотечного входа. Для того, чтобы добиться успешного автоматизированные чип-след эксперименты, это также важно использовать высококачественные стриженой хроматина препараты и чип-след класса антител в каждом эксперименте система использует технологию магнитной шарик на основе и обеспечивает гибкость, чтобы изменить основные экспериментальные параметры, такие как инкубации время для пальто антителчисле и шаги иммунопреципитации или модификации условий моющих позволяет исследователю провести все необходимые эксперименты для чип-след оптимизации. Автоматизированная система "открыт" платформа, которая также позволяет сравнивать несколько реагентов параллельно для оптимизации экспериментальных условий для каждой отдельной линии клеток и антител и позволяет прямое сравнение различных типов и концентраций хроматина, различных антител и даже различных типов магнитных бусы.
Одним из ограничений автоматизированной системы является необходимость автоматизации всех протоколов в объемах, которые варьируются от 5 мкл до 200 мкл. Тем не менее, миниатюризация экспериментов в этой автоматизированной платформы также позволяет экономить расходы на реагенты.
В дополнение к протоколам, описанным в данном исследовании, система является гибкой, а также позволяет автоматизировать множество других приложений на основе магнитного бисера, таких как иммунопреципитации ай захват метилированной ДНК (MEDIP и MethylCap технологии), иммунопреципитацией hydroxylmethylated ДНК (hMEDIP), последовательный иммунопреципитации хроматина (ReChIP), РНК иммунопреципитации (РНК-IP), бисульфит преобразования и очистки ДНК анализов.
Раскрытие информации
The authors of this article, at the time of its writing, are employed by Diagenode S.A. and Diagenode, Inc., the manufacturer of the automated system described.
Благодарности
This work was supported by the BLUERPINT EU grant (BLUEPRINT – A BLUEPRINT of Haematopoietic Epigenomes). We also thank the Walloon Region (DG06) for its financial support.
Материалы
| Name | Company | Catalog Number | Comments |
| PBS | Life technologies | 14190-094 | |
| Trypsin-EDTA | Sigma | T3924-100ML | |
| Formaldehyde 37% | Sigma | F8775-25 | |
| 1.25 M Glycine Solution | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL2 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Shearing Buffer iS1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Protease Inhibitors Mix (200x) | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| HBSS (no calcium, no magnesium, no phenol red) | Life technologies | 14175-053 | |
| Lysis Buffer tL1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ChIP Buffer tC1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ideal ChIP-seq kit | Diagneode | C01010051 | |
| ChIP-Buffer H | Diagenode | C01010020 | Component of the Auto Histone ChIP-seq kit |
| Auto Histone ChIP-seq kit | Diagenode | C01010020 | |
| Auto True Micro ChIP kit | Diagenode | C01010130 | |
| H3K79me3 polyclonal antibody-Classic | Diagenode | C15310068 | |
| H3K27me3 polyclonal antibody-Classic | Diagenode | C15410069 | |
| H3K4me3 polyclonal antibody-Classic | Diagenode | C15410030 | |
| H3K4me2 polyclonal antibody-Classic | Diagenode | C15410035 | |
| H3K9ac polyclonal antibody-Premium | Diagenode | C15410004 | |
| H3K9/14ac polyclonal antibody-Premium | Diagenode | C15410200 | |
| H3K36me3 polyclonal antibody-Premium | Diagenode | C15410058 | |
| H3K9me3 polyclonal antibody-Premium | Diagenode | C15410193 | |
| Rabbit IgG | Diagenode | C15410206 | |
| Protein-A coated paramagnetic beads | Diagenode | C01010020 | |
| Auto IPure | Diagenode | C03010010 | |
| MicroChIP DiaPure columns | Diagenode | C03040001 | |
| Universal SyberGreenMaster Mix 1.25 ml | Diagenode | DMMLD2D100 | |
| Quant-IT dsDNA | Invitrogen | Q32854 | |
| Illumina Sample Preparation kit fro Genomic DNA | Illumina | FC-121-3001 | |
| Illumina True-seq kit ChIP library Prep kit | Illumina | IP-202-1012 | |
| MicroPlex Library Preparation Kit | Diagenode | C05010010 | |
| Agencourt AMPure XP beads | Beckman Coulter | A63881 | |
| Illumina Library prep Quantification kit | Kapa Biosystems | KK4844 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | |
| Bioruptor Plus | Diagenode | B01020001 | |
| Bioruptor Pico | Diagenode | B01060001 | |
| Qubit system | Invitrogen | Q32857 | |
| Illumina Hiseq systems | Illumina |
Ссылки
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Adli, M., Bernstein, B. E. Whole-genome chromatin profiling from limited numbers of cells using nano-ChIP-seq. Nature Protocols. 6, 1656-1668 (2011).
- Blecher-Gonen, R., et al. High-throughput chromatin immunoprecipitation for genome-wide mapping of in vivo protein-DNA interactions and epigenomic states. Nature Protocols. 8, 539-554 (2013).
- Aldridge, S., et al. AHT-ChIP-seq: a completely automated robotic protocol for high-throughput chromatin immunoprecipitation. Genome Biology. 14, 124 (2013).
- Farias-Hesson, E., et al. Semi-automated library preparation for high-throughput DNA sequencing platforms. Journal of Biomedicin., & Biotechnology. 2010, (2010).
- Callejas, S., Alvarez, R., Benguria, A., Dopazo, A. AG-NGS: a powerful and user-friendly computing application for the semi-automated preparation of next-generation sequencing libraries using open liquid handling platforms. BioTechniques. 56, 28-35 (2014).
- Zang, C., et al. A clustering approach for identification of enriched domains from histone modification ChIP-Seq data. Bioinformatics. 25, 1952-1958 (2009).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9, 137 (2008).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Molecular Cell. 38, 576-589 (2010).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics (Oxford, England). 26, 841-842 (2010).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены