Method Article
Automatizando Experiments chip-seq para gerar perfis epigenéticos em 10.000 células HeLa
Neste Artigo
Resumo
Methods for mapping in vivo protein-DNA interactions are becoming crucial for every aspect of genomic research but they are laborious, costly, and time consuming. Here a commercially available robotic liquid handling system that automates chromatin immunoprecipitation for mapping in vivo protein-DNA interactions with limited amounts of cells is presented.
Resumo
Chromatin immunoprecipitation followed by next generation sequencing (ChIP-seq) is a technique of choice for studying protein-DNA interactions. ChIP-seq has been used for mapping protein-DNA interactions and allocating histones modifications. The procedure is tedious and time consuming, and one of the major limitations is the requirement for high amounts of starting material, usually millions of cells. Automation of chromatin immunoprecipitation assays is possible when the procedure is based on the use of magnetic beads. Successful automated protocols of chromatin immunoprecipitation and library preparation have been specifically designed on a commercially available robotic liquid handling system dedicated mainly to automate epigenetic assays. First, validation of automated ChIP-seq assays using antibodies directed against various histone modifications was shown, followed by optimization of the automated protocols to perform chromatin immunoprecipitation and library preparation starting with low cell numbers. The goal of these experiments is to provide a valuable tool for future epigenetic analysis of specific cell types, sub-populations, and biopsy samples.
Introdução
Como Next-Generation Sequencing (NGS) tecnologias se tornaram comuns e mais acessível, o principal método para o mapeamento de todo o genoma de interações proteína-ADN agora é imunoprecipitação de cromatina seguida de detecção NGS (CHIP-seq), que permite a descoberta do fator de transcrição sites ou padrões de modificações de histonas vinculativo. ChIP-seq é vantajosa no fornecimento de dados de alto rendimento de todo o genoma que pode ser utilizado para análise quantitativa e qualitativa das interacções proteína-ADN pela medição dos fragmentos de ADN enriquecidos. No entanto, existem algumas desvantagens em experiências ChIP-seq padrão, tais como a dificuldade de obter material suficiente para criar uma biblioteca de sequenciação.
ChIP experiências são divididos em seis passos básicos incluindo as regiões de ligação 1) reticulação de ADN-proteína 2) preparação da amostra que inclui a lise celular e corte a cromatina por sonicação, 3) a formação de imunocomplexos,4) precipitação dos imunocomplexos, 5) lavagem dos imunocomplexos, e 6) eluição do material enriquecido e análise por qPCR e NGS.
O sucesso de um ensaio de chip é dependente de três factores principais: uma boa preparação de cromatina, a quantidade de antigénio na amostra original, e a especificidade e afinidade do anticorpo para o seu antigénio cognato. Uma das principais limitações é a necessidade de quantidades elevadas a partir de números de células, a fim de se obter suficiente ADN enriquecido para criar uma biblioteca de sequenciação. Para os cientistas que trabalham com quantidades de amostras limitadas, como amostras de biópsia ou sub-populações de células, experimentos chip-seq são muito desafiador. Estudos recentes têm mostrado que os ensaios de ChIP-seq pode ser realizada quando se trabalha com uma baixa quantidade de células de 1, 2. Diagenode desenvolveu um sistema de manipulação robótica líquido que pode automatizar completamente as experiências ChIP-seq quando começando com um número limitado de células.
Automation fornecemuitas vantagens sobre a preparação manual de amostras de chip-seq, uma vez que diminui o erro humano, reduz a variabilidade, e reduz o custo experimental. Protocolos semi-automatizados para imunoprecipitação da cromatina e preparação biblioteca foram relatados, mas nenhum desses estudos tem mostrado dados ao usar números de celulares de baixo 3, 4, 5, 6.
Neste trabalho um fluxo de trabalho automatizado completo é descrito tanto para imunoprecipitação e preparação biblioteca ensaios de cromatina em um sistema de tratamento de líquido robótica que utiliza tecnologia baseada em grânulo magnético e que pode resolver vários parâmetros na otimização de protocolo. Aqui, experimentos chip seq automatizados foram realizados com sucesso em um número limitado de células com o objetivo de simplificar, padronizar e fornecendo uma solução confiável para estudar perfis epigenéticos em populações de células pequenas. O protocolo ChIP automatizado descrito neste trabalho foi otimizado em células HeLa utilizando anticorpos específicos de histonas e reagentes but o fluxo de trabalho pode ser adaptado para outras linhas de células e anticorpos com optimização experimental correspondente.
Protocolo
1. As experiências ChIP Padrão
- Recolha de células, e a reticulação do DNA-proteína.
- Cultivar células HeLa-S3 a uma confluência de 80% -90%. Remover o meio de cultura, lava-se a placa duas vezes com 10 ml de solução salina tamponada 1x com fosfato (PBS), e adicionar Tripsina-EDTA (1x) para o prato de cultura. Incubar durante um máximo de 2 minutos para separar as células do prato. Recolher as células e lava-se duas vezes com 10 ml de PBS.
NOTA: Prolongamento do tempo de incubação vai levar a danos celulares. - Centrifuga-se as células durante 5 min a 500 x g e ressuspender as células em 20 ml de PBS. Prossiga para a contagem das células.
- Centrifuga-se as células durante 5 min a 500 xg, descartar o sobrenadante e adicionar 500 ul de PBS. O número óptimo de células para o passo de fixação é de 10 milhões de células por 500 ul de PBS.
- Adicionar 13.5 ul de formaldeído a 37% fresco em cada alíquota de 500 ul de suspensão de células. Fixar as células durante 8 minutos à temperatura ambiente.
- Adicionar 57 ul de 1,25 M de glicina solutião para parar a fixação. Incubar durante 5 min à temperatura ambiente com mistura constante num vortex suave. Trabalhar em gelo a partir deste ponto em diante.
- Centrifuga-se as células a 500 xg durante 5 min a 4 ° C e desprezar o sobrenadante sem perturbar o sedimento celular.
- Lavar as células duas vezes com 1 ml de PBS. Descartar o sobrenadante e suavemente manter o sedimento de células em gelo.
- Cultivar células HeLa-S3 a uma confluência de 80% -90%. Remover o meio de cultura, lava-se a placa duas vezes com 10 ml de solução salina tamponada 1x com fosfato (PBS), e adicionar Tripsina-EDTA (1x) para o prato de cultura. Incubar durante um máximo de 2 minutos para separar as células do prato. Recolher as células e lava-se duas vezes com 10 ml de PBS.
- A lise celular e cromatina tosquia
- Adicionar 10 ml de tampão de gelo iL1 lise frio ao sedimento de células (1 ml de tampão de lise por 1 milhão de células, é a proporção óptima). Pipeta cima e para baixo várias vezes e incubar durante 10 min a 4 ° C com mistura suave.
- Centrifugar o lisado durante 5 min a 500 xg e 4 ° C. Descartar o sobrenadante.
- Adicionar 10 ml de tampão de lise frio gelo il2 aos lisados e misture delicadamente por pipetagem cima e para baixo. Incubar os lisados durante 10 minutos a 4 ° C.
- Centrifugar durante 5 minutos a 500 xg e 4 ° C e desprezar o sobrenadante.
- Prepare o buffer de corte completa adição do cocktail inibidor da protease 200x (PIC) para o tampão de cisalhamento IS1. Manter o tampão em gelo, durante 5 min e depois trabalhar em gelo. Adicionar 1 ml de tampão completo corte is1 a cada 10 milhões de células de pelotização e misture delicadamente por pipetagem cima e para baixo. Antes de sonicação incubar as amostras em gelo durante 10 min para reduzir a viscosidade da amostra.
- Cisalhamento alíquotas de 300 ul de cromatina por ultra-sons utilizando um sonicador de banho de água durante 2 a 3 conjuntos de 10 ciclos cada. Um ciclo é composto por 30 seg "ON" e 30 segundos "OFF" em um cenário de alta potência. Em alternativa, utilizar um dispositivo de pico-sonicação com um curto tempo de sonicação de 5 a 10 ciclos de 30 seg "ON", 30 seg "OFF". Resumidamente vortex e girar os tubos entre as corridas. Ao usar outros tipos de sonicators, siga as instruções do fabricante correspondente para cromatina cisalhamento.
- Centrifugar a cromatina tosquiada a 16.000 xg durante 10 min e collect o sobrenadante para ser usado imediatamente no passo de IP. Alternativamente, a cromatina armazenar a -80 ° C durante até 2 meses para uso futuro.
- Analisar a cromatina eficiência de corte antes da etapa immunoprecipitation usando 1-1,5% de gel de agarose TAE ou bionalyzer. Tamanhos dos fragmentos de cromatina óptimas variam entre 100-600 pb.
2. Baixo Experiments chip Cell
- Recolha de células e ADN-proteína de reticulação
- Cultivar células HeLa-S3 a uma confluência de 80% -90%. Remover o meio de cultura, lava-se a placa duas vezes com 10 ml de solução salina tamponada 1x com fosfato (PBS), e adicionar 1x tripsina-EDTA para o prato de cultura. Incubar durante um máximo de 2 minutos para separar as células do prato.
NOTA: Prolongamento do tempo de incubação vai levar a danos celulares. - Recolher as células por adição de 1 ml de meio de cultura contendo soro para um tubo de centrifugação de 1 ml. Contar as células.
- Centrifuga-se as células durante 5 min a 500 x g. Traga o número de célulasa 10.000 células por ml de meio de cultura para a fixação.
- Adicionar 27 mL de 36,5% de formaldeído preparado fresco em cada tubo para a fixação. Inverter o tubo de duas ou três vezes e incuba-se 10 min à temperatura ambiente.
- Adicionar 115 uL de 1,25 M de solução de glicina para a amostra, inverter o tubo de duas ou três vezes e incuba-se 5 minutos à TA. Trabalhar em gelo a partir deste ponto em diante.
- Centrifuga-se as células a 300 xg durante 10 min a 4 ° C. Descartar o sobrenadante lentamente.
- Lave as células com 1 ml de gelo frio HBSS com PIC (200x, concentração final 1x). Inverter o tubo de duas ou três vezes para ressuspender as células e centrifugar a 300 xg durante 10 min a 4 ° C. Descartar o sobrenadante e suavemente manter o sedimento de células em gelo.
- Cultivar células HeLa-S3 a uma confluência de 80% -90%. Remover o meio de cultura, lava-se a placa duas vezes com 10 ml de solução salina tamponada 1x com fosfato (PBS), e adicionar 1x tripsina-EDTA para o prato de cultura. Incubar durante um máximo de 2 minutos para separar as células do prato.
- A lise celular e cromatina tosquia
- Adicionar 25 uL de Tampão de Lise completa de TL1 (Tampão de Lise TL1 + PIC) por 10.000 células e agitar manualmente o fundo do tubo para ressuspender as células. Incubar em gelo durante 5 min.
- Adicionar 75 ul de HBSS completo (HBSS + PIC) de tampão em cada alíquota contendo 10.000 células.
- Cisalhamento 100 mL alíquotas de 10.000 células por sonicação durante 5 séries de 5 de cada ciclo. Um ciclo é composto por 30 seg "ON" e 30 segundos "OFF" na configuração de alta potência. Em alternativa, utilizar um dispositivo de pico-sonicação usado com um menor tempo de sonicação de 5 ciclos de 30 s "ON", 30 seg "OFF". Tamanhos dos fragmentos de cromatina óptimas variam entre 100-600 pb. Note-se que as preparações de cromatina, e tipos de células diferentes sonicadores exigem experiências de optimização de corte separados.
- Centrifugar a cromatina tosquiada a 14.000 xg durante 10 min para eliminar o material insolúvel e recolher o sobrenadante para ser usado imediatamente no passo de IP. Alternativamente, a cromatina armazenar a -80 ° C durante até 2 meses para uso futuro.
- Analisar a cromatina eficiência de corte antes da etapa de imunoprecipitação utilizando 1-1,5% de géis de agarose ou TAEo bionalyzer. Amostras Tratar com RNase antes de agarose gel análise, a fim de melhorar a avaliação visual de cisalhamento. Tamanhos dos fragmentos de cromatina óptimas variam entre 100-600 pb.
3. Chromatin Immunoprecipitation e Biblioteca Prep
- Por padrão automatizado experimentos ChIP
- Adicionar 120 ul de tampão de ChIP H (ChIP Tampão H + PIC) a 100 ul cortadas cromatina. Use 200 mL para o IP e manter 2 l para 20 l como amostra de entrada.
- Selecione o chip 200 protocolo ul automatizada no instrumento de automação. A taxa de transferência do protocolo é de 1 a 16 amostras por corrida.
- Executar uma experiência ChIP automatizado usando, cromatina correspondente a 1-2 milhões de células, 1-2 mg de anti-H3K79me3, grau -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 e H3K9me3 ChIP-seq Os anticorpos policlonais de coelho. Quantidades de anticorpos óptimas variam de acordo com a modificação de histonas e a afinidade e especificaçãoificity do anticorpo correspondente.
- Utilize quantidades iguais de não-imune IgG de coelho como um anticorpo de controlo de isotipo. Como alternativa, use pérolas não revestidos ou anticorpo específico bloqueado como controles de chip. Adicionar 20 ul de proteína A esferas magnéticas revestidas para cada reacção.
- Use as histonas automatizado chip seq reagentes para realizar experimentos automatizados chip com anti-H3K79me3 e policlonais -H3K4me2. Utilizar os reagentes ideais kit chip-seq para realizar experimentos chip com anti-H3K27me3, -H3K4me3, -H3K9ac, -H3K9 / 14ac, -H3K36me3 e -H3K9me3.
- Selecione o protocolo ChIP automatizado seguindo as instruções do software implementado no dispositivo de automação. Defina os parâmetros experimentais chip para 4 horas para a etapa de revestimento de anticorpos e 15 horas para a etapa de imunoprecipitação. O passo de reticulação reversa tem lugar no instrumento automatizado, a 65 ° C durante 4 h.
- Purifica-se o ADN com ligações cruzadas inversa no sistema automatizado. Select automated protocolos de purificação de DNA com um protocolo ou kit de purificação de DNA usando magnética talão de base. Elui-se o ADN em 25 ul de água.
- Quantificar o ADN imunoprecipitado por extracção de 10% do ADN imunoprecipitado. O rendimento de ADN imunoprecipitado depende da qualidade da cromatina e do anticorpo, o tipo de célula e a modificação da histona-alvo. Quantificar o ADN utilizando um kit de ensaio de acordo com as instruções do fabricante.
- Analisar a qualidade do ADN imunoprecipitado por PCR quantitativo utilizando iniciadores para, pelo menos, um controlo positivo e um negativo regiões genómicas. Não utilize mais do que 10% do ADN imunoprecipitado total para avaliar enriquecimentos ChIP.
- Preparar as reacções qPCR. Adicionar 10 ml de uma mistura principal 2x SyberGreen qPCR, 1 ml de mistura de iniciadores, 1-5 ul de DNA imunoprecipitado ou entrada e água estéril até 20 l de volume de reacção final. O programa qPCR inclui um passo de desnaturação inicial a 95 ° Cpara 5-10 min, dependendo do fornecedor da polimerase Taq e as temperaturas de recozimento deve ser definido de acordo com os primers selecionados.
- Utilização automatizado protocolos biblioteca prep compatíveis com os reagentes comercialmente disponíveis de preparação biblioteca Ilumina-ChIP seq para construir as bibliotecas utilizando tanto de chips de DNA, bem como o ADN de entrada salvos da mesma preparação de cromatina. Use 10-20 ng de ADN imunoprecipitado a partir de cada anticorpo para a preparação da biblioteca. Prepare-se para 16 bibliotecas automatizadas por corrida.
- Sequenciar as bibliotecas e gerar clusters de acordo com as instruções do fabricante Illumina. Realizar análise primária bioinformática (filtragem cluster, chamada base, etc.) seguindo o padrão Illumina gasoduto, filtro e alinhar a lê para a mais recente montagem do genoma humano (versão atual é GRCh38) com o alinhador ELAND. Use sicer 7 ou 8 para MACS pico chamado e realizar as análises, a jusante de picos wom Homer 9, 10 ou BEDTools software preferido.
- Para o número automatizado experimentos chip de baixo consumo de células
- Adicionar 120 mL de tampão chip termina TC1 (chip de buffer tc1 + PIC) para 100 l cortadas cromatina. Use 200 mL para o IP e manter 20 l como entrada.
- Selecione automatizado ChIP 200 protocolo ul no sistema de automação. A taxa de transferência do protocolo é de 1 a 16 amostras por corrida.
- Executar uma experiência ChIP-seq automatizado utilizando reagentes ChIP automatizados e anticorpos da classe ChIP otimizados para trabalhar em quantidades baixas de cromatina. Use cromatina correspondente a 10.000 células e 100.000 células, 0,5 ug de anti-H3K27me3, 0,25 mg -H3K4me3, 0,1 g -H3K27ac, 0,25 mg -H3K9me3 anticorpos policlonais de coelho coelho premium chip seq grau. Quantidades de anticorpos óptimas variam de acordo com a modificação de histonas e a afinidade e especificidade do anticorpo correspondente.
- Use uma quantidade igual de rab não imuneIgG pouco como um anticorpo de controlo de isotipo. Como alternativa, use pérolas não revestidos ou anticorpo específico bloqueado como controles de chip. Adicionar 10 ul de proteína A esferas magnéticas revestidas para cada reacção.
- Selecione o protocolo ChIP automatizado seguindo as instruções do software implementado no dispositivo de automação. Defina os parâmetros experimentais chip para 4 horas para a etapa de revestimento de anticorpos e 15 horas para a etapa de imunoprecipitação. O passo de reticulação reversa tem lugar no instrumento automatizado, a 65 ° C durante 4 h.
- Purifica-se o ADN com ligações cruzadas reversa utilizando colunas de giro seguindo as instruções do fabricante e elui-se em volumes de 6 ul de 25 ul de água.
- Quantificar o ADN utilizando um estojo de ensaio comercial. Analisar os resultados por qPCR utilizando primers para regiões controlo positivas e negativas para avaliar a qualidade de chips.
- Use um kit de preparação biblioteca com biblioteca otimizada reagentes de preparação para preparar bibliotecas com baixo quan DNAtidades. Use 30 pg e 300 pg de chip de DNA (correspondente a 10.000 e 100.000 células experimentos, respectivamente) para a preparação da biblioteca. Prepare bibliotecas utilizando o protocolo automatizado compatível com os reagentes de preparação da biblioteca. A biblioteca prep débito automático é de 1 a 48 bibliotecas por corrida.
- Após o reparo final dos modelos de ADN de cadeia dupla, ligar os adaptadores de haste-laçada cliváveis contendo os locais dos primers de sequenciamento. Seguindo o passo de extensão de ADN, amplificar a amostra com o método de amplificação de alta fidelidade descrito no protocolo do kit preparação biblioteca.
- Após a amplificação biblioteca, quantificar e purificar as bibliotecas seguindo as orientações do kit de preparação da biblioteca. Note-se que a seleção de tamanho após a purificação não é necessário.
- Sequenciar as bibliotecas e gerar clusters de acordo com as instruções do fabricante. Realizar análise primária bioinformática (filtragem cluster, chamada base, etc.) seguindo o estandegasoduto fabricante ard, filtrar e alinhar a lê para a mais recente montagem do genoma humano (versão atual é GRCh38) com o alinhador ELAND 7. Use sicer 8 ou 9 para MACS pico chamado e realizar as análises, a jusante de picos com Homer, BEDTools 10, ou qualquer software preferido.

Figura 1. Imagens do software que mostra como configurar experimentos automatizados ficha no IP-Star Compact. O software oferece a flexibilidade para selecionar a quantidade de amostras por prazo, bem como para alterar os parâmetros experimentais chave (anticorpo de revestimento, IP e lavagens ) de acordo com o pesquisador precisa. O procedimento automatizado permite testar diferentes condições em paralelo (isto é, diferentes tipos e quantidades de anticorpos, os tipos e quantidades diferentes de células e mesmo t diferenteypes e quantidades de esferas magnéticas no mesmo prazo. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 2. Imagens do software que mostra como configurar preparação automatizada biblioteca para a próxima geração de sequenciamento utilizando o kit biblioteca no sistema de automação. Por favor, clique aqui para ver uma versão maior desta figura.
Resultados
Otimizando experimentos chip-seq automatizados para oito diferentes marcadores de histonas
A fim de desenvolver e validar os protocolos ChIP automatizados, anticorpos da classe de chip seq que foram previamente validados em experimentos chip-Seq manuais com sucesso (dados não mostrados) foram selecionados. Os seguintes anticorpos da classe de chip seguintes foram escolhidos para este estudo: anti-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 e -H3K9me3. A especificidade de todos os anticorpos da classe ChIP-seq foi confirmado previamente por dot blot, matrizes de péptidos e experiências de Western Blot (dados não mostrados). Experiências piloto ChIP-qPCR com quantidades crescentes de anticorpo foram realizadas para determinar a sensibilidade dos anticorpos (Figura 3). qPCR com, pelo menos, duas positivas e duas metas de controle negativos foram analisados e perfis com enriquecimentos de positivo sobre o alvo negativo maior do que cinco vezes são qualificados para o seqüenciamento exper iments. É importante a realização de experimentos chip e chip-seq com uma alta qualidade de cromatina cortado. Todas as experiências ChIP mostrados nesta publicação foram realizadas utilizando cromatina fresco. É também possível congelar as células fixas à temperatura de -80 ° C e prosseguir com a preparação da cromatina e de cisalhamento em um dia diferente. No entanto, a cromatina preparada a partir de células fixadas congelados podem se comportar de forma diferente de cromatina preparada na hora e, portanto, condições de sonicação pode precisar de ser otimizado para cada preparação da cromatina. Ao trabalhar com diferentes tipos de células, tampões de corte com diferentes composições de detergente (SDS) pode ser usado. Os tipos celulares, tais como linhas de células primárias ou células cultivadas em suspensão de células são difíceis de cortar e exigirá de elevadas concentrações de SDS (1%), enquanto que as linhas celulares que são fáceis de cortar, tais como HeLa, as concentrações baixas de SDS (0,1%) no buffers de cisalhamento.
res.jpg "/>
Figura 3. Validação de anticorpos ChIP de grau utilizando o sistema de automação. Chip foi realizada com anti-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, H3K9 / 14ac, anticorpos policlonais de coelho -H3K36me3 e -H3K9me3 na cromatina tosquiada de 1 milhão de células HeLa-S3, dependendo das modificações de histonas. Protocolos ChIP automatizado, com volumes de trabalho de 200 ul foram utilizados no instrumento de automação para experimentos de titulação de anticorpos. Quantidades de anticorpos de 1, 2, 5 e 10 mg foram testadas por experimento chip e 2 ug IgG foram utilizados como controle negativo em cada experimento. Enriquecimentos foram avaliadas por qPCR. Os resultados são apresentados como% de entrada (a quantidade relativa de DNA imunoprecipitou comparação com DNA de entrada após análise qPCR). Por favor, clique aqui para ver uma versão maior desta figura.
Depois de validar e determinar optima l quantidades de anticorpos ChIP de grau para ser usado no sistema de automação, experiências ChIP-seq automáticos foram realizados a fim de gerar perfis de sequenciação para cada modificação das histonas (Figura 4).

Figura perfis chip-seq 4. histona gerada por experiências chip-seq automatizadas. A figura mostra os perfis de chip-seq em diferentes regiões genômicas para H3K4me3, H3K9ac e H3K9 / 14acH3K4me2 H3K79me3 e H3K36me3. 4A mostra a distribuição de pico ao longo do completo X cromossômica e 4B a distribuição de uma região de 75 kb em torno do gene GAPDH. 4C mostra os perfis de H3K27me3, H3K36me3 H3K4me3 e numa região de 500 kb em torno do gene MYT1 e 4D mostra a distribuição de H3K9me3 num ZNF12 região circundante 200 kb."target =" _ large.jpg blank "> Clique aqui para ver uma versão maior desta figura.
Histona perfis epigenéticos para seis modificações de histonas diferentes associadas com a expressão do gene foram gerados (H3K4me3, H3K9ac, H3K9 / 14ac, H3K4me2, H3K79me3 e H3K36me3). Figura 4A mostra perfis ChIP-seq ao longo do cromossomo X para os diferentes marcadores de histonas. A correlação altamente pico observado entre os seis perfis diferentes de histonas indica os recursos de sistema automatizado para gerar dados precisos e confiáveis. Figura 4B, 4C e 4D mostram a distribuição de picos para diferentes modificações de histonas em regiões genômicas específicas.
Experimentos de imunoprecipitação da cromatina automatizados para baixo para 200 células
A quantidade mínima de células que podem ser utilizados em ensaios ChIP depende da qualidade da cromatina, o specificity e sensibilidade do anticorpo e a abundância da proteína ou modificação da histona estudado. Seleccionar anticorpos grau boas ChIP-seq é importante quando se trabalha com uma quantidade limitada de amostra e a selecção de reagentes optimizados e transportadores diferentes melhora a eficiência da recuperação de ADN e contribuir para o sucesso da experiência ChIP. Para determinar a quantidade mínima de células que o protocolo de ChIP automatizado pode processar, diferentes quantidades de cromatina, do anticorpo, e as esferas magnéticas foram testados no sistema automatizado de IP-Star ChIP utilizando reagentes especificamente optimizadas para trabalhar com baixas quantidades de cromatina.
Em primeiro lugar, a partir de cromatina de 10.000 células a ultra-sons como descrito no protocolo. Os resultados foram confirmados por ChIP qPCR (Figura 5A), que mostra os enriquecimentos significativas com anticorpo H3K4me3 em regiões de controlo positivo e sinal negligenciável nas regiões de controlo negativo. Para efeito de comparação e à prova de coerência, additionadados obtidos com H3K27ac l, H3K9me3 e anticorpos H3K27me3, utilizando 10.000 células é fornecido.
ChIP experimentos foram realizados em seguida automatizados para demonstrar as capacidades do sistema automatizado para trabalhar com baixas quantidades de células usando o mesmo anticorpo H3K4me3. O chip automatizado bem executada, demonstrada por uma série de dez reacções IP que foram altamente reprodutíveis e comparável com os resultados ChIP manuais (Figura 5B). Experimentos manuais e automatizados foram realizados e os benefícios dos protocolos automatizados foram vistos na redução da experiência para experiência variabilidade (Figura 5C).
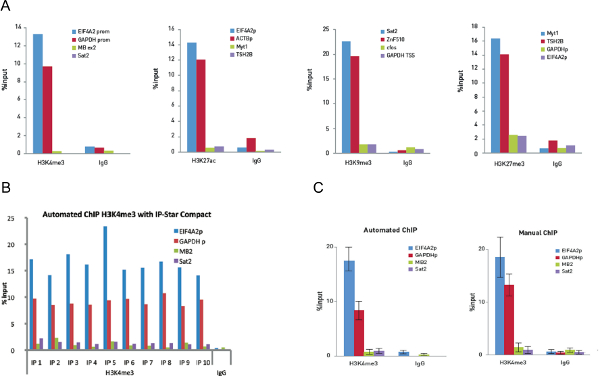
Figura 5. Otimização de experimentos chip e Auto chip em 10.000 células experimentos ChIP manual foram realizadas em 10.000 células e usando 0,25 mg de H3K4me3, 0,1 g de H3K27ac, 0,5 mg de H3K9me3 e 0,25 ug de anticorpos H3K27me3. Quantidades iguais de IgG de coelho foram utilizados como controlo. A qPCR foi realizado com os iniciadores para dois loci positivo e negativo para dois loci cada ensaio ChIP. A Figura 5A mostra a recuperação, expresso como uma percentagem do consumo de (a quantidade relativa de ADN imunoprecipitado em comparação com o ADN de entrada após análise qPCR). A Figura 5B mostra 10 reacções ChIP executado no IP-Star compacta com 0,25 ug H3K4me3 anticorpo policlonal e 0,25 ug de anticorpo IgG de coelho como controlo negativo. Então análise qPCR foi realizada com primers para o positivo promotor loci EIF4A2 e GAPDH TSS eo exon2 loci Myoglobin negativo e SAT2. A figura mostra a recuperação, expressa como uma percentagem de entrada (valor relativo de ADN imunoprecipitado em comparação com o ADN de entrada após análise qPCR). A Figura 5C mostra os dados H3K4me3 ChIP de 10 experiências de ChIP manuais em comparação com as 10 experiências ChIP automatizados. As barras de erro representam s desvios tandard de cada uma das dez repetições. Por favor, clique aqui para ver uma versão maior desta figura.
A fim de compreender a sensibilidade dos protocolos ChIP automatizados, as experiências foram realizadas usando uma quantidade de células que variou de 100.000 para baixo para 200 células por IP. O anticorpo anti-H3K27me3 foi utilizado, pois é uma modificação muito comum histona. A utilização de outros anticorpos de histonas ou não-histonas pode exigir células mais ou menos, dependendo da abundância do epitopo e a qualidade do anticorpo. Os experimentos foram validados por PCR quantitativa e verificou-se que, ao reduzir quantidades de grânulos e fundo anticorpo nas experiências é reduzido permitindo resultados bem sucedidos ChIP-qPCR com tão pouco quanto 200 células de anticorpo (Figura 6).
iles / ftp_upload / 52150 / 52150fig6highres.jpg "/>
Figura 6. automatizado ensaios chip em 200 células. As células Hela-S3 e anticorpo dirigido contra H3K27me3. Cromatina foi tosquiada de 1 milhão de células e diluições em série desta cromatina (de 100.000 a 200 equivalentes de células) foram utilizados por reacção ChIP. 1 ug de H3K27me3 e 10 ul de proteína A-revestido esferas magnéticas foram usadas no experimento 100.000 células, 0,5 ug de H3K27me3 e 10 ul de pérolas de 10.000 e 1.000 células, e 0,25 ug de H3K27me3 e 5 ul de pérolas com 500 e 200 células. 1 mg e 0,5 mg de IgG de coelho foram utilizados como anticorpo controle negativo quando a realização de experimentos com 100.000 células e 1.000 células, respectivamente. 6A mostra a ocupação de genes TSH2B e GAPDH em% sobre a entrada. 6B mostra ocupação relativa do TSH2B versus controle GAPDH negativo região genômica.
Downstream análise dos resultados ChIP-seq sobre10.000 células
A fim de avaliar a qualidade global de experiências ChIP-seq automatizado com baixo número de células de partida, automatizado ensaios ChIP-seguintes foram realizados com 0,25 ug do anticorpo H3K4me3 em 10.000 células HeLa e as experiências foram usadas em ChIP 100.000 células HeLa-S3 como controlo positivo para a experiência. As bibliotecas automatizadas foram preparadas usando a biblioteca MicroPlex reagentes preparação adaptadas às bibliotecas preparadas com baixas quantidades de DNA. Note-se que, embora seja possível realizar experiências bem sucedidas ChIP-automatizados com menos do que 10.000 células, as quantidades de ADN puxado para baixo, não será suficiente para preparar bibliotecas utilizando os reagentes do kit. Geração e sequenciação de aglomerados foram realizados de acordo com as instruções do fabricante. As análises de bioinformática após o seqüenciamento mostrar resultados em circulação a partir do número de células de amostras de baixa CHIP. O conjunto de dados de 30 pg (correspondendo a 10.000 células de material de partida ) Contém ruído de fundo baixo e picos de enriquecimento altamente fiáveis, que são confirmados por tanto o conjunto de dados de 300 pg (correspondendo a 100.000 células de material de partida) e o conjunto de dados H3K4me3 gerado pelo instituto largo para o projecto CODIFICAR o qual foi utilizado como uma referência externa. É importante notar o Top dados proporção 40 sobreposição, que refere-se a um método padrão utilizado no projeto ENCODE 11, na qual o chip-seq é considerado reprodutível se comparar dois conjuntos de dados não existe, pelo menos, uma sobreposição de 80% do melhor 40% dos picos classificados por pontuação significado. O conjunto de dados de 30 pg preenche estes critérios quando comparado ao tanto o conjunto de dados de 300 pg (considerando todos os seus picos, e não apenas o melhor 40%) e os dados do Instituto Broad (Tabela 1). O conjunto de dados de 300 pg mostra picos quase idênticos aos dados do Instituto Broad com um Rácio sobreposição de 40 a 98% (Figura 7).
ighres.jpg "/>
Figura 7. ensaios chip e geração biblioteca em 10.000 células experimentos ChIP-seq foram gerados em 10.000 e 100.000 células HeLa-S3 usando anticorpo H3K4me3 (0,25 mg / mL). As 35 marcas pb foram mapeados para o genoma humano com o alinhador ELAND. Durante o pico subsequente chamando sicer poderia identificar com fiabilidade os enriquecimentos de baixos números de células, bem como a partir de milhões de células. Os conjuntos de dados foram analisados e comparados uns com os outros e para os dados de referência gerados pelo instituto largo. As amostras de células baixas são consistentes e têm muito alta similaridade. A amostra de 30 pg preenche os critérios ENCODE 11 (min. 80% do top 40% dos picos devem sobrepor-se). Por favor, clique aqui para ver uma versão maior desta figura.
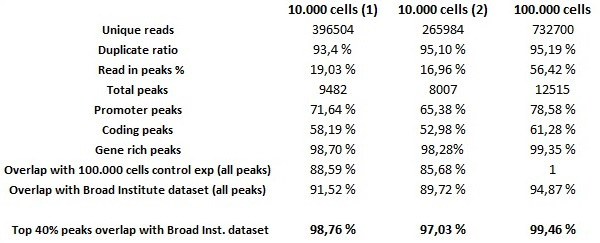
Tabela 1.
Discussão
Immunoprecipitation cromatina seguida de seqüenciamento é agora um procedimento padrão. Aqui um protocolo ChIP-seq automatizado que pode gerar perfis epigenética cromatina com tão poucos como 10.000 células de material de partida é apresentado.
Automatizando chip e preparação biblioteca ensaios permite padronizar o procedimento de otimização chip e reduzir a variabilidade experimental. O sistema de manuseio de líquidos aqui apresentada elimina muitos dos procedimentos manuais associadas com o chip reduzindo as mãos no tempo para apenas 30 min, minimiza a perda de amostra, e permite que precisa ChIP-seq com apenas alguns picogramas de entrada da biblioteca. A fim de realizar experimentos chip-seq automatizados de sucesso, é também crucial para usar preparações cromatina cortados alta qualidade e anticorpos da classe de chip seq em cada experimento O sistema utiliza tecnologia baseada em grânulo magnético e oferece flexibilidade para mudar principais parâmetros experimentais, tais como incubação tempo para o revestimento de anticorpoing e as etapas de imunoprecipitação ou modificação das condições de lavagem, permitindo que o pesquisador para realizar todas as experiências necessárias para a otimização ChIP-seq. O sistema automatizado é uma plataforma de "aberto", que também permite a comparação de vários reagentes em paralelo para a optimização das condições experimentais de cada linha celular individual e anticorpo e permite uma comparação directa de diferentes tipos e concentrações de cromatina, anticorpos diferentes e mesmo diferentes tipos de magnético grânulos.
Uma das limitações do sistema automatizado é a necessidade de automatizar todos os protocolos em volumes que variam de 5 ul a 200 ul. Contudo, a miniaturização dos experimentos nesta plataforma automatizada também permite poupar custos em reagentes.
Em adição com os protocolos descritos no presente estudo, o sistema também é adaptável e automatiza uma variedade de outras aplicações baseadas grânulo magnético, tais como uma imunoprecipitaçãond captura de DNA metilado (tecnologias MEDIP e MethylCap), imunoprecipitação de DNA hydroxylmethylated (hMEDIP), cromatina imunoprecipitação sequencial (ReChIP), immunoprecipitation RNA (RNA-IP), conversão de bissulfito, e ensaios de purificação de DNA.
Divulgações
The authors of this article, at the time of its writing, are employed by Diagenode S.A. and Diagenode, Inc., the manufacturer of the automated system described.
Agradecimentos
This work was supported by the BLUERPINT EU grant (BLUEPRINT – A BLUEPRINT of Haematopoietic Epigenomes). We also thank the Walloon Region (DG06) for its financial support.
Materiais
| Name | Company | Catalog Number | Comments |
| PBS | Life technologies | 14190-094 | |
| Trypsin-EDTA | Sigma | T3924-100ML | |
| Formaldehyde 37% | Sigma | F8775-25 | |
| 1.25 M Glycine Solution | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL2 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Shearing Buffer iS1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Protease Inhibitors Mix (200x) | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| HBSS (no calcium, no magnesium, no phenol red) | Life technologies | 14175-053 | |
| Lysis Buffer tL1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ChIP Buffer tC1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ideal ChIP-seq kit | Diagneode | C01010051 | |
| ChIP-Buffer H | Diagenode | C01010020 | Component of the Auto Histone ChIP-seq kit |
| Auto Histone ChIP-seq kit | Diagenode | C01010020 | |
| Auto True Micro ChIP kit | Diagenode | C01010130 | |
| H3K79me3 polyclonal antibody-Classic | Diagenode | C15310068 | |
| H3K27me3 polyclonal antibody-Classic | Diagenode | C15410069 | |
| H3K4me3 polyclonal antibody-Classic | Diagenode | C15410030 | |
| H3K4me2 polyclonal antibody-Classic | Diagenode | C15410035 | |
| H3K9ac polyclonal antibody-Premium | Diagenode | C15410004 | |
| H3K9/14ac polyclonal antibody-Premium | Diagenode | C15410200 | |
| H3K36me3 polyclonal antibody-Premium | Diagenode | C15410058 | |
| H3K9me3 polyclonal antibody-Premium | Diagenode | C15410193 | |
| Rabbit IgG | Diagenode | C15410206 | |
| Protein-A coated paramagnetic beads | Diagenode | C01010020 | |
| Auto IPure | Diagenode | C03010010 | |
| MicroChIP DiaPure columns | Diagenode | C03040001 | |
| Universal SyberGreenMaster Mix 1.25 ml | Diagenode | DMMLD2D100 | |
| Quant-IT dsDNA | Invitrogen | Q32854 | |
| Illumina Sample Preparation kit fro Genomic DNA | Illumina | FC-121-3001 | |
| Illumina True-seq kit ChIP library Prep kit | Illumina | IP-202-1012 | |
| MicroPlex Library Preparation Kit | Diagenode | C05010010 | |
| Agencourt AMPure XP beads | Beckman Coulter | A63881 | |
| Illumina Library prep Quantification kit | Kapa Biosystems | KK4844 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | |
| Bioruptor Plus | Diagenode | B01020001 | |
| Bioruptor Pico | Diagenode | B01060001 | |
| Qubit system | Invitrogen | Q32857 | |
| Illumina Hiseq systems | Illumina |
Referências
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Adli, M., Bernstein, B. E. Whole-genome chromatin profiling from limited numbers of cells using nano-ChIP-seq. Nature Protocols. 6, 1656-1668 (2011).
- Blecher-Gonen, R., et al. High-throughput chromatin immunoprecipitation for genome-wide mapping of in vivo protein-DNA interactions and epigenomic states. Nature Protocols. 8, 539-554 (2013).
- Aldridge, S., et al. AHT-ChIP-seq: a completely automated robotic protocol for high-throughput chromatin immunoprecipitation. Genome Biology. 14, 124 (2013).
- Farias-Hesson, E., et al. Semi-automated library preparation for high-throughput DNA sequencing platforms. Journal of Biomedicin., & Biotechnology. 2010, (2010).
- Callejas, S., Alvarez, R., Benguria, A., Dopazo, A. AG-NGS: a powerful and user-friendly computing application for the semi-automated preparation of next-generation sequencing libraries using open liquid handling platforms. BioTechniques. 56, 28-35 (2014).
- Zang, C., et al. A clustering approach for identification of enriched domains from histone modification ChIP-Seq data. Bioinformatics. 25, 1952-1958 (2009).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9, 137 (2008).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Molecular Cell. 38, 576-589 (2010).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics (Oxford, England). 26, 841-842 (2010).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados