Method Article
Método EPA 1615. La medición de enterovirus y norovirus Ocurrencia en agua por cultura y RT-qPCR. Parte III. Detección de virus por RT-qPCR
En este artículo
Resumen
Here we present a procedure to quantify enterovirus and norovirus in environmental and drinking waters using reverse transcription-quantitative PCR. Mean virus recovery from groundwater with this standardized procedure from EPA Method 1615 was 20% for poliovirus and 30% for murine norovirus.
Resumen
EPA Method 1615 measures enteroviruses and noroviruses present in environmental and drinking waters. This method was developed with the goal of having a standardized method for use in multiple analytical laboratories during monitoring period 3 of the Unregulated Contaminant Monitoring Rule. Herein we present the protocol for extraction of viral ribonucleic acid (RNA) from water sample concentrates and for quantitatively measuring enterovirus and norovirus concentrations using reverse transcription-quantitative PCR (RT-qPCR). Virus concentrations for the molecular assay are calculated in terms of genomic copies of viral RNA per liter based upon a standard curve. The method uses a number of quality controls to increase data quality and to reduce interlaboratory and intralaboratory variation. The method has been evaluated by examining virus recovery from ground and reagent grade waters seeded with poliovirus type 3 and murine norovirus as a surrogate for human noroviruses. Mean poliovirus recoveries were 20% in groundwaters and 44% in reagent grade water. Mean murine norovirus recoveries with the RT-qPCR assay were 30% in groundwaters and 4% in reagent grade water.
Introducción
PCR cuantitativa (qPCR, ver materiales suplementarios para las definiciones de los términos utilizados en este manuscrito) y transcriptasa reversa-qPCR (RT-qPCR) son herramientas valiosas para la detección y cuantificación de virus entéricos humanos en el medio ambiente y bebiendo aguas, y en especial para muchos virus que hacen no replicar o replicar mal en sistemas de cultivo celular. Ambas herramientas han demostrado que muchos tipos de virus están presentes en aguas ambientales y de agua potable en todo el mundo 6.1. Su uso junto con la secuenciación de los fragmentos genómicos amplificados durante las investigaciones brote de la enfermedad ha proporcionado evidencia de la transmisión del virus a base de agua, ya que han demostrado que el virus encontrado en el agua potable es idéntico al cobertizo por los pacientes de brotes 7-10.
Tanto qPCR y RT-qPCR son herramientas de salud pública útiles. Por ejemplo, los datos de los estudios realizados por la Agencia de Protección Ambiental (EPA) mostraron una relación fuerte seamediciones de los indicadores de interpolación por qPCR y efectos sobre la salud en las aguas de recreo. Como resultado, finales de 2012 Criterios de Calidad del Agua Recreativa de la EPA incluye un método qPCR para vigilar las playas recreativas 11,12. Borchardt y sus colegas también encontraron una fuerte relación entre la gastroenteritis aguda en las comunidades que utilizan las aguas subterráneas sin tratar y virus en las aguas subterráneas, medida por RT-qPCR 1.
El propósito de este trabajo es describir el componente de ensayo molecular del Método EPA 1615 13,14. Este ensayo usa RT-qPCR para proporcionar una estimación cuantitativa de enterovirus y norovirus copias genómicas (CG) por litro basado en el volumen original de agua del medio ambiente o beber pasado a través de un filtro de electropositivo. Una visión general del procedimiento molecular se muestra en la Figura sección 1. Protocolo 1 detalla los procedimientos para la preparación de la curva estándar. Estos estándares se preparan a partir de un reactivo que contiene un RNUna copia de la secuencia diana para todos los conjuntos de cebador / sonda. Sección 2 describe el procedimiento de concentración terciario. Sección 3 da el procedimiento para la extracción de ARN de las muestras de agua y control concentrado. El ARN de cada muestra de prueba se transcribe invertir utilizando ensayos por triplicado y cebadores aleatorios para cebar la transcripción (Sección 4). El ADNc de cada reacción de transcripción inversa se divide en cinco ensayos específicos del virus separadas que se analizan por triplicado mediante qPCR (Sección 5; Figura 2). El ensayo usa cebadores y sondas de la literatura científica (Tabla 1) que están diseñados para detectar muchos enterovirus y norovirus y un reactivo que contiene hepatitis G ARN para identificar muestras de ensayo que son inhibidores de la RT-qPCR 15.
Protocolo
Nota: Las hojas de datos utilizar para rastrear todos los pasos del protocolo; hojas de datos de ejemplo se dan en el material complementario Tablas S2-S4.
1. Preparación de la curva estándar
- Preparar una solución de trabajo del reactivo curva estándar (por ejemplo, Armored RNA EPA-1615) diluyéndola partir de la concentración suministrado por el fabricante a una concentración de 2,5 x 10 8 partículas / ml (2,5 x 10 8 GC / ml) utilizando TSM tampón III. Divida la madre de trabajo en 250 alícuotas mu l utilizando 1,5 ml tubos de microcentrífuga y se almacena a -20 ° C.
NOTA: Ver protocolo de materiales suplementarios Paso S1 para obtener instrucciones sobre la preparación de las reservas de trabajo de virus y plásmidos para su uso como reactivos curva estándar alternativos. - Descongelar una o más de las alícuotas del stock de trabajo. Preparar cinco diluciones seriadas de 10 veces utilizando 1,5 ml tubos de microcentrífuga, dando concentraciones de 2,5 x 10 7, 2.5 x 10 6, 2.5 x10 5, 2,5 x 10 4 y 2.5 x 10 3 GC / ml.
- Preparar la primera dilución añadiendo 25 l de la / ml madre de trabajo 2,5 x 10 8 GC 225 l de tampón de TSM III. Mezclar durante 5-15 segundos utilizando un mezclador vortex.
- Preparar la siguiente dilución añadiendo 25 l de la dilución preparada en el paso 1.2.1 a 225 l de tampón de TSM III. Mezclar de nuevo y seguir un proceso similar para preparar los próximos tres diluciones de 10 veces.
- Extraer el ARN de la madre de trabajo curva estándar y los cinco diluciones inmediatamente utilizando el procedimiento de la Sección 3.
2. Concentración terciaria
- Preparar un concentrador centrífugo (corte de peso molecular 30.000) para cada muestra recogida mediante la adición de al menos 10 ml de 1x PBS, 0,2% de albúmina de suero bovino (BSA) a la cámara de muestra superior. Asegúrese de que la solución ha llenado la cámara de concentración canal delgada, y luego celebrar O / N a 4 ° C.
- Deseche el fluido. Enjuague el concentrador de una vez con al menos 10 ml de agua estéril de grado reactivo para eliminar el exceso de BSA y luego deseche el agua.
- Añadir una cantidad de concentrado de agua secundaria de cada muestra de prueba igual a S, el ensayo Volumen de muestra en concentradores centrífugos separadas.
- Calcular S utilizando la ecuación 1,
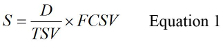
Donde D es el volumen de la muestra original de agua ensayada, TSV es el volumen total de la muestra y FCSV es el concentrado de volumen de la muestra final. Ver materiales suplementarios S2 para un ejemplo del cálculo del S.
- Calcular S utilizando la ecuación 1,
- Centrifugar cada muestra de ensayo a 3.000-6.000 xg a 4 ° C en un rotor de cubeta oscilante durante 20 - 30 min. Compruebe el volumen en la cámara de concentración canal delgada.
- Si el volumen es mayor que 400 l, centrifugar de nuevo durante 20 min o más. Continuar centrifugación hasta que la muestra in the cámara de concentración canal delgada se ha reducido a menos de 400 l. No retire el sobrenadante.
- Lavar los lados del concentrador centrífugo con 1 ml de fosfato de sodio 0,15 M estéril, pH 7-7.5 para aumentar la recuperación de virus. Centrifugar de nuevo a 3.000-6.000 xg y 4 ° C hasta que la muestra se ha reducido a menos de 400 l. Repita este paso de lavado una vez adicional.
- Usando una micropipeta 100-200 l, medir y transferir cada muestra se concentró a un tubo de microcentrífuga de 1,5 ml (es decir, la transferencia de 200 l al tubo de microcentrífuga y luego medir el concentrado restante mediante el ajuste de la micropipeta hasta que el líquido restante puede ser completamente elaborado cuidadosamente en la punta de pipeta). Añadir fosfato de sodio 0,15 M, pH 7-7.5, para ajustar el volumen final a 400 ± 2 l.
- Extraer el ácido nucleico de inmediato por continuar con el paso 3. Mantenga las muestras concentradas que no puede ser processed inmediatamente a 4 ° C por no más de 24 horas.
3. Nucleic Acid Aislamiento
- Añadir 200 l de tampón de extracción preparado como se describe en el Paso 3.3 y 200 l de concentrado terciaria de cada muestra de prueba de la Etapa 2.5 o dilución curva estándar de la Etapa 1.3 para separar la etiqueta 1,5 ml tubos de microcentrífuga. Congele restante concentrados terciarios en o por debajo de -70 ° C. Extraer los ácidos nucleicos a partir de cada muestra según las instrucciones del fabricante de extracción de ácido nucleico kit para el protocolo de giro para muestras de sangre con las siguientes excepciones.
- No añadir proteasa a las muestras de agua o utilizar el tampón de extracción se suministra con el kit de extracción de ácido nucleico.
- Preparar tampón de extracción con ARN transportador
- Añadir 310 l de tampón de dilución de ARN transportador a un vial que contiene 310 g de ARN portador. Mezcle para disolver y luego dividir en 6 alícuotas que contienen alrededor de 50 l. Conservar a -20DO.
- Añadir 28 l de ARN transportador descongelado por ml del tampón de extracción para obtener una concentración de ARN portador de 0,027 g / l. Utilice el tampón de extracción de ARN modificado de soporte en lugar del que se suministra con el kit de extracción.
- Preparar una solución de tampón de elución principal mediante la adición de ribonucleasa (RNasa) Inhibidor hasta una concentración final de 400 unidades / ml para el tampón de elución suministrado con el kit de extracción.
- Eluir el ARN de la unión de ácidos nucleicos columna de centrifugación mediante la colocación de 50 l de tampón de elución con inhibidor de RNasa en la columna. Espere 1 min, y luego centrifugar a 6000 xg durante 1 min a TA.
- Repita el paso 3.4.1 y luego retire y deseche la columna.
- Preparar alícuotas de los extractos de ARN a partir del paso 3.4.2. Preparar 6 alícuotas de la solución de trabajo curva estándar y cada dilución curva estándar que contiene por lo menos 15 l cada uno. Preparar 4 alícuotas de todas las demás muestras de ARN que contienen al menos22 l cada uno. Almacenar una alícuota de cada muestra y la curva estándar de los controles a 4 ° C si pueden ser procesadas por transcripción inversa dentro de 4 horas; de lo contrario, almacenar todos alícuotas en o por debajo de -70 ° C.
4. transcripción inversa (RT)
- Preparar 100 mM de soluciones madre de cada cebador de oligonucleótido y la sonda enumerados en la Tabla 1 mediante la adición de un volumen de agua de calidad para PCR a cada vial utilizando una cantidad (en microlitros) igual a diez veces el número de nanomoles (nmoles) de oligonucleótido presentes en el vial (como se muestra en la etiqueta o en la hoja de especificaciones del fabricante (por ejemplo, volver a suspender una cartilla que contiene 36,3 nmol en 363 l). Mezclar hasta disolver.
- Diluir los 100 M soluciones 1:10 con agua PCR-grado para preparar 10 M soluciones de trabajo.
- Preparar RT Master Mix 1 y 2 en una habitación limpia con la guía en la Tabla 2. Pipetear 16,5 l de RT Master Mix 1 a cada placa de PCR pocillo usando una pipeta multicanal.
- Thaw, si congela, los extractos de ácidos nucleicos de cada muestra de campo y laboratorio matriz de la muestra fortificada (LFSM; es decir, la muestra matriz de agua sin semillas).
- Diluir cada campo y muestra LFSM 1: 5 y 1:25 en tampón de elución que contiene 400 unidades / ml de inhibidor de RNasa.
- Thaw, si congela, pero no lo diluya Lab fortificada en blanco (LFB, es decir, un control positivo de la calidad utilizando agua sin semillas grado reactivo), Laboratorio Blanco de reactivo (LRB, es decir, un control negativo calidad utilizando agua de grado reactivo), Evaluación del Desempeño (PE , es decir, las muestras de agua de grado reactivo sembrado utilizan para evaluar el rendimiento de laboratorio antes del inicio de un estudio), Test Performance (PT, es decir, las muestras de agua de grado reactivo sembrado con títulos desconocidos a un analista que se utilizan para evaluar el rendimiento de laboratorio durante un estudio ), el control de la extracción negativo NA lotes, o ARN extraído del conjunto curva estándar.
- Coloque 6.7 l de la ARN a partir de cada muestra de prueba, control, y la curva estándar en pocillos de placas de PCR separadas, utilizando pocillos por triplicado para las muestras de ensayo y controles y pocillos duplicados para las curvas de calibración (véase la figura S1 para un ejemplo placa de RT).
- Coloque 6.7 l de tampón de elución en pocillos de placas de PCR separadas para controles sin plantilla (NTC). Incluir 2-8 NTC por placa RT, utilizando dos para la primera muestra y luego dos más por cada cuarta muestra adicional.
- Distribuir la extracción negativo y controles NTC en toda la placa.
- Selle la placa de PCR con una placa resistente sellador térmico. Mezcle las muestras durante 5-10 segundos y centrifugar a ≥ 500 xg brevemente.
- Incubar la placa durante 4 min a 99 ° C y luego enfriar rápidamente a 4 ° C en un ciclador térmico. Centrifugar de nuevo en ≥ 500 xg brevemente.
- Retire con cuidado la junta de la placa y luego añadir 16,8 l de RT Master Mix 2 a cada pocillo. Seal de la placa de nuevo con un sellador de placa resistente al calor, seguido de la mezcla y una breve centrifugación a ≥ 500 x g.
- Colocar la placa en un ciclador térmico y una duración de 15 min a 25 ° C, seguido por 60 min a 42 ° C, 5 min a 99 ° C, y luego por un ciclo de 4 ° C espera.
- Proceso de inmediato o dentro de 8 horas por qPCR (Paso 5), o almacenar las muestras en o por debajo de -70 ° C hasta que se puede procesar. Almacene las muestras que se pueden procesar en 8 horas a 4 ° C.
5. En tiempo real PCR cuantitativa (qPCR)
- Determinar el valor medio de la hepatitis G Cq para cada lote de reactivo G hepatitis antes de ejecutar cualquier muestras de ensayo.
- Ejecutar un ensayo de RT utilizando 10 réplicas preparados como se describe para los controles NTC (Paso 4.1.1). Ejecutar el ensayo G qPCR hepatitis como se describe a continuación (Pasos 5.2 a 5.5.3). Calcular el valor medio Cq de las 10 repeticiones.
- Ajuste la hepatitis G cantidad de reactivo en RT Master Mix 1 (Tabla 2), Si es necesario, para obtener un valor medio Cq entre 25 y 32 unidades. Compensar la cantidad subir o bajar al cambiar el volumen de agua añadida para mantener la RT Master Mix 1 volumen final de 16,5 l por ensayo.
- Confirme los ajustes por Pasos 5.1.1 a 5.1.2 y el cambio Tabla 2 repitiendo para reflejar las cantidades ajustadas.
- Preparar maestros mezclas qPCR en una habitación limpia utilizando las guías en la Tabla 3 para enterovirus, Tabla 4 y Tabla 5 para el norovirus genogrupo I, Tabla 6 para el norovirus genogrupo II, Tabla 7 para el norovirus murino (norovirus genogrupo V), y en la Tabla 8 para la hepatitis G. Mix y cada mezcla maestra centrifugar a ≥ 500 xg brevemente.
- Añadir las mezclas de reacción de PCR a los pocillos apropiados de una placa de reacción óptica etiquetados, utilizando 14 l por pocillo y las placas separadas para cada ensayo qPCR (ver Figura S2 para un posible diseño para un ensayo de qPCR basado en el diseño de RT en la Figura S1).
- Descongele la placa RT desde el paso 4.8 a temperatura ambiente, si se congela. Mezclar con un mezclador plato y centrifugar a ≥ 500 xg brevemente.
- Dispensar 6 l de la cDNA apropiada a los pocillos apropiados de la placa de reacción óptica. Mezclar las muestras en la placa de reacción óptica y centrifugar a ≥ 500 xg brevemente.
- Ejecute la hepatitis G ensayo qPCR en el campo sin diluir y diluidos y muestras LFSM antes de ejecutar todos los otros ensayos de qPCR. Utilice la dilución más baja del terreno o de la muestra LFSM que es <1 Cq valor mayor que el valor medio de la hepatitis G Cq para los ensayos de qPCR enterovirus y norovirus.
- Configurar el software termociclador de PCR cuantitativa de acuerdo con las instrucciones del fabricante. Identificar las muestras de la curva estándar como las normas y para cada dilución curva estándar, introduzca los valores de copia genómicas que se muestran en la Tabla 9.
- Ejecutar la placa en la cuantitativa termociclador de PCR durante 10 minutos a 95 ° C, seguido por 45 ciclos de 15 segundos a 95 ° C y 1 min a 60 ° C.
- Determina si cada curva estándar cumple con los valores aceptables indicados en la tabla 10. Vea la sección materiales suplementarios S3 para ejemplos.
- Calcula la desviación estándar global (DesviaciónEstándar) para la curva estándar utilizando la ecuación 2,
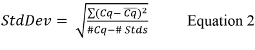
donde Cq es el valor reportado para cada réplica curva estándar, Cq es el valor medio para cada conjunto de réplicas, #Cq es el número total de valores Cq para todas las repeticiones de control estándar que tienen valores positivos (es decir, no indeterminada), y # Stds es el número de controles estándar que tienen valores positivos. - Si el software termociclador de PCR cuantitativa no calcula la pendiente de cada curva estándar, calcular la pendiente utilizando la ecuación 3,60;
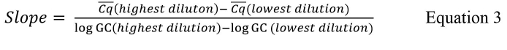
donde Cq es el valor medio de las diluciones altas y más bajas utilizadas log GC es el logaritmo del valor copia genómica de las diluciones altas y más bajas utilizadas en la Tabla 9. - Calcular el valor de R 2 utilizando la ecuación 4.
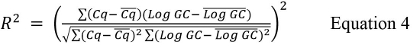
donde Cq es la media de todos los valores de la CQ y registro de GC es el valor medio de registro GC para cada réplica. - Calcular la eficiencia% utilizando la ecuación 5:
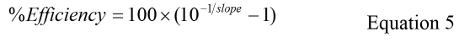
- Calcula la desviación estándar global (DesviaciónEstándar) para la curva estándar utilizando la ecuación 2,
- Registre los valores GC calculados por el software termociclador para todas las muestras de prueba basados en las curvas de calibración que cumplen los criterios especificados en la Tabla 10 y los valores medios de GC para cada muestra. Vuelva a ejecutar las muestras con las curvas de calibración que no cumplan con los criterios establecidos en Tabla 10 o cuando los controles negativos (LRB, el control de la extracción negativo NA lotes o NTC) son positivos. Vuelva a procesar las muestras que no cumplen con los criterios o tienen controles positivos falsos durante la repetición.
- Determinar la GC por litro (GC L) para cada muestra de ensayo usando la Ecuación 6:
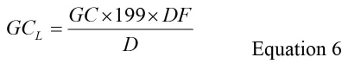
donde GC es el número de copias del genoma media desde el paso 5.7, el factor '199' es el factor de dilución total de las reducciones de volumen que se producen durante la concentración terciario, extracción de RNA y pasos de RT-qPCR, DF es el factor de dilución que compensa la inhibición y D es el volumen de la muestra original de agua ensayada en litros. Ver materiales suplementarios sección S4 para un ejemplo del cálculo del GC L. - Calcule el GC total de LFB y LRB muestras multiplicando el valor medio de GC desde el Paso 5,5 por 199 y dividiendo por 0,3.
Resultados
La recuperación total del virus se determinó a través de campo vinculado y muestras de agua subterránea LFSM. Un total de siete conjuntos de muestras se analizaron mediante dos conjuntos recogidos en distintas ocasiones a partir de tres plantas de tratamiento públicos y un conjunto de muestras recogidas del pozo privado. Niveles de semillas para las muestras LFSM eran 3 x 10 6 MPN de Sabin poliovirus serotipo 3 y 5 x 10 6 PFU de norovirus murino. Norovirus murino fue utilizado como un sustituto en la evaluación método debido a la falta de existencias de norovirus humanos con una concentración de virus suficiente para muestras LFSM. Para muestras de agua subterránea la recuperación media fue de poliovirus 20%, con un error estándar de 2%, mientras que 14 significa la recuperación norovirus murino fue 30%, con un error estándar de 3% (Figura 3). La muestra habitual campo de las aguas subterráneas para cada LFSM no tenía enterovirus detectable o norovirus.
Muestras LFB y LRB se midieron utilizando wat sin semillas y cabeza de serie de grado reactivoer. Todos los LRB muestras fueron negativas (datos no mostrados). Poliovirus de recuperación promedio de 44% con un error estándar de 1% (Figura 3), mientras que la recuperación norovirus murino promedio de 4% con un error estándar de 0.5%.
RT-qPCR requiere el uso de reactivos adecuados curva estándar. La Figura 4 muestra una curva estándar típica para enterovirus y norovirus GII. La curva GII norovirus cumple con los criterios de desempeño curva estándar (Cuadro 10) con un valor de R 2 de 0,9987, una desviación estándar global de 0,14, y la eficiencia del 101%. Norovirus GIA y curvas GIB (no se muestra) son casi idénticas a la de GII norovirus. La curva de enterovirus cumple con los criterios de desempeño método con un valor de R 2 de 0,9874, una desviación estándar global de 0,58, y la eficiencia del 103%, pero tiene unos cien veces menos sensibilidad y por lo tanto un límite de detección más alto que las curvas de norovirus.
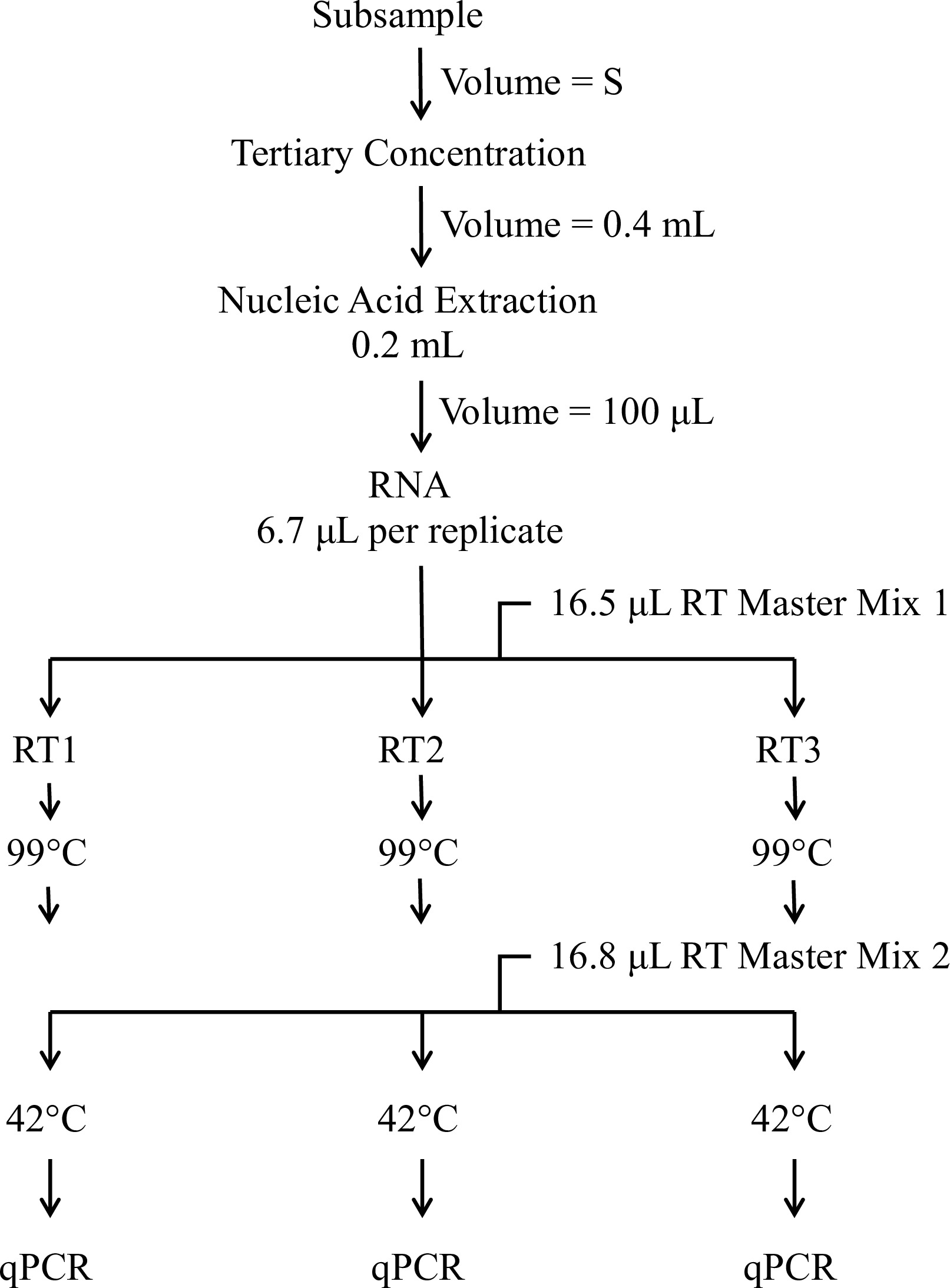
Figura 1. Vista general del Procedimiento Molecular. El procedimiento molecular incluye concentración de la muestra adicional más allá de la realizada para medir el virus infeccioso, extracción de ácidos nucleicos, a dos pasos del protocolo de transcripción inversa (RT), y la PCR cuantitativa (qPCR). El volumen de partida (S) representa una proporción método definido de la muestra original de agua.
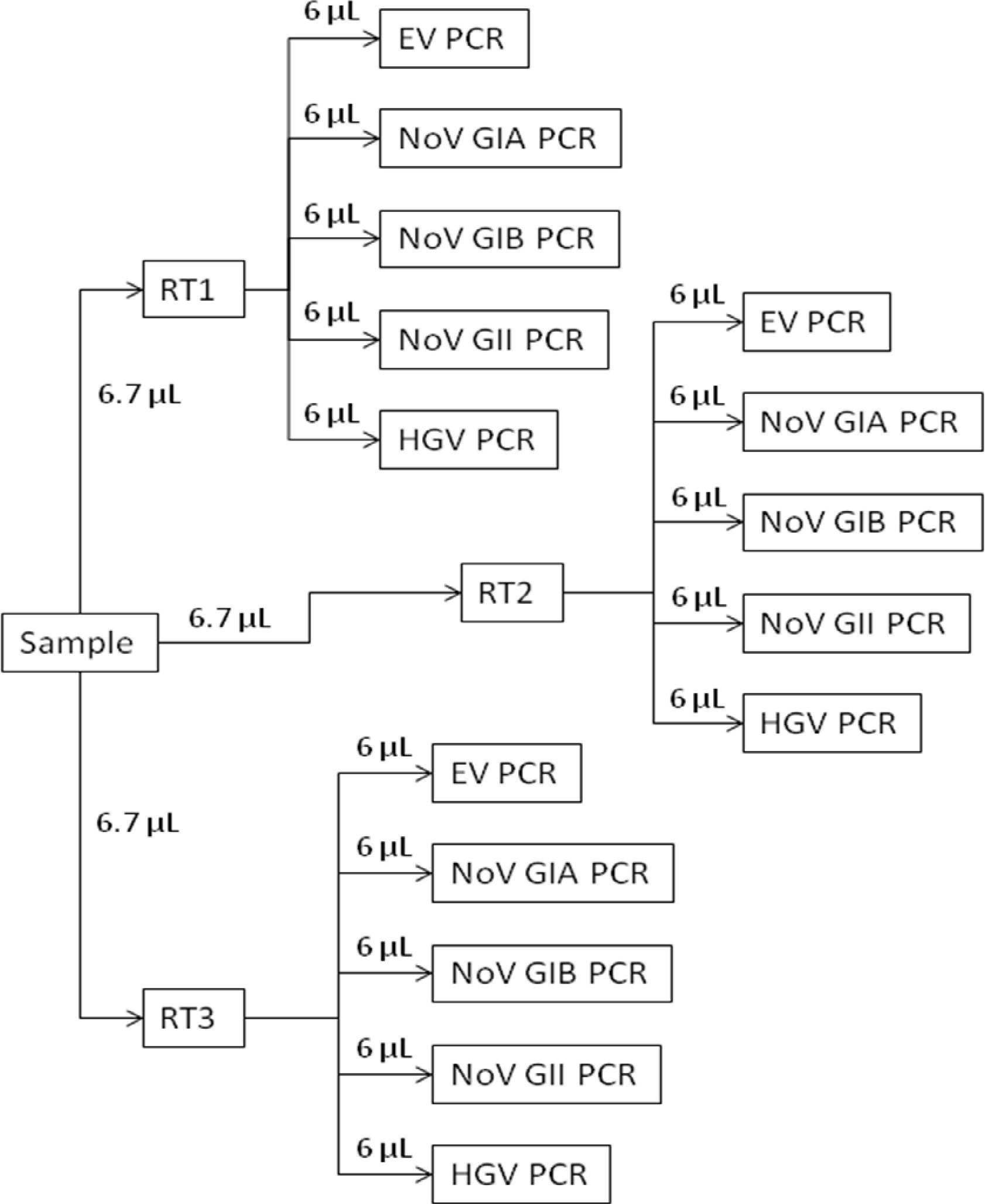
Figura 2. RT-qPCR esquemática visión general. Cada ARN muestra de prueba extraída se transcribe invertir utilizando ensayos por triplicado (RT1, RT2 y RT3). El ADNc a partir de cada uno de los ensayos de RT por triplicado a continuación, se analiza en busca de virus específicos utilizando enterovirus separado (EV PCR), genogrupo norovirus I (NOV GIA PCR y el NoV GIB PCR), norovirus genogrupo II (NoV GII PCR), y los ensayos de la hepatitis G (HGV PCR).
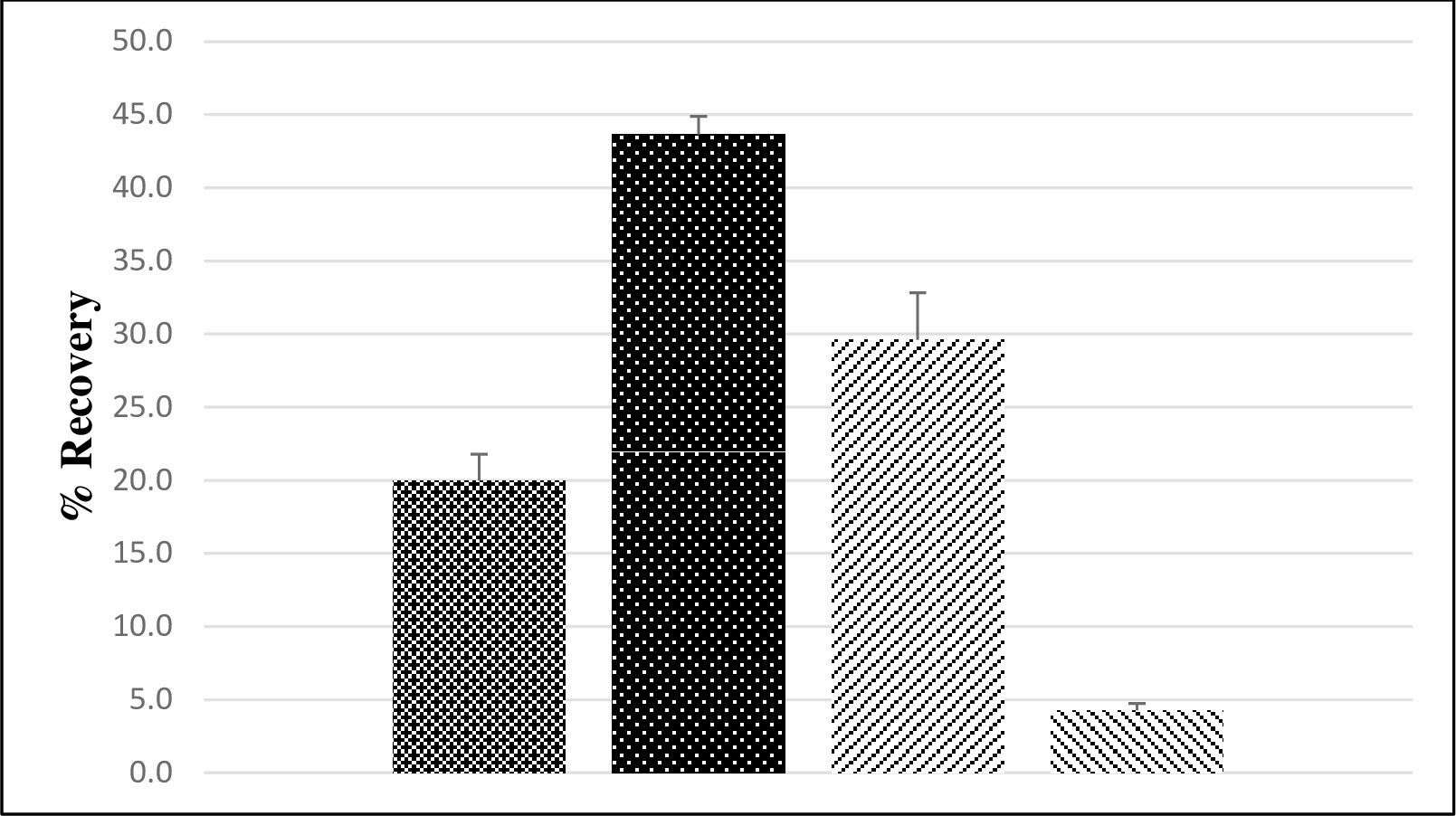
Figura 3. Media poliovirus y murino norovirus Recuperación (%) de la Zona y el Agua. El porcentaje de recuperación promedio de grado reactivo se muestra para poliovirus de suelo (  ; n = 7) y de grado reactivo (
; n = 7) y de grado reactivo (  ; n = 12) de agua y para el norovirus murino de suelo (
; n = 12) de agua y para el norovirus murino de suelo (  ; n = 7) y de grado reactivo (
; n = 7) y de grado reactivo (  ; n = 12) agua (1), donde "n" es el número de muestras de agua separadas procesados. Las barras de error representan el error estándar.
; n = 12) agua (1), donde "n" es el número de muestras de agua separadas procesados. Las barras de error representan el error estándar.

Figura 4. enterovirus y norovirus GII curva estándar. Curvas estándar típicas para enterovirus y norovirus GII se muestran. Las fórmulas que dan pendiente y R 2 valores para cada curva se calculan por el termociclador.
1. Archivo Suplementario Por favor haga clic aquí para descargar este archivo.
| Grupo Virus | Primer / Nombre de la sonda (1) | Secuencia (2) | Referencia | |
| Enterovirus | ||||
| Entf (EV-L) | CCTCCGGCCCCTGAATG | 20 | ||
| EntR (EV-R) | ACCGGATGGCCAATCCAA | 20 | ||
| ENTP (Ev-sonda) | 6FAM-CGGAACCGACTACTTTGGGTGTCCGT-TAMRA | 21 | ||
| Norovirus GIA | ||||
| NorGIAF (JJV1F) | GCCATGTTCCGITGGATG | 22 | ||
| NorGIAR (JJV1R) | TCCTTAGACGCCATCATCAT | 22 | ||
| NorGIAP (JJV1P) | 6FAM-TGTGGACAGGAGATCGCAATCTC-TAMRA | 22 | ||
| Norovirus GIB | ||||
| NorGIBF (QNIF4) | CGCTGGATGCGNTTCCAT | 23 | ||
| NorGIBR (NV1LCR) | CCTTAGACGCCATCATCATTTAC | 23 | ||
| NorGIBP (NV1LCpr) | 6FAM-TGGACAGGAGAYCGCRATCT-TAMRA | 23 | ||
| El norovirus GII | ||||
| NorGIIF (QNIF2d) | ATGTTCAGRTGGATGAGRTTCTCWGA | 25 | ||
| NorGIIR (COG2R) | TCGACGCCATCTTCATTCACA | 25 | ||
| NorGIIP (QNIFS) | 6FAM-AGCACGTGGGAGGGCGATCG-TAMRA | 25 | ||
| Norovirus GV | ||||
| MuNoVF1 | AGATCAGCTTAAGCCCTATTCAGAAC | 14 | ||
| MuNoVR1 | CAAGCTCTCACAAGCCTTCTTAAA | 14 | ||
| MuNoVP1 | VIC-TGGCCAGGGCTTCTGT-MGB | 14 | ||
| Hepatitis G | ||||
| HepF (5'-NCR cebador directo) | CGGCCAAAAGGTGGTGGATG | 19 | ||
| HEPR (5'-NCR cebador inverso) | CGACGAGCCTGACGTCGGG | 19 | ||
| Hepp (hepatitis G Sonda TaqMan | 6FAM-AGGTCCCTCTGGCGCTTGTGGCGAG-TAMRA | 1 | ||
Tabla 1. Los cebadores y sondas TaqMan para la detección de virus por RT-qPCR.
(1) Método 1615 de imprimación y nombres de sonda son las tres primeras letras del nombre del virus concatenado a F, R o P para adelante, atrás, y la sonda. El norovirus genogrupo se designa mediante la adición de GI y GII a los nombres. Los dos conjuntos de primer GI norovirus también se distinguen usando A y B. Primer sonda y los nombres de las referencias principales se dan en los padresheses.
(2) La orientación de las secuencias de cebador y sonda es 5 'a 3'. Los siguientes indicadores de bases degeneradas se utilizan: N-una mezcla de los cuatro nucleótidos; R-A + G; Y-T + C; W-A + T; y la I-inosina.
| Ingrediente | Volumen por reacción de (l) (2) | La concentración final | Volumen por Master Mix (l) (3) |
| RT Master Mix 1 | |||
| Cebador aleatorio | 0.8 | 10 ng / l (c. 5,6 M) | 84 |
| Hepatitis G Armored RNA (4) | 1 | 105 | |
| Agua de calidad PCR | 14.7 | 1543.5 | |
| Atal | 16.5 | 1732.5 | |
| RT Master Mix 2 | |||
| 10x PCR Buffer II | 4 | Tris 10 mM, pH 8,3, KCl 50 mM | 420 |
| 25 mM MgCl 2 | 4.8 | 3 mM | 504 |
| DNTPs 10 mM- | 3.2 | 0,8 mm | 336 |
| 100-mM de DTT | 4 | 10 mM | 420 |
| Inhibidor de RNasa | 0.5 | 0,5 unidades / l | 52.5 |
| Superíndice II RT | 0.3 | 1,6 unidades / l | 31.5 |
| Total | 16.8 | 1764 | |
Tabla 2. RT Master Mix 1 y 2 (1).
(1) Preparar RT Maestro Mezclas en un clhabitación ean, es decir, una habitación donde no se realizan procedimientos moleculares y microbiológicos.
(2) El volumen final de ensayo RT es 40-l.
(3) Los volúmenes muestran se basan en 105 ensayos. Esto es suficiente para una placa de PCR de 96 pocillos con los ensayos adicionales añadidos para dar cuenta de las pérdidas. La cantidad puede hacerse a escala arriba o hacia abajo de acuerdo con el número de muestras y controles que será analizada.
(4) Determinar la cantidad de reactivo G hepatitis incluir en el RT Master Mix 1 como se describe en materiales suplementarios Paso S4.
| Ingrediente | Volumen por reacción de (l) (2) | La concentración final | Volumen por Master Mix (l) (3) |
| 2x LightCycler 480 Sondas Master Mix | 10 | Propiedad | 1050 |
| ROX referenciacolorante (4) | 0.4 | 0,5 mM | 42 |
| Agua de calidad PCR | 1 | 105 | |
| Entf 10 mM | 0.6 | 300 nM | 63 |
| 10 M EntR | 1.8 | 900 nM | 189 |
| 10 M ENTP | 0.2 | 100 nM | 21 |
| Total | 14 | 1470 |
Tabla 3. PCR Master Mix para Enterovirus (EV) ensayo (1).
(1) Preparar todos los Maestros mezclas de PCR en una habitación limpia.
(2) El volumen final de ensayo qPCR es de 20 l.
(3) Los volúmenes muestran se basan en 105 ensayos. Esto es suficiente para una placa de PCR de 96 pocillos con los ensayos adicionales añadidos para dar cuenta de las pérdidas. La cantidad puede escalar hacia arriba o hacia abajo en función del número de muestras y controles queanalizar.
(4) Sustituto de agua de calidad PCR para este reactivo al utilizar instrumentos que no lo requieran.
| Ingrediente | Volumen por reacción de (l) (2) | La concentración final | Volumen por Master Mix (l) (3) |
| 2x LightCycler 480 Sondas Master Mix | 10 | Propiedad | 1050 |
| Tinte referencia ROX (4) | 0.4 | 0,5 mM | 42 |
| Agua de calidad PCR | 1.4 | 147 | |
| 10 M NorGIAF | 1 | 500 nM | 105 |
| 10 M NorGIAR | 1 | 500 nM | 105 |
| 10 M NorGIAP | 0.2 | 100 nM | 21 |
| Total | 14 | 1470 |
Tabla 4. PCR Master Mix para Norovirus GIA (NOV GIA) Ensayo (1).
Ver Tabla 3 para las notas (1) - (4).
| Ingrediente | Volumen por reacción (l) (2) | Concentración final | Volumen por Master Mix (l) (3) |
| 2x LightCycler 480 Sondas Master Mix | 10 | Propiedad | 1050 |
| Tinte referencia ROX (4) | 0.4 | 0,5 mM | 42 |
| Agua de calidad PCR | 0.3 | 31.5 | |
| 10 M NorGIBF | 1 | 500 nM | 105 |
| 10 M NorGIBR | 1.8 | 900 nM | 189 |
| 10 M NorGIBP | 0.5 | 250 nM | 52.5 |
| Total | 14 | 1470 |
Tabla 5. PCR Master Mix para Norovirus GIB (NOV GIB) Ensayo (1).
Ver Tabla 3 para las notas (1) - (4).
| Ingrediente | Volumen por reacción (l) (2) | Concentración final | Volumen por Master Mix (l) (3) |
| 2x LightCycler 480 Sondas Master Mix | 10 | Propiedad | 1050 |
| Tinte referencia ROX (4) | 0.4 | 0,5 mM | 42 |
| Agua de calidad PCR | 0.3 | 31.5 | |
| 10 M NorGIIF | 1 | 500 nM | 105 |
| 10 M NorGIIR | 1.8 | 900 nM | 189 |
| 10 M NorGIIP | 0.5 | 250 nM | 52.5 |
| Total | 14 | 1470 |
Tabla 6. PCR Master Mix para norovirus GII (NOV GII) Ensayo (1).
Ver Tabla 3 para las notas (1) - (4).
| Ingrediente | Volumen por reacción (l) (2) | Concentración final | Volumen por Master Mix (l) (3) |
| 2x LightCycler 480 Sondas Master Mix | 10 | Propiedad | 1050 |
| Tinte referencia ROX (4) | 0.4 | 0,5 mM | 42 |
| Agua de calidad PCR | 0.3 | 31.5 | |
| 10 M MuNoVF1 | 1 | 500 nM | 105 |
| 10 M MuNoVR1 | 1.8 | 900 nM | 189 |
| 10 M MuNoVP1 | 0.5 | 250 nM | 52.5 |
| Total | 14 | 1470 |
Tabla 7. PCR Master Mix para murino norovirus Ensayo (1).
Ver Tabla 3 para las notas (1) - (4).
| Ingrediente | Volumenpor reacción (l) (2) | Concentración final | Volumen por Master Mix (l) (3) |
| 2x LightCycler 480 Sondas Master Mix | 10 | Propiedad | 1050 |
| Tinte referencia ROX (4) | 0.4 | 0,5 mM | 42 |
| Agua de calidad PCR | 1.4 | 147 | |
| 10 M HepF | 1 | 500 nM | 105 |
| 10 M HEPR | 1 | 500 nM | 105 |
| 10 M Hepp | 0.2 | 100 nM | 21 |
| Total | 14 | 1470 |
Tabla 8. PCR Master Mix para la hepatitis G (HGV) Ensayo (1).
Ver Tabla 3 para las notas (1) - (4).
| Concentración Curva Estándar | Las copias genómicas por RT-qPCR ensayo (1, 2) |
| 2,5 x 10 8 | 502500 |
| 2,5 x 10 7 | 50250 |
| 2,5 x 10 6 | 5025 |
| 2,5 x 10 5 | 502.5 |
| 2,5 x 10 4 | 50.25 |
| 2,5 x 10 3 | 5,025 |
Tabla 9. Copias curva estándar genómica.
(1) Identificar los pozos de la curva estándar como las normas y colocar las copias genómicas por valores de ensayo de RT-qPCR en el lugar adecuado en el software termociclador.
(2) Una curva estándar aceptable tendrá una eficiencia del 70% -110%, Un valor de R 2> 0,97 y una desviación estándar global de <0,5 para el norovirus y <1,0 para enterovirus.
| Criterios | Valor Aceptable | |
| Norovirus | Enterovirus | |
| Desviación Estándar general | <0,5 | <1,0 |
| R 2 | > 0.97 | > 0.97 |
| Eficiencia | 70% a 115% | 70% a 115% |
Tabla 10. Criterios de curva estándar de aceptación (1).
(1) Las curvas de calibración con% de eficiencia de 70% -110% son aceptables, pero los valores en el rango de 90% -115% son ideales. Los valores inferiores a 90% pueden indicar errores de pipeteo o dilución.
| QA Componente | La media de la gama de Recuperación (%) | Coeficiente de variación (%) |
| Laboratorio Blanco de reactivo; muestras PT o PE negativos | 0 | N / A (1) |
| Lab fortificada en blanco; Lab matriz de la muestra fortificada | 5-200 | N / A |
| Muestras PT positiva y PE | 15-175 | ≤130 |
Tabla 11. Método 1615 Criterios de Desempeño.
(1) No aplicable.
| Medios de comunicación | Composición |
| Fosfato de sodio 0,15 M, pH 7.0-7.5 | Preparar fosfato 0,15 M de sodio disolviendo 40,2 g de fosfato de sodio, dibásico (Na 2 HPO 4 · 7H 2 O) en un volumen final de 1 L dH 2 O. Ajustar el pH a 7,0-7,5 con HCl. Autoclave a 121 ° C, 15 psi durante 15 min. Tienda solución de fosfato de sodio a temperatura ambiente durante un máximo de 12 meses. |
| 5% de BSA | Preparar disolviendo 5 g de BSA en 100 ml de dH 2 O. Esterilizar haciendo pasar la solución a través de un filtro de esterilización de 0,2 micras. |
| PBS, 0,2% de BSA | Preparar mediante la adición de 4 ml de 5% de BSA a 96 ml de PBS. Esterilizar haciendo pasar la solución a través de un filtro de esterilización de 0,2 micras. |
| Búfer TSM III | Disolver 1,21 g de base Trisma, 5,84 g de NaCl, 0,203 g de MgCl 2, 1 ml de gelatina Prionex y 3 ml Mikrozid III en 950 ml de agua de grado reactivo. Ajustar el pH a 7,0 y luego llevar el volumen final a 1 L. Esterilizar haciendo pasar la solución a través de un filtro de esterilización de 0,2 micras. |
| 0,525% de hipoclorito de sodio (NaClO) | Preparar una solución de NaClO 0,525% diluyendo lejía doméstica 1:10 en dH2O Guarde 0,525% soluciones NaClO durante un máximo de 1 semana a temperatura ambiente. |
| Tiosulfato de sodio 1-M (Na 2 S 2 O 3) pentahidratado | Preparar una solución 1 M disolviendo 248,2 g de Na 2 S 2 O 3 en 1 L de dH 2 O. Tienda tiosulfato de sodio para un máximo de 6 meses a temperatura ambiente. |
Tabla 12. Tabla de Medios.
Discusión
Estudios nacionales a gran escala de la contaminación viral de las fuentes de agua y beber requieren el uso de múltiples laboratorios analíticos. Bajo estas condiciones, se necesita un método estándar para asegurar que los datos generados por los múltiples laboratorios es comparable. Hay muchos métodos moleculares publicados para la detección de virus, pero muy pocos métodos moleculares normalizados. Método EPA 1615 es un método estandarizado diseñado específicamente para la detección de enterovirus y norovirus en matrices de agua por RT-qPCR. Métodos moleculares estandarizados están disponibles para la detección de virus en los alimentos (CEN / ISO TS 15.216-1 y CEN / ISO TS 15216-2; 07 de abril 2013) 16,17 y se han aplicado a la detección de virus de hepatitis A y el norovirus en primavera agua 16. Todos los métodos estándar deben incluir controles de calidad, los criterios para minimizar internacional y la variación intra-laboratorio y datos falsos positivos debido a la contaminación de laboratorio. Para reducir aún más datos falsos, EMétodo PA 1615 sigue la orientación de la EPA sobre los métodos moleculares, 18 que estipula la separación de trabajo durante el proceso y una forma de flujo de trabajo. Incluye una hepatitis G 1,19 de control y procedimientos para minimizar los resultados negativos falsos debido a inhibidores de la RT-qPCR 15 interna. Utiliza ensayos cuantitativos junto con volúmenes estandarizados, tanto de agua muestreado y analizado el agua de modo que todos los datos de campo se expresa en copias genómicas por litro del campo o beber agua muestreada. Aunque solo tubo eficiente (una etapa) ensayos de RT-PCR están disponibles comercialmente, el método utiliza intencionadamente ensayos separados. Esto tiene la desventaja de reducir al mínimo la cantidad de muestra que puede ensayarse en cada reacción, pero da una mayor flexibilidad en el uso de múltiples conjuntos de cebadores. Ensayos de RT-qPCR están limitados por y sólo tan bueno como los cebadores y sondas utilizados y probablemente ningún conjunto de cebadores detectarán todas las variantes del virus dentro de un grupo. El conjunto de cebadores enterovirus fue elegido porquese dirige a la región 5 'no codificante conservada, 20,21 detecta una amplia variedad de serotipos de enterovirus y virus detectados por ella están asociados con efectos en la salud por el consumo de aguas subterráneas sin tratar 1. Dos conjuntos de cebadores se utilizan para la detección de genogrupo I norovirus 22,23. El primero fue elegido debido a la fuerte correlación entre los efectos de salud en los niños pequeños y detecta virus 1. El segundo genogrupo I conjunto de cebadores y el conjunto de cebadores utilizado para genogrupo II norovirus fueron escogidos porque detectan la más amplia variedad de cepas 24,25.
A pesar de las ventajas principales de los procedimientos de RT-qPCR y qPCR para la detección de ARN viral en el agua, hay varias limitaciones. En primer lugar, ambas partículas de virus infecciosos y no infecciosos, incluidos los inactivado por los desinfectantes, se pueden amplificar por estos procedimientos. Los resultados de Borchardt sugieren que esto es un problema menor para las aguas subterráneas sin tratar facuíferos rom similares a los de las comunidades estudiadas que para las aguas superficiales desinfectados 1. Para los virus cultivables este problema se puede superar usando PCR en combinación con la cultura 26,27. El problema también ha sido abordado por algunos virus mediante el uso de agentes de reticulación de ácido nucleico 28-30. Este último enfoque es más eficaz para los virus inactivados por hipoclorito y no eficaces para aquellos inactivado por UV.
Una segunda limitación de estos procedimientos moleculares es que el volumen de la muestra concentrada que se puede ensayar típicamente es mucho menor que la utilizada para procedimientos de cultivo de 6,31. Este problema es a menudo manejado por cualquiera de sustitución de un procedimiento basado en el polietilenglicol para la concentración secundaria estándar por floculación orgánica, lo que permite que la muestra se resuspendió en un volumen más pequeño, o mediante la adición de una etapa de concentración de la muestra terciaria 6,32,33 . Método 1615 utiliza Centrifultrafiltración ugal para proporcionar concentración terciario. Ultrafiltración centrífuga elimina el agua y los componentes de menos de 30.000 Daltons resultantes tanto en concentración de cualquier virus en muestras de ensayo y una reducción en pequeñas inhibidores de peso molecular de ensayos moleculares. Este terciarios resultados etapa de concentración en un factor de concentración total de> 10 5 para cualquier virus que estaba presente en el agua están probando.
Una tercera limitación es la presencia de inhibidores de procedimientos moleculares en muestras ambientales. Aunque se han desarrollado numerosos enfoques para eliminar los inhibidores, ningún enfoque es eficaz para todas las matrices de agua y tipos de virus 6,34,35, haciendo que el uso de controles internos diseñados para estimar el nivel de inhibición esencial. El reactivo de la hepatitis G utilizado en este método satisface este requisito al proporcionar un nivel constante de ARN viral en todas las reacciones y un ensayo de RT-qPCR para la estimación de la inhibición. Cuando el mejor de tél inhibidor enfoques de eliminación no logran eliminar la inhibición, concentrados de muestra se pueden diluir, siempre y cuando las concentraciones de virus son más altas que las concentraciones de inhibidor 14,15.
El procedimiento descrito en este documento curva estándar tiene tanto ventajas como una limitación importante. Una ventaja es que el reactivo utilizado materiales de todos los componentes necesarios en un solo reactivo, lo que permite un único control que se utilizará para todos los ensayos. Este reactivo es especialmente una ventaja para los ensayos de norovirus. Partículas norovirus sólo se pueden obtener a partir de individuos infectados por lo que es muy difícil obtener partículas virales para su uso como estándares. Una ventaja más importante es que proporciona un estándar para todos los virus de ARN de ARN específicas en un reactivo, como teniendo un nivel de ARN es esencial para la cuantificación exacta de ARN 36. Sin embargo, su capacidad de cuantificar virus precisión está limitada por el hecho de que los efectos de matriz no se tienen en cuenta. Esto significa que copia genómicavalores de números no pueden considerarse absoluta y sólo deben considerarse en términos relativos. Se recomienda que un número suficiente de curva estándar alícuotas de valores de trabajo (paso 1.2) estar preparado para cubrir los estudios completos. Por ejemplo, cada 250 l alícuota proporciona suficiente reactivo para 6 placas de RT. Si se sabe que un estudio requerirá análisis de 500 muestras, se necesitaría un mínimo de 12 alícuotas (500 muestras / 7 muestras por placa RT / 6 placas RT por alícuota).
Método EPA 1615 es un método basado en el rendimiento. Muchos fabricantes hacen reactivos equivalentes a los especificados en este documento y estos reactivos pueden ser sustituidos, siempre y cuando se cumplan los criterios de rendimiento. La curva patrón, que también sirve como un control de RT-qPCR positivo, es de valor en solucionar problemas de rendimiento. El rendimiento puede disminuir debido a la degradación del ARN, la vida útil de reactivo, el fracaso de los congeladores, la calibración del instrumento, y el error técnico. Problemas de rendimiento deben ser sospechosoed si las curvas de calibración diferentes de la mostrada en la Figura 4 o si no cumplen con la especificación de rendimiento para las curvas estándar. El ensayo de RT-qPCR es bastante robusto; completo fracaso es probablemente debido al manejo inadecuado de ARN o error técnico (por ejemplo, un reactivo faltante). Gran se debe tener cuidado en el manejo de muestras de ARN entre la extracción y la etapa de RT para reducir la degradación del ARN de las ribonucleasas.
Recuperaciones poliovirus de las aguas subterráneas y de grado reactivo y recuperaciones de norovirus murino de las aguas subterráneas se reunieron el Método EPA 1615 los criterios de aceptación de rendimiento (Cuadro 11) y son similares a los reportados por otros 33,37,38. Recuperaciones norovirus murino de muestras LFB eran mucho más bajos que los de los virus de la polio y no habrían cumplido con los criterios de aceptación-poliovirus específico. Las razones de la recuperación norovirus murino menor de agua grado reactivo son desconocidos. Similar a los resultados del presente documento, Karim y colleagues reportaron una recuperación para el norovirus GI.1 del 4% del agua del grifo 39. Lee et al. 37 informó recuperaciones medias para norovirus murino y GII.4 norovirus humana de 18% y 26% de agua destilada utilizando filtros de disco, respectivamente. Usando condiciones similares a Lee y colegas, Kim y Ko observaron recuperaciones de 46% y 43% para estos virus, respectivamente 38. Gibbons et al. 40 obtuvieron alrededor de 100% de recuperación de GII.4 norovirus humano a partir de agua de mar, pero Kim y Ko 38 encontraron que la adición de sal al agua destilada a concentraciones similares o superiores a agua de mar reduce significativamente la recuperación del virus murino y resultó en una reducción de dos veces en la recuperación GII.4.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
The authors thank Dr. Eric Rhodes for preparing the clones used in the development of Standard curve reagent, Brian McMinn for assistance in sample processing, Larry Wymer for statistical analysis, Dr. Mark Borchardt, U.S. Department of Agriculture, Marshfield, WI, for supplying the Sabin poliovirus serotype 3 used in this study and Dr. H. W. Virgin, Washington University, St. Louis, MO, for murine norovirus. The authors also acknowledge Gretchen Sullivan for assistance in preparation of stock laboratory reagents, Dr. Mohammad Karim for propagation of murine norovirus stocks, and local private well owners and utilities for allowing us to collect water samples. Although this work was reviewed by EPA and approved for publication, it may not necessarily reflect official Agency policy. Mention of trade names or commercial products does not constitute endorsement or recommendation for use.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 ml tube chamber | Diversified Biotech | CHAM-3000 | |
| 1 °C cool brick | Diversified Biotech | BRIK-2501 | |
| 10x PCR Buffer II and 25-mM MgCl2 | Life Technologies | N8080130 | |
| -20 °C freezer | VWR | 97043-346 | Must be a manual defrost freezer |
| -70 °C or colder freezer | Thermo Scientific | MBF700LSAO-E | |
| 96-well chamber | Diversified Biotech | CHAM-1000 | |
| Absolute ethanol | Fisher Scientific | BP2818-100 | |
| Armored RNA EPA-1615 | Asuragen | Custom order | Used for quantifying the RT-qPCR assay |
| Armored RNA Hepatitis G virus | Asuragen | 42024 | |
| Autoclave | Steris | Amsco Lab Series | |
| Biosafety cabinet | NuAir Laboratory Equipment Supply | Labgard 437 ES | |
| Bovine serum albumin (BSA) | Affymetrix | 10856 | Crystalline grade or better |
| Buffer AVE | Qiagen | 1026956 | Carrier RNA dilution buffer |
| Buffer AVL | Qiagen | 19073 | Extraction buffer |
| Carrier RNA | Qiagen | Not applicable | Use carrier RNA supplied with Buffer AVL |
| Centrifuge bottles | Fisher Scientific | 05-562-23 or 05-562-26 | |
| Centrifuge rotors | Beckman Coulter | 339080, 336380 | |
| Cool safe box | Diversified Biotech | CSF-BOX | |
| Dithiothreitol (DTT) | Promega | P1171 | |
| LightCycler® 480 Probes Master kit | Roche Diagnostics | 4707494001 | |
| Microcentrifuge tubes | Fisher Scientific | 02-682-550 | Use ribonuclease- and deoxyribonuclease-free tubes with snap caps |
| Microcide III | Fitzgerald | 99R-103 | |
| Microseal 'A' film | Bio-Rad Laboratories | HSA5001 | Heat resistant |
| Microseal 'F' film | Bio-Rad Laboratories | MSA1001 | Freezer resistant |
| Mini-plate spinner | Labnet International | MPS1000 | |
| Multichannel pipette | Rainin | L8-20 | |
| Multi-tube chamber | Diversified Biotech | CHAM-5000 | |
| Optical reaction plate | Life Technologies | 4314320 | |
| PCR nucleotide mix | Promega | U1515 | |
| PCR plate | Bio-Rad Laboratories | HSS9601 | |
| PCR-grade water | Roche | 3315932001 | |
| Phosphate buffered saline (PBS) | U.S. Biological | D9820 | |
| Plate mixer | Scientific Industries | MicroPlate Genie | |
| Prionex gelatin | Sigma Aldrich | G0411 | |
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Includes Buffers AW1 (first wash buffer), AW2 (second wash buffer), AE (elution buffer); ethanol must be added to Buffers AW1 and AW2 before use; Do not use Buffer AL supplied with the kit |
| Quantitative PCR thermal cycler | Life Technologies | 4351405 | |
| Random primer | Promega | C1181 | |
| Reagent Reservoir | Fisher Scientific | 21-381-27E | |
| Refrigerated centrifuge | Beckman Coulter | 367501 | |
| RNase Inhibitor | Promega | N2515 or N2615 | |
| ROX reference dye | Life Technologies | 12223 | |
| SuperScript II or III Reverse Transcriptase | Life Technologies | 18064-022 or 18080044 | |
| Surgical gloves | Fisher Scientific | 19-058-800 | |
| Thermal cycler | Life Technologies | 4314879 | |
| Trisma base | Sigma Aldrich | T1503 | |
| Vivaspin 20 centrifugal concentrator units | Sartorius-Stedim | VS2022 |
Referencias
- Borchardt, M. A., Spencer, S. K., Kieke, B. A., Lambertini, E., Loge, F. J. Viruses in nondisinfected drinking water from municipal wells and community incidence of acute gastrointestinal illness. Environ. Health Perspect. 120 (9), 1272-1279 (2012).
- Ye, X. Y., et al. Real-time PCR detection of enteric viruses in source water and treated drinking water in Wuhan, China. Curr. Microbiol. 65 (3), 244-253 (2012).
- Williamson, W. M., et al. Enteric viruses in New Zealand drinking-water sources. Water Sci. Technol. 63 (8), 1744-1751 (2011).
- Lambertini, E., Borchardt, M. A., Kieke, B. A., Spencer, S. K., Loge, F. J. Risk of viral acute gastrointestinal illness from nondisinfected drinking water distribution systems. Environ. Sci. Technol. 46 (17), 9299-9307 (2012).
- Chigor, V. N., Okoh, A. I. Quantitative RT-PCR detection of hepatitis A virus, rotaviruses and enteroviruses in the Buffalo River and source water dams in the Eastern Cape Province of South Africa. Int. J. Environ. Res. Public Health. 9 (11), 4017-4032 (2012).
- Fout, G. S., Martinson, B. C., Moyer, M. W., Dahling, D. R. A multiplex reverse transcription-PCR method for detection of human enteric viruses in groundwater. Appl. Environ. Microbiol. 69 (6), 3158-3164 (2003).
- Hewitt, J., Bell, D., Simmons, G. C., Rivera-Aban, M., Wolf, S., Greening, G. E. Gastroenteritis outbreak caused by waterborne norovirus at a New Zealand ski resort. Appl. Environ. Microbiol. 73 (24), 7853-7857 (2007).
- Jack, S., Bell, D., Hewitt, J. Norovirus contamination of a drinking water supply at a hotel resort. New Zealand Med. J. 126 (1387), 98-107 (2013).
- Anderson, A. D., et al. A waterborne outbreak of Norwalk-like virus among snowmobilers-Wyoming, 2001. J. Infect. Dis. 187 (2), 303-306 (2003).
- Parshionikar, S. U., et al. Waterborne outbreak of gastroenteritis associated with a norovirus. Appl. Environ. Microbiol. 69 (9), 5263-5268 (2003).
- U.S. Environmental Protection Agency Office of Water. . 820-F-12-058: Recreational Water Quality Criteria. , 1-63 (2012).
- Wade, T. J., et al. Rapidly measured indicators of recreational water quality and swimming-associated illness at marine beaches: a prospective cohort study. Environ. Health. 9, 66 (2010).
- Fout, G. S., et al. . Method 1615: Measurement of enterovirus and norovirus occurrence in water by culture and RT-qPCR (EPA/600/R-10/181). , 1-91 (2012).
- Cashdollar, J. L., et al. Development and Evaluation of EPA Method 1615 for Detection of Enterovirus and Norovirus in Water. Appl. Environ. Microbiol. 79 (1), 215-223 (2013).
- Gibson, K. E., Schwab, K. J., Spencer, S. K., Borchardt, M. A. Measuring and mitigating inhibition during quantitative real time PCR analysis of viral nucleic acid extracts from large-volume environmental water samples. Water Res. 46 (13), 4281-4291 (2012).
- Fuentes, C., et al. Standardized multiplex one-step qRT-PCR for hepatitis A virus, norovirus GI and GII quantification in bivalve mollusks and water. Food Microbiol. 40, 55-63 (2014).
- Coudray, C., Merle, G., Martin-Latil, S., Guillier, L., Perelle, S. Comparison of two extraction methods for the detection of hepatitis A virus in lettuces using the murine norovirus as a process control. J. Virol. Methods. 193 (1), 96-102 (2013).
- Sen, K., et al. . EPA 815-B-04-001: Quality assurance/quality control guidance for laboratories performing PCR analyses on environmental samples. , 1-56 (2004).
- Schlueter, V., Schmolke, S., Stark, K., Hess, G., Ofenloch-Haehnle, B., Engel, A. M. Reverse transcription-PCR detection of hepatitis G virus. J. Clin. Microbiol. 34 (11), 2660-2664 (1996).
- De Leon, R., Shieh, C., Baric, R. S., Sobsey, M. D. Detection of enteroviruses and hepatitis A virus in environmental samples by gene probes and polymerase chain reaction. Proc. Water Qual. Technol. Conf. , 833-853 (1990).
- Monpoeho, S., et al. Quantification of enterovirus RNA in sludge samples using single tube real-time RT-PCR. BioTechniques. 29 (1), 88-93 (2000).
- Jothikumar, N., et al. Rapid and sensitive detection of noroviruses by using TaqMan-based one-step reverse transcription-PCR assays and application to naturally contaminated shellfish samples. Appl. Environ. Microbiol. 71 (4), 1870-1875 (2005).
- da Silva, A. K., et al. Evaluation of removal of noroviruses during wastewater treatment, using real-time reverse transcription-PCR: different behaviors of genogroups I and II. Appl. Environ. Microbiol. 73 (24), 7891-7897 (2007).
- Butot, S., et al. Evaluation of various real-time RT-PCR assays for the detection and quantitation of human norovirus. J. Virol. Methods. 167 (1), 90-94 (2010).
- Loisy, F., et al. Real-time RT-PCR for norovirus screening in shellfish. J. Virol. Methods. 123 (1), 1-7 (2005).
- Reynolds, K. A., Gerba, C. P., Pepper, I. L. Detection of infectious enteroviruses by an integrated cell culture-PCR procedure. Appl. Environ. Microbiol. 62 (4), 1424-1427 (1996).
- Ming, H. X., Zhu, L., Zhang, Y. Rapid quantification of infectious enterovirus from surface water in Bohai Bay, China using an integrated cell culture-qPCR. Mar. Pollut. Bull. 62 (10), 2047-2054 (2011).
- Parshionikar, S., Laseke, I., Fout, G. S. Use of propidium monoazide in reverse transcriptase PCR to distinguish between infectious and noninfectious enteric viruses in water samples. Appl. Environ. Microbiol. 76 (13), 4318-4326 (2010).
- Sanchez, G., Elizaquivel, P., Aznar, R. Discrimination of infectious hepatitis A viruses by propidium monoazide real-time RT-PCR. Food Environ. Virol. 4 (1), 21-25 (2012).
- Kim, S. Y., Ko, G. Using propidium monoazide to distinguish between viable and nonviable bacteria, MS2 and murine norovirus. Lett. Appl. Microbiol. 55 (3), 182-188 (2012).
- Fout, G. S., Schaefer, F. W., Messer, J. W., Dahling, D. R., Stetler, R. E. . EPA/600/R-95/178: ICR Microbial Laboratory Manual, I.1-ApD-23. , (1996).
- Abbaszadegan, M., Stewart, P., LeChevallier, M. A strategy for detection of viruses in groundwater by PCR. Appl. Environ. Microbiol. 65 (2), 444-449 (1999).
- Lambertini, E., et al. Concentration of enteroviruses, adenoviruses, and noroviruses from drinking water by use of glass wool filters. Appl. Environ. Microbiol. 74 (10), 2990-2996 (2008).
- Rodriguez, R. A., Thie, L., Gibbons, C. D., Sobsey, M. D. Reducing the effects of environmental inhibition in quantitative real-time PCR detection of adenovirus and norovirus in recreational seawaters. J. Virol. Methods. 181 (1), 43-50 (2012).
- Iker, B. C., Bright, K. R., Pepper, I. L., Gerba, C. P., Kitajima, M. Evaluation of commercial kits for the extraction and purification of viral nucleic acids from environmental and fecal samples. J. Virol. Methods. 191 (1), 24-30 (2013).
- Fey, A., et al. Establishment of a real-time PCR-based approach for accurate quantification of bacterial RNA targets in water, using Salmonella as a model organism. Appl. Environ. Microbiol. 70 (6), 3618-3623 (2004).
- Lee, H., et al. Evaluation of electropositive filtration for recovering norovirus in water. J. Water Health. 9 (1), 27-36 (2011).
- Kim, M., Ko, G. Quantitative characterization of the inhibitory effects of salt, humic acid, and heavy metals on the recovery of waterborne norovirus by electropositive filters. J. Water Health. 11 (4), 613-622 (2013).
- Karim, M. R., Rhodes, E. R., Brinkman, N., Wymer, L., Fout, G. S. New electropositive filter for concentrating enteroviruses and noroviruses from large volumes of water. Appl. Environ. Microbiol. 75 (8), 2393-2399 (2009).
- Gibbons, C. D., Rodriguez, R. A., Tallon, L., Sobsey, M. D. Evaluation of positively charged alumina nanofibre cartridge filters for the primary concentration of noroviruses, adenoviruses and male-specific coliphages from seawater. J. Appl. Microbiol. 109 (2), 635-641 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados