Method Article
엔테로 바이러스 및 문화와 RT-qPCR에 의해 물에서 노로 바이러스의 발생의 EPA 방법 1615 측정. 파트 III. RT-qPCR에 의한 바이러스 검출
요약
Here we present a procedure to quantify enterovirus and norovirus in environmental and drinking waters using reverse transcription-quantitative PCR. Mean virus recovery from groundwater with this standardized procedure from EPA Method 1615 was 20% for poliovirus and 30% for murine norovirus.
초록
EPA Method 1615 measures enteroviruses and noroviruses present in environmental and drinking waters. This method was developed with the goal of having a standardized method for use in multiple analytical laboratories during monitoring period 3 of the Unregulated Contaminant Monitoring Rule. Herein we present the protocol for extraction of viral ribonucleic acid (RNA) from water sample concentrates and for quantitatively measuring enterovirus and norovirus concentrations using reverse transcription-quantitative PCR (RT-qPCR). Virus concentrations for the molecular assay are calculated in terms of genomic copies of viral RNA per liter based upon a standard curve. The method uses a number of quality controls to increase data quality and to reduce interlaboratory and intralaboratory variation. The method has been evaluated by examining virus recovery from ground and reagent grade waters seeded with poliovirus type 3 and murine norovirus as a surrogate for human noroviruses. Mean poliovirus recoveries were 20% in groundwaters and 44% in reagent grade water. Mean murine norovirus recoveries with the RT-qPCR assay were 30% in groundwaters and 4% in reagent grade water.
서문
정량 PCR (qPCR에,이 논문에서 사용하는 용어의 정의에 대한 보충 자료 참조) 전사-qPCR에 (RT-qPCR에) 물을 검출하는 환경에서 인간의 장내 바이러스를 정량화 및 음주에 대한 가치있는 도구이며, 특히 이렇게 많은 바이러스에 대한 역 복제 또는 세포 배양 시스템에서 복제하지 저조한. 두 도구는 많은 바이러스 종류는 세계 1-6에 걸쳐 환경 마시는 물에 존재하는 것을 증명하고있다. 그들이 마시는 물에서 발견 된 바이러스가 발병 환자 7-10에 의한 창고 동일 것으로 나타났습니다로 질병 발생을 조사하는 동안 증폭 된 유전자 단편의 염기 서열과 결합 된 이들의 사용은 수 인성 바이러스 전송에 대한 증거를 제공하고 있습니다.
qPCR에와 RT-qPCR에 모두 유용 공중 보건의 도구입니다. 예를 들어, 미국 환경 보호국 (EPA)에 의해 수행 연구의 데이터는 강한 관계가 될 수 있었다qPCR에 레크리에이션 바다에서 건강에 미치는 영향에 의해 트윈 지표 측정. 그 결과, EPA의 최종 2,012 레크리에이션 수질 기준 오락 해변 (11, 12)를 모니터링하기위한 qPCR의 방법을 포함한다. RT-qPCR에 1로 측정 Borchardt와 동료들은 또한 지하수에서 처리되지 않은 지하수 및 바이러스를 사용하여 지역 사회에서 급성 위장염 사이에 강한 관계를 발견했다.
이 논문의 목적은 EPA 방법 1615 13, 14의 분자 분석 구성 요소를 설명하는 것입니다. 이 분석은 전기 양성 필터를 통해 장내 바이러스와 전달 된 환경이나 식수의 원래 볼륨에 따라 리터당 노로 바이러스 게놈 사본 (GC)의 정량적 추정치를 제공하기 위해 RT-qPCR에 사용합니다. 분자 절차의 개요는 표준 곡선을 준비 1. 프로토콜 부분을 그림 1 세부 절차를 나타낸다. 이 기준은 RN이 포함 된 시약에서 준비모든 프라이머 / 프로브 세트의 표적 서열의 카피. 제 2 차 농도 절차를 설명합니다. 섹션 3은 농축 및 제어 샘플로부터 RNA를 추출하기위한 절차를 제공한다. 각각의 시료에서 RNA를 역 세중 분석 및 주요 전사 (4 절)에 임의의 프라이머를 사용하여 전사한다. (도 2 제 5 항) 각 역전사 반응에서 cDNA를 qPCR에 의해 중으로 분석 다섯 별도의 바이러스 별 분석으로 분할된다. 분석은 여러 엔테로 바이러스 및 noroviruses 및 RT-qPCR에 15 저해되어 시료를 식별 간염 G RNA를 함유하는 시약을 검출하도록 설계 과학 문헌 (표 1)에서의 프라이머 및 프로브를 사용한다.
프로토콜
주 : 사용 데이터 시트가 프로토콜의 모든 단계를 추적하는 단계; 예를 들어, 데이터 시트는 보충 자료 테이블 S2-S4에 제시되어있다.
1. 표준 곡선 준비
- (예를 들어, 장갑 RNA EPA-1615) 2.5 × 108 입자 / ㎖ (2.5 × 10 8 GC / ㎖) TSM의 사용 농도로 제조자에 의해 공급되는 농도로 희석하여 표준 곡선 시약 작업 스톡을 제조 III 버퍼입니다. -20 ℃에서 1.5 ml의의 microcentrifuge 튜브 및 저장소를 사용하여 250 μL 분취 액으로 작업 주식을 나눈다.
참고 : 다른 표준 곡선 시약으로 사용하기위한 준비 작업 바이러스의 주식과 플라스미드에 대한 지침은 보충 자료 프로토콜 단계 (S1)를 참조하십시오. - 작업 스톡 분취 량 중 하나 이상을 해동. 2.5 × 10 (7)의 농도를 제공, 2.5 × 10 (6), 2.5 × 1.5 ml의 마이크로 원심 튜브를 이용하여 5 ~ 10 배 연속 희석을 준비105, 2.5 × 104, 2.5 × 103 GC / ㎖.
- TSM III 버퍼 225 μL에 2.5 × 10 8 GC / ㎖ 작업 주식의 25 μl를 추가하여 최초의 희석을 준비합니다. 볼텍스 혼합기를 사용하여 5 ~ 15 초 동안 혼합한다.
- TSM III 완충액 225 μL 단계 1.2.1에서 제조 한 희석액 25 μl를 첨가하여 희석 다음을 준비한다. 다시 혼합하고, 다음 세 10 배 희석액을 조제 유사한 과정을 계속한다.
- 표준 곡선 작업 재고에서 RNA와 바로 제 3의 절차를 사용하여 다섯 희석의 압축을 풉니 다.
2. 고등 농도
- 상부 샘플 챔버로 1X PBS 중 적어도 10 ㎖, 0.2 % 소 혈청 알부민 (BSA)을 첨가하여 각각의 샘플 채취 용 원심 농축기 (30,000 분자량 컷)을 준비한다. 이 솔루션은 얇은 채널 농도 챔버를 채운 확인하고 4 ℃에서 O / N을 누릅니다.
- 유체를 폐기하십시오. 과량의 BSA를 제거하고 물을 폐기 멸균 시약 등급 이상의 물 10 mL로 농축 한 번 씻어.
- S, 원심 분리 농축기로 상기 분석 샘플 량과 동일한 각 시험 샘플로부터 이차 물 농축액의 양을 추가한다.
- 식 (1) 사용 (S)를 계산,
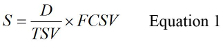
D는있는 분석 원래 물 샘플의 볼륨 인 경우, TSV는 전체 샘플 볼륨과 FCSV는 최종 농축 샘플 볼륨입니다. S.의 계산의 예를 들면 기업 재료 S2보기
- 식 (1) 사용 (S)를 계산,
- 원심 분리기 (20)에 대한 스윙 버킷 회에서 4 ° C에서 3,000~6,000 XG에 각각의 테스트 샘플 - 30 분. 얇은 채널 농도 챔버에서 볼륨을 확인합니다.
- 볼륨이 20 분 이상 다시보다 400 μL, 원심 분리기 인 경우. 일의 샘플까지 원심 계속전자 얇은 채널 농도 실 미만 400 μL로 감소되었다. 상층 액을 제거하지 마십시오.
- 바이러스의 회수를 증가시키기 위해 멸균 0.15 M 인산 나트륨 1 ㎖, pH를 7-7.5로 원심 농축기의 측면을 세척한다. 원심 분리기는 다시 3,000~6,000 XG, 4 ° C에서 샘플 미만 400 μL로 감소 될 때까지. 이 세척 단계를 하나의 추가 시간을 반복합니다.
- , 100-200 μL의 마이크로 피펫을 사용하여 조심스럽게 측정하여 1.5 ml의 마이크로 원심 튜브 (즉, 마이크로 원심 튜브에 200 μL를 전송하고 나머지 유체가 완전히 인출 될 때까지 다음, 마이크로 피펫을 조정함으로써, 농축 잔액을 측정하기 위해 각각의 농축 시료를 전송할 ) 피펫 팁으로. 400 ± 2 μL에 최종 볼륨을 조정, 0.15 M 인산 나트륨, pH를 7-7.5을 추가합니다.
- 즉시 3. processe 수없는 농축 샘플을 잡고 단계로 진행하여 핵산을 추출d를 즉시 이하 24 시간 동안 4 ° C에서.
3. 핵산 분리
- 1.5 ml의 마이크로 원심 튜브를 라벨 분리하는 단계 1.3 단계 2.5 표준 곡선 희석에서 각각의 시료에서 단계에 차 농축액 3.3 200 μl를 기술 된 바와 같이 제조 된 추출 버퍼 200 μl를 추가합니다. 또는 -70 ℃ 이하 차 농축 나머지 동결. 다음을 제외 혈액 샘플 스핀 프로토콜 핵산 추출 키트 제조자의 지시에 따라 각 시료로부터 핵산을 추출한다.
- 물 샘플에 단백질 분해 효소를 추가하거나 핵산 추출 키트와 함께 제공되는 추출 버퍼를 사용하지 마십시오.
- 캐리어 RNA로 추출 버퍼를 준비
- 캐리어 RNA의 310 μg의 들어있는 유리 병에 캐리어 RNA 희석 버퍼의 310 μl를 추가합니다. 용해 후 50 μl를 포함하는 6 분취 량으로 나누어 섞는다. -20에서 보관기음.
- 0.027 μg의 / μL의 캐리어 RNA 농도를 얻기 위해 추출 완충액 1 ㎖ 당 해동 캐리어 RNA의 28 μL를 추가한다. 추출 키트와 함께 제공하는 대신에, 캐리어 - 개정 RNA 추출 완충액을 사용한다.
- 리보 뉴 클레아 제를 첨가하여 용출 완충제 마스터 용액을 조제 (의 RNase) 억제제 추출 키트와 함께 공급 된 용출 버퍼 400 단위 / ml의 최종 농도.
- 컬럼으로의 RNase 억제제 용출 완충액 50 μl를 배치하여 스핀 컬럼 바인딩에서 용출 된 핵산 RNA. 1 분 동안 기다린 후 실온에서 1 분 동안 6,000 XG에 원심 분리기.
- 단계를 반복 3.4.1 다음 제거하고 열을 버린다.
- 단계 3.4.2에서 RNA 추출물의 분취 량을 준비합니다. (6) 표준 곡선 작업 주식의 분취 적어도 15 μL를 각각 포함하는 각 표준 곡선의 희석을 준비합니다. 적어도 포함하는 모든 다른 RNA 샘플 4 분취 준비22 μL 각. 이들은 4 시간 이내에 역전사에 의해 처리 될 수있는 경우에 4 ° C에서 각각의 시료와 표준 곡선 분취 컨트롤 중 하나를 저장; 그렇지 않으면, 또는 -70 ℃ 이하 모든 분취를 저장합니다.
4. 역전사 (RT)
- 열 번 같음 (마이크로 리터 단위) 존재 올리고 뉴클레오티드 nanomoles (nmol의) 개수를 양을 사용하여 각 바이알 PCR 등급 물의 부피를 첨가하여 표 1에 나열된 각 올리고 뉴클레오티드 프라이머 및 프로브의 100 μM 스톡 용액을 제조 유리 병 라벨에 또는 예를 들어 제조업체의 사양서 (에서와 같이 바이알 (,) 363 μL에서 36.3 nmol의 포함하는 프라이머를 재현 탁. 용해 섞는다.
- 솔루션을 작업 10 μm의를 준비하는 PCR 등급 물 100 μm의 해결책 1:10 희석.
- 2. 피펫을 표 가이드를 사용하여 클린 룸에서 RT Maste의 16.5 μl를 RT 마스터 믹스 1과 2를 준비각 PCR 플레이트 R 믹스 1은 잘 멀티 채널 피펫을 사용하여.
- 해동, 냉동 경우, 각 필드의 샘플 및 실험실 강화 샘플 매트릭스에서 핵산 추출물 (LFSM, 즉, 시드 물 매트릭스 샘플).
- RNA 분해 효소 억제제의 400 단위 / ㎖를 함유하는 용출 완충액으로 5 1시 25분 : 각 필드 LFSM 샘플 1 희석.
- 실험실 시약 블랭크 (LRB; 즉, 시약 등급의 물을 사용하여 음의 품질 관리), 성능 평가 (PE, 해동, 냉동되었지만 랩 요새 빈 (즉, 시딩 된 시약 등급의 물을 사용하여 긍정적 인 품질 관리 LFB)를 희석하지 ; 즉, 시드 시약 등급의 물 샘플은 연구 시작에 앞서 실험 성능), 성능 시험 (PT를 평가하는 데, 즉, 연구 기간 동안 실험실 성능을 평가하는 데 사용되는 분석이 알려지지 역가 시드 시약 등급의 물 샘플 ), NA 배치 부정적인 추출 제어, 또는 표준 곡선 세트에서 추출 된 RNA.
- 별도의 PCR 판 우물에 모든 테스트 샘플, 제어 및 표준 곡선에서 RNA의 6.7 μl를 놓고 테스트 샘플과 컨트롤에 대한 세중의 우물을 사용하여 표준 곡선 (RT 플레이트, 예를 들어 그림 S1 참조) 우물을 복제.
- 아니 템플릿 컨트롤 (NTC)에 대해 별도의 PCR 판 우물에 용출 버퍼의 6.7 μl를 놓습니다. 첫 번째 샘플에 대한이 모든 네 번째 추가 샘플에 대한 다음 두 가지 이상을 사용하여 RT 접시 당 2-8 NTC를 포함합니다.
- 판 전체에 부정적인 추출 및 NTC 컨트롤을 배포합니다.
- 내열 접시 실러와 PCR 플레이트를 밀봉합니다. 5 ~ 10 초 동안 샘플을 혼합 한 후 ≥ 500 XG 간단히 원심 분리기.
- 99 ° C에서 4 분의 판을 품어 후 열 자전거 타는 4 ℃로 급속 냉각. 원심 분리기 다시 ≥ 500 XG 간단히에서.
- 조심스럽게 플레이트 씰을 제거하고 각 웰에 RT 마스터 믹스 2의 16.8 μl를 추가합니다. 괜찮다알 혼합하여 내열성 플레이트 실러 다시 판 및 ≥ 500 X g에서 간단한 원심 분리.
- 4 ° C 유지주기에 의해 다음 열 자전거 타는 사람에 접시를 놓고 99 ° C에서 42 ° C에서 60 분 다음 25 ℃에서 15 분, 5 분을 실행합니다.
- 프로세스를 즉시 또는 qPCR에 (5 단계), 또는 저장 샘플 8 시간 내에서 이하는 -70 ° C 그들은 처리 할 수있을 때까지. 4 ° C에서 8 시간 내에 처리 할 수있는 저장 샘플.
5. 실시간 정량 PCR (qPCR에)
- 이전의 모든 테스트 샘플을 실행하기 간염 G 시약의 각 로트의 평균 간염 G 된 CQ 값을 결정합니다.
- NTC 제어한다 (단계 4.1.1)에서 기술 한 바와 같이 제조하여 10 회 반복을 RT 분석을 실행. (5.5.3 5.2 단계) 아래에 설명 된대로 간염 G qPCR에 분석을 실행합니다. 10 복제의 평균 된 CQ 값을 계산합니다.
- RT 마스터 믹스 1 간염 G 시약의 양 (표 2를 조정), 필요한 경우, 32 대 (25)와 사이의 평균 된 CQ 값을 얻었다. 물의 양을 변화시킴으로써 승강 량을 보상하여 분석 당 16.5 μL에서 RT 마스터 믹스 한 최종 용적을 유지했다.
- 조정 된 수량을 반영하기 위해 5.1.2 및 변경 테이블 2 단계 5.1.1를 반복하여 모든 조정을 확인합니다.
- 장내 표 3에서 가이드를 사용하여 클린 룸에서 qPCR에 마스터 믹스를 준비, 간염 표 4 쥐 노로 바이러스 (노로 바이러스의 유전자형의 V)에 대한 노로 바이러스의 유전자형 II, 표 7에 대한 노로 바이러스 유전자형 I, 표 6 표 5, 표 8 G. 믹스는 각 마스터 믹스 한 다음 ≥ 500 XG 간단히 원심 분리기.
- 각 qPCR에 분석을 위해 잘 분리 판 당 14 μl를 사용하여 표시 광 반응 판의 해당 우물에 PCR 마스터 믹스를 추가 (그림 S2 그림 (S1)에서 RT 레이아웃에 따라 qPCR에 분석)에 대한 가능한 레이아웃.
- 냉동 경우, 실온에서 단계 4.8에서 RT 판을 녹여. 접시 믹서를 사용하여 혼합 한 후 ≥ 500 XG 간단히 원심 분리기.
- 광 반응 판의 적당한 웰에 해당하는 cDNA 6 μL 분주. ≥ 500 XG 잠깐의 광 반응 플레이트와 원심 분리기에서 샘플을 섞는다.
- 다른 모든 qPCR에 분석을 실행하기 전에 원액 희석 필드와 LFSM 샘플에 간염 G qPCR에 분석을 실행합니다. 필드 또는이다 LFSM 샘플 <장내 바이러스와 노로 바이러스 qPCR에 분석의 의미 간염 G 된 CQ 값보다 큰 1 된 CQ 값의 가장 낮은 희석을 사용합니다.
- 제조업체의 지시에 따라 정량적 PCR 열 순환기 소프트웨어를 설정한다. 표준으로 표준 곡선 샘플을 확인하고 각각의 표준 곡선의 희석, 표 9의 게놈 복사 값을 입력합니다. 95 ℃, 60 ℃에서 1 분 15 초로 45 사이클,이어서 95 ° C에서 10 분 동안 정량적 PCR 열 순환기 판을 실행.
- 각각의 표준 곡선은 표 10에 주어진 허용 값을 충족하는지 여부를 결정합니다. 예제 보충 자료 섹션 (S3)를 참조하십시오.
- 수학 식 2를 사용하여 표준 곡선에 대한 전체 표준 편차 (STDDEV)을 계산,
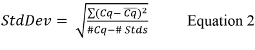
Cq와 각 표준 곡선 복제에 대해보고 된 값이고, Cq와는 복제의 각 세트에 대한 평균 값이 #Cq는 총 양의 값 (즉, 미결정 생략) 모두 표준 제어 복제 용 된 CQ 값의 개수, 및 #입니다 성병은 양의 값을 가지고 표준 컨트롤 수있다. - 정량적 PCR 열 순환기 소프트웨어는 각각의 표준 곡선에 대한 기울기를 산출하지 않는 경우, 수학 식 3을 이용하여 기울기 계산(60);
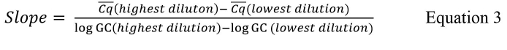
Cq와 사용 된 최고 및 최저 희석 평균값이며 로그 여기서 GC는 표 9에서 사용 된 최고 및 최저 희석을위한 게놈 사본의 로그 값이다. - 식 4를 사용하여 R 2 값을 계산합니다.
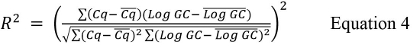
Cq와 모든 된 CQ 값의 평균이며 로그 GC 각 복제에 대한 평균 GC 로그 값이다. - 식 (5)를 사용하여 %의 효율을 계산한다 :
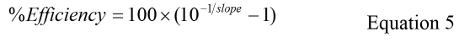
- 수학 식 2를 사용하여 표준 곡선에 대한 전체 표준 편차 (STDDEV)을 계산,
- 표 10에 명시된 기준 및 각 샘플에 대한 평균 값을 GC 충족 표준 곡선에 기초하여 모든 테스트 샘플에 대한 열 순환기 소프트웨어 GC에 의해 산출 된 값을 기록한다. 의 기준에 맞지 않는 표준 곡선과 어떤 샘플을 다시 실행 표 10 또는 부정적인 컨트롤 (LRB, NA 배치 부정적인 추출 제어, 또는 NTC)는 긍정적이다. 다시 실행 중 기준을 충족하지 못하는 거짓 양성 대조군을 가지고있는 샘플을 다시 처리합니다.
- 수학 식 (6) 이용하여 각 시료에 대한 GC 리터당 (GC에 L)을 결정 :
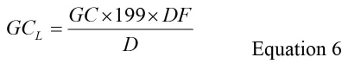
GC는 단계 570에서 평균 게놈 카피 수이고, 요소 '199'급 농도 중에 발생하는 부피 감소를위한 총 희석 인자, RNA를 추출하고 RT-qPCR의 단계, DF는 억제 보상 희석 인자 , D는 리터에있는 분석 원래 물 샘플의 볼륨입니다. GC (L)의 계산의 예에 대한 보충 자료 부 S4 참조. - 단계 (199)에 의해 5.5에서 GC 평균 값을 0.3로 승산 나누어 총 LFB의 GC 및 LRB 샘플을 계산한다.
결과
전체 바이러스 복구 쌍이 필드 LFSM 지하수 샘플을 사용하여 측정 하였다. 일곱 샘플 세트의 총 세 공용 처리장 및 개인 웰로부터 수집 한 샘플 세트로부터 분리 된 경우에 수집 된 두 세트를 사용하여 분석 하였다. LFSM 샘플 종자 수준 × 106 뮤린 노로 바이러스의 MPN 세이빈의 폴리오 바이러스 혈청 형 3, 5 × 106 PFU 3이었다. 뮤린 노로 인해 LFSM 샘플 충분한 바이러스 농도 인간 노로 주식 부족 평가 방법에 대용으로 사용 하였다. 지하수 시료에 대한 평균 폴리오 바이러스 복구는 2 %의 표준 오차, 14 동안은 쥐 노로 바이러스 복구가 3 % (그림 3)의 표준 오차, 30 %였다 평균 20 %였다. 각 LFSM에 대한 일반 필드 지하수 샘플을 검출 가능한 장 바이러스 나 노로 바이러스 없었다.
LFB과 LRB 샘플 시드와 시드 화 시약 급 와트를 사용하여 측정 하였다어. 모든 샘플 LRB (데이타 미기재) 음성이었다. 쥐 노로 바이러스 복구가 0.5 %의 표준 오차 4 %를 평균하는 동안 소아마비 바이러스 복구, 1 % (그림 3)의 표준 오차 44 %를 평균.
RT-qPCR에 도표 4. 적절한 표준 곡선 시약의 사용을 필요로 장내 바이러스와 노로 바이러스 GII의 일반적인 표준 곡선을 보여줍니다. 노로 바이러스의 GII 곡선은 0.9987의 R 2 값, 0.14의 전체 표준 편차, 101 %의 효율로 표준 곡선의 성능 기준 (표 10)을 충족합니다. 노로 바이러스 GIA와 GIB 곡선 (도시하지 않음) 우둔함 바이러스 GII의 그것과 거의 동일하다. 엔테로 곡선은 0.9874의 R 2 값, 0.58의 전체 표준 편차, 103 %의 효율에있어서의 성능 기준을 충족하지만, 배 이하 백에 대한 민감도 때문에 노로 곡선보다 높은 검출 한계를 갖는다.
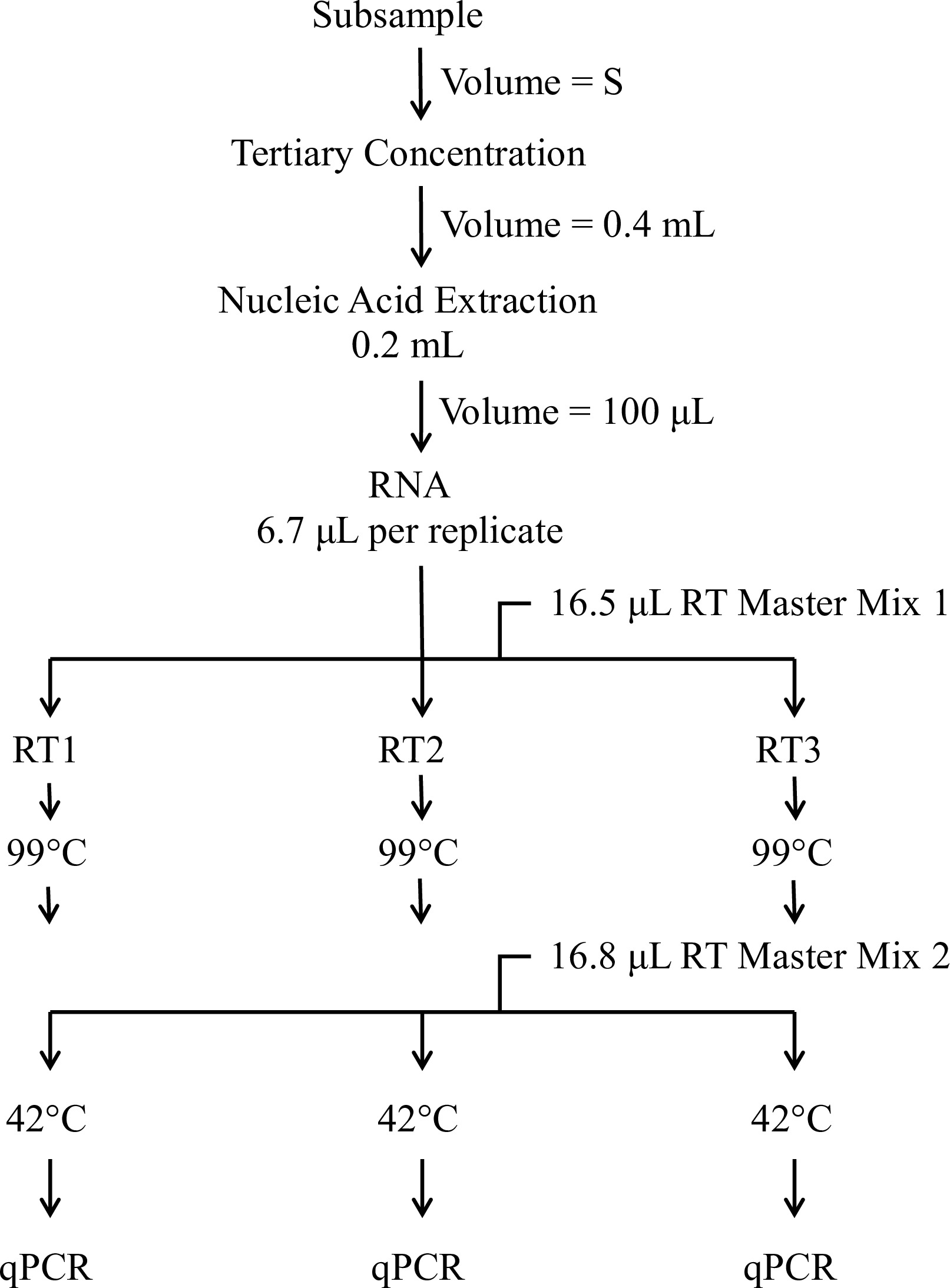
도 분자 절차 1. 개관. 분자량 절차는 감염성 바이러스, 핵산의 추출을 측정하기 위해 수행 된 이후 추가로 시료 농도를 포함하는, 두 단계는 전사 (RT) 프로토콜, 및 정량적 PCR (qPCR에)를 역방향. 출발 체적 (S)은 원래 물 시료에있어서 정의 비율을 나타낸다.
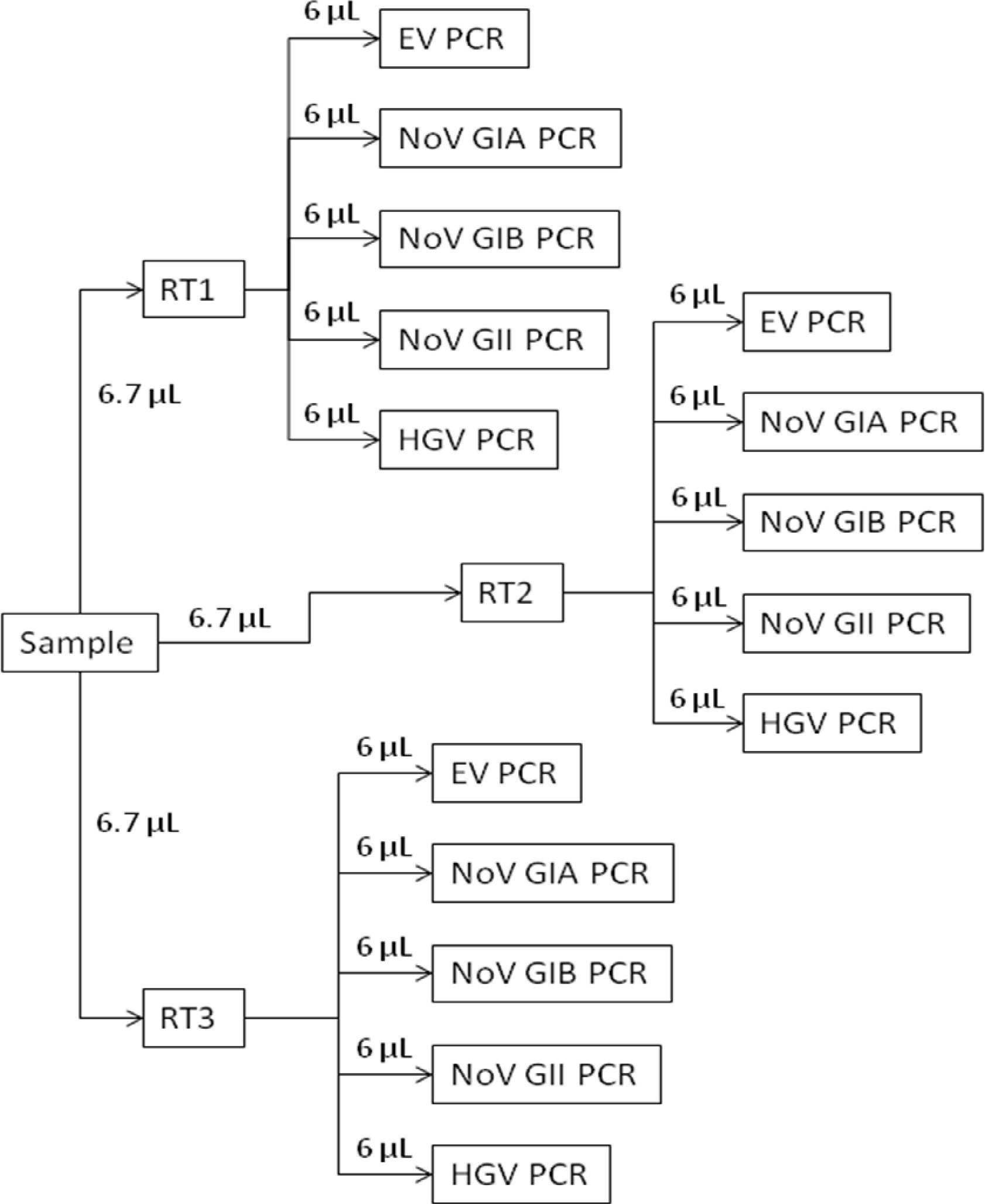
도 2. RT-qPCR의 개요 개략적. 추출한 각 시험품 RNA가 역 중으로 분석 (RT1, RT2 및 RT3)를 이용하여 전사된다. 다음 별도의 엔테로 바이러스 (EV PCR)을 이용하여 특정 바이러스에 대한 분석 세중 RT 분석, 노로 바이러스의 유전자형 I (NOV GIA PCR 및 NOV GIB PCR), 노로 바이러스의 유전자형 II에서 각각의 cDNA (NOV GII PCR), 간염 G (HGV PCR) 분석.
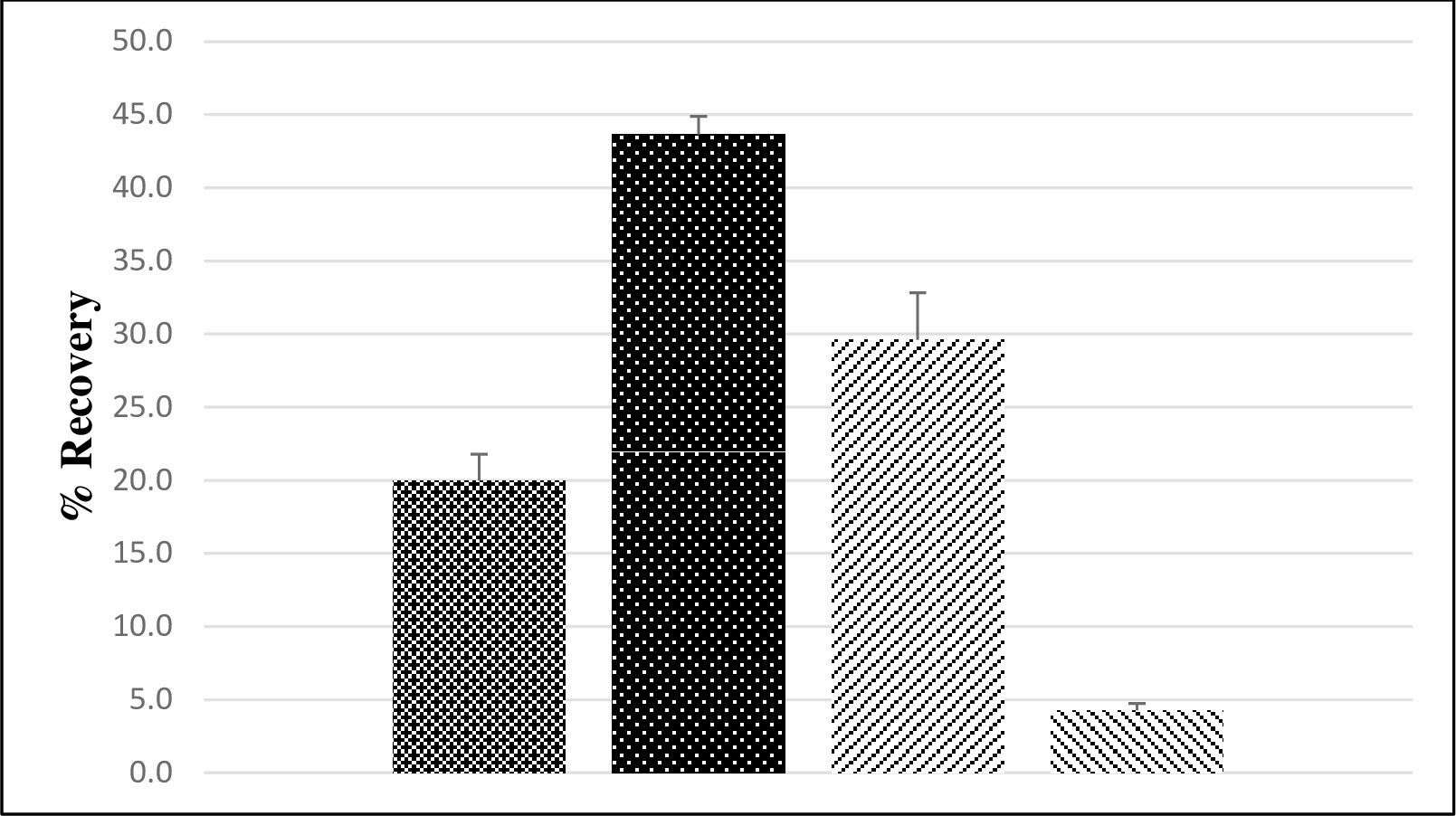
그림 3. 평균 폴리오 바이러스와 지상에서 쥐 노로 바이러스 복구 (%) 및 시약 급 물. 평균 회수율은 지상에서 폴리오 바이러스 (에 대해 표시됩니다  ; N = 7) 및 시약 등급에서 (
; N = 7) 및 시약 등급에서 (  ; N = 12) 물과 지상에서 쥐 노로 바이러스에 대한 (
; N = 12) 물과 지상에서 쥐 노로 바이러스에 대한 (  ; N = 7) 및 시약 등급에서 (
; N = 7) 및 시약 등급에서 (  ; N = 12), 물 (1), 여기서 "n"은 별도의 처리 물 샘플의 수이다. 오차 막대는 표준 오류를 나타냅니다.
; N = 12), 물 (1), 여기서 "n"은 별도의 처리 물 샘플의 수이다. 오차 막대는 표준 오류를 나타냅니다.

그림 4. 엔테로 바이러스 및 노로 바이러스 GII 표준 곡선. 엔테로 바이러스에 대한 일반적인 표준 곡선과 노로 바이러스 GII이 표시됩니다. 기울기 및 R 각 곡선의 2 값을주는 공식은 열 자전거 타는 사람에 의해 계산된다.
보충 파일 1 이 파일을 다운로드하려면 여기를 클릭하십시오.
| 바이러스 그룹 | 프라이머 / 프로브 이름 (1) | 순서 (2) | 참고 | |
| 엔테로 바이러스 | ||||
| EntF (EV-L) | CCTCCGGCCCCTGAATG | (20) | ||
| ENTR (EV-R) | ACCGGATGGCCAATCCAA | (20) | ||
| EntP (EV-프로브) | 6FAM-CGGAACCGACTACTTTGGGTGTCCGT-TAMRA | (21) | ||
| 노로 바이러스 GIA | ||||
| NorGIAF (JJV1F) | GCCATGTTCCGITGGATG | (22) | ||
| NorGIAR (JJV1R) | TCCTTAGACGCCATCATCAT | (22) | ||
| NorGIAP (JJV1P) | 6FAM-TGTGGACAGGAGATCGCAATCTC-TAMRA | (22) | ||
| 노로 바이러스 GIB | ||||
| NorGIBF (QNIF4) | CGCTGGATGCGNTTCCAT | (23) | ||
| NorGIBR (NV1LCR) | CCTTAGACGCCATCATCATTTAC | (23) | ||
| NorGIBP (NV1LCpr) | 6FAM-TGGACAGGAGAYCGCRATCT-TAMRA | (23) | ||
| 노로 바이러스 GII | ||||
| NorGIIF (QNIF2d) | ATGTTCAGRTGGATGAGRTTCTCWGA | (25) | ||
| NorGIIR (COG2R) | TCGACGCCATCTTCATTCACA | (25) | ||
| NorGIIP (QNIFS) | 6FAM-AGCACGTGGGAGGGCGATCG-TAMRA | (25) | ||
| 노로 바이러스 GV | ||||
| MuNoVF1 | AGATCAGCTTAAGCCCTATTCAGAAC | (14) | ||
| MuNoVR1 | CAAGCTCTCACAAGCCTTCTTAAA | (14) | ||
| MuNoVP1 | VIC-TGGCCAGGGCTTCTGT-MGB | (14) | ||
| 간염 G | ||||
| HepF (5'-NCR 정방향 프라이머) | CGGCCAAAAGGTGGTGGATG | (19) | ||
| HEPR (5'-NCR 역방향 프라이머) | CGACGAGCCTGACGTCGGG | (19) | ||
| HEPP (간염 G의 TaqMan 프로브 | 6FAM-AGGTCCCTCTGGCGCTTGTGGCGAG-TAMRA | 1 | ||
표 1. 프라이머 및 RT-qPCR에 의한 바이러스 탐지 용의 TaqMan 프로브.
(1) 방법 1,615 프라이머 및 프로브 이름은 순방향, 역방향, 및 프로브 F, R, P 또는 연접 된 바이러스 이름의 처음 세 문자이다. 노로 바이러스의 유전자형은 이름에 GI과 GII을 추가하여 지정됩니다. 또한 기본 참조에서 A와 B 프라이머 및 프로브 이름을 사용하여 구별되는 두 개의 노로 바이러스 위장관 프라이머 세트는 부모에 제시되어있다heses.
(2) 프라이머 및 프로브 서열의 방향은 '3'5입니다. 다음 퇴화 기본 지표가 사용된다 : 네 개의 뉴클레오티드의 N-혼합물 R-A + G; Y-T + C; W-+ T; 그리고 I-이노신.
| 성분 | 반응 당 볼륨 (μL) (2) | 최종 농도 | 마스터 믹스 (μL) 당 볼륨 (3) |
| RT 마스터 믹스 1 | |||
| 랜덤 프라이머 | 0.8 | 10 NG / μL (C. 5.6 μM) | (84) |
| 간염 G 기갑 RNA (4) | 1 | (105) | |
| PCR 등급 물 | 14.7 | 1543.5 | |
| 에탈 | 16.5 | 1732.5 | |
| RT 마스터 믹스 2 | |||
| 10 배 PCR 버퍼 II | 4 | 10 mM의 트리스, 산도 8.3, 50 mM의 KCl을 | (420) |
| 25-MM의 MgCl 2 | 4.8 | 3 밀리미터 | (504) |
| 10-MM의 dNTPs | 3.2 | 0.8 밀리미터 | (336) |
| 100-MM DTT | 4 | 10 mM의 | (420) |
| 의 RNase 억제제 | 0.5 | 0.5 단위 / μL | 52.5 |
| 첨자 II RT | 0.3 | 1.6 단위 / μL | 31.5 |
| 합계 | 16.8 | 1764 | |
표 2. RT 마스터 믹스 1 및 2 (1).
(1) (CL)에 RT 마스터 믹스를 준비합니다즉 EAN 룸, 분자 및 미생물 학적 절차가 수행되지 않는 방.
(2) RT 최종 분석 부피는 40 μL이다.
(3) 볼륨 (105)에 기초하여 분석된다 나타낸다. 여분의 분석은 손실을 고려하여 추가로이 96 웰 PCR 플레이트에 충분하다. 양 분석 될 샘플 및 컨트롤의 개수에 따라 스케일 업 또는 다운 할 수도있다.
(4) 보충 자료 스텝 S4에 기재된 바와 같이 RT 마스터 믹스 1에 포함 간염 G 시약의 양을 결정한다.
| 성분 | 반응 당 볼륨 (μL) (2) | 최종 농도 | 마스터 믹스 (μL) 당 볼륨 (3) |
| 배의 LightCycler 480 프로브 마스터 믹스 | (10) | 소유권 | 1050 |
| ROX 참조염료 (4) | 0.4 | 0.5 밀리미터 | (42) |
| PCR 등급 물 | 1 | (105) | |
| 10 μM의 EntF | 0.6 | 300 nm의 | (63) |
| 10 μm의 ENTR | 1.8 | 900 nm의 | 189 |
| 10 μm의 EntP | 0.2 | 100 nm의 | (21) |
| 합계 | (14) | 1470 |
표 3. PCR 마스터 믹스 엔테로 바이러스 (EV) 분석 (1).
(1) 클린 룸의 모든 PCR 마스터 믹스를 준비합니다.
(2) 최종의 qPCR 분석 부피는 20 μL이다.
(3) 볼륨 (105)에 기초하여 분석된다 나타낸다. 여분의 분석은 손실을 고려하여 추가로이 96 웰 PCR 플레이트에 충분하다. 양 샘플 것 컨트롤의 개수에 따라 스케일 업 또는 다운 할 수도분석.
이 시약 (4) 대체 PCR 등급 물을 필요로하지 않는 악기를 사용하여.
| 성분 | 반응 당 볼륨 (μL) (2) | 최종 농도 | 마스터 믹스 (μL) 당 볼륨 (3) |
| 배의 LightCycler 480 프로브 마스터 믹스 | (10) | 소유권 | 1050 |
| 참조 ROX 염료 (4) | 0.4 | 0.5 밀리미터 | (42) |
| PCR 등급 물 | 1.4 | 147 | |
| 10 μm의 NorGIAF | 1 | 500 nm의 | (105) |
| 10 μm의 NorGIAR | 1 | 500 nm의 | (105) |
| 10 μm의 NorGIAP | 0.2 | (10)0 nm의 | (21) |
| 합계 | (14) | 1470 |
표 4. 노로 바이러스 GIA에 대한 PCR 마스터 믹스 (NOV GIA) 분석 (1).
표 3의 각주 참조 (1) - (4).
| 성분 | 반응 당 볼륨 (μL) (2) | 최종 농도 | 마스터 믹스 (μL) 당 볼륨 (3) |
| 배의 LightCycler 480 프로브 마스터 믹스 | (10) | 소유권 | 1050 |
| 참조 ROX 염료 (4) | 0.4 | 0.5 밀리미터 | (42) |
| PCR 등급 물 | 0.3 | 31.5 | |
| 10 μm의 NorGIBF | 1 | 500 nm의 | (105) |
| 10 μm의 NorGIBR | 1.8 | 900 nm의 | 189 |
| 10 μm의 NorGIBP | 0.5 | 250 nm의 | 52.5 |
| 합계 | (14) | 1470 |
표 5. 노로 바이러스 GIB에 대한 PCR 마스터 믹스 (NOV GIB) 분석 (1).
표 3의 각주 참조 (1) - (4).
| 성분 | 반응 당 볼륨 (μL) (2) | 최종 농도 | 마스터 믹스 (μL) 당 볼륨 (3) |
| 배의 LightCycler 480 프로브 마스터 믹스 | (10) | 소유권 | 1050 |
| 참조 ROX 염료 (4) | 0.4 | 0.5 밀리미터 | (42) |
| PCR 등급 물 | 0.3 | 31.5 | |
| 10 μm의 NorGIIF | 1 | 500 nm의 | (105) |
| 10 μm의 NorGIIR | 1.8 | 900 nm의 | 189 |
| 10 μm의 NorGIIP | 0.5 | 250 nm의 | 52.5 |
| 합계 | (14) | 1470 |
표 6. 노로 바이러스 GII에 대한 PCR 마스터 믹스 (NOV GII) 분석 (1).
표 3의 각주 참조 (1) - (4).
| 성분 | 반응 당 볼륨 (μL) (2) | 최종 농도 | 마스터 믹스 (μL) 당 볼륨 (3) |
| 배의 LightCycler 480 프로브 마스터 믹스 | (10) | 소유권 | 1050 |
| 참조 ROX 염료 (4) | 0.4 | 0.5 밀리미터 | (42) |
| PCR 등급 물 | 0.3 | 31.5 | |
| 10 μm의 MuNoVF1 | 1 | 500 nm의 | (105) |
| 10 μm의 MuNoVR1 | 1.8 | 900 nm의 | 189 |
| 10 μm의 MuNoVP1 | 0.5 | 250 nm의 | 52.5 |
| 합계 | (14) | 1470 |
표 7. PCR 마스터 믹스 쥐 노로 바이러스 분석 (1).
표 3의 각주 참조 (1) - (4).
| 성분 | 음량반응 당 (μL) (2) | 최종 농도 | 마스터 믹스 (μL) 당 볼륨 (3) |
| 배의 LightCycler 480 프로브 마스터 믹스 | (10) | 소유권 | 1050 |
| 참조 ROX 염료 (4) | 0.4 | 0.5 밀리미터 | (42) |
| PCR 등급 물 | 1.4 | 147 | |
| 10 μm의 HepF | 1 | 500 nm의 | (105) |
| 10 μm의 HEPR | 1 | 500 nm의 | (105) |
| 10 μm의 HEPP | 0.2 | 100 nm의 | (21) |
| 합계 | (14) | 1470 |
표 8. 간염 G에 대한 PCR 마스터 믹스 (HGV) 분석 (1).
만나다 각주 표 3 (1) - (4).
| 표준 곡선 농도 | RT-qPCR에 분석 당 게놈 복사 (1, 2) |
| 2.5 × 10 (8) | 502500 |
| 2.5 × 10 (7) | 50250 |
| 2.5 × 106 | 5025 |
| 2.5 × 105 | 502.5 |
| 2.5 × 104 | 50.25 |
| 2.5 × 10 3 | 5.025 |
표 9. 표준 곡선 게놈 복사합니다.
(1) 표준과 표준 곡선의 우물을 확인하고 열 순환기 소프트웨어의 적절한 장소에 RT-qPCR에 분석 값에 따라 게놈 사본을 배치합니다.
(2) 상 허용되는 표준 곡선은 70 % -110 %의 효율을 갖, R 2 값> 0.97, 및 노로 바이러스에 대한의 전체 표준 편차 <0.5 엔테로 바이러스에 대한 <1.0.
| 기준 | 허용되는 값 | |
| 노로 바이러스 | 엔테로 바이러스 | |
| 전체 표준 편차 | <0.5 | <1.0 |
| R 2 | > 0.97 | > 0.97 |
| 능률 | 115 % 70 % | 115 % 70 % |
표 10. 표준 곡선 허용 기준 (1).
(1) 70 % -110 %의 %의 효율이 표준 곡선은 허용하지만, 90 % -115 %의 범위의 값은 이상적이다. 90 % 미만의 값은 피펫 또는 희석 오류가있을 수 있습니다.
| 품질 보증 구성 요소 | 평균 복구 범위 (%) | 변동 계수 (%) |
| 실험실 시약 빈; 부정적인 PT 또는 PE 샘플 | 0 | N / A (1) |
| 연구소 빈 강화; 연구소 강화 샘플 매트릭스 | 5-200 | N / A |
| 긍정적 인 PT 및 PE 샘플 | 15-175 | ≤130 |
표 11. 방법 1615 성능 기준.
(1) 해당 사항 없음.
| 미디어 | 구성 |
| 0.15 M 인산 나트륨, pH가 7.0-7.5 | 1 L의 DH의 최종 부피 인산 나트륨 이염 기성 (NA 2 HPO 4 · 7H 2 O) 40.2 g을 용해하여 0.15 M 인산 나트륨을 제조 2 O HCl을 7.0-7.5로 pH를 조정합니다. 121 ° C를 15 분 15 PSI에 오토 클레이브. 12 개월까지 실온에서 보관 인산 나트륨 용액. |
| 5 % BSA | DH 2 O. 100ml에 BSA 5g을 용해하여 준비 0.2 ㎛의 멸균 필터를 통해 용액을 통과시킴으로써 멸균. |
| PBS, 0.2 % BSA | PBS 96 ml의 5 % BSA 4㎖를 첨가하여 준비한다. 0.2 ㎛의 멸균 필터를 통해 용액을 통과시킴으로써 멸균. |
| TSM III 버퍼 | 1.21 G Trisma베이스, 5.84 g의 염화나트륨, 0.203 그램의 MgCl 2, 1 ml의 Prionex 젤라틴, 950 ml의 시약 등급의 물에 3 ㎖ Microcide III를 녹인다. 7.0의 pH를 조정 한 후 0.2 ㎛의 멸균 필터를 통해 용액을 통과시킴으로써 하나 L. 살균 최종 부피를 가지고. |
| 0.525 % 차아 염소산 나트륨 (차아 염소산 나트륨) | D에서 가정용 표백제 1:10 희석하여 0.525 % 차아 염소산 나트륨 용액을 제조H 2 O 실온에서 최대 1 주간 0.525 % 차아 염소산 나트륨 용액을 저장합니다. |
| 1 M 나트륨 티오 설페이트 (NA 2 S 2 O 3) 수화물 | DH 2 O 1 L에 S 2 O 3 나 2의 248.2 g을 용해하여 1 M 용액을 준비 실온에서 최대 6 개월 동안 저장 티오 황산나트륨. |
미디어의 표 12 표.
토론
소스 마시는 물의 바이러스 오염의 대규모 국가 연구는 여러 분석 실험실의 사용을 필요로한다. 이러한 조건 하에서 표준 방법은 여러 실험실에서 생성 된 데이터가 비교 될 수 있도록 요구된다. 바이러스 검출을위한 많은 출판 분자 방법,하지만 거의 표준화 된 분자 방법이 있습니다. EPA 방법 1615 구체적으로 RT-qPCR에 의한 물 행렬에 장내 바이러스와 노로 바이러스의 검출을 위해 설계된 표준화 된 방법이다. 표준화 된 분자 방법은 식품에서 바이러스 탐지에 사용할 수있는, 16, 17 (15216-2 CEN / ISO TS 15216-1 및 CEN / ISO TS 2013년 4월 7일) 및 간염의 검출 바이러스와 노로 바이러스 봄에 적용된 물 (16). 모든 표준 방법은 품질 성능 제어 및 기준 상호 최소화하고 내 실험실 변화 인해 실험실 오염 거짓 긍정적 인 데이터를 포함해야합니다. 또한, 전자를 거짓 데이터를 줄이기 위해PA 방법 1,615 처리 한 방법 워크 플로우 동안 작업의 분리를 규정 분자 방법에 대한 EPA의 지침, (18)을 따른다. 그것은 형 간염 G에게 1,19 내부 통제 인한 RT-qPCR에 (15)의 억제제 거짓 부정적인 결과를 최소화하기위한 절차를 포함한다. 이는 모든 필드 데이터의 필드 리터 또는 마시는 물 샘플 당 게놈 사본에서 발현되도록 분석 모두 샘플링 물과의 표준화 된 볼륨과 함께 정량 분석법을 사용한다. 효율적인 단일 튜브 (원스텝) RT-PCR 분석은 시판되고 있지만, 본 방법은 의도적으로 분리 된 분석법을 사용한다. 이는 각 반응에서 분석 될 수있는 샘플들의 양을 최소화하는 단점이 있지만, 다중 프라이머 세트의 사용에 더 많은 유연성을 제공한다. 프라이머 및 프로브를 사용하고 가능성이 더 프라이머 세트는 그룹 내의 모든 바이러스 변종을 감지하지 않습니다으로 RT-qPCR의 분석에 의해 만 좋은 제한됩니다. 장내 프라이머 세트가 있기 때문에 선택되었다이는 (20, 21)이 장내 바이러스 혈청 형의 다양한 검출 보존 5'- 비 코딩 영역을 표적으로하고, 그것에 의해 검출 된 바이러스는 미처리 한 지하수의 소비에서 건강에 미치는 영향과 관련된다. 두 프라이머 세트는 내가 22, 23을 noroviruses 유전자형의 검출에 사용된다. 첫 번째는 어린 아이들의 건강에 미치는 영향 사이의 강한 상관 관계로 인해 선택 및 바이러스 1 검출되었다. 그들은 균주 (24, 25)의 폭 넓은 다양성을 감지하기 때문에 설정 프라이머 두 번째 유전자형과 유전자형 II noroviruses에 사용되는 프라이머 세트가 선택되었다.
물에서 바이러스 성 RNA를 검출 qPCR에와 RT-qPCR에 절차의 주요 장점에도 불구하고, 몇 가지 제한 사항이 있습니다. 제, 소독제 불 활성화를 포함하여 모두 감염성 및 비 감염성 바이러스 입자는, 이러한 절차에 의해 증폭 될 수있다. Borchardt의 결과는 이것이 미처리 지하수 f를위한 문제의 적은 것을 제안지역 사회에서 유사한 롬 대수층은 소독 지표수 1보다 공부했다. 배양 바이러스 예방 프로그램으로이 문제는 문화 (26, 27)와 함께 PCR을 이용하여 극복 될 수있다. 문제는 또한 핵산 가교제 28 내지 30의 이용을 통해 몇몇 바이러스 해결되었다. 이 후자의 방법은 바이러스 차아 염소산에 의해 불 활성화 및 자외선에 의해 불 활성화에게는 효과적이지 더 효과적입니다.
이들 분자 절차 번째 제한은 통상적으로 분석 할 수있다 농축 샘플의 부피는 배양 과정 6,31 위해 사용 된 것보다 훨씬 더 작다는 것이다. 이 문제는 종종 어느 샘플이 작은 부피에 재현 탁하고, 또는 삼차 시료 농도 단계 6,32,33을 첨가 할 수 있도록 유기 응집에 의한 표준 이차 농도에 대한 폴리에틸렌 글리콜 기반의 절차를 치환하여 취급 . 방법 1615 centrif를 사용ugal 한외 여과는 차의 농도를 제공합니다. 원심 한외 여과 물 및 시료의 모든 바이러스의 양 농도 및 분자 분석의 작은 분자량 억제제의 감소의 결과로 이하의 성분을 제거 30,000 달톤. 물에 존재했던 어떤 바이러스> 10 (5)의 전체 집중 계수에이 차 농축 단계의 결과는 테스트되고.
세 번째 제한은 환경 시료에서 분자 절차의 억제제의 존재이다. 억제제를 제거하기 위해 다양한 방법이 개발되었지만, 어떠한 방식은 필수적 억제의 레벨을 추정하기위한 내부 제어의 활용, 모든 물 행렬 및 바이러스 유형 6,34,35 효과적 없다. 이 방법에 사용 간염 G는 모든 시약 및 반응 억제를 추정하기위한 RT-qPCR의 분석에서 바이러스 RNA의 일정한 레벨을 제공함으로써 이러한 요구를 만족시킨다. 때 T의 최고그 시료를 농축 한 바이러스 농도가 억제제 농도 14,15 이상만큼 희석 될 수 있고, 제거 접근법 억제가 제거되지 억제제.
본원에 기술 된 표준 곡선 절차 주요 장점과 한계를 모두 갖는다. 장점은 시약이 단일 제어가 모든 분석에 사용될 수 있도록, 하나의 시약의 공급 모든 필요한 부품을 사용한다는 것이다. 이 시약은 특히 노로 바이러스 시험 법에 대한 장점이다. 노로 바이러스 입자는 단지 매우 어려운 기준으로 사용하기 위해 바이러스 입자를 얻을 수있다 감염된 개체로부터 얻어 질 수있다. 더 중요한 장점은 RNA 표준 RNA (36)의 정확한 정량을 위해 필수적이다 갖는 것으로는, 하나의 시약의 모든 표적 RNA 바이러스에 대한 RNA 표준을 제공한다는 것이다. 그러나, 바이러스를 정확하게 계량하는 능력은 매트릭스 효과가 고려되지 않는다는 사실에 의해 제한된다. 이것은 그 게놈 사본을 의미합니다숫자 값은 절대 간주 될 수없고 단지 상대적인 관점에서 고려되어야한다. 그것은 표준 곡선 작업입니다 씩 충분한 수의가 (1.2 단계) 완료 연구를 충당하기 위해 준비하는 것이 좋습니다. 예를 들어, 각각의 250 μL 분취 6 RT 플레이트 충분한 시약을 제공한다. 이 연구는 500 샘플의 분석을 필요로하는 것이 공지되어있는 경우, 12 분취 량의 최소값 (500 샘플 RT 플레이트 당 / 7의 샘플 / RT 분취 량 당 6 판)을 필요로 할 것이다.
EPA 방법 1615은 성능 기반 방법입니다. 많은 제조업 자들은 여기에 명시 동등한 시약을 이들 시약만큼 성능 기준을 충족으로 치환 될 수있다. 또한 긍정적 인 RT-qPCR의 제어 역할을 표준 곡선은 성능 문제를 해결 가치이다. 성능으로 인해 RNA의 분해, 시약의 유효 기간, 냉동고의 고장, 장비 교정 및 기술 오류로 거부 할 수 있습니다. 성능 문제가 의심해야그들은 표준 곡선에 대한 성능 규격을 만족하지 않는 경우는 ED 표준 곡선은도 4에 도시 된 것과 다를 경우 나. RT-qPCR의 분석은 매우 강력합니다; 완전한 실패로 인해 RNA 또는 기술적 오류 (예를 들어, 누락 된 시약)의 부적절한 취급에 가능성이있다. 세심 추출 및 리보 뉴 클레아로부터 RNA의 분해를 감소시키는 단계 사이 RT RNA 샘플을 처리에주의해야한다.
지상 및 시약 등급의 물과 지하수에서 쥐 노로 바이러스의 회복에서 소아마비 바이러스의 회복은 EPA 방법 1,615 성능 허용 기준 (표 11)를 만나 다른 33,37,38에 의해보고 된 것과 유사하다. LFB 샘플에서 쥐 노로 바이러스 회복은 소아마비 바이러스보다 훨씬 낮았다과 소아마비 바이러스 별 허용 기준을 충족하지 않을 것입니다. 시약 등급의 물에서 낮은 쥐 노로 바이러스 복구하는 이유는 알려져 있지 않다. 여기에 결과와 유사하게, 카림과 collea에는 동급 수돗물 39에서 4 %의 노로 바이러스의 GI.1에 대한 복구를보고했다. 리 등. 37 뮤린 노로 18 %의 인간의 GII.4 노로 각각 디스크 필터를 사용하여 증류수에 대한 평균 26 % 회수율을보고했다. 리와 동료에게 유사한 조건을 사용하여, 김과 코는 각각 38,이 바이러스를 46 %와 43 %의 회수율을 관찰했다. 기븐스 외. (40)는 해수로부터 인간 노로 바이러스의 GII.4 100 % 회수 주위 수득하지만 김과 코 (38)의 농도와 유사한 또는 쥐 바이러스 상당히 감소 회수율 해수 이상과 증류수로 염의 첨가를 초래한다는 발견 GII.4 복구 약 두 배 감소.
공개
The authors have nothing to disclose.
감사의 말
The authors thank Dr. Eric Rhodes for preparing the clones used in the development of Standard curve reagent, Brian McMinn for assistance in sample processing, Larry Wymer for statistical analysis, Dr. Mark Borchardt, U.S. Department of Agriculture, Marshfield, WI, for supplying the Sabin poliovirus serotype 3 used in this study and Dr. H. W. Virgin, Washington University, St. Louis, MO, for murine norovirus. The authors also acknowledge Gretchen Sullivan for assistance in preparation of stock laboratory reagents, Dr. Mohammad Karim for propagation of murine norovirus stocks, and local private well owners and utilities for allowing us to collect water samples. Although this work was reviewed by EPA and approved for publication, it may not necessarily reflect official Agency policy. Mention of trade names or commercial products does not constitute endorsement or recommendation for use.
자료
| Name | Company | Catalog Number | Comments |
| 1.5 ml tube chamber | Diversified Biotech | CHAM-3000 | |
| 1 °C cool brick | Diversified Biotech | BRIK-2501 | |
| 10x PCR Buffer II and 25-mM MgCl2 | Life Technologies | N8080130 | |
| -20 °C freezer | VWR | 97043-346 | Must be a manual defrost freezer |
| -70 °C or colder freezer | Thermo Scientific | MBF700LSAO-E | |
| 96-well chamber | Diversified Biotech | CHAM-1000 | |
| Absolute ethanol | Fisher Scientific | BP2818-100 | |
| Armored RNA EPA-1615 | Asuragen | Custom order | Used for quantifying the RT-qPCR assay |
| Armored RNA Hepatitis G virus | Asuragen | 42024 | |
| Autoclave | Steris | Amsco Lab Series | |
| Biosafety cabinet | NuAir Laboratory Equipment Supply | Labgard 437 ES | |
| Bovine serum albumin (BSA) | Affymetrix | 10856 | Crystalline grade or better |
| Buffer AVE | Qiagen | 1026956 | Carrier RNA dilution buffer |
| Buffer AVL | Qiagen | 19073 | Extraction buffer |
| Carrier RNA | Qiagen | Not applicable | Use carrier RNA supplied with Buffer AVL |
| Centrifuge bottles | Fisher Scientific | 05-562-23 or 05-562-26 | |
| Centrifuge rotors | Beckman Coulter | 339080, 336380 | |
| Cool safe box | Diversified Biotech | CSF-BOX | |
| Dithiothreitol (DTT) | Promega | P1171 | |
| LightCycler® 480 Probes Master kit | Roche Diagnostics | 4707494001 | |
| Microcentrifuge tubes | Fisher Scientific | 02-682-550 | Use ribonuclease- and deoxyribonuclease-free tubes with snap caps |
| Microcide III | Fitzgerald | 99R-103 | |
| Microseal 'A' film | Bio-Rad Laboratories | HSA5001 | Heat resistant |
| Microseal 'F' film | Bio-Rad Laboratories | MSA1001 | Freezer resistant |
| Mini-plate spinner | Labnet International | MPS1000 | |
| Multichannel pipette | Rainin | L8-20 | |
| Multi-tube chamber | Diversified Biotech | CHAM-5000 | |
| Optical reaction plate | Life Technologies | 4314320 | |
| PCR nucleotide mix | Promega | U1515 | |
| PCR plate | Bio-Rad Laboratories | HSS9601 | |
| PCR-grade water | Roche | 3315932001 | |
| Phosphate buffered saline (PBS) | U.S. Biological | D9820 | |
| Plate mixer | Scientific Industries | MicroPlate Genie | |
| Prionex gelatin | Sigma Aldrich | G0411 | |
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Includes Buffers AW1 (first wash buffer), AW2 (second wash buffer), AE (elution buffer); ethanol must be added to Buffers AW1 and AW2 before use; Do not use Buffer AL supplied with the kit |
| Quantitative PCR thermal cycler | Life Technologies | 4351405 | |
| Random primer | Promega | C1181 | |
| Reagent Reservoir | Fisher Scientific | 21-381-27E | |
| Refrigerated centrifuge | Beckman Coulter | 367501 | |
| RNase Inhibitor | Promega | N2515 or N2615 | |
| ROX reference dye | Life Technologies | 12223 | |
| SuperScript II or III Reverse Transcriptase | Life Technologies | 18064-022 or 18080044 | |
| Surgical gloves | Fisher Scientific | 19-058-800 | |
| Thermal cycler | Life Technologies | 4314879 | |
| Trisma base | Sigma Aldrich | T1503 | |
| Vivaspin 20 centrifugal concentrator units | Sartorius-Stedim | VS2022 |
참고문헌
- Borchardt, M. A., Spencer, S. K., Kieke, B. A., Lambertini, E., Loge, F. J. Viruses in nondisinfected drinking water from municipal wells and community incidence of acute gastrointestinal illness. Environ. Health Perspect. 120 (9), 1272-1279 (2012).
- Ye, X. Y., et al. Real-time PCR detection of enteric viruses in source water and treated drinking water in Wuhan, China. Curr. Microbiol. 65 (3), 244-253 (2012).
- Williamson, W. M., et al. Enteric viruses in New Zealand drinking-water sources. Water Sci. Technol. 63 (8), 1744-1751 (2011).
- Lambertini, E., Borchardt, M. A., Kieke, B. A., Spencer, S. K., Loge, F. J. Risk of viral acute gastrointestinal illness from nondisinfected drinking water distribution systems. Environ. Sci. Technol. 46 (17), 9299-9307 (2012).
- Chigor, V. N., Okoh, A. I. Quantitative RT-PCR detection of hepatitis A virus, rotaviruses and enteroviruses in the Buffalo River and source water dams in the Eastern Cape Province of South Africa. Int. J. Environ. Res. Public Health. 9 (11), 4017-4032 (2012).
- Fout, G. S., Martinson, B. C., Moyer, M. W., Dahling, D. R. A multiplex reverse transcription-PCR method for detection of human enteric viruses in groundwater. Appl. Environ. Microbiol. 69 (6), 3158-3164 (2003).
- Hewitt, J., Bell, D., Simmons, G. C., Rivera-Aban, M., Wolf, S., Greening, G. E. Gastroenteritis outbreak caused by waterborne norovirus at a New Zealand ski resort. Appl. Environ. Microbiol. 73 (24), 7853-7857 (2007).
- Jack, S., Bell, D., Hewitt, J. Norovirus contamination of a drinking water supply at a hotel resort. New Zealand Med. J. 126 (1387), 98-107 (2013).
- Anderson, A. D., et al. A waterborne outbreak of Norwalk-like virus among snowmobilers-Wyoming, 2001. J. Infect. Dis. 187 (2), 303-306 (2003).
- Parshionikar, S. U., et al. Waterborne outbreak of gastroenteritis associated with a norovirus. Appl. Environ. Microbiol. 69 (9), 5263-5268 (2003).
- U.S. Environmental Protection Agency Office of Water. . 820-F-12-058: Recreational Water Quality Criteria. , 1-63 (2012).
- Wade, T. J., et al. Rapidly measured indicators of recreational water quality and swimming-associated illness at marine beaches: a prospective cohort study. Environ. Health. 9, 66 (2010).
- Fout, G. S., et al. . Method 1615: Measurement of enterovirus and norovirus occurrence in water by culture and RT-qPCR (EPA/600/R-10/181). , 1-91 (2012).
- Cashdollar, J. L., et al. Development and Evaluation of EPA Method 1615 for Detection of Enterovirus and Norovirus in Water. Appl. Environ. Microbiol. 79 (1), 215-223 (2013).
- Gibson, K. E., Schwab, K. J., Spencer, S. K., Borchardt, M. A. Measuring and mitigating inhibition during quantitative real time PCR analysis of viral nucleic acid extracts from large-volume environmental water samples. Water Res. 46 (13), 4281-4291 (2012).
- Fuentes, C., et al. Standardized multiplex one-step qRT-PCR for hepatitis A virus, norovirus GI and GII quantification in bivalve mollusks and water. Food Microbiol. 40, 55-63 (2014).
- Coudray, C., Merle, G., Martin-Latil, S., Guillier, L., Perelle, S. Comparison of two extraction methods for the detection of hepatitis A virus in lettuces using the murine norovirus as a process control. J. Virol. Methods. 193 (1), 96-102 (2013).
- Sen, K., et al. . EPA 815-B-04-001: Quality assurance/quality control guidance for laboratories performing PCR analyses on environmental samples. , 1-56 (2004).
- Schlueter, V., Schmolke, S., Stark, K., Hess, G., Ofenloch-Haehnle, B., Engel, A. M. Reverse transcription-PCR detection of hepatitis G virus. J. Clin. Microbiol. 34 (11), 2660-2664 (1996).
- De Leon, R., Shieh, C., Baric, R. S., Sobsey, M. D. Detection of enteroviruses and hepatitis A virus in environmental samples by gene probes and polymerase chain reaction. Proc. Water Qual. Technol. Conf. , 833-853 (1990).
- Monpoeho, S., et al. Quantification of enterovirus RNA in sludge samples using single tube real-time RT-PCR. BioTechniques. 29 (1), 88-93 (2000).
- Jothikumar, N., et al. Rapid and sensitive detection of noroviruses by using TaqMan-based one-step reverse transcription-PCR assays and application to naturally contaminated shellfish samples. Appl. Environ. Microbiol. 71 (4), 1870-1875 (2005).
- da Silva, A. K., et al. Evaluation of removal of noroviruses during wastewater treatment, using real-time reverse transcription-PCR: different behaviors of genogroups I and II. Appl. Environ. Microbiol. 73 (24), 7891-7897 (2007).
- Butot, S., et al. Evaluation of various real-time RT-PCR assays for the detection and quantitation of human norovirus. J. Virol. Methods. 167 (1), 90-94 (2010).
- Loisy, F., et al. Real-time RT-PCR for norovirus screening in shellfish. J. Virol. Methods. 123 (1), 1-7 (2005).
- Reynolds, K. A., Gerba, C. P., Pepper, I. L. Detection of infectious enteroviruses by an integrated cell culture-PCR procedure. Appl. Environ. Microbiol. 62 (4), 1424-1427 (1996).
- Ming, H. X., Zhu, L., Zhang, Y. Rapid quantification of infectious enterovirus from surface water in Bohai Bay, China using an integrated cell culture-qPCR. Mar. Pollut. Bull. 62 (10), 2047-2054 (2011).
- Parshionikar, S., Laseke, I., Fout, G. S. Use of propidium monoazide in reverse transcriptase PCR to distinguish between infectious and noninfectious enteric viruses in water samples. Appl. Environ. Microbiol. 76 (13), 4318-4326 (2010).
- Sanchez, G., Elizaquivel, P., Aznar, R. Discrimination of infectious hepatitis A viruses by propidium monoazide real-time RT-PCR. Food Environ. Virol. 4 (1), 21-25 (2012).
- Kim, S. Y., Ko, G. Using propidium monoazide to distinguish between viable and nonviable bacteria, MS2 and murine norovirus. Lett. Appl. Microbiol. 55 (3), 182-188 (2012).
- Fout, G. S., Schaefer, F. W., Messer, J. W., Dahling, D. R., Stetler, R. E. . EPA/600/R-95/178: ICR Microbial Laboratory Manual, I.1-ApD-23. , (1996).
- Abbaszadegan, M., Stewart, P., LeChevallier, M. A strategy for detection of viruses in groundwater by PCR. Appl. Environ. Microbiol. 65 (2), 444-449 (1999).
- Lambertini, E., et al. Concentration of enteroviruses, adenoviruses, and noroviruses from drinking water by use of glass wool filters. Appl. Environ. Microbiol. 74 (10), 2990-2996 (2008).
- Rodriguez, R. A., Thie, L., Gibbons, C. D., Sobsey, M. D. Reducing the effects of environmental inhibition in quantitative real-time PCR detection of adenovirus and norovirus in recreational seawaters. J. Virol. Methods. 181 (1), 43-50 (2012).
- Iker, B. C., Bright, K. R., Pepper, I. L., Gerba, C. P., Kitajima, M. Evaluation of commercial kits for the extraction and purification of viral nucleic acids from environmental and fecal samples. J. Virol. Methods. 191 (1), 24-30 (2013).
- Fey, A., et al. Establishment of a real-time PCR-based approach for accurate quantification of bacterial RNA targets in water, using Salmonella as a model organism. Appl. Environ. Microbiol. 70 (6), 3618-3623 (2004).
- Lee, H., et al. Evaluation of electropositive filtration for recovering norovirus in water. J. Water Health. 9 (1), 27-36 (2011).
- Kim, M., Ko, G. Quantitative characterization of the inhibitory effects of salt, humic acid, and heavy metals on the recovery of waterborne norovirus by electropositive filters. J. Water Health. 11 (4), 613-622 (2013).
- Karim, M. R., Rhodes, E. R., Brinkman, N., Wymer, L., Fout, G. S. New electropositive filter for concentrating enteroviruses and noroviruses from large volumes of water. Appl. Environ. Microbiol. 75 (8), 2393-2399 (2009).
- Gibbons, C. D., Rodriguez, R. A., Tallon, L., Sobsey, M. D. Evaluation of positively charged alumina nanofibre cartridge filters for the primary concentration of noroviruses, adenoviruses and male-specific coliphages from seawater. J. Appl. Microbiol. 109 (2), 635-641 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유