Method Article
Generación rápida y eficiente de las células madre pluripotentes recombinante humana mediada por la recombinasa Cambio en el cassette
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí se presenta un método de edición gen rápida y eficiente basado en RMCE en el locus de AAVS1 humanas pluripotentes células madre (hPSCs) que mejora los sistemas anteriormente descritos. Usando esta técnica, líneas isogénicas se pueden generar rápida y fiable para los estudios comparativos adecuados, facilitar la investigación transgénesis mediada con hPSCs.
Resumen
Incluso con la revolución de las tecnologías de reconocimiento de genes dirigidos por CRISPR-Cas9, la modificación genética de las células madre pluripotentes humanas (hPSCs) sigue siendo lento. Los estudios comparativos que utilizan líneas recombinantes con transgenes integrados en loci puerto seguro, podrá beneficiarse de los enfoques que utilizan recombinasas específicas de sitio específico, como Cre o FLPe, que son más rápidos y menos propensos a los efectos desviado. Se han descrito Tales métodos, aunque no lo hacen significativamente gen superan la orientación en la mayoría de los aspectos. El uso de nucleasas de dedos de zinc, que previamente creó una línea de células maestras en el locus de AAVS1 hPSCs que contiene una GFP-higromicina-tk que expresa casete, flanqueado por secuencias FRT heterotípicos. A continuación, se describen los procedimientos para llevar a cabo el intercambio de casete FLPe recombinasa mediada (RMCE) usando esta línea. El maestro clínea ell se transfecta con un vector donante RMCE, que contiene un promotor de resistencia a puromicina, y con FLPe recombinasa. Aplicación del programa de selección tanto positiva (puromicina) y negativo (UIAF) conduce a la selección de RMCE sin integraciones aleatorias. RMCE genera líneas transgénicas policlonales pluripotentes caracterizado completamente en el 15 d con una eficiencia del 100%. A pesar de la recientemente descrita limitaciones del locus AAVS1, la facilidad del sistema allana el camino para HPSC transgénesis en los entornos isogénicas, es necesario para los estudios comparativos, y permite a las pantallas de genética semi-alto rendimiento para la ganancia / pérdida de análisis de funciones que de otro modo ser altamente tiempo.
Introducción
Targeted recombinación del genoma ha facilitado los rápidos avances en muchas áreas de la investigación. En ratones, la sinergia entre la edición del genoma y la investigación con células madre ha permitido una mayor comprensión del complejo mecanismo de la función de genes y la regulación de genes. Se espera que tales avances en las células humanas pluripotentes madre (hPSCs), así, a pesar de que desde hace muchos años desde que el primer aislamiento de células madre embrionarias humanas (hESCs), y más tarde humana células madre pluripotentes inducidas (hiPSCs), edición gen ha constituido un obstáculo técnico . Los recientes avances en la ingeniería del genoma utilizando nucleasas-cinc dirigidos Finger nucleasas (ZFNs), la transcripción nucleasas efectoras del tipo activador de (TALEN), o en clúster intercaladas con regularidad corto repeticiones palindrómicas / proteína CRISPR-asociado 9 (CRISPR / Cas9) nos han permitido superar estas dificultades, la toma de recombinación dirigida un proceso eficiente de 1 - 3.
loci específicos en el mouse genoma que permiten la expresión transgénica estable, fiable y ubicuo en la ausencia de efectos adversos como Rosa26, HPRT1, o Col1A1, se han convertido en herramientas esenciales en la realización de estudios genéticos. loci de salvaguarda permiten el análisis comparables entre diferentes líneas de ratones en contextos isogénicas. Esto no se puede lograr usando métodos de integración aleatoria estándar, que se asocian con limitaciones bien conocidas como la mutagénesis de inserción, los efectos dependientes de la dosis, o la expresión del transgen abigarrada. Gen de facilitar la edición y la flexibilidad en los loci puerto seguro se incrementa mediante el uso de recombinasas específicas de sitio específico como Cre o flipasa (FLPe), que reconocen específicamente secuencias diana (loxP o FRT, respectivamente) y catalizan la recombinación eficiente entre los objetivos idénticos. Debido a estas características, la edición gen de la recombinasa mediada por el uso de secuencias loxP o FRT en preintegradas loci puerto seguro es una herramienta común utilizada en el ratón transgénesis. Además de la inserción de casete o escisión mediada entre secuencias diana idénticas, el uso de sitios loxP o FRT incompatibles permite el intercambio de casete recombinasa mediada (RMCE) 4.
En humanos, los intentos de identificar loci de puerto seguro han llevado a cabo. La HPRT ortólogos de ratón, a pesar de su asociación con el síndrome de Lesch-Nyhan pérdida de función, y ROSA26 han sido blanco de hPSCs. HPRT se informó de silenciar rápidamente la expresión del transgen en el CES y, al igual ROSA26, su capacidad para sostener la expresión del transgen en terminales células diferenciadas no se investigó 5-7. Debido a una mutación nula homocigótica del gen de CCR5 parece ser bien tolerada en los seres humanos, se evaluó su valor como puerto seguro. CCR5 era específico y informó para sostener la expresión transgénica estable en diferentes líneas celulares humanas, incluyendo CES 8,9. Sin embargo,este último no se ha demostrado a nivel clonal durante el cultivo a largo plazo, y la expresión del transgen en todas partes, no se demostró en progenie diferenciada de las tres capas germinales. AAVS1, el sitio de integración natural de la Adeno Asociadas tipo Virus 2 (AAV), se puso a prueba en hESCs así, debido a la resistencia informado a silenciar transgén 10. Más tarde, muchos grupos utilizaron el locus AAVS1 y describen la expresión transgénica estable en hPSCs no diferenciadas, así como en su progenie diferenciada de las tres capas germinales, tanto in vitro como in vivo 2,8,11,12. Los resultados recientes, sin embargo, estos hallazgos matiz, ya que se encontró el locus AAVS1 para ejercer la inhibición variable de transgenes in vitro en hESCs indiferenciada y en la progenie de hepatocitos 13.
Los estudios de investigación adicionales utilizando enfoques de integración al azar y los métodos para determinar la integración de copia única destinada para encontrar genositios de integración de micro resistentes al silenciamiento de transgenes durante la expansión y diferenciación hPSCs 14,15. En general, hasta ahora, ningún sitio genómico ha sido completamente validado como un puerto seguro en hPSCs y su progenie; la identificación de un sitio apropiado para la expresión transgénica estable en todas partes, no sólo en hPSCs sino también en sus progenies diferenciadas in vitro e in vivo, queda por resolver. Entre todos los loci estudiados ya pesar de sus limitaciones, AAVS1 sigue siendo la mejor caracterizada y más utilizada en la investigación de células madre.
RMCE se ha realizado con éxito en hPSCs en algunos de estos loci 6,7,14,16, utilizando la mayoría de la recombinasa Cre, aunque hay indicios de que FLPe es más eficiente que Cre 17. En todos estos casos, se usó un casete de resistencia a los medicamentos positivo para la selección de las colonias recombinantes. Aunque tuvo éxito, estos procedimientos no constituyen un avance técnico más gene- normaprocedimientos de edición utilizando ZFNs, Talens o CRISPR / Cas9, como un único procedimiento de selección de antibióticos no descarta integraciones aleatorias y requiere la detección de colonias para identificar clones correctamente orientados.
En este procedimiento, se describen los métodos para llevar a cabo RMCE en hPSCs en el locus AAVS1 usando una combinación de positivo (dentro de la casete de entrada) y negativo (dentro de la casete preintegradas) selecciones que permitan la generación de líneas transgénicas policlonales en ± 15 días con una eficiencia del 100% y libre de eventos de integración al azar. Por lo tanto, este método representa un avance más allá de las tecnologías actualmente RMCE descritos en hPSCs.
Protocolo
1. Preparación de la HPSC Línea Celular Maestro que contiene FRT-para la transfección
- Cultura células madre / IPSC líneas de células primarios bajo procedimientos estándar dentro o fuera de fibroblastos embrionarios de ratón inactivados (IMEF), utilizando células madre o medio IPSC sin alimentador, respectivamente (Tabla 1). Preparar 2 (en alimentadores) o 1 (pozos libres de alimentador) de hPSCs en una placa de 6 pocillos para cada condición experimental.
- Tenga en cuenta las culturas, y cuando las células de los 60 años - El 70% de confluencia, placa resistente a los medicamentos IMEF (iDR4). Brevemente, recubrir los pocillos necesarios de una placa de 12 pocillos con 0,5 ml de solución de gelatina al 0,1% y se incuba durante 5 min a TA. Plate 125.000 iDR4s por pocillo e incubar O / N con medio IMEF (Tabla 1).
2. Transfección HPSC por Nucleofection
- El día siguiente, pre-incubar cultivos de células madre / IPSC con medio fresco, incluyendo inhibidor 10 M asociada a Rho de la proteína quinasa (ROCK) (Y-27632), durante 1 hora a 37 ° C. norteext, tome solución de transfección de células madre de la nevera para pre-calentar a temperatura ambiente.
- Preparar 1 ml de nucleofection medio de baño (Tabla 1) por condición nucleofection. Tomar medio IMEF fuera de los pozos, se lava con 1 ml de PBS temperatura ambiente, y añadir 500 l de nucleofection medio de baño. La transferencia de los otros 500 l a tubos de 1,5 mL estériles y almacenar a 37 ° C hasta el momento de chapado. NOTA: Un experimento típico RMCE contiene tres condiciones diferentes con relaciones moleculares de donante RMCE: vectores pFLPe de 2: 0, 2: 1, y 0: 1.
- Separar células madre / IPSC en una suspensión de células individuales.
- Quítese los medios de comunicación, se lava con 2 ml de PBS RT, y se añade 1 ml de tripsina al 0,05% o accutase en alimentador o libres de alimentador culturas HPSC, respectivamente. Incubar durante 7 (tripsina) o y 2 - 5 (accutase) minutos a 37 ° C. Para el tratamiento Accutase - células deben permanecer sueltos, pero adjunto.
- Retirar con cuidado la placa de la incubadora y sinllowing que las células se separan, eliminar el agente de disociación mediante aspiración. Añadir 1 ml de células madre de medios y disociar las células sueltas lavando suavemente los medios de comunicación sobre la superficie de la placa, evitando la formación de espuma. Asegúrese de que las células están en una sola suspensión celular.
- Recoger las células en un 15 o 50 ml tubo limpio y estéril. Lavar los pocillos con 1 ml de medio de células madre y recoger las células en el mismo 15 o 50 ml tubo. Tomar una parte alícuota de 50 l para contar (durante el paso siguiente) usando un dispositivo de recuento celular. Anotar el volumen de recogida y calcular el número total de células. Centrifugar las células a 300 xg durante 5 min a TA.
- Resuspender el sedimento celular a una suspensión de 10 6 células / ml con PBS utilizando una pipeta serológica ml 5 y pipeteo suave. Evitar la formación de espuma. Pasar 2 ml por cada condición experimental (aproximadamente 2 x 10 6 células) para limpiar, tubos de 15 ml estériles.
- Si bien la centrifugación, prepare el RMCE donante-plásmidos pFLPe se mezcla en un volumen máximo de 10 mL.
- Transferir 2,5 g de pFLPe (6,5 Kb) a un tubo limpio, estéril 1,5 ml. Añadir una proporción 2: 1 molecular del vector donante RMCE 13 al vector pFLPe. Por ejemplo, aproximadamente 10 mg de un vector de 12 Kb RMCE donante.
- Proceder con nucleofection, evitando la formación de espuma en todas las etapas.
NOTA: Algunos protocolos de transfección HPSC aplican un paso adicional antes de agotamiento del IMEF el paso 2.4. Sin embargo, en la práctica, IMEF es una fracción celular de menor importancia en la única suspensión que no altera significativamente la eficacia de transfección, ni va a crear contaminación de las células recombinantes, ya que no son proliferativas. Además, es esencial para proceder rápidamente durante el proceso de transfección para aumentar la viabilidad. Por estas razones, no se aplica el agotamiento del IMEF.- Retire la placa iDR4 y los tubos de 1,5 mL con el medio chapadode la incubadora. Un tubo a la vez, pipetear de la PBS con cuidado, sin alterar el sedimento celular. Eliminar la mayor cantidad posible. Resuspender el precipitado con 100 l de solución de transfección pipeteando suavemente. Transferir la suspensión celular al tubo de 1,5 ml que contiene las mezclas de plásmidos y mezclar suavemente.
- Transferir la mezcla de células-ADN a la cubeta transfector, teniendo cuidado de no introducir burbujas. Introducir la cubeta en el dispositivo nucleofector y aplicar A13 programa (células cultivadas en los alimentadores) o F16 (alimentador libre).
NOTA: Otros métodos de transfección o nucleofector dispositivos pueden requerir la optimización de estos programas o condiciones de transfección. - Volver a tomar la cubeta y, utilizando las pipetas de transferencia desechables disponibles en el kit nucleofection, recoger su contenido mediante la aplicación de 0,5 ml del medio de recubrimiento y aspirar todo el volumen. Llevar a cabo este paso de forma rápida, pero con cuidado y en un solo movimiento.
- Placa gota a gota en el 12-well placa que contiene el iDR4 y 500 l de medio de baño. Repita los pasos 2.6.1 - 2.6.4 para la siguiente condición experimental.
- Coloque la placa de cultivo en un estante en la incubadora y se mueven lentamente hacia atrás y hacia atrás y de lado a lado para distribuir uniformemente la suspensión celular. Incubar durante 24 h antes de la siguiente cambio de medios de comunicación para permitir que las células se recuperen en presencia de 10 mM inhibidor de roca (Y-27632).
3. selección positiva y negativa de las células sometidas a RMCE
- medios de cultivo de cambio al día (1 ml / pocillo), y 2 - 3 días post-transfección, comienzan selección con 100 ng / ml de puromicina.
NOTA: Las concentraciones óptimas de reactivos de selección positivos y negativos deben ser determinados experimentalmente para cada línea de células maestras HPSC recién generado. Realizar una curva de muerte 13, usando la línea de células maestras para determinar la dosis baja (concentración a la que la toxicidad visual mínimo es evidente después de 7d de la selección, pero la cultura no es demasiado grande), la dosis óptima (la concentración más baja a la que todas las células están muertas después de 7 d de selección), y la dosis alta (concentración que provocó toxicidad evidente, matando a las células después de 2 - 3 d) tanto para puromicina y Fialuridine (FIAU).- Aspirar el medio células madre. Lavar con 1 ml de PBS. Añadir medio hESC con 100 ng / ml de puromicina.
- Observar el crecimiento celular para que la muerte y el crecimiento están equilibrados. Cambiar los medios de comunicación con puromicina al día, después del paso 3.1.1. Cuando la tasa de crecimiento supera a la muerte celular, aumentar la concentración de puromicina en bloques de 25 - 50 ng / ml hasta un máximo de 250 ng / mL. Continuar la selección de puromicina 5 - 7 d.
- 3 - 4 días después de comenzar la selección de puromicina, alrededor del día 6 después de la transfección, comenzará la selección con 0,5 M UIAF. Cambiar los medios de comunicación todos los días y mantener UIAF por no más de 7 días continuos.
4. La expansión y caracterización de líneas RMCE
- Tras la finalización de la selección, en torno a D14 - 15, las colonias resistentes RMCE están presentes y constituyen la nueva línea RMCE. Dividir a granel en una proporción 1: 2 siguiendo los procedimientos estándar (paso 1, P1).
- Placa de 2 pocillos de una placa de 12 pocillos (dentro o fuera de los alimentadores, dependiendo de las condiciones de cultivo habituales de la línea celular maestra original). Utilice un pocillo para una mayor expansión, almacenamiento o disposición experimental (sin descripción adicional de estos procedimientos se realiza en este protocolo), y el otro para la caracterización.
- Cuando las células de P1 están dispuestos para dividir, disociar el bien para la caracterización en una única suspensión celular como se describe en 2.4 y recoger las células en un tubo de 15 ml. NOTA: Use las células de la HPSC WT (sin modificación genética) y la línea de células maestras como controles negativos y positivos, respectivamente, para la caracterización.
- Centrifugar a 300 xg durante 5 min a TA. Resuspender en 1 ml de PBS y se divide la muestra en dos por citometría de flujoy el análisis de ADN 13.
- La citometría de flujo análisis de 13 (300 L):
- Añadir suero de ternera fetal a las células en PBS hasta una concentración final de 5%. Transferir las células a un tubo de FACS limpio con un filtro de células y mantenerlas en hielo.
NOTA: La viabilidad celular es alto en estas condiciones, por lo que es posible, pero no necesario, utilizar una mancha fluorescente de células no viables (por ejemplo, 7-AAD o PI). - Proceder de inmediato a análisis en un citómetro de flujo analizador de células y registrar 20.000 - 30.000 eventos. Para el análisis, establecer las puertas anteriores GFP o TDT fondo de fluorescencia usando la muestra de control negativo WT.
- Añadir suero de ternera fetal a las células en PBS hasta una concentración final de 5%. Transferir las células a un tubo de FACS limpio con un filtro de células y mantenerlas en hielo.
- Análisis de ADN 13 (700 l):
- Transferir las células a un tubo limpio de 1,5 ml. Centrifugar a 300 xg durante 5 min a RT y aspirar el sobrenadante. Proceder a la extracción de ADN utilizando un kit comercial y siga las instrucciones del fabricante. Almacenar el sedimento seco a -20 ° C durante latEl análisis er.
- Medir la concentración de ADN.
- Preparar reacciones estándar de PCR de acuerdo con las condiciones de la Figura 3 y la Tabla 2 muestras Run PCR 13 en un gel de agarosa al 2% que contenía 1x mancha de gel de ADN a 150 V durante 15 - 25 min.. Analizar el funcionamiento de un analizador de gel.
Resultados
Usando AAVS1 ZNFs específicos de, líneas completamente caracterizados de células primarios células madre / IPSC que contienen secuencias diana FRT heterotípicos se generaron y se describen 13. Las líneas de células maestras que contienen FRT-mantuvieron la pluripotencia y la integridad del genoma después del tratamiento ZFN y de manera estable expresan GFP in vitro e in vivo. RMCE se realiza mediante co-transfección de las líneas de células primarios con FLPe recombinasa y vectores donantes RMCE (Figura 1A). Vectores RMCE contienen secuencias FRT idénticos a los de la línea de células maestras AAVS1 que flanquean el transgén Figura 1B supervisa RMCE mediante el que expresan constitutivamente TDT pZ:.. F3-P CAGGS tdTPH-F Después de la transfección, HPSC formar pequeños grupos de células distribuidas uniformemente a lo largo la capa iDR4 MEF, y se transfectaron HPSC expresan tanto GFP así como transitoria TDT (Figura 1B, D 2). Las células se dejaron recuperar durante 2-3d después de la transfección antes de iniciar la selección. La selección positiva se empezó a usar una dosis baja de puromicina con el fin de favorecer la inserción de los donantes RMCE. Puromicina se aplica suavemente al principio, porque la muerte celular masiva inicial podría dar lugar a células individuales se someten a recombinación, haciéndolos incapaces de sobrevivir al proceso. En los siguientes días, la concentración de puromicina necesita ser levantada por etapas hasta la dosis óptima con el fin de permitir que las células recombinantes para crecer en pequeñas colonias, mientras que las células nonrecombined mueren gradualmente.
3-4 d después de iniciar la selección positiva (alrededor de 6 D después de la transfección), selección negativa se inicia con el fin de seleccionar sólo para los eventos de recombinación mediados por FRT. RMCE causa la pérdida de la timidina quinasa (Tk) gen suicida y por tanto la sensibilidad a la Fialuridine (FIAU). La selección negativa aplicada en este punto, en el que pequeños grupos de 2 - 4 de GFP - células / TDT + (RMCE) soncapaz de soportar concentraciones óptimas de FIAU (Figura 1B, D 6), impide la integración aleatoria, ya que en este tipo de eventos, el gen Tk en el locus AAVS1 no se ve afectado. La aparición de simultánea FRT-mediada y la integración aleatoria es muy poco probable, como se demuestra por la ausencia de eventos de integración aleatorios más tarde durante la caracterización (Figura 2B).
La GFP RMCE mixta - colonias / TDT + siga creciendo, mientras que cada vez más homogénea, de modo que para D de 9 - 10 después de la transfección, algunas colonias RMCE mixtos no totalmente seleccionados se pueden encontrar (Figura 1B). GFP - colonias / TDT + RMCE sólo están presentes cuando el donante y tanto RMCE FLPe (2: 1) se utilizan, mientras que RMCE no se produce en ausencia de FLPe (2: 0). Selección completa con UIAF hasta el día 13 - 15 después de la transfección da lugar a una GFP homogénea - / + TDT culturmi. RMCE produce una media de 12,8 ± 6,8 (n = 6) colonias Puro R / R FIAU en 15 días 13. La citometría de flujo caracterización de la línea RMCE recién generado (las colonias RMCE Puro R / R FIAU combinados en una población celular clonal) confirma que el 100% de las células presentan la RMCE GFP - / TDT + fenotipo (Figura 2A). Cuando se utilizan los donantes RMCE sin expresión constitutiva de un indicador fluorescente, la línea Puro R / R UIAF RMCE HPSC resultante es homogénea GFP -.
PCR caracterización de la línea RMCE no clonal demuestra el intercambio de casete completo (no hay rastros de la casete de la línea de células maestras pueden ser detectados) y que el programa de selección utilizado genera líneas libres de la integración aleatoria (tanto del donante RMCE y la FLPe que expresan vector) (Figura 2B). Estos resultados fueron probados aún más por sblot UR y, además, se demostró que las líneas RMCE mantienen su pluripotencia 13. Esto hace que ya no sea necesario para llevar a cabo ya sea la evaluación de todo el genoma de la integración aleatoria o la prueba de pluripotencia durante los experimentos RMCE de rutina. En resumen, el uso de este protocolo, HPSC líneas celulares transgénicas en el locus AAVS1 libre de la integración aleatoria pueden ser generados fácilmente en 15 d con una eficiencia del 100% y sin la necesidad de una amplia caracterización.

Figura 1: Descripción esquemática de RMCE (A) Línea de tiempo del programa de selección RMCE.. Izquierda: la línea de células maestro (MCL), que alberga un casete FRT-flanqueado (triángulos) que expresan GFP e higromicina-tk (ligada por 2A péptidos de autoescisión), se transfecta por nucleofection (NF) con el vector FLPe-expresar y el vector donante RMCE, whICH contiene un promotor puromicina gen de resistencia (empalme aceptor - SA- Puro R) y un casete experimental variable (X). Derecha: nueva línea RMCE (RMCEL) obtenido después de la finalización de la selección con puromicina (triángulo rojo) y la UIAF (línea azul). (B) RMCE usando un constitutiva TDT-expresión de los donantes y diferentes proporciones de ADN del donante y el ADN FLPe (0: 1, 2: 1, 2: 0) supervisado por microscopía de fluorescencia (GFP - fluorescencia verde, TDT - fluorescencia roja) a diferentes puntos de tiempo. D 2 y 6: escala barras representan 100 micras. D 10: barras de escala representan 200 micras. Por favor, haga clic aquí para ver una versión más grande de esta figura.
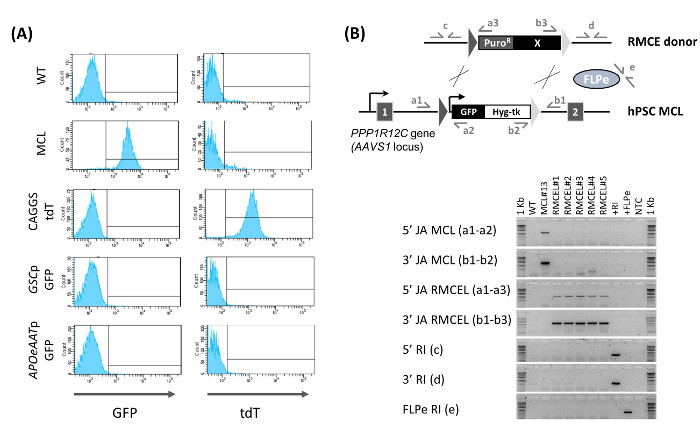
Figura 2:. Caracterización de RMCE Lines (A) Determinación de RMCE porcitometría de flujo. GFP y expresión TDT se muestra por la línea WT, la línea de células maestras (MCL), y la línea RMCE recién generado en D 15 post-nucleofection utilizando un vector constitutiva TDT-expresión (CAGGS TDT), o de los donantes con las buenas prácticas agrarias impulsadas por un promotor goosecoid (GSCP GFP, marcador de endodermo definitivo) o un promotor AAT (APOeAATp GFP, marcador de hepatocitos). (B) La confirmación de RMCE (5 '/ 3' JA RMCEL), ausencia de (MCL) casete de la línea de células maestro (5 '/ 3' JA MCL), y la falta de integración aleatoria (5 '/ 3' / FLPe RI ) por PCR utilizando los pares de cebadores representados. ADN de WT, MCL, líneas RMCE (1 - 5) se utilizaron, vector RMCE donante (+ RI), FLPe vector de expresión y control sin molde (NTC). Figura modificado a partir de (Ordovás et al., 2015) 13. Haga clic aquí para ver una versión más grande de esta figura.
dentro-page = "1"> 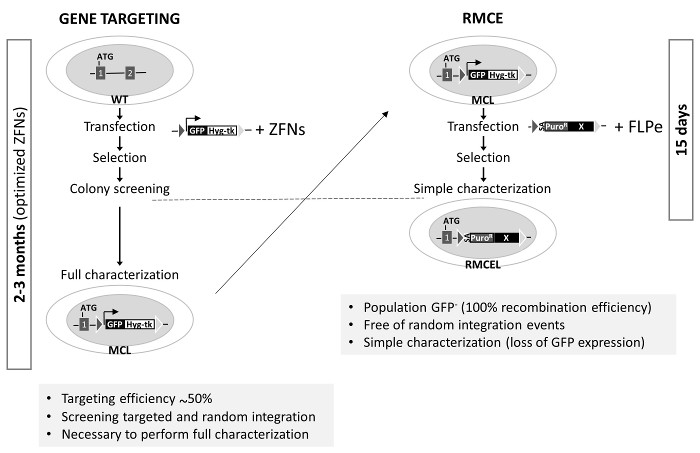
Figura 3. Generación de la línea celular de Maestro-RMCE adecuada y comparación de la orientación de genes con RMCE en AAVS1 Izquierda:. Orientación de genes utiliza células WT no modificados que son cotransfectadas con un donante (casete se describe en la Figura 1A para la generación de la línea de células maestras , MCL) y ZFNs optimizados específicos. El proceso para completar la caracterización completa puede durar hasta 3 meses. Derecha: RMCE se lleva a cabo por cotransfección del donante con vectores FLPe, y se genera una línea totalmente caracterizado 15 d. Eficiencia en la focalización de genes en AAVS1 es a partir de estudios anteriores 1,2. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Tabla 1. Composición de Medios.
| Ensayo | Adelante | Marcha atrás | amplicón | Ciclo de PCR | ||
| 5'JA MCL | CACTTTGAGCTCTACTGGCTTC | CGTTACTATGGGAACATACGTCA | 1,1 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 68 ° C (-0.5 ° C / ciclo), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ºC, 5' | ||
| 3'JA MCL | TAACTGAAACACGGAAGGAG | AAGGCAGCCTGGTAGACA | 1,4 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 68 ° C (-0.5 ° C / ciclo), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ºC, 5' | ||
| 5'JA RMCEL | CACTTTGAGCTCTACTGGCTTC | CATGTTAGAAGACTTCCTCTGC | 1,1 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 68 ºC (-0,5 ºC / ciclo), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ºC, 30 '' -72 ºC, 1 '30' 'X25] - 72 ºC, 5' | ||
| 3'JA RMCEL | TTCACTGCATTCTAGTTGTGG | AAGGCAGCCTGGTAGACA | 1,5 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 68 ° C (-0.5 ° C / ciclo), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ºC, 5' | ||
| DONANTE 5'RI | GTACTTTGGGGTTGTCCAG | TTGTAAAACGACGGCCAG | 0,5 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ºC, 5' | ||
| DONANTE 3'RI | CCTGAGTTCTAACTTTGGCTC | ACACAGGAAACAGCTATGAC | 0,5 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ºC, 5' | ||
| RI FLPe | CCTAGCTACTTTCATCAATTGTG | GTATGCTTCCTTCAGCACTAC | 0.65 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ºC, 5' | ||
Tabla 2. Los conjuntos de cebadores utilizados para la PCR genotipificación. Modificado de Ordovás et al., 2015 13.
Discusión
métodos de edición de genes en loci de puerto seguro siguen siendo una herramienta esencial para el desarrollo de la transgénesis en hPSCs. Aunque el carácter de puerto seguro de AAVS1 recientemente ha sido cuestionada por algunas aplicaciones 13,18, este lugar sigue siendo actualmente el mejor caracterizado en líneas celulares derivadas de seres humanos. La conciencia de sus limitaciones en hPSCs puede ayudar a obtener datos fiables. Por lo tanto, AAVS1 todavía se espera que sea un sitio útil, por ejemplo, para estudios de ganancia y la pérdida de función o expresión inducible / constitutiva de factores que requieren un contexto isogénica dentro y entre los fondos genéticos determinados.
El locus AAVS1 ha sido el objetivo de muchos grupos que utilizan ZFNs, TALEN o CRISPR / Cas9 1,2,19. Estas nucleasas aumentan significativamente la eficacia de la recombinación homóloga en un locus determinado. Sin embargo, el proceso para seleccionar y caracterizar completamente clones correctamente orientados, de forma aleatoria integración y para mantener la pluripotencia y la integridad del genoma puede tardar hasta 3 meses (utilizando herramientas de edición de genes optimizados) (Figura 3). Los dos últimos pueden ser causados por posibles mutaciones generadas por la actividad nucleasa desviado. El sistema RMCE utilizado aquí, sin embargo, ofrece un método rápido, eficiente y elegante para lograr este objetivo en sólo dos semanas, una vez que las secuencias diana-recombinasa específica se preintegradas en el locus AAVS1. El uso de la selección positiva / negativa y un programa de selección apropiada son los principales factores que contribuyen a la simplificación de la edición de genes en el locus AAVS1 por RMCE.
caracterización genotípica de las nuevas líneas transgénicas se reduce de manera significativa (sin detección clonal es necesario), y la caracterización asociada a la actividad nucleasa fuera del objetivo se hace prescindible debido a la especificidad de FLPe para frs. La caracterización también se puede reducir en los experimentos de rutina RMCE demostrando lapérdida completa de la expresión de GFP desde la línea de células maestras (Figura 2A), ya que el intercambio de casete completa y la falta de integración aleatoria ya han sido suficientemente demostrado por PCR (Figura 2B) y Southern blot 13. El uso de la selección positiva / negativa también constituye la principal diferencia con los informes anteriores. Varios grupos que anteriormente descritos RMCE en hPSCs, ya sea en el AAVS1 o otros loci, emplean sólo una única etapa de selección positiva 7,14,16. Esto no constituye una ventaja técnica importante sobre la edición gen realizado con nucleasas, porque la detección de colonias tiene que ser realizado igualmente con el fin de demostrar la integración y la ausencia de integración aleatoria correcta a nivel clonal.
El uso de este sistema de RMCE en múltiples líneas / IPSC de células madre requiere pre-integración del casete descrito contiene FRT en el locus AAVS1 de cada línea independiente. Sin embargo, una vez generado,su rapidez y simplicidad hace que sea posible el desarrollo de las pantallas de genética semi-alto rendimiento en los entornos isogénicas definidas para las aplicaciones mencionadas anteriormente que de otro modo sería técnicamente mucho tiempo. Además, todos los vectores RMCE se construyen dentro del vector gen de la orientación pZ AAVS1 utilizado para dirigir el locus con ZFNs, pero también se informó de Talens o CRISPR / Cas9. dualidad del vector RMCE es altamente útil para generar varias líneas para un transgén determinado en hPSCs con o sin FRT. Por ejemplo, un hit demostró con éxito en una pantalla genética determinada llevada a cabo por RMCE lo ideal sería que tenga que ser probado en múltiples líneas HPSC para confirmar los resultados. En ausencia de varias líneas de células primarios RMCE-adecuado, gen objetivo directo de la exitosa usando las nucleasas se pudo realizar. La utilidad del carácter dual de los vectores RMCE ha sido probado por nuestro grupo 20. En este estudio, la corrección de genes se realizó en derivados de pacienteLa demencia frontotemporal (DFT) iPSCs que llevan una mutación que causa progranulin (PGRN) deficiencia de expresión. Gen de complementación mediante la inserción ZFNs mediada en el locus AAVS1 de un casete que restauró los niveles de PGRN, corrigió el fenotipo defectuoso en corticogenesis asociado con la presencia de la mutación. Además, una línea comparable fue generada por RMCE en hESCs WT para ser utilizado como control para la expresión PGRN exógeno.
En ausencia de colonias recombinantes, comprobar la eficacia de transfección del vector donante RMCE. Las eficacias de transfección superiores a 30% de rendimiento los resultados indicados anteriormente. Una menor eficiencia de transfección disminuyen la eficiencia de recombinación. Finalmente, el sistema RMCE se ha generado en el locus AAVS1, pero es aplicable a cualquier otro locus.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
L.O. was funded by IWT/OZM/090838, IACS BPAMER3/08/04, and the Government of Aragon FMI048/08; RB by IWT fellowship SB-121396; M.P. by FWO 1288714N; and R.S. by the Dutch Diabetes Foundation. N.H., J.V., K.C., and Q.C. were supported by IWT fellowships SB-121396, SB-101230, SB-91228, and SB-093228, respectively. Funding to C.M.V. was from FWO G.0667.07, G.0975.11, and KU Leuven (EIW-B4855-EF/05/11, ETH-C1900-PF, EME-C2161-GOA/11/012), IWT-HEPSTEM, BELSPO-IUAP-DEVREPAIR, FP7-HEMIBIO (266777).
Materiales
| Name | Company | Catalog Number | Comments |
| DR4 Mouse Embryonic Fibroblasts (MEF) | AMS bio | GSC-6204G | hPSC culture on feeers |
| CF-1 MEF, mitomycin-C treated | AMS Bio | GSC-6001M | |
| EmbryoMax 0.1% Gelatin Solution | Millipore | ES-006-B | iMEF plating |
| DMEM/F-12, HEPES | Gibco | 31330-038 | |
| KnockOut Serum Replacement | Gibco | 10828-028 | |
| L-Glutamine | Sigma Aldrich | G8540 | For hESC medium |
| L-Glutamine 200 mM | Gibco | 25030-081 | For iMEF medium |
| 2-Mercaptoethanol 50 mM | Gibco | 31350-010 | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140-035 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| Human basic FGF | Peprotech | 100-18C | |
| Y-27632 in Solution | VWR | 688001-500 | |
| DMEM High Glucose | Gibco | 41965-039 | |
| Fetal Bovine Serum | Sigma Aldrich | F7524 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300-054 | hPSC culture on feeders |
| Matrigel hESC qualified | VWR | 734-1440 | |
| mTeSR1 complete medium kit | Stem cell technologies | 05850 | For Feeder free culture |
| StemPro Accutase Cell Dissociation Reagent | Gibco | A1110501 | For feeder free culture |
| Y-27632 in solution | VWR | 56726 | |
| Human Stem Cell Nucleofector Kit 2 | Lonza | VVPH-5022 | |
| Puromycine Dihydrochloride | Gibco | A11138-03 | |
| FIAU (Fialuridine) | ABX | 2910 | |
| Hygromycin B | Sigma Aldrich | H3274 | |
| pFLPe | Modified to from pCAGGS-FLPe (MES4488, Open Biosystems) to remove puromycin | ||
| RMCE donors | - | - | Modified from pZ donor AAVS1 puromycin vector (PZD0020-1KT, Sigma Aldrich)13 |
| 5mL Round Bottom Polystyrene Test Tube, with Cell Strainer | Falcon | 352235 | |
| PureLink Genomic DNA kit | Invitrogen | K182001 | |
| GoTaq Flexi DNA Polymerase | Promega | M8298 | |
| Agarose | Sigma | A9539 | |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | |
| Nucleofector 2b Device | Lonza (Amaxa) | AAB-1001 | |
| NucleoCounter NC-100 | Chemometec | NC-100 | |
| BD FACS Canto | BD Biosciences | 337175 | |
| NucleoCassette | Chemometec | 941-0002 |
Referencias
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat. Biotechnol. 29 (8), 731-734 (2011).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat. Biotechnol. 27 (9), 851-857 (2009).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Baer, A., Bode, J. Coping with kinetic and thermodynamic barriers: RMCE, an efficient strategy for the targeted integration of transgenes. Curr. Opin. Biotechnol. 12 (5), 473-480 (2001).
- Sakurai, K., et al. Efficient integration of transgenes into a defined locus in human embryonic stem cells. Nucleic Acids Res. 38 (7), e96 (2010).
- Irion, S., et al. Identification and targeting of the ROSA26 locus in human embryonic stem cells. Nat. Biotechnol. 25 (12), 1477-1482 (2007).
- Di Domenico, A. I., Christodoulou, I., Pells, S. C., McWhir, J., Thomson, A. J. Sequential genetic modification of the hprt locus in human ESCs combining gene targeting and recombinase-mediated cassette exchange. Cloning Stem Cells. 10 (2), 217-230 (2008).
- Lombardo, A., et al. Site-specific integration and tailoring of cassette design for sustainable gene transfer. Nat. Methods. 8 (10), 861-869 (2011).
- Lombardo, A., et al. Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery. Nat. Biotechnol. 25 (11), 1298-1306 (2007).
- Simth, J. R., et al. Robust , Persistent Transgene Expression in Human Embryonic Stem Cells Is Achieved with AAVS1-Targeted Integration. Stem Cells. 26, 496-504 (2008).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Qian, K., et al. A simple and efficient system for regulating gene expression in human pluripotent stem cells and derivatives. Stem Cells. 32 (5), 1230-1238 (2014).
- Ordovás, L., et al. Efficient Recombinase-Mediated Cassette Exchange in hPSCs to Study the Hepatocyte Lineage Reveals AAVS1 Locus-Mediated Transgene Inhibition. Stem Cell Reports. 5 (5), 918-931 (2015).
- Du, Z. -. W., Hu, B. -. Y., Ayala, M., Sauer, B., Zhang, S. -. C. Cre recombination-mediated cassette exchange for building versatile transgenic human Embryonic Stem Cells lines. Stem Cells. 27, 1032-1041 (2009).
- Papapetrou, E. P., et al. Genomic safe harbors permit high β-globin transgene expression in thalassemia induced pluripotent stem cells. Nat. Biotechnol. 29 (1), 73-78 (2011).
- Ramachandra, C. J., et al. Efficient recombinase-mediated cassette exchange at the AAVS1 locus in human embryonic stem cells using baculoviral vectors. Nucleic Acids Res. 39 (16), e107 (2011).
- Takata, Y., Kondo, S., Goda, N., Kanegae, Y., Saito, I. Comparison of efficiency between FLPe and Cre for recombinase-mediated cassette exchange in vitro and in adenovirus vector production. Genes to Cells. 16 (7), 765-777 (2011).
- Mizutani, T., Li, R., Haga, H., Kawabata, K. Transgene integration into the human AAVS1 locus enhances myosin II-dependent contractile force by reducing expression of myosin binding subunit 85. Biochem. Biophys. Res. Commun. 465 (2), 270-274 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Raitano, S., et al. Restoration of progranulin expression rescues cortical neuron generation in an induced pluripotent stem cell model of frontotemporal dementia. Stem Cell Reports. 4 (1), 16-24 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados