Method Article
Eine schnelle und effiziente Erzeugung von rekombinanten humanen pluripotenten Stammzellen durch Rekombinase-vermittelten Kassettenaustausch in der
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier berichten wir eine schnelle und effiziente Gen Editierverfahren basierend auf RMCE im AAVS1 Locus der menschlichen pluripotenten Stammzellen (hPSCs), die auf die zuvor beschriebenen Systeme verbessert. Mit dieser Technik können isogene Linien schnell und zuverlässig für die richtige Vergleichsstudien erzeugt, erleichtert Transgenität vermittelte Forschung mit hPSCs.
Zusammenfassung
Auch bei der Revolution von Gen-Targeting - Technologien geführt von CRISPR-Cas9, der genetischen Veränderung von menschlichen pluripotenten Stammzellen (hPSCs) ist immer noch zeitaufwendig. Vergleichende Studien, die rekombinante Linien mit Transgene integriert in den sicheren Hafen loci nutzen könnten von Ansätzen profitieren, die ortsspezifische gezielte Rekombinasen verwenden, wie Cre oder FLPe, die schneller sind und weniger anfällig für Off-Target-Effekte. Derartige Verfahren sind beschrieben worden, obwohl sie in den meisten Aspekten Targeting nicht wesentlich übertreffen Gen tun. Mit Zink-Finger - Nukleasen, wir zuvor erstellt eine Master - Zelllinie in AAVS1 Locus von hPSCs , die ein GFP-Hygromycin-tk enthält Kassette exprimieren, flankiert von heterotypic FRT - Sequenzen. Hier beschreiben wir die Verfahren FLPe Rekombinase-vermittelten Kassettenaustausch (RMCE) mit dieser Linie durchzuführen. Der Master-cell Leitung ist mit einem RMCE Donor-Vektor transfiziert, der ein promotor Puromycin-Resistenz enthält und mit FLPe Rekombinase. Die Anwendung sowohl eine positive (Puromycin) und negativen (FIAU) Auswahlprogramm führt zur Auswahl von RMCE ohne Zufallsintegrationen. RMCE erzeugt vollständig charakterisiert pluripotenten polyklonalen transgenen Linien in 15 d mit 100% Wirkungsgrad. Trotz der kürzlich Einschränkungen des AAVS1 Locus beschrieben, ebnet die Leichtigkeit des Systems den Weg für HPSC Transgene in isogenen Einstellungen ist, die für vergleichende Studien und ermöglicht semi-Hochdurchsatz - genetischen Screens für Gewinn / Verlust der Funktionsanalyse, die sonst als sehr zeitaufwendig.
Einleitung
Gezielte Genom Rekombination hat in vielen Bereichen der Forschung rasche Fortschritte erleichtert. Bei Mäusen hat sich für ein besseres Verständnis des komplexen Mechanismus der Genfunktion und Genregulation erlaubt die Synergie zwischen Genom-Bearbeitung und Stammzellforschung. Dieser Fortschritt ist in der humanen pluripotenten Stammzellen (hPSCs) als auch zu erwarten, obwohl seit vielen Jahren seit der ersten Isolierung von humanen embryonalen Stammzellen (hES-Zellen), und später menschlichen induzierten pluripotenten Stammzellen (hiPSCs), Gen-Bearbeitung eine technische Hürde gebildet hat . Jüngste Fortschritte in der Genomtechnik durch gezielte Nukleasen-Zink-Finger-Nukleasen (ZFNs), Transkriptionsaktivator- artigen Effektor Nukleasen (TALEN) oder Clustered regelmäßig voneinander beabstandete kurze Palindrom Wiederholungen / CRISPR-assoziierten Protein 9 (CRISPR / Cas9) haben uns erlaubt, diese zu überwinden 3 - Schwierigkeiten, Rekombination , ein effizientes Verfahren 1 gezielt zu machen.
Spezifische Loci in der mouse Genom, mit denen stabile, zuverlässige und allgegenwärtige Transgen - Expression in Abwesenheit von Nebenwirkungen wie Rosa26, HPRT1 oder Col1A1 haben wesentlich geworden Werkzeuge bei der Durchführung von genetischen Untersuchungen. Sicherer Hafen loci ermöglichen vergleichbare Analyse zwischen verschiedenen Linien von Mäusen in isogenen Kontexten. Das kann nicht mit Standard-Zufallsintegrationsverfahren erreicht, die mit den bekannten Einschränkungen wie Insertionsmutagenese, dosisabhängige Effekte, oder bunten Transgen-Expression assoziiert sind. Gene Bearbeitung einfach und flexibel in sicheren Hafen loci wird durch die Verwendung von ortsspezifische gezielte Rekombinasen wie Cre oder Flippase (FLPe) erhöht, die spezifisch Zielsequenzen (loxP oder FRT, jeweils) zu erkennen und effiziente Rekombination zwischen identischen Zielen katalysieren. Aufgrund dieser Eigenschaften ist Rekombinase-vermittelte Gen Bearbeitung mit loxP oder FRT-Sequenzen in vorintegrierten sicheren Hafen Loci ein übliches Werkzeug in der Maus verwendet transGenesis. Zusätzlich zur Kassetteneinschub- oder zwischen identischen Zielsequenzen vermittelte Exzision gestattet die Verwendung von inkompatiblen loxP oder FRT - Stellen für die Rekombinase-vermittelten Kassettenaustausch (RMCE) 4.
In der menschlichen Versuche, sicheren Hafen Loci durchgeführt wurden zu identifizieren. Die Maus orthologous HPRT trotz seiner Verbindung mit dem Verlust der Funktion Lesch-Nyhan - Syndrom und ROSA26 haben in hPSCs gezielt worden. HPRT schnell Transgen - Expression berichtet wurde ESC zum Schweigen zu bringen und, wie ROSA26, seine Fähigkeit , die Transgen - Expression zu erhalten in 7 - terminal differenzierten Zellen wurde nicht 5 untersucht. Da eine homozygote Nullmutation des CCR5 - Gens beim Menschen gut verträglich zu sein scheint, wurde sein Wert als sicheren Hafen beurteilt. CCR5 wurde gezielt und berichtet stabile Transgen - Expression in verschiedenen menschlichen Zelllinien zu erhalten, einschließlich der WSR 8,9. Aber,letzterer wurde nicht auf der klonalen Ebene während einer Langzeitkultur erwiesen und ubiquitäre Transgen - Expression wurde nicht in differenzierte Nachkommen der drei Keimschichten. AAVS1, die natürliche Integrationsstelle des Adeno Associated Virus Typ 2 (AAV), wurde gezeigt , getestet in hESCs als auch, aufgrund von Berichten über Widerstand gegen Transgen - Silencing 10. Später viele Gruppen verwendet , um die AAVS1 Locus und beschrieben stabile transgene Expression in undifferenzierten hPSCs sowie in ihren differenzierten Nachkommen aller drei Keimschichten, sowohl in vitro als auch in vivo 2,8,11,12. Die jüngsten Ergebnisse Nuance dennoch diese Ergebnisse, da der AAVS1 Locus gefunden wurde in vitro in undifferenzierten hES und in Hepatozyten Nachkommen 13 variable Transgen Hemmung auszuüben.
Weitere Screening-Studien zufällige Integration Ansätze und Methoden unter Verwendung von Einzelkopie-Integration zu bestimmen, richtet geno zu findenmic Integrationsstellen resistent gegen Transgen - Silencing bei hPSCs Expansion und Differenzierung 14,15. Insgesamt bisher wurde kein Genomstelle vollständig als sicherer Hafen in hPSCs und ihre Nachkommen validiert; Identifizierung einer geeigneten Stelle für die ubiquitäre stabilen Transgen - Expression, nicht nur in hPSCs sondern auch in ihren differenzierten Nachkommen in vitro und in vivo bleibt, gelöst werden. Unter allen untersuchten Loci und trotz ihrer Grenzen bleibt AAVS1 die am besten charakterisierte und am häufigsten verwendeten in der Stammzellenforschung.
RMCE wurde in hPSCs in einigen dieser Loci 6,7,14,16 unter Verwendung meist Cre - Rekombinase erfolgreich durchgeführt, auch wenn es Anzeichen dafür gibt , dass FLPe effizienter als Cre 17 ist. In all diesen Fällen wird eine positive Arzneimittelresistenzkassette wurde für die Selektion von rekombinanten Kolonien verwendet. Obwohl erfolgreich, sie bilden diese Verfahren keinen technischen Fortschritt gegenüber Standard-Gen-Bearbeitungsverfahren ZFNs, Talens oder CRISPR / Cas9, als ein einziges Antibiotikum Auswahlverfahren schließt nicht aus, zufällige Integrationen und erfordert Kolonie-Screening korrekt zu identifizieren, gezielt Klone verwenden.
In diesem Verfahren beschreiben wir Verfahren RMCE in hPSCs in der AAVS1 Locus unter Verwendung einer Kombination von positiv (innerhalb der ankommenden Kassette) und negativen (innerhalb der vorintegrierten Kassette) Selektionen durchzuführen , die für die Erzeugung von polyklonalen transgenen Linien in ± 15 Tage ermöglichen , mit 100% Effizienz und frei von zufälligen Integration Veranstaltungen. Daher stellt diese Methode Fortschritt gegenüber gegenwärtig beschriebenen RMCE Technologien in hPSCs.
Protokoll
1. Herstellung der FRT-enthaltenden HPSC Master-Zelllinie für die Transfection
- Kultur hESC / iPSC Master - Zelllinien unter Standardverfahren an oder aus inaktivierten embryonalen Maus - Fibroblasten (Imef) unter Verwendung von hESC oder Feeder frei iPSC Medium (Tabelle 1). Bereiten Sie 2 (auf Zubringer) oder 1 (Feeder frei) Vertiefungen hPSCs in einer 6-Well-Platte für jeden experimentellen Zustand.
- Beachten Sie die Kulturen, und wenn die Zellen erreichen 60 bis 70% Konfluenz, Platte arzneimittelresistente Imef (iDR4). Kurz gesagt, beschichten die erforderlichen Vertiefungen einer 12-Well-Platte mit 0,5 ml 0,1% igen Gelatinelösung und 5 min bei RT inkubiert. Platte 125.000 iDR4s pro Vertiefung und Inkubation O / N mit Imef Medium (Tabelle 1).
2. HPSC Transfektion von Nucleofection
- Am folgenden Tag HES / Ik Kulturen mit frischem Medium vorge inkubieren, einschließlich 10 & mgr; M Rho-associated protein kinase (ROCK) -Inhibitor (Y-27632), für 1 h bei 37 ° C. Next, nehmen hESC Transfektion Lösung aus dem Kühlschrank vorab auf Raumtemperatur erwärmt.
- Bereiten Sie 1 ml Nukleofektion Plating Medium (Tabelle 1) pro Nukleofektion Zustand. Nehmen Sie Imef Medium aus den Vertiefungen, Waschen mit 1 ml Raumtemperatur PBS und fügen 500 ul Nukleofektion Plating Medium. Übertragen Sie die anderen 500 ul in sterile 1,5 ml-Röhrchen und lagern Sie sie bei 37 ° C bis zur Beschichtung. HINWEIS: Ein typisches Experiment RMCE enthält drei verschiedenen Bedingungen mit molekularem Verhältnisse von RMCE Donor: pFLPe Vektoren von 2: 0, 2: 1 und 0: 1.
- Nehmen Sie hESC / Ik in einer einzigen Zellsuspension.
- Nehmen Sie die Medien aus, waschen Sie es mit 2 ml RT PBS und 1 mL Trypsin 0,05% oder Accutase auf den Zubringer oder Feeder-frei HPSC Kulturen sind. Inkubieren 7 (Trypsin) oder und 2 bis 5 (Accutase) min bei 37 ° C. Für die Accutase Behandlung - Zellen müssen locker bleiben, aber angebracht.
- Entfernen Sie vorsichtig die Platte aus dem Inkubator und ohne einllowing die Zellen zu lösen, entfernen Sie den Dissoziierungsmittels durch Aspiration. 1 mL hESC Medien und distanzieren die losen Zellen durch sanft die Medien auf der Plattenoberfläche Spülung, die Vermeidung Schäumen. Stellen Sie sicher, dass die Zellen in einer einzigen Zellsuspension sind.
- Sammeln Sie die Zellen in einem sauberen und sterilen 15 oder 50 ml-Röhrchen. Waschen der Vertiefungen mit 1 ml HES-Medien und die Zellen in der gleichen 15 oder 50 ml-Röhrchen sammeln. Nehmen ein 50 ul Aliquot (im nächsten Schritt) zum Zählen mit einem Zell Zählvorrichtung. Beachten Sie die Lautstärke der Sammlung nach unten und berechnen Gesamtzellzahl. Spin down die Zellen bei 300 g für 5 min bei RT.
- Resuspendieren des Zellpellets zu einer Suspension von 10 6 Zellen / ml mit PBS wurde ein 5 ml serologische Pipette und vorsichtiges Pipettieren verwendet. Schaumbildung vermeiden. Übertragung von 2 ml pro Versuchsbedingung (etwa 2 x 10 6 Zellen) zu reinigen, sterile 15 ml - Röhrchen.
- Während der Zentrifugation vorpare die RMCE Spender- pFLPe Plasmide in einem maximalen Volumen von 10 ul vermischt.
- Übertragen 2,5 ug pFLPe (6,5 Kb) in einen sauberen, sterilen 1,5 - ml - Röhrchen. Eine 2: 1 - Molekülverhältnis des RMCE Donorvektor 13 zur pFLPe Vektor. Beispielsweise etwa 10 & mgr; g eines 12 Kb RMCE Donorvektor.
- Fahren Sie mit Nukleofektion, die Vermeidung in allen Schritten Schäumen.
HINWEIS: Einige HPSC Transfektionsprotokolle gelten eine zusätzliche Imef Abreicherungsschritt vor dem Schritt 2.4. Doch in der Praxis ist Imef eine kleine Zellfraktion in der Einzel Suspension, die nicht wesentlich Transfektionseffizienz verändern, noch wird es verunreinigen rekombinanten Zellen schaffen, da sie nicht-proliferative. Darüber hinaus ist es wichtig, schnell während des Transfektionsprozess fortfahren Lebensfähigkeit zu erhöhen. Aus diesen Gründen wird Imef Erschöpfung nicht angewendet.- Entfernen Sie die iDR4 Platte und die 1,5-ml-Röhrchen mit dem Plattierungsmediumaus dem Inkubator. Ein Rohr zu einer Zeit, die pipettieren PBS sorgfältig ohne Störung der Zellpellet ab. Zu entfernen, so viel wie möglich. Das Pellet mit 100 & mgr; l der Transfektion Lösung durch vorsichtiges Pipettieren. Übertragen Sie die Zellsuspension auf die 1,5 ml-Röhrchen, die Plasmid-Mischungen enthalten, und vorsichtig mischen.
- Übertragen Sie die Zell-DNA-Gemisch in die Transfektor Küvette, die Aufmerksamkeit nicht Blasen einzuführen. Führen Sie die Küvette in den Nucleofector Gerät und Anwendung Programm A13 (Zellen, die auf Zubringer) oder F16 (Feeder-frei).
Hinweis: andere Transfektionsverfahren oder Nucleofector Geräte können Optimierung dieser Programme oder Transfektion Bedingungen erfordern. - Erneute Teilnahme an der Küvette und unter Verwendung der Einweg-Transferpipetten in der Nukleofektion-Kit, sammeln ihr Inhalt durch die Anwendung von 0,5 ml des Beschichtungsmedium und Absaugen ganze Volumen. Führen Sie diesen Schritt schnell, aber sanft und in einer Bewegung.
- Platte tropfenweise in 12-well Platte die iDR4 und 500 & mgr; l Plating Medium enthält. Wiederholen Sie die Schritte 2.6.1 - 2.6.4 für die nächste Versuchsbedingung.
- Legen Sie die Kulturschale auf einem Regal im Inkubator und bewegen Sie ihn langsam hin und her und von Seite zu Seite zu gleichmäßig die Zellsuspension zu verteilen. Inkubieren für 24 h vor der nächsten Medienwechsel, damit die Zellen in Gegenwart von 10 uM ROCK-Inhibitor (Y-27632) zu gewinnen.
3. Positive und Negative Selektion von Zellen, die eine RMCE
- Ändern Kulturmedien täglich (1 ml / Well), und 2 - 3 d nach der Transfektion starten Auswahl mit 100 ng / ml Puromycin.
HINWEIS: Optimale Konzentrationen von positiven und negativen Selektionsreagenzien bestimmt werden müssen experimentell für jede neu HPSC Master-Zelllinie erzeugt. Führen Sie einen Kill - Kurve 13, die Master - Zelllinie mit der niedrigen Dosis zu bestimmen (Konzentration , bei der minimale visuelle Toxizität nach einer scheinbaren 7d der Auswahl, aber die Kultur ist nicht bewachsen), optimale Dosis (die niedrigste Konzentration, bei der alle Zellen tot sind nach 7 d Selektions) und hohe Dosis (Konzentration, die offensichtlich Toxizität verursacht, tötete alle Zellen nach 2 - 3 d) für beide Puromycin und Fialuridine (FIAU).- Saugen Sie das HES-Medium. Waschen mit 1 ml PBS. In hES-Medium mit 100 ng / ml Puromycin.
- Beachten Sie das Zellwachstum, so dass der Tod und das Wachstum ausgeglichen sind. Ändern Medien mit Puromycin täglich nach Schritt 3.1.1. 50 ng / ml bis zu einem Maximum von 250 ng / mL - wenn Wachstumsrate Zelltod einholt, Puromycin Konzentration in Blöcken von 25 erhöhen. Weiter Puromycin-Selektion für 5 bis 7 d.
- 3 bis 4 d nach Puromycin-Selektion starten, um den Tag 6 nach der Transfektion starten Auswahl mit 0,5 uM FIAU. Ändern Sie den Medien täglich und halten FIAU nicht länger als 7 Tage kontinuierlich.
4. Erweiterung und Charakterisierung von RMCE Linien
- Nach Abschluss der Auswahl, um D14 - 15, sind beständig RMCE Kolonien vorhanden und die neue RMCE Linie bilden. Split in loser Schüttung in einem Verhältnis 1: 2 nach Standardverfahren (Passage 1, P1).
- Plate 2 Vertiefungen einer 12-Well-Platte (ein oder aus Speiser, abhängig von den regulären Kulturbedingungen der ursprünglichen Master-Zelllinie). Verwenden Sie eine gut für den weiteren Ausbau, Lagerung oder Versuchsaufbau (keine zusätzliche Beschreibung dieser Verfahren in diesem Protokoll durchgeführt wird), und die andere für die Charakterisierung.
- Wenn Zellen von P1 bereit sind zu teilen, trennen die gut für die Charakterisierung in einer einzigen Zellsuspension, wie in 2.4 und sammeln die Zellen in einem 15 ml-Röhrchen beschrieben. HINWEIS: Verwenden Sie Zellen aus dem HPSC WT (keine genetische Modifikation) und Master-Zelllinie als negative und positive Kontrollen, jeweils für die Charakterisierung.
- Spin down bei 300 g für 5 min bei RT. Resuspendieren in 1 ml PBS und spaltete die Probe in zwei für die Durchflusszytometrieund DNA - Analyse 13.
- Durchflusszytometrie - Analyse 13 (300 & mgr; l):
- Hinzufügen fötalem Kälberserum zu den Zellen in PBS bis zu einer Endkonzentration von 5%. Übertragen Sie die Zellen in einen sauberen FACS-Röhrchen mit einer Zelle Sieb und halten sie auf dem Eis.
HINWEIS: Die Lebensfähigkeit der Zellen wird unter diesen Bedingungen hoch ist , so ist es möglich, aber nicht notwendig, eine Fluoreszenzfarbstoff für die nicht lebensfähigen Zellen zu verwenden (beispielsweise 7-AAD oder PI). - Gehen Sie sofort auf die Analyse in einem Durchflusszytometer Zellanalysator und aufzeichnen 20.000 - 30.000 Veranstaltungen. Zur Analyse setzen die Tore über GFP oder TDT Hintergrund florescence die WT negativen Kontrollprobe verwendet wird.
- Hinzufügen fötalem Kälberserum zu den Zellen in PBS bis zu einer Endkonzentration von 5%. Übertragen Sie die Zellen in einen sauberen FACS-Röhrchen mit einer Zelle Sieb und halten sie auf dem Eis.
- DNA - Analyse 13 (700 & mgr; l):
- Übertragung der Zellen auf ein sauberes 1,5-ml-Röhrchen. Spin down bei 300 g für 5 min bei RT und den Überstand aspirieren. Gehen Sie zur DNA-Extraktion eines kommerziellen Kits und den Anweisungen des Herstellers folgen. Lagern Sie das trockene Pellet bei -20 ° C für later Analyse.
- Messen Sie DNA-Konzentration.
- Bereiten Standard - PCR - Reaktionen entsprechend den Bedingungen in 3 und Tabelle 2 13 Run PCR - Proben auf einem 2% Agarosegel , enthaltend 1x DNA - Gel - Färbung bei 150 V für 15 - 25 Min . . Analysieren Sie den Lauf in einem Gel-Analysator.
Ergebnisse
Mit AAVS1 -spezifische ZNFs, vollständig charakterisiert hESC / iPSC Master - Zelllinien heterotypic FRT - Zielsequenzen enthalten , wurden erzeugt und 13 beschrieben. Die FRT haltigen Master - Zelllinien aufrechterhalten Pluripotenz und Genomintegrität nach der Behandlung ZFN und exprimiert stabil GFP in vitro und in vivo. RMCE wird durch Co - Transfektion der Stammzelllinien mit FLPe Rekombinase und RMCE Spendervektoren (1A) durchgeführt. RMCE Vektoren enthalten identischen FRT - Sequenzen zu den in der AAVS1 Stammzelllinie flankieren das Transgen 1B überwacht RMCE die konstitutiv exprimierenden TDT pZ verwendet:.. F3-P CAGGS tdTPH-F Nach der Transfektion HPSC bilden kleine Gruppen von gleichmäßig verteilten Zellen über die iDR4 MEF Schicht und transfiziert HPSC exprimieren sowohl GFP als auch transient TdT (1B, D 2). Die Zellen werden für 2 erholen - 3d nach der Transfektion vor der Auswahl beginnen. Positive Selektion gestartet wird zunächst eine niedrige Dosis von Puromycin, um unter Verwendung von RMCE Spender Insertion zu begünstigen. Puromycin wird sanft am Anfang angewendet, weil anfängliche massiven Zelltod zu einzelnen Zellen, die eine Rekombination führen könnten, so dass sie nicht in der Lage, den Prozess zu überleben. In den folgenden Tagen muss Puromycin Konzentration schrittweise erhöht bis die optimale Dosis, um die rekombinanten Zellen zu ermöglichen, werden in kleine Kolonien zu wachsen, während die nonrecombined Zellen allmählich sterben.
3 bis 4 d nach positiver Selektion Einleitung (etwa D 6 nach der Transfektion), negative Selektion ist zu wählen, um nur für FRT-vermittelte Rekombinationsereignisse begonnen. RMCE verursacht den Verlust der Thymidinkinase (Tk) Suizidgen und somit die Empfindlichkeit auf Fialuridine (FIAU). Negative Auswahl an dieser Stelle angelegt, in denen kleine Gruppen von 2 bis 4 GFP - / TDT + (RMCE) Zellen sindkönnen optimale Konzentrationen von FIAU (1B, D 6), verhindert, zufällige Integration zu widerstehen , da in solchen Fällen nicht beeinflußt das TK - Gen in dem Locus AAVS1 bleibt. Auftreten von gleichzeitigen FRT-vermittelten und zufällige Integration ist sehr unwahrscheinlich, da durch das Fehlen von zufälligen Integrationsereignisse später während der Charakterisierung (2B) gezeigt.
Die gemischte RMCE GFP - / TDT + Kolonien weiter wachsen , während homogenere werden, so daß von D von 9 bis 10 nach Transfektion einige nicht vollständig ausgewählten gemischten RMCE Kolonien (1B) befindet. GFP - / TDT + RMCE Kolonien sind nur vorhanden , wenn die beiden RMCE Donor- und FLPe (2: 1) verwendet werden, während RMCE in Abwesenheit von FLPe tritt nicht auf (2: 0). Volle Auswahl mit FIAU bis Tag 13. - 15. nach der Transfektion führt zu einer homogenen GFP - / TDT + culture. RMCE ergibt einen Durchschnitt von 12,8 ± 6,8 (n = 6) Puro R / R FIAU Kolonien in 15 Tagen 13. Durchflusszytometrie Charakterisierung der neu erzeugten RMCE Linie (die Puro R / R FIAU RMCE Kolonien in einer Zellpopulation kombiniert nonclonal) bestätigt , dass 100% der Zellen präsentieren den RMCE GFP - / TDT + Phänotyp (2A). Wenn RMCE Donatoren ohne konstitutive Expression eines fluoreszierenden Reporter verwendet werden, ist die resultierende Puro R / R FIAU RMCE HPSC Linie homogen GFP -.
PCR-Charakterisierung der nonclonal RMCE Linie zeigt den vollständigen Kassettenaustausch (keine Spuren der Kassette der Master-Zelllinie nachgewiesen werden kann) und dass die Auswahl verwendete Programm generiert zufällige Integration freien Leitungen (sowohl des RMCE Spender und FLPe-exprimierenden Vektor) (2B). Diese Ergebnisse wurden durch die bewährteouthern Blot und zusätzlich haben wir gezeigt , dass die RMCE Linien bleiben pluripotent 13. Dadurch ist es nicht mehr erforderlich, entweder die genomweite Auswertung von Zufallsintegration oder die Pluripotenz Test während der Routine RMCE Experimente durchzuführen. Zusammengefasst dieses Protokoll verwenden, frei von zufälligen Integration HPSC transgenen Zelllinien in der AAVS1 Locus leicht in 15 d mit 100% Effizienz und ohne die Notwendigkeit von umfangreichen Charakterisierung erzeugt werden kann.

Abbildung 1: Schematische Übersicht über RMCE (A) Zeitleiste des RMCE Auswahlprogramm.. Links: die Master-Zelllinie (MCL), eine FRT-flankierten Kassette beherbergt (Dreiecke), die GFP und Hygromycin-tk (von 2A Selbst spalten Peptide gebunden) exprimieren, transfiziert wird durch Nukleofektion (NF) mit dem FLPe-exprimierenden Vektor und der RMCE Spender Vektor, which enthält ein promotor Puromycin - Resistenzgen (Splicing Akzeptor - SA- Puro R) und eine variable experimentellen Kassette (X). Rechts: neue RMCE Linie (RMCEL) nach Abschluss der Selektion erhalten mit Puromycin (rotes Dreieck) und FIAU (blaue Linie). (B) RMCE unter Verwendung eines konstitutiven TDT-exprimierenden Donor- und unterschiedliche Verhältnisse von Donor - DNA und FLPe DNA (0: 1, 2: 1, 2: 0) , überwacht durch Fluoreszenzmikroskopie (GFP - grüne Fluoreszenz, TDT - rote Fluoreszenz) bei unterschiedlichen Zeitpunkte. D 2 und 6: Maßstabsbalken repräsentieren 100 & mgr; m. D 10: Maßstabsbalken repräsentieren 200 & mgr; m. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
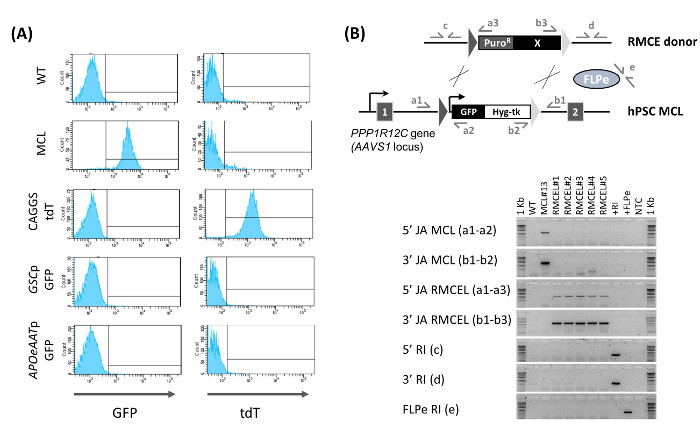
Abbildung 2:. Charakterisierung von RMCE Linien (A) Bestimmung der RMCE durchDurchflusszytometrie. GFP und TDT Ausdruck ist für die WT Linie, Master - Zelllinie (MCL), und neu generierte RMCE Linie bei D 15 Post-Nukleofektion mit einem konstitutiven Vektor TDT-Ausdruck (CAGGS TDT) oder Spender mit GFP gezeigt durch einen goosecoid Promotor angetrieben (GSCP GFP, endgültige Endoderm Marker) oder ein AAT - Promotor (APOeAATp GFP, Hepatozyten - Marker). (B) Bestätigung der RMCE (5 '/ 3' JA RMCEL), das Fehlen der Master - Zelllinie (MCL) Kassette (5 '/ 3' JA MCL), und der Mangel an zufälligen Integration (5 '/ 3' / FLPe RI ) durch PCR unter Verwendung der Primerpaare dargestellt. DNA von WT, MCL, RMCE Linien (1 - 5), Spender RMCE Vektor (+ RI), FLPe Expressionsvektor und keine Vorlage Kontrolle (NTC) verwendet wurden. Abbildung modifiziert aus (Ordovas et al., 2015) 13. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
innerhalb-page = "1"> 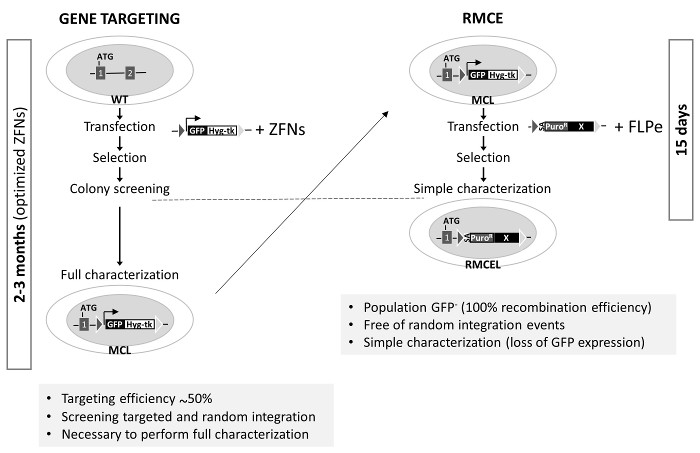
Abbildung 3. Die Erzeugung der RMCE-geeigneten Master - Zelllinie und Vergleich von Gen - Targeting mit RMCE in AAVS1 . Links: Das Gen - Targeting verwendet unmodifizierten WT - Zellen , die mit einem Spender (Kassette beschrieben cotransfiziert in 1A für die Erzeugung der Master - Zelllinie MCL), und spezifisch optimiert ZFNs. Der Prozess zu vervollständigen die vollständige Charakterisierung bis zu 3 Monate in Anspruch nimmt. Rechts: RMCE wird durch Co-Transfektion des Spenders mit FLPe Vektoren durchgeführt wird, und eine vollständig charakterisiert Linie wird in 15 d erzeugt. Gene Targeting - Effizienz in AAVS1 ist aus früheren Studien 1,2. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Tabelle 1. Medien Zusammensetzung.
| Probe | Vorwärts | Umkehren | Amplicon | PCR - Zyklus | ||
| 5'JA MCL | CACTTTGAGCTCTACTGGCTTC | CGTTACTATGGGAACATACGTCA | 1.1 Kb | 95 ºC, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / Zyklus), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ° C, 30 '' - 72 ºC, 1 '30' '] X25 - 72 ºC, 5' | ||
| 3'JA MCL | TAACTGAAACACGGAAGGAG | AAGGCAGCCTGGTAGACA | 1.4 Kb | 95 ºC, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / Zyklus), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ° C, 30 '' - 72 ºC, 1 '30' '] X25 - 72 ºC, 5' | ||
| 5'JA RMCEL | CACTTTGAGCTCTACTGGCTTC | CATGTTAGAAGACTTCCTCTGC | 1.1 Kb | 95 ºC, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / Zyklus), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ° C, 30 '' -72 ºC, 1 '30' '] X25 - 72 ºC, 5' | ||
| 3'JA RMCEL | TTCACTGCATTCTAGTTGTGG | AAGGCAGCCTGGTAGACA | 1.5 Kb | 95 ºC, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / Zyklus), 1' 30 ''] X15 - [95 ºC, 30 '' - 58 ° C, 30 '' - 72 ºC, 1 '30' '] X25 - 72 ºC, 5' | ||
| 5'RI SPENDER | GTACTTTGGGGTTGTCCAG | TTGTAAAACGACGGCCAG | 0,5 Kb | 95 ºC, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ºC, 30' '] X25 - 72 ºC, 5' | ||
| 3'RI SPENDER | CCTGAGTTCTAACTTTGGCTC | ACACAGGAAACAGCTATGAC | 0,5 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ºC, 30' '] X25 - 72 ºC, 5' | ||
| RI FLPe | CCTAGCTACTTTCATCAATTGTG | GTATGCTTCCTTCAGCACTAC | 0,65 Kb | 95 ºC, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ºC, 30' '] X25 - 72 ºC, 5' | ||
Tabelle 2. Primer - Sets verwendet für die PCR - Genotypisierung. Geändert von Ordovas et al., 2015 13.
Diskussion
Gene Bearbeitungsmethoden in sicheren Hafen loci bleiben ein wichtiges Instrument Transgene in hPSCs zu entwickeln. Obwohl der sichere Hafen Charakter AAVS1 hat vor kurzem für einige Anwendungen 13,18 in Frage gestellt wurde, bleibt diese Stelle derzeit in Menschen stamm gekennzeichnet beste Zelllinien. Das Bewusstsein für ihre Grenzen in hPSCs kann helfen, zuverlässige Daten zu erhalten. Daher wird AAVS1 noch eine nützliche Website zu erwarten sind , beispielsweise für die Verstärkungs- und loss-of-function - Studien oder induzierbare / konstitutive Expression von Faktoren , die eine isogene Kontext innerhalb und zwischen bestimmten genetischen Hintergründen erforderlich.
Die AAVS1 Locus wurde von vielen Gruppen ins Visier genommen ZFNs, TALEN oder CRISPR / Cas9 1,2,19 verwenden. Diese Nukleasen erhöhen signifikant die Effizienz der homologen Rekombination in einem bestimmten Ort. Doch in den Prozessbildschirm und vollständig zu charakterisieren richtig gezielt Klone, frei von zufälligen integRation und Pluripotenz und Genomintegrität aufrecht zu erhalten bis zu 3 Monate in Anspruch nehmen (Abbildung 3) (optimierte Gen - Editing - Tools verwenden). Die letzten beiden können durch mögliche Mutationen durch Off-Target-Nukleaseaktivität erzeugt verursacht werden. Das RMCE System hier verwendet, jedoch bietet eine schnelle, effiziente und elegante Methode , um dieses Ziel in nur zwei Wochen erreichen, sobald Rekombinase-spezifischen Zielsequenzen in den AAVS1 Locus vorintegrierten werden. Verwendung von positiven / negativen Selektion und einem geeigneten Selektionsprogramm sind die wichtigsten Faktoren für die Vereinfachung der Gen Bearbeitung im AAVS1 Locus durch RMCE beiträgt.
Genotypischen Charakterisierung der neuen transgenen Linien signifikant reduziert (ohne klonale Screening notwendig ist), und die Charakterisierung verbundenen Off-Target-Nuclease-Aktivität entbehrlich aufgrund der Spezifität der FLPe für FRT gemacht. Die Charakterisierung kann auch in der Routine RMCE Experimente reduziert werden, indem der Nachweisvollständigen Verlust der GFP - Expression von der Master - Zelllinie (2A), da volle Kassettenaustausch und der Mangel an zufälligen Integration bereits ausreichend nachgewiesen durch PCR (2B) und Southern - Blot - 13 ist. Die Verwendung von positiven / negativen Selektion stellt auch die größte Unterschied zu früheren Berichten. Mehrere Gruppen , die in hPSCs zuvor RMCE beschrieben, entweder in der AAVS1 oder andere Loci, nur eine einzige positive Selektionsschritt 7,14,16 eingesetzt. Dies stellt keine großen technischen Vorteil gegenüber Gen-Bearbeitung mit Nukleasen ausgeführt darstellen, weil Kolonie-Screening ebenso durchgeführt, um muss korrekte Integration und das Fehlen von zufälligen Integration auf der klonalen Ebene zu demonstrieren.
Die Nutzung dieser RMCE System in mehreren hESC / iPSC Linien erfordert preintegration der beschriebenen FRT enthaltenden Kassette im AAVS1 Locus jeder unabhängigen Linie. Wenn jedoch einmal erzeugt,seine Schnelligkeit und Einfachheit macht es möglich, halb hohen Durchsatz genetischen Screens in definierten isogenen Einstellungen für Anwendungen zu entwickeln, sonst technisch sehr zeitaufwendig wäre darüber erwähnt werden. Darüber hinaus sind alle RMCE Vektoren innerhalb des pZ AAVS1 Gen-Targeting - Vektor konstruiert , verwendet , um den Locus mit ZFNs Ziel, aber TALENS oder CRISPR / Cas9 wurden ebenfalls berichtet. Die Dualität der RMCE Vektor ist sehr nützlich mehrere Zeilen für einen bestimmten Transgen in hPSCs mit oder ohne FRT zu erzeugen. Beispielsweise in einer bestimmten genetischen Screen durchgeführt, indem RMCE müssten idealerweise getestet werden, in mehreren Linien HPSC einen Treffer erfolgreich demonstriert, um die Ergebnisse zu bestätigen. In Abwesenheit von mehreren RMCE-geeigneten Master-Zelllinien, direkte Gen des Treffers Targeting Nukleasen mit durchgeführt werden konnte. Der Nutzen des dualen Charakter der RMCE Vektoren wurde von unserer Gruppe 20 belegt. In dieser Studie wurde Genkorrektur durchgeführt in Patienten stammFrontotemporale Demenz (FTD) iPSCs , die eine Mutation verursacht progranulin (PGRN) Ausdruck Mangel tragen. Gene Komplementation durch ZFNs vermittelte Insertion in den Locus AAVS1 einer Kassette , die PGRN Pegel wiederhergestellt, korrigiert , um die defekten Phänotyp in Kortikogenese mit dem Vorhandensein der Mutation in Verbindung gebracht. Zusätzlich wurde eine vergleichbare Linie von RMCE in WT hESCs erzeugt für exogene PGRN Ausdruck als Kontrolle verwendet werden.
In Abwesenheit von rekombinanten Kolonien, überprüfen Transfektionseffizienz des RMCE Donorvektor. Transfektionseffizienzen überlegen zu 30% ergeben sich die oben angegebenen Ergebnisse. Niedrigere Transfektionseffizienzen verringern Rekombination Effizienz. Schließlich hat die RMCE System im AAVS1 Locus erzeugt worden ist , aber es ist anwendbar auf jeden anderen Locus.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
L.O. was funded by IWT/OZM/090838, IACS BPAMER3/08/04, and the Government of Aragon FMI048/08; RB by IWT fellowship SB-121396; M.P. by FWO 1288714N; and R.S. by the Dutch Diabetes Foundation. N.H., J.V., K.C., and Q.C. were supported by IWT fellowships SB-121396, SB-101230, SB-91228, and SB-093228, respectively. Funding to C.M.V. was from FWO G.0667.07, G.0975.11, and KU Leuven (EIW-B4855-EF/05/11, ETH-C1900-PF, EME-C2161-GOA/11/012), IWT-HEPSTEM, BELSPO-IUAP-DEVREPAIR, FP7-HEMIBIO (266777).
Materialien
| Name | Company | Catalog Number | Comments |
| DR4 Mouse Embryonic Fibroblasts (MEF) | AMS bio | GSC-6204G | hPSC culture on feeders |
| CF-1 MEF, mitomycin-C treated | AMS Bio | GSC-6001M | |
| EmbryoMax 0.1% Gelatin Solution | Millipore | ES-006-B | iMEF plating |
| DMEM/F-12, HEPES | Gibco | 31330-038 | |
| KnockOut Serum Replacement | Gibco | 10828-028 | |
| L-Glutamine | Sigma Aldrich | G8540 | For hESC medium |
| L-Glutamine, 200 mM | Gibco | 25030-081 | For iMEF medium |
| 2-Mercaptoethanol, 50 mM | Gibco | 31350-010 | |
| MEM Non-Essential Amino Acids Solution (100x) | Gibco | 11140-035 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| Human basic FGF | Peprotech | 100-18C | |
| Y-27632 in Solution | VWR | 688001-500 | |
| DMEM High Glucose | Gibco | 41965-039 | |
| Fetal Bovine Serum (FBS) | Sigma Aldrich | F7524 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300-054 | hPSC culture on feeders |
| Matrigel, hESC qualified | VWR | 734-1440 | |
| mTeSR1 complete medium kit | Stem cell technologies | 05850 | For Feeder-free culture |
| StemPro Accutase Cell Dissociation Reagent | Gibco | A1110501 | For feeder-free culture |
| Y-27632 in solution | VWR | 56726 | |
| Human Stem Cell Nucleofector Kit 2 | Lonza | VVPH-5022 | |
| Puromycine Dihydrochloride | Gibco | A11138-03 | |
| FIAU (Fialuridine) | ABX | 2910 | |
| Hygromycin B | Sigma Aldrich | H3274 | |
| pFLPe | Modified to from pCAGGS-FLPe (MES4488, Open Biosystems) to remove puromycin | ||
| RMCE donors | - | - | Modified from pZ donor AAVS1 puromycin vector (PZD0020-1KT, Sigma Aldrich)13 |
| 5 mL Round Bottom Polystyrene Test Tube, with Cell Strainer | Falcon | 352235 | |
| PureLink Genomic DNA kit | Invitrogen | K182001 | |
| GoTaq Flexi DNA Polymerase | Promega | M8298 | |
| Agarose | Sigma | A9539 | |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | |
| Nucleofector 2b Device | Lonza (Amaxa) | AAB-1001 | |
| NucleoCounter NC-100 | Chemometec | NC-100 | |
| BD FACS Canto | BD Biosciences | 337175 | |
| NucleoCassette | Chemometec | 941-0002 |
Referenzen
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat. Biotechnol. 29 (8), 731-734 (2011).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat. Biotechnol. 27 (9), 851-857 (2009).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Baer, A., Bode, J. Coping with kinetic and thermodynamic barriers: RMCE, an efficient strategy for the targeted integration of transgenes. Curr. Opin. Biotechnol. 12 (5), 473-480 (2001).
- Sakurai, K., et al. Efficient integration of transgenes into a defined locus in human embryonic stem cells. Nucleic Acids Res. 38 (7), e96(2010).
- Irion, S., et al. Identification and targeting of the ROSA26 locus in human embryonic stem cells. Nat. Biotechnol. 25 (12), 1477-1482 (2007).
- Di Domenico, A. I., Christodoulou, I., Pells, S. C., McWhir, J., Thomson, A. J. Sequential genetic modification of the hprt locus in human ESCs combining gene targeting and recombinase-mediated cassette exchange. Cloning Stem Cells. 10 (2), 217-230 (2008).
- Lombardo, A., et al. Site-specific integration and tailoring of cassette design for sustainable gene transfer. Nat. Methods. 8 (10), 861-869 (2011).
- Lombardo, A., et al. Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery. Nat. Biotechnol. 25 (11), 1298-1306 (2007).
- Simth, J. R., et al. Robust , Persistent Transgene Expression in Human Embryonic Stem Cells Is Achieved with AAVS1-Targeted Integration. Stem Cells. 26, 496-504 (2008).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Qian, K., et al. A simple and efficient system for regulating gene expression in human pluripotent stem cells and derivatives. Stem Cells. 32 (5), 1230-1238 (2014).
- Ordovás, L., et al. Efficient Recombinase-Mediated Cassette Exchange in hPSCs to Study the Hepatocyte Lineage Reveals AAVS1 Locus-Mediated Transgene Inhibition. Stem Cell Reports. 5 (5), 918-931 (2015).
- Du, Z. -W., Hu, B. -Y., Ayala, M., Sauer, B., Zhang, S. -C. Cre recombination-mediated cassette exchange for building versatile transgenic human Embryonic Stem Cells lines. Stem Cells. 27, 1032-1041 (2009).
- Papapetrou, E. P., et al. Genomic safe harbors permit high β-globin transgene expression in thalassemia induced pluripotent stem cells. Nat. Biotechnol. 29 (1), 73-78 (2011).
- Ramachandra, C. J., et al. Efficient recombinase-mediated cassette exchange at the AAVS1 locus in human embryonic stem cells using baculoviral vectors. Nucleic Acids Res. 39 (16), e107(2011).
- Takata, Y., Kondo, S., Goda, N., Kanegae, Y., Saito, I. Comparison of efficiency between FLPe and Cre for recombinase-mediated cassette exchange in vitro and in adenovirus vector production. Genes to Cells. 16 (7), 765-777 (2011).
- Mizutani, T., Li, R., Haga, H., Kawabata, K. Transgene integration into the human AAVS1 locus enhances myosin II-dependent contractile force by reducing expression of myosin binding subunit 85. Biochem. Biophys. Res. Commun. 465 (2), 270-274 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Raitano, S., et al. Restoration of progranulin expression rescues cortical neuron generation in an induced pluripotent stem cell model of frontotemporal dementia. Stem Cell Reports. 4 (1), 16-24 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten