Method Article
A partir de un producto natural a su cluster biosintético Gen: Una demostración del uso de Polyketomycin
En este artículo
Resumen
Aquí se presenta un protocolo detallado de (A) la identificación de un producto natural con actividad antibiótica, (B) la purificación del compuesto, (C) el primer modelo de su biosíntesis, (D) la secuenciación del genoma / -Minería y la ( E) verificación de la agrupación de genes biosintéticos.
Resumen
Cepas de Streptomyces son conocidos por su capacidad de producir una gran cantidad de diferentes compuestos con diversas actividades biológicas. El cultivo bajo diferentes condiciones a menudo conduce a la producción de nuevos compuestos. Por lo tanto, los cultivos de producción de las cepas se extraen con acetato de etilo y los extractos crudos se analizaron por HPLC. Además, los extractos se ensayaron para determinar su bioactividad mediante diferentes ensayos. Para elucidación de la estructura del compuesto de interés se purifica mediante una combinación de diferentes métodos de cromatografía.
La secuenciación del genoma junto con la minería genoma permite la identificación de un grupo de genes de biosíntesis de productos naturales usando diferentes programas de ordenador. Para confirmar que el grupo de genes correcto ha sido identificado, los experimentos de inactivación de genes que se han realizado. Los mutantes resultantes se analizan para la producción del producto natural particular. Una vez que el grupo de genes correcta ha sido inactivado, lacepa debe dejar de producir el compuesto.
El flujo de trabajo se muestra para el polyketomycin compuesto antibacteriano producido por Streptomyces diastatochromogenes Tü6028. Hace unos diez años, cuando la secuenciación del genoma era todavía muy caro, la clonación y la identificación de un grupo de genes fue un proceso muy lento. la secuenciación del genoma rápida, combinados con la minería genoma acelera el proceso de identificación de cluster y abre nuevas maneras de explorar la biosíntesis y para generar nuevos productos naturales por métodos genéticos. El protocolo descrito en este documento puede ser asignado a cualquier otro compuesto derivado de una cepa de Streptomyces u otro microorganismo.
Introducción
productos naturales de plantas y microorganismos han sido siempre una fuente importante para el desarrollo de fármacos y la investigación clínica. El primer antibiótico penicilina fue descubierta en 1928 a partir de un hongo por Alexander Fleming 1. Hoy en día, muchos más productos naturales se utilizan en el tratamiento clínico.
Un género conocido por su capacidad de producir varias clases de metabolitos secundarios con diferentes bioactividades es Streptomyces. Streptomyces son bacterias Gram-positivas y pertenecen a la clase de Actinobacteria y el orden Actinomycetales. Casi dos tercios de los antibióticos de uso clínico se derivan de los Actinomycetales, sobre todo a partir de Streptomyces, como la anfotericina 2, 3 daptomicina o tetraciclina 4. Dos premios Nobel se han concedido en el campo de la investigación de antibióticos Streptomyces. El primero fue a Selman Waksman para el descubrimiento de la estreptomicina, la primera de unaantibiótica eficaz contra la tuberculosis. 5 En 2015, como parte del Premio Nobel de Fisiología y Medicina, el descubrimiento de avermectinas de S. avermitilis se adjudicó también. La avermectina se utiliza para el tratamiento de enfermedades parasitarias 6,7.
El enfoque tradicional para el descubrimiento de productos naturales en microorganismos tales como Streptomyces generalmente implica el cultivo de la cepa bajo diferentes condiciones de crecimiento, así como la extracción y el análisis de metabolitos secundarios. Ensayos de bioactividad (por ejemplo, ensayos para actividad antibacteriana y contra el cáncer) se llevan a cabo para detectar la actividad del compuesto. Finalmente, el compuesto de interés se aísla y la estructura química se aclara.
Las estructuras de los productos naturales a menudo están compuestos de restos individuales que se están formando moléculas complejas. Hay algunas, pero limitadas, las principales rutas biosintéticas que conducen a la construcción de bloqueks, que se utilizan para la biosíntesis de productos naturales. Las principales vías de biosíntesis de poliquétidos son las vías, las vías que conducen a terpenoides y alcaloides, vías utilizando aminoácidos, y las vías que conducen a restos de azúcar. Cada vía se caracteriza por un conjunto de enzimas específicas 8. Basándose en la estructura del compuesto, estas enzimas biosintéticas se pueden predecir.
Hoy en día, el análisis estructural detallado de un compuesto en combinación con la secuenciación de próxima generación y el análisis bioinformático puede ayudar a identificar la agrupación de genes biosintéticos responsables. La información de clúster abre nuevas vías para futuras investigaciones producto natural. Esto incluye la expresión heteróloga para aumentar el rendimiento del producto natural, modificación compuesto blanco de la deleción del gen o la alteración y la biosíntesis combinatoria con genes de otras vías.
Polyketomycin fue aislado independientemente del caldo de cultivo dedos cepas, Streptomyces sp. MK277-AF1 9 y Streptomyces diastatochromogenes Tü6028 10. La estructura se elucidó por análisis de RMN y de rayos X. Polyketomycin se compone de un decaketid tetracíclico y un ácido salicílico de dimetilo, unidos por los dos restos desoxiazúcar ß-D-amicetose y α-L-axenose. Se muestra actividad citotóxica y antibióticos, incluso contra cepas Gram-positivos resistentes a múltiples fármacos, tales como MRSA 11.
Una biblioteca de cósmidos genómica de S. diastatochromogenes Tü6028 se generó y se proyectó hace muchos años. El uso de sondas de genes específicos de la agrupación de genes polyketomycin con un tamaño de 52,2 kb, que contiene 41 genes, fue identificado después de varios meses de intenso trabajo 12. Recientemente, se obtuvo un proyecto de secuencia del genoma de S. diastatochromogenes que conduce a la identificación rápida de la agrupación de genes biosintéticos polyketomycin. En este panorama, un método que ayuda a identificar un producto natural y dilucidar se describirá su agrupación de genes biosintéticos, utilizando polyketomycin como un ejemplo.
Aquí explicamos los pasos individuales que conducen de un producto natural a su agrupación de genes biosintéticos se muestra para polyketomycin producido por Streptomyces diastatochromogenes Tü6028. El protocolo comprende la identificación y purificación de un producto natural con propiedades antibióticas. Además el análisis estructural y la comparación con los resultados de genoma plomo minería a la identificación de la agrupación de genes biosintéticos. Este procedimiento se puede aplicar a cualquier otro compuesto derivado de una cepa de Streptomyces o cualquier otro microorganismo.
Protocolo
1. Identificación de un producto natural con Antibiótico Propiedad
- Cultivar un microorganismo en diferentes condiciones (por ejemplo, tiempo, temperatura, pH, medios de comunicación) después de la "OSMAC (una cepa de muchos compuestos) enfoque" 13. Seleccionar un medio en el que se observa la producción de un compuesto.
- Cultivar Streptomyces diastatochromogenes Tü6028 bajo las siguientes condiciones
- Crecer Streptomyces diastatochromogenes cepa Tü6026 sobre placas MS (harina de soja 20 g, D-manitol 20 g, MgCl2 10 mM, 1,5% de agar, el agua del grifo 1 l). Inocular un pequeño bucle de las esporas de esta cepa en 20 ml de medio TSB líquido (caldo de soja tríptico 30 g, agua del grifo 1 L, pH 7,2; medio de precultivo) en un matraz Erlenmeyer con una espiral. Agitar el matraz en un agitador rotatorio (28 ° C, 180 rpm, 2 días).
- Inocular el cultivo principal en 100 ml de medio de HA (glucosa 4 g, extracto de levadura 4 g, maltaextraer 10 g, agua del grifo 1 L, pH 7,2) con 2 ml del precultivo. Cultivar la cepa a 28 ° C durante 6 días en un agitador rotatorio a 180 rpm.
- Preparación del extracto crudo
- Cosecha células por centrifugación (10 min, 3.000 xg).
- Para los pasos siguientes manejar disolventes orgánicos bajo una campana de humos.
- Para la extracción de compuesto a partir del micelio, Resuspender las células en un doble volumen de acetona y agitar en un tubo para 30 min, 180 rpm. Se filtra el líquido a través de papel de filtro comercial y se evapora la acetona mediante evaporador rotatorio a 40 ° C y 550 bar. Se disuelve el extracto en 20 ml de agua: acetato de etilo (1: 1) y agitar en un embudo de separación durante 30 minutos a 180 rpm.
- Para la extracción compuesto del medio de cultivo, ajustar el caldo de cultivo a pH 4,0 mediante la adición de HCl 1 M. Añadir 100 ml de acetato de etilo y agitador en un embudo de separación durante 30 minutos, 180 rpm.
- Recoger la fase de acetato de etilo y se evapora por la podredumbre evaporación ary a 40 ° C y 240 bar.
- El análisis del extracto en bruto mediante HPLC
- Disolver los extractos en 1 ml de MeOH, se filtra a través de un filtro de 0,45 micras de tamaño de poro y ejecutar cromatografía líquida de alta resolución (HPLC) 14.
- En el caso de polyketomycin, equipar el sistema de HPLC con una precolumna C18 (4,6 x 20 mm 2) y una columna C18 (4,6 x 100 mm 2). Utilice un gradiente lineal de acetonitrilo en ácido acético + 0,5% que van desde 20% a 95% en H 2 O + 0,5% (velocidad de flujo: 0,5 ml min -1) acético.
NOTA: Polyketomycin tiene un tiempo de retención de 25,9 min (véase la Figura 1A). El espectro de UV / vis tiene máximos a 242 nm, 282 nm, 446 nm y mínimos a 262 nm y 359 nm (véase la Figura 1B). En el modus negativo una masa de 863.2 [MH] - es detectable (véase la Figura 1C).
"Src =" / files / ftp_upload / 54952 / 54952fig1.jpg "/>
Figura 1: LC / MS Análisis de Polyketomycin. (A) HPLC cromatograma (λ = 430 nm) del extracto crudo después del cultivo de Streptomyces diastatochromogenes Tü6028 durante 6 días. Polyketomycin tiene un tiempo de retención de 25,9 min UV (B) / vis espectros de Polyketomycin. (C) Los espectros de masas de Polyketomycin en el modus negativo. Pico principal con m / z 863,2 [MH] -. Haga clic aquí para ver una versión más grande de esta figura.
- Identificación de la actividad antibiótica utilizando el ensayo de difusión en disco
- Inocular cepas de prueba como Gram-positivos subtilis bacteria Bacillus (en medio LB; LB 20 g, agua del grifo 1 L, pH 7,4) y otras cepas de Streptomyces (en medio TSB; caldo de soja tríptico 30 g, agua del grifo 1 L, pH 70.2), las bacterias Gram-negativas Escherichia coli (en medio LB) y las cepas fúngicas (en medios tales como el medio YPD; extracto de levadura 10 g, peptona 20 g, glucosa 20 g, grifo de agua 1 L, pH 7,4). Tomar 100 l de prueba de tensión pre-cultivo y repartirlos sobre las respectivas placas de agar.
- Se disuelve el extracto en bruto o compuesto purificado en 500 l de metanol (alternativamente agua o DMSO) y la pipeta 20 hasta 50 l en discos de papel estéril. discos de papel seco durante 30 minutos bajo la mesa de trabajo y ponerlos en las placas con cultivos de ensayo. Preparar un control negativo (solvente) y un control positivo (antibiótico, por ejemplo, apramicina [1 mg]).
- Las placas se incuban en condiciones adecuadas hasta que las cepas de prueba se cultivan de forma visible y determinar la zona de inhibición, si es aparente. Incubar E. coli y Bacillus sp. a 37 ° C durante 16 h, Streptomyces sp. a 28 ° C durante 2-4 días, cepa fúngica (depende principalmente de la cepa de prueba exacta) unat 30 ° C durante 2 días).
2. La extracción a gran escala, purificación y elucidación de la estructura del Compuesto
- Cultivar S. diastatochromogenes en medio de 5 l HA (glucosa 4 g, extracto de levadura 4 g, extracto de malta 10 g, el agua del grifo 1 L, pH 7,2). Inocular 2 ml del cultivo previo en 30 x 500 ml frascos Erlenmeyer conteniendo 150 ml de medio de HA. Incubar la cepa a 28 ° C durante 6 días en un agitador rotatorio a 180 rpm.
- Cosecha células por centrifugación (10 min, 15.000 xg, RT) y los compuestos de extractos utilizando acetato de etilo (ver sección 1.3).
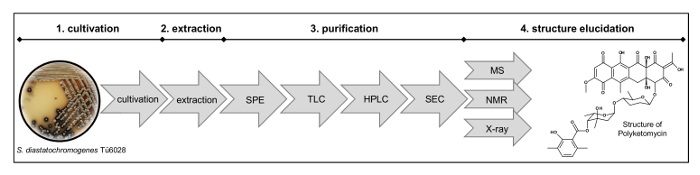
Figura 2: Flujo de trabajo para la elucidación estructural. El procedimiento comprende (1) el cultivo de la cepa, (2) extracción, (3) la purificación por extracción en fase sólida (SPE), cromatografía de capa fina (TLC), cromatografía líquida de alto rendimiento preparativa (HPLC), el tamañocromatografía de exclusión (SEC) y (4) elucidación de la estructura por análisis de masas (MS), resonancia magnética nuclear (RMN) y mediciones de rayos X. Haga clic aquí para ver una versión más grande de esta figura.
- Purificación de Polyketomycin
NOTA: El proceso de purificación y elucidación de la estructura se muestra en la Figura 2.- Fraccionar el extracto en bruto mediante una columna de extracción en fase sólida C18 (SPE) usando un gradiente de 10% -stepwise MeOH que van desde 30% a 100% de MeOH en H 2 O, 100 ml para cada condición.
- Se purifica la fracción que contiene el compuesto de nuevo por cromatografía preparativa en capa fina (TLC) 15 usando CH 2 Cl 2 / MeOH (7: 1) como sistema de elución.
- Se purifica el compuesto por HPLC preparativa 16. Equipar el sistema de HPLC con una precolumna C18 (5 m; 9,4 x 20 mm) y unacolumna principal (5 m; 9,4 x 150 mm). Utilice un gradiente de ácido acético acetonitrilo + 0,5% que van desde 20% a 95% en ácido acético H 2 O + 0,5% (velocidad de flujo: 2,0 ml / min).
- Para eliminar los disolventes y otras impurezas pequeñas, lleve a cabo una última etapa de purificación por cromatografía de exclusión de tamaño utilizando una columna en MeOH 17. Recoger el compuesto puro final y evaporar MeOH por evaporación rotativa a 40 ° C y 240 bar.
- estructura elucidación
- Se disuelve el compuesto puro (más de 2 mg) en 600 a 700 l (dependiendo de la máquina) de DMSO-d 6, ficha 1D de RMN (1 H, 13 C) y RMN 2D (HSQC, HMBC, 1 H- 1 H COSY) los espectros de RMN en un espectrómetro 18. Expresar los desplazamientos químicos en los valores de delta (ppm) mediante el uso de DMSO-d 6.
- Registrar un espectro de masas de alta resolución (HRESI-MS) 19 de polyketomycin utilizando una alta resoluciónespectrómetro de masas.
- Elucidar la estructura por la interpretación de los resultados del análisis de los datos de RMN y MS 10.
3. Proponer biosintética Modelo del Nuevo compuesto aislado
- Analizar la estructura del compuesto aislado y predecir las enzimas, que pudieran estar implicados en su biosíntesis. Asignarlos a policétido (tipo I, II o III), la síntesis de péptido no ribosomal, lantipeptide, terpenos, o azúcar vía metabolismo 8.
- ejemplo polyketomycin
- Subdividir la estructura en mitades individuales obvias: fracción tetracíclicos, dos monosacáridos y el ácido salicílico dimetilo.
- Descubre, donde los restos pueden ser derivadas de: El resto tetracíclico podría derivarse de un tipo de poliquétido sintasa II y el resto de ácido salicílico de dimetilo puede ser derivado por un iterativo policétido sintasa de tipo I. Los dos restos de azúcares, que son 6-desoxiazúcares , podrían ser SYNTHESIzeta a partir de glucosa implica un-glucosa-4,6-deshidratasa TDP durante la biosíntesis y unido probablemente por dos glicosiltransferasas (véase la Figura 3) 8.
- Predecir los genes putativos de la agrupación. Piense de enzimas que están implicadas en la síntesis de los restos individuales, en la conexión de las unidades, así como en la modificación y la adaptación de la molécula. La agrupación de genes biosintéticos de Polyketomycin muy probablemente contiene los genes que codifican una policétido sintasa tipo II (por lo tanto mínimo un ACP, KS α und KS β para conectar unidades de elongación), un proceso iterativo tipo policétido sintasa I (al menos ACP, AT, KS), una TDP-glucosa-4,6-deshidratasa (necesaria para el paso: la glucosa → desoxiglucosa) y dos glicosiltransferasas (adjuntando dos monómeros de azúcar a la aglicona) 8.
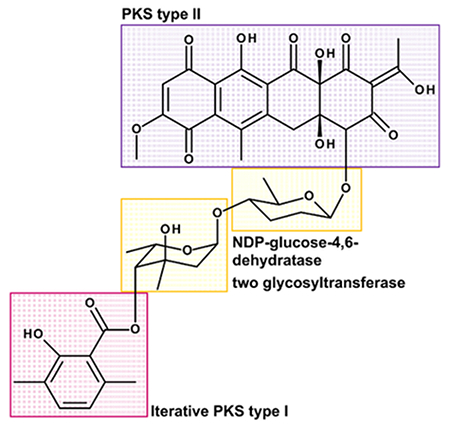
Figura 3: Estructura del Polyketomycin Divided en simples bloques de construcción. Polyketomycin se compone de un decaketid tetracíclico (PKS de tipo II) y un ácido salicílico de dimetilo (iterativo tipo PKS I), unidos por los dos restos desoxiazúcar β-D-amicetose y α-L-axenose (NDP-glucosa-4,6- deshidratasa y dos glicosiltransferasa necesario). Haga clic aquí para ver una versión más grande de esta figura.
4. Secuenciación del Genoma / Minería
- Secuenciación de próxima generación
- Secuenciar el ADN genómico mediante tecnologías de secuenciación de próxima generación, como Illumina, 454 pirosecuenciación 20 o sólido. Alinear solo lee a una secuencia de referencia o ensamblar de novo.
NOTA: El genoma de S. diastatochromogenes Tü6028 se secuenció en el Centro de Biotecnología (CeBiTec) en la Universidad de Bielefeld. Todas las lecturas fueron reunidos para un proyecto de genomade 7,9 Mb.
- Secuenciar el ADN genómico mediante tecnologías de secuenciación de próxima generación, como Illumina, 454 pirosecuenciación 20 o sólido. Alinear solo lee a una secuencia de referencia o ensamblar de novo.
- la minería genoma
- Búsqueda de marcos de lectura abiertos (ORF) por el uso de, por ejemplo, NCBI anotación del genoma procariótico Pipeline 21,22, RAST (rápida anotación utilizando la tecnología del subsistema) 23,24,25, Prokka (rápida anotación del genoma procariótico) 26 o 27 GenDB. Estos programas también proponen sus funciones. El análisis de la secuencia de S. diastatochromogenes Tü6028 proyecto genoma dado lugar a la identificación de más de 7.000 ORF.
- Ejecutar BLAST específica (Basic Local Alignment Search Tool) análisis para obtener más información como alineaciones con otros genes similares y dominios catalíticos 28,29,30.
- Para la identificación de los programas dirigidos por el grupo de genes de metabolitos secundarios como antiSMASH 31,32,33, 34 y NaPDoS NRPSpredictor 35,36. En el proyecto de genoma de Streptomyces diastatochromogenes antiSMASH 31,32,33 identicado 22 grupos de genes.
- Analizar los grupos de genes putativos para su vía (s) enzimática (policétido sintasa (tipo I, II o III), la sintetasa no ribosómica de péptidos, lanthipeptide, terpenoides, el metabolismo del azúcar ...). Búsqueda de clúster (s) que contiene genes que podrían estar implicados en la síntesis del compuesto (ver sección 3). En S. diastatochromogenes anotada grupo 2 contiene policétido sintasa tipo II genes, tres policétido sintasa de tipo I genes, un gen TDP-glucosa-4,6-deshidratasa y dos genes de glicosiltransferasa (véase la Figura 4).
- Centrarse en los genes individuales dentro del grupo. Para el tipo I y PKS NRPS la especificidad de aciltransferasas y dominios de adenilación, y por lo tanto la incorporación de las unidades extensoras sola, puede ser predicho. También comparar la orden de la unidad extensora incorporado con la molécula. antiSMASH 31,32,33 también muestra las agrupaciones similares de compuestos ya conocidos, con un enlace a la base de datos MIBiG 37.
- Comparación de la estructura del compuesto con estos otros compuestos y comprobar si hay similitudes.

Figura 4: Salida de antiSMASH Polyketomycin biosintética de Gene Cluster y visión general de otros clusters en S. diastatochromogenes Tü6028. (A) Descripción general de los grupos de genes biosintéticos predichos en el genoma de S. diastatochromogenes Tü6028; (B) Grupo 2 Polyketomycin agrupación de genes biosintéticos con determinados genes. Haga clic aquí para ver una versión más grande de esta figura.
5. Verificación del cluster biosintético de Gene
- Búsqueda de un gen con funciones obvias e importantes (esenciales) para la biosíntesis del compuesto. Para la verificación de la polyketogrupo de genes micina la pokPI gen, que codifica para la α cetosintasa de la PKS de tipo II, se seleccionó y se inactiva por un fuera de -deletion marco (véase la figura 5B). Alternativamente, la interrupción del gen mediante la clonación de un fragmento interno (véase la Figura 5C).
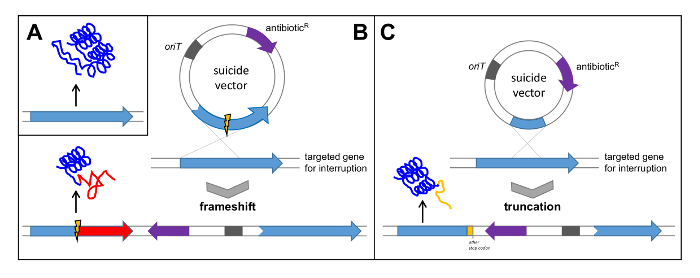
Figura 5: Verificación de una agrupación de genes mediante entrecruzamiento sencillo. (A) gen nativo conduce a la traducción de una proteína funcional; (B) Clonación del gen con deleción interna en un vector suicida conduce a un único cruce que resulta en un cambio de marco en el gen diana y la posterior traducción de la proteína no funcional; (C) Clonación de un fragmento interno del gen en un vector suicida conduce a un truncamiento del gen y la posterior traducción de una proteína no funcional. oriT: origen de tRANSFERENCIA; R antibióticos: resistencia a los antibióticos. Haga clic aquí para ver una versión más grande de esta figura.
- Clonación del constructo de un solo cruce
- Amplificar un fragmento que contiene pokPI por PCR con los cebadores 38 y pokPI_for pokPI_rev.
- Producto Clone PCR en el vector suicidio pKC1132 39 (apramicina R [50 mg / ml]). El vector suicida no es capaz de replicarse en la cepa de Streptomyces y por lo tanto tiene que integrar en el gen diana mediante recombinación homóloga para proporcionar apramicina-resistencia. Transferir el vector en E. coli huésped de clonación mediante la transformación de choque térmico 40.
- Aislar el vector mediante lisis alcalina 41.
- Digerir el ADN del vector con una sola enzima de restricción de corte que corta dentro del fragmento. El gen pokPIe se digirió con enzima Kpn I.
- Tratar DNA vector digerido con el ADN grande de la polimerasa I (Klenow) fragmento, que tiene '→ 3' actividad 5 polimerasa y 3 '→ 5' exonucleasa-actividad. Blunting de los extremos cohesivos y religación posterior conduce a la pérdida de cuatro bases. Compruebe si hay pérdida de estas bases por la transformación pasos en E. coli XL1 Blue, recogiendo colonias individuales, aislar su ADN plásmido, 41 y analizar más a fondo por la digestión de restricción y secuenciación.
- Conjugación del cruce de un solo constructo en Streptomyces
- Transferir el vector suicida que contiene el gen pokPI (pKC1132_ pokPI DEL) con deleción en E. coli ET 12567 pUZ8002 42 (kanamicina R) por transformación de choque térmico 40. Se incuban las células en 100 ml de medio LB (kanamicina 50 mg ml -1, apramicina 50 mg ml -1 ) Durante la noche a 37 ° C. células de la cosecha por centrifugación (3.000 xg, 10 min, 4 ° C) y lavar células dos veces por resuspensión en 50 ml de medio LB. Por último, volver a suspender las células en 2 x 500 l de medio LB.
- Mezclar 500 l E. coli pUZ8002 pKC1132_ pokPI con la cultura del 500 l Streptomyces diastatochromogenes (alternativamente usar esporas). Extender la mezcla en placas MS (harina de soja 20 g, D-manitol 20 g, MgCl2 10 mM, 1,5% de agar, el agua del grifo 1 l). Incubar las placas durante 20 horas a 28 ° C.
- Superponer cada placa con apramicina (1,25 mg) y fosfomicina (5 mg) disuelto en 1 ml de agua y dejar secar. Se incuban las placas durante varios días a 28 ° C hasta que exconjugantes son visibles.
- Compruebe mutantes entrecruzamiento sencillo
- Inocular exconjugantes individuales en 20 ml de medio TSB (apramicina 50 mg ml -1) y los incuban a 28 ° C durante tres días y 180 rpm.
- Aislar el ADN genómico 43 y comprobar la interrupción del gen pokPI por PCR.
- Se inoculan los clones individuales con la interrupción del gen de tipo salvaje cepa verificada y Streptomyces en 100 ml de medio de HA y los incuban durante 6 días a 28 ° C y 180 rpm. Se extrae el extracto crudo (ver sección 1.3). Verificar la producción de compuestos mediante análisis por HPLC-MS (ver sección 1.4). El pico correspondiente en el cromatograma de HPLC y la masa del compuesto no deberían ser detectables más (véase la Figura 6).
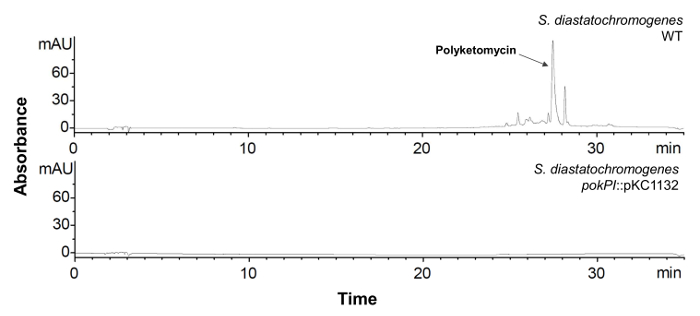
Figura 6: Análisis por HPLC de S. diastatochromogenes con inactivada pokPI génica. Cromatograma HPLC (λ = 430 nm) del extracto crudo de S. diastatochromogenes WT (arriba) y la cepa mutante con interrumpido pokPI -Gén (abajo). La cepa mutante no produce polyketomycin más.Haga clic aquí para ver una versión más grande de esta figura.
Resultados
En este resumen se describen los pasos individuales de la identificación de un antibiótico que lleva a su agrupación de genes biosintéticos. Hace muchos años hemos clonado de una biblioteca de cósmidos, fagos empaquetados en ellos, transducidas células huésped de E. coli, y tuvo que examinar miles de colonias para identificar los clones que tienen regiones de solapamiento de clúster polyketomycin. La secuenciación de los cósmidos era también un proceso difícil y costoso 12.
Con el fin de llevar a cabo más estudios sobre la cepa secuenciado todo el genoma de Streptomyces diastatochromogenes Tü6028. Con el proyecto de secuencia del genoma que identifica fácilmente la agrupación de genes biosintéticos de polyketomycin y otros grupos que codifican compuestos prometedores. La Figura 7 compara el método de la "vieja" de la identificación del cluster biosintético por clonación de una biblioteca de cósmidos y la detección elaborative, yel método "nuevo" mediante la secuenciación de todo el genoma con la posterior explotación minera genoma en una escala de tiempo áspero. Las nuevas tecnologías de secuenciación y nuevos programas de minería genoma acelerar todo el proceso.
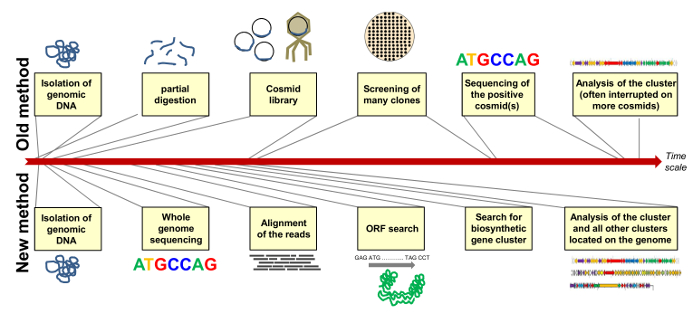
Figura 7: Comparación del Método y "viejo" "Nuevo" de la asignación de un gene cluster biosintético. El método "viejo" comprende la clonación de una biblioteca de cósmidos con la selección de clones positivos y secuenciación del cósmido (s) respectiva (arriba); El método "nuevo" incluye secuenciación del genoma completo y -Minería para identificar todos los grupos de genes de metabolitos secundarios situados en el genoma de la cepa (abajo). Duración de los pasos individuales se muestran en una escala de tiempo áspero. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En este laboratorio una biblioteca de cósmidos genómica de Streptomyces diastatochromogenes se generó y se proyectó hace muchos años, lo que resulta en la identificación de la agrupación de genes polyketomycin través de un proceso extremadamente lento. Caracterización de los genes individuales fuera posible usando deleciones de genes específicos y el análisis de los mutantes resultantes 12. Recientemente, se obtuvo un proyecto de secuencia del genoma de S. diastatochromogenes que permite la identificación rápida de la agrupación de genes biosintéticos polyketomycin. Podríamos detectar fácilmente los genes biosintéticos, aunque el proyecto de secuencia del genoma contiene todavía muchos contigs. El proceso descrito se puede lograr en pocos meses. Sin embargo, el procedimiento comprende muchos pasos. medidas individuales pueden fallar varias veces, la prevención de la progresión a etapas posteriores:
El género Streptomyces es conocida por su capacidad de producir compuestos bioactivos. Mientras que llevan a menudo más de 20 biografíasynthetic grupos de genes, por lo general sólo una o dos compuestos se producen bajo condiciones de laboratorio. La aplicación del enfoque OSMAC (cultivo de una cepa en condiciones diferentes) para despertar grupos de genes silenciosos a veces puede no ser suficiente. La manipulación genética de los genes reguladores, tales como los genes reguladores pleiotrópicos ADPA 44 y bldA 45,46, es también un método eficaz para activar la producción de otros metabolitos secundarios.
Para la elucidación de la estructura del compuesto, por ejemplo, por análisis de RMN, por lo general más de 2 mg de compuesto purificado es necesario. Por lo tanto, a menudo se requiere la fermentación de la cultura más de 10 L. Sin un fermentador que es capaz de mantener condiciones de oxígeno, pH y temperatura, puede ser un reto en un pequeño laboratorio para manejar esta cantidad de cultivo y la extracción posterior. Durante la purificación, el compuesto puede cambiar debido a la oxidación, la radiación o la temperatura.También, se utilizan las más etapas de purificación, mayor será la probabilidad de degradación.
Al analizar la estructura del producto natural, y los grupos en el genoma, a veces no es tan fácil de identificar el clúster correspondiente. En primer lugar, si sólo hay un proyecto de secuencia del genoma alguna parte del clúster puede no aparecer. En segundo lugar, no todos los genes que son necesarios para la biosíntesis están en el clúster. En tercer lugar, a veces un grupo se divide en dos partes separadas una de otra por muchos kilobases. En cuarto lugar, puede ser difícil decidir cuál es el grupo de genes correspondiente. En caso de gran tipo PKS I o sistemas NRPS, donde es posible calcular el número de unidades de elongación en función del número de módulos, o incluso identificar las unidades extensoras individuales por análisis de los dominios de selección, se convierte fácilmente. Sin embargo, en el caso de trabajar de forma iterativa enzimas de la predicción de los compuestos sintetizados a menudo no es posible, especialmente si la straen cuenta con más de 40 grupos de genes. En quinto lugar, la naturaleza es muy complejo y lleno de compuestos desconocidos. A menudo la biosíntesis es una mezcla de diferentes vías. Si el nuevo compuesto no es identificado todavía, o no relacionado con otro compuesto, puede ser difícil identificar el clúster, para proponer un modelo de biosíntesis y para demostrarlo.
Una vez identificado el clúster, la técnica de cruce individual es un método rápido y bueno para verificar la hipótesis. PCR, clonación en un vector suicida, conjugación, la selección de clones positivos, y el ensayo de producción son los únicos pasos necesarios. Una desventaja de esta técnica es que la integración del vector en el cromosoma no es estable debido a más eventos de recombinación. Por lo tanto, con el fin de analizar más a fondo los genes individuales, se requiere que las supresiones de genes precisos. También puede ser difícil de manipular cepas de Streptomyces en el nivel genético.
El procedimiento descrito se puede asignar to cualquier otro compuesto producido por una cepa de Streptomyces u otro microorganismo. El conocimiento acerca de un grupo de genes de biosíntesis y su compuesto sintetizado nos da más oportunidades para modificar moléculas ya existentes con el fin de mejorarlos para la lucha contra los patógenos resistentes a múltiples fármacos.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
S. Zhang is funded by China Scholarship Council. The authors are very grateful to former people working on polyketomycin project in this lab and Prof. Dr. Hans-Peter Fiedler, University of Tübingen, for providing the polyketomycin producer. The research was supported by the DFG (RTG 1976).
Materiales
| Name | Company | Catalog Number | Comments |
| agar | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 5210.4 | |
| agarose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6352.4 | |
| D-mannitol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4175.1 | |
| glucose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6780.1 | |
| LB | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X964.3 | |
| malt extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X976.2 | |
| MgCl2 | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2189.1 | |
| Peptone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | ||
| soy flour | W.Schoenemberger GmbH, Magstadt, Germany | Hensec-Vollsoja | |
| tryptic soy broth | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X938.3 | Caso-Bouillon |
| yeast extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2363.2 | |
| Solvents | |||
| Acetic acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 3738.5 | |
| Acetone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 9372.2 | |
| Acetonitrile | Avantor Performance Materials B.V., Deventer, The Netherlands | JT-9012-03 | |
| Dichlorofrom | Fisher Scientific GmbH, Schwerte, Germany | 1530754 | |
| DMSO (Dimethyl sulfoxide) | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4720.1 | |
| DMSO-d6 (Dimethyl sulfoxide-d6) 99.9 atom% D | ARMAR Chemicals, Döttingen, Switzerland | 15200.204 | |
| Ethyl acetate | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6784.4 | |
| Hydrochloric acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6331.4 | |
| Methanol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 7342.1 | |
| Enzymes | |||
| restriction enzymes | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| polymerase | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| DNA Polymerase I Large (Klenow) Fragment | Promega GmbH, Mannheim, Germany | ||
| Antibiotics | |||
| apramycin | AppliChem GmbH, Darmstadt, Germany | A7682.0005 | |
| fosfomycin | Sigma-Aldrich Chemie GmbH, Taufkirchen, Germany | P5396-50G | |
| kanamycin | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | T832.2 | |
| Plasmid/Vectors | |||
| pKC1132 | Bierman et al. 1992 | ||
| Primer | |||
| pokPI_for | TGATGGTGCCGCTGGCCATGG | Primer to amplify fragment containing pokPI gene | |
| pokPI_rev | AGCGTTCACTGTTCCGCCCGAC | ||
| Bacterial strains | |||
| Bacillus subtilis COHN ATCC6051 | Gram-positive test strain | ||
| Escherichia coli ET12567 pUZ8002 | MacNeil et al. 1992 | strain for conjugation | |
| Escherichia coli XL1 Blue | Agilient Technologies, Santa Clara, USA | Gram-negative test strain + cloning host | |
| Streptomyces diastatochromogenes Tü6028 | Paululat et al., 1999 | Polyketomycin producer | |
| Online services | |||
| antismash (Antibiotics and Secondary Metabolite Analysis Shell) | http://antismash.secondarymetabolites.org/ | Detection of secondary metabolite gene cluster | |
| BLAST (Basic Local Alignment Search Tool) | http://blast.ncbi.nlm.nih.gov/Blast.cgi | finds regions of similarity between biological sequences | |
| GenDB | https://www.uni-giessen.de/fbz/fb08/Inst/bioinformatik/software/gendb | Annotation of ORFs | |
| MiBIG (Minimum Information about a Biosynthetic Gene cluster) | http://mibig.secondarymetabolites.org/ | Database of biosyntetic gene clusters | |
| NaPDoS | http://napdos.ucsd.edu/ | Detection of seconary metabolite gene cluster | |
| NCBI Prokaryotic Genome Annotation Pipeline | http://www.ncbi.nlm.nih.gov/genome/annotation_prok/ | Annotation of ORFs | |
| NRPSpredictor | http://nrps.informatik.uni-tuebingen.de/ | Detection of NRPS domains | |
| Prokka (rapid prokaryotic genome annotation) | http://www.vicbioinformatics.com/software.prokka.shtml | Annotation of ORFs | |
| RAST (rapid annotation using subsystems technology) | http://rast.nmpdr.org/ | Annotation of ORFs | |
| Other programs | |||
| Chem Station Rev. A.09.03 | Agilent Technologies, Waldbronn, Germany | Handling program for HPLC | |
| Clone Manager Suite 7 | Scientific and Educational Software, Cary, USA | Designing Cloning Experiment | |
| Newbler v2.8 | Roche Diagnostics | Alignment of sequencing reads | |
| Machines | |||
| Centrifuge Avanti J-6000, Rotor JA-10 | Beckman Coulter GmbH, Krefeld, Germany | ||
| HPLC/MS | Agilent Technologies, Waldbronn, Germany | ||
| _Autosampler: G1313A | Agilent Technologies, Waldbronn, Germany | ||
| _Pre-column: XBridge C18 (20 mm x 4.6 mm; Particle size: 3.5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Column:Xbridge C18 (100 mm × 4.6 mm; Particle size: 3.5 μm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Pre-Column: Zorbax B-C18 (9.4 x 150 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Column: Zorbax B-C18 (9.4 x 20 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Degasser: G1322A | Agilent Technologies, Waldbronn, Germany | ||
| _Quarternary pump: G1311A | Agilent Technologies, Waldbronn, Germany | ||
| _Diode array detector (DAD )G1315B (λ = 254 nm and 400 nm) | Agilent Technologies, Waldbronn, Germany | ||
| _Quadrupole mass detector (MSD) G1946D(2-3000 m/z) | Agilent Technologies, Waldbronn, Germany | ||
| rotary evaporator | |||
| _heating bath Hei-VAP Value/G3 | Heidolph Instruments GmbH & Co.KG, Schwabach, Germany | ||
| _vacuum pump system SC 920 G | KNF Global Strategies AG, Sursee, Switzerland | ||
| Other material | |||
| Sephadex LH20 | GE Healthcare, | ||
| Chromafil PVDF-45/15MS (pore size 0.45 µm; filter Ø15 mm) | MACHEREY-NAGEL GmbH & Co. KG, Düren, Germany | ||
| SPE column Oasis HLB 20 35 cc (6g) | Waters GmbH, Eschborn, Germany | ||
| E. coli | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Bacillus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Streptomyces sp. | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: TSB, Composition: CASO Boullion, Amount to 1 L H2O: 30 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Yeast extract, Amount to 1 L H2O: 10 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Peptone, Amount to 1 L H2O: 20 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Glucose, Amount to 1 L H2O: 20 g | |
| for agar plates add 2% agar | |||
Referencias
- Fleming, A. On the antibacterial action of cultures of a penicillin, with special reference to their use in isolation of B. influenzae. Br J Exp Pathol. 10 (3), 226-236 (1929).
- Lemke, A., Kiderlen, A. F., Kayser, O. Amphotericin B. Appl. Microbiol. Biotechnol. 68 (2), 151-162 (2005).
- Kirkpatrick, P., Raja, A., LaBonte, J., Lebbos, J. Daptomycin. Nat. Rev. Drug Discov. 2 (12), 943-944 (2003).
- Zakeri, B., Wright, G. D. Chemical biology of tetracycline antibiotics. Biochem. cell Biol. 86 (2), 124-136 (2008).
- Schatz, A., Bugle, E., Waksman, S. A. Streptomycin, a Substance Exhibiting Antibiotic Activity Against Gram-Positive and Gram-Negative Bacteria. Exp. Biol. Med. 55 (1), 66-69 (1944).
- Burg, R. W., et al. Avermectins, new family of potent anthelmintic agents: producing organism and fermentation. Antimicrob. Agents Chemother. 15 (3), 361-367 (1979).
- Egerton, J. R., Ostlind, D. A., et al. Avermectins, new family of potent anthelmintic agents: efficacy of the B1a component. Antimicrob. Agents Chemother. 15 (3), 372-378 (1979).
- Walsh, C. T., Fischbach, M. A. Natural products version 2.0: connecting genes to molecules. J. Am. Chem. Soc. 132 (8), 2469-2493 (2010).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces sp. MK277-AF1. II. Structure determination. J. Antibiot. (Tokyo). 51 (1), 26-32 (1998).
- Paululat, T., Zeeck, A., Gutterer, J. M., Fiedler, H. P. Biosynthesis of polyketomycin produced by Streptomyces diastatochromogenes.Tü6028. J. Antibiot. (Tokyo). 52 (2), 96-101 (1999).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces. sp. MK277-AF1. I. Taxonomy, production, isolation, physico-chemical properties and biological activities. J. Antibiot. (Tokyo). 51 (1), 21-25 (1998).
- Daum, M., et al. Organisation of the biosynthetic gene cluster and tailoring enzymes in the biosynthesis of the tetracyclic quinone glycoside antibiotic polyketomycin. Chembiochem. 10 (6), 1073-1083 (2009).
- Bode, H. B., Bethe, B., Höfs, R., Zeeck, A. Big effects from small changes: possible ways to explore nature's chemical diversity. Chembiochem. 3 (7), 619-627 (2002).
- Wolfender, J. -. L. HPLC in Natural Product Analysis: The Detection Issue. Planta Med. 75 (07), 719-734 (2009).
- Stahl, E. . Thin-layer chromatography. A laboratory handbook. , (1967).
- Snyder, L. R., Kirkland, J. J., Glajch, J. L. Preparative HPLC Separation. Pract. HPLC Method Dev. , 616-642 (2012).
- Granath, K. A., Kvist, B. E. Molecular weight distribution analysis by gel chromatography on sephadex. J. Chromatogr. A. 28, 69-81 (1967).
- Jackman, L. M., Sternhell, S. Application of Nuclear Magnetic Resonance Spectroscopy in Organic Chemistry. Int. Ser. Org. Chem. , (1969).
- Xian, F., Hendrickson, C. L., Marshall, A. G. High resolution mass spectrometry. Anal. Chem. 84 (2), 708-719 (2012).
- Metzker, M. L. Sequencing technologies - the next generation. Nat. Rev. Genet. 11 (1), 31-46 (2010).
- Tatusova, T., et al. Prokaryotic Genome Annotation Pipeline. NCBI Handb. 2nd Ed. , (2013).
- Angiuoli, S. V., et al. Toward an online repository of Standard Operating Procedures (SOPs) for (meta)genomic annotation. OMICS. 12 (2), 137-141 (2008).
- Aziz, R. K., et al. The RAST Server: rapid annotations using subsystems technology. BMC Genomics. 9, 75 (2008).
- Overbeek, R., et al. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST). Nucleic Acids Res. 42, 206-214 (2014).
- Brettin, T., et al. RASTtk: a modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes. Sci. Rep. 5, 8365 (2015).
- Seemann, T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Meyer, F. GenDB--an open source genome annotation system for prokaryote genomes. Nucleic Acids Res. 31 (8), 2187-2195 (2003).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. J. Mol. Biol. 215 (3), 403-410 (1990).
- Madden, T. The BLAST Sequence Analysis Tool. NCBI Handb. 2nd Ed. , (2003).
- Marchler-Bauer, A., et al. CDD: NCBI's conserved domain database. Nucleic Acids Res. 43, 222-226 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Res. 39, 339-346 (2011).
- Blin, K., et al. antiSMASH 2.0--a versatile platform for genome mining of secondary metabolite producers. Nucleic Acids Res. 41, 204-212 (2013).
- Weber, T., et al. antiSMASH 3.0 - a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Res. 43, 237-243 (2015).
- Ziemert, N., et al. The natural product domain seeker NaPDoS: a phylogeny based bioinformatic tool to classify secondary metabolite gene diversity. PLoS One. 7 (3), 34064 (2012).
- Rausch, C., Weber, T., Kohlbacher, O., Wohlleben, W., Huson, D. H. Specificity prediction of adenylation domains in nonribosomal peptide synthetases (NRPS) using transductive support vector machines (TSVMs). Nucleic Acids Res. 33 (18), 5799-5808 (2005).
- Röttig, M., et al. NRPSSpredictor2--a web server for predicting NRPS adenylation domain specificity. Nucleic Acids Res. 39, 362-367 (2011).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nat. Chem. Biol. 11 (9), 625-631 (2015).
- Mullis, K., et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb. Symp. Quant. Biol. 51, 263-273 (1986).
- Bierman, M., et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp. Gene. 116 (1), 43-49 (1992).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli.using the heat shock method. J. Vis. Exp. (6), e253 (2007).
- Bimboim, H. C., Doly, J. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Res. 7 (6), 1513-1523 (1979).
- MacNeil, D. J., et al. Analysis of Streptomyces avermitilis. genes required for avermectin biosynthesis utilizing a novel integration vector. Gene. 111 (1), 61-68 (1992).
- Pospiech, A., Neumann, B. A versatile quick-prep of genomic DNA from Gram-positive bacteria. Trends Genet. 11 (6), 217-218 (1995).
- Makitrynskyy, R., et al. Pleiotropic regulatory genes bldA, adpA and absB are implicated in production of phosphoglycolipid antibiotic moenomycin. Open Biol. 3 (10), 130121 (2013).
- Kalan, L., et al. A cryptic polyene biosynthetic gene cluster in Streptomyces calvus is expressed upon complementation with a functional bldA gene. Chem. Biol. 20 (10), 1214-1224 (2013).
- Gessner, A., et al. Changing Biosynthetic Profiles by Expressing bldA in Streptomyces Strains. ChemBioChem. 16 (15), 2244-2252 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados