Method Article
에서 Polyketomycin를 사용하여 데모 : 그 생합성 유전자 클러스터에 천연 제품에서
요약
다음은 (A) 항균 활성을 갖는 천연 제품의 식별, (B) 화합물의 정제 (C)의 생합성의 첫 모델 (D) 게놈 시퀀싱 / -mining 및 상세한 프로토콜을 제시 ( E) 생합성 유전자 클러스터의 확인.
초록
스트렙토 균주 다양한 생리 활성을 가진 다른 화합물을 많이 생성하는 그들의 능력에 대해 공지되어있다. 상이한 조건에서 배양들은 신규 화합물의 생산 효율을 높일 수있다. 따라서, 생산 균주의 배양 물을 에틸 아세테이트로 추출하고, 조 추출물을 HPLC에 의해 분석된다. 또한, 추출물 다른 분석하여 자신의 생체 활성에 대해 시험한다. 구조 해명 들어 관심의 화합물은 다른 크로마토 그래피 방법의 조합에 의해 정제한다.
게놈 마이닝 결합 게놈 서열은 다른 컴퓨터 프로그램을 사용 천연물 생합성 유전자 클러스터의 식별을 허용한다. 유전자 비활성화 실험을 수행 할 수 있고, 정확한 유전자 클러스터가 발견되었음을 확인한다. 생성 된 돌연변이 체는 특히 천연 제품의 생산에 대해 분석한다. 정확한 유전자 클러스터가 비활성화되면,균주의 화합물을 제조하는 데 실패한다.
워크 플로는 스트렙토 마이 세스 (Streptomyces) diastatochromogenes Tü6028에 의해 생성 된 항균 화합물 polyketomycin에 대해 표시됩니다. 게놈 시퀀싱은 여전히 매우 비싼 때 약 10 년 전, 유전자 클러스터의 복제 및 식별은 매우 시간이 많이 소요되는 과정이었다. 게놈 마이닝과 함께 빠른 게놈 시퀀싱 클러스터 식별 재판을 가속화하고 새로운 방법 생합성을 탐구하고 유전 적 방법으로 새로운 천연 제품을 생성하기 위해 열립니다. 이 논문에 기재된 프로토콜은 방선균 또는 다른 미생물 유래의 다른 화합물에 할당 될 수있다.
서문
식물과 미생물로부터 천연 제품은 항상 임상 약물 개발 및 연구를위한 중요한 원천이었다. 최초의 항생제 페니실린은 알렉산더 플레밍 (1)에 의해 곰팡이에서 1928 년에 발견되었다. 요즘, 많은 천연 제품은 임상 치료에 사용된다.
다른 생리 활성 이차 대사 산물의 다양한 종류를 생산하는 자사의 능력에 대해 알려진 하나 속 스트렙토 마이 세스입니다. 스트렙토 마이 세스 (Streptomyces)은 그람 양성 세균과 방선균의 클래스와 순서 방선균 목에 속한다. 임상 적으로 사용되는 항생제의 거의 3 분의 2는 3 테트라 사이클린 4 daptomycin, 암포 테리 신 2와 같이 주로 스트렙토 마이 세스 (Streptomyces)에서, 방선균 목에서 파생됩니다. 두 노벨상은 스트렙토 마이 세스 (Streptomyces) 항생제 연구의 분야에서 수상했습니다. 첫번째는 스트렙토 마이신의 발견은 먼저 대한 셀만 브와 왁스 갔다결핵에 대한 효과적인 tibiotic. 5 2015 년, 생리학 및 의학 노벨상의 일환으로, S.의 avermitilis에서 avermectin에의 발견은 물론 수상했다. avermectin에 기생 -6,7- 질환의 치료에 사용된다.
예컨대 스트렙토 같은 미생물 천연 제품의 발견에 대한 전통적인 접근은 일반적으로 다른 성장 조건 하에서 배양 균주뿐 아니라 추출 및 이차 대사 산물의 분석을 포함한다. 생체 활성 분석 (항균 항암 활성에 대한 분석은 예) 화합물의 활성을 검출하기 위해 수행된다. 마지막으로, 관심있는 화합물을 분리하였고, 화학 구조가 밝혀져있다.
천연물의 구조는 종종 복잡한 분자 형성되어 단일 부분으로 구성된다. 블록 건물로 이어지는 몇 가지 있지만, 제한, 주요 생합성 경로가 있습니다자연 산물의 생합성에 사용되는 KS. 주요 생합성 경로는 설탕 부분으로 이어지는 폴리 케 티드 경로, 아미노산을 사용 테르 페 노이드 및 알칼로이드, 경로으로 이어지는 경로 및 경로입니다. 각 경로는 특정 효소 (8)의 집합을 특징으로한다. 화합물의 구조를 기반으로 이러한 생합성 효소가 예측 될 수있다.
오늘날, 차세대 시퀀싱 및 생물 정보학적인 분석과 함께 화합물의 상세한 구조 분석을 담당 생합성 유전자 클러스터를 식별 할 수있다. 클러스터 정보는 더 자연 제품 연구에 대한 새로운 방법을 연다. 이 유전자 삭제 또는 변경 및 기타 경로에서 유전자 조합 생합성에 의해 천연 제품의 수율, 목표 화합물의 변형을 증가시키는 이종 표현을 포함한다.
Polyketomycin은의 배양액으로부터 독립적으로 분리 하였다두 균주 스트렙토 마이 세스 특검팀. MK277-AF1 9 스트렙토 마이 세스 (Streptomyces) diastatochromogenes Tü6028 10. 구조는 NMR 및 X 선 분석에 의해 밝혀졌다. Polyketomycin는 β-D-amicetose와 α-L-axenose 두 디 옥시 당 부분으로 연결하는 테트라 decaketid 및 다이 메틸 살리실산으로 구성되어있다. 그것은 심지어 MRSA (11) 그람 양성 다제 내성 균주에 대해, 세포 독성 항생제 활동을 표시합니다.
S. diastatochromogenes Tü6028의 게놈 코스 미드 라이브러리를 생성하고 몇 년 전에 상영되었다. (41) 유전자를 포함, 52.2 킬로바이트의 크기로 polyketomycin 유전자 클러스터를 특정 유전자를 프로브를 사용하여 강렬한 작품 12의 몇 달 후 확인되었다. 최근, S.의 diastatochromogenes 초안 게놈 서열은 polyketomycin 생합성 유전자 클러스터의 빠른 확인 선도 얻었다. 이러한 개요에서, 방법은 아이덴 돕는천연물을 tify 및 생합성 유전자 클러스터는 예를 들어 polyketomycin하여 설명한다 명료.
여기에서 우리는 스트렙토 마이 세스 (Streptomyces) diastatochromogenes Tü6028에 의해 생성 polyketomycin에 대해 도시의 생합성 유전자 클러스터에 천연 제품에서 이어지는 하나의 단계를 설명합니다. 프로토콜은 항생 물질 특성을 갖는 천연 생성물의 동정 및 정제를 포함한다. 또한 구조 분석 및 생합성 유전자 클러스터의 식별에 게놈 마이닝 리드의 결과와 비교. 이 절차는 방선균 또는 다른 미생물 유래의 다른 화합물에도 적용 할 수있다.
프로토콜
항생제 속성을 가진 천연 제품 1. 확인
- 은 "OSMAC (하나의 변형 많은 화합물) 방식"(13) 다음 조건이 다른 미생물 (예를 들면 시간, 온도, pH, 매체)를 배양. 화합물의 생산이 관찰 된 하나의 매체를 선택한다.
- 다음과 같은 조건에서 스트렙토 마이 세스 (Streptomyces) diastatochromogenes Tü6028 육성
- MS 판에 스트렙토 마이 세스 (Streptomyces) diastatochromogenes Tü6026 변형 성장 (대두 가루 20g, D- 만니톨 20g,의 MgCl 2 10 밀리미터, 1.5 % 한천, 수돗물 1 L). 나선형으로 삼각 플라스크에, 작은 20 mL의 액체 TSB 배지에이 균주의 포자의 루프 (예비 배양 매체 트립신 콩 국물 30g, 수돗물 1 L, pH를 7.2)을 접종한다. 회전 진탕 (28 ° C, 180 rpm으로 2 일)에 플라스크를 흔들어.
- 100 mL의 HA 배지 (포도당 4g, 효모 추출물 4g, 맥아의 주요 문화를 접종예비 배양 2 mL의 10g, 수돗물 1 L, pH를 7.2) 압축을 풉니 다. 문화 180 rpm으로 회전 진탕 6 일 동안 28 ° C에서 변형.
- 조 추출물의 제조
- 원심 분리하여 수확 세포 (10 분, 3000 XG).
- 다음 단계는 흄 후드 아래에 유기 용매를 처리합니다.
- 균사체에서 화합물의 추출,에 resuspend 아세톤의 두 가지 볼륨 세포와는 30 분, 180 rpm으로의 튜브에 흔들. 상업적 여과지에 액을 여과하고, 40 ℃, 550 바에서 회전 증발기로 아세톤을 증발시켰다. 아세트산 에틸 20 ㎖의 물에 녹인 추출물 (1 : 1), 180 rpm으로 30 분 동안 분별 깔대기에서 흔들어.
- 배양 배지에서 화합물 추출을 위해, 1 M HCl을 첨가하여 pH를 4.0으로 배양액을 조정한다. 100 mL의 에틸 아세테이트를 추가하고, 30 분, 180 rpm으로의 분리 깔때기에 흔들어.
- 에틸 아세테이트 상을 수집하고 부패에 의해 증발 40 ° C와 240 바에서 진 증발.
- HPLC에 의해 조 추출물의 분석
- 1 mL의 메탄올에 녹인 추출물을 0.45 ㎛의 공극 크기의 필터를 통해 필터링하고, 고속 액체 크로마토 그래피 (HPLC) (14)을 실행.
- polyketomycin의 경우, C18의 프리 컬럼 (4.6 × 20 ㎜ × 2) 및 C18 컬럼 (4.6 × 100 ㎜ × 2)으로 HPLC 시스템을 장비. H 20 % 내지 95 %까지의 아세토 니트릴 + 0.5 % 아세트산의 선형 구배를 사용하여 2 O + 0.5 % 아세트산 (유량 : 0.5 mL의 최소 -1).
주 : Polyketomycin는 25.9 분의 체류 시간을 갖는다 (도 1a 참조). 자외선 / 마주 스펙트럼은 262 nm 내지 359 nm에서 242 nm의, 282 nm의, 446 nm의 최소값에서 최대가 (그림 1B 참조). 부정적인 잠정 863.2 [MH]의 질량에서 - (그림 1C 참조) 검출입니다.
"SRC ="/ 파일 / ftp_upload / 54952 / 54952fig1.jpg "/>
그림 1 : Polyketomycin의 LC / MS 분석. 육일에 대한 스트렙토 마이 세스 (Streptomyces) diastatochromogenes Tü6028의 재배 후 조 추출물의 (A) HPLC 크로마토 그램 (λ = 430 ㎚). Polyketomycin은 / 힘 스펙트럼 Polyketomycin 25.9 분 (B) UV의 체류 시간을 가진다. 음의 잠정의 Polyketomycin의 (C) 질량 스펙트럼. m / z 863.2와 주 피크 [MH] -. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 항생제 활성 식별 디스크 확산 분석을 이용하여
- 그람 양성 세균 바실러스 서브 틸리 스 (Bacillus subtilis) 등의 시험 균주를 접종 (LB 배지, LB 20g, 수돗물 1 L, pH를 7.4) TSB 배지에서 다른 스트렙토 마이 세스 (Streptomyces) 균주 (; 트립신 콩 국물 30g, 수돗물 1 L, pH가 70.2), 그람 음성 세균 대장균 LB 배지에서 대장균 () 및 YPD 매체와 같은 미디어에 곰팡이 균주 (효모 추출물 10g, 펩톤 20g, 포도당 20g, 탭 물 1 L, pH를 7.4). 시험 균주 예비 배양의 100 μL를 가지고 각각의 한천 플레이트에 그들을 확산.
- 무균 종이 디스크에 메탄올 (또는 물 또는 DMSO) 및 피펫 20-50 μL의 500 μL의 조 추출물 또는 정제 된 화합물을 녹여. 워크 벤치에서 30 분 동안 건조 종이 디스크 및 시험 문화와 함께 접시에 넣어. 음성 대조군 (용매)과 양성 대조군을 준비 (항생제, 예를 들어, 아 프라 마이신 [1 밀리그램).
- 시험 균주가 눈에 띄게 성장 될 때까지 적절한 조건에서 번호판을 품어 명백한 경우, 억제 영역을 결정합니다. 대장균 및 바실러스 SP를 품어. 16 시간 동안 37 ° C, 스트렙토 마이 세스 특검팀에서. 2-4일 28 ° C에서, 곰팡이 균주 (정확한 시험 균주에 주로 의존)을t 2 일간 30 °의 C).
화합물 2. 대규모 추출, 정제 및 구조 해명
- (물 1 L, pH가 7.2을 누른 포도당 4g, 효모 추출물 4g, 맥아 추출물 10g) 5 L의 HA 매체에 S.의 diastatochromogenes 육성. 150 mL의 HA 매체를 포함하는 30 X 500 mL의 삼각 플라스크에 예비 배양 2 mL를 접종. 180 rpm으로 회전 진탕 6 일 동안 28 ° C에서 긴장을 품어.
- 원심 분리에 의해 수확 된 세포 (10 분, 5000 XG, RT)과 에틸 아세테이트를 사용하여 추출 화합물 (1.3 장 참조).
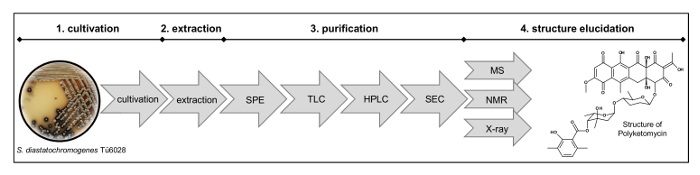
그림 2 : 구조 해명에 대한 워크 플로우. 프로세스는 변형 (1)의 배양 (2) 추출, 고체 상 추출 (SPE), 박층 크로마토 그래피 (TLC), 분 취용 고성능 액체 크로마토 그래피 (HPLC)의 크기에 의해 (3) 정제 포함질량 분석 (MS), 핵 자기 공명 (NMR) 및 X 선 측정에 의한 배제 크로마토 그래피 (SEC), (4) 구조 해명. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- Polyketomycin의 정제
참고 : 정제 및 구조를 해명하는 과정은 그림 2에 표시됩니다.- H 2 O 30 % 내지 100 %의 MeOH 범위의 10 % 메탄올 구배 -stepwise 각 조건에 대해 100 ㎖를 사용하여 C18 고체상 추출 (SPE) 컬럼에 의해 조 추출물 분획.
- (1 7)로서 용출 시스템 분취 박층 크로마토 그래피 (TLC) (15) 사용을 CH2Cl2 / 메탄올에 의해 상기 화합물을 함유하는 분획을 정제한다.
- 제조용 HPLC (16)에 의해 화합물을 정제 하였다. 와; C18의 프리 컬럼 (9.4 × 20 μm의 5 ㎜)으로 HPLC 시스템을 착용주요 열 (5 μm의, 9.4 × 150 mm)이다. H 20 % 내지 95 % O 2 + 0.5 % 아세트산 징 아세토 니트릴 + 0.5 % 아세트산의 구배를 사용하는 (유속 : 2.0 mL / 분).
- 용매 및 다른 작은 불순물을 제거하고, 메탄올 (17)의 열을 이용하여 크기 배제 크로마토 그래피에 의해 최종 정제 단계를 수행한다. 최종 순수한 화합물을 수집하고 40 ° C와 240 바에서 회전 증발하여 메탄올을 증발.
- 구조 해명
- DMSO-d를 6 (기계에 따라 다름) 600-700 μL의 순수한 화합물 (이상 2 mg)을 기록 1D NMR (1 H, 13 C) 및 2D NMR (HSQC, HMBC, 1 H-1 H를 녹이고 NMR 분광계 (18)에 COSY) 스펙트럼. DMSO-d를 6을 사용하여 δ 값 (PPM) 화학 변화를 표현한다.
- 고해상도 질량 스펙트럼 (HRESI-MS) 고해상도를 사용 polyketomycin 19을 녹음질량 분석기.
- NMR 및 MS 데이터 분석 (10)의 결과 해석에 의해 구조를 해명.
3. 새 격리 된 화합물의 생합성 모델 제안
- 단리 된 화합물의 구조를 분석하고 그 생합성에 관여 할 수있는 효소를 예측한다. 폴리 케 티드 (타입 I, II 또는 III), 비 - 리보솜 펩티드 합성, lantipeptide, 테르 페 노이드, 또는 당 대사 경로 (8)에 할당.
- 예 polyketomycin
- 테트라 부분, 두 개의 단당류와 다이 메틸 살리실산 : 분명 하나의 부분으로 구조를 세분화.
- 잔기는로부터 유도 될 수있는 위치를 알아낼 다음 테트라 잔기는 폴리 케 티드 신타 형태로부터 유도 될 수도 II 및 디메틸 살리실산 부분은 반복 폴리 케 티드 신타 형 I. 의해 유도 될 수있는 6 deoxysugars은 두 당 잔기 , synthesi 될 수 있습니다데이빗은 생합성 동안에 TDP 글루코스 -4,6- 아제 두 글리코 의해 아마도 첨부 관련된 포도당 (도 3 참조) 8.
- 클러스터의 추정 유전자를 예측하고있다. 단위가 연결뿐만 아니라, 수정 및 분자에 맞추어 단일 부분의 합성에 관여하는 효소를 생각한다. Polyketomycin의 생합성 유전자 클러스터는 아마 (확장 장치를 연결 따라서 최소 ACP, KS α 싶게 KS β)가 폴리 케 티드 합성 효소 타입 II를 코딩하는 유전자를 포함하는 반복적 인 폴리 케 티드 신타 아제 타입 I (적어도 ACP, AT, KS)와, TDP 글루코스 -4,6- 아제 (단계 필요한 : 글루코스 → 글루코오스) 두 글리코 (어글리 콘 두 당 단량체 부착) 8.
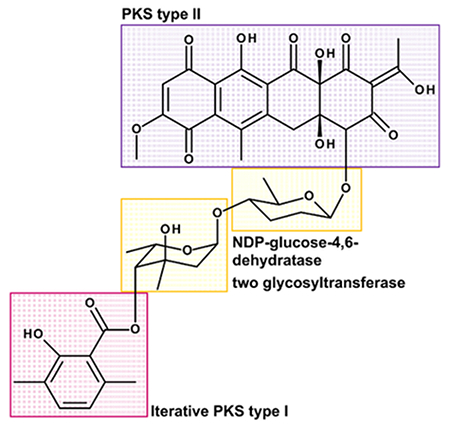
그림 3 : Polyketomycin divId는의 구조단일 빌딩 블록으로 에드. Polyketomycin은 β-D-amicetose 및 α-L-axenose (NDP 포도당 - 4,6- 개의 디 옥시 당 잔기가 결합하는 테트라 decaketid (PKS 유형 II) 및 다이 메틸 살리실산 (반복 PKS 타입 I)로 구성된다 ) 탈수와 두 개의 글 라이코 실 트랜스퍼 라제이 필요합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. 게놈 시퀀싱 / 광업
- 차세대 시퀀싱
- Illumina 사, 454 pyrosequencing 또는 고체 (20)와 같은 차세대 시퀀싱 기술에 의해 게놈 DNA 시퀀스. 하나의 정렬 기준 순서로 읽거나 새로이 조립한다.
참고 : S. diastatochromogenes Tü6028의 게놈은 빌레펠트 대학의 생명 공학 센터 (CeBiTec)에서 염기 서열했다. 모든 초안 게놈에 조립 된 읽기7.9 메가의.
- Illumina 사, 454 pyrosequencing 또는 고체 (20)와 같은 차세대 시퀀싱 기술에 의해 게놈 DNA 시퀀스. 하나의 정렬 기준 순서로 읽거나 새로이 조립한다.
- 게놈 마이닝
- 예를 NCBI 원핵 생물 게놈 주석 파이프 라인 (21, 22)에 대해, RAST는 23,24,25, Prokka (빠른 원핵 생물 게놈 주석) 26 GenDB 27 (빠른 주석이 서브 시스템 기술을 사용)의 사용에 의해 오픈 리딩 프레임 (ORF를) 검색합니다. 이 프로그램은 또한 기능을 제안한다. S.의 분석은 7,000 개의 ORF의 식별을 주도 Tü6028 초안 게놈 시퀀스를 diastatochromogenes.
- 실행 특정 BLAST (기본 로컬 맞춤 검색 도구) 분석은 다른 유사한 유전자와 촉매 도메인 28,29,30와 정렬 같은 더 많은 정보를 얻을 수 있습니다.
- antiSMASH 31,32,33, NaPDoS 34 NRPSpredictor 35, 36 등의 보조 대사 유전자 클러스터 실행 프로그램의 식별하십시오. 스트렙토 마이 세스 (Streptomyces) diastatochromogenes antiSMASH 31,32,33 식별자의 초안 게놈에서22 유전자 클러스터를 얘기들이었다.
- 이들 효소 경로 (들)에 대한 추정되는 유전자 클러스터 (폴리 케 티드 신타 아제 (타입 I, II 또는 III), 비 - 리보솜 펩티드 신타 제, lanthipeptide, 테르 페 노이드, 당 대사 ...)을 분석. 화합물의 합성에 관여 할 수있는 유전자를 포함하는 클러스터 (들)의 검색 (섹션 3 참조). S.의 diastatochromogenes에서 주석 클러스터 2는 폴리 케 티드 신타 제 II 형 유전자 세 폴리 케 티드 신타 아제 타입 I 유전자하는 TDP 글루코스 -4,6- 데 히드라 타 아제 유전자와 두 전이 효소 유전자 (도 4 참조)을 포함한다.
- 클러스터 내의 단일 유전자에 초점을 맞 춥니 다. PKS 타입 I 및 NRPS acyltransferases 및 adenylation 도메인의 특이성, 따라서 하나의 확장 유닛의 혼입 예측 될 수있다. 또한 분자로 혼입 확장 유닛의 순서를 비교한다. antiSMASH 31,32,33 또한 MIBiG 데이터베이스 (37)에 대한 링크와 함께, 이미 공지 된 화합물의 유사한 클러스터를 보여줍니다.
- 이러한 다른 화합물과 화합물의 구조를 비교하고 유사성을 확인합니다.

그림 4 : S.의 Polyketomycin 생합성 유전자 클러스터 및 기타 클러스터의 개요 antiSMASH 출력 Tü6028을 diastatochromogenes. (A)는 S.의 게놈의 예상 생합성 유전자 클러스터의 개요는 Tü6028 diastatochromogenes; 표적 유전자 (B)이 클러스터 Polyketomycin 생합성 유전자 클러스터. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
생합성 유전자 클러스터 5. 확인
- 화합물의 생합성에 대한 분명하고 중요한 (필수) 기능을 가진 유전자를 검색합니다. polyketo의 검증을위한PKS 타입 II에서 ketosynthase의 α를 코딩하는 유전자 pokPI 마이신 유전자 클러스터는, 프레임 -deletion의 출력 (도 5b 참조)에 의해 선택된 불 활성화시켰다. 대안 적으로, 내부 단편을 클로닝하여 유전자를 인터럽트 (도 5c 참조).
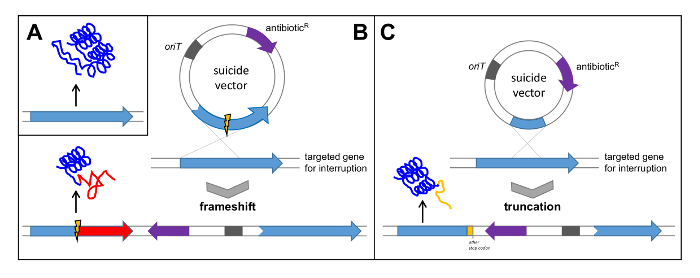
그림 5 : 단일 크로스 오버에 의한 유전자 클러스터의 확인. (A) 네이티브 유전자는 기능성 단백질의 번역을 유도; 자살 벡터로 내부 삭제와 유전자의 (B) 복제는 대상 유전자 및 비 기능성 단백질의 후속 번역의 프레임 변화의 결과로 하나의 크로스 오버로 연결; 자살 벡터로 유전자의 내부 단편 (C) 복제는 유전자와 비 기능성 단백질의 후속 번역의 절단에 이르게. 기술 Orit : t의 기원를 ransfer; 항생제 R : 항생제 내성. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 단일 교차 구조의 클로닝
- 프라이머 pokPI_for 및 pokPI_rev와 PCR (38)에 의해 pokPI을 포함하는 단편을 증폭.
- 자살 벡터 내로 클론 PCR 생성물 pKC1132 39 (R 아 프라 마이신 [50 ㎎ / ㎖). 자살 벡터는 방선균에서 복제 할 수없고, 따라서 아 프라 마이신 내성을 제공하는 상 동성 재조합에 의해 표적 유전자에 통합한다. 열 충격 변환 (40)에 의해 호스트를 복제 대장균에 벡터를 전송합니다.
- 알칼리 용해 (41)에 의해 벡터를 분리합니다.
- 프래그먼트 내 절단 한 커터 제한 효소와 벡터 DNA를 소화. pokPI 세대E는 분해시켰다 효소 KPN의 I.
- 5 '→ 3'중합 효소 활성 및 3 '→ 5'엑소 뉴 클레아 활동이 큰 DNA 중합 효소 I (클레 나우) 조각으로 분해 벡터 DNA를 취급합니다. 끈적 끈적한 끝의 둔화 이후 재결합 네 기지의 손실로 연결됩니다. 자신의 플라스미드 DNA, (41)을 분리하고 제한 소화 및 시퀀싱에 의해 더 분석, 하나의 식민지를 따기, 대장균 XL1 블루로 단계를 변환하여 이러한 기지의 손실을 확인합니다.
- 단일 크로스 오버의 활용 스트렙토 마이 세스 (Streptomyces)로 구성
- 열 충격 변환 (40)에 의해 대장균 ET12567 pUZ8002 42 (카나마이신 R)에 삭제와 pokPI 유전자 (pKC1132_ pokPI 델)을 포함하는 자살 벡터를 전송합니다. 100 mL의 LB 배지에서 세포를 품어 (카나마이신 50 μg의 용액 -1, 아 프라 마이신 50 μg의 용액 -1 ) 하룻밤에 37 ° C. 원심 분리하여 수확 세포 (3,000 XG, 10 분, 4 ° C) 및 50 mL의 LB 배지에서 재 부유에 의해 두 번 세포를 씻으십시오. 마지막으로, LB 미디어의 2 × 500 μL에에 resuspend 세포.
- 믹스 500 μL 대장균 pUZ8002 pKC1132_ pokPI 델 500 μL 스트렙토 마이 세스 (Streptomyces) diastatochromogenes 문화 (또는 포자를 사용). MS는 접시에 혼합물 (대두 가루 20g, D- 만니톨 20g,의 MgCl 2 10 밀리미터가 1.5 % 한천, 수돗물 1 L)을 확산. 28 ° C에서 20 시간 동안 번호판을 품어.
- 아 프라 마이신 (1.25 mg)을 물 1 mL에 용해 fosfomycin (5 mg)을 각 플레이트를 오버레이하고 건조 할 수 있습니다. exconjugants이 표시 될 때까지 28 ° C에서 며칠 동안 번호판을 품어.
- 단일 교차 돌연변이를 확인
- 20 ㎖ TSB 배지 (50 mg의 용액 아 프라 마이신 -1)에서 단일 exconjugants 접종 사흘 180 rpm으로 28 ° C에서 그들을 품어.
- 게놈 DNA (43)을 분리 및 PCR에 의해 pokPI 유전자의 중단을 확인합니다.
- 100 mL의 HA 매체에서 확인 된 유전자 중단 및 스트렙토 마이 세스 (Streptomyces)의 야생형 균주와 단일 클론을 접종하고 28 ° C, 180 rpm에서 육일 그들을 품어. 조 추출물 (섹션 1.3 참조) 압축을 풉니 다. HPLC-MS 분석에 의해 화합물 생산을 확인합니다 (섹션 1.4 참조). HPLC 크로마토 그램의 피크와 대응하는 화합물의 질량이 더 이상 검출이어야한다 (도 6 참조).
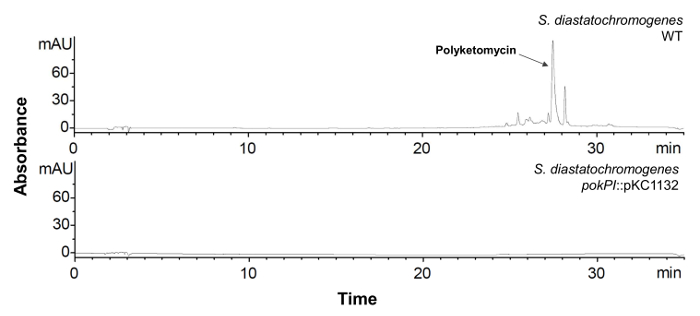
그림 6 : 비활성화 된 pokPI 유전자와 S.의 diastatochromogenes의 HPLC 분석. S.의 조 추출물의 HPLC 크로마토 그램 (λ = 430 nm의)는 중단 pokPI -gen (아래)와 WT (위)와 돌연변이 균주를 diastatochromogenes. 돌연변이 균주는 더 이상 polyketomycin 생성하지 않습니다.이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
이 개요에서 우리는 그 생합성 유전자 클러스터에 이르는 항생제의 식별에서 하나의 단계를 설명합니다. 몇 년 전 우리가 파지로 그들을 패키지 코스 미드 라이브러리를 복제, 대장균 숙주 세포를 형질 도입하고, polyketomycin 클러스터의 중복 영역을 갖는 클론을 식별하기 위해 식민지의 수천을 선별했다. 코스 미드의 시퀀싱도 어렵고 비용 처리 (12)이었다.
변형에 대한 추가 연구를 수행하기 위해 우리는 스트렙토 마이 세스 (Streptomyces) diastatochromogenes Tü6028의 전체 게놈 염기 서열. 초안 게놈 시퀀스를 통해 우리가 쉽게 polyketomycin 유망한 화합물을 인코딩 다른 클러스터의 생합성 유전자 클러스터를 확인했다. 도 7은 코스 미드 라이브러리 스크리닝 정교한 클로닝하여 생합성 클러스터를 식별하는 "이전"방법을 비교하고,거친 시간 척도에 후속 게놈 마이닝과 전체 게놈 시퀀싱에 의해 "새로운"방법. 새로운 시퀀싱 기술 및 새로운 게놈 마이닝 프로그램은 전체 프로세스의 속도.
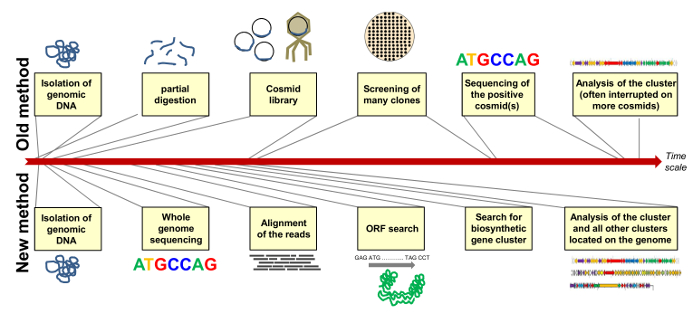
그림 7 : 생합성 유전자 클러스터 할당의 "이전"과 "새로운"방법의 비교. "오래된"방법은 양성 클론의 선택과 각 코스 미드 (들)의 시퀀스와 코스 미드 라이브러리의 클로닝 (위)와; "새로운"방법은 전체 게놈 시퀀싱 및 변형 (아래)의 게놈에있는 모든 보조 대사 유전자 클러스터를 식별하기 위해 -mining를 포함한다. 단일 단계의 소요 시간은 대략적인 시간 스케일에 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
이 실험에서는 스트렙토 diastatochromogenes 게놈의 코스 미드 라이브러리를 생성하고 매우 시간 소모적 인 과정을 통해 polyketomycin 유전자 클러스터의 식별 결과 오래전 스크리닝 하였다. 단일 유전자의 특성은 타겟 유전자 삭제하고, 생성 된 돌연변이 체 (12)의 분석을 통해 가능했다. 최근, S.의 diastatochromogenes 초안 게놈 서열은 polyketomycin 생합성 유전자 클러스터의 빠른 확인을 가능하게 얻었다. 초안 게놈 시퀀스는 여전히 많은 콘티가 포함되어 있지만, 우리는 쉽게 생합성 유전자를 검출 할 수있다. 설명 된 과정은 개월 이내에 달성 될 수있다. 그러나, 절차는 다수의 단계를 포함한다. 단일 단계는 다음 단계로의 진행을 방지하는 여러 번 실패 할 수있다 :
속은 스트렙토 생리 활성 화합물을 생성하는 능력에 대해 공지되어있다. 그들은 종종 20 개 이상의 바이오스를 수행하는 동안일반적으로 하나 또는 두 화합물은 실험실 조건에서 일어난다 ynthetic 유전자 클러스터. 침묵 유전자 클러스터를 깨울 수있는 OSMAC 방식 (다른 조건에서 하나의 균주의 배양)의 적용은 때로는 충분하지 않을 수 있습니다. 예다면 발현 성 조절 유전자 ADPA 44, 45, 46 등 bldA 조절 유전자의 유전자 조작은, 다른 이차 대사 산물의 생산을 활성화 할 수있는 효과적인 방법이다.
NMR 분석에 의해 예 화합물의 구조 규명을 위해, 정제 된 화합물을 일반적으로 2 개 이상의 밀리그램이 필요하다. 따라서, 10 개 이상의 L 문화의 발효는 종종 필요합니다. 산소, pH 및 온도 조건을 유지 할 수있는 발효기없이, 문화 및 후속 추출이 양을 처리 할 수있는 작은 실험실에서 어려울 수 있습니다. 정제 중에, 상기 화합물은 산화에 의한 방사선 또는 온도로 변경 될 수있다.또한, 이상의 정제 단계는 높은 분해의 가능성을 사용한다.
천연 생성물의 구조 및 게놈 클러스터를 분석 할 때, 때때로 해당하는 클러스터를 식별하는 것을 용이하지 않다. 단지 초안 게놈 시퀀스가있는 경우 첫째, 클러스터의 일부가 누락 될 수 있습니다. 둘째, 생합성에 필요한 모든 유전자 클러스터에 있지. 셋째, 때때로 많은 클러스터 킬로베이스가 서로 분리 된 두 부분으로 분할된다. 넷째, 적절한 유전자 클러스터 어느 하나를 결정하기 어려울 수있다. 이 모듈의 개수에 기초하여 확장 유닛의 개수를 계산, 또는 상기 선택 영역을 분석하여 하나의 확장 유닛을 식별 할 수있다 큰 PKS 유형 I 또는 NRPS 시스템의 경우에, 쉽게집니다. 그러나, 반복적으로 작동하는 경우에, 합성 화합물의 예측 특히 STRA 경우가 종종 불가능 효소에 40 개 이상의 유전자 클러스터를 보유하고 있습니다. 다섯째, 자연은 매우 복잡하고 아직 알 수없는 화합물이 가득합니다. 종종 다른 생합성 경로의 혼합물이다. 신규 화합물은 다른 화합물에 관한 아직 식별 여부되지 않는다면 생합성 모델을 제안하고 증명 클러스터를 식별하기 어려울 수있다.
클러스터가 식별되면, 단일 크로스 오버 기술은 가정을 확인하는 것이 빠른 방법이다. PCR, 자살 벡터, 접합, 긍정적 인 클론의 선택, 생산 분석에 복제에 필요한 유일한 단계입니다. 이 기법의 단점은 염색체 내로 벡터의 통합으로 인해 상기 재조합으로 안정하지 않다는 것이다. 따라서, 상기 하나의 유전자를 분석하기 위하여, 정확한 유전자 결실이 필요하다. 또한 그것은 유전자 수준에서 스트렙토 마이 세스 (Streptomyces) 균주를 조작하는 까다로운 일이 될 수 있습니다.
설명 된 절차는 t을 할당 할 수 있습니다방선균 또는 다른 미생물에 의해 생성 된 임의의 다른 화합물을 오. 생합성 유전자 클러스터 및 합성 화합물에 대한 지식은 우리에게 다제 내성 병원균과의 싸움을 위해 그들을 향상을 목표로 기존의 분자를 수정할 수있는 추가 기회를 제공합니다.
공개
The authors have nothing to disclose.
감사의 말
S. Zhang is funded by China Scholarship Council. The authors are very grateful to former people working on polyketomycin project in this lab and Prof. Dr. Hans-Peter Fiedler, University of Tübingen, for providing the polyketomycin producer. The research was supported by the DFG (RTG 1976).
자료
| Name | Company | Catalog Number | Comments |
| agar | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 5210.4 | |
| agarose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6352.4 | |
| D-mannitol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4175.1 | |
| glucose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6780.1 | |
| LB | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X964.3 | |
| malt extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X976.2 | |
| MgCl2 | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2189.1 | |
| Peptone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | ||
| soy flour | W.Schoenemberger GmbH, Magstadt, Germany | Hensec-Vollsoja | |
| tryptic soy broth | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X938.3 | Caso-Bouillon |
| yeast extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2363.2 | |
| Solvents | |||
| Acetic acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 3738.5 | |
| Acetone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 9372.2 | |
| Acetonitrile | Avantor Performance Materials B.V., Deventer, The Netherlands | JT-9012-03 | |
| Dichlorofrom | Fisher Scientific GmbH, Schwerte, Germany | 1530754 | |
| DMSO (Dimethyl sulfoxide) | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4720.1 | |
| DMSO-d6 (Dimethyl sulfoxide-d6) 99.9 atom% D | ARMAR Chemicals, Döttingen, Switzerland | 15200.204 | |
| Ethyl acetate | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6784.4 | |
| Hydrochloric acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6331.4 | |
| Methanol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 7342.1 | |
| Enzymes | |||
| restriction enzymes | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| polymerase | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| DNA Polymerase I Large (Klenow) Fragment | Promega GmbH, Mannheim, Germany | ||
| Antibiotics | |||
| apramycin | AppliChem GmbH, Darmstadt, Germany | A7682.0005 | |
| fosfomycin | Sigma-Aldrich Chemie GmbH, Taufkirchen, Germany | P5396-50G | |
| kanamycin | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | T832.2 | |
| Plasmid/Vectors | |||
| pKC1132 | Bierman et al. 1992 | ||
| Primer | |||
| pokPI_for | TGATGGTGCCGCTGGCCATGG | Primer to amplify fragment containing pokPI gene | |
| pokPI_rev | AGCGTTCACTGTTCCGCCCGAC | ||
| Bacterial strains | |||
| Bacillus subtilis COHN ATCC6051 | Gram-positive test strain | ||
| Escherichia coli ET12567 pUZ8002 | MacNeil et al. 1992 | strain for conjugation | |
| Escherichia coli XL1 Blue | Agilient Technologies, Santa Clara, USA | Gram-negative test strain + cloning host | |
| Streptomyces diastatochromogenes Tü6028 | Paululat et al., 1999 | Polyketomycin producer | |
| Online services | |||
| antismash (Antibiotics and Secondary Metabolite Analysis Shell) | http://antismash.secondarymetabolites.org/ | Detection of secondary metabolite gene cluster | |
| BLAST (Basic Local Alignment Search Tool) | http://blast.ncbi.nlm.nih.gov/Blast.cgi | finds regions of similarity between biological sequences | |
| GenDB | https://www.uni-giessen.de/fbz/fb08/Inst/bioinformatik/software/gendb | Annotation of ORFs | |
| MiBIG (Minimum Information about a Biosynthetic Gene cluster) | http://mibig.secondarymetabolites.org/ | Database of biosyntetic gene clusters | |
| NaPDoS | http://napdos.ucsd.edu/ | Detection of seconary metabolite gene cluster | |
| NCBI Prokaryotic Genome Annotation Pipeline | http://www.ncbi.nlm.nih.gov/genome/annotation_prok/ | Annotation of ORFs | |
| NRPSpredictor | http://nrps.informatik.uni-tuebingen.de/ | Detection of NRPS domains | |
| Prokka (rapid prokaryotic genome annotation) | http://www.vicbioinformatics.com/software.prokka.shtml | Annotation of ORFs | |
| RAST (rapid annotation using subsystems technology) | http://rast.nmpdr.org/ | Annotation of ORFs | |
| Other programs | |||
| Chem Station Rev. A.09.03 | Agilent Technologies, Waldbronn, Germany | Handling program for HPLC | |
| Clone Manager Suite 7 | Scientific and Educational Software, Cary, USA | Designing Cloning Experiment | |
| Newbler v2.8 | Roche Diagnostics | Alignment of sequencing reads | |
| Machines | |||
| Centrifuge Avanti J-6000, Rotor JA-10 | Beckman Coulter GmbH, Krefeld, Germany | ||
| HPLC/MS | Agilent Technologies, Waldbronn, Germany | ||
| _Autosampler: G1313A | Agilent Technologies, Waldbronn, Germany | ||
| _Pre-column: XBridge C18 (20 mm x 4.6 mm; Particle size: 3.5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Column:Xbridge C18 (100 mm × 4.6 mm; Particle size: 3.5 μm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Pre-Column: Zorbax B-C18 (9.4 x 150 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Column: Zorbax B-C18 (9.4 x 20 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Degasser: G1322A | Agilent Technologies, Waldbronn, Germany | ||
| _Quarternary pump: G1311A | Agilent Technologies, Waldbronn, Germany | ||
| _Diode array detector (DAD )G1315B (λ = 254 nm and 400 nm) | Agilent Technologies, Waldbronn, Germany | ||
| _Quadrupole mass detector (MSD) G1946D(2-3000 m/z) | Agilent Technologies, Waldbronn, Germany | ||
| rotary evaporator | |||
| _heating bath Hei-VAP Value/G3 | Heidolph Instruments GmbH & Co.KG, Schwabach, Germany | ||
| _vacuum pump system SC 920 G | KNF Global Strategies AG, Sursee, Switzerland | ||
| Other material | |||
| Sephadex LH20 | GE Healthcare, | ||
| Chromafil PVDF-45/15MS (pore size 0.45 µm; filter Ø15 mm) | MACHEREY-NAGEL GmbH & Co. KG, Düren, Germany | ||
| SPE column Oasis HLB 20 35 cc (6g) | Waters GmbH, Eschborn, Germany | ||
| E. coli | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Bacillus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Streptomyces sp. | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: TSB, Composition: CASO Boullion, Amount to 1 L H2O: 30 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Yeast extract, Amount to 1 L H2O: 10 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Peptone, Amount to 1 L H2O: 20 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Glucose, Amount to 1 L H2O: 20 g | |
| for agar plates add 2% agar | |||
참고문헌
- Fleming, A. On the antibacterial action of cultures of a penicillin, with special reference to their use in isolation of B. influenzae. Br J Exp Pathol. 10 (3), 226-236 (1929).
- Lemke, A., Kiderlen, A. F., Kayser, O. Amphotericin B. Appl. Microbiol. Biotechnol. 68 (2), 151-162 (2005).
- Kirkpatrick, P., Raja, A., LaBonte, J., Lebbos, J. Daptomycin. Nat. Rev. Drug Discov. 2 (12), 943-944 (2003).
- Zakeri, B., Wright, G. D. Chemical biology of tetracycline antibiotics. Biochem. cell Biol. 86 (2), 124-136 (2008).
- Schatz, A., Bugle, E., Waksman, S. A. Streptomycin, a Substance Exhibiting Antibiotic Activity Against Gram-Positive and Gram-Negative Bacteria. Exp. Biol. Med. 55 (1), 66-69 (1944).
- Burg, R. W., et al. Avermectins, new family of potent anthelmintic agents: producing organism and fermentation. Antimicrob. Agents Chemother. 15 (3), 361-367 (1979).
- Egerton, J. R., Ostlind, D. A., et al. Avermectins, new family of potent anthelmintic agents: efficacy of the B1a component. Antimicrob. Agents Chemother. 15 (3), 372-378 (1979).
- Walsh, C. T., Fischbach, M. A. Natural products version 2.0: connecting genes to molecules. J. Am. Chem. Soc. 132 (8), 2469-2493 (2010).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces sp. MK277-AF1. II. Structure determination. J. Antibiot. (Tokyo). 51 (1), 26-32 (1998).
- Paululat, T., Zeeck, A., Gutterer, J. M., Fiedler, H. P. Biosynthesis of polyketomycin produced by Streptomyces diastatochromogenes.Tü6028. J. Antibiot. (Tokyo). 52 (2), 96-101 (1999).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces. sp. MK277-AF1. I. Taxonomy, production, isolation, physico-chemical properties and biological activities. J. Antibiot. (Tokyo). 51 (1), 21-25 (1998).
- Daum, M., et al. Organisation of the biosynthetic gene cluster and tailoring enzymes in the biosynthesis of the tetracyclic quinone glycoside antibiotic polyketomycin. Chembiochem. 10 (6), 1073-1083 (2009).
- Bode, H. B., Bethe, B., Höfs, R., Zeeck, A. Big effects from small changes: possible ways to explore nature's chemical diversity. Chembiochem. 3 (7), 619-627 (2002).
- Wolfender, J. -. L. HPLC in Natural Product Analysis: The Detection Issue. Planta Med. 75 (07), 719-734 (2009).
- Stahl, E. . Thin-layer chromatography. A laboratory handbook. , (1967).
- Snyder, L. R., Kirkland, J. J., Glajch, J. L. Preparative HPLC Separation. Pract. HPLC Method Dev. , 616-642 (2012).
- Granath, K. A., Kvist, B. E. Molecular weight distribution analysis by gel chromatography on sephadex. J. Chromatogr. A. 28, 69-81 (1967).
- Jackman, L. M., Sternhell, S. Application of Nuclear Magnetic Resonance Spectroscopy in Organic Chemistry. Int. Ser. Org. Chem. , (1969).
- Xian, F., Hendrickson, C. L., Marshall, A. G. High resolution mass spectrometry. Anal. Chem. 84 (2), 708-719 (2012).
- Metzker, M. L. Sequencing technologies - the next generation. Nat. Rev. Genet. 11 (1), 31-46 (2010).
- Tatusova, T., et al. Prokaryotic Genome Annotation Pipeline. NCBI Handb. 2nd Ed. , (2013).
- Angiuoli, S. V., et al. Toward an online repository of Standard Operating Procedures (SOPs) for (meta)genomic annotation. OMICS. 12 (2), 137-141 (2008).
- Aziz, R. K., et al. The RAST Server: rapid annotations using subsystems technology. BMC Genomics. 9, 75 (2008).
- Overbeek, R., et al. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST). Nucleic Acids Res. 42, 206-214 (2014).
- Brettin, T., et al. RASTtk: a modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes. Sci. Rep. 5, 8365 (2015).
- Seemann, T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Meyer, F. GenDB--an open source genome annotation system for prokaryote genomes. Nucleic Acids Res. 31 (8), 2187-2195 (2003).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. J. Mol. Biol. 215 (3), 403-410 (1990).
- Madden, T. The BLAST Sequence Analysis Tool. NCBI Handb. 2nd Ed. , (2003).
- Marchler-Bauer, A., et al. CDD: NCBI's conserved domain database. Nucleic Acids Res. 43, 222-226 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Res. 39, 339-346 (2011).
- Blin, K., et al. antiSMASH 2.0--a versatile platform for genome mining of secondary metabolite producers. Nucleic Acids Res. 41, 204-212 (2013).
- Weber, T., et al. antiSMASH 3.0 - a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Res. 43, 237-243 (2015).
- Ziemert, N., et al. The natural product domain seeker NaPDoS: a phylogeny based bioinformatic tool to classify secondary metabolite gene diversity. PLoS One. 7 (3), 34064 (2012).
- Rausch, C., Weber, T., Kohlbacher, O., Wohlleben, W., Huson, D. H. Specificity prediction of adenylation domains in nonribosomal peptide synthetases (NRPS) using transductive support vector machines (TSVMs). Nucleic Acids Res. 33 (18), 5799-5808 (2005).
- Röttig, M., et al. NRPSSpredictor2--a web server for predicting NRPS adenylation domain specificity. Nucleic Acids Res. 39, 362-367 (2011).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nat. Chem. Biol. 11 (9), 625-631 (2015).
- Mullis, K., et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb. Symp. Quant. Biol. 51, 263-273 (1986).
- Bierman, M., et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp. Gene. 116 (1), 43-49 (1992).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli.using the heat shock method. J. Vis. Exp. (6), e253 (2007).
- Bimboim, H. C., Doly, J. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Res. 7 (6), 1513-1523 (1979).
- MacNeil, D. J., et al. Analysis of Streptomyces avermitilis. genes required for avermectin biosynthesis utilizing a novel integration vector. Gene. 111 (1), 61-68 (1992).
- Pospiech, A., Neumann, B. A versatile quick-prep of genomic DNA from Gram-positive bacteria. Trends Genet. 11 (6), 217-218 (1995).
- Makitrynskyy, R., et al. Pleiotropic regulatory genes bldA, adpA and absB are implicated in production of phosphoglycolipid antibiotic moenomycin. Open Biol. 3 (10), 130121 (2013).
- Kalan, L., et al. A cryptic polyene biosynthetic gene cluster in Streptomyces calvus is expressed upon complementation with a functional bldA gene. Chem. Biol. 20 (10), 1214-1224 (2013).
- Gessner, A., et al. Changing Biosynthetic Profiles by Expressing bldA in Streptomyces Strains. ChemBioChem. 16 (15), 2244-2252 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유