Method Article
D'un produit naturel à son cluster Gène biosynthétique: Une démonstration utilisant Polyketomycin de
Dans cet article
Résumé
Nous présentons ici un protocole détaillé de (A) l'identification d'un produit naturel avec une activité antibiotique, (B) la purification du composé, (C) le premier modèle de sa biosynthèse, (D) le séquençage du génome / -mining et ( E) la vérification du groupe de gènes de biosynthèse.
Résumé
Souches de Streptomyces sont connus pour leur capacité à produire un grand nombre de composés différents avec divers bioactivité. La culture dans des conditions différentes conduit souvent à la production de nouveaux composés. Par conséquent, les cultures des souches de production sont extraites avec de l'acétate d'éthyle et les extraits bruts sont analysés par HPLC. En outre, les extraits sont testés quant à leur activité biologique par différents dosages. Pour la détermination de la structure du composé d'intérêt est purifié par une combinaison de différentes méthodes de chromatographie.
Le séquençage du génome couplé à l'extraction du génome permet l'identification d'un groupe de gènes de biosynthèse de produits naturels en utilisant des programmes informatiques différents. Pour confirmer que le groupe de gènes correct a été identifié, des expériences d'inactivation de gènes doivent être effectuées. Les mutants résultants sont analysés pour la production du produit naturel en particulier. Une fois que le groupe de gènes correct a été inactivé, lasouche devrait échouer pour produire le composé.
Le flux de travail est montré pour la polyketomycin composé antibactérien produit par Streptomyces Tü6028. Il y a une dizaine d'années, lorsque le séquençage du génome était encore très cher, le clonage et l'identification d'un groupe de gènes est un processus très chronophage. séquençage du génome rapide combiné avec l'extraction du génome accélère le procès de l'identification de cluster et ouvre de nouvelles façons d'explorer la biosynthèse et de générer de nouveaux produits naturels par des méthodes génétiques. Le protocole décrit dans le présent document peut être attribué à un autre composé dérivé d'une souche de Streptomyces ou d' un autre micro - organisme.
Introduction
Produits naturels des plantes et des micro-organismes ont toujours été une source importante pour le développement de médicaments cliniques et de recherche. Le premier Pénicilline antibiotique a été découvert en 1928 à partir d' un champignon par Alexander Fleming 1. De nos jours, beaucoup plus de produits naturels sont utilisés dans le traitement clinique.
Un genre connu pour sa capacité de produire divers types de métabolites secondaires avec différentes activités biologiques est Streptomyces. Streptomyces sont des bactéries Gram-positives et appartiennent à la classe des Actinobactéries et l'ordre actinomycetales. Près des deux tiers des antibiotiques utilisés en clinique sont dérivés de actinomycetales, principalement à partir de Streptomyces, comme l' amphotéricine 2, 3 ou daptomycine tétracycline 4. Deux prix Nobel ont été décernés dans le domaine de Streptomyces recherche sur les antibiotiques. Le premier est allé à Selman Waksman pour la découverte de la streptomycine, le premier d'untibiotic efficace contre la tuberculose. 5 En 2015, dans le cadre du Prix Nobel de physiologie et de médecine, la découverte de l' avermectine de S. avermitilis a été attribué aussi bien. Avermectine est utilisé pour le traitement des maladies parasitaires 6,7.
L'approche traditionnelle de la découverte de produits naturels tels que des micro - organismes Streptomyces implique généralement la culture de la souche dans des conditions de croissance différentes, ainsi que l' extraction et l' analyse des métabolites secondaires. Des essais de bioactivité (par exemple , des dosages pour l' activité anti - bactérienne et anti - cancéreuse) sont réalisés pour détecter l'activité du composé. Enfin, le composé d'intérêt est isolée et la structure chimique est élucidée.
Les structures des produits naturels sont souvent composées de fragments individuels qui se forment des molécules complexes. Il y a quelques, mais limitées, les grandes voies de biosynthèse conduisant à la construction de blocks, qui sont utilisés pour la biosynthèse des produits naturels. Les principales voies de biosynthèse sont les voies de polykétides, les voies menant à des terpénoïdes et des alcaloïdes, des voies en utilisant des acides aminés, et les voies conduisant à des fragments de sucre. Chaque voie est caractérisée par un ensemble d'enzymes spécifiques 8. Sur la base de la structure du composé, ces enzymes biosynthétiques peuvent être prédites.
De nos jours, l'analyse structurale détaillée d'un composé en combinaison avec le séquençage de la prochaine génération et l'analyse bioinformatique peut aider à identifier le groupe de gènes de biosynthèse responsable. Les informations de cluster ouvre de nouvelles voies pour de nouvelles recherches sur les produits naturels. Cela comprend l'expression hétérologue pour augmenter le rendement du produit naturel, la modification du composé visé par délétion du gène ou de la modification et de la biosynthèse combinatoire avec des gènes provenant d'autres voies.
On a isolé Polyketomycin indépendamment dans le bouillon de culture dedeux souches, Streptomyces sp. MK277 AF1 9 et 10 Tü6028 Streptomyces. La structure a été élucidée par analyse par RMN et aux rayons X. Polyketomycin se compose d'un decaketid tétracyclique et un acide salicylique de diméthyle, lié par les deux fragments désoxyose ß-D-amicetose et α-L-axenose. Il affiche cytotoxique et une activité antibiotique, même contre les souches multirésistantes Gram-positives telles que le SARM 11.
Une bibliothèque de cosmide génomique de S. Tü6028 a été généré et projeté il y a plusieurs années. Utilisation de gène spécifique des sondes du groupe de gènes polyketomycin avec une taille de 52,2 kb, contenant 41 gènes, a été identifié après plusieurs mois de travail intense 12. Récemment, un projet de séquençage du génome de S. diastatochromogenes a été obtenu conduisant à l'identification rapide du groupe de gènes de biosynthèse polyketomycin. Dans cette vue d'ensemble, une méthode aidant à identifier un produit naturel et élucider son groupe de gènes de biosynthèse sera décrit, en utilisant polyketomycin comme un exemple.
Ici , nous expliquons les étapes simples qui mènent à partir d' un produit naturel à son groupe de gènes de biosynthèse montré pour polyketomycin produit par Streptomyces Tü6028. Le protocole comprend l'identification et la purification d'un produit naturel dont les propriétés antibiotiques. En outre l'analyse structurelle et la comparaison avec les résultats du génome plomb minier à l'identification du groupe de gènes de biosynthèse. Cette procédure peut être appliquée à tout autre composé dérivé d'une souche de Streptomyces , ou tout autre micro - organisme.
Protocole
1. Identification d'un produit naturel avec des antibiotiques de la propriété
- Cultiver un micro - organisme dans des conditions différentes (par exemple le temps, la température, le pH, les médias) à la suite de la "OSMAC (une souche de nombreux composés) approche" 13. Sélectionner un milieu dans lequel la production d'un composé est observée.
- Cultiver Streptomyces Tü6028 dans les conditions suivantes
- Cultiver la souche Tü6026 de Streptomyces sur des plaques de sclérose en plaques (farine de soja 20 g, D-mannitol 20 g, MgCl2 10 mM, agar 1,5%, l' eau du robinet 1 L). Inoculer une petite boucle des spores de cette souche dans 20 ml de milieu TSB liquide (soja tryptique bouillon 30 g, l'eau du robinet 1 L, pH 7,2, milieu de préculture) dans une fiole Erlenmeyer avec une spirale. Agiter le flacon sur un agitateur rotatif (28 ° C, 180 rpm, 2 jours).
- Inoculer la culture principale dans un milieu HA 100 ml (4 g de glucose, extrait de levure 4 g de maltextrait 10 g, eau du robinet 1 litre, pH 7,2) avec 2 ml de la préculture. Culture de la souche à 28 ° C pendant 6 jours sur un agitateur rotatif à 180 tours par minute.
- Préparation de l'extrait brut
- Récolte des cellules par centrifugation (10 min, 3000 xg).
- Pour les prochaines étapes manipuler des solvants organiques sous une hotte.
- Pour l'extraction du composé à partir du mycélium, remettre les cellules dans un double volume d'acétone et agiter dans un tube pendant 30 min, 180 tours par minute. Filtrer le liquide à travers un papier filtre du commerce et on évapore l'acétone à l'évaporateur rotatif à 40 ° C et 550 bars. Dissoudre l'extrait dans 20 ml d'eau: acétate d'éthyle (1: 1) et l'agiter dans une ampoule à décanter pendant 30 min à 180 rpm.
- Pour l'extraction du composé à partir du milieu de culture, ajuster le bouillon de culture à pH 4,0 par addition de HCl 1 M. Ajouter de l'acétate d'éthyle 100 ml et l'agiter dans une ampoule à décanter pendant 30 min, 180 rpm.
- Recueillir la phase d'acétate d'éthyle et évaporer par la pourriture évaporation ary à 40 ° C et 240 bar.
- L' analyse de l'extrait brut par HPLC
- Dissoudre les extraits dans 1 ml de MeOH, filtrer à travers un filtre de taille de 0,45 um de pores et exécuter la chromatographie liquide à haute performance (CLHP) 14.
- Dans le cas de polyketomycin, d' équiper le système HPLC avec une précolonne C18 (4,6 x 20 mm 2) et une colonne C18 (4,6 x 100 mm 2). Utiliser un gradient linéaire d'acétonitrile + acide acétique à 0,5% allant de 20% à 95% en H (débit: 0,5 ml min -1) d'acide acétique 2 O + 0,5%.
NOTE: Polyketomycin a un temps de rétention de 25,9 min (voir la figure 1A). Le spectre UV / vis présente des maxima à 242 nm, 282 nm, 446 nm et minima à 262 nm et 359 nm (voir figure 1B). Dans le mode négatif , une masse de 863,2 [MH] - est détectable (voir la figure 1C).
"Src =" / files / ftp_upload / 54952 / 54952fig1.jpg "/>
Figure 1: LC / MS Analyse des Polyketomycin. (A) Chromatogramme CLHP (λ = 430 nm) de l'extrait brut après la culture de Streptomyces Tü6028 pendant 6 jours. Polyketomycin a un temps de rétention de 25,9 min (B) , UV / vis des spectres de Polyketomycin. (C) Les spectres de masse de Polyketomycin dans le mode négatif. Pic principal avec m / z 863,2 [MH] -. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Identification de l'activité antibiotique utilisant un test de diffusion sur disque
- Inoculer souches d'essai comme subtilis bactéries Bacillus Gram positif (en milieu LB, LB 20 g, l' eau du robinet 1 L, pH 7,4) et d' autres souches de Streptomyces (en milieu TSB; bouillon de soja tryptique 30 g, l' eau du robinet 1 L, pH 70,2), les bactéries Gram-négatives Escherichia coli (dans du milieu LB) et des souches fongiques (dans des milieux tels que le milieu YPD, extrait de levure 10 g, 20 g de peptone, glucose 20 g, eau du robinet 1 litre, pH 7,4). Prendre 100 pi d'essai souche préculture et les répartir sur des plaques d'agar respectives.
- Dissoudre l'extrait brut ou composé purifié dans 500 pi de methanol (alternativement l'eau ou le DMSO) et pipette 20-50 pi sur des disques de papier stériles. disques de papier sec pour 30 min sous la table de travail et les mettre sur les plaques avec des cultures d'essai. Préparer un témoin négatif (solvant) et un contrôle positif (antibiotique, par exemple apramycine [1 mg]).
- Incuber les plaques dans des conditions adéquates jusqu'à ce que les souches d'essai sont visiblement cultivés et déterminer la zone d'inhibition, si apparente. Incuber E. coli et Bacillus sp. à 37 ° C pendant 16 h, Streptomyces sp. à 28 ° C pendant 2-4 jours, la souche fongique (principalement dépendante de la souche de test exact) unt 30 ° C pendant 2 jours).
2. Grande Extraction d'échelle, purification et structure Elucidation du composé
- Cultiver S. diastatochromogenes dans 5 L milieu de HA (glucose 4 g, extrait de levure 4 g, extrait de malt 10 g, eau du robinet 1 L, pH 7,2). Inoculer 2 ml de la préculture en 30 x 500 flacons Erlenmeyer ml contenant 150 ml milieu HA. Incuber la souche à 28 ° C pendant 6 jours sur un agitateur rotatif à 180 tours par minute.
- cellules de récolte par centrifugation (10 min, 15 000 xg, RT) et les composés d'extrait en utilisant l'acétate d'éthyle (voir section 1.3).
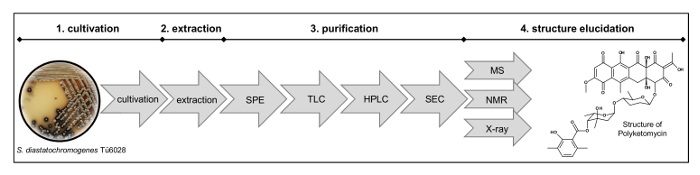
Figure 2: Flux de travail pour la structure Elucidation. Le procédé comprend (1) la culture de la souche, (2) d'extraction, (3) la purification par extraction en phase solide (SPE), la Chromatographie sur couche mince (CCM), Chromatographie liquide haute performance preparative (HPLC), la tailleChromatographie d'exclusion (SEC) et (4) détermination de la structure par analyse de masse (MS), la résonance magnétique nucléaire (RMN) et des mesures radiographiques. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Purification de Polyketomycin
Remarque: Le procédé de purification et la détermination de la structure est représentée sur la figure 2.- Fractionner l' extrait brut par une colonne C18 extraction en phase solide (SPE) en utilisant un gradient de MeOH -stepwise 10% allant de 30% à 100% de MeOH dans H 2 O, 100 mL pour chaque condition.
- On purifie la fraction contenant le composé en outre par chromatographie préparative sur couche mince (CCM) en utilisant 15 CH 2 Cl 2 / MeOH (7: 1) comme système d'élution.
- Purifier le composé par HPLC préparative 16. Équiper le système HPLC avec une précolonne C18 (5 um, 9,4 x 20 mm) etcolonne principale (5 um, 9,4 x 150 mm). (Débit: 2,0 ml / min) en utilisant un gradient d'acétonitrile + acide acétique à 0,5% allant de 20% à 95% dans l' acide acétique H 2 O + 0,5%.
- Pour éliminer les solvants et d' autres petites impuretés, effectuer une dernière étape de purification par chromatographie d'exclusion stérique en utilisant une colonne 17 dans du MeOH. Recueillir le composé pur final et évaporer MeOH un évaporateur rotatif à 40 ° C et 240 bar.
- Structure elucidation
- On dissout le composé pur (plus de 2 mg) dans 600-700 ul (dépendant de la machine) de DMSO - d6, fiche 1D RMN (1 H, 13 C) et la RMN 2D (HSQC, HMBC, 1 H- 1 H COSY) les spectres sur un spectromètre de RMN 18. Exprimer les déplacements chimiques en valeurs ô (ppm) en utilisant DMSO - d6.
- Enregistrer un spectre de masse à haute résolution (HRESI-MS) 19 de polyketomycin utilisant une haute résolutionspectromètre de masse.
- Elucider la structure par l' interprétation des résultats de l' analyse des données RMN et MS 10.
3. Proposer biosynthétique Modèle du Nouveau composé isolé
- Analyser la structure du composé isolé et prévoir des enzymes qui pourraient être impliquées dans sa biosynthèse. Attribuer leur polycétide (type I, II ou III), la synthèse des peptides non-ribosomique, lantipeptide, terpénoïde, ou le sucre métabolisme voie 8.
- Exemple polyketomycin
- Subdiviser la structure en fractions simples évidentes: groupement tétracyclique, deux monosaccharides et l'acide salicylique de diméthyle.
- Savoir, où les fragments peuvent être obtenus à partir: Le fragment tétracyclique peut être dérivé d'un type polycétide synthase II et le fragment d'acide salicylique de diméthyle peut être dérivé d'un polycétide de type synthase itérative I. Les deux fragments de sucre, qui sont de 6 désoxysucres , pourrait être SYNTHESIzed à partir de glucose comportant un TDP-glucose-4,6-déshydratase lors biosynthèse et fixé probablement par deux glycosyltransférases (voir figure 3) 8.
- Prédire gènes putatifs dans le cluster. Penser à des enzymes qui sont impliquées dans la synthèse des fragments simples, en reliant les unités, ainsi que dans la modification et l'adaptation de la molécule. Le groupe de gènes biosynthétique de Polyketomycin contient très probablement des gènes codant pour un type polycétide synthase II (donc au moins un PVA, KS α und KS β pour le raccordement des unités d'extension), une méthode itérative de type polycétide synthase I (au moins ACP, AT, KS), un TDP-glucose-4,6-déshydratase (nécessaire pour l' étape: glucose → désoxyglucose) et deux glycosyltransférases (fixation de deux monomères de sucre vers l'aglycone) 8.
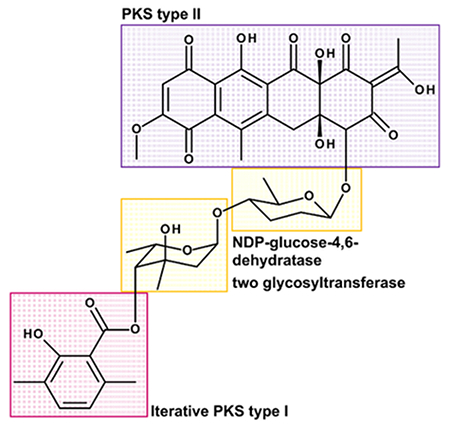
Figure 3: Structure de Polyketomycin Divided en simples blocs de construction. Polyketomycin se compose d'un decaketid tétracyclique (PKS de type II) et d'un acide salicylique de diméthyle (itératif de type PKS I), reliés par les deux groupements désoxyose β-D-amicetose et α-L-axenose (PDN-glucose-4,6- déshydratase et deux glycosyltransférase requis). S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
4. Genome Sequencing / Mines
- Séquençage de nouvelle génération
- Séquencer l'ADN génomique par les technologies de séquençage de prochaine génération comme Illumina, 454 pyroséquençage ou SOLiD 20. Alignez seul lit à une séquence de référence ou d' assembler de novo.
NOTE: Le génome de S. Tü6028 a été séquencée au Center for Biotechnology (CeBiTec) à l'Université de Bielefeld. Toutes les lectures ont été assemblés à un projet de génomede 7,9 Mb.
- Séquencer l'ADN génomique par les technologies de séquençage de prochaine génération comme Illumina, 454 pyroséquençage ou SOLiD 20. Alignez seul lit à une séquence de référence ou d' assembler de novo.
- L' exploitation minière du génome
- Rechercher des cadres de lecture ouverts (ORF) par l'utilisation de par exemple NCBI procaryotes Genome Annotation Pipeline 21,22, RAST (annotation rapide en utilisant la technologie du sous - système) 23,24,25, Prokka (rapide annotation du génome procaryote) 26 ou GenDB 27. Ces programmes proposent également leurs fonctions. L'analyse de la S. diastatochromogenes Tü6028 projet de séquençage du génome a conduit à l'identification de plus de 7000 ORFs.
- BLAST spécifique Run (Basic Local Alignment Search Tool) analyse pour obtenir plus d' informations , comme les alignements avec d' autres gènes similaires et domaines catalytiques 28,29,30.
- Pour l'identification des gènes de métabolite programmes cluster run secondaires comme antiSMASH 31,32,33, NaPDoS 34 et NRPSpredictor 35,36. Dans le projet de génome de antiSMASH 31,32,33 identi Streptomycesfiés 22 groupes de gènes.
- Analyser les groupes de gènes putatifs pour leur voie (s) enzymatique (polycétide synthase (type I, II ou III), synthétase non-ribosomique peptide, lanthipeptide, terpénoïdes, le métabolisme du sucre ...). Recherche de groupe (s) contenant des gènes qui pourraient être impliqués dans la synthèse du composé (voir section 3). Dans S. diastatochromogenes groupe annotée 2 contient polycétide type synthase gènes II, trois gènes de polycétide de type synthase I, un gène TDP-glucose-4,6-déshydratase et deux gènes de glycosyltransférase (voir Figure 4).
- Focus sur des gènes uniques au sein du cluster. PKS pour le type I et NRPS la spécificité des acyltransférases et des domaines de adénylation, et donc l'incorporation d'unités d'extension unique, peut être prédite. Vous pouvez également comparer l'ordre de l'unité d'extension incorporé à la molécule. antiSMASH 31,32,33 montre également des grappes similaires de composés déjà connus, avec un lien vers la base de données MIBiG 37.
- Comparer la structure du composé avec ces autres composés et vérifier les similitudes.

Figure 4: Sortie antiSMASH de Polyketomycin biosynthétique Gene Cluster et vue d' ensemble des autres clusters dans S. diastatochromogenes Tü6028. (A) Vue d'ensemble des groupes de gènes biosynthétiques prédites dans le génome de S. diastatochromogenes Tü6028; (B) Cluster 2 groupe de gènes de biosynthèse Polyketomycin avec des gènes ciblés. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
5. Vérification du Cluster Gène biosynthétique
- Recherche d'un gène avec des fonctions évidentes et importantes (essentielles) pour la biosynthèse du composé. Pour la vérification de la polyketogroupe de gènes mycin le pokPI gène codant pour l'α de cétosynthase du type II PKS, a été sélectionné et inactivé par un hors du cadre -deletion (voir la figure 5B). Vous pouvez également interrompre le gène par clonage d' un fragment interne (voir la figure 5C).
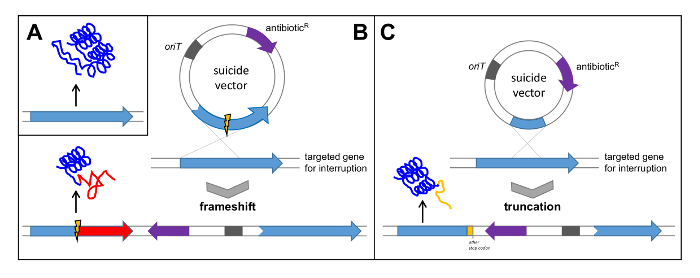
Figure 5: Vérification d'un groupe de gènes par simple Crossover. (A) gène natif conduit à la traduction d'une protéine fonctionnelle; (B) Clonage du gène par deletion interne dans un vecteur suicide conduit à un croisement entraîne un décalage de cadre dans le gène ciblé et la traduction subséquente de la protéine non fonctionnelle; (C) Clonage d'un fragment interne du gène dans un vecteur suicide conduit à une troncature du gène et la traduction subséquente d'une protéine non fonctionnelle. oriT: origine de transfert; antibiotique R: résistance aux antibiotiques. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Clonage de la construction simple croisement
- Amplifier un fragment contenant pokPI par PCR avec des amorces 38 pokPI_for et pokPI_rev.
- Produit Clone PCR dans le vecteur suicide pKC1132 39 (apramycine R [50 mg / mL]). Le vecteur de suicide ne sont pas capables de se répliquer dans la souche Streptomyces et a donc à intégrer dans le gène ciblé par recombinaison homologue pour fournir apramycine-résistance. Transférer le vecteur dans E. coli hôte de clonage par le choc transformation de chaleur 40.
- Isoler le vecteur par lyse alcaline 41.
- Digérer l'ADN vecteur avec une seule enzyme de restriction de coupe qui coupe à l'intérieur du fragment. Le pokPI genE a été digéré par enzyme Kpn I.
- Traiter l'ADN du vecteur digéré avec grande ADN polymerase I (Klenow) fragment, ce qui a 5 '→ 3' activité de polymerase et 3 '→ 5' exonucléase activité. Désaffûtant des extrémités collantes et religation subséquente conduit à la perte de quatre bases. Vérifier la perte de ces bases par la transformation des étapes dans E. coli XL1 Blue prélèvement de colonies individuelles, à isoler leur ADN plasmidique, 41 et en outre l' analyse par digestion de restriction et séquençage.
- Conjugaison du simple croisement construire dans Streptomyces
- Transférer le vecteur suicide contenant le gène pokPI (pKC1132_ pokPI del) avec suppression dans E. coli ET12567 pUZ8002 42 (kanamycine R) par le choc de transformation de chaleur 40. Incuber les cellules dans 100 ml LB médias (kanamycine 50 ug mL -1, apramycine 50 pg ml -1 ) Pendant la nuit à 37 ° C. récolte des cellules par centrifugation (3000 xg, 10 min, 4 ° C) et laver les cellules deux fois par remise en suspension dans 50 ml de milieu LB. Enfin, remettre les cellules dans 2 x 500 ul de milieu LB.
- Mélanger 500 ul E. coli pUZ8002 pKC1132_ pokPI del avec la culture de 500 ul Streptomyces (alternativement utiliser des spores). Étendre le mélange sur des plaques MS (farine de soja 20 g, D-mannitol 20 g, MgCl2 10 mM, agar 1,5%, l' eau du robinet 1 L). Incuber les plaques pendant 20 h à 28 ° C.
- Superposez chaque plaque avec apramycine (1,25 mg) et fosfomycine (5 mg) dissous dans 1 ml d'eau et les laisser sécher. Laisser incuber les plaques pendant plusieurs jours à 28 ° C jusqu'à ce que exconjuguants sont visibles.
- Vérifiez mutants crossovers simples
- Inoculer exconjugants unique dans 20 mL du milieu TSB (apramycine 50 mg mL -1) et les incuber à 28 ° C pendant trois jours et 180 tours par minute.
- Isoler l' ADN génomique 43 et vérifier l' interruption du gène pokPI par PCR.
- Inoculer clones individuels avec interruption génique vérifiée et Streptomyces souche de type sauvage dans 100 ml milieu HA et incuber pendant 6 jours à 28 ° C et 180 rpm. Extraire l'extrait brut (voir section 1.3). Vérifiez la production de composé par analyse HPLC-MS (voir section 1.4). Le pic correspondant dans le chromatogramme HPLC et la masse du composé ne devrait pas être plus détectable (voir Figure 6).
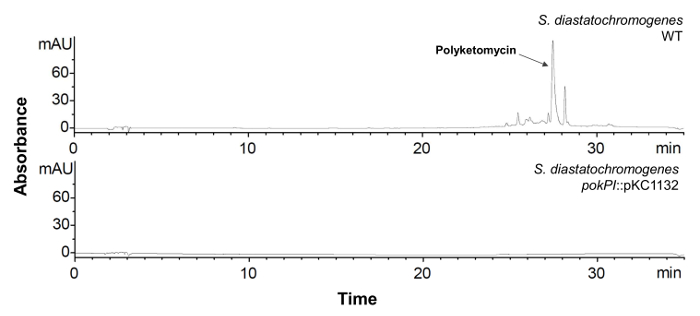
Figure 6: Analyse par HPLC de S. diastatochromogenes avec inactivé pokPI Gene. HPLC chromatogramme (λ = 430 nm) d'extrait brut de S. diastatochromogenes WT ( en haut) et de la souche mutante avec interrompue pokPI -Gen (ci - dessous). La souche mutante ne produit plus polyketomycin.S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Résultats
Dans cet aperçu, nous décrivons les étapes simples de l'identification d'un antibiotique conduisant à son groupe de gènes de biosynthèse. Il y a plusieurs années nous avons cloné une bibliothèque de cosmides, les emballés dans des phages, transduction E. cellules hôtes coli, et a dû examiner des milliers de colonies pour identifier les clones ayant des régions qui se chevauchent de grappe polyketomycin. Le séquençage des cosmides a également été un processus difficile et coûteux 12.
Afin de mener d' autres études sur la souche nous avons séquencé la totalité du génome de Streptomyces Tü6028. Avec le projet de séquençage du génome nous avons facilement identifié le groupe de gènes de biosynthèse de polyketomycin et d'autres groupes codant pour des composés prometteurs. La figure 7 compare la méthode "ancienne" pour identifier le cluster de biosynthèse par clonage d'une banque de cosmides et de dépistage élaboratif etla «nouvelle» méthode par séquençage du génome entier à l'exploitation du génome ultérieur sur une échelle de temps rugueux. Les nouvelles technologies de séquençage et de nouveaux programmes d'exploration de génome d'accélérer l'ensemble du processus.
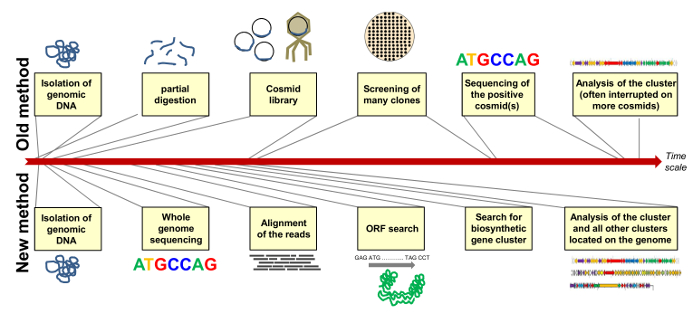
Figure 7: Comparaison de la «vieille» et «nouvelle» méthode d'attribution d' un gène biosynthétique Cluster. La méthode "ancienne" comprend le clonage d'une banque de cosmides avec la sélection des clones positifs et le séquençage du cosmide (s) respectif (ci-dessus); La méthode «nouvelle» comprend le séquençage du génome entier et -mining pour identifier tous les groupes de gènes de métabolites secondaires situés sur le génome de la souche (ci-dessous). Durée des étapes simples sont présentées sur une échelle de temps rugueux. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Discussion
Dans ce laboratoire une bibliothèque de cosmide génomique de Streptomyces diastatochromogenes a été généré et projeté il y a plusieurs années, ce qui entraîne l'identification du groupe de gènes polyketomycin à travers un processus extrêmement fastidieux. Caractérisation des gènes uniques sont possibles en utilisant des délétions de gènes cibles et l' analyse des mutants résultant 12. Récemment, un projet de séquençage du génome de S. diastatochromogenes a été obtenue permettant l'identification rapide du groupe de gènes de biosynthèse polyketomycin. Nous pourrions facilement détecter les gènes de biosynthèse, bien que le projet de séquençage du génome contient encore beaucoup de contigs. Le procédé décrit peut être réalisé en quelques mois. Cependant, le procédé comprend plusieurs étapes. étapes simples peuvent échouer à plusieurs reprises, ce qui empêche la progression vers les étapes suivantes:
Le genre Streptomyces est connu pour sa capacité à produire des composés bioactifs. Alors qu'ils transportent souvent plus de 20 biosclusters de gènes ynthetic, habituellement un ou deux composés sont produits dans des conditions de laboratoire. L'application de l'approche OSMAC (culture d'une souche dans des conditions différentes) pour réveiller groupes de gènes silencieux peut parfois ne pas être suffisant. La manipulation génétique des gènes de régulation, tels que les gènes régulateurs pléiotropiques ADPA 44 et bldA 45,46, est aussi une méthode efficace pour activer la production d'autres métabolites secondaires.
Pour l'élucidation de la structure du composé, par exemple , par analyse RMN, habituellement plus de 2 mg de composé purifié est nécessaire. Par conséquent, la fermentation de plus de 10 litres de la culture est souvent nécessaire. Sans un fermenteur qui est capable de maintenir l'oxygène, du pH et de la température, il peut être difficile dans un petit laboratoire pour traiter cette quantité de la culture et l'extraction ultérieure. Au cours de la purification, le composé peut être modifié en raison de l'oxydation, le rayonnement ou la température.En outre, plus les étapes de purification sont utilisées, plus le risque de dégradation.
Lors de l'analyse de la structure du produit naturel, et les grappes dans le génome, il est parfois pas facile d'identifier le cluster approprié. Tout d'abord, s'il y a seulement un projet de séquençage du génome une partie de la grappe peut être manquante. D'autre part, tous les gènes qui sont nécessaires pour la biosynthèse ne sont pas dans le cluster. En troisième lieu, un cluster est parfois divisé en deux parties séparées l'une de l'autre par plusieurs kilobases. Quatrièmement, il peut être difficile de décider lequel est le groupe de gènes approprié. Dans le cas d'un grand type PKS systèmes NRPS I ou, où il est possible de calculer le nombre d'unités d'extension en fonction du nombre de modules, ou même d'identifier les unités d'extension simples par analyse des domaines de la sélection, il se transforme facilement. Cependant, dans le cas d'enzymes de travailler itérativement la prédiction des composés synthétisés est souvent pas possible, surtout si le straen a plus de 40 groupes de gènes. Cinquièmement, la nature est très complexe et plein de composés encore inconnus. Souvent, la biosynthèse est un mélange de différentes voies. Si le nouveau composé est pas encore identifié, ou non liés à un autre composé, il peut être difficile d'identifier le cluster, de proposer un modèle de biosynthèse et pour le prouver.
Une fois que le cluster est identifié, la technique de croisement unique est une bonne et rapide méthode pour vérifier l'hypothèse. La PCR, le clonage dans un vecteur suicide, la conjugaison, la sélection des clones positifs, et le dosage de la production sont les seules étapes requises. Un inconvénient de cette technique est que l'intégration du vecteur dans le chromosome est instable en raison d'autres événements de recombinaison. Par conséquent, afin d'analyser davantage les gènes individuels, des deletions de gènes précises sont nécessaires. En outre , il peut être difficile de manipuler des souches de Streptomyces sur le plan génétique.
La procédure décrite peut être affectée to tout autre composé produit par une souche de Streptomyces ou d' un autre micro - organisme. La connaissance d'un groupe de gènes de biosynthèse et son composé synthétisé nous donne de nouvelles possibilités pour modifier déjà les molécules existantes dans le but de les améliorer pour la lutte contre les agents pathogènes multirésistantes.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
S. Zhang is funded by China Scholarship Council. The authors are very grateful to former people working on polyketomycin project in this lab and Prof. Dr. Hans-Peter Fiedler, University of Tübingen, for providing the polyketomycin producer. The research was supported by the DFG (RTG 1976).
matériels
| Name | Company | Catalog Number | Comments |
| agar | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 5210.4 | |
| agarose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6352.4 | |
| D-mannitol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4175.1 | |
| glucose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6780.1 | |
| LB | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X964.3 | |
| malt extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X976.2 | |
| MgCl2 | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2189.1 | |
| Peptone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | ||
| soy flour | W.Schoenemberger GmbH, Magstadt, Germany | Hensec-Vollsoja | |
| tryptic soy broth | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X938.3 | Caso-Bouillon |
| yeast extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2363.2 | |
| Solvents | |||
| Acetic acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 3738.5 | |
| Acetone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 9372.2 | |
| Acetonitrile | Avantor Performance Materials B.V., Deventer, The Netherlands | JT-9012-03 | |
| Dichlorofrom | Fisher Scientific GmbH, Schwerte, Germany | 1530754 | |
| DMSO (Dimethyl sulfoxide) | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4720.1 | |
| DMSO-d6 (Dimethyl sulfoxide-d6) 99.9 atom% D | ARMAR Chemicals, Döttingen, Switzerland | 15200.204 | |
| Ethyl acetate | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6784.4 | |
| Hydrochloric acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6331.4 | |
| Methanol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 7342.1 | |
| Enzymes | |||
| restriction enzymes | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| polymerase | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| DNA Polymerase I Large (Klenow) Fragment | Promega GmbH, Mannheim, Germany | ||
| Antibiotics | |||
| apramycin | AppliChem GmbH, Darmstadt, Germany | A7682.0005 | |
| fosfomycin | Sigma-Aldrich Chemie GmbH, Taufkirchen, Germany | P5396-50G | |
| kanamycin | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | T832.2 | |
| Plasmid/Vectors | |||
| pKC1132 | Bierman et al. 1992 | ||
| Primer | |||
| pokPI_for | TGATGGTGCCGCTGGCCATGG | Primer to amplify fragment containing pokPI gene | |
| pokPI_rev | AGCGTTCACTGTTCCGCCCGAC | ||
| Bacterial strains | |||
| Bacillus subtilis COHN ATCC6051 | Gram-positive test strain | ||
| Escherichia coli ET12567 pUZ8002 | MacNeil et al. 1992 | strain for conjugation | |
| Escherichia coli XL1 Blue | Agilient Technologies, Santa Clara, USA | Gram-negative test strain + cloning host | |
| Streptomyces diastatochromogenes Tü6028 | Paululat et al., 1999 | Polyketomycin producer | |
| Online services | |||
| antismash (Antibiotics and Secondary Metabolite Analysis Shell) | http://antismash.secondarymetabolites.org/ | Detection of secondary metabolite gene cluster | |
| BLAST (Basic Local Alignment Search Tool) | http://blast.ncbi.nlm.nih.gov/Blast.cgi | finds regions of similarity between biological sequences | |
| GenDB | https://www.uni-giessen.de/fbz/fb08/Inst/bioinformatik/software/gendb | Annotation of ORFs | |
| MiBIG (Minimum Information about a Biosynthetic Gene cluster) | http://mibig.secondarymetabolites.org/ | Database of biosyntetic gene clusters | |
| NaPDoS | http://napdos.ucsd.edu/ | Detection of seconary metabolite gene cluster | |
| NCBI Prokaryotic Genome Annotation Pipeline | http://www.ncbi.nlm.nih.gov/genome/annotation_prok/ | Annotation of ORFs | |
| NRPSpredictor | http://nrps.informatik.uni-tuebingen.de/ | Detection of NRPS domains | |
| Prokka (rapid prokaryotic genome annotation) | http://www.vicbioinformatics.com/software.prokka.shtml | Annotation of ORFs | |
| RAST (rapid annotation using subsystems technology) | http://rast.nmpdr.org/ | Annotation of ORFs | |
| Other programs | |||
| Chem Station Rev. A.09.03 | Agilent Technologies, Waldbronn, Germany | Handling program for HPLC | |
| Clone Manager Suite 7 | Scientific and Educational Software, Cary, USA | Designing Cloning Experiment | |
| Newbler v2.8 | Roche Diagnostics | Alignment of sequencing reads | |
| Machines | |||
| Centrifuge Avanti J-6000, Rotor JA-10 | Beckman Coulter GmbH, Krefeld, Germany | ||
| HPLC/MS | Agilent Technologies, Waldbronn, Germany | ||
| _Autosampler: G1313A | Agilent Technologies, Waldbronn, Germany | ||
| _Pre-column: XBridge C18 (20 mm x 4.6 mm; Particle size: 3.5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Column:Xbridge C18 (100 mm × 4.6 mm; Particle size: 3.5 μm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Pre-Column: Zorbax B-C18 (9.4 x 150 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Column: Zorbax B-C18 (9.4 x 20 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Degasser: G1322A | Agilent Technologies, Waldbronn, Germany | ||
| _Quarternary pump: G1311A | Agilent Technologies, Waldbronn, Germany | ||
| _Diode array detector (DAD )G1315B (λ = 254 nm and 400 nm) | Agilent Technologies, Waldbronn, Germany | ||
| _Quadrupole mass detector (MSD) G1946D(2-3000 m/z) | Agilent Technologies, Waldbronn, Germany | ||
| rotary evaporator | |||
| _heating bath Hei-VAP Value/G3 | Heidolph Instruments GmbH & Co.KG, Schwabach, Germany | ||
| _vacuum pump system SC 920 G | KNF Global Strategies AG, Sursee, Switzerland | ||
| Other material | |||
| Sephadex LH20 | GE Healthcare, | ||
| Chromafil PVDF-45/15MS (pore size 0.45 µm; filter Ø15 mm) | MACHEREY-NAGEL GmbH & Co. KG, Düren, Germany | ||
| SPE column Oasis HLB 20 35 cc (6g) | Waters GmbH, Eschborn, Germany | ||
| E. coli | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Bacillus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Streptomyces sp. | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: TSB, Composition: CASO Boullion, Amount to 1 L H2O: 30 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Yeast extract, Amount to 1 L H2O: 10 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Peptone, Amount to 1 L H2O: 20 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Glucose, Amount to 1 L H2O: 20 g | |
| for agar plates add 2% agar | |||
Références
- Fleming, A. On the antibacterial action of cultures of a penicillin, with special reference to their use in isolation of B. influenzae. Br J Exp Pathol. 10 (3), 226-236 (1929).
- Lemke, A., Kiderlen, A. F., Kayser, O. Amphotericin B. Appl. Microbiol. Biotechnol. 68 (2), 151-162 (2005).
- Kirkpatrick, P., Raja, A., LaBonte, J., Lebbos, J. Daptomycin. Nat. Rev. Drug Discov. 2 (12), 943-944 (2003).
- Zakeri, B., Wright, G. D. Chemical biology of tetracycline antibiotics. Biochem. cell Biol. 86 (2), 124-136 (2008).
- Schatz, A., Bugle, E., Waksman, S. A. Streptomycin, a Substance Exhibiting Antibiotic Activity Against Gram-Positive and Gram-Negative Bacteria. Exp. Biol. Med. 55 (1), 66-69 (1944).
- Burg, R. W., et al. Avermectins, new family of potent anthelmintic agents: producing organism and fermentation. Antimicrob. Agents Chemother. 15 (3), 361-367 (1979).
- Egerton, J. R., Ostlind, D. A., et al. Avermectins, new family of potent anthelmintic agents: efficacy of the B1a component. Antimicrob. Agents Chemother. 15 (3), 372-378 (1979).
- Walsh, C. T., Fischbach, M. A. Natural products version 2.0: connecting genes to molecules. J. Am. Chem. Soc. 132 (8), 2469-2493 (2010).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces sp. MK277-AF1. II. Structure determination. J. Antibiot. (Tokyo). 51 (1), 26-32 (1998).
- Paululat, T., Zeeck, A., Gutterer, J. M., Fiedler, H. P. Biosynthesis of polyketomycin produced by Streptomyces diastatochromogenes.Tü6028. J. Antibiot. (Tokyo). 52 (2), 96-101 (1999).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces. sp. MK277-AF1. I. Taxonomy, production, isolation, physico-chemical properties and biological activities. J. Antibiot. (Tokyo). 51 (1), 21-25 (1998).
- Daum, M., et al. Organisation of the biosynthetic gene cluster and tailoring enzymes in the biosynthesis of the tetracyclic quinone glycoside antibiotic polyketomycin. Chembiochem. 10 (6), 1073-1083 (2009).
- Bode, H. B., Bethe, B., Höfs, R., Zeeck, A. Big effects from small changes: possible ways to explore nature's chemical diversity. Chembiochem. 3 (7), 619-627 (2002).
- Wolfender, J. -. L. HPLC in Natural Product Analysis: The Detection Issue. Planta Med. 75 (07), 719-734 (2009).
- Stahl, E. . Thin-layer chromatography. A laboratory handbook. , (1967).
- Snyder, L. R., Kirkland, J. J., Glajch, J. L. Preparative HPLC Separation. Pract. HPLC Method Dev. , 616-642 (2012).
- Granath, K. A., Kvist, B. E. Molecular weight distribution analysis by gel chromatography on sephadex. J. Chromatogr. A. 28, 69-81 (1967).
- Jackman, L. M., Sternhell, S. Application of Nuclear Magnetic Resonance Spectroscopy in Organic Chemistry. Int. Ser. Org. Chem. , (1969).
- Xian, F., Hendrickson, C. L., Marshall, A. G. High resolution mass spectrometry. Anal. Chem. 84 (2), 708-719 (2012).
- Metzker, M. L. Sequencing technologies - the next generation. Nat. Rev. Genet. 11 (1), 31-46 (2010).
- Tatusova, T., et al. Prokaryotic Genome Annotation Pipeline. NCBI Handb. 2nd Ed. , (2013).
- Angiuoli, S. V., et al. Toward an online repository of Standard Operating Procedures (SOPs) for (meta)genomic annotation. OMICS. 12 (2), 137-141 (2008).
- Aziz, R. K., et al. The RAST Server: rapid annotations using subsystems technology. BMC Genomics. 9, 75 (2008).
- Overbeek, R., et al. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST). Nucleic Acids Res. 42, 206-214 (2014).
- Brettin, T., et al. RASTtk: a modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes. Sci. Rep. 5, 8365 (2015).
- Seemann, T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Meyer, F. GenDB--an open source genome annotation system for prokaryote genomes. Nucleic Acids Res. 31 (8), 2187-2195 (2003).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. J. Mol. Biol. 215 (3), 403-410 (1990).
- Madden, T. The BLAST Sequence Analysis Tool. NCBI Handb. 2nd Ed. , (2003).
- Marchler-Bauer, A., et al. CDD: NCBI's conserved domain database. Nucleic Acids Res. 43, 222-226 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Res. 39, 339-346 (2011).
- Blin, K., et al. antiSMASH 2.0--a versatile platform for genome mining of secondary metabolite producers. Nucleic Acids Res. 41, 204-212 (2013).
- Weber, T., et al. antiSMASH 3.0 - a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Res. 43, 237-243 (2015).
- Ziemert, N., et al. The natural product domain seeker NaPDoS: a phylogeny based bioinformatic tool to classify secondary metabolite gene diversity. PLoS One. 7 (3), 34064 (2012).
- Rausch, C., Weber, T., Kohlbacher, O., Wohlleben, W., Huson, D. H. Specificity prediction of adenylation domains in nonribosomal peptide synthetases (NRPS) using transductive support vector machines (TSVMs). Nucleic Acids Res. 33 (18), 5799-5808 (2005).
- Röttig, M., et al. NRPSSpredictor2--a web server for predicting NRPS adenylation domain specificity. Nucleic Acids Res. 39, 362-367 (2011).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nat. Chem. Biol. 11 (9), 625-631 (2015).
- Mullis, K., et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb. Symp. Quant. Biol. 51, 263-273 (1986).
- Bierman, M., et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp. Gene. 116 (1), 43-49 (1992).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli.using the heat shock method. J. Vis. Exp. (6), e253 (2007).
- Bimboim, H. C., Doly, J. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Res. 7 (6), 1513-1523 (1979).
- MacNeil, D. J., et al. Analysis of Streptomyces avermitilis. genes required for avermectin biosynthesis utilizing a novel integration vector. Gene. 111 (1), 61-68 (1992).
- Pospiech, A., Neumann, B. A versatile quick-prep of genomic DNA from Gram-positive bacteria. Trends Genet. 11 (6), 217-218 (1995).
- Makitrynskyy, R., et al. Pleiotropic regulatory genes bldA, adpA and absB are implicated in production of phosphoglycolipid antibiotic moenomycin. Open Biol. 3 (10), 130121 (2013).
- Kalan, L., et al. A cryptic polyene biosynthetic gene cluster in Streptomyces calvus is expressed upon complementation with a functional bldA gene. Chem. Biol. 20 (10), 1214-1224 (2013).
- Gessner, A., et al. Changing Biosynthetic Profiles by Expressing bldA in Streptomyces Strains. ChemBioChem. 16 (15), 2244-2252 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon