Method Article
Из натурального продукта до его биосинтетического кластера генов: демонстрация Использование Polyketomycin из
В этой статье
Резюме
Здесь мы представляем подробный протокол (А) определение натурального продукта с антибиотической активностью, (Б) очистки соединения, (C) первая модель биосинтеза, (D) секвенирование генома / -mining и ( е) проверка кластера генов биосинтетических.
Аннотация
Streptomyces штаммы известны своей способностью производить множество различных соединений с различными биоактивности. Культивирование в различных условиях, часто приводит к получению новых соединений. Таким образом, продуктивных культур штаммов экстрагируют этилацетатом, и сырые экстракты анализируют с помощью ВЭЖХ. Кроме того, экстракты испытаны на их биологическую активность различными анализами. Для установления структуры соединение интерес очищают с помощью комбинации различных методов хроматографии.
Секвенирование генома в сочетании с добычей полезных ископаемых генома позволяет идентифицировать кластер генов биосинтетических натуральный продукт с использованием различных компьютерных программ. Для того, чтобы подтвердить, что правильный кластер генов был идентифицирован, по инактивации генов эксперименты должны быть выполнены. Полученные мутанты анализируются для производства конкретного природного продукта. После того, как правильный кластер генов был инактивирован, тоштамм должен не в состоянии произвести соединение.
Рабочий процесс показан для антибактериальной соединения polyketomycin производимого Streptomyces diastatochromogenes Tü6028. Около десяти лет назад, когда секвенирование генома была все еще очень дорого, клонирование и идентификация кластера генов был очень трудоемкий процесс. Быстрое секвенирование генома в сочетании с добычей полезных ископаемых генома ускоряет судебный процесс по делу идентификации кластера и открывает новые пути для изучения биосинтеза и генерировать новые натуральные продукты с помощью генетических методов. Протокол , описанный в данном документе , могут быть отнесены к любым другим соединением , полученных из штамма Streptomyces или другого микроорганизма.
Введение
Натуральные продукты из растений и микроорганизмов всегда были важным источником для клинического разработки лекарственных средств и научных исследований. Первый антибиотик пенициллин был обнаружен в 1928 г. из гриба Александром Флемингом 1. В настоящее время, многие другие натуральные продукты используются в клинической терапии.
Один род известен своей способностью производить различные виды вторичных метаболитов с различными биоактивности является Streptomyces. Streptomyces являются грамположительные бактерии и принадлежат к классу Actinobacteria и порядка Actinomycetales. Почти две трети клинически используемых антибиотиков получены из Actinomycetales, в основном , из Streptomyces, как амфотерицин 2, 3 или даптомицин тетрациклина 4. Два Нобелевские премии были удостоены в области исследований Streptomyces антибиотиков. Первый из них пошел в Зельман Ваксман за открытие стрептомицина, первого вtibiotic эффективен против туберкулеза. 5 В 2015 году в рамках Нобелевской премии по физиологии и медицине, открытие авермектина из S.avermitilis , был награжден , а также. Авермектин используется для лечения паразитарных заболеваний 6,7.
Традиционный подход к открытию природных продуктов в таких микроорганизмов, как Streptomyces обычно включает в себя культивирование штамма при различных условиях роста, а также извлечение и анализ вторичных метаболитов. Биоактивность анализы (например , анализы для антибактериальной и противоопухолевой активностью) выполняются для определения активности соединения. И, наконец, интерес соединение выделяют и химическая структура выяснены.
Структуры натуральных продуктов часто состоят из единичных фрагментов, которые, образующих сложные молекулы. Есть несколько, но ограниченные, основные биосинтетические пути, ведущие к созданию блокакс, которые используются для биосинтеза натуральных продуктов. Основные пути биосинтеза являются поликетидный дорожки, пути, ведущие к терпеноидов и алкалоидов, путей с использованием аминокислот, а также пути, ведущие к сахарных звеньев. Каждый путь характеризуется набором специфических ферментов 8. На основании структуры соединения, эти ферменты биосинтеза можно предсказать.
В настоящее время, детальный структурный анализ соединения в сочетании с секвенирования следующего поколения и биоинформатики анализа может помочь определить ответственного биосинтетическую кластера генов. Информация кластера открывает новые пути для дальнейших исследований натуральных продуктов. Это включает в себя гетерологичную экспрессию, чтобы увеличить выход природного продукта, целевое соединение модификации путем делеции гена или изменения и комбинаторной биосинтеза с генами из других путей.
Polyketomycin был выделен независимо от культуральной средыдва штамма, Streptomyces Sp. MK277-AF1 9 и Streptomyces diastatochromogenes Tü6028 10. Структура была выяснена с помощью ЯМР и рентгеноструктурного анализа. Polyketomycin состоит из тетрациклическое decaketid и диметиловый салициловой кислоты, связанный с двумя deoxysugar фрагментами бета-D-amicetose и & alpha; -L-axenose. Он отображает цитотоксическое и антибиотической активностью, даже по отношению к грамположительным с множественной лекарственной устойчивостью штаммов , таких как MRSA 11.
Геномную космида библиотека S. diastatochromogenes Tü6028 был сгенерирован и просеивают много лет назад. Использование специфического гена зондов кластера генов polyketomycin с размером 52,2 кб, содержащий 41 генов, был идентифицирован после нескольких месяцев напряженной работы 12. Недавно была получена последовательность генома проекта С. diastatochromogenes что приводит к быстрой идентификации polyketomycin биосинтетического кластера генов. В этом обзоре, метод помогает идентифы натуральный продукт, и выяснить его биосинтетических генов кластера будет описано ниже, с использованием polyketomycin в качестве примера.
Здесь мы объясняем отдельные шаги , которые ведут из натурального продукта до его биосинтетического кластера генов , показанного на polyketomycin производства Streptomyces diastatochromogenes Tü6028. Протокол включает в себя идентификацию и очистку природного продукта с антибиотическими свойствами. Далее структурный анализ и сравнение с результатами генома добычи приводят к идентификации биосинтетического кластера генов. Эта процедура может быть применена к любым другим соединением , полученных из штамма Streptomyces или любого другого микроорганизма.
протокол
1. Идентификация Натуральный продукт с Антибиотик собственности
- Выращивают микроорганизма при различных условиях (например , время, температура, рН, СМИ) после "OSMAC (один штамм-много соединений) подход" 13. Выберите одну среду, в которой наблюдается получения соединения.
- Выращивают Streptomyces diastatochromogenes Tü6028 при следующих условиях
- Grow Streptomyces diastatochromogenes штамма Tü6026 на MS планшетах (соевая мука 20 г D-маннита 20 г, MgCl 2 , 10 мМ, 1,5% агар, водопроводная вода 1 л). Привить небольшую петлю спор этого штамма в 20 мл жидкости TSB среды (трипсином соевый бульон 30 г, водопроводная вода 1 л, рН 7,2; предкультуральной среды) в колбе Эрленмейера с спирали. Взболтать колбу на роторной качалке (28 ° C, 180 оборотов в минуту, 2 дня).
- Привить основной культуры в 100 мл среды HA (глюкоза 4 г, дрожжевой экстракт 4 г, солодэкстракт 10 г, водопроводную воду 1 л, рН 7,2) с 2 мл предварительной культуры. Культура деформация при 28 ° С в течение 6 дней на роторной качалке при 180 оборотах в минуту.
- Приготовление сырого экстракта
- Урожай клетки центрифугированием (10 мин, 3000 XG).
- Для последующих шагов обрабатывать органические растворители под вытяжкой.
- Для извлечения соединения из мицелия, ресуспендирования клеток в двукратном объеме ацетона и трясти в трубке в течение 30 мин, 180 оборотов в минуту. Фильтр жидкости через коммерческие бумаги фильтруют и выпаривают ацетон в роторном испарителе при 40 ° С и 550 бар. Растворить экстракт в 20 мл воды: этилацетат (1: 1) и трясти ее в делительную воронку в течение 30 мин при 180 оборотах в минуту.
- При экстракции соединения из культуральной среды, регулировать культурального бульона до рН 4,0 добавлением 1 М HCl. Добавить 100 мл этилацетата, а трясти ее в делительную воронку в течение 30 мин, 180 оборотов в минуту.
- Собирают этилацетатной фазы и испаряются гнилью ичных испарения при 40 ° C и 240 бар.
- Анализ неочищенного экстракта с помощью высокоэффективной жидкостной хроматографии
- Растворите экстракты в 1 мл MeOH, фильтровать их через фильтр с размером пор 0,45 мкм пор и запустить высокоэффективной жидкостной хроматографии (ВЭЖХ) 14.
- В случае polyketomycin, оборудовать систему ВЭЖХ с С18 предколонкой (4,6 х 20 мм 2) и колонке С18 (4,6 х 100 мм 2). С помощью линейного градиента ацетонитрила + 0,5% уксусной кислоты в диапазоне от 20% до 95% в H 2 O + 0,5% (расход: 0,5 мл мин -1) уксусной кислоты.
Примечание: Polyketomycin имеет время удерживания 25,9 мин (см Фигура 1А). UV / VIS спектр имеет максимумы при 242 нм, 282 нм, 446 нм и минимумы при 262 нм и 359 нм (рис 1В). В отрицательном модусе массой от 863,2 [МН] - можно обнаружить (см рисунок 1С).
"SRC =" / файлы / ftp_upload / 54952 / 54952fig1.jpg "/>
Рисунок 1: LC / MS анализ Polyketomycin. (А) хроматограмма высокоэффективной жидкостной хроматографии (λ = 430 нм) неочищенного экстракта после культивирования Streptomyces diastatochromogenes Tü6028 в течение 6 дней. Polyketomycin имеет время удерживания 25,9 мин (В) UV / VIS спектры Polyketomycin. (C) Масс - спектры Polyketomycin в отрицательном модусе. Основной пик с т / г 863,2 [MH] -. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
- Определение активности антибиотика с помощью диффузионного диска анализа
- Привить тестовые штаммы , такие как грамположительных бактерий Bacillus Сенная (в LB среде; LB 20 г, водопроводная вода 1 л, рН 7,4) и другие штаммы Streptomyces (в TSB среде; трипсином соевого бульона , 30 г, водопроводная вода 1 л, рН 70,2), грамотрицательные бактерии кишечной палочки (в LB среде) и грибковые штаммы (в средах , таких как YPD среде, дрожжевой экстракт 10 г, пептон 20 г, глюкоза 20 г, водопроводная вода 1 л, рН 7,4). Взять 100 мкл тестируемого штамма предкультурой и распределить их на соответствующие чашки с агаром.
- Растворение сырого экстракта или очищенного соединения в 500 мкл метанола (в качестве альтернативы воде или ДМСО) и пипетка 20-50 мкл на стерильные бумажные диски. Сухие бумажные диски в течение 30 мин под верстак и поместить их на пластины с тест-культур. Приготовьте отрицательный контроль (растворитель) и положительного контроля (антибиотик, например , апрамицин [1 мг]).
- Инкубируйте пластин при соответствующих условиях, пока тестовые штаммы не заметно выросли и определяют зону ингибирования, если очевидно. Выдержите E.coli и Bacillus зр. при 37 ° С в течение 16 ч, Streptomyces Sp. при 28 ° С в течение 2-4 дней, грибковый штамм (в основном зависит от точного критерия деформации) ат 30 ° С в течение 2-х дней).
2. Крупномасштабная добыча, очистка и структура Выяснение соединения
- Выращивают S. diastatochromogenes в 5 л HA среды (глюкоза 4 г, дрожжевой экстракт 4 г, солодовый экстракт 10 г, водопроводная вода 1 л, рН 7,2). Инокулируйте 2 мл предварительной культуры в 30-х 500 колб Эрленмейера мл, содержащих 150 мл среды HA. Выдержите деформация при 28 ° С в течение 6 дней на ротационном шейкере со скоростью 180 оборотов в минуту.
- Урожай клетки центрифугированием (10 мин, 15000 XG, RT) и экстракт соединений с использованием этилацетата (смотри раздел 1.3).
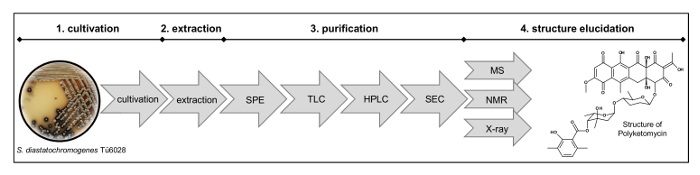
Рисунок 2: Поток операций для установления структуры. Способ включает (1) культивирование штамма, (2) экстракции, (3) очистка с помощью твердофазной экстракции (SPE), тонкослойной хроматографии (ТСХ), препаративной высокоэффективной жидкостной хроматографии (ВЭЖХ), размерхроматографии с исключением (SEC), и (4) СТРОЕНИЯ с помощью масс-спектрального анализа (МС), ядерного магнитного резонанса (ЯМР) и измерений рентгеновского излучения. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
- Очистка Polyketomycin
ПРИМЕЧАНИЕ: Процесс очистки и установления структуры показана на рисунке 2.- Фракционирования сырой экстракт по колонке С18 твердофазной экстракции (SPE) с использованием 10% MeOH -stepwise градиент в диапазоне от 30% до 100% MeOH в H 2 O, 100 мл для каждого условия.
- Очищают соединение фракции , содержащей далее с помощью препаративной тонкослойной хроматографии (ТСХ) с использованием 15 CH 2 Cl 2 / MeOH (7: 1) в качестве системы элюции.
- Очищают соединение с помощью препаративной ВЭЖХ 16. Оборудуйте систему ВЭЖХ с С18 предколонкой (5 мкм, 9,4 х 20 мм) иосновная колонка (5 мкм, 9,4 х 150 мм). Используйте градиент ацетонитрила + 0,5% уксусной кислоты в диапазоне от 20% до 95% в H 2 O + 0,5% уксусной кислоты (скорость потока: 2,0 мл / мин).
- Для удаления растворителей и других мелких примесей, выполнить последнюю стадию очистки с помощью вытеснительной хроматографии с использованием колонки в MeOH 17. Собирают окончательное чистое соединение и испаряться МеОН на роторном испарителе при 40 ° С и 240 бар.
- Структура выяснение
- Растворите чистое соединение (более 2 мг) в 600-700 мкл ( в зависимости от машины) или ДМСО - d6, запись 1D ЯМР (1 Н, 13 С) и 2D - ЯМР (HSQC, НМВС, 1 Н- 1 H COSY) спектры на спектрометре ЯМР 18. Экспресс химические сдвиги в & delta значений (частей на миллион), используя ДМСО - d6.
- Запись масс - спектр высокого разрешения (HRESI-MS) 19 polyketomycin с использованием высокого разрешениямасс-спектрометр.
- Поясним структуру путем интерпретации результатов анализа ЯМР и МС 10.
3. предложить биосинтетических модель нового выделенного соединения
- Анализ структуры выделенного соединения и предсказать ферменты, которые могут быть вовлечены в его биосинтеза. Назначают их поликетидный (тип I, II или III), нерибосомальных синтеза пептидов, lantipeptide, терпеноида или метаболизм сахара пути 8.
- Пример polyketomycin
- Подразделите структуру в очевидных одиночных фрагментов: тетрациклическое части, две моносахаридов и диметилового салициловой кислоты.
- Выясните, где фрагменты могут быть получены из: тетрациклический фрагмент могут быть получены из типа поликетидсинтаза II и диметиловый фрагмент салициловой кислоты могут быть получены с помощью итерационной поликетидный типа синтазы I. Две сахарные группы, которые являются 6-дезоксисахаров , может быть synthesiЗед из глюкозы с участием TDP-глюкоза-4,6-дегидратазе в процессе биосинтеза и приложенные к нему , вероятно , двумя гликозилтрансферазами (смотри рисунок 3) 8.
- Предсказать предположительные генов в кластере. Подумайте ферментов, которые участвуют в синтезе отдельных фрагментов, при установлении соединения блоков, а также в модификации и адаптации молекулы. Ген биосинтетическая кластер Polyketomycin наиболее вероятно , содержит гены , кодирующие тип поликетидсинтаза II (поэтому минимальный АСР, KS α унд KS & beta для подключения расширителей единиц), итеративный типа поликетидсинтаза I ( по крайней мере , ACP, AT, KS), A TDP-глюкоза-4,6-дегидратазы (необходимо для стадии: глюкоза → дезоксиглюкозы) и два гликозилтрансферазы (имеется два сахарных мономеров с агликоном) 8.
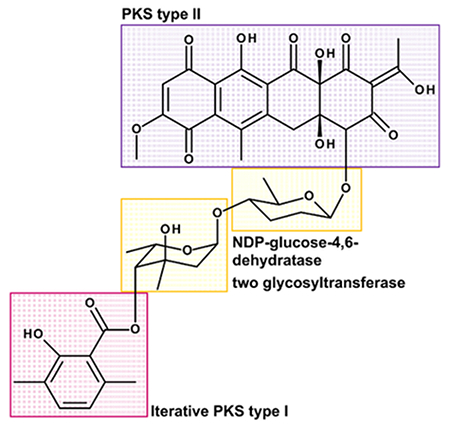
Рисунок 3: Структура Polyketomycin дивидеэд в единые строительные блоки. Polyketomycin состоит из тетрациклическое decaketid (ПКС тип II) и диметиловый салициловой кислоты (итеративный ПКС тип I), связанный с двумя deoxysugar остатками β-D-amicetose и & alpha; -L-axenose (NDP-глюкоза-4,6- дегидратазе и требуется два гликозилтрансферазу). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
4. секвенированию генома / Mining
- Следующее поколение секвенирования
- Последовательность геномной ДНК с помощью технологии секвенирования следующего поколения , как Illumina, 454 Пиросеквенирование или SOLiD 20. Выравнивание одного читает эталонной последовательности или собрать заново.
ПРИМЕЧАНИЕ: геном S. diastatochromogenes Tü6028 секвенировали в Центре биотехнологии (CeBiTec) в Университете Билефельда. Все читает были собраны на проект генома7,9 Mb.
- Последовательность геномной ДНК с помощью технологии секвенирования следующего поколения , как Illumina, 454 Пиросеквенирование или SOLiD 20. Выравнивание одного читает эталонной последовательности или собрать заново.
- добыча Геном
- Поиск открытых рамок считывания (ORFs) путем использования, например , NCBI прокариотических генома Аннотация трубопровода 21,22, РАСТ (быстрое аннотаций с использованием подсистемы технологии) 23,24,25, Prokka (быстрое прокариотической геном аннотацию) 26 или GENDB 27. Эти программы также предлагают свои функции. Анализ S. diastatochromogenes Tü6028 последовательность генома проект привел к идентификации более чем 7000 ORF , .
- Запуск конкретных BLAST (Basic Local Alignment Search Tool) анализ , чтобы получить более подробную информацию , как выравниваний с другими подобными генами и каталитических доменов 28,29,30.
- Для идентификации гена метаболит программ вторичного кластера бегут как antiSMASH 31,32,33, NaPDoS 34 и NRPSpredictor 35,36. В проекте генома Streptomyces diastatochromogenes antiSMASH 31,32,33 IdentiFied 22 кластеров генов.
- Анализ мнимые генных кластеров для их ферментативного пути (ов) (поликетидсинтаза (тип I, II или III) нерибосомальных пептид-синтетазы, lanthipeptide, терпеноидов, метаболизм сахара ...). Поиск кластера (ов), содержащих гены, которые могут быть вовлечены в синтезе соединения (смотри раздел 3). В S. diastatochromogenes аннотированный кластер 2 содержит поликетидный Type - синтазы гены II, три поликетидный типа синтазы I генов, ген TDP-глюкоза-4,6-дегидратазы и два гена гликозилтрансферазу (смотри рисунок 4).
- Фокус на отдельных генов в кластере. Для типа I PKS и NRPS специфичность ацилтрансферазы и adenylation доменов, и, таким образом, включение одного расширители единиц, могут быть предсказаны. Также сравните порядок объединенного блока расширителя с молекулой. antiSMASH 31,32,33 также показывает аналогичные кластеры уже известных соединений, со ссылкой на базу данных MIBiG 37.
- Сравните структуру соединения с этими другими соединениями и проверить наличие сходства.

Рисунок 4: antiSMASH Выход Polyketomycin кластера генов биосинтеза и Обзор других кластеров в S. diastatochromogenes Tü6028. (А) Обзор прогнозируемых биосинтетических кластеров генов в геноме S. diastatochromogenes Tü6028; (Б) Кластер 2 Polyketomycin генов биосинтетических кластера с целевыми генами. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
5. Проверка кластера биосинтетического гена
- Поиск гена с очевидными и важными (существенных) функций для биосинтеза соединения. Для проверки polyketoMYCIN кластер генов ген pokPI, кодирующую ketosynthase альфа от типа ПКС II, был выбран и инактивируют путем из кадра -deletion (смотри рисунок 5B). В качестве альтернативы, прервать ген путем клонирования внутренний фрагмент (см рисунок 5C).
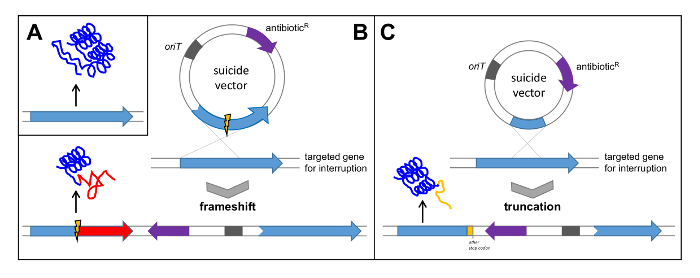
Рисунок 5: Проверка кластера генов с помощью единого Crossover. (А) нативный ген приводит к переводу функционального белка; (Б) Клонирование гена с внутренней делецией в вектор суицида приводит к одному кроссовером , что приводит к сдвигу рамки в целевом гене и последующего перевода неактивный белок; (С) Клонирование внутреннего фрагмента гена в вектор суицида приводит к усечения гена и последующего перевода нефункционального белка. Орит: происхождение тransfer; R антибиотик: устойчивость к антибиотикам. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
- Клонирование одного кроссовера конструкции
- Амплифицировать фрагмент , содержащий pokPI с помощью ПЦР с праймерами 38 pokPI_for и pokPI_rev.
- Продукт ПЦР - клон в векторе самоубийства pKC1132 39 (апрамицин R [50 мг / мл]). Вектор самоубийство не способен к репликации в штамме Streptomyces и , таким образом , должен интегрировать в геном целевой путем гомологичной рекомбинации , чтобы обеспечить апрамицину резистентность. Перенос вектора в E.coli хоз ине клонировани путем теплового шока трансформации 40.
- Изолировать вектор с помощью щелочного лизиса 41.
- Дайджест векторной ДНК с одним режущим ферментом рестрикции, который разрезает внутри фрагмента. PokPI поколенияе расщепляли фермент Kpn I.
- Лечить переваренной векторной ДНК с большой ДНК-полимеразы I (Кленова) фрагмент, который имеет 5 '→ 3' полимеразной активностью и 3 '→ 5' экзонуклеазной-активность. Притупление липких концов и последующее повторное лигирование приводит к потере четырех оснований. Проверьте потери этих баз путем преобразования шагов в Е.coli XL1 Blue, собирание отдельных колоний, изолируя их ДНК плазмиды, 41 и дальнейшего анализа с помощью расщепления рестриктазами и секвенирования.
- Сопряжение одноленточного кроссовера конструкции в Streptomyces
- Перенести вектор самоубийство , содержащий ген pokPI (pKC1132_ pokPI Del) с удалением в E.coli ET12567 pUZ8002 42 (канамицин R) с помощью теплового шока трансформации 40. Инкубируйте клетки в 100 мл LB среды (канамицин 50 мкг мл -1, апрамицин 50 мкг мл -1 ) В течение ночи при 37 ° С. Урожай клетки центрифугированием (3000 XG, 10 мин, 4 ° С) и мыть клетки дважды ресуспендирования в 50 мл LB-носителей. И, наконец, ресуспендирования клеток в 2 х 500 мкл LB сред.
- Смешайте 500 мкл кишечной палочки pUZ8002 pKC1132_ pokPI дель 500 мкл Streptomyces diastatochromogenes культуры ( в качестве альтернативы использовать споры). Распространение смесь на MS планшетах (соевая мука 20 г D-маннита 20 г, MgCl 2 , 10 мМ, 1,5% агар, водопроводная вода 1 л). Инкубируйте пластин в течение 20 ч при 28 ° С.
- Перекрытие каждую пластину с апрамицину (1,25 мг) и фосфомицина (5 мг), растворенный в 1 мл воды и дайте им высохнуть. Инкубируйте пластин в течение нескольких дней при температуре 28 ° C до эксконъюгантов не видно.
- Проверка одного кроссовера мутантов
- Привить одиночные эксконъюгантов в 20 мл среды TSB (апрамицин 50 мг мл -1) и инкубировать их при 28 ° С в течение трех дней и 180 оборотов в минуту.
- Изолировать геномную ДНК 43 и проверить прерывание гена pokPI с помощью ПЦР.
- Инокулируйте отдельные клоны с проверенными прерыванием гена и Streptomyces дикого типа штамма в 100 мл среды HA и инкубировать их в течение 6 дней при 28 ° С и 180 оборотах в минуту. Извлечение неочищенного экстракта (смотри раздел 1.3). Проверка производства соединение с помощью анализа ВЭЖХ-МС (см раздел 1.4). Соответствующий пик в хроматограмме ВЭЖХ и массу соединения не должно быть обнаружено больше (рисунок 6).
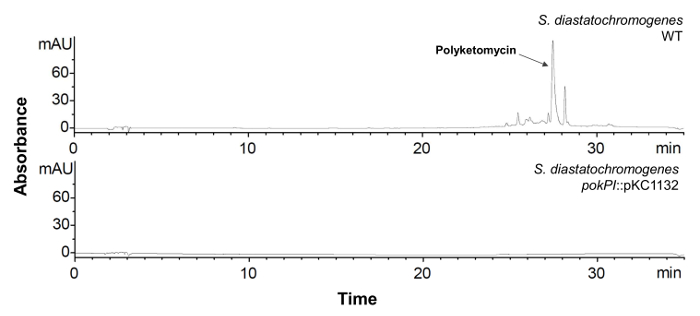
Рисунок 6: Анализ ВЭЖХ С. diastatochromogenes с инактивированной pokPI гена. Хроматограмма ВЭЖХ (λ = 430 нм) неочищенного экстракта S. diastatochromogenes WT (сверху) и мутантного штамма с прерванного pokPI -GEN (ниже). Мутантный штамм не производит polyketomycin больше.Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Результаты
В этом обзоре мы опишем отдельные шаги от идентификации антибиотика, ведущего к его биосинтетического кластера генов. Много лет назад мы клонировали библиотеку космиды, оформляли их в фагов, трансдуцированная E. клетки - хозяева кишечной палочки, и должен был экран тысячи колоний для идентификации клонов , имеющих перекрывающиеся области polyketomycin кластера. Секвенирование космиды был также трудным и дорогостоящим процессом 12.
Для проведения дальнейших исследований штамма мы упорядочили весь геном Streptomyces diastatochromogenes Tü6028. С проектом последовательности генома мы легко идентифицировать биосинтетических генов кластера polyketomycin и других кластеров, кодирующих перспективных соединений. Рисунок 7 сравнивает "старый" способ идентификации биосинтетического кластера путем клонирования библиотеки космиды и пояснительный скрининга, и«новый» метод, с помощью всего генома с последующей добычей генома на шероховатой временной шкале. Новые технологии секвенирования и новые программы горнодобывающие генома ускорить весь процесс.
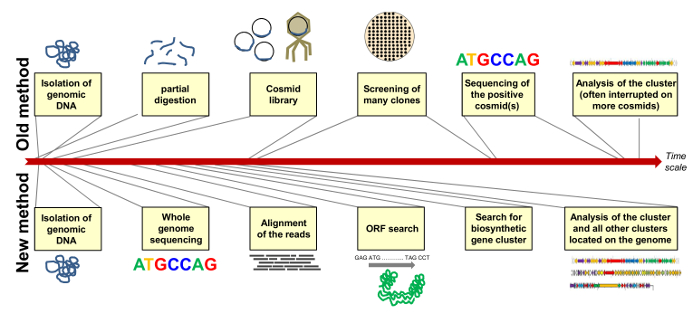
Рисунок 7: Сравнение "старой" и "Новый" Метод Назначение биосинтетических кластера генов. "Старый" способ включает клонирование библиотеки космиды с выделением положительных клонов и последовательности соответствующих космиды (ов) (выше); "Новый" метод включает в себя весь секвенирование генома и -mining для выявления всех вторичных генов кластеры метаболита, расположенные на геном штамма (ниже). Продолжительность отдельных стадий показаны на шероховатой временной шкале. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Обсуждение
В этой лаборатории был создан геномный космида библиотека Streptomyces diastatochromogenes и просеивают много лет назад, в результате идентификации кластера генов polyketomycin через процесс чрезвычайно много времени. Характеристика отдельных генов было возможно с помощью целевых делеции и анализ полученных мутантов 12 генов. В последнее время , последовательность проект генома S. diastatochromogenes получали позволяя быстро идентифицировать polyketomycin биосинтетического кластера генов. Мы могли бы легко обнаружить гены биосинтетических, хотя последовательность проект генома содержит еще много контигов. Описанный процесс может быть достигнуто в течение нескольких месяцев. Тем не менее, эта процедура включает в себя множество этапов. Одиночные шаги может произойти сбой в несколько раз, предотвращая прогрессирование последующих стадий:
Род Streptomyces известен своей способностью продуцировать биологически активных соединений. В то время как они несут часто более 20 биозуynthetic кластеры генов, как правило, только один или два соединения, полученные в лабораторных условиях. Применение подхода OSMAC (выращивание одного штамма в различных условиях), чтобы проснуться молчащие кластеры генов иногда может быть недостаточно. Генетические манипуляции регуляторных генов, таких как плейотропных регуляторных генов ADPA 44 и bldA 45,46, также является эффективным методом для активизации производства других вторичных метаболитов.
Для выяснения структуры жилого комплекса , например , с помощью ЯМР - анализа, как правило , более 2 мг очищенного соединения необходимо. Таким образом, ферментация более 10 л культуры часто требуется. Без ферментере, который способен поддерживать кислорода, рН и температурные условия, это может оказаться непростой задачей в небольшой лаборатории, чтобы справиться с этой суммы культуры и последующего извлечения. Во время очистки, соединение может быть изменено из-за окисления, радиации или температуры.Кроме того, используются более стадий очистки, тем выше вероятность ухудшения.
При анализе структуры природного продукта, а также кластеры в геноме, иногда это не так легко идентифицировать соответствующий кластер. Во-первых, если есть только последовательность генома проекта некоторая часть кластера может отсутствовать. Во-вторых, не все гены, необходимые для биосинтеза в кластере. В-третьих, иногда кластер разделяется на две части, отделенные друг от друга многими т.п.н.. В-четвертых, это может быть трудно решить, какой из них является подходящим кластера генов. В случае большого типа ПКС I или NRPS системах, где можно рассчитать количество наполнительного звеньев на основе количества модулей, или даже идентифицировать отдельные единицы расширителей с помощью анализа выбирающего доменов, получается легко. Тем не менее, в случае итеративно работающих ферментов Предсказание синтезированных соединений часто не представляется возможным, особенно если страв более 40 кластеров генов. В-пятых, природа очень сложна и полна еще неизвестных соединений. Часто биосинтез представляет собой смесь различных путей. Если новое соединение еще не идентифицированы, или не связанные с другим соединением, оно может быть трудно идентифицировать кластер, чтобы предложить модель биосинтез и доказать.
После того, как кластер идентифицируется, единственный метод кроссовера является хорошим и быстрый способ, чтобы проверить гипотезу. ПЦР, клонирование в вектор самоубийства, сопряжением, отбор позитивных клонов, и производство анализа являются единственными шаги, которые требуются. Одним из недостатков этого метода является то, что интеграция вектора в хромосому не является стабильным из-за дальнейших событий рекомбинации. Поэтому для того, чтобы дальнейшего анализа отдельных генов, точные делеции генов необходимы. Кроме того, это может быть сложно манипулировать штаммов Streptomyces на генетическом уровне.
Описанная процедура может быть назначена TO любое другое соединение , полученное штамма Streptomyces или другого микроорганизма. Знание о биосинтетического кластера генов и его синтезированного соединения дает нам дополнительные возможности для изменения уже существующих молекул с целью улучшения их для борьбы с множественной лекарственной устойчивостью патогенных микроорганизмов.
Раскрытие информации
The authors have nothing to disclose.
Благодарности
S. Zhang is funded by China Scholarship Council. The authors are very grateful to former people working on polyketomycin project in this lab and Prof. Dr. Hans-Peter Fiedler, University of Tübingen, for providing the polyketomycin producer. The research was supported by the DFG (RTG 1976).
Материалы
| Name | Company | Catalog Number | Comments |
| agar | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 5210.4 | |
| agarose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6352.4 | |
| D-mannitol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4175.1 | |
| glucose | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6780.1 | |
| LB | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X964.3 | |
| malt extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X976.2 | |
| MgCl2 | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2189.1 | |
| Peptone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | ||
| soy flour | W.Schoenemberger GmbH, Magstadt, Germany | Hensec-Vollsoja | |
| tryptic soy broth | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | X938.3 | Caso-Bouillon |
| yeast extract | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 2363.2 | |
| Solvents | |||
| Acetic acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 3738.5 | |
| Acetone | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 9372.2 | |
| Acetonitrile | Avantor Performance Materials B.V., Deventer, The Netherlands | JT-9012-03 | |
| Dichlorofrom | Fisher Scientific GmbH, Schwerte, Germany | 1530754 | |
| DMSO (Dimethyl sulfoxide) | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 4720.1 | |
| DMSO-d6 (Dimethyl sulfoxide-d6) 99.9 atom% D | ARMAR Chemicals, Döttingen, Switzerland | 15200.204 | |
| Ethyl acetate | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6784.4 | |
| Hydrochloric acid | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 6331.4 | |
| Methanol | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | 7342.1 | |
| Enzymes | |||
| restriction enzymes | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| polymerase | New England Biolabs GmbH, Frankfurt am Main, Germany | ||
| DNA Polymerase I Large (Klenow) Fragment | Promega GmbH, Mannheim, Germany | ||
| Antibiotics | |||
| apramycin | AppliChem GmbH, Darmstadt, Germany | A7682.0005 | |
| fosfomycin | Sigma-Aldrich Chemie GmbH, Taufkirchen, Germany | P5396-50G | |
| kanamycin | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | T832.2 | |
| Plasmid/Vectors | |||
| pKC1132 | Bierman et al. 1992 | ||
| Primer | |||
| pokPI_for | TGATGGTGCCGCTGGCCATGG | Primer to amplify fragment containing pokPI gene | |
| pokPI_rev | AGCGTTCACTGTTCCGCCCGAC | ||
| Bacterial strains | |||
| Bacillus subtilis COHN ATCC6051 | Gram-positive test strain | ||
| Escherichia coli ET12567 pUZ8002 | MacNeil et al. 1992 | strain for conjugation | |
| Escherichia coli XL1 Blue | Agilient Technologies, Santa Clara, USA | Gram-negative test strain + cloning host | |
| Streptomyces diastatochromogenes Tü6028 | Paululat et al., 1999 | Polyketomycin producer | |
| Online services | |||
| antismash (Antibiotics and Secondary Metabolite Analysis Shell) | http://antismash.secondarymetabolites.org/ | Detection of secondary metabolite gene cluster | |
| BLAST (Basic Local Alignment Search Tool) | http://blast.ncbi.nlm.nih.gov/Blast.cgi | finds regions of similarity between biological sequences | |
| GenDB | https://www.uni-giessen.de/fbz/fb08/Inst/bioinformatik/software/gendb | Annotation of ORFs | |
| MiBIG (Minimum Information about a Biosynthetic Gene cluster) | http://mibig.secondarymetabolites.org/ | Database of biosyntetic gene clusters | |
| NaPDoS | http://napdos.ucsd.edu/ | Detection of seconary metabolite gene cluster | |
| NCBI Prokaryotic Genome Annotation Pipeline | http://www.ncbi.nlm.nih.gov/genome/annotation_prok/ | Annotation of ORFs | |
| NRPSpredictor | http://nrps.informatik.uni-tuebingen.de/ | Detection of NRPS domains | |
| Prokka (rapid prokaryotic genome annotation) | http://www.vicbioinformatics.com/software.prokka.shtml | Annotation of ORFs | |
| RAST (rapid annotation using subsystems technology) | http://rast.nmpdr.org/ | Annotation of ORFs | |
| Other programs | |||
| Chem Station Rev. A.09.03 | Agilent Technologies, Waldbronn, Germany | Handling program for HPLC | |
| Clone Manager Suite 7 | Scientific and Educational Software, Cary, USA | Designing Cloning Experiment | |
| Newbler v2.8 | Roche Diagnostics | Alignment of sequencing reads | |
| Machines | |||
| Centrifuge Avanti J-6000, Rotor JA-10 | Beckman Coulter GmbH, Krefeld, Germany | ||
| HPLC/MS | Agilent Technologies, Waldbronn, Germany | ||
| _Autosampler: G1313A | Agilent Technologies, Waldbronn, Germany | ||
| _Pre-column: XBridge C18 (20 mm x 4.6 mm; Particle size: 3.5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Column:Xbridge C18 (100 mm × 4.6 mm; Particle size: 3.5 μm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Pre-Column: Zorbax B-C18 (9.4 x 150 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _semi-prep Column: Zorbax B-C18 (9.4 x 20 mm; Particle size: 5 µm) | Agilent Technologies, Waldbronn, Germany | ||
| _Degasser: G1322A | Agilent Technologies, Waldbronn, Germany | ||
| _Quarternary pump: G1311A | Agilent Technologies, Waldbronn, Germany | ||
| _Diode array detector (DAD )G1315B (λ = 254 nm and 400 nm) | Agilent Technologies, Waldbronn, Germany | ||
| _Quadrupole mass detector (MSD) G1946D(2-3000 m/z) | Agilent Technologies, Waldbronn, Germany | ||
| rotary evaporator | |||
| _heating bath Hei-VAP Value/G3 | Heidolph Instruments GmbH & Co.KG, Schwabach, Germany | ||
| _vacuum pump system SC 920 G | KNF Global Strategies AG, Sursee, Switzerland | ||
| Other material | |||
| Sephadex LH20 | GE Healthcare, | ||
| Chromafil PVDF-45/15MS (pore size 0.45 µm; filter Ø15 mm) | MACHEREY-NAGEL GmbH & Co. KG, Düren, Germany | ||
| SPE column Oasis HLB 20 35 cc (6g) | Waters GmbH, Eschborn, Germany | ||
| E. coli | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Bacillus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: LB, Composition: LB, Amount to 1 L H2O: 20 g | |
| Streptomyces sp. | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: TSB, Composition: CASO Boullion, Amount to 1 L H2O: 30 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Yeast extract, Amount to 1 L H2O: 10 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Peptone, Amount to 1 L H2O: 20 g | |
| fungus | Carl Roth GmbH + Co. KG, Karlsruhe, Germany | Medium: YPD, Composition: Glucose, Amount to 1 L H2O: 20 g | |
| for agar plates add 2% agar | |||
Ссылки
- Fleming, A. On the antibacterial action of cultures of a penicillin, with special reference to their use in isolation of B. influenzae. Br J Exp Pathol. 10 (3), 226-236 (1929).
- Lemke, A., Kiderlen, A. F., Kayser, O. Amphotericin B. Appl. Microbiol. Biotechnol. 68 (2), 151-162 (2005).
- Kirkpatrick, P., Raja, A., LaBonte, J., Lebbos, J. Daptomycin. Nat. Rev. Drug Discov. 2 (12), 943-944 (2003).
- Zakeri, B., Wright, G. D. Chemical biology of tetracycline antibiotics. Biochem. cell Biol. 86 (2), 124-136 (2008).
- Schatz, A., Bugle, E., Waksman, S. A. Streptomycin, a Substance Exhibiting Antibiotic Activity Against Gram-Positive and Gram-Negative Bacteria. Exp. Biol. Med. 55 (1), 66-69 (1944).
- Burg, R. W., et al. Avermectins, new family of potent anthelmintic agents: producing organism and fermentation. Antimicrob. Agents Chemother. 15 (3), 361-367 (1979).
- Egerton, J. R., Ostlind, D. A., et al. Avermectins, new family of potent anthelmintic agents: efficacy of the B1a component. Antimicrob. Agents Chemother. 15 (3), 372-378 (1979).
- Walsh, C. T., Fischbach, M. A. Natural products version 2.0: connecting genes to molecules. J. Am. Chem. Soc. 132 (8), 2469-2493 (2010).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces sp. MK277-AF1. II. Structure determination. J. Antibiot. (Tokyo). 51 (1), 26-32 (1998).
- Paululat, T., Zeeck, A., Gutterer, J. M., Fiedler, H. P. Biosynthesis of polyketomycin produced by Streptomyces diastatochromogenes.Tü6028. J. Antibiot. (Tokyo). 52 (2), 96-101 (1999).
- Momose, I., et al. Polyketomycin, a new antibiotic from Streptomyces. sp. MK277-AF1. I. Taxonomy, production, isolation, physico-chemical properties and biological activities. J. Antibiot. (Tokyo). 51 (1), 21-25 (1998).
- Daum, M., et al. Organisation of the biosynthetic gene cluster and tailoring enzymes in the biosynthesis of the tetracyclic quinone glycoside antibiotic polyketomycin. Chembiochem. 10 (6), 1073-1083 (2009).
- Bode, H. B., Bethe, B., Höfs, R., Zeeck, A. Big effects from small changes: possible ways to explore nature's chemical diversity. Chembiochem. 3 (7), 619-627 (2002).
- Wolfender, J. -. L. HPLC in Natural Product Analysis: The Detection Issue. Planta Med. 75 (07), 719-734 (2009).
- Stahl, E. . Thin-layer chromatography. A laboratory handbook. , (1967).
- Snyder, L. R., Kirkland, J. J., Glajch, J. L. Preparative HPLC Separation. Pract. HPLC Method Dev. , 616-642 (2012).
- Granath, K. A., Kvist, B. E. Molecular weight distribution analysis by gel chromatography on sephadex. J. Chromatogr. A. 28, 69-81 (1967).
- Jackman, L. M., Sternhell, S. Application of Nuclear Magnetic Resonance Spectroscopy in Organic Chemistry. Int. Ser. Org. Chem. , (1969).
- Xian, F., Hendrickson, C. L., Marshall, A. G. High resolution mass spectrometry. Anal. Chem. 84 (2), 708-719 (2012).
- Metzker, M. L. Sequencing technologies - the next generation. Nat. Rev. Genet. 11 (1), 31-46 (2010).
- Tatusova, T., et al. Prokaryotic Genome Annotation Pipeline. NCBI Handb. 2nd Ed. , (2013).
- Angiuoli, S. V., et al. Toward an online repository of Standard Operating Procedures (SOPs) for (meta)genomic annotation. OMICS. 12 (2), 137-141 (2008).
- Aziz, R. K., et al. The RAST Server: rapid annotations using subsystems technology. BMC Genomics. 9, 75 (2008).
- Overbeek, R., et al. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST). Nucleic Acids Res. 42, 206-214 (2014).
- Brettin, T., et al. RASTtk: a modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes. Sci. Rep. 5, 8365 (2015).
- Seemann, T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Meyer, F. GenDB--an open source genome annotation system for prokaryote genomes. Nucleic Acids Res. 31 (8), 2187-2195 (2003).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. J. Mol. Biol. 215 (3), 403-410 (1990).
- Madden, T. The BLAST Sequence Analysis Tool. NCBI Handb. 2nd Ed. , (2003).
- Marchler-Bauer, A., et al. CDD: NCBI's conserved domain database. Nucleic Acids Res. 43, 222-226 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Res. 39, 339-346 (2011).
- Blin, K., et al. antiSMASH 2.0--a versatile platform for genome mining of secondary metabolite producers. Nucleic Acids Res. 41, 204-212 (2013).
- Weber, T., et al. antiSMASH 3.0 - a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Res. 43, 237-243 (2015).
- Ziemert, N., et al. The natural product domain seeker NaPDoS: a phylogeny based bioinformatic tool to classify secondary metabolite gene diversity. PLoS One. 7 (3), 34064 (2012).
- Rausch, C., Weber, T., Kohlbacher, O., Wohlleben, W., Huson, D. H. Specificity prediction of adenylation domains in nonribosomal peptide synthetases (NRPS) using transductive support vector machines (TSVMs). Nucleic Acids Res. 33 (18), 5799-5808 (2005).
- Röttig, M., et al. NRPSSpredictor2--a web server for predicting NRPS adenylation domain specificity. Nucleic Acids Res. 39, 362-367 (2011).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nat. Chem. Biol. 11 (9), 625-631 (2015).
- Mullis, K., et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb. Symp. Quant. Biol. 51, 263-273 (1986).
- Bierman, M., et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp. Gene. 116 (1), 43-49 (1992).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli.using the heat shock method. J. Vis. Exp. (6), e253 (2007).
- Bimboim, H. C., Doly, J. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Res. 7 (6), 1513-1523 (1979).
- MacNeil, D. J., et al. Analysis of Streptomyces avermitilis. genes required for avermectin biosynthesis utilizing a novel integration vector. Gene. 111 (1), 61-68 (1992).
- Pospiech, A., Neumann, B. A versatile quick-prep of genomic DNA from Gram-positive bacteria. Trends Genet. 11 (6), 217-218 (1995).
- Makitrynskyy, R., et al. Pleiotropic regulatory genes bldA, adpA and absB are implicated in production of phosphoglycolipid antibiotic moenomycin. Open Biol. 3 (10), 130121 (2013).
- Kalan, L., et al. A cryptic polyene biosynthetic gene cluster in Streptomyces calvus is expressed upon complementation with a functional bldA gene. Chem. Biol. 20 (10), 1214-1224 (2013).
- Gessner, A., et al. Changing Biosynthetic Profiles by Expressing bldA in Streptomyces Strains. ChemBioChem. 16 (15), 2244-2252 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены