Method Article
Un método de extracción fraccionada simple para el análisis comprensivo de metabolitos, lípidos y proteínas a partir de una sola muestra
En este artículo
Resumen
Se presenta un protocolo para la extracción completa de lípidos, metabolitos y proteínas de tejidos biológicos utilizando una muestra.
Resumen
La comprensión de los sistemas biológicos complejos requiere la medición, el análisis y la integración de múltiples clases de compuestos de la célula viva, usualmente determinada por transcriptomic, proteómica, metabolómica y lipidomic mediciones. En este protocolo, introducimos un método simple para la extracción reproducible de metabolitos, lípidos y proteínas de tejidos biológicos usando una sola alícuota por muestra. El método de extracción se basa en un sistema de metil -tert -butil-éter: metanol: agua para el reparto líquido: líquido de los metabolitos hidrófobos y polares en dos fases inmiscibles junto con la precipitación de proteínas y otras macromoléculas como un gránulo sólido. Este método, por lo tanto, proporciona tres fracciones diferentes de composición molecular específica, que son totalmente compatibles con tecnologías comunes de "alto rendimiento", tales como cromatografía líquida (LC) o cromatografía de gases (GC) acoplada a espectrómetros de masas. A pesar de que el método fue initDesarrollado para el análisis de diferentes muestras de tejido vegetal, ha demostrado ser totalmente compatible para la extracción y análisis de muestras biológicas de sistemas tan diversos como algas, insectos y tejidos de mamíferos y cultivos celulares.
Introducción
La biología de los sistemas, que surgió a mediados del siglo pasado 1 y fue adelantada por el análisis a gran escala de los conjuntos de datos genómicos y transcriptómicos 2 , 3 , se ha convertido en un nuevo e indispensable enfoque para el análisis de sistemas biológicos complejos 4 , 5 . El principal objetivo de la biología de sistemas es descifrar las interacciones y dependencias de los componentes en los sistemas biológicos y vincular el vínculo entre los genotipos, su realización, las transformaciones moleculares y los fenotipos resultantes. En consecuencia, la integración de conjuntos de datos completos, producidos por los diversos enfoques analíticos a gran escala, a saber, genómica, transcriptómica, metabolómica, lipidómica y proteómica y su análisis computacional se ha convertido en un requisito previo para la descripción y comprensión de sistemas biológicos complejos.
Bas Sobre la enorme diversidad química y la complejidad de los componentes biológicos de cualquier sistema vivo, la producción de grandes y completos conjuntos de datos "ómicos" depende en gran medida de la calidad del método de extracción aplicado 9 . Además de la calidad del método de extracción, la economía del método es importante; Esto significa que sería deseable obtener tanta información molecular de la menor entrada posible de la muestra. Frecuentemente, las cantidades de muestra pueden ser limitantes y, por lo tanto, es altamente deseable hacer uso de un método de extracción, que puede derivar tantas clases moleculares a partir de una sola extracción de una muestra dada. Esto significa que en lugar de utilizar varios métodos de extracción especializados para la extracción de diferentes clases de compuestos de diferentes alícuotas de muestra de la misma muestra, se emplea un método de extracción secuencial que fracciona los constituyentes moleculares de una alícuota en diferentes fracciones moleculares.
El método común utilizado para estos métodos de extracción fraccionada se basa en el método de dos fases de extracción de lípidos de Folch et al, desarrollado en 1957 10. Este método se basa en un cloroformo: metanol / agua de partición de metabolitos polares e hidrófobos y fue Con la evolución de la biología de sistemas multi-ómicos, el método de Folch se mejoró adicionalmente, paso a paso, mediante su utilización para la partición de muestras de proteínas y metabolitos polares y lípidos para el análisis de lípidos de alta calidad. Metabolómica a base de cromatografía de gases y líquidos y lipidómica de compuestos polares e hidrófobos, además de la proteómica líquida a base de cromatografía 11 , 12 , 13 , 14. Desafortunadamente, todos estos métodos se basan en un método de extracción a base de cloroformo que no sólo Conduce al indeseable foRmación de la pastilla de proteína como una interfase entre la fase polar y la fase lipídica, pero que también es un disolvente indeseable desde una perspectiva de química verde 15 , 16 . Sin embargo, el disolvente metil tert -butil éter (MTBE) supera ambos de estos problemas antes mencionados y es un sustituto adecuado para el cloroformo. Con base en estos requerimientos, decidimos establecer un MTBE: metanol: método de extracción basado en agua, el cual cumple con todas las especificaciones antes mencionadas y por lo tanto funciona como un punto de partida ideal para un análisis multi-ómico integral 16 .Este protocolo guía al usuario paso a paso a través del flujo de trabajo simple, rápido y reproducible de la preparación de la muestra, incluyendo la resolución de problemas comunes. Además, introduciremos brevemente datos analíticos ejemplares de la cromatografía líquida de ultra-rendimiento-espectrometría de masas (UPLC-MS) -basLipidómica, metabolómica y proteómica perfil de muestras de tejidos vegetales. A pesar de que los ejemplos dados se derivan de 50 mg de una muestra de tejido foliar de Arabidopsis thaliana , este protocolo se ha utilizado para varias otras muestras y tejidos biológicos, incluyendo algas 17 , 18 , insectos 19 y células, órganos y tejidos de mamíferos 20 , 22 . El alcance del protocolo de extracción presentado es proporcionar una descripción clara y detallada del manejo de la muestra previa a la extracción y del propio procedimiento de extracción. A pesar de que ofrecemos tres breves ejemplos de aplicación analítica, se puede obtener información detallada sobre el manejo de los datos antes y después de la analítica en nuestras publicaciones anteriores 16 , 23 , 24 , 25 , 26 .
Protocolo
Precaución : El metanol (MeOH) y el metil -tert -butiléter (MTBE), utilizados durante la extracción, son inflamables y pueden presentar irritación respiratoria, ocular o cutánea, por exposición prolongada y / o contacto. Por favor, manipúelas cuidadosamente en una campana extractora y use los procedimientos de seguridad adecuados durante la extracción (bata de laboratorio, gafas de seguridad, guantes, etc. ). El nitrógeno líquido y el hielo seco, usados en varias etapas de este protocolo, pueden causar quemaduras graves por contacto prolongado con la piel. Por favor guárdelas cuidadosamente usando guantes y gafas protectoras. Los usuarios pueden utilizar diferentes productos químicos, reactivos o estándares internos para el análisis de muestras, algunos de los cuales pueden ser tóxicos. Examine las hojas de datos de seguridad de los productos químicos correspondientes.
1. Recolección y recolección de muestras biológicas
- Prepare tubos de recolección rotulados.
NOTA: Aquí, coseche las muestras biológicas en envases rotulados, de 2 ml, de fondo redondo y segurosK que contenían dos bolas metálicas de 5 mm de diámetro para el homogeneizador de tejidos. - Preparar una llenado de nitrógeno líquido dewar.
- Cosecha de la muestra biológica y congelación instantánea del tejido en nitrógeno líquido. Realice este paso tan pronto como sea posible en pocos segundos para evitar los cambios metabólicos inducidos por la herida.
Nota: Para fines de demostración, use hojas de roseta de Arabidopsis thaliana (Col-0) de tipo silvestre de 30 días de edad, cultivadas en suelo bajo condiciones de días largos. - Mantenga las muestras recolectadas en hielo seco para estancias cortas o almacénelas a -80 ° C por períodos más largos.
2. Molienda y trastorno de tejidos
- Pre-enfriar los porta-tubos del homogeneizador de tejidos en nitrógeno líquido durante al menos 10 min. Si no hay disponible un homogeneizador de tejidos, utilice morteros y pilones limpios y preenfriados.
- Tomar las muestras del nitrógeno líquido, hielo seco o -80 ° C congelador y colocarlos en el pre-enfriadoPorta-tubos.
- Coloque rápidamente los porta tubos en el homogeneizador de tejidos.
- Moler el material biológico en un polvo fino y homogéneo. Utilice 20 Hz durante 1 min para las hojas.
Nota; El tiempo y la velocidad de homogeneización pueden variarse dependiendo del tejido, asegurarse de que la muestra se homogeneiza en un polvo fino y que este polvo se mantiene congelado en cada paso de la homogeneización. - Tome las muestras biológicas de los porta-tubos y manténgalas congeladas hasta una extracción posterior.
3. Pesaje de los tejidos
- Utilice un balance analítico con suficiente precisión para las cantidades de muestra requeridas.
- Prepare un tubo de microcentrífuga de 2 ml de fondo redondo, de seguridad.
- Pre-enfriar los tubos y espátulas en nitrógeno líquido.
- Alícuota la cantidad requerida de polvo de tejido en el tubo de microcentrífuga de seguridad de 2 ml.
Precaución: Evite cualquier descongelación del material vegetal minimizando el tiempoLas muestras. - Devolver las muestras alicuotadas inmediatamente después de pesar en nitrógeno líquido.
- Registre para cada muestra el peso exacto. Utilice 10-50 mg ± 10% para la mayoría de los tejidos vegetales.
- Almacenar las muestras alícuotas a -80 ° C hasta su extracción.
4. Configuración del reactivo
- Utilizar una mezcla de extracción de metil terc -butil éter (MTBE) / metanol (MeOH).
- Para la preparación de 100 ml de disolvente de extracción, añadir 75 ml de MTBE a 25 ml de MeOH para preparar una mezcla de MTBE: MeOH (3: 1, vol / vol).
- Agregar normas internas para la normalización del post análisis de acuerdo con las necesidades analíticas. Típicamente, agregar 50 μl de 1,2-diheptadecanoil- sn -glicero-3-fosfocolina (1 mg / ml en cloroformo) como patrón interno para el análisis de lípidos basado en UPLC-MS, a~nadiendo 50 μl de sorbitol 13C 1 mg / ml en agua) como normas internas para el análisis basado en GC-MS deMetabolitos. Las normas internas para el análisis de metabolitos basados en UPLC-MS son 50 μl de corticosterona (1 mg / ml en metanol) y 25 μL de ampicilina (1 mg / ml en metanol).
- Transferir el disolvente a una botella de vidrio limpia que se enjuagó con una mezcla de MTBE: MeOH.
- Conservar la mezcla de extracción hasta 1 semana a 4 ° C.
NOTA: No almacene la mezcla de extracción por períodos más largos para mantener resultados reproducibles.
- Para inducir la separación de fases, utilizar agua (H2O) / metanol (MeOH).
- Para la preparación de 100 ml de H2O: MeOH, añadir 75 ml de H2O a 25 ml de MeOH para preparar H $ ₂ $ O: MeOH (3: 1, vol / vol).
- Transferir el disolvente a una botella de vidrio limpia que se enjuagó con una mezcla de H $ ₂ $ O: MeOH. Este disolvente se puede almacenar durante varias semanas a temperatura ambiente.
5. Extracción de Muestras
- Pre-enfriar la extracción m(MTBE: MeOH, 3: 1, vol / vol) a -20 ° C usando un sistema de refrigeración de líquido o un congelador de -20 ° C.
- Sacar las muestras alícuotas una por una y agregar 1 ml de la mezcla de extracción previamente enfriada a cada tubo de muestra. Precaución: Realice este paso rápidamente debido a la baja viscosidad del MTBE.
- Se mezcla inmediatamente en un mezclador de vórtice hasta que el tejido se homogeneiza bien dentro de la mezcla de extracción.
NOTA: Este paso es muy importante, ya que se requiere precipitar las proteínas e inactiva sus actividades enzimáticas. - Incubar todas las muestras en un agitador orbital a 100 rpm durante 45 min a 4 ° C.
- Sonicar las muestras durante 15 minutos en un baño de sonicación enfriado con hielo.
6. Fraccionamiento por separación de fases
- Añadir 650 μl de H 2 O: MeOH (3: 1, vol / vol) a cada tubo de muestra.
- Mezclar bien por vórtex durante 1 min.
- Centrifugar las muestras a una velocidad de 20.000 x g durante 5 min a 4 ° C.
NOTA: Después de este paso, hay dos fases líquidas inmiscibles con un gránulo sólido en el fondo del tubo.
Precaución: Manipular los tubos con cuidado para evitar la mezcla de las dos fases líquidas y evitar la alteración de la pastilla precipitada.
7. Alícuota de fracciones polares e hidrófobas
- Transferir 500 μL del disolvente de la fase superior, que contiene lípidos, a un tubo de microcentrífuga de 1,5 ml marcado.
NOTA: Las muestras de lípidos divididas en alícuotas pueden concentrarse directamente para el análisis inmediato de UPLC-MS (etapa 8.1) o almacenarse durante varias semanas a -80ºC. - Una vez que se eliminan los 500 μl de la muestra, se retira la fase lipídica restante usando una pipeta de 200 μl.
- Transferir 400 μl del disolvente de la fase inferior (metabolitos polares y semi-polares) en un tubo de microcentrífuga de 1,5 ml marcado. Las muestras polares alícuotas se pueden concentrar directamente para análisis UPLC-MS inmediato (paso 8.2) o almacenarse paraR varias semanas a -80 ° C.
- Tome una alícuota adicional de 200 μl para realizar análisis adicionales, por ejemplo, análisis de metabolitos a base de cromatografía de gases como se describe con precisión 16 .
- Eliminar el resto de la fase acuosa por pipeteado del exceso de volumen.
- Lavar la proteína obtenida, el almidón, el sedimento de la pared celular con 500 mu l de metanol agitando a fondo durante 1 minuto.
- Centrifugar las muestras a una velocidad de 10.000 x g durante 5 min a 4 ° C.
- Realizar la extracción de proteínas y la digestión (etapa 11) o el análisis de almidón / pared celular como se describe anteriormente [ 16] .
NOTA: Si no se utilizan inmediatamente, estos gránulos se pueden almacenar durante varias semanas a -80 ° C.
8. Concentración y almacenamiento de fracciones
- Evaporar el disolvente a partir de muestras de lípidos (del paso 7.1) en un concentrador de vacío sin calentamiento (durante 1-2 horas) o preferiblemente usar un nitrEvaporador de flujo de oxígeno para evitar las modificaciones oxidativas de los lípidos.
NOTA: Las muestras secas deben ser analizadas inmediatamente. Para el almacenamiento, dejar las muestras en solución de MTBE, idealmente en viales de vidrio (paso 7.1). - Evaporar el disolvente de las muestras acuosas (de la etapa 7.3 o 7.4) durante la noche en un concentrador de vacío sin calentamiento. NOTA: las muestras secas pueden almacenarse durante varias semanas a -80 ° C antes del análisis.
9. Análisis de lípidos usando UPLC-MS 24
- Se vuelven a suspender las fracciones de lípidos secas (del paso 8.1) en 400 μl de acetonitrilo: 2-propanol (7: 3, vol / vol).
- Transferir el líquido suficiente a los viales de vidrio y tapar firmemente.
- Coloque los viales de vidrio en un inyector automático refrigerado (4 ° C).
- Inyectar 2 μL por muestra y separar los lípidos en una columna de fase inversa (RP) C8 mantenida a 60 ° C usando un sistema UPLC funcionando a un caudal de 400 μl / min.
- Utilice el teléfono móvilFases descritas en la Tabla 1 para la separación cromatográfica.
- Adquirir los espectros de masas en modo de ionización positiva y negativa utilizando un instrumento MS adecuado que cubra el rango de masa entre 150 y 1.500 m / z.
10. Análisis de metabolitos polares y semi-polares utilizando UPLC-MS 25 .
- Vuelva a suspender la fase polar (del paso 8.2) en 200 μl de metanol UPLC: agua (1: 1, vol / vol).
- Transferir el líquido suficiente a los viales de vidrio y tapar firmemente.
- Coloque los viales de vidrio en un inyector automático refrigerado (4 ° C).
- Inyectar 2 μL de cada muestra y separar los metabolitos en una columna RP C18 mantenida a 40 ° C usando un sistema UPLC funcionando a un caudal de 400 μl / min.
- Utilizar las fases móviles para la separación cromatográfica con los parámetros dados en la Tabla 2 .
- Adquirir espectros de masas de exploración completa en modo de ionización positiva y negativaUtilizando un espectrómetro de masas adecuado que cubra un rango de masa entre 50 y 1.500 m / z.
11. Extracción, digestión y análisis de proteínas 16
- Vuelva a suspender el sedimento lavado de proteína / almidón / pared celular (del paso 7.8) en 200 μl del tampón de extracción de proteínas de elección. Nota: Utilizamos tampón urea / tiourea (urea 5 M, tiourea 2 mM, DTT 15 mM, CHAPS al 2% y inhibidores de la proteasa y fosfatasa) 27 .
- Sonicar las muestras durante 10 min en un baño sónico enfriado con hielo.
- Incubar las muestras durante 30 minutos en un agitador orbital (100 rpm) a temperatura ambiente.
- Centrifugar las proteínas disueltas a 10.000 x g durante 5 min.
- Recoger el sobrenadante de la proteína en un nuevo tubo.
- Determinar la concentración de proteínas del sobrenadante recogido [ 28] .
- Digerir 50 μg de proteína en solución con un protocolo de elección. Típicamente, utilice la mezcla de Trypsin / Lys-C accordiNg al manual de instrucciones.
- Después de la digestión, realizar la desalación de los péptidos antes de la espectrometría de masas utilizando puntas de la etapa C18 y eluir los péptidos digeridos [ 29] .
- Concentrar las muestras hasta la sequedad en un concentrador de vacío sin calefacción.
- Se vuelven a suspender las muestras en tampón de carga apropiado (por ejemplo acetonitrilo al 5%, ácido fórmico al 0,5%) y se analizan las mezclas de péptidos por LC-MS / MS usando un espectrómetro de masas de alta resolución conectado a un sistema nano LC.
NOTA: En el conjunto de datos de proteómica ejemplar presentado en este protocolo, se utilizó un gradiente como se describe en la Tabla 3 . - Establecer el espectrómetro de masas, utilizando una estrategia de la parte superior 15, donde una exploración completa (FS) fue seguido por un máximo de 15 datos dependientes MS / MS escanea, a los siguientes parámetros: El FS fue en el rango de masa 200-2000 m / Una resolución de 70.000 con un valor diana de 3x106 iones. Obtenga los escaneados MS / MS dependientes de los datos por higher-eneRgy disociación colisionaria (HCD). Establecer los valores objetivo de la MS / MS a 1e 5 iones, con un tiempo de llenado iónico máximo de 50 ms, una ventana de aislamiento de 4,0 m / z, la energía de colisión normalizada (NCE) del 30% y una relación de relleno inferior del 1%. Mida los iones MS / MS con una resolución de 17.500 y la exclusión dinámica se fija en 60 s.
Resultados
Los conjuntos completos de datos multi-ómicos son invaluables para la comprensión de sistemas biológicos complejos. La estrategia para un experimento biológico exitoso generalmente comienza a partir de un diseño experimental, una configuración del experimento y un desempeño satisfactorios, seguida de la recolección de muestras, extracción, adquisición de datos analíticos, procesamiento de datos sin procesar, análisis de datos estadísticos, identificación de metabolitos relevantes e interpretación de datos biológicos Incluyendo el mapeo de rutas y la visualización ( Figura 1 ).
En el protocolo de extracción que aquí se presenta, nos centramos en los pasos de recolección de muestras, manipulación y extracción, que se describen en la descripción detallada del flujo de trabajo en la Figura 2 . Para fines de demostración, se seleccionaron 50 mg de tejido foliar de Arabidopsis. Este material fue cosechado, molido y extraído antes de someterlo a tresEjemplares UPLC-MS plataformas analíticas, proporcionando datos que pueden utilizarse para el análisis lipídico, metabolómico y proteómico objetivo y no segmentado. Las Figuras 2 y 6 incluyen adicionalmente imágenes representativas de cómo, bajo condiciones estándar, el solvente de extracción debe ser similar. Además, se muestran ejemplos de muestras que contienen cantidades excesivas de macromoléculas precipitadas (proteínas y almidón) y muestras con homogeneización inadecuada de muestras ( Figura 3 ). La solución de problemas para estos dos problemas comunes se presenta en breve en la Figura 3, pero también se discute con más detalle en nuestra publicación anterior [ 16] .
Las figuras 4 y 5 describen ejemplos de tres cromatogramas analíticos derivados del lípido, el polar / semMetabolitos i-polares y análisis de proteínas. Los lípidos, que se tomaron de la fase superior MTBE ( Figura 2 ], se analizaron por cromatografía líquida de alta resolución de fase inversa (RP) C8 acoplada a espectrometría de masas de alta resolución. Los lípidos pueden ser adquiridos usando modos de ionización MS positivos y negativos ( Figura 4 , panel superior) 16 , 24 .
Los metabolitos primarios y secundarios polares y semi-polares se analizaron a partir de la fase polar (agua / metanol) ( Figura 2 ) mediante la fase inversa (RP) C18 UPLC-MS 25 . El método ilustrado, que utiliza la cromatografía de fase reversa, es altamente compatible con el análisis de los metabolitos semipolares (a saber, los metabolitos del metabolismo secundario de las plantas), que pueden analizarse utilizando modos de ionización positiva y negativa en el MS( Figura 4 , panel inferior) 16 . Los metabolitos más hidrófilos de esta fracción (azúcares, aminoácidos polares, etc.) que no muestran buena retención sobre el material en fase reversa pueden analizarse por otros métodos analíticos tales como GC-MS 16 o cromatografía líquida de interacción hidrofílica 30 .
Las proteínas, que se recuperaron de la pastilla sólida en el fondo del tubo de extracción ( Figura 2 ), se digirieron en solución y se analizaron usando una pistola LC-MS ( Figura 5 ), mientras que el protocolo para la extracción de almidón y pared celular Material se puede obtener de nuestro protocolo publicado anteriormente [ 16] .
En resumen, más de 200 especies lipídicas, 50 metabolitos semi-polares anotados y varios miles de proteínasNs se pueden identificar rutinariamente a partir de muestras del tipo utilizado en nuestro ejemplo. Además, el método mostró amplia aplicabilidad utilizando diferentes tejidos, órganos y material de cultivo celular ( Figura 6 ).

Figura 1: Flujo de trabajo general para análisis de omics no segmentados a gran escala. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2 : Preparación de muestras y flujo de trabajo de extracción para el análisis de lípidos, metabolitos y proteínas a partir de una única alícuota de una muestra biológica.Haga clic aquí para ver una versión más grande de esta figura.
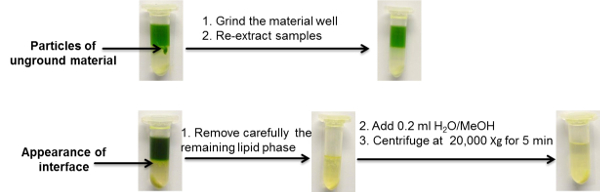
Figura 3 : Ejemplos ilustrativos de problemas comúnmente observados usando protocolos de extracción de partición de dos fases. Haga clic aquí para ver una versión más grande de esta figura.
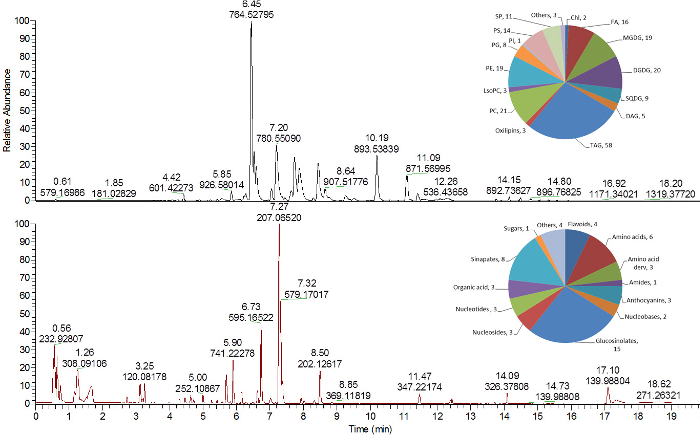
Figura 4 : Cromatogramas representativos de lípidos y metabolitos semipolares de extractos de hoja de Arabidopsis thaliana . Cromatograma de pico baseS de lípidos (panel superior) y metabolitos semipolares (panel inferior) analizados en modo de ionización positiva 16 . Los gráficos circulares en la esquina superior derecha de cada cromatograma muestran el número de lípidos y metabolitos identificados asignados a diferentes clases de productos químicos. Chl, clorofilas; DAG, diacilglicérido; DGDG, digalactosildiacilglicerol; FA, ácido graso; LysoPC, lisofosfatidilcolina; MGDG, monogalactosildiacilglicerol; PC, fosfatidilcolina; PE, fosfatidiletanolamina; PG, fosfatidilglicerol; PI, fosfatidilinositol; PS, fosfatidilserina; SP, esfingolípido; SQDG, sulfoquinovosyldiacylglycerol; TAG, triacilglicérido. Haga clic aquí para ver una versión más grande de esta figura.
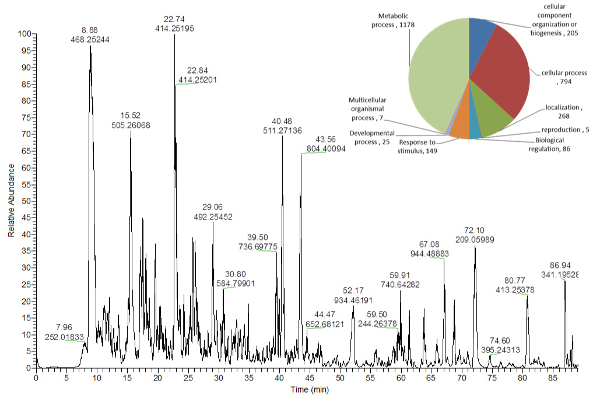
Figura 5 Trong>: Cromatograma de Pico Base Representativo de Péptidos de Extractos de Hoja de Arabidopsis thaliana .
El gráfico circular muestra en la esquina superior derecha indica el número de proteínas identificadas asignadas a diferentes procesos biológicos [ 16] . Haga clic aquí para ver una versión más grande de esta figura.

Figura 6 : Ejemplos de extracción representativos de diversos tipos de tejidos de Arabidopsis thaliana de tipo salvaje .
Todas las muestras se extrajeron utilizando 50 mg de peso fresco de los tejidos indicados."> Haga clic aquí para ver una versión más grande de esta figura.
| Tiempo (min) | % De Tampón A a Tampón B | |
| 0 a 1 min | 45% A | Tampón A: Acetato de NH _ {4} 1 M, ácido acético al 0,1% en agua UPLC de grado MS |
| 1 a 4 min | Gradiente lineal de 45% A a 25% A | Tampón B: 1% de NH _ {4} - Acetato 1M, ácido acético al 0,1% en acetonitrilo / isopropanol 7: 3, (v: v) |
| 4 a 12 min | Gradiente lineal de 25% A a 11% A | Caudal 400 μL / min |
| 12 a 15 min | Gradiente lineal de 11% A a 0% A | Volumen de inyección 2 μL |
| 15 a 19,5 min | Lavar la columna durante 4,5 min con 0% de A | |
| 19,50 a 19,51 min | Regresar al 45% A | |
| 19,51 a 24 min | Equilibrar con 45% de A |
Tabla 1: Parámetros de gradiente para RP-UPLC Separación de lípidos. RP, fase inversa. UPLC, cromatografía líquida de ultra-rendimiento.
| Tiempo (min) | % De Tampón A a Tampón B | |
| 0 a 1 min | 99% A | Tampón A: ácido fórmico al 0,1% en agua de grado UPLC |
| 1 a 11 min | Gradiente lineal de 99% A a 60% A | Tampón B: ácido fórmico al 0,1% en UPLC grad acetonitrilo |
| 11 a 13 min | Gradiente lineal de 60% A a 30% A | Caudal 4001 L / min |
| 13 a 15 min | Gradiente lineal desde 30% A hasta 1% A | Volumen de inyección 2 μL |
| 15 a 16 min | Lavar la columna durante 1 min con 1% de A | |
| 16 a 17 min | Gradiente lineal desde1% A hasta 99% A | |
| 17 a 20 min | Equilibrar durante 3 min a 99% A |
Tabla 2: Parámetros de gradiente para RP-UPLC Separación de metabolitos polares y semi-polares. RP, fase inversa. UPLC, cromatografía líquida de ultra-rendimiento.
| Tiempo (min) | % Tampón B a Tampón A | |
| 0 a 5 min | Gradiente lineal de 0 a 10% | Tampón A: ácido fórmico al 0,1% en agua de grado UPLC |
| 5 a 80 min | Gradiente lineal de 10% a 40% | Tampón B: ácido fórmico al 0,1% en acetonitrilo al 60% de grado UPLC |
| 80 a 85 min | Gradiente lineal de 40% a 60% | Caudal 300 nL / min |
| 85 a 86 min | Gradiente lineal de 60% a 95% | Volumen de inyección 5 μL |
| 86 a 91 min | La columna de lavado durante 5 min con 95% | |
| 91 a 92 min | Gradiente lineal del 95% al 0% | |
| 93 a 110 min | Equilibrar la columna durante 17 min a 0% |
Tabla 3: Parámetros de gradiente para la separación Nano-LC de péptidos. LC, cromatografía líquida.
Discusión
En este artículo, describimos e ilustramos un protocolo de extracción simple y altamente aplicable para análisis lipídomico, metabolómico y proteómico completo a partir de una sola muestra de hoja de 50 mg. El método se ha utilizado previamente en varios estudios, que han sido publicados en diversos artículos 17 , 18 , 19 , 20 , 21 , 22 , 23 , 24 , 25 , 26 , 31 , 32 , 33 , 34 , 35 , 36 , 37. Y probado, además de su línea directaFlujo de trabajo y alta aplicabilidad para ser robustos y reproducibles.
Las aplicaciones aquí proporcionadas muestran algunos métodos de rutina para la selección inicial de una muestra biológica compleja. Estos conjuntos de datos metabolómicos y lipidómicos ilustrados a gran escala pueden proporcionar información completa sobre los cambios amplios o específicos en el metabolismo del sistema biológico analizado, mientras que los datos obtenidos a partir del análisis de proteínas proporcionan información sobre las variaciones cuantitativas (abundancia) y cualitativas ) Cambios de enzimas, proteínas estructurales o factores de transcripción (TFs) que controlan las funciones celulares y la maquinaria. De acuerdo con esto, los datos integrales de los ómicos tienen el potencial de revelar información inicial sobre los posibles cambios inducidos por las perturbaciones genéticas o bióticas y / o abióticas de un sistema biológico, al dilucidar los cambios moleculares de diversas moléculas asociadas a vías metabólicas o procesos celulares específicos.
Por supuesto, A largo plazo, es absolutamente esencial, para un análisis exitoso de biología de sistemas, maximizar el número de entidades moleculares analizadas y anotadas, permitiendo el seguimiento de las funciones y actividades celulares lo más completamente posible. Con este fin, las fracciones obtenidas podrían aplicarse adicionalmente a diversos métodos analíticos, dirigiéndose a otros compuestos o clases de compuestos ( Figura 4 ).
Dicho esto, hay que mencionar que la estrategia de análisis global de los datos obtenidos puede ser seguida por dos estrategias diferentes: por un lado, hemos estado enfatizando la elucidación de funciones celulares por cuantificación de compuestos conocidos. Por otra parte, muchos de los metabolitos y lípidos medidos todavía no se conocen ni anotan. Estas, sin embargo, las mediciones compuestas sin anotar también contienen un montón de información significativa, que, puede ser utilizado por métodos estadísticos para la clasificación o discriminación bEntre grupos o tratamientos 20 , 21 , 22 .
Sin embargo, estos compuestos desconocidos, especialmente los que son relevantes para la clasificación de grupos o que actúan como biomarcadores, necesitan ser identificados. Este proceso de identificación es, por desgracia, bastante tedioso y no puede lograrse sin medidas o estrategias analíticas adicionales 38 . Como puede verse en la figura 4 , el número de compuestos sin anotar es bastante alto (en realidad, la gran mayoría). Sin embargo, como se mencionó anteriormente, estos picos cromatográficos pueden ser manejados dentro del análisis de datos y por lo tanto las entidades afectadas significativamente pueden ser elucidadas y sometidas a estrategias de identificación adicionales.
En resumen, podemos concluir que el protocolo aquí presentado ofrece varias ventajas para la biología de sistemas experimentales, así como para aplicaciones estadísticas clásicasAciones.
En primer lugar, ya que todas las fracciones se extraen de una sola muestra, la variación entre los diferentes conjuntos de datos experimentales (lípidos, metabolitos, proteínas) se reduce significativamente ya que cada conjunto de datos se deriva de la misma alícuota de la muestra. Esto conduce claramente a una mayor comparabilidad de los resultados obtenidos.
En segundo lugar, el método es fácilmente escalable y lo hace por lo tanto altamente compatible con cantidades de muestra pequeñas a grandes. Usamos rutinariamente 10-100 mg de muestras de tejido, pero los estudios lipidómicos exitosos también se han realizado en tan sólo 20 semillas de Arabidopsis [ 31] . Especialmente la compatibilidad con pequeñas cantidades de muestras hace que este método sea aplicable si se dispone de cantidades limitadas de tejidos o muestras biológicas. Sin embargo, incluso si se dispone de suficiente material de muestra, el método presentado aquí ofrece la ventaja de utilizar estas muestras en un número mayor de repeticiones experimentales en lugar de uCantarlos para diferentes procedimientos de extracción. Esto permite un mejor y más refinado análisis estadístico de datos.
En tercer lugar, dado que el método se basa en un fraccionamiento líquido-líquido de moléculas polares y no polares, proporciona, a diferencia de los métodos sencillos de extracción en una fase ( por ejemplo extracciones con metanol), una importante etapa de descomplexación en el procedimiento. Esta descomplexación de muestra eficiente conduce a una purificación parcial de las fracciones individuales debido a la separación de moléculas que interfieren químicamente entre sí. Por consiguiente, el proceso de separación química no sólo proporciona una ventaja práctica para la distribución alícuota sistemática de las muestras extraídas en diferentes clases químicas, sino que también mejora las medidas analíticas individuales, ya que elimina compuestos contaminantes de las diferentes fracciones. Claramente, podemos observar que especialmente los lípidos, que están divididos a la fase orgánica y que usualmente afectan negativamente a laAnálisis cromatográfico de compuestos polares, estará casi completamente ausente de la fracción polar. Lo mismo es cierto para el análisis de los lípidos hidrófobos, que se agotarán de los compuestos polares. Además de la purificación de compuestos polares y no polares entre sí, se agotan y se recogen las proteínas y otras macro-moléculas de la muestra, que no sólo proporciona una fracción separada, que puede utilizarse para análisis de proteína, almidón y pared celular 16 , sino también Conduce a una muestra más limpia dentro de las fracciones individuales. Esto es especialmente relevante, ya que se sabe que la presencia de macromoléculas grandes, conduce a daños o al menos una vida más corta de las columnas analíticas.
Por último, pero no menos importante, el método de extracción de MTBE descrito, que se basa en el disolvente de sustitución de cloroformo menos peligroso y más favorable 15 , ya ha sido demostrado por varios estudios de nuestro grupo,Para diferentes muestras biológicas de plantas 16 , algas 17 , 18 , moscas 19, pero también varios tejidos, órganos o células de mamíferos 20 , 21 , 22 .
Divulgaciones
Los autores no tienen nada que revelar
Agradecimientos
MS cuenta con el apoyo de una beca de doctorado del programa GERLS-DAAD. Quisiéramos agradecer al Dr. Andrew Wiszniewski por leer y comentar sobre el manuscrito. Estamos muy agradecidos a todos los miembros del laboratorio de Giavalisco en el Instituto Max-Planck de Fisiología de Plantas Moleculares, Golm, Alemania por su ayuda.
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents and standards | |||
| Ampicillin | Sigma Aldrich | A9393-5G | Internal standard for metabolites |
| Corticosterone | Sigma Aldrich | 27840-500MG | Internal standard for metabolites, HPLC grade |
| 13C Sorbitol | Sigma Aldrich | 605514 | Internal standard for metabolites, ISOTEC® Stable Isotopes |
| 1,2-diheptadecanoyl-sn-glycero-3-phosphocholine (17:0 PC) | Avanti Polar Lipids | 850360P | Internal standard for lipids |
| Methanol (MeOH) | Biosolve Chemicals | 13684102 | ULC-MS grade |
| Water | Biosolve Chemicals | 23214102 | ULC-MS grade |
| Methyl tert-butyl ether (MTBE) | Biosolve Chemicals | 13890602 | HPLC grade |
| Trypsin/Lys-C mix | Promega | V5072 | Enzymatic digestion of proteins |
| Equipment | |||
| Balance | Sartorius Corporation | 14 557 572 | |
| Tissue grinding mixer mill | Retsch, Mixer Mill MM 300 | 20.746.0001 | |
| Mortar and pestle | Sigma Aldrich | Z247464-1EA | |
| Vortex mixer | Vortex-Genie 2, Model G560 | SI-0236 | |
| Vacuum concentrator | Scan Speed Maxi Vac Alpha Evaporators | 7.008.500.002 | |
| 2 mL Safe-lock microcentrifuge tubes | Eppendorf | 30120094 | Used for sample extarction |
| 1.5 mL Safe-lock microcentrifuge tubes | Eppendorf | 30120086 | Used for fractions |
| Shaker | Eppendorf Thermomixer 5436 | 2050-100-05 | |
| Sonicator | USC 300 TH | 142-0084 | |
| Refrigerated microcentrifuge | Eppendorf, model 5427R | 22620701 | |
| UPLC system | Waters Acquity UPLC system (Waters, Machester, UK) | ||
| MS system | Exactive, Orbitrap-type, MS (Exactive, Thermo-Fisher, Bremen, Germany). | ||
| Reversed Phase (RP) Bridged Ethyl Hybrid (BEH) C8 column (100 mm × 2.1 mm containing 1.7 μm diameter particles) | Waters, Machester, UK | 186002878 | Analysis of lipids |
| RP High Strength Silica (HSS) T3 column (100 mm × 2.1 mm containing 1.8 μm diameter particles) | Waters, Machester, UK | 186003539 | Analysis of metabolites |
| Q ExactivePlus high resolution mass spectrometer connected to an EASY-nLC 1000 system | Thermo-Fisher, Bremen, Germany | Analysis of peptides |
Referencias
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Yang, M. Q., et al. The emerging genomics and systems biology research lead to systems genomics studies. BMC Genomics. 15, 1 (2014).
- Sheth, B. P., Thaker, V. S. Plant systems biology: insights, advances and challenges. Planta. 240 (1), 33-54 (2014).
- Ideker, T., Galitski, T., Hood, L. A new approach to decoding life: Systems biology. Annu Rev Genom Hum G. 2, 343-372 (2001).
- Somvanshi, P. R., Venkatesh, K. V. A conceptual review on systems biology in health and diseases: from biological networks to modern therapeutics. Syst Synth Biol. 8 (1), 99-116 (2014).
- Oliver, S. G., Winson, M. K., Kell, D. B., Baganz, F. Systematic functional analysis of the yeast genome. Trends Biotechnol. 16 (9), 373-378 (1998).
- Tweeddale, H., Notley-McRobb, L., Ferenci, T. Effect of slow growth on metabolism of Escherichia coli, as revealed by global metabolite pool ("metabolome") analysis. J Bacteriol. 180 (19), 5109-5116 (1998).
- Kim, H. K., Verpoorte, R. Sample preparation for plant metabolomics. Phytochemical analysis : PCA. 21 (1), 4-13 (2010).
- Folch, J., Lees, M., Stanley, G. H. S. A Simple Method for the Isolation and Purification of Total Lipides from Animal Tissues. J Biol Chem. 226 (1), 497-509 (1957).
- Weckwerth, W., Wenzel, K., Fiehn, O. Process for the integrated extraction, identification and quantification of metabolites, proteins and RNA to reveal their co-regulation in biochemical networks. Proteomics. 4 (1), 78-83 (2004).
- Wienkoop, S., et al. Integration of metabolomic and proteomic phenotypes: analysis of data covariance dissects starch and RFO metabolism from low and high temperature compensation response in Arabidopsis thaliana. Mol Cell Proteomics. 7 (9), 1725-1736 (2008).
- Valledor, L., et al. A universal protocol for the combined isolation of metabolites, DNA, long RNAs, small RNAs, and proteins from plants and microorganisms. Plant J. 79 (1), 173-180 (2014).
- Nakayasu, E. S., et al. MPLEx: a Robust and Universal Protocol for Single-Sample Integrative Proteomic, Metabolomic, and Lipidomic Analyses. mSystems. 1 (3), (2016).
- Alfonsi, K., et al. Green chemistry tools to influence a medicinal chemistry and research chemistry based organisation. Green Chem. 10 (1), 31-36 (2008).
- Salem, M. A., Juppner, J., Bajdzienko, K., Giavalisco, P. Protocol: a fast, comprehensive and reproducible one-step extraction method for the rapid preparation of polar and semi-polar metabolites, lipids, proteins, starch and cell wall polymers from a single sample. Plant Methods. 12, 45 (2016).
- Hemme, D., et al. Systems-wide analysis of acclimation responses to long-term heat stress and recovery in the photosynthetic model organism Chlamydomonas reinhardtii. Plant Cell. 26 (11), 4270-4297 (2014).
- Sharma, D. K., et al. UPLC-MS analysis of Chlamydomonas reinhardtii and Scenedesmus obliquus lipid extracts and their possible metabolic roles. J Appl Phycol. 27 (3), 1149-1159 (2015).
- Delgado, R., Munoz, Y., Pena-Cortes, H., Giavalisco, P., Bacigalupo, J. Diacylglycerol activates the light-dependent channel TRP in the photosensitive microvilli of Drosophila melanogaster photoreceptors. J Neurosci. 34 (19), 6679-6686 (2014).
- Bozek, K., et al. Organization and evolution of brain lipidome revealed by large-scale analysis of human, chimpanzee, macaque, and mouse tissues. Neuron. 85 (4), 695-702 (2015).
- Khrameeva, E. E., et al. Neanderthal ancestry drives evolution of lipid catabolism in contemporary Europeans. Nat Commun. 5, 3584 (2014).
- Bozek, K., et al. Exceptional evolutionary divergence of human muscle and brain metabolomes parallels human cognitive and physical uniqueness. PLoS Biol. 12 (5), 1001871 (2014).
- Krueger, S., Steinhauser, D., Lisec, J., Giavalisco, P. Analysis of subcellular metabolite distributions within Arabidopsis thaliana leaf tissue: a primer for subcellular metabolomics. Methods Mol Biol. 1062, 575-596 (2014).
- Hummel, J., et al. Ultra performance liquid chromatography and high resolution mass spectrometry for the analysis of plant lipids. FrontPlant Sci. 2, 54 (2011).
- Giavalisco, P., et al. Elemental formula annotation of polar and lipophilic metabolites using (13) C, (15) N and (34) S isotope labelling, in combination with high-resolution mass spectrometry. Plant J. 68 (2), 364-376 (2011).
- Caldana, C., et al. Systemic analysis of inducible target of rapamycin mutants reveal a general metabolic switch controlling growth in Arabidopsis thaliana. Plant J. 73 (6), 897-909 (2013).
- Giavalisco, P., et al. Proteome analysis of Arabidopsis thaliana by two-dimensional gel electrophoresis and matrix-assisted laser desorption/ionisation-time of flight mass spectrometry. Proteomics. 5 (7), 1902-1913 (2005).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature protocols. 2 (8), 1896-1906 (2007).
- Spagou, K., et al. Hydrophilic interaction chromatography coupled to MS for metabonomic/metabolomic studies. J Sep Sci. 33 (6-7), 716-727 (2010).
- Hauck, O. K., et al. Uric acid accumulation in an Arabidopsis urate oxidase mutant impairs seedling establishment by blocking peroxisome maintenance. Plant Cell. 26 (7), 3090-3100 (2014).
- Avin-Wittenberg, T., et al. Global analysis of the role of autophagy in cellular metabolism and energy homeostasis in Arabidopsis seedlings under carbon starvation. Plant Cell. 27 (2), 306-322 (2015).
- Watanabe, M., et al. Comprehensive dissection of spatiotemporal metabolic shifts in primary, secondary, and lipid metabolism during developmental senescence in Arabidopsis. Plant physiol. 162 (3), 1290-1310 (2013).
- Lambers, H., et al. Proteaceae from severely phosphorus-impoverished soils extensively replace phospholipids with galactolipids and sulfolipids during leaf development to achieve a high photosynthetic phosphorus-use-efficiency. New Phytol. 196 (4), 1098-1108 (2012).
- Degenkolbe, T., et al. Differential remodeling of the lipidome during cold acclimation in natural accessions of Arabidopsis thaliana. Plant J. 70, 972-982 (2012).
- Bromke, M. A., et al. Liquid chromatography high-resolution mass spectrometry for fatty acid profiling. Plant J. 81 (3), 529-536 (2015).
- Li, N., et al. FAX1, a novel membrane protein mediating plastid fatty acid export. PLoS Biol. 13 (2), 1002053 (2015).
- Kueger, S., Steinhauser, D., Willmitzer, L., Giavalisco, P. High-resolution plant metabolomics: from mass spectral features to metabolites and from whole-cell analysis to subcellular metabolite distributions. Plant J. 70 (1), 39-50 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados