Method Article
La proyección de imagen seguida de la célula de seguimiento para la biología de la célula de Monitor y la progresión de linaje de poblaciones neuronales múltiples en vivo
* Estos autores han contribuido por igual
En este artículo
Resumen
Se describe un protocolo robusto para monitorear las poblaciones neuronales de Time-lapse vídeo-microscopía seguido basado en software de post-processing. Este método representa una poderosa herramienta para identificar acontecimientos biológicos en una población seleccionada durante los experimentos de imagen vivo.
Resumen
Comprensión de los mecanismos que controlan eventos críticos biológicos de las poblaciones neuronales de la célula, tales como proliferación, diferenciación o las decisiones de destino de la célula, será crucial para diseñar estrategias terapéuticas para muchas enfermedades que afectan al sistema nervioso. Los métodos actuales para rastrear poblaciones celulares dependen de sus resultados finales en imágenes fijas y generalmente no proporcionan suficiente resolución temporal para identificar características de comportamiento en las células. Por otra parte, las variaciones en la muerte celular, heterogeneidad conductual dentro de una población celular, dilución, difusión o la baja eficiencia de los marcadores utilizados para analizar las células son todas desventajas importantes que conducirán a una lectura incompleta o incorrecta de los resultados. Por el contrario, realizar la proyección de imagen viva y célula bajo condiciones apropiadas de seguimiento representa una poderosa herramienta para seguimiento de cada uno de estos eventos. Aquí, un time-lapse protocolo de video-microscopía, seguido de postproceso, se describe para el seguimiento de poblaciones neuronales con la resolución de la célula, utilizando software específico. Los métodos descritos permiten a los investigadores abordar cuestiones esenciales con respecto a la progresión de la célula biología y linaje de distintas poblaciones neuronales.
Introducción
Para poder desarrollar estrategias terapéuticas nuevas y más eficaces para regenerar las poblaciones neuronales, primero debemos entender los mecanismos básicos que mantienen las células con un potencial regenerativo de nervios. Lograr este objetivo requiere un amplio conocimiento de los factores que regulan el equilibrio entre la quietud, proliferación/diferenciación, el modo y momento de división, duración del ciclo celular, capacidades migratorias, viabilidad, etcetera. Aunque es un enfoque técnico que se ha empleado por muchos años1, proyección de imagen en vivo y observación directa siguen siendo la mejor opción para controlar los eventos mencionados. A diferencia de muchos otros enfoques centrados en lecturas de punto final, viva imagen y célula de seguimiento proporciona información a lo largo de la longitud de un experimento de2,3,4,5, 6. por lo tanto, la adición de resolución temporal permite la muerte celular, celular heterogéneo comportamiento o las decisiones del destino de la célula, así como muchos otros eventos críticos para identificar que de lo contrario podrían pasar inadvertido. Idealmente, estas características de las células deben ser vigiladas mejor a la única célula nivel en vivo, donde ambos intrínseca (célula Autónoma) y extrínseca señales (lugar de la célula) se toman en cuenta.
Sin embargo, aunque en la en vitro situación eventos ocurren en un ambiente que no reproducen el medio natural, las condiciones de cultivo baja densidad normalmente utilizadas en estos protocolos son más convenientes revelar características intrínsecas de la células. Por otra parte, un control más simple del medio circundante, modificando simplemente el medio de crecimiento, puede constituir una herramienta valiosa para investigar el papel individual de cada factor extrínseco que define el nicho de nervios, así como los factores ambientales que pueden ser inducida en escenarios patológicos7,8,9,10,11,12,13. Por lo tanto, cuando se configura correctamente, como en el protocolo propuesto aquí, en proyección de imagen proporciona una solución factible en vitro para frente a la mayoría de las preguntas enumeradas anteriormente.
En Resumen, este protocolo describe el hardware, software, condiciones de cultivo y los principales pasos necesarios para realizar con éxito un experimento imagen vivo seguido por célula de seguimiento. Este enfoque ofrece información valiosa que ayuda a revelar aspectos fundamentales de la biología y de la progresión de linaje, de varias poblaciones neuronales.
Protocolo
Las secciones siguientes describen los pasos necesarios para realizar la proyección de imagen vivo seguida de seguimiento de la célula de varias poblaciones neuronales (figura 1). Todos los procedimientos que implican animales descritos en el presente Protocolo deben ser llevado a cabo con arreglo a las directrices del Consejo Internacional para la ciencia de animales de laboratorio (ICLAS).
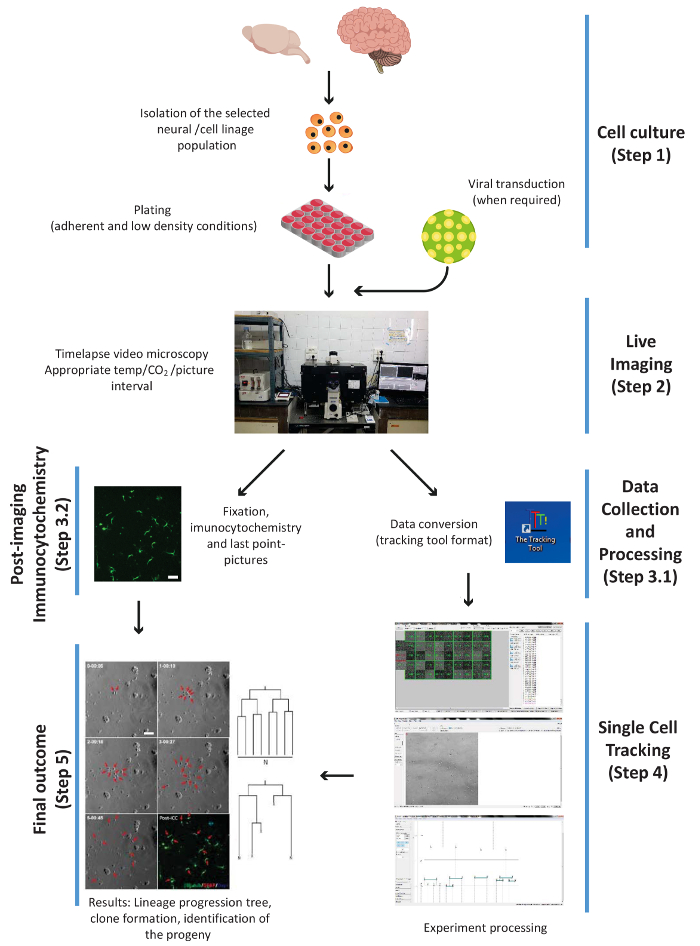
Figura 1. Esquema que ilustra los principales pasos experimentales del procedimiento, es decir: de la célula cultura, proyección de imagen de vivo, PICC y recopilación de datos, célula de seguimiento y el resultado final. Los pasos están numerados según el flujo de trabajo del protocolo. Haga clic aquí para ver una versión más grande de esta figura.
1. célula cultura: Aislamiento y la galjanoplastia de la población Neural o linaje de la célula
Nota: Junto con este protocolo, se dan ejemplos de su aplicación a poblaciones celulares distintas para validar su utilidad para analizar la biología de las células neuronales. Estos incluyen: células madre neuronales adultas (aNSCs) deriva el ratón zona subependimaria (SEZ) (para un aislamiento detallado protocolo véase14); Astrocitos corticales postnatales para estudiar la reprogramación neuronal (para un aislamiento detallado protocolo véase15); Astrocitos cerebelosos postnatales (para una detallada aislamiento método véase16); y línea celular de Neuroblastoma de ratón Neuro-2a (N2a).
- Las células directamente en placas de 24 pocillos recubiertas poly-D-lisina de la semilla. Use 1 mL de medio de cultivo por pozo. Incubar las placas a 37 ° C y 8% CO2 para aNSCs, o en 37 ° C y 5% CO2 para la línea celular astrocitos por 2 h antes de la proyección de imagen de vivo. Evitar el uso de cubreobjetos para evitar el movimiento no deseado como se desplaza la platina del microscopio motorizado que hace inviable la célula de seguimiento.
Nota: Las densidades celulares y medios de cultivo empleados en los experimentos son: 30-40.000 células/pozo para aNSCs en Dulbecco de modificado medio (mezcla de nutrientes de DMEM:F12) del águila; 20.000 células/pozo de las células N2a en medio DMEM alta glucosa, 80.000 células/pocillo de astrocitos cerebelosos en medio DMEM alta glucosa; y 55-65.000 células/pozo de astrocitos posnatales en medio de la mezcla de nutrientes DMEM:F12. - Estandarizar el protocolo de la cultura mediante el ajuste de la densidad celular de la cultura para el menor número de células viables. Sin embargo, la densidad de células debe ser suficientemente alta para mantener la viabilidad de la cultura.
Nota: Si la densidad celular es demasiado alta, el resto de suciedad o mala disociación (matas) puede obstaculizar el seguimiento de las células.
2. proyección de imagen de Time-lapse vídeo-microscopía en vivo
- Activar los sistemas de incubación, microscopio, cámara y hardware. La temperatura y presión a 37 ° C y 8% CO2 para aNSCs, o a 37 ° C y 5% CO2 para la línea de los astrocitos de la célula de aire. Permita que la temperatura y niveles de CO2 para estabilizar para 1-2 h.
Nota: Es necesario un equipo específico para realizar análisis de vídeo Time-lapse, incluyendo: fase de campo brillante contraste/la fluorescencia Microscopios con componentes motorizados; dispositivos de incubación que controlan la temperatura, el CO2 y la humedad; y finalmente, fiable y lo suficientemente poderoso hardware y software capaces de adquirir y manejar el volumen de imágenes obtuvieron durante experimentos de proyección de imagen Vives (por favor verifique la Tabla de materiales). - Una vez que las células se unen firmemente a la placa (2 h después de la galjanoplastia), utiliza un rotulador permanente para hacer una pequeña marca en el fondo de un pozo que no se utilizará para el seguimiento, es decir, un pozo que no contiene células.
Nota: Esta marca se utilizará como una referencia a las coordenadas xyz de cero, y puede ser utilizado en cualquier momento durante o después del experimento o entre los cambios del medio, para volver a la posición cero. - Coloque la placa en cámara de incubación del microscopio y sujete la placa a la etapa para evitar cualquier movimiento indeseado durante el desplazamiento de fase motorizado del microscopio.
- Permitir que la temperatura del medio de cultivo celular se equilibren en la cámara durante aproximadamente 20 minutos. Este paso evitará una pérdida de enfoque durante la grabación debido a la dilatación de los componentes.
- Inicie el software de imágenes en vivo y seleccionar el módulo Time-lapse para establecer el experimento.
- Establecer la duración total de la experiencia y los ciclos de adquisición de imagen en el "menú de la ficha de calendario". Debido a la fototoxicidad inherente de la transmisión o la luz de la fluorescencia utilizado, definir un intervalo adecuado de equilibrio entre la resolución temporal del análisis y la posible muerte de la célula.
Nota: por ejemplo, un total de 120 h fue seleccionado para las culturas aNSC, adquiriendo brightfield fotos cada 5 minutos considera que la adquisición de 120 h de una sola película en esta configuración requiere 120-150 gigabytes de espacio de almacenamiento libre en el dispositivo de la computadora. - Seleccionar las posiciones de la imagen definidas por el x y y coordenadas y la distancia focal (la coordenada z) en el "menú de pestaña puntos xyz". Incluir el punto de referencia (coordenadas xyz cero) como la posición inicial con el fin de recuperar las coordenadas en cualquier momento.
- Seleccione el tipo de adquisición en el "menú de pestaña longitud de onda en selección", trasmitida solo o en combinación con epifluorescencia excitación cuando sea necesario. Seleccione el tiempo de exposición. Tener en cuenta que la exposición excesiva a transmitido, y sobre todo fluorescente, puede comprometer la viabilidad celular (como se indicó anteriormente).
- Para aNSCs, cerebelosos astrocitos y células N2a, seleccione brightfield (tiempo de exposición de 10-50 ms).
- Para astrocitos corticales transduced seleccione brightfield (tiempo de exposición de 10-50 ms) en combinación con fluorescencia roja/verde, según el reportero utilizado para el experimento (longitud de onda de excitación de rojo: 550 nm y 400 ms de tiempo de exposición; excitación verde longitud de onda: 460-500 nm y tiempo de exposición de 100 ms).
- Definir el nombre del experimento y la carpeta donde se almacenarán las imágenes. Guarde la lista de posiciones para volver a cargar el experimento en cualquier momento y una vez que se han establecido todas las condiciones, ejecute el experimento haciendo clic en el botón "ejecutar ahora".
- Detener el experimento y volver a ajustar las condiciones de enfoque haga clic en el "botón z sobreescribe" una vez al día hasta que se termine el experimento. Si se necesitan cambios en el medio durante la proyección de imagen vivo, detener el experimento y recuperar la placa de la cámara de lapso de tiempo.
Nota: Los cambios en el pH del medio debido a la muerte celular o proliferación excesiva, así como las variaciones de temperatura, pueden afectar el enfoque correcto del microscopio de las células. Para cultivos sensibles (como aNSCs) recomendamos el uso de medio suplementado con ácido-(2-hydroxyethyl)-1--1-ácido 4 (HEPES) (concentración final: 1 mM).
3. la proyección de imagen inmunocitoquímica (PICC), recopilación de datos y procesamiento
- Una vez terminado el experimento, hacer una pausa en el software y recuperar la placa de fijación y PICC, como se describe en los siguientes pasos.
- Realizar la fijación celular: lavar las células una vez con solución fisiológica 1 mL tamponada fosfato (PBS) y agregar 500 μl de paraformaldehido (PFA) (4% en PBS), incubando 10 minutos a temperatura ambiente (RT).
PRECAUCIÓN: Paraformaldehido es un fijador fuerte y debe manipularse con cuidado para evitar el contacto con la piel o los ojos. Debe ser manipulado solamente dentro de una campana de humos. - Lavar las células 3 veces con 1 mL de PBS y agregar 500 μl de la solución de bloqueo (PBS con un 2% (peso/vol) de albúmina de suero bovino (BSA) y 0.2% (vol/vol) de un surfactante no iónico). Incubar 1 h a TA.
- Retirar la solución de bloqueo y añadir 250-400 μL de la solución de anticuerpos primarios. Incubar 2 h a TA. La solución de anticuerpos primarios contiene anticuerpos primarios diluidos en PBS con un 2% (peso/vol) de BSA y 0.2% (vol/vol) de un surfactante no iónico. Anticuerpos utilizados en los experimentos descritos aquí: GFAP (1: 500), βIII-tubulina (1:1, 000) y α-tubulina (1:1, 000). Como esto se lleva a cabo directamente en el pozo, mayores volúmenes de las soluciones se requieren (250-400 μL) para cubrir todas las celdas.
- Lavar tres veces con 1 mL de PBS y añadir 250-400 μL de la solución de anticuerpos secundarios (diluida como se describe en el paso 3.4). Anticuerpos secundarios utilizados en los experimentos descritos aquí: anti-Mouse fluoresceína (FITC) (1: 800), anti-conejo Cy3 (1: 500). Incubar 1 h a temperatura ambiente en la oscuridad.
- Lavar tres veces en 1 mL de PBS. Mantener las células en 1 mL de PBS para los pasos subsiguientes del protocolo.
- Coloque la placa en la platina del microscopio y fije firmemente a la etapa para evitar movimientos no deseados durante el desplazamiento de la platina del microscopio motorizado.
- Recuperar el xyz con la marca hecha en el paso 2.2 de posición cero y vuelva a la posición de este punto de referencia pulsando el botón "Compensar todos X, Y, Z". Ajustar la distancia focal para cada posición.
- Adquirir una ronda final de las imágenes, configurar las condiciones necesarias para la emisión de fluorescencia en "menú de ficha para selección de longitud de onda" con el fin de detectar los antígenos que se previamente focalizados en el PICC.
- Brevemente, además de brightfield, activar FITC (excitación: 495 nm) y Cy3 (excitación: 550 nm) opciones de adquisición del software. Utilice 10-50 ms para la exposición de brightfield de 400 ms para detectar los fluoróforos y presione el botón de "bucle de tiempo 1", para adquirir una ronda final en cuadros.
Nota: La intensidad de la fluorescencia puede diferir dependiendo de los resultados de la PICC. Ajustar el tiempo de exposición para obtener la calidad de imagen óptima.
- Brevemente, además de brightfield, activar FITC (excitación: 495 nm) y Cy3 (excitación: 550 nm) opciones de adquisición del software. Utilice 10-50 ms para la exposición de brightfield de 400 ms para detectar los fluoróforos y presione el botón de "bucle de tiempo 1", para adquirir una ronda final en cuadros.
- Seleccione la opción de archivo y exportación de software y exportar las imágenes de Tagged Image File Format (Tiff) o formato de Joint Photographic Experts Group (Jpeg) a una carpeta de destino predefinido.
- Convertir las imágenes exportadas en el formato requerido por el software de seguimiento: la herramienta de seguimiento de17 (tTt). Para ello, definir la entrada de carpeta en la "herramienta de conversión de tTt" ventana, así como los marcadores utilizados para las posiciones (xy), canales (c) y puntos de tiempo (t) de salida y presione el botón "convertir imágenes".
Nota: Las imágenes deben cambiar el nombre de acuerdo con los ajustes específicos y se deben almacenar en carpetas individuales para cada posición utilizado en el experimento. Instrucciones para la instalación, requisitos, cambio de nombre de imágenes de posiciones y el uso de la herramienta de seguimiento están disponibles para descargar en: https://www.bsse.ethz.ch/csd/software/tTt-and-qtfy.html.
4. célula seguimiento
- Después de retitular los datos, ejecutar el software de tTt. Seleccione una carpeta de trabajo usuario nombre y tTt.
Nota: Seguimiento del carpeta de trabajo herramienta tendrá todos los datos analizados y los resultados exportados. La carpeta de trabajo debe tener el nombre de tTtexport, que contiene las subcarpetas llamadas "AVIexport", "Configuraciones", "TreeExport" y "tTtfiles". - Selecciona el experimento que se carguen en la "ventana de carpeta seleccione experimento", que indica la ruta de la carpeta donde se almacena el experimento, y luego haga clic en el botón "Experimento de la carga".
- Ejecutar el convertidor de archivo de registro para transformar las imágenes cargadas en un formato que puede ser leído por el software de seguimiento (para experimentos cargados por primera vez, esto se pedirá automáticamente por el software).
- Seleccione una posición para seguimiento haciendo clic en su símbolo (después de conversión, un resumen de las posiciones registradas durante los experimentos se mostrará en la "ventana de diseño de posición"). Cada posición será representado por un símbolo que consiste en un cuadro de la posición y el número correspondiente (ver figura 1).
- Una vez se ha seleccionado la posición y se muestra una lista de las imágenes disponibles a la derecha de la "ventana de diseño de posición", selecciónelas y haga clic en el botón "Cargar imágenes".
- Una vez que la carga es completa y la "ventana del Editor de celda" aparece, seleccione la longitudes de onda y el intervalo de imagen para ser rastreados en la "ventana del Editor de la célula". Longitud de onda 0 corresponde a brightfield, 1 corresponde al FITC, Cy3, 2 y 3 para DAPI. En los experimentos descritos aquí, se utilizó el intervalo de 1, es decir, todas las imágenes cargadas. Para aclarar, intervalo 2 significa carga de cada segundo de imagen.
- Una vez que las imágenes hayan sido cargadas, volver a la "ventana de diseño de posición" y haga doble clic en el icono que representa la posición previamente cargada. La "ventana de la película" aparecerá que permite el seguimiento de la célula a realizar.
- Siguiendo las instrucciones de la herramienta de seguimiento, se procederá a seguimiento. Seleccionar el canal 0 (correspondiente a brightfield) y ajustar el brillo y contraste ("ajuste botón gamma"). Iniciar el rastreo pulsando la tecla F2.
Nota: Durante el seguimiento, la célula de seguimiento seguirán colocando el puntero del mouse en él y pulsando la tecla "0". La división celular, apoptosis celular y celular perdido botones están disponibles para controlar estos eventos específicos de la célula.
5. resultado
- Una vez concluida la célula de seguimiento y la progenie identificada, salvar el experimento (pestaña Editor celular ventana/archivo / guardar árbol actual) y proceder a exportar los resultados.
- Exportación de los árboles de linaje y datos de la celda en el "menú de exportación", situado en la "ventana del Editor de celda". Además, exportar las imágenes de la célula y películas a través del "menú de exportación" accesible a través de la "ventana de la película". Las imágenes, árboles de linaje, datos y películas se exportarán a la carpeta de trabajo de tTt.
Resultados
El método descrito permite preguntas críticas sobre la biología de la célula de varias poblaciones neuronales para resolverse. Por ejemplo, ha sido posible controlar la progresión de la estirpe neurogénica y oligodendrogliogenic de aNSCs7,8,14,18. Mediante el seguimiento solo aNSCs y su progenie (figura 2A, B), fue posible demostrar aNSCs aislados en vitro mantener su naturaleza neurogénica, en su mayoría generan neuroblastos, y que siguen una secuencia de propuestas en vivo19 pero no demostrado en el nivel unicelular. Además, este sistema de cultivo permitió asimétrica las divisiones de célula visualizar por primera vez en el linaje aNSC de SEZ (figura 2B), proporcionando un modelo único para estudiar NSC auto-renovación8,14. Asimismo y sin importar el linaje analizados, fue posible obtener datos valiosos en cuanto a crecimiento de la célula, las rondas de división, viabilidad celular o duración del ciclo celular.
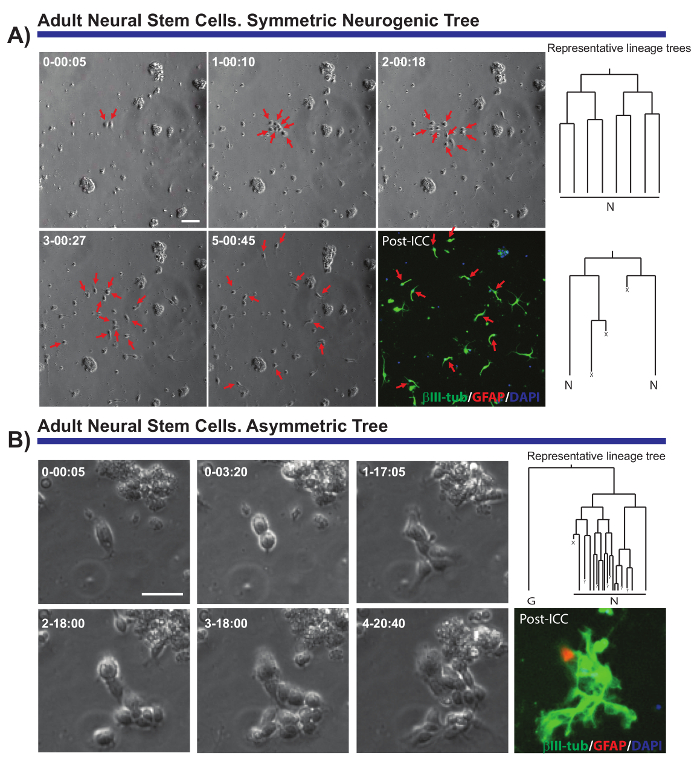
Figura 2. Ejemplo de aNSCs aislado del SEZ y analizada por la proyección de imagen viva y célula de seguimiento. Imágenes de contraste de fase representan la progresión de la copia en diferentes puntos temporales (día-h: min). La última imagen corresponde a la proyección de imagen de la inmunocitoquímica (PICC) para la proteína ácida fibrilosa Glial (GFAP, rojo), βIII-tubulina (verde) y 4', 6-diamidino-2-phenylindole (DAPI, azul). (A) análisis de árboles neurogénicas simétricos a través de diferentes rondas de amplificación divisiones generan neuroblastos poste-mitotic. Flechas rojas indican las células incluidas en los árboles simétricos. A la derecha, se muestran los árboles linaje corresponde a los clones y generado por el software de tTt. (B) ejemplo de un progenitor genera un árbol neurogénico asimétrico, con una rama en divisiones de amplificación para producir neuroblastos mientras la otra da lugar a quieto GFAP positivas las células a través de un evento potencial de autorenovación. A la derecha, se muestra el árbol del linaje generado por el software de tTt. En todos los árboles del linaje: "N" representa poste-mitotic neuroblastos; "G", quietas células GFAP-positivas; "X", la muerte celular; y "?" un celular perdido. Representa la barra de escala 50 μm. haga clic aquí para ver una versión más grande de esta figura.
La proyección de imagen en vivo y unicelular seguimiento análisis también proporciona una lectura exacta de la capacidad migratoria de una población neuronal. Dicha información fue obtenida de astrocitos cerebelosos postnatales presentados a un arañazo herida ensayo20, generando información con respecto a la distancia promedio recorrida por los astrocitos en el cierre de la herida (figura 3). Por otra parte, era posible ver que algunos de los astrocitos dividieron durante el proceso de curación, mientras que otros permanecen inalteradas durante todo el experimento. Sorprendentemente, aquellos que encuentren parecen exhibir comportamiento migratorio más prolífico que sus homólogos no se dividen (viajando dos veces como mucho en promedio). Este fenómeno sugiere una heterogeneidad muy interesante en la capacidad de los astrocitos forman una cicatriz sobre la lesión, que habría sido diluido hacia fuera en la lectura de un experimento de análisis de punto final clásico.
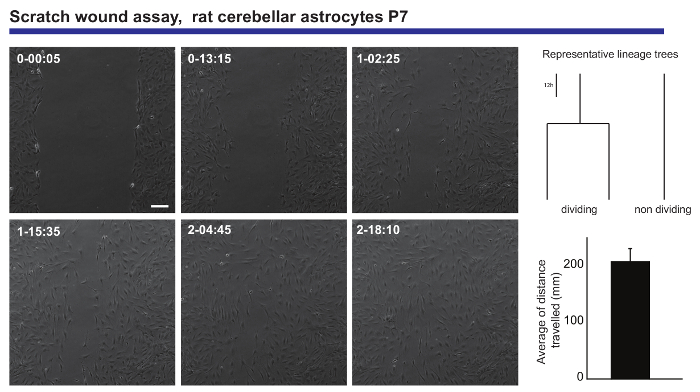
Figura 3. Análisis del comportamiento migratorio de astrocitos cerebelosos postnatales en un arañazo herida ensayo. Imágenes de contraste de fase representan la herida en diferentes puntos temporales (día-h: min). Árboles de linaje, generados por el software de tTt, ilustran el comportamiento del representante, en términos de división celular, de los astrocitos al cerrar la herida. Histograma muestra la distancia promedio recorrida por los astrocitos por la célula de seguimiento (media ± SEM). Representa la barra de escala 50 μm. haga clic aquí para ver una versión más grande de esta figura.
Otra característica interesante de los experimentos de microscopía de vídeo Time-lapse es la capacidad para comparar la proliferación y diferenciación en una población celular. Probamos las células N2a plateadas bajo condiciones que promueven la proliferación (en presencia de 10% suero bovino fetal (FBS)) o diferenciación (en presencia de 0,5% FBS + 10 μm del ácido araquidónico). Fue posible seguir la progresión del linaje de estas células bajo condiciones proliferativas (Figura 4A), mientras que la diferenciación de las células no proliferan y forman neuritas (Figura 4B). Notable, célula de seguimiento permitió colonias con diferentes proliferación capacidades para distinguirse y neurita alargamiento (y retracción) a ser evaluados, proporcionando datos precisos y cuantitativos que pueden ser exportados posteriormente.
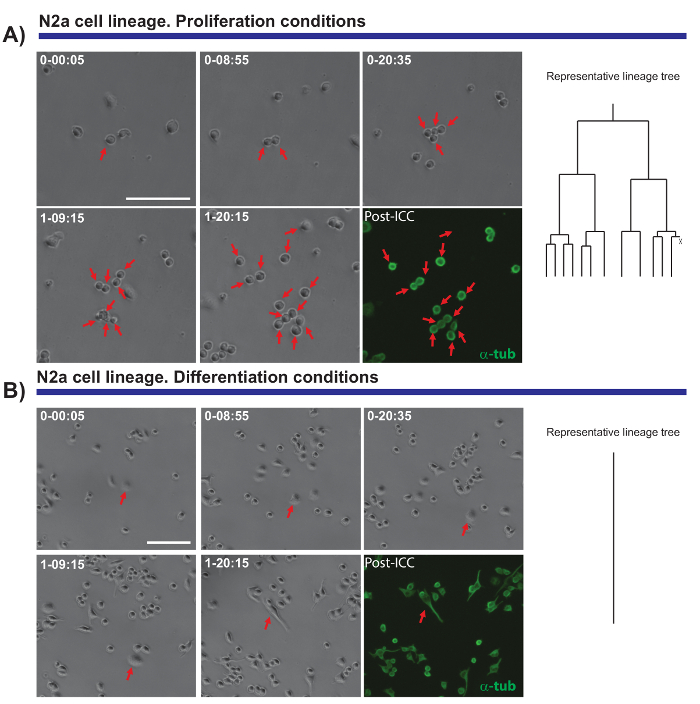
Figura 4. El monitoreo de N2a biología de la célula en proliferación (A) o condiciones de diferenciación (B). Imágenes de contraste de fase que representan la progresión de la copia en diferentes puntos temporales (día-h: min). La última imagen corresponde a la inmunocitoquímica la proyección de imagen (PICC) de α-tubulina (verde). (A) célula de seguimiento permite que las rondas de división a ser monitoreados, así como la heterogeneidad en la respuesta proliferativa de células diferentes. A la derecha, el árbol del linaje generado por el software de tTt ilustra el comportamiento proliferativo de las células N2a. (B) las células en condiciones de diferenciación salir del ciclo celular y generan neuritas, un proceso que se puede medir con eficacia por el análisis de la proyección de imagen. Solo rastreo celular, representada por el árbol del linaje a la derecha, ilustra cómo las células N2a salir del ciclo celular y detener la división celular en condiciones de diferenciación. Barra de escala representa 50 μm.Haga clic aquí para ver una versión más grande de esta figura.
Finalmente, la proyección de imagen en vivo y célula de seguimiento es muy útil para monitorizar los cambios morfológicos y moleculares cuando las células se someten a la reprogramación. En proyección de imagen de astrocitos postnatales transduced con homólogo Achaete-scute 1 (Ascl1) proporciona datos valiosos con respecto a los cambios morfológicos que ocurren durante la reprogramación o el bloqueo de la división celular cuando los astrocitos están siendo reprogramada (véase Figura 5). Por otra parte, cuando Ascl1 transducción se combina con la transducción de la construcción de codificación para la proteína de fluorescencia verde (GFP) bajo el control del promotor doble Cortin (DCX), es posible definir el momento exacto cuando neuronal marcadores específicos comienzan a expresarse en las células reprogramadas (figura 5). Time-lapse vídeo-microscopía también permite que el número de células que completan con éxito reprogramación para ser cuantificado y Comparado con las células que mueren durante este proceso. Supervisión de acontecimientos condujeron a la identificación de las críticas "checkpoints" en las células que fueron reprogramadas con éxito9.
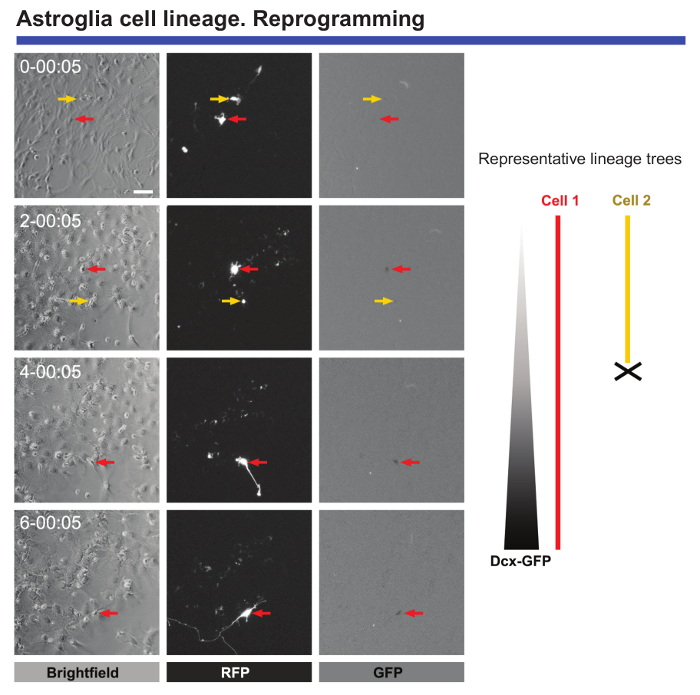
Figura 5. Análisis de los astrocitos corticales postnatales sujetado a reprogramación neuronal. Reprogramación se indujo mediante transducción con vectores de proteína (RFP) de fluorescencia rojo Ascl1 pro-neurogénica. Conversión neuronal fue monitoreado por la transducción de señales con un vector de codificación de GFP bajo el control de un promotor DCX. Imágenes de contraste de fase muestran la progresión de la reprogramación en diferentes puntos temporales (día-h: min). Imágenes de fluorescencia de la RFP y GFP expresión, respectivamente. La proyección de imagen en vivo y célula de seguimiento permitió acontecimientos cruciales que deben seguirse, tales como cambios morfológicos, la ausencia de división celular durante la reprogramación, muerte celular, y se puede definir la hora exacta en las células reprogramadas comienzan expresar marcadores neuronales . Barra de escala representa 80 μm. haga clic aquí para ver una versión más grande de esta figura.
Discusión
Uno de los valores más importantes de la proyección de imagen viva es la posibilidad de realizar seguimiento de linaje exacto, dilucidar aspectos críticos de la evolución del linaje en una población neuronal. Seguimiento de linaje se define como la identificación y seguimiento de toda la progenie de un solo progenitor, del fundador del clon a clon posterior formado21. Notable, métodos alternativos para linaje rastreo (p. ej., transducción viral o reportero multicolor construcciones21) tienen un inconveniente fundamental, por el que el resultado final se basa en imágenes fijas y no necesariamente constituyen toda la secuencia. Esto significa que la muerte celular, heterogeneidad en el comportamiento de la población celular, dilución, difusión o pobre eficiencia de los marcadores, junto con otros importantes desventajas, conducen a una lectura incompleta o incorrecta de los resultados2. Además, la proyección de imagen vivo permite al investigador analizar características importantes de la biología de poblaciones neuronales, como el modo y el momento de la división celular, crecimiento celular, migración, proliferación y diferenciación, la duración del ciclo celular, la neurita formación, complejidad y longitud, la célula selección de destino (diferenciación) o conversión (reprogramación).
Además, en proyección de imagen puede ser fácilmente complementado con otros análisis que se pretende obtener datos de las células tales como, por ejemplo, la secuencia de RNA. Sin embargo lograr el beneficio combinado de proyección de imagen de vivo y otras técnicas requiere que esas células previamente monitoreadas en las películas son identificadas más adelante volver a y recogidas individualmente para el análisis secundario. Esto puede lograrse mediante el uso de microscopios que incluyen coordenadas posicionales, aplicando reporteros fluorescentes de células específicas o análisis de la distribución de grupos de células como referencias. De hecho, la combinación del transcriptoma perfil y comportamiento de las células individuales pueden representar una ruta de gran alcance para aclarar nuevas señales moleculares implicadas en la biología de las células.
Uno de los principales problemas que pueden poner en peligro un experimento vivo de imágenes es una densidad de cultura celular inadecuada. Como se indicó anteriormente, en alta densidad puede afectar el exceso de suciedad o mala disociación (formación de grumos) la calidad y la resolución espacial de las imágenes, haciendo inviable célula de seguimiento. Por lo tanto, las condiciones de las poblaciones celulares distintas bajo estudio deben ajustarse en el menor número de células posible sin comprometer la viabilidad del cultivo celular.
La frecuencia de adquisición de la imagen también es crucial y debe ser ajustada con cuidado, especialmente cuando se utiliza iluminación de fluorescencia. Exposición excesiva a transmite y sobre todo luz de la fluorescencia puede comprometer la viabilidad celular. Por otra parte, una excesiva demora entre la captura de las imágenes puede interferir con la resolución temporal del análisis.
Otro paso crítico durante el experimento directo de proyección de imagen es el ajuste periódico de enfoque. Fallo en el correcto ajuste/re-establecer de la distancia focal puede obstaculizar el seguimiento de la célula. Por otra parte, es necesario comprobar cuidadosamente que la cámara de incubación conserva la temperatura adecuada, humedad y niveles de CO2 , que modifica las variaciones no deseadas que pueden inducir la muerte celular.
Por último, una vez que el PICC ha llevado a cabo, es importante recuperar correctamente la posición de xyz cero antes de la última ronda de adquisición de imágenes. Incorrecto volver a configurar del xyz posición cero resultará difícil hacer coincidir las imágenes de contraste de fase y de la inmunofluorescencia, que impiden la identificación de la progenie de la célula.
Aunque este enfoque tiene muchas facetas positivas, aún persisten algunas limitaciones a la proyección de imagen viva de las poblaciones neuronales. Por ejemplo, la densidad de célula bajo requerida hacer seguimiento solo acertado de la célula de aNSCs hace que sea imposible emplear ensayos bioquímicos, como la de Western Blot14. Además, monitoreo rápido dividir poblaciones como cerebelosos astrocitos o células N2a es temporal restringida como a menudo es muy difícil de rastrear las células como las culturas cerca de confluencia. Además, muchos métodos de cultivo, así como las restricciones biológicas inherentes asociadas con el aislamiento de las células, a menudo comprometen la viabilidad celular durante largos periodos, limitando la duración de los experimentos de imagen vivo. Finalmente, aislar las células de su ambiente natural tiene efectos positivos y negativos. Células aisladas de su nicho fisiológico pueden no recibir señales importantes que modulan su comportamiento, mientras que al mismo tiempo, representa un medio poderoso para probar el efecto de esas señales individualmente en la progresión del linaje de nervios específicos poblaciones.
Dadas las limitaciones descritas anteriormente, es claro que el escenario metodológico perfecto para llevar a cabo experimentos de seguimiento de celular directo imágenes y solo bajo condiciones fisiológicas normales en vivo. Sin embargo, las técnicas actuales son incapaces de seguir las células durante largos períodos de tiempo en regiones profundas del cerebro2. Por lo tanto, el futuro de la proyección de imagen vivo debe centrarse en la superación de esta limitación, con el objetivo de analizar completamente la biología celular de las células en vivo con lo más pequeñas posible interferencia del entorno fisiológico3.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Beatriz Gascon por su asistencia y obras de arte en la figura 1. También agradecemos a Dr. C. Norris por su ayuda. El trabajo presentado aquí fue apoyado por becas de investigación, "Red de excelencia Consolider-Ingenio Español Ion canal iniciativa" (BFU2015-70067REDC), MEC (BFU2014-53654-P), BRADE-CM (S2013/hielo-2958), UCM-Santander (PR26/16-18B-3) y Fundación Ramon Programa de Areces Grant (PR2018/16-02). Felipe Ortega reconoce el Ramón y Cajal programa del Ministerio de economía y competitividad (MEC: RYC-2013-13290).
Materiales
| Name | Company | Catalog Number | Comments |
| Poly-D-lysine | Sigma | P0899 | Working solution 0.02 mg mL-1 |
| 24 wells plate | Falcon | 352047 | |
| Dulbecco’s modified Eagle’s medium (DMEM): F12 Nutrient Mixture medium (-L Glutamine) | Invitrogen | 21331-020 | |
| DMEM High Glucose medium | Sigma | D6546 | |
| Bovine Serum Albumin | Sigma | A6003 | |
| Triton X-100 | Merck | 11869 | non-ionic surfactant |
| Mouse anti-β III Tubulin | Sigma | T8660 | |
| Rabbit anti-GFAP | DakoCytomation | Z0334 | |
| Mouse anti-α Tubulin | Sigma | T5168 | |
| Anti-Mouse FITC | Jackson Laboratories | 715-095-150 | |
| Anti-Rabbit Cy3 | Jackson Laboratories | 711-165-152 | |
| Brightfield/Phase contrast/fluoresence microscope | Nikon | TE-2000-E | |
| CFI PLAN FLUOR DLLL 10X objetives | Nikon | Ref 280MRH10101 | |
| CFI SUPER PLAN FLUOR ELWD AMD 20X objetives | Nikon | Ref 280MRH48230 | |
| pE-300 LED fluorescence | Cool LED | Ref Number 1981 | |
| 310M-201 Incubation system (temperature) | OKO-Lab | Serial Nº VOF007307 | |
| Pro-ScanII Motorized stage system | Prior | Serial Nº 60018 | |
| High precision microscope camera version 4.2 | ANDOR Zyla | VSC-03650 | |
| Specifc software for live imaging with timelapse module: NIS-Elements AR4.5 | Nikon | NIS-Elements AR4.5 -Hasp ID: 13CE819E | |
| OKO touch Incubation system (CO2) | OKO-lab | Serial number 1716 | |
| Murine Neuro-2a Neuroblastoma Cell line | ATCC | ATCCCCL131 | |
| HEPES buffer solution 1 M | Invitrogen | 15630-056 |
Referencias
- Conklin, E. G. The Mutation Theory From the Standpoint of Cytology. Science. 21 (536), 525-529 (1905).
- Ortega, F., Costa, M. R. Live Imaging of Adult Neural Stem Cells in Rodents. Front Neurosci. 10, 78 (2016).
- Skylaki, S., Hilsenbeck, O., Schroeder, T. Challenges in long-term imaging and quantification of single-cell dynamics. Nat Biotechnol. 34 (11), 1137-1144 (2016).
- Schroeder, T. Tracking hematopoiesis at the single cell level. Ann N Y Acad Sci. 1044, 201-209 (2005).
- Schroeder, T. Imaging stem-cell-driven regeneration in mammals. Nature. 453 (7193), 345-351 (2008).
- Etzrodt, M., Endele, M., Schroeder, T. Quantitative single-cell approaches to stem cell research. Cell Stem Cell. 15 (5), 546-558 (2014).
- Ortega, F., et al. Oligodendrogliogenic and neurogenic adult subependymal zone neural stem cells constitute distinct lineages and exhibit differential responsiveness to Wnt signalling. Nat Cell Biol. 15 (6), 602-613 (2013).
- Costa, M. R., et al. Continuous live imaging of adult neural stem cell division and lineage progression in vitro. Development. 138 (6), 1057-1068 (2011).
- Gascon, S., et al. Identification and Successful Negotiation of a Metabolic Checkpoint in Direct Neuronal Reprogramming. Cell Stem Cell. 18 (3), 396-409 (2016).
- Karow, M., et al. Reprogramming of pericyte-derived cells of the adult human brain into induced neuronal cells. Cell Stem Cell. 11 (4), 471-476 (2012).
- Kleiderman, S., et al. Conversion of Nonproliferating Astrocytes into Neurogenic Neural Stem Cells: Control by FGF2 and Interferon-gamma. Stem Cells. 34 (12), 2861-2874 (2016).
- Bunk, E. C., et al. Prox1 Is Required for Oligodendrocyte Cell Identity in Adult Neural Stem Cells of the Subventricular Zone. Stem Cells. 34 (8), 2115-2129 (2016).
- Aravantinou-Fatorou, K., et al. CEND1 and NEUROGENIN2 Reprogram Mouse Astrocytes and Embryonic Fibroblasts to Induced Neural Precursors and Differentiated Neurons. Stem Cell Reports. 5 (3), 405-418 (2015).
- Ortega, F., et al. Using an adherent cell culture of the mouse subependymal zone to study the behavior of adult neural stem cells on a single-cell level. Nat Protoc. 6 (12), 1847-1859 (2011).
- Heinrich, C., et al. Generation of subtype-specific neurons from postnatal astroglia of the mouse cerebral cortex. Nat Protoc. 6 (2), 214-228 (2011).
- Jimenez, A. I., et al. Potentiation of ATP calcium responses by A2B receptor stimulation and other signals coupled to Gs proteins in type-1 cerebellar astrocytes. Glia. 26 (2), 119-128 (1999).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotechnol. 34 (7), 703-706 (2016).
- Ortega, F., Berninger, B., Costa, M. R. Primary culture and live imaging of adult neural stem cells and their progeny. Methods Mol Biol. 1052, 1-11 (2013).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annu Rev Neurosci. 32, 149-184 (2009).
- Yu, A. C., Lee, Y. L., Eng, L. F. Astrogliosis in culture: I. The model and the effect of antisense oligonucleotides on glial fibrillary acidic protein synthesis. J Neurosci Res. 34 (3), 295-303 (1993).
- Kretzschmar, K., Watt, F. M. Lineage tracing. Cell. 148 (1-2), 33-45 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados