Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluar In Vitro el daño de ADN utilizando el ensayo cometa
En este artículo
Resumen
El ensayo cometa es un método eficiente para detectar daños en el ADN como sola y doble hebra de ADN se rompe. Se describen ensayos cometa alcalino y neutral para medir el daño del ADN en células de cáncer para evaluar el efecto terapéutico de la quimioterapia.
Resumen
Daños en el ADN es un fenómeno común para cada celda durante su vida útil y se definición como una alteración de la estructura química de la DNA genomic. Tratamientos contra el cáncer, tales como radio y quimioterapia, introduce gran cantidad de daño de ADN adicional, llevando a la detención del ciclo celular y apoptosis para limitar la progresión del cáncer. Evaluación cuantitativa del daño de la DNA durante la terapia experimental del cáncer es un paso clave para justificar la eficacia de un agente genotóxico. En este estudio, nos centramos en un análisis de electroforesis unicelular, también conocido como el ensayo del cometa, que puede cuantificar solo y doble cadena ADN rompe en vitro. El ensayo cometa es un método de cuantificación de daños de ADN que es eficiente y fácil de realizar, y tiene demandas de tiempo y dentro de presupuesto bajo y alta reproducibilidad. Aquí, destacamos la utilidad del ensayo cometa para un estudio preclínico por evaluar el efecto genotóxico de la terapia de combinación de olaparib/temozolomida a las células de glioma U251.
Introducción
El análisis del cometa primero fue desarrollado por Ostling y Johanson en 1984 demostrando que la migración del ADN de fragmentos de los núcleos en un estado neutro1. La técnica fue desarrollada más tarde por Singh et al., mostrando que una condición alcalina aumenta sustancialmente la especificidad y la reproducibilidad del ensayo2. Desde entonces, el análisis del cometa neutro se utiliza sobre todo para detectar roturas de la DNA de doble cadena, mientras que el ensayo del cometa alcalino es más sensible para cantidades más pequeñas de daño en el ADN, incluyendo solo y doble cadena de ADN rompe, sitios álcali lábil, DNA-DNA o Cross-linking de la proteína ADN y rupturas monocatenarias de DNA asociadas con la supresión incompleta de reparación sitios3,4. Ambos ensayos permiten visualización de ADN fragmentado y proporcionan una manera sencilla de evaluar cuantitativamente el daño de la DNA. El análisis del cometa es considerado como un método sensible para estudios toxicológicos genéticos in vitro e in vivo y es aplicable a las áreas de investigación, tales como la selección precoz candidato a fármaco, monitoreo ambiental, control biológico humano, y fundamental de investigación en daño de la DNA y reparación5.
El principio del ensayo es que bajo un campo eléctrico, el DNA fragmentado migra fuera del cuerpo de nucleoid (también conocido como la "cabeza de cometa") y forma una mancha de ADN en el gel de agarosa (también conocido como la "cola de cometa"). Con nucleótidos la coloración, el grado de daño del ADN puede cuantificarse mediante el análisis de "cometas" formados por esta electroforesis unicelular. Cálculo del momento de cola más puede ayudar a comparar los daños en el ADN entre los diferentes grupos experimentales. En comparación con los métodos tradicionales de detección de daño de ADN, el análisis del cometa es directa, sensible, barato y relativamente sencillo.
Radioterapia y quimioterapias son estrategias comunes para el tratamiento del cáncer mediante la generación de hebra y ADN de doble cadena se rompe en los cromosomas6. El avance reciente de inhibidores de la reparación de ADN permite un más eficaz efecto genotóxico por quimioterapia de combinación y por lo tanto, reduce los efectos secundarios sistémicos tales como anemia, infección y la médula ósea supresión7, 8. en este estudio, mostramos la investigación de un inhibidor de poli (ADP-ribosa) polimerasa (PARP), olaparib (Ola)9. PARP es una proteína nuclear abundante y es responsable de la reparación de la base de la supresión de ADN mediante la formación de un polímero de poli (ADP-ribosa)10. La temozolomida (TMZ) es un agente alquilante oral disponible y ha sido ampliamente utilizado para el tratamiento de pacientes de glioma. Utilizando el ensayo cometa para cuantificar daños en el ADN, demostramos que combina olaparib con temozolomida profundamente mejora el daño de la DNA en células de glioma, que sugiere terapia combinada olaparib/temozolomida es una estrategia eficaz para tratar el glioma, en comparación con temozolomida sólo11.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. preparar reactivos
- 1 x PBS
- diluir 100 mL 10 x PBS con 900 mL dH 2 O y ajustar el pH a 7.4 con un medidor de pH. Almacenar a temperatura ambiente.
- Solución de lisis (LS)
- preparar 2.5 M NaCl, 100 mM disódico EDTA, 10 mM Tris base y 200 mM NaOH en 900 mL dH 2 O; comúnmente tarda unos 20 minutos para permitir que la mezcla se disuelva completamente. Ajustar el pH a 10 con un medidor de pH. Agregar 1% Sodio Lauril sarcosinato y 1% Tritón X-100 y ajustar el volumen final a 1000 mL. Refrigerar a 4 ° C durante al menos 30 minutos antes de su uso.
- (AES) de la solución de electroforesis alcalina, pH > 13
- preparar 200 mM NaOH y 1 mM EDTA disódico en 800 mL dH 2 O. ajustar el pH y que sea pH > 13. Ajustar el volumen final a 1000 mL. Hacer fresco antes de usarlo y enfriar a 4 ° C durante al menos 30 minutos antes de su uso.
- Solución de electroforesis neutro (NES)
- preparar 1.000 mL de tampón de electroforesis neutral mezclando 100 mM Tris base y 300 mM acetato de sodio en 1.000 mL dH 2 O. Ajuste el pH a 9.0 con ácido acético glacial. Refrigerar a 4 ° C durante al menos 30 minutos antes de su uso.
- Solución de precipitación de DNA (DPS)
- preparación de stock de 10 mL de 7,5 M amonio acetato. Para 50 mL de solución de precipitación de DNA, mezcla de acetato de amonio de 7.5 M 6,7 mL con etanol al de 95% 43,3 mL. Almacenar a temperatura ambiente.
- Solución de tinción
- Agregar 1 μl 10.000 x mancha fluorescente verde de ácido nucleico (p. ej., SYBR Green) en 30 mL de Tris-EDTA buffer (10 mM de Tris-HCl, 1 mM EDTA disódico, pH 7.4) y tienda a 4 ° C. proteger de la luz.
- baja de 1% agarosa de fusión
- derretir 1% bajo derretir la agarosa de punto (1 g en 100 mL dH 2 O) en el microondas. Remolino de la agarosa cada 15-20 s para asegurarse que la agarosa está completamente fundido. Colocar la agarosa en baño de agua de 37 ° C por al menos 20 min antes del uso.
- Pre-warm pipetas
- cortar los extremos estrechos de P200 pipetas de 3 mm y caliente a 37 ° C antes de pipetear agarosa.
2. Preparar diapositivas Comet
- diapositiva capa
- derretimiento 1% agarosa (1 g en 100 mL dH 2 O) en el microondas durante 2-3 minutos o hasta que la agarosa está completamente fundido. Sumergir el portaobjetos de cristal en la agarosa y limpie un lado de la diapositiva usando un paño libre de pelusas.
- Colocar el portaobjetos sobre una superficie plana para secar al aire o calentar a 50 ° C para un secado más rápido, una película transparente de la agarosa debe ser formada después del secado. Colocar los portaobjetos recubiertos en 37 ° C antes de su uso.
- Preparación de suspensiones celulares solo
- cultura y tratar las células de glioma
- cultura las células U251 MG en medio DMEM-jamón F-12 suplementado con 10% FBS, 100 U/mL de penicilina y estreptomicina 10 de μg/mL en 37 & # 176; C con 5% CO 2.
- Digerir las células utilizando tripsina de 1 mL por 3 min y neutralizar la tripsina con medio DMEM-jamón F-12 FBS. Recoger en tubo de 15 mL, centrifugado a 300 x g durante 4 minutos, aspirar el medio y suspender las células en 2 x 10 5 células/mL en PBS 1 x.
Nota: La muestra de células se debe prepararse inmediatamente antes de iniciar el ensayo y todas las muestras deben ser manipuladas en un ambiente oscuro o atenuado para evitar daños en el ADN de luz. - La suspensión de células se combinan con agarosa al 1% fundido bajo punto de fusión (a 37 ° C) en una proporción de 1:10 (v/v), mezcle suavemente transfiriendo hacia arriba y hacia abajo y pipetear inmediatamente 30 μL sobre un portaobjetos. Use el lado de la punta de la pipeta a la mezcla de agarosa/célula para asegurar la formación de una fina capa.
- Colocar la diapositiva plana a 4 ° C en la oscuridad para 10 minutos aumentando el tiempo de gelificación a 30 min mejora la adherencia de las muestras en ambientes de alta humedad.
- Sumerja el portaobjetos en 4 ° C LS en la oscuridad durante 1 hora hasta toda la noche.
- cultura y tratar las células de glioma
3. Sola célula de electroforesis
- proceder a alcalino (paso 3.2) o neutro (paso 3.3) cometa ensayo
- para el ensayo cometa alcalino
- suavemente quitar diapositivas del LS, drenar el exceso de tampón y sumergir suavemente en AES durante 1 h a 4 ° C para permitir el desenrollar de la DNA. Guardar las diapositivas en la oscuridad.
- Añadir AES previamente enfriado en la bandeja de electroforesis diapositiva, no exceda 0,5 cm por encima de las diapositivas (esto depende del tamaño de las unidades de electroforesis), coloque el portaobjetos dentro y cubierta con una tapa. Ajuste la tensión de alimentación a 1 V/cm (la longitud entre los electrodos) y correr durante 30 min a 4 ° C.
- Solución de electroforesis exceso de drenaje de diapositiva. Cuidadosamente sumerja los portaobjetos en dH 2 O por 5 min a temperatura ambiente.
- Cuidadosamente sumerja los portaobjetos en etanol al 70% por 5 min a temperatura ambiente. Proceda al paso 4.
- Para el ensayo cometa neutro
- retire suavemente las diapositivas del LS, drenar el exceso de tampón y sumergir suavemente en NES por 30 min a 4 ° C. Mantenga el carro en la oscuridad.
- Añadir tampón de electroforesis neutro previamente enfriado en la bandeja de electroforesis diapositiva, no exceda 0,5 cm por encima de diapositivas (esto depende del tamaño de las unidades de electroforesis), coloque el portaobjetos dentro y cubierta con una tapa. Ajuste la tensión de alimentación a 1 V/cm (la longitud entre los electrodos) y correr por 45 min a 4 ° C.
- Descarga el exceso de tampón de las diapositivas. Cuidadosamente sumerja los portaobjetos en DPS durante 30 min a temperatura ambiente.
- Suavemente sumergir el portaobjetos en etanol al 70% durante 30 min a temperatura ambiente. Proceda al paso 4.
4. Diapositivas de la cometa de la mancha
- secar el portaobjetos a 37 ° C durante 10-15 min en la oscuridad.
- Lugar 50-100 μl verde fluorescente de ácido nucleico coloración de la solución en cada secado agarosa y tinción durante 15 min a temperatura ambiente en la oscuridad.
- Las diapositivas brevemente en dH 2 O y seque completamente a 37 ° C en la oscuridad. Proceder a la adquisición de imágenes y análisis.
5. Adquisición y análisis de imágenes
Nota: la visualización y cuantificación de roturas del ADN se basan en la microscopía de epifluorescencia y el software de ensayo de cometa (véase Tabla de materiales) 12 .
- Coloque los portaobjetos en el microscopio con un soporte de diapositivas. Asegúrese de que el gel de agarosa es frente a la lente del objetivo. Captura al azar de imágenes de las diapositivas manchadas cometa utilizando un microscopio de fluorescencia con un objetivo de 10 x. Evitar los bordes y las áreas alrededor de las burbujas de aire.
- Asegúrese de que cada cola de la cometa se distribuye horizontalmente. Cabezas de la cometa deben originar de la izquierda y la cola de la derecha.
- Guardar cada imagen en un formato binario de TIF con la brillante mancha de ADN y fondo oscuro. Carga de imágenes a los software que utilizan el " seleccionar los archivos para analizar " botón, que se encuentra a la izquierda de la barra de herramientas. Aparecerá una ventana de vista de la imagen ( figura 1).
- Dibujar un marco de medición en la pantalla y ajuste su tamaño conforme la cometa de la célula. Haga clic en el " ajuste " botón para configurar el umbral de la cabeza, la cometa y la cola según la imagen, a continuación, haga clic en el " iniciar medidas " botón ( figura 1).
- Seleccionar una celda usando el marco y activar la medición haciendo clic con el ratón en el " ensayo del cometa " botón; una intensidad imagen demuestra para arriba en el " perfiles " ventana con los parámetros de medición seleccionados. Los resultados se pueden guardar haciendo clic en el " almacenar resultado " botón ( figura 1).
Nota: El software calcula los parámetros incluyendo la longitud de la cola del cometa, el porcentaje de la cola de ADN, el momento de la cola (TM) y el momento de cola oliva (OTM). Los momentos de cola se calculan por las fórmulas como sigue:

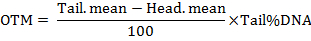
- analizar por lo menos 50 células por tratamiento.
Access restricted. Please log in or start a trial to view this content.
Resultados
El presente Protocolo describe un flujo de trabajo paso a paso para la ejecución del ensayo de cometa y análisis de datos (figura 1). Resultados de los ensayos de comet alcalino y neutro mostraron que la cola del cometa de doxorrubicina-U251 las células tratadas (1 μm, 20 h) era más largo y tenía mayor intensidad de ADN, lo que sugiere una sustancial acumulación de ADN fragmentado debido a la quimioterapia (figura 2).
...Access restricted. Please log in or start a trial to view this content.
Discusión
El ensayo cometa es una herramienta eficaz para medir solo y doble cadena roturas del ADN a nivel celular. El ensayo se ha aplicado extensamente como un "estándar de oro" en los estudios sobre genotoxicidad y biomonitoreo13, que van desde lesiones de base ADN reticulaciones, desarrollo de fármacos y sitios sensibles de álcali. En el presente estudio, hemos mostrado dos protocolos distintos paso a paso para los ensayos de comet alcalino y neutro, respectivamente. Combinación de electroforesis u...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Esta investigación fue apoyada por el programa de investigación intramuros de los NIH, el NCI y el CCR. Todos los autores recibieron beca de investigación intramuros de NIH, NCI y CCR.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 10x PBS(Ca++, Mg++ free) | TEKnova | P0196 | |
| NaCl | Sigma | S5886 | |
| EDTA | TEKnova | E0308 | |
| Trizma base | Sigma | T1503 | |
| NaOH | Sigma | 72068 | |
| Sodium lauryl sarcosinate | Sigma | L7414 | |
| Triton X-100 | Sigma | 93443 | |
| Sodium acetate | Sigma | 32318 | |
| Glacial acetic acid | Sigma | 695092 | |
| Ammonium acetate | Sigma | A1542 | |
| SYBR Green | Invitrogen | S33102 | |
| Low melting point agarose | Invitrogen | 16520 | |
| Agarose | Invitrogen | 16500 | |
| 95% ethanol | WARNER-GRAHAM | #64-17-5 | |
| Trypsin | GIBICO | 25300-054 | |
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| Glass tissue slides | ELECTRON MICROSCOPY SCIENCES | 63422-11 | |
| Kimwipes | KIMberly-Clark | ||
| 1.5 mL Microcentrifuge Tubes | DENVILLE | ||
| Pipette Tips | SHARP | ||
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Microwave | Avanti | ||
| Waterbath | PRECISION | ||
| Horizontal electrophoresis chamber | TREVIGEN | Cometassay ES II | |
| Power supply | Bio-Rad | ||
| Incubator | Quincy Lab | Model 12-140E | |
| Fluorescent microscope | Zeiss | LSM700 | |
| Micropipettor | Eppendorf |
Referencias
- Ostling, O., Johanson, K. J. Microelectrophoretic study of radiation-induced DNA damages in individual mammalian cells. Biochem Biophys Res Commun. 123 (1), 291-298 (1984).
- Singh, N. P., McCoy, M. T., Tice, R. R., Schneider, E. L. A simple technique for quantitation of low levels of DNA damage in individual cells. Exp Cell Res. 175 (1), 184-191 (1988).
- Tice, R. R., et al. Single cell gel/comet assay: guidelines for in vitro and in vivo genetic toxicology testing. Environ Mol Mutagen. 35 (3), 206-221 (2000).
- Shah, A. J., Lakkad, B. C., Rao, M. V. Genotoxicity in lead treated human lymphocytes evaluated by micronucleus and comet assays. Indian J Exp Biol. 54 (8), 502-508 (2016).
- Azqueta, A., Collins, A. R. The essential comet assay: a comprehensive guide to measuring DNA damage and repair. Arch Toxicol. 87 (6), 949-968 (2013).
- Goldstein, M., Kastan, M. B. The DNA damage response: implications for tumor responses to radiation and chemotherapy. Annu Rev Med. 66, 129-143 (2015).
- Gavande, N. S., et al. DNA repair targeted therapy: The past or future of cancer treatment? Pharmacol Ther. 160, 65-83 (2016).
- Torgovnick, A., Schumacher, B. DNA repair mechanisms in cancer development and therapy. Front Genet. 6, 157(2015).
- Weston, V. J., et al. The PARP inhibitor olaparib induces significant killing of ATM-deficient lymphoid tumor cells in vitro and in vivo. Blood. 116 (22), 4578-4587 (2010).
- Brown, J. S., O'Carrigan, B., Jackson, S. P., Yap, T. A. Targeting DNA Repair in Cancer: Beyond PARP Inhibitors. Cancer Discov. 7 (1), 20-37 (2017).
- Lu, Y., et al. Chemosensitivity of IDH1-Mutated Gliomas Due to an Impairment in PARP1-Mediated DNA Repair. Cancer Res. 77 (7), 1709-1718 (2017).
- Konca, K., et al. A cross-platform public domain PC image-analysis program for the comet assay. Mutat Res. 534 (1-2), 15-20 (2003).
- Valverde, M., Rojas, E. Environmental and occupational biomonitoring using the Comet assay. Mutat Res. 681 (1), 93-109 (2009).
- Collins, A. R. The comet assay for DNA damage and repair: principles, applications, and limitations. Mol Biotechnol. 26 (3), 249-261 (2004).
- Karbaschi, M., Cooke, M. S. Novel method for the high-throughput processing of slides for the comet assay. Sci Rep. 4, 7200(2014).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados