Method Article
Enriquecimiento de fosfopéptidos acoplada con espectrometría de masa libre de etiqueta cuantitativa para investigar la Phosphoproteome en cáncer de próstata
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo describe un procedimiento para extraer y enriquecer fosfotoproteida de líneas celulares de cáncer de próstata o tejidos para un análisis de la phosphoproteome través de proteómica masa basado en la espectrometría.
Resumen
Fosfoproteómico consiste en el estudio a gran escala de proteínas fosforiladas. Fosforilación de la proteína es un paso crítico en muchas vías de transducción de señal y es regulada firmemente por cinasas y fosfatasas. Por lo tanto, caracterizar la phosphoproteome puede proporcionar penetraciones en la identificación de nuevas dianas y biomarcadores para terapia oncológica. Espectrometría de masas proporciona un modo globalmente detectar y cuantificar miles de eventos de fosforilación único. Sin embargo, fosfotoproteida es mucho menos abundante que no fosfotoproteida, haciendo el análisis bioquímico más desafiante. Para superar esta limitación, se requieren métodos para enriquecer fosfotoproteida antes de los análisis de espectrometría de masas. Describimos un procedimiento para extraer y asimilar las proteínas del tejido para producir péptidos, seguidos por un enriquecimiento de fosfotirosina (pY) y péptidos de la fosfoserina-treonina (pST) utilizando un anticuerpo-basado o dióxido de titanio (TiO2)-base método de enriquecimiento. Después de la preparación de la muestra y espectrometría de masas, posteriormente identificar y cuantificar mediante cromatografía líquida-espectrometría y software de análisis fosfotoproteida.
Introducción
Un estimado 165.000 nuevos casos y aproximadamente 29.000 muertes ocurrirá en 2018 debido a cáncer de próstata, que representa el cáncer más común y la segunda causa de muerte relacionada con cáncer en hombres en los Estados Unidos1. Primeras etapas del cáncer de próstata son tratables con terapia de radiación o resección de la enfermedad órgano-confinada, donde la tasa de recurrencia de 10 años es entre 20% y 40% para los pacientes que se someten a prostatectomía y entre 30% y 50% para los pacientes que reciben radiación terapia2. Porque el cáncer de próstata se basa en andrógenos de señalización para el crecimiento, terapias de la castración quirúrgica y química se emplean también para pacientes de alto riesgo. Sin embargo, la recaída ocurre cuando el cáncer ya no responde a terapia de privación de andrógenos evidenciada por recurrencia bioquímica, donde el antígeno prostático específico en suero se levanta otra vez. En este punto en la progresión, las metástasis son a menudo detectadas así. Esta etapa avanzada, llamada cáncer de próstata metastásico resistente a la castración, representa la forma letal de la enfermedad donde el pronóstico es un tiempo de supervivencia media de menos de dos años3. Pocas opciones de tratamiento están disponibles en fase de enfermedad, incluyendo antiandrógenos de segunda generación como enzalutamide y abiraterona, quimioterapia basada en taxanos como docetaxel. A pesar de los tratamientos disponibles, la enfermedad a menudo progresa. Por lo tanto, el descubrimiento y desarrollo de modalidades de tratamiento nuevo son necesarias para mejorar la atención de pacientes de cáncer de próstata con enfermedad avanzada.
Espectrometría de masas (MS)-basado los acercamientos proporcionan un análisis global del proteoma mediante la detección de cientos de miles de analitos péptido4. En particular, el descubrimiento proteómica, también conocido como adquisición de datos dependientes (DDA), puede producir la identificación y cuantificación de miles de péptidos4,5. Descubrimiento basado en MS proteómica puede ser delineado más en proteómica de arriba hacia abajo, donde se caracterizan las proteínas intactas, y la proteómica (también conocido como escopeta) de abajo a arriba, donde se analizan los péptidos para caracterizar las proteínas5. Así, en proteomics de la escopeta, un paso de la proteólisis ocurre en la preparación de la muestra anterior el análisis de MS que desdoblan las proteínas en péptidos. Al final, se realiza una búsqueda de base de datos para asignar los péptidos a las proteínas para la identificación. Etiqueta-libre, así como varios isótopos etiquetado[por ejemplo, el isótopo estable que etiqueta por los aminoácidos en cultura de célula (SILAC)] métodos pueden utilizarse para comparar cuantitativamente péptidos entre muestras6,7. Etiquetado técnicas isotópicas son el estándar de oro, libre de etiqueta métodos han demostrado similar cuantificación exactitudes8,9 y tienen compensaciones comparables entre sensibilidad y especificidad10. Cuantificación de etiqueta-libre proporciona una mayor cobertura y permite comparaciones entre muchas más muestras, mientras que los métodos basados en la etiqueta están limitados por costos y multiplexación capacidad6,7,8.
Además, escopeta MS también puede utilizarse para interrogar (PTMs) las modificaciones post-traduccionales como fosforilación11. Debido a la naturaleza estequiométrica inferior de fosfotoproteida en comparación con péptidos total, se emplean varios métodos para enriquecer para fosfotoproteida, incluyendo inmunoprecipitación basados en anticuerpos de péptidos del phosphotyrosine (pY), dióxido de titanio (TiO2 ) e inmovilizados metal afinidad cromatografía (IMAC)5,12. Ya que la fosforilación de la proteína es un paso clave en muchos célula de señalización de vías, escopeta fosfoproteómico permite a los investigadores investigar cambios en diferentes tipos de cáncer, incluyendo mama13,14de próstata, renal15, de señalización de la célula y ovario,16,17 para comprender mejor la biología del cáncer y para identificar nuevas dianas potenciales para la terapia.
Este método de phosphoproteomic de fusil libre de etiqueta fue construido y refinado basado en trabajo previo por el grupo de Graeber18,19,20. Este protocolo se inicia describiendo la extracción y digestión de proteínas y fosfoproteínas de tejido en péptidos. A continuación detallamos el enriquecimiento de pY péptidos usando anticuerpos específicos fosfotirosina y TiO2. También describimos el enriquecimiento de los péptidos de la fosfoserina-treonina (pST) mediante el fuertes del intercambio catiónico (SCX) seguido de TiO2. Este protocolo concluye con la presentación de las muestras a un centro de MS y el uso de software de análisis de MS para identificar y cuantificar fosfotoproteida y sus correspondientes fosfoproteínas. La aplicación de este protocolo puede extenderse más allá del cáncer de próstata en otros campos fuera de Oncología y cáncer.
Protocolo
Experimentos con tumores xenoinjerto fueron aprobados por la Rutgers Universidad institucional Animal Care y uso como conjunto adelante bajo los lineamientos de los institutos nacionales de salud.
1. extracción de proteínas
- Preparar el tampón de lisis (tabla 1). (El volumen depende el número de muestras a ser cosechado). Para muestras de célula en vitro , proceda al paso 1.2. Para el tejido del tumor, proceda al paso 1.3.
-
Recolección de células
- Recoger las células en un tubo cónico de 50 mL y vuelta a 700 x g durante 5 min a 4 ° C. Deseche el sobrenadante y mantener el diábolo en el hielo. Repita este paso para todos los platos recoger las células en un perdigón. (Normalmente, unos 5 platos 15 cm casi confluentes de células se necesitan para 5 mg de proteína, pero este puede ser dependiente en la línea celular y debe determinarse empíricamente por cada investigador.)
- Lavar el pellet con 30 mL de refrigerados con tampón fosfato salino (PBS) y vuelta a 700 x g durante 5 min a 4 ° C antes de aspirar el PBS. Añadir 1,5 mL de tampón de lisis por 5 mg de proteína utilizada para el precipitado de células. Pipeta de arriba a abajo un par de veces. Vaya al paso 1.4.
-
Recolección de tejidos
- Peso del tumor y añadir 2 mL de tampón de lisis helada por cada 100 mg de tejido en un tubo de ensayo de la cultura. (Por lo general, es necesario 50 a 150 mg de peso húmedo de tejido).
- Homogeneizar el lisado usando un homogeneizador de mano o sobremesa (pulso 2 x 15 s.) Limpie el homogenizador antes de la primera muestra y entre muestras mediante el uso de lejía del 10%, etanol al 70% y agua desionizada en sucesión.
- Para reducir y alkylate, calentar las muestras homogeneizadas a 95 ° C durante 5 minutos. Luego enfriarlos en hielo durante 15 minutos. Someter a ultrasonidos en el hielo, el lisado x 3 (es decir, el pulso durante 30 s con 60 s pausas entre pulsos). La muestra no debe ser viscoso o clumpy en este punto. Calienta el lisado a 95 ° C por 5 min21.
- Centrifugadora lisado en la sonicación mismo tubo usando un rotor de columpio cubo 3.500 x g a 15 ° C durante 15 minutos recoger el sobrenadante y desechar el precipitado.
- Determinar la concentración de proteína mediante la realización de un ensayo de Bradford22. Si es necesario, diluir el lisado a 5 mg/mL con un tampón de lisis. Almacenar a-20 ° C.
Nota: El experimento puede hacer una pausa aquí. Congelar las muestras a-80 ° C y continuar en una fecha posterior.
2. lisada digestión
- Diluir la muestra de 12-fold mediante el uso de 100 mM Tris (pH = 8.5) para reducir la cantidad de guanidinio. Diluir todas las muestras para el mismo volumen para minimizar los efectos de la digestión desigual. Ahorrar 12,5 μg de los digeridos lisado para confirmar en un gel teñido de Coomassie23.
- Para 5 mg de proteína, añadir 10 μg de lisil endopeptidasa (Lys-C) e incubar a temperatura ambiente para ajustar pH 5-6 h. 8.0 por adición de 1 M untitrated Tris (pH ~ 11).
- Preparar 1 mg/mL de L-1-tosylamido-2-FENILETILO clorometil cetona (TPCK)-tratado de tripsina en HCl 1 m m (con 20 m de CaCl2). Añadir la tripsina en una proporción de tripsina: proteína de 1: 100 e incubar a 37 ° C durante 3 h.
- Añadir la misma cantidad de tripsina fresco como en el paso 2.3. Incubar a 37 ° C durante la noche.
- Ahorrar 12,5 μg de la digerida lisado para confirmar la digestión completa en un gel teñido de Coomassie23.
3. Invierta la extracción en fase
- Registrar el volumen lisado. Filtrar la muestra mediante un filtro de corte 15 mL 10 kDa. Centrifugar la muestra a 3.500 x g utilizando el swing cubo rotor (o 3.500 g de x en un rotor de ángulo fijo) a 15 ° C hasta que el volumen retenido es de menos de 250 μl (esto tarda unos 45-60 min). Recoge el flujo a través y deseche el retenido.
Nota: El experimento puede hacer una pausa aquí. Congelar las muestras a-80 ° C y continuar en una fecha posterior. - Para acidificar la muestra, agregar aproximadamente 20 μl de 5% de ácido trifluoroacético (TFA) por mL de lisado. Mézclelos bien y medir el pH de la muestra mediante el uso de las tiras de pH. Ajustar el pH a 2.5 usando 5% TFA.
- Conecte el extremo corto de una columna de C-18 a un colector de vacío. Establecer el vacío entre 17 y 34 kPa (o según las instrucciones del fabricante). El uso de pipetas de vidrio , mojadas la columna con 3 mL de 100% acetonitrilo (ACN). No deje que la columna seca.
- Usando pipetas de vidrio , equilibrar la columna con 6 mL de 0.1% TFA aplica como 2 x 3 mL. Carga de la muestra acidificada en la columna. No agregue más de 3 mL a la vez. Ajuste el vacío para llegar a cerca de 1 a 2 gotas por segundo.
- Usando pipetas de vidrio , lavar la columna con 9 mL de 0.1% TFA aplica como 3 x 3 mL. Eluir la columna con 2 mL de 40% ACN, 0.1% TFA. Recoja dos fracciones de 2 mL en tubos de cultivo de cristal . Deseche el columna.
- Cubrir los tubos de elución con parafilm y 3-5 agujeros en la cubierta utilizando una aguja de 20G. Congelar el eluido en hielo seco durante al menos 30 minutos hasta que esté totalmente sólido.
- Liofilizar las fracciones durante la noche. Al día siguiente, asegúrese de que las muestras estén completamente secas antes de parar el liofilizador. Guarde los tubos en un tubo cónico de 50 mL con delicadas toallitas a-80 ° C.
Nota: El experimento puede hacer una pausa aquí.
4. inmunoprecipitación y enriquecimiento de pY péptidos24
- Resuspender el polvo liofilizado con 0,5 mL de tampón de Unión helada inmunoprecipitación (IP) en cada fracción. Piscina las fracciones al transferir el volumen de resuspensión de 0,5 mL de la fracción segunda a la primera fracción y guarde la pipeta. Vortex vigorosamente (en vez de pipeteo arriba y abajo) para asegurarse de que la muestra esté completamente disuelta antes de transferirla a un criotubo de 3,6 mL tapón de rosca.
- Como en el paso 4.1., enjuague los tubos de liofilización con otro 0,5 mL de tampón de unión de IP (tabla 1) en cada tubo. Transferir la solución al tubo de tapón de rosca de 3,6 mL utilizando la misma punta de pipeta para minimizar cualquier pérdida de muestra. Repita el enjuague 1 x más, haciendo que el volumen de resuspensión final 2 mL (5 mg de proteína). Medir el pH de la muestra para asegurarse de que es aproximadamente de 7,4. Si es demasiado ácida, iterativamente agregar 10 μl de 1 M Tris (pH untitrated, ~ 11). Si es demasiado básico, iterativamente agregar 10 μl de HCl diluido (1:25 ó 1: 100).
-
Lavado preliminar de los granos de pY (para 5 mg de a partir de lisado)
- por ejemplo se necesitan 25 μg de anticuerpo 4 10 y 12,5 μg de anticuerpo 27B10.4. Después de usar una p200 pipeta con una punta de corte para transferencia de los anticuerpos en los tubos de microcentrífuga separado, lavar los anticuerpos con 450 μl de tampón de unión de helada IP 2 x. Les centrifugar a 100 x g durante 1 min a 4 ° C y aspirar hacia fuera el sobrenadante.
- Resuspender los granos a una concentración stock de 0.5 mg/mL con buffer de enlace IP. (No no agitar con vortex los granos). Después de tomar la mezcla necesaria (50 μl de mezcla de anticuerpos de 10 4 y 25 μl 27B10.4 anticuerpos de la mezcla por ejemplo) en un solo tubo, girar por los tubos de centrífuga común a 200 x g durante 1 min a 4 ° C. Lavar las paredes con sobrenadante antes de devolver los granos para almacenamiento en el refrigerador.
- Añadir granos de pre-lavado pY a la solución de muestra resuspendidos en los criotubos de tapón de rosca. Les incube a 4 ° C en un rotador de extremo a extremo toda la noche.
- Coloque los criotubos de tapón en un tubo de centrífuga de 50 mL con un delicado paño. Desactivación de las cuentas a 100 x g durante 1 minuto guarde el sobrenadante, que se utilizará para enriquecer para péptidos de pST. El enriquecimiento para pST comienza en el paso 7 y puede realizarse en paralelo para el procesamiento de péptidos de pY.
- Resuspender los granos con 300 μL de tampón de enlace IP. Transferir a un tubo de microcentrífuga de 2 mL y los hace girar hacia abajo a 100 x g durante 1 min a 4 ° C.
- Enjuague la incubación del tubo 3 x con 200 μL de tampón de enlace IP. Transferir el contenido al mismo tubo de microcentrífuga cada vez. Luego los hace girar hacia abajo.
- Lavar los granos en la microcentrífuga tubo 3 veces con 500 μl de tampón de unión de IP y los hace girar hacia abajo a 100 x g durante 1 minuto. Luego lavar los granos 4 x con 450 μl de 25 mM NH4HCO3, pH 7,5 y vuelta les abajo a 100 x g durante 1 min uso un fresco 25 mM NH4HCO3 solución de polvo cada vez.
- Centrifugue los granos a 1.500 x g durante 1 minuto uso una punta de carga de gel para eliminar completamente el sobrenadante sumergiendo la punta de la punta de carga de gel ligeramente por debajo de la superficie de los granos.
- Añadir 4 x el volumen de grano de 0.1% TFA para los granos (es decir, añadir 300 μL de 0.1% TFA de 75 μg de mezcla de grano de pY). Mezclar bien e incubar la mezcla en un Termomezcladores a 1.000 rpm durante 15 min a 37 ° C.
- La resuspensión de transferencia a un filtro de 0.2 μm spin. Rápidamente girar hacia abajo el tubo de elución y el volumen residual la transferencia al mismo filtro spin utilizando una pipeta P10. Desactivación del filtro spin 850 x g durante 1 minuto transferencia la elución a un tubo de microcentrífuga de baja unión a proteínas . Vacío durante la noche se concentran el eluido a sequedad a 40 ° C y con un tiempo de calor de 300 min.
Nota: El experimento puede hacer una pausa aquí. Congelar las muestras a-80 ° C y continuar en una fecha posterior.
5. dióxido de titanio enriquecimiento25 de pY péptidos
- Resuspender el secado abajo fosfotoproteida en 200 μL de 50% ACN, 0.1% TFA. Vórtice y ellos centrifugar a 10.000 x g durante 30 s. repetir este 1 x para resuspenderlos bien.
- Preparación de los granos de TiO2 en consejos que tienen una capacidad para 200 μL muestras.
- Golpee suavemente en el lado pequeño de la punta de la punta para mover el material a tal fin. Enjuague la punta mediante la adición de 200 μL del 100% ACN, seguido por invertir la punta y sacudiendo el extremo pequeño para mover el líquido hacia la tapa.
- Usando una cuchilla de afeitar, corte la punta pequeña de la punta y coloque sobre un tubo de unión de proteínas baja. (Evite usar tubos de poliestireno como el TiO2 se pegará a los lados del tubo.) Retire la tapa e inserte una micropipeta para sumergirse a la ACN restantes. Repetir el lavado con 200 μL del 100% ACN. Los granos de TiO2 se encuentran ahora en el tubo de unión de proteínas baja para los siguientes pasos.
- Requisito de TiO2 con 500 μl de ACN 100% 2 x. Pipeta para mezclar los granos con el solvente. Les centrifugar a 100 x g durante 1 minuto.
- Condición de TiO2 con 500 μl de tampón de fosfato de sodio 0.2m (pH ~ 7) x 2. Lavar los granos con 300 μL de equilibrio buffer x 3. Porque el TiO2 es muy denso, los granos se repone rápidamente.
- Añadir 400 μL de 50% ACN, 0.1% TFA en el tubo de baja unión a proteínas, seguida de adición 84 μl de ácido láctico. Transferencia fosfotoproteida resuspendido en el tubo de unión a proteínas bajo e Incube por 1 h a temperatura ambiente usando un rotor de extremo a extremo.
- Centrifugue los granos a 100 x g durante 1 min para los pellets. Lavar con 300 μL de tampón de equilibrado (tabla 1) 2 x y los hace girar hacia abajo a 100 x g durante 1 minuto.
- Enjuagar los granos con 300 μL de lavado Tampón x 2. Transfieren a un filtro de centrifugado de 0.2 μm. Los hace girar a 1.500 x g durante 1 minuto.
- Transferencia de la unidad de filtro a un tubo limpio de 1.5 mL de unión a proteínas baja. Eluir el contenido 2 x con 200 μL de 0.9% NH3 H2O. medida del pH con las tiras de pH, que debe estar entre 10 y 11. Vacío se concentran el eluido a sequedad durante la noche para evaporar el amoniaco.
6. péptidos de pY para MS Analyses de la desalación
- Reconstituir el fosfotoproteida con 15 μl de 0.1% TFA por Vortex y centrifugar a 10.000 x g por 30 s para suspender de nuevo los. Repita este 1 x para resuspenderlos bien. No pipetear arriba y abajo.
- Limpiar la muestra usando una punta de C-18 con una capacidad de enlace de 5 μg y seguir el protocolo del fabricante.
- Secar completamente el volumen de elución por concentración del vacío. Esto tarda 1-2 h. Resuspender el fosfotoproteida secado en 12,5 μl de solución de espectrometría de masas (ver tabla 1) (o como recomendada por MS del investigador proteómica core facility). Vortex y centrifugar brevemente la solución abajo a 10.000 x g por 30 s. repetir este 2 x para resuspenderlos bien. Las muestras están listas para envío a un centro de espectrometría de masas (paso 11).
Nota: Los siguientes pasos que a continuación se relacionan con enriquecimiento de péptido de pST sólo.
7. Invierta la extracción en fase de péptidos de pST
- Medir la concentración de péptido del sobrenadante adquirido paso 4.6 por realizar un análisis de péptidos. Una cantidad suficiente para espectrometría de masas pST es de 2,5 mg.
- Ajustar el pH a 3.5 con 5% TFA.
- Conecte el extremo corto de una columna de C-18 a un colector de vacío. Establecer el vacío entre 17 y 34 kPa (o según las instrucciones del fabricante). Moje la columna con 3 mL de 100% ACN. No deje que la columna seca.
- Equilibrar la columna con 6 mL de 0.1% TFA aplica como 2 x 3 mL. Carga de la muestra acidificada en la columna. No agregue más de 3 mL a la vez. Ajuste el vacío para llegar a unos 1-2 gotas por segundo.
- Lavar la columna con 9 mL de 0.1% TFA aplica como 3 x 3 mL. Eluir la columna con 2 mL de 40% ACN, 0.1% TFA. Recoja dos fracciones de 2 mL en tubos de cultivo de cristal. Deseche el columna.
- Cubrir los tubos de elución con parafilm y 3-5 agujeros en la cubierta utilizando una aguja de 20G. Congelar el eluido en hielo seco durante al menos 30 minutos hasta que esté totalmente sólido.
- Liofilizar las fracciones seleccionadas durante la noche. Al día siguiente, asegúrese de que las muestras estén completamente secas antes de parar el liofilizador. Guarde los tubos en un tubo cónico de 50 mL con delicadas toallitas a-80 ° C.
Nota: El experimento puede hacer una pausa aquí.
8. fuerte del intercambio catiónico (SCX) de péptidos de pST
- Resuspender los péptidos liofilizados en 2 mL de Buffer A (tabla 1). Las fracciones de cada muestra de la piscina. (La solución debe estar turbia).
- Preparar el múltiple de vacío. Conectar una columna SCX a una jeringa de 3 mL con el émbolo retirado. Establecer el vacío entre 17 y 34 kPa (o según las instrucciones del fabricante).
- Acondicionar la columna SCX con 4 mL de ACN, seguido de 4 mL de Buffer A.
- 2 mL de la muestra de la carga del paso 8.1 y recoger el eluido inmediatamente. Carga 3 mL de tampón a: b (80.9:19.1) y recoger el eluido. Piscina de los eluatos de cada muestra y alícuota les en tubos con 2 mL baja unión a proteínas.
- Vacío se concentran todas las muestras hasta que quede aproximadamente el 30% del volumen. (Este paso dura aproximadamente 2-4 h.) Piscina las alícuotas en 1 tubo bajo de unión a proteínas para cada muestra.
- Conecte el extremo corto de una columna de C-18 a un colector de vacío. Establecer el vacío entre 17 y 34 kPa (o según las instrucciones del fabricante). Moje la columna con 3 mL de 100% ACN 2 x. No deje que la columna seca.
- Equilibrar la columna con 3 mL de 0.1% TFA 2 x. Cargar la muestra en la columna. No agregue más de 3 mL a la vez. Ajuste el vacío para llegar a unos 1-2 gotas por segundo.
- Lavar la columna con 3 mL de 0.1% TFA 2 x. Eluir la columna con 4 mL 50% ACN, 0.1% TFA.
9. dióxido de titanio enriquecimiento de péptidos de pST
-
Preparación de los granos de TiO2 en consejos que tienen una capacidad para 200 muestras μl
- Golpee suavemente en el lado de punta pequeña de la punta para mover los granos para ello. Retire la tapa y verter los granos en un tubo cónico de polipropileno de 15 mL.
- Enjuague la punta mediante la adición de 200 μL del 100% ACN, invirtiendo la punta un par de veces y sacudiendo el extremo pequeño para mover el líquido hacia la tapa. Usando una cuchilla de afeitar, corte la punta pequeña de la punta y colocar sobre el tubo cónico de polipropileno de 15 mL. Retire la tapa e inserte una micropipeta para sumergirse a la ACN restantes. Repetir el lavado con 200 μL del 100% ACN. El TiO2 cuentas ahora se encuentran en el tubo cónico de 15 mL para los siguientes pasos.
- Requisito de TiO2 con 500 μl de ACN 100% 2 x. Pipeta para mezclar los granos con el solvente. Les centrifugar a 100 x g durante 1 minuto.
- Condición de TiO2 con 500 μl de tampón de fosfato de sodio 0.2m (pH ~ 7) dos veces. Lavar los granos con 300 μL de equilibrio buffer x 3.
- Transferencia fosfotoproteida eluída en el tubo cónico de polipropileno de 15 mL. Añadir 560 μl de ácido láctico e incubar durante 1 h a temperatura ambiente usando un rotor de extremo a extremo.
- Centrifugar la mezcla a 100 x g durante 1 min para que sedimenten los granos. Lavar con 300 μL de tampón de equilibrado (tabla 1) 3 x. Los hace girar hacia abajo a 100 x g durante 1 minuto.
- Enjuagar los granos con 300 μL de lavado Tampón x 2. Transfieren a un filtro de centrifugado de 0.2 μm. Los hace girar hacia abajo a 1.500 x g durante 1 minuto.
- Transferencia de la unidad de filtro a un tubo limpio de 1.5 mL de unión a proteínas baja. Eluir el contenido 2 x con 200 μL de 0.9% NH3 H2O. Deje la solución reposar en fosfotoproteida por 2 min antes de los liberador de. Medir el pH, que debe ser entre 10 y 11.
- Vacío se concentran el eluido a sequedad durante la noche para evaporar el amoniaco.
10. péptidos de pST de la desalación para análisis de MS
- Golpee suavemente en el lado pequeño de la punta de la punta para mover el material a tal fin. Enjuague la punta mediante la adición de 200 μL del 100% ACN, seguido por invertir la punta y sacudiendo el extremo pequeño para mover el líquido hacia la tapa.
- Usando una cuchilla de afeitar, cortar la punta pequeña de la punta y coloque en un tubo cónico de polipropileno de 15 mL. Retire la tapa e inserte una micropipeta para sumergirse a la ACN restantes. Repetir el lavado con 200 μL del 100% ACN. Los granos de TiO2 se encuentran ahora en el tubo cónico de polipropileno de 15 mL para los siguientes pasos.
- Limpiar la muestra usando una punta de C-18 con una capacidad de 100 μg. (siga las instrucciones del fabricante).
- Secar completamente el volumen de elución por concentración del vacío. Esto toma 1-2 h.
- Resuspender el fosfotoproteida secado en 12,5 μl de solución de espectrometría de masas (o recomendación de instalaciones centrales para del investigador MS proteómica). Vortex y centrifugar a 10.000 x g por 30 s. Repita 2 x para resuspenderlos bien. (No pipetear arriba y abajo.)
11. Análisis de espectrometría de masas
- Enviar las muestras al centro de base MS proteómica para realizar MS líquida de cromatografía en tandem (LC-MS/MS) usando la configuración recomendada. Ejemplo con los siguientes parámetros (ver tabla 2 para el resumen):
- Carga 5 μl de las muestras en una columna de la trampa (2 cm de largo x 75 μm de diámetro) y lávelos con 0.1% TFA por 5 min con un caudal de 5 μl/min.
- Traer la trampa con una columna analítica de nano (20 cm x 75 μm) con un caudal de 300 nL/min.
- Los gradientes lineales segmentados (un porcentaje de 0.16% de ácido fórmico, 80% ACN en 0.2% de ácido fórmico) son diferentes entre las muestras de pY y pST:
- Para las muestras de pY, procederá a la elución con un gradiente de 4-15% en 5 min, 15-50% en 40 min y 50-90% en 5 minutos.
- Para las muestras de pST, procederá a la elución con un gradiente de 4-15% en 30 min, 15-25% en 40 minutos, 25-50% en el minuto 44 y 50-90% en el minuto 11.
- Adquirir datos de MS en el modo de adquisición de datos-dependiente con una serie cíclica de un análisis completo con una resolución de 120.000 seguido por MS/MS (HCD, energía de la colisión relativa del 27%) de los iones más intensos 20 y una duración de la dinámica de exclusión de 20 s.
- A la MS ejecutar terminación, importar los archivos raws de MS en un programa de software de análisis de MS para identificar y cuantificar fosfotoproteida. (MaxQuant software8,26,27 fue utilizado en este experimento. Salvo que se especifique en el cuadro 3, los valores iniciales fueron utilizados.)
Resultados
Este protocolo describe detalladamente un método para extracción de proteínas y la digestión seguida por enriquecimiento de fosfopéptidos y posterior análisis de MS (figura 1). Las composiciones de todos los buffers y soluciones que se utilizan en el presente Protocolo se enumeran en la tabla 1. El uso secuencial de Lys-C y tripsina proporciona una digestión eficiente. Un tinción de Coomassie gel de pre digerida lisado confirma la presencia de proteínas, mientras que la coloración de la digerida lisado confirma la digestión completa (figura 2A). Para una digestión completa, no bandas deberían aparecer por encima de 15 kDa, excepto el 30 kDa y 23,3 kDa bandas para Lys-C y tripsina, respectivamente. La adición de Lys-C también reduce el número de divisiones perdidas (figura 2B). Porque pY péptidos representan sólo el 2% de los phosphoproteome28, inmunoprecipitación de los péptidos de pY usando un anticuerpo específico de pY es el primer paso de enriquecimiento de péptido de pY. El sobrenadante resultante se convierte en la entrada para el enriquecimiento de péptido de pST. La inmunoprecipitación de pY efectivamente separa pY péptidos péptidos pST donde en promedio 85% de lo fosfotoproteida identificada a partir de la preparación de pY pY (Figura 3A) y más del 99% de lo fosfotoproteida identificada a partir de la preparación de pST son pST (figura 3B). Dióxido de titanio se utiliza para enriquecer para fosfotoproteida en ambas preparaciones. El porcentaje esperado de péptidos en la preparación de MS listo que se fosforila es entre 30-50% (Figura 4A). La variabilidad en el porcentaje de enriquecimiento de fosfopéptidos puede ser mayor en la preparación de pY como resultado hay muchos menos péptidos de pY que péptidos de pST. En términos de fosfopéptidos especies, la mayoría de lo fosfotoproteida detectado tiene un grupo fosforilo de simple o doble (Figura 4B).
Después de realizar la espectrometría de masas, se cargan los archivos crudos de MS en un software de análisis de MS. La configuración de los parámetros utilizada en el experimento se enumeran en la tabla 3 pero variará de software a software y puede variar de una versión a. Los parámetros que se enumeran no quedaron como predeterminado, incluyendo un FDR corte del 1% para juego de péptido-espectro (PSM) con una calificación mínima de Andrómeda del 40 para la identificación de péptidos modificados27. Establecer un corte de la probabilidad de localización de más de 0,75 filtra aproximadamente 5% de los péptidos de pY y 15% y 34% de la pS y pT péptidos, respectivamente (figura 5A). Después de aplicar estos filtros, el número esperado de identificaciones de fosfopéptidos en el final del análisis de MS es de aproximadamente 300 pY péptidos (de 5 mg de la proteína de partida) y unos 7.500 péptidos pS y pT 640 péptidos (de 2,5 mg del péptido a partir de cantidad) de las preparaciones de enriquecimiento respectivo (figura 5B). El número de repeticiones y la variabilidad de la intensidad de la señal de fosfopéptidos determinan la alimentación adecuada para las comparaciones estadísticas. En cuatro experimentos independientes con grupos biológicos duplicados o triplicados, se calcularon los coeficientes de porcentaje de variación (% CV) de fosfotoproteida detectado. Distribución de la variabilidad (p. ej., pST grupos 1-5 en la figura 5) indica que la recogida de muestras, preparación y espectrometría de masas funciona era constante. Por otro lado, las distribuciones de mayor variabilidad (p. ej., pST Grupo 6 en figura 5) indica datos más ruidosos que requerirían cambios de doblez más grandes para detectar diferencias significativas en el análisis diferencial aguas abajo.
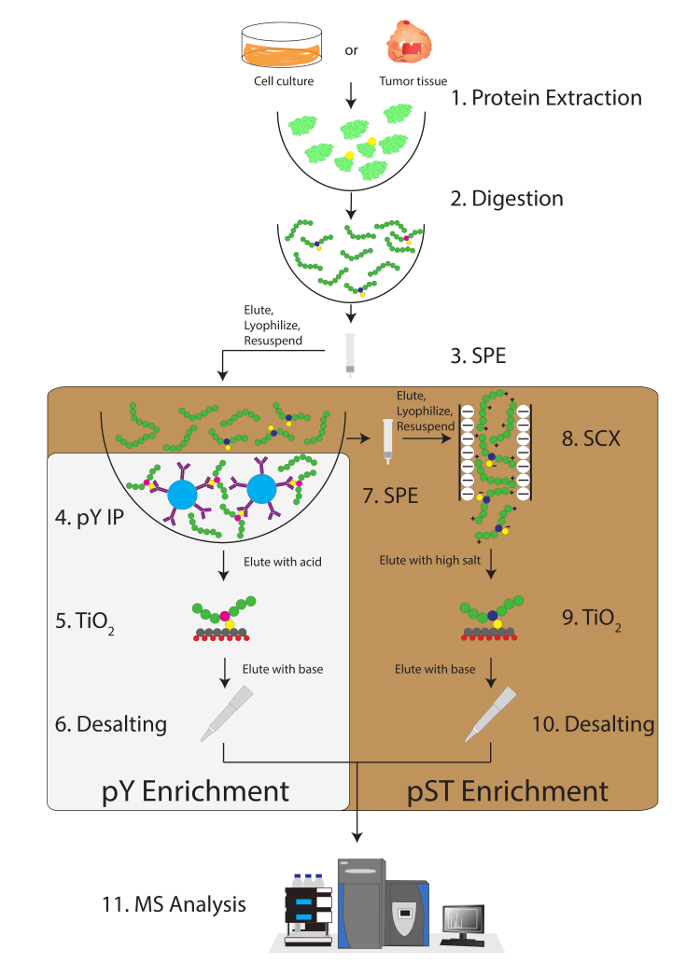
Figura 1: Diagrama de flujo de trabajo. Proteínas de las muestras extraídas y digeridas. Péptidos son extraídos por extracción en fase sólida (SPE), y péptidos de fosfotirosina (pY) son immunoprecipitated. En paralelo, los péptidos de la fosfoserina-treonina (pST) se enriquecen desde el sobrenadante en el paso de la inmunoprecipitación de pY. Intercambio catiónico fuerte (SCX) se realiza en el sobrenadante para eliminar los péptidos altamente cargadas para reducir los iones supresión12. Ambas preparaciones se someten a enriquecimiento de fosfopéptidos vía la dióxido de titanio (TiO2). Después de la limpieza de la muestra, líquido cromatografía-tandem espectrometría de masas (LC-MS/MS) se realiza para medir la abundancia de fosfopéptidos. Los datos en bruto se cargan luego en un software de análisis de MS para identificar fosfotoproteida. Haga clic aquí para ver una versión más grande de esta figura.
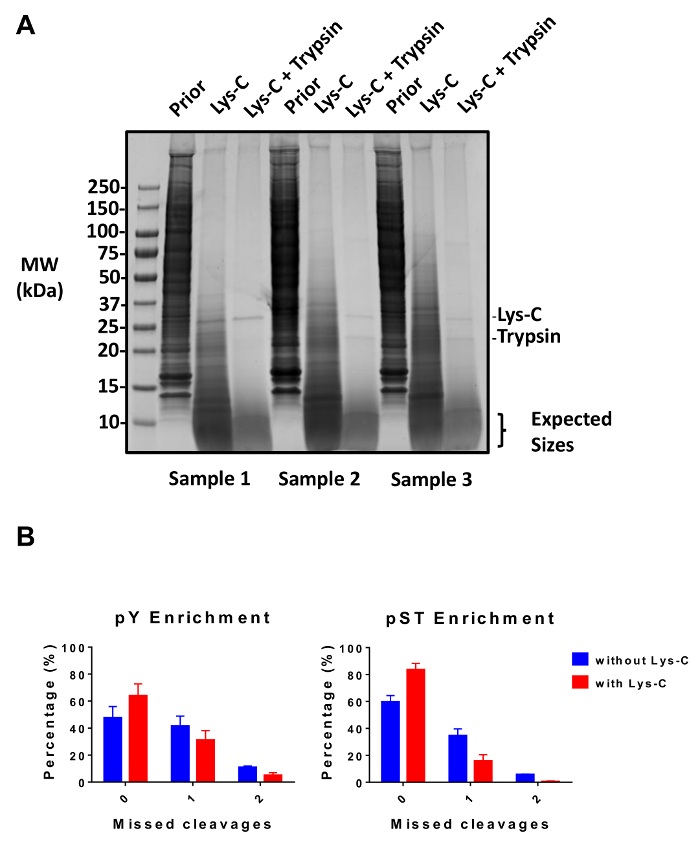
Figura 2: evaluación de la digestión de. (A) tres muestras con 12,5 μg de lisado digestión pre- digestión, post-Lys-C, y digestión de la tripsina se muestran. Una prueba de gel colorante Coomassie indica una digestión limpia después del uso secuencial de Lys-C y tripsina. Los marcadores de peso molecular (MW) tamaño están en kilodaltons (kDa). (B) A reducción de divisiones perdidas se observa después de Lys-C fue agregado al Protocolo. El porcentaje de fosfotoproteida sin perdidas divisiones aumentó de 48% a 64% y del 60% al 84% en promedio para las preparaciones de enriquecimiento pY y pST, respectivamente. Los gráficos de resumen los datos obtenidos en dos experimentos realizados sin Forfait Lis y cinco experimentos llevados a cabo con Lys-C. Las barras de error son desviaciones que representan pY 38 y 38 muestras de pST de 2 experimentos separados (sin Forfait Lis) y pY 62 y 60 muestras de pST de 5 experimentos independientes (con Lys-C). Haga clic aquí para ver una versión más grande de esta figura.
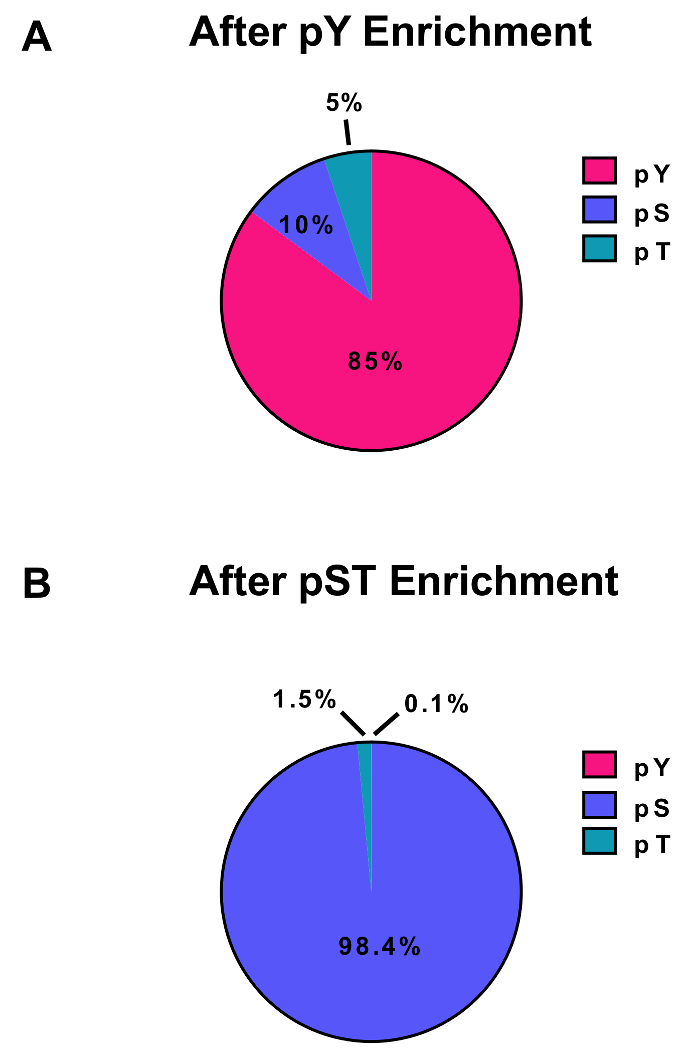
Figura 3: enriquecimiento de pY y pST fosfotoproteida. Estos paneles muestran los porcentajes de pSTY fosfotoproteida de (A) el pY o (B) las preparaciones de enriquecimiento del pST. El enriquecimiento de pY pY inmunoprecipitación y dióxido de titanio resultó en 85% fosfotoproteida ser para pY péptidos, mientras que sólo el 0.1% de la fosfotoproteida en el enriquecimiento de pST son pY. Estos valores fueron dibujados de examinar la fosfo (archivo STY)Sites.txt de un experimento representativo después de filtrar los contaminantes, secuencias de reversas y fosfotoproteida con probabilidades de localización inferior a 0.75. Haga clic aquí para ver una versión más grande de esta figura.
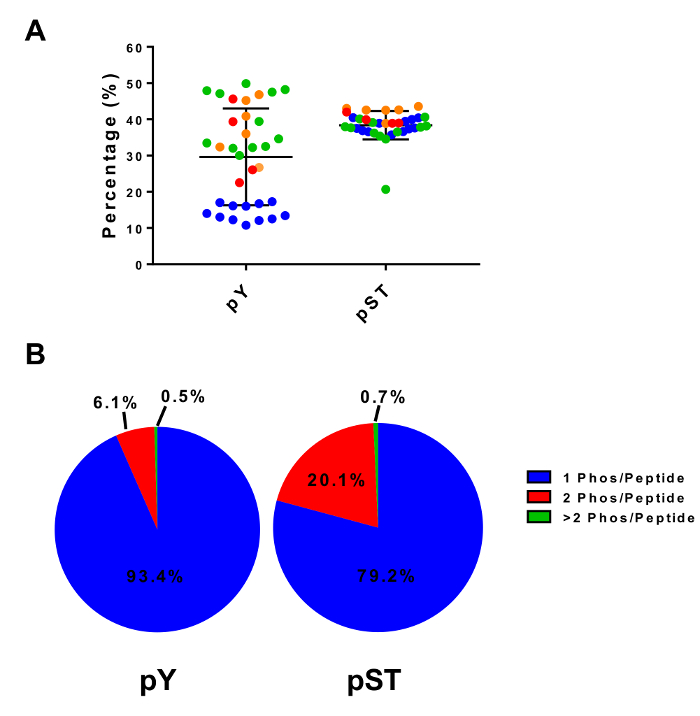
Figura 4: enriquecimiento de fosfopéptidos con dióxido de titanio. (A) se muestra el porcentaje de fosfotoproteida detectado (en relación al total péptidos) de muestras en cuatro experimentos separados. (B) este panel muestra la composición media de mono, doble y multi-phosphorylated péptidos en cuatro experimentos separados. Las barras de error en el panel de A son las desviaciones estándar. Haga clic aquí para ver una versión más grande de esta figura.
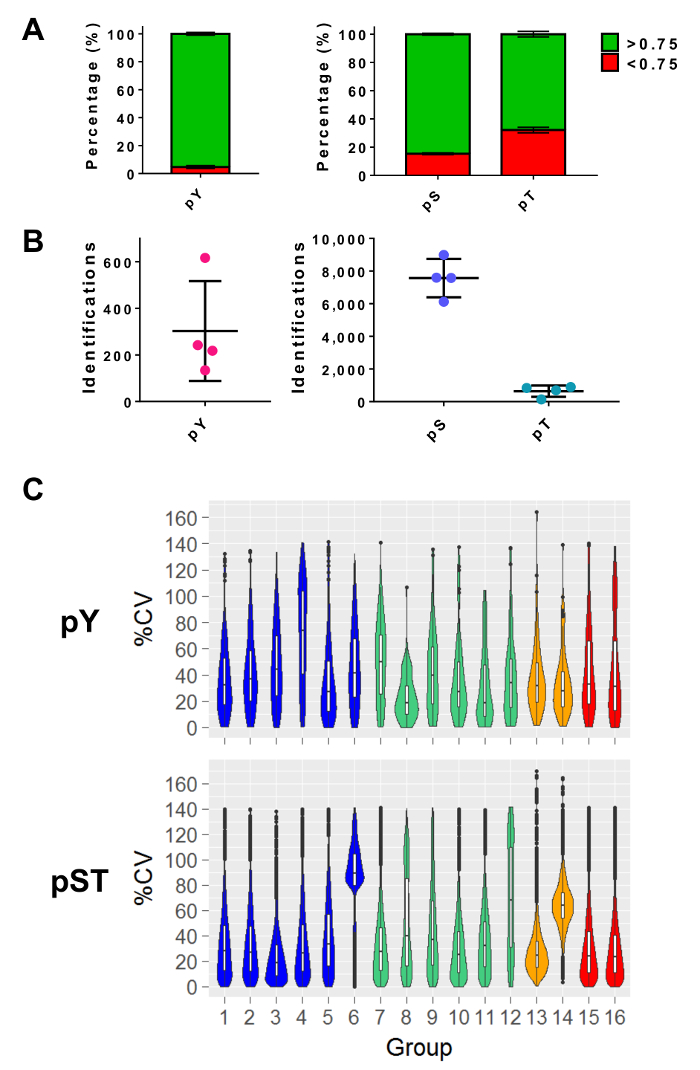
Figura 5: espera identificaciones phosphoresidue. (A) este panel muestra las probabilidades de localización de fosforilación de IDs de enriquecimiento pY (izquierda) y el enriquecimiento de pST (derecha). El porcentaje medio de IDs que cumplen con el límite de la probabilidad de > 0,75 es 93%, 75% y 52% en pY, pS y pT, respectivamente. (B) la media de número de ID con un > 0.75 probabilidad de localización es 300 para pY, 7.500 de pS y 640 para el pT. (C) este panel muestra diagramas de violín del coeficiente porcentual de variación (% CV) de la fosfotoproteida. Sólo se realizó una evaluación del % CV si se detectó un valor de intensidad de señal en cada repetición biológica o grupo por triplicado. Datos fue tomados de cuatro experimentos separados. Las barras de error en paneles A y B son las desviaciones estándar de pY 34 y 34 muestras de pST de 4 experimentos independientes. Haga clic aquí para ver una versión más grande de esta figura.
| Tampón de | Volumen | Composición | |
| Tampón de lisis de cloruro de guanidinio de 6 M | 50 mL | Cloruro de guanidinio de 6 M, 100 mM tris pH 8.5, fosfina tris (2-carboxietil) de 10 mM, 40 mM cloroacetamida, 2 mM ortovanadato de sodio, pirofosfato de sodio de 2,5 mM, glicerofosfato-β de 1 mM, 500 mg n-octil glucósido ultra puro con agua al volumen | |
| pirofosfato de sodio 100 mM | 50 mL | 2,23 g sodio pirofosfato decahidrato, agua ultrapura a volumen | |
| Glicerofosfato-β de 1M | 50 mL | 15,31 g β-glicerofosfato, agua ultrapura a volumen | |
| 5% de ácido trifluoroacético | 20 mL | Añadir 1 mL de 100% de ácido trifluoroacético en 19 mL de agua ultra pura | |
| 0.1% de ácido trifluoroacético | 250 mL | Añadir 5 mL 5% de ácido trifluoroacético a 245 mL de agua ultra pura | |
| tampón de elución de pY | 250 mL | 0,1% ácido trifluoroacético ácido, 40% acetonitrilo, agua ultrapura a volumen | |
| tampón de elución de pST | 250 mL | 0,1% ácido trifluoroacético ácido, 50% acetonitrilo, agua ultrapura a volumen | |
| Buffer de enlace de IP | 200 mL | 50 mM tris, pH 7.4, 50 mM cloruro sódico y agua ultrapura a volumen | |
| bicarbonato de amonio 25 mM, pH 7.5 | 10 mL | 19,7 mg se disuelven en 10 mL de agua ultra pura estéril, pH a 7.5 con 1 N de ácido clorhídrico (~ 10-15 μl/10 ml de solución), hacer fresco | |
| 1M amortiguador de fosfatos, pH 7 | 1.000 mL | fosfato de biácido del sodio 423 mL de 1 M, 577 mL 1 M sodio hidrógeno fosfato | |
| Tampón de equilibrado | 14 mL | 6,3 mL acetonitrilo, 280 μl 5% de ácido trifluoroacético, ácido láctico de 1740 μl, 5,68 mL de agua ultra pura | |
| Tampón de lavado | 20 mL | 9 mL acetonitrilo, 400 μL 5% de ácido trifluoroacético, 10,6 mL de agua ultra pura | |
| Solución de espectrometría de masas | 10 mL | 500 acetonitrilo μl, 200 μL 5% de ácido trifluoroacético, 9,3 mL de agua ultra pura | |
| Buffer A | 250 mL | fosfato monopotásico de 5 mM (pH 2.65), 30% de acetonitrilo, cloruro de potasio 5 mM, agua ultra-pure volumen | |
| Tampón B | 250 mL | fosfato monopotásico de 5 mM (pH 2.65), 30% de acetonitrilo, 350 mM cloruro de potasio, agua ultrapura a volumen | |
| 0.9% de hidróxido de amonio | 10 mL | Hidróxido de amonio 300 μL 29,42%, 9,7 mL de agua ultra pura | |
Tabla 1: soluciones y Buffers. Esta tabla muestra las composiciones de los buffers y soluciones utilizadas en el presente Protocolo.
| Configuración de LC-MS/MS | ||
| Parámetro | pY posición | pST ajuste |
| Muestra (μL) de carga | 5 | |
| Tasa de flujo de carga (μL/min) | 5 | |
| Gradiente de flujo (nL/min) | 300 | |
| Degradado lineal (porcentaje 0.16% ácido fórmico, 80% ACN en 0.2% de ácido fórmico) | 4 - 15% durante 5 min. | 4 - 15% por 30 min. |
| 15 - 50% durante 40 min. | 15 - 25% durante 40 min. | |
| 50 - 90% por 5 min. | 25 - 50% para 44 min | |
| 50 - 90% para los 11 min. | ||
| Resolución de escaneado completo | 120.000 | |
| Número de iones más intensos seleccionado | 20 | |
| Energía de la colisión relativa (%) (HCD) | 27 | |
| Exclusión dinámica (s) | 20 | |
Tabla 2: configuración de LC-MS. Este es un ejemplo de configuración de LC-MS en un experimento de phosphoproteomic típica escopeta. Las muestras fueron cargadas a una columna de la trampa. La trampa fue traída en línea con una columna analítica. Estos ajustes fueron optimizados para usar el sistema LC-MS enumerado en la tabla de materiales y reactivos. Estos ajustes deberían ajustarse a otros sistemas de LC-MS.
| Configuración de los parámetros de MaxQuant | ||
| Ajuste | Acción | |
| Parámetros específicos de grupo | ||
| Tipo | Tipo | Seleccione estándar |
| Multiplicidad | El valor 1 | |
| Modo de digestión | Enzima | Seleccione tripsina/P |
| Max. perdidas las divisiones | La posición 2 | |
| Modificaciones | Modificaciones variables | Añadir fosfo (orzuelo) |
| Cuantificación de etiqueta-libre | Cuantificación de etiqueta-libre | Seleccione LFQ |
| Cuenta de relación LFQ min. | El valor 1 | |
| LFQ rápido | Compruebe fuera de | |
| Varios | Volver a cuantificar | Compruebe fuera de |
| Parámetros globales | ||
| Secuencias de | Archivos FASTA | Fasta seleccione archivo descargado de UniProt |
| Modificaciones fijadas | Añadir Carbamidomethyl (C) | |
| Identificación de ADV. | Partido entre carreras | Compruebe fuera de |
| Ventana de tiempo de partido | En 5 min | |
| Ventana de tiempo de alineación | En 20 min | |
| Coinciden con características no identificadas | Compruebe fuera de | |
| Cuantificación de proteína | Cuenta de relación min. | El valor 1 |
| Ubicaciones de carpeta | Modificar en consecuencia | |
Tabla 3: configuración de software de análisis de MS. En MaxQuant, los parámetros específicos de grupo y globales en esta tabla fueron seleccionados o regulados. Todos los demás parámetros permanecieron en defecto. Estos experimentos se realizaron usando la versión 1.5.3.30. Los parámetros pueden variar de una versión a y de software a software.
Discusión
Antes de utilizar este protocolo para enriquecer para fosfotoproteida, una consideración cuidadosa del diseño experimental es fundamental. Utilizando repeticiones biológicas es un uso más rentable de los recursos de la espectrometría de masas que repeticiones técnicas. El número de repeticiones necesarias dependerá en parte la variabilidad de los datos. Un estudio reciente demostró que, mientras que el número de repeticiones más allá de los tres sólo marginalmente incrementa el número de identificación, el número de identificaciones significativas entre los grupos aumenta con más repeticiones10.
Debido a la menor abundancia de fosfoproteínas en la célula, cantidades suficientes de proteína a partir son necesarios para obtener una phosphoproteome global de muestras en modo de detección de cáncer de próstata. En estos experimentos, se utilizaron 5 mg de proteína. Aproximadamente cinco platos de 15 cm casi confluentes de células proporcionan suficiente proteína como entrada en este protocolo, aunque se trata de células dependientes de la línea. En cuanto a tejido del tumor, el rendimiento esperado de la proteína es alrededor 6-8% del peso del tejido. En el ajuste en vitro , una muestra de control positivo a tener en cuenta es la adición de vanadato de 1 mM por 30 min antes de la cosecha de las células. Vanadato, un competitivo fosfotirosil fosfatasa inhibidor, preservará la fosforilación de tirosina, aumentando así el número de pY péptido identificaciones29.
Digestión limpia es un paso clave para maximizar la identificación fosfopéptidos. Además de la prueba de tinción de Coomassie, el porcentaje de perdidas divisiones en los datos puede utilizarse para evaluar la eficiencia de la digestión (figura 2). Software de control de calidad está disponible que analiza divisiones perdidas y otras métricas de evaluación de calidad de datos de MS30. Mientras que la tripsina es que las proteasas más comunes, alternativas están disponibles5 a dirección cobertura las brechas en el proteoma donde péptidos trípticos óptima no pueden ser generado31. La configuración del software de análisis de MS entonces tendría que ser modificado en consecuencia para ajustar los cambios en las proteasas.
El protocolo emplea inmunoprecipitación (para el enriquecimiento de pY) así como dióxido de titanio (TiO2) enriquecer para fosfotoproteida. Alternativas para enriquecer para péptidos incluyen cromatografía de afinidad metálica inmovilizados (IMAC), otros óxidos de metal para la cromatografía de la afinidad del óxido de metal (MOAC) tales como hidróxido de aluminio y la captura de la afinidad de iones metálicos basados en polímeros (PolyMAC) 5,12. Estudios anteriores han demostrado que los métodos de enriquecimiento diferentes enriquecen para las diferentes poblaciones de fosfotoproteida32. Por ejemplo, IMAC enriquece más péptidos fosforilados multi mientras MOAC enriquece preferentemente de péptidos fosforilados mono33. Los Resultados de la representante de este protocolo refleja esta observación (Figura 4B). Una reciente publicación demostró que combinar la IMAC y MOAC utilizando un material híbrido potencialmente podría proporcionar una mayor cobertura de fosfopéptidos especies34. Así, este protocolo podría modificarse para utilizar otros métodos de enriquecimiento en paralelo para permitir un análisis más exhaustivo de la phosphoproteomic.
La suite de software de26 MaxQuant se utiliza para analizar los datos de MS en este protocolo, pero también hay aplicaciones comerciales35 para la cuantificación e identificación de fosfopéptidos. Para la identificación de fosfopéptidos, se aplica un corte de la probabilidad de la localización. Este filtro se realiza para seleccionar para fosfotoproteida con una alta confianza (es decir, mayor que 0.75) en la identificación de phosphoresidue10,28. En otras palabras, la probabilidad sumada de todos los otros residuos que potencialmente podría contener el phospho-grupo es menor que 0.25. Esta corte podría plantearse para aumentar el rigor de la selección de fosfopéptidos. Con relación al número de identificación, el número esperado de péptidos pY es cientos, mientras que el número esperado de péptidos de pST es alta miles. Estos valores reflejan la distribución de phosphoproteome previamente observado que alrededor del 2%, 12% y 86% de los phosphosites son pY, pT y pS, respectivamente28.
Si las medidas de enriquecimiento pY y pST se realizan en paralelo, los pasos de preparación de la muestra en el protocolo pueden realizarse en seis días. Por la vinculación con la poderosa herramienta de MS, protocolos enriquecimiento de fosfopéptidos como esta proporcionan un enfoque global de científicos recopilar datos para analizar la phosphoproteome en sus campos de investigación respectivos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a los miembros del laboratorio de Drake por sus consejos y entrada en el manuscrito. También agradecemos a los miembros de la biológica masa espectrometría instalación de Facultad Robert Wood Johnson Medical y Rutgers, la estatal Universidad de Nueva Jersey, para asesoramiento y realización de espectrometría de masas en nuestras muestras. Larry C. Cheng es apoyado por el Instituto Nacional de General médica Ciencias de los institutos nacionales de salud con el número de concesión GM008339 T32. Thomas G. Graeber es apoyado por el NCI/NIH (esporas en próstata cáncer P50 CA092131; P01 CA168585) y una sociedad americana del cáncer Research Scholar Award (RSG-12-257-01-TBE). Justin M. Drake es apoyado por el Departamento de defensa de la próstata cáncer de investigación programa de W81XWH-15-1-0236, próstata cáncer Fundación Premio al investigador joven, la Fundación de salud de Nueva Jersey y un premio precisión medicina iniciativa piloto de la Rutgers Instituto del cáncer de Nueva Jersey.
Materiales
| Name | Company | Catalog Number | Comments |
| Ultra-Low Temperature Freezer | Panasonic | MDF-U76V | |
| Freezer -20 °C | VWR | scpmf-2020 | |
| Swing rotor bucket | ThermoFisher Scientific | 75004377 | |
| Vacuum manifold | Restek | 26080 | |
| Lyophilizer | Labconco | 7420020 | |
| CentriVap Benchtop Vacuum Concentrator | Labconco | 7810010 | |
| End-over-end rotator | ThermoFisher Scientific | 415110Q | |
| Razor blade | Fisher Scientific | 620177 | |
| Amicon Ultra-15 Centrifugal Filter Units | Millipore Sigma | UFC901024 | |
| Glass culture tubes | Fisher Scientific | 14-961-26 | |
| Parafilm | Fisher Scientific | 13-374-12 | |
| 20G needle | BD | B305175 | |
| Kimwipes | Fisher Scientific | 06-666A | |
| Screw cap cryotube | ThermoFisher Scientific | 379189 | |
| Nunc 15 mL conical tubes | ThermoFisher Scientific | 12-565-268 | |
| Gel loading tips | Fisher Scientific | 02-707-181 | |
| Millipore 0.2 µm spin filter | Millipore Sigma | UFC30GVNB | |
| Low protein-binding Eppendorf tubes | Eppendorf | 22431081 | |
| anti-Phosphotyrosine, Agarose, Clone: 4G10 | Millipore Sigma | 16101 | |
| 27B10.4 antibody | Cytoskeleton | APY03-beads | |
| Peptide assay kit | Thermo Scientific | 23275 | Step 7 |
| TopTip | PolyLC Inc | TT200TIO.96 | Steps 5 and 9 |
| SCX columns (PolySULFOETHYL A) | PolyLC Inc | SPESE1203 | |
| 3 mL syringe | BD | 309657 | |
| Trifluoracetic Acid (TFA) | Fisher Scientific | PI-28904 | |
| Acetonitrile (ACN) | Fisher Scientific | A21-1 | |
| Lactic acid | Sigma-Aldrich | 69785-250ML | |
| Ammonium Hydroxide | Fisher Scientific | A669S-500 | |
| Potassium Phosphate Monobasic | Fisher Scientific | BP362-500 | |
| Potassium Chloride | Fisher Scientific | BP366-500 | |
| Calcium Chloride Dihydrate | Fisher Scientific | BP510-500 | |
| Tris Base | Fisher Scientific | BP152-5 | |
| Trypsin, TPCK Treated | Worthington Biochemicals | LS003740 | |
| Lysyl Endopeptidase | Wako Pure Chemical Industries, Ltd. | 125-05061 | |
| MonoTip | PolyLC Inc | TT200TIO.96 | Step 10 |
| ZipTip | MilliporeSigma | ZTC18S096 | Step 6 |
| nanoEase, MZ peptide BEH C18, 130A, 1.7 μm, 75 μm x 20 cm | Waters | 186008794 | Step 11: analytical column |
| Acclaim PepMap 100 C18 LC Columns | ThermoFisher Scientific | 164535 | Step 11: trap column |
| Ultimate 3000 RLSCnano System | Dionex | ULTIM3000RSLCNANO | Step 11 |
| Q Exactive HF | ThermoFisher Scientific | IQLAAEGAAPFALGMBFZ | Step 11 |
| MilliQ water | deionized water used to prepare all solutions and bufferes | ||
| Sonic Dismembrator | Fisher Scientific | FB-120 | sonicator |
| Polytron System PT | Kinematica AG | PT 10-35 GT | homogenizer |
Referencias
- Siegel, R. L., Miller, K. D., Jemal, A. Cancer statistics, 2018. CA: A Cancer Journal for Clinicians. 68 (1), 7-30 (2018).
- Paller, C. J., Antonarakis, E. S. Management of biochemically recurrent prostate cancer after local therapy: evolving standards of care and new directions. Clinical Advances in Hematology & Oncology. 11 (1), 14-23 (2013).
- Lowrance, W. T., Roth, B. J., Kirkby, E., Murad, M. H., Cookson, M. S. Castration-resistant prostate cancer: AUA guideline amendment 2015. The Journal of Urology. 195 (5), 1444-1452 (2016).
- Domon, B., Aebersold, R. Options and considerations when selecting a quantitative proteomics strategy. Nature Biotechnology. 28 (7), 710-721 (2010).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Bantscheff, M., Schirle, M., Sweetman, G., Rick, J., Kuster, B. Quantitative mass spectrometry in proteomics: a critical review. Analytical and Bioanalytical Chemistry. 389 (4), 1017-1031 (2007).
- Bantscheff, M., Lemeer, S., Savitski, M. M., Kuster, B. Quantitative mass spectrometry in proteomics: critical review update from 2007 to the present. Analytical and Bioanalytical Chemistry. 404 (4), 939-965 (2012).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Rubbi, L., et al. Global phosphoproteomics reveals crosstalk between Bcr-Abl and negative feedback mechanisms controlling Src signaling. Science Signaling. 4 (166), ra18 (2011).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045 (2018).
- Rush, J., et al. Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nature Biotechnology. 23 (1), 94-101 (2005).
- Fila, J., Honys, D. Enrichment techniques employed in phosphoproteomics. Amino Acids. 43 (3), 1025-1047 (2012).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Drake, J. M., et al. Phosphoproteome integration reveals patient-specific networks in prostate cancer. Cell. 166 (4), 1041-1054 (2016).
- Lue, H. W., et al. Metabolic reprogramming ensures cancer cell survival despite oncogenic signaling blockade. Genes & Development. 31 (20), 2067-2084 (2017).
- Francavilla, C., et al. Phosphoproteomics of primary cells reveals druggable kinase signatures in ovarian cancer. Cell Reports. 18 (13), 3242-3256 (2017).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Skaggs, B. J., et al. Phosphorylation of the ATP-binding loop directs oncogenicity of drug-resistant BCR-ABL mutants. Proceedings of the National Academy of Sciences of the United States of America. 103 (51), 19466-19471 (2006).
- Zimman, A., et al. Activation of aortic endothelial cells by oxidized phospholipids: a phosphoproteomic analysis. Journal of Proteome Research. 9 (6), 2812-2824 (2010).
- Zimman, A., Berliner, J. A., Graeber, T. G. Phosphoproteomic analysis of aortic endothelial cells activated by oxidized phospholipids. Methods in Molecular Biology. , 53-69 (2013).
- Humphrey, S. J., Azimifar, S. B., Mann, M. High-throughput phosphoproteomics reveals in vivo insulin signaling dynamics. Nature Biotechnology. 33 (9), 990-995 (2015).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Meyer, T. S., Lamberts, B. L. Use of coomassie brilliant blue R250 for the electrophoresis of microgram quantities of parotid saliva proteins on acrylamide-gel strips. Biochimica el Biophysica Acta. 107 (1), 144-145 (1965).
- Bergstrom Lind, S., et al. Immunoaffinity enrichments followed by mass spectrometric detection for studying global protein tyrosine phosphorylation. Journal of Proteome Research. 7 (7), 2897-2910 (2008).
- Pinkse, M. W., Uitto, P. M., Hilhorst, M. J., Ooms, B., Heck, A. J. Selective isolation at the femtomole level of phosphopeptides from proteolytic digests using 2D-NanoLC-ESI-MS/MS and titanium oxide precolumns. Analytical Chemistry. 76 (14), 3935-3943 (2004).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Cox, J., et al. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Olsen, J. V., et al. Global, in vivo, and site-specific phosphorylation dynamics in signaling networks. Cell. 127 (3), 635-648 (2006).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. The Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Bielow, C., Mastrobuoni, G., Kempa, S. Proteomics quality control: quality control software for MaxQuant results. Journal of Proteome Research. 15 (3), 777-787 (2016).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Bodenmiller, B., Mueller, L. N., Mueller, M., Domon, B., Aebersold, R. Reproducible isolation of distinct, overlapping segments of the phosphoproteome. Nature Methods. 4 (3), 231-237 (2007).
- Leitner, A., Sturm, M., Lindner, W. Tools for analyzing the phosphoproteome and other phosphorylated biomolecules: a review. Analytica Chimica Acta. 703 (1), 19-30 (2011).
- Yang, D. S., et al. Design and synthesis of an immobilized metal affinity chromatography and metal oxide affinity chromatography hybrid material for improved phosphopeptide enrichment. Journal of Chromatography A. 1505, 56-62 (2017).
- Al Shweiki, M. R., et al. Assessment of label-free quantification in discovery proteomics and impact of technological factors and natural variability of protein abundance. Journal of Proteome Research. 16 (4), 1410-1424 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados