Method Article
Métodos de transferencia de energía por resonancia de una sola molécula para la investigación en tiempo real de la resolución de Holliday Junction por GEN1
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí se presenta un protocolo para la realización de la transferencia de energía de resonancia de una sola molécula de la resonancia de la unidad para estudiar la resolución hj. La excitación alterna de dos colores se utiliza para determinar las constantes de disociación. El smFRET de lapso de tiempo de un solo color se aplica en ensayos de escisión en tiempo real para obtener la distribución del tiempo de permanencia antes de la resolución de HJ.
Resumen
Los métodos a granel miden el comportamiento del conjunto de moléculas, en el que las tasas de reacción individuales de los pasos subyacentes se promedian en toda la población. La transferencia de energía de resonancia de una sola molécula (smFRET) proporciona un registro de los cambios de conformación que tienen lugar las moléculas individuales en tiempo real. Por lo tanto, smFRET es potente en la medición de cambios estructurales en la enzima o sustrato durante la unión y la catálisis. Este trabajo presenta un protocolo para la toma de imágenes de una sola molécula de la interacción de una unión Holliday de cuatro vías (HJ) y una endonucleasa I (GEN1), una enzima de recombinación homóloga citosolílica. También se presentan los protocolos experimentales smFRET de excitación alterna de un solo color y dos colores (ALEX) para seguir la resolución del HJ por GEN1 en tiempo real. La cinética de la dimitación GEN1 se determina en el HJ, que se ha sugerido para desempeñar un papel clave en la resolución del HJ y ha permanecido esquiva hasta ahora. Las técnicas descritas aquí se pueden aplicar ampliamente para obtener valiosas perspectivas mecanicistas de muchos sistemas de ADN enzimático.
Introducción
Los métodos de una sola molécula basados en la detección de fluorescencia proporcionan altas relaciones señal-ruido1. FRET es una técnica espectroscópica que puede medir distancias en el rango de 1-10 nm, haciendo esta técnica como una regla molecular para medir distancias en el rango de nanómetros2,3. El espectro de absorción del aceptador tiene una superposición espectral parcial con el espectro de emisión del donante en el extremo de longitud de onda más corto. FRET está mediada por la transferencia de energía sin radiación entre un donante y un par de aceptadores, mientras que la eficiencia de la transferencia de energía depende de la distancia y la orientación del aceptador4.
Se han implementado varios enfoques para minimizar el fondo y mejorar la eficiencia de detección de la señal de fluorescencia5,6. Un enfoque es la microscopía confocal, en la que un agujero restringe el punto de excitación a un tamaño por debajo del límite de difracción7. Otro enfoque es la fluorescencia de reflexión interna total (TIRF), que es una técnica de iluminación de campo amplio en la que la luz se dirige fuera del eje por encima de un ángulo crítico8. La luz se refleja totalmente internamente en la interfaz entre el vidrio y la solución acuosa, generando una onda evanescente que sólo ilumina los fluoróforos unidos a la superficie del vidrio y evita que el fondo de los fluoróforos en el resto de la solución.
En la microscopía confocal, las moléculas pueden ser libremente difuminadas o inmovilizadas en la superficie. La resolución temporal alcanzada puede ser dentro de microsegundos a varios milisegundos9. La detección confocal para una sola molécula se realiza mediante diodo de avalancha de un solo fotón (SPAD) y escaneo punto por punto de la región de interés10. En TIRF, una serie temporal de unos pocos cientos de moléculas inmovilizadas en la superficie es registrada por un detector acoplado de carga bidimensional (CCD) sensible a la posición. El CCD amplifica la señal de fluorescencia ya sea por pantalla de fósforo intensificada y placa de microcanal o multiplicación en chip de fotoelectrones (EMCCD). La resolución temporal depende de la velocidad de lectura y la eficiencia cuántica del CCD y, por lo general, del orden de unas pocas decenas de milisegundos6.
HJ es un intermediario central en reparación y recombinación de ADN11,12,13,14. HJ tiene dos hilos de cruce continuos y dos que se conectan entre las hebras continuas sin intersecar entre sí. HJ existe en solución como conformadores apilados por X, que se someten a la isomerización continua por las hebras continuas que se convierten en cruce y las hebras de cruce se vuelven continuas en el otro conformador15. La preferencia isómero del HJ depende de la secuencia central y del entorno iónico y ha sido ampliamente estudiada por FRET16,17,18,19.
GEN120 es una proteína monomérica en la solución21 y requiere dimerización para aparar el HJ, permitiendo así una separación adecuada de las hebras recombinadas22,23. La preferencia conformadora de apilamiento del HJ influye en el resultado de la recombinación genética estableciendo la orientación de la resolución por el HJ resolvases24. Comprender cómo GEN1 une el HJ, coordina las dos incisiones y asegura que su resolución completa ha sido todo bajo estudio intensivo21,22,23,25,26 ,27,28,29,30.
En este estudio, se utiliza una configuración TIRF basada en objetivos como se describió anteriormente31. La excitación alterna de dos colores (ALEX) se aplica para determinar los cambios de conformación tras la interacción de GEN1 con el fluoróforo con la etiqueta HJ. ALEX produce histogramas 2D basados en dos parámetros ratiométricos de eficiencia FRET E, que depende de la distancia donante-aceptador, y el parámetro S de estequiometría, que mide la estequiometría donante-aceptador32. ALEX permite la clasificación de especies fluorescentes basadas en las estequiometrías de los fluoróforos, incluyendo subpoblaciones sólo de donantes, sólo para pagos y mixtas. ALEX puede extender el uso de FRET a toda la gama y puede detectar diferencias en el brillo y el oligomerización de los fluoróforos, así como monitorear las interacciones entre macromoléculas y ligandos33.
Se constata que GEN1 logra resolver constantemente el HJ durante la vida útil del complejo GEN1-HJ. Los cambios de conformación dependientes del tiempo se derivan de los rastros de tiempo de moléculas individuales, mientras que los histogramas representan la distribución de las poblaciones subyacentes. Utilizando EL FRET de un solo color de lapso de tiempo, se demuestran velocidades de ajuste rápidas y bajas de velocidad lenta para el dimer GEN1, que aumentan la afinidad del dimer GEN1 ensamblado en el primer producto de incisión.
Protocolo
1. Preparación de cubiertas funcionales en superficie
-
Limpieza
- Coloque cinco tapas (24 mm x 60 mm) en etanol dentro de un frasco de Coplin. Sonicar en etanol entonces en 1 M de hidróxido de potasio durante 30 min durante 3x. Lavar en acetona 3x y luego decantar.
-
Silanización
- Preparar una solución de 2.8% 3-aminopropyltriethoxysilane (APTES) en acetona. Selle la botella APTES con una película de parafina y guárdela a 4oC.
NOTA: Use gafas de seguridad y trabaje bajo una campana de humos. El recipiente de la solución de silano debe estar completamente seco y enjuagado con acetona inmediatamente antes y después de verter la solución de silano en el frasco. - Vierta 70 ml de la solución APTES del 2,8% en el frasco de Coplin que contiene los cubreobjetos. Agitar el frasco durante 4 minutos en un agitador orbital.
- Deje reposar el frasco en el banco durante 5 minutos, sonicar durante 1 min, y finalmente mantener el frasco en el banco durante otros 10 minutos para que el silano reaccione con los grupos hidroxilo en la superficie de vidrio.
- Atemple la reacción mediante la adición de 1 L de agua desionizada vertiendo agua directamente en el frasco para un intercambio rápido de disolventes. Enjuague los portaobjetos 3veces en agua agitando lateralmente el frasco sobre una superficie plana.
- Saque los labios de las cubiertas del frasco y colóquelos en una bandeja de papel de aluminio. Hornee los labios de las cubiertas en un horno a 110 oC durante 30 minutos para secar los labios de las cubiertas y curar el silano. Deje la bandeja en el banco para que los cubreobjetos se enfríen a temperatura ambiente.
- Preparar una solución de 2.8% 3-aminopropyltriethoxysilane (APTES) en acetona. Selle la botella APTES con una película de parafina y guárdela a 4oC.
-
PEGylation
- Caliente el PEG y el PEG biotinilados, almacenados a -20 oC a temperatura ambiente (RT) para evitar la condensación de humedad al abrir el recipiente.
- Coloque cinco tapas con la superficie silanizada hacia arriba en una caja. Coloque dos resbalones de vidrio de cubierta (22 mm x 22 mm) como espaciadores a lo largo de los bordes de las cubiertas silanizadas.
- Una vez calentadas, haga las soluciones de PEG y PEG biotiniladas en una proporción de 1:100 en 1 ml de solución fresca de bicarbonato sódico de 0,1 M añadiendo 1,5 mg de PEG biotinilado y 150 mg de PEG en un tubo de 1,5 ml.
- Vortex el tubo para disolver PEG y girar hacia abajo para eliminar las burbujas de aire.
NOTA: En el futuro de este paso, sea rápido, porque PEG se hidroliza en solución dentro de una escala de tiempo de min. - Aplicar rápidamente 100 l de la solución PEG en cada cubreportada. Tome otro cubreobjetos horneados y coloque su superficie silanizada superior boca abajo en la parte superior de la cubierta con la solución PEG, por lo tanto, formando un sándwich de vidrio-solución de vidrio en el que los cubres no silanizados de 22 mm x 22 mm permiten que los dos cubres funcionalizados se separen fácilmente.
- Incubar los labios de las cubiertas durante la noche (16 h) en la oscuridad y en RT. Una vez completada la incubación, desmonte los labios de las cubiertas y, a continuación, enjuague 10 veces con agua desionizada lavándolo desde el costado con una botella de chorro.
- Seque los labios de las cubiertas bajo un flujo de nitrógeno seco. Almacene los cubreobjetos secos al vacío.
NOTA: Las diapositivas se pueden utilizar durante 1 mes sin degradación de la calidad.
2. Preparación de la célula de flujo
-
Célula de flujo monocanal
- Taladre dos orificios con 1,22 mm de diámetro en la parte media de una corredera de cuarzo (50 mm x 20 mm) con los centros situados 37 mm de separación y 6,5 mm desde el borde de la corredera(Figura 1A).
- Corte un canal de 41 mm x 2,25 mm en una pieza de 50 mm x 20 mm de una hoja de doble adhesivo utilizando una cortadora electrónica.
- Retire el lado de plástico de la cubierta protectora y alinee los bordes de la pieza con los bordes de la corredera de cuarzo. Retire las burbujas de aire atrapadas presionando suavemente con un par de pinzas de politetrafluoroetileno.
- Retire el lado del papel de la pieza adhesiva. Monte la pieza en la superficie funcionalizada del cubreobjetos.
- Corte el tubo de polietileno (I.D. 1,22 mm) en una longitud de 11 cm para la entrada y de 25 cm para la salida. Inserte el tubo en los orificios previamente perforados como entrada y salida para la celda de flujo.
- Utilice pegamento epoxi de 5 min para sellar alrededor de los bordes de la interfaz de tapa de cuarzo y alrededor de los tubos para la entrada y salida.
- Utilice la celda de flujo inmediatamente una vez que se seque o almacene bajo vacío seco para su uso posterior.
- Disolver la avidina en PBS a una concentración de 0,03 mg/ml. Filtrar a través del filtro de jeringa de 0,2 m.
- Flujo de avidina en la célula de flujo utilizando una jeringa de 1 ml. Use otra jeringa llena de tampón para lavar el exceso de avidina. Tenga cuidado de no introducir burbujas de aire mientras intercambia las jeringas.
-
Célula de flujo de múltiples canales
- Taladre seis orificios con un diámetro de 1,22 mm en cada uno de los lados largos de una corredera de cuarzo (76 mm x 25 mm)(Figura 1B). Haga los agujeros 4,5 mm desde el borde de la corredera y 9,3 mm de distancia. Asegúrese de que la distancia entre los centros de cada par de orificios sea de 15 mm.
- Corte seis canales (20 mm x 2,25 mm) en una pieza de cinta adhesiva doble de 76 mm x 25 mm utilizando la cortadora electrónica.
- Retire el lado de plástico de la cubierta protectora y alinee los bordes de la pieza adhesiva con los bordes de la corredera de cuarzo. Retire las burbujas de aire atrapadas presionando suavemente con un par de pinzas de politetrafluoroetileno.
- Retire el lado del papel de la pieza adhesiva y colóquela en la superficie funcionalizada del cubreobjetos.
NOTA: A veces, despegar el lado del papel y fijarlo a la corredera de cuarzo funciona bien en la celda de flujo multicanal. - Corte los tubos de entrada (11 cm) y los tubos de salida (25 cm) para los seis canales. Prepare la celda de flujo como se describe en los pasos 2.1.6–2.1.9.
- Conecte la salida del primer canal a la bomba. Coloque la entrada en un tubo de 0,5 ml con SSS.
NOTA: La longitud del tubo de entrada se elige para maximizar el número de eventos en los experimentos de escisión realizados bajo flujo continuo mediante la sincronización del tiempo de entrada de enzimas en la célula de flujo y el inicio de la imagen disminuyendo así el fotoblanqueo prematuro de la fluoróforos. - Muévase a un nuevo canal desconectando la salida del canal utilizado. Cierre la toma de corriente con un tapón hecho de una aguja de jeringa sellada con pegamento en la pieza de plástico. Cierre la entrada del canal utilizado.
3. Preparación del sistema de barrido de oxígeno (OSS)
- Disolver 0,2 g de ácido carboxílico de estado triple (triplete que minimiza el parpadeo de los fluoróforos) en 800 ml de metanol.
- Añadir 6 ml de H2O desionizado y añadir 1 N NaOH en sentido de gota hasta que se disuelva. Filtrar a través de un filtro de jeringa, hacer en 1 ml alícuotas, y almacenar a -80 oC. La concentración de la población es de 100 m.
- Preparar una solución fresca de ácido 3,4-dihroxybenzoic (PCA) disolviendo 61 mg de polvo de PCA en 4 ml de ddH2O. La concentración de la población es de 100 nM.
- Añadir 58 l de 10 N NaOH en sentido de gota, asegurándose de vórtice después de cada caída hasta que el PCA se disuelva completamente (pH n.o 9).
- Disolver 5,3 mg de protocateuato 3,4-dioxigenasa (3,4-PCD) en 7 ml de búfer de almacenamiento PCD (Tabla 1). 3,4-PCD elimina el oxígeno de los tampones de unión/escisión catalizando la oxidación del ácido protocatechuico34.
- Divida la solución PCD en alícuotas de 1 ml. La concentración de stock es de 1 M. Congele las alícuotas en nitrógeno líquido y almacene a -80 oC para almacenamiento a largo plazo o a -20 oC para almacenamiento a corto plazo.
- Prepare un búfer de enlace nuevo (Tabla 1). Sustituya 2 mM CaCl2 por 2 mM MgCl2 para experimentos de escisión smFRET.
- Prepare 1 ml del búfer de imágenes (Tabla 1). Mantenga el tampón de imágenes en el hielo hasta que se introduzca en la célula de flujo para mantener la actividad del sistema de barrido de oxígeno.
4. Preparación de HJs etiquetados fluorescentemente
- Reconstituir los oligos liofilizados(Tabla 2) en el buffer de Tris-EDTA (Tabla 1) a una concentración de 100 M.
- Preparar la unión sintética mezclando porciones equimolares de 3 l de cada uno de los oligos X0 enumerados en la Tabla 1.
- Anneal calentando a 95oC durante 5 min seguido de un enfriamiento lento a RT a una velocidad de 1 oC/min. Utilice un bloque de calor o un termociclador PCR para lograr la velocidad de enfriamiento deseada.
- Cargue la mezcla en 8 cm x 8 cm de gel de poliacrilamida 10% Tris-borate-EDTA. Aplicar 100 V y ejecutar el gel durante 2 h. Las bandas se ven claramente a los ojos, y su color es púrpura.
- Excise la banda del sustrato recocido con una cuchilla limpia. Transfiera la pieza de gel a un tubo autoclave de 1,5 ml.
- Aplastar la pieza de gel dentro del tubo con un émbolo limpio y luego añadir 100 l de tampón TE100(Tabla 1).
- Extraer el HJ agitando el tubo a 20oC a 1.500 rpm en un termomezclador durante 2 h o incubar durante la noche a 4oC.
- Realizar precipitación de etanol en la solución que contiene el sustrato35.
- Resuspender el sustrato en 20 s de tampón TE100 (Tabla 1). La concentración final es de 1–3 m. Aliquot 2 l en cada tubo y almacenar a -20 oC.
5. Expresión de proteínas y purificación de GEN1
- Construir el plásmido para la expresión de GEN1 humano truncado1,2,3,4,5 con hexa histidina-etiqueta en el C-terminus20 por PCR del vector de entrada.
NOTA: El etiquetado N-terminal daría lugar a la inactivación de GEN1. La cola C no estructurada hace que la purificación de toda la longitud GEN1 sea significativamente más difícil. Además, se informó de que la longitud completa de GEN1 presentaba menos actividad que la versión23truncada. - Transforme el vector de expresión en e. coli BL21-CodonPlus (DE3)-RIPL strain.
- Inocular las células transformadas en dos matraces de 6 L cada uno que contienen 2 L de medios de caldo de Luria a 37 oC con temblores a 180 rpm hasta que se alcance una OD600 de 0,8.
- Enfriar el cultivo a 16 oC e inducir la expresión GEN1 con 0,1 mM isopropyl--d-thiogalactopyranoside (IPTG) durante 48 h.
- Cosecha las células girando hacia abajo a 4oC a 1000 x g en una centrífuga. Cada litro de cultivo produce 5–6 g del pellet.
- Deseche el sobrenadante y resuspenda las células peletizadas en el tampón de lisis(Tabla 1) usando 4 ml/g de células.
- Realice la lisis celular usando un disruptor celular a 30 kPsi y luego gire hacia abajo a 10,000 x g durante 1 h a 4 oC. Recoger el sobrenadante y filtrarlo en hielo usando filtros de 0,45 m.
- Realice la purificación de proteínas utilizando FPLC pasando el filtrado a través de una columna Ni-NTA de 5 ml a un caudal de 2,5 ml/min utilizando el tampón A (Tabla 1).
- Lavar con volúmenes de 15 columnas (CV). Elute con un gradiente lineal de Tampón A y 500 mM de Imidazol sobre 20 CV en fracciones de 5 ml. GEN1 elutes de la columna a alrededor de 100 mM de Imidazol.
- Pipetear alas de 10 l de las fracciones recogidas, añadir el mismo volumen de 2x SDS de colorante de carga a cada alícuota. Desnaturalizar las muestras calentando a 90 oC durante 5 min, enfriar y espinar las muestras.
- Cargue las muestras en un gel Bis-Tris al 10%. Ejecutar el gel durante 30–45 min a 200 V. Mancha usando Coomassie Azul Brillante, luego destaína. Recoja las fracciones que contienen GEN1 purificado.
- Reducir la concentración de sal de las fracciones combinadas a 100 mM mediante dilución utilizando el tampón C (Tabla 1).
- Pasar la proteína baja de sal a través de una columna de heparina de 5 ml a un caudal de 3 ml/min utilizando el tampón B (Tabla 1).
- Lavar con 10 CV. Elute usando un gradiente de 20 CV con Buffer B y 1 M NaCl. Recoger fracciones de 5 ml en las que GEN1 eluja alrededor de 360 mM NaCl.
- Compruebe las fracciones eluted para las fracciones GEN1 purificadas como se describe en el paso 5.8. Combine esas fracciones y diluya a 100 mM de NaCl usando el Tampón C.
- Cargue la proteína sal baja en una columna de intercambio catiónico a un caudal de 1 ml/min utilizando el búfer B.
- Eluir por un gradiente de 40 CV usando Buffer B y 1 M NaCl. Recoger fracciones de 1,7 ml en las que GEN1 eluja alrededor de 300 mM de NaCl.
- Compruebe la pureza de GEN1 en las fracciones eluted como se describe en el paso 5.8.
- Combine las fracciones más puras y el dializ a 4 oC contra el búfer de almacenamiento(Tabla 1). Realice al menos un intercambio del búfer durante la diálisis.
- Mida la concentración de proteínas de 0,5–1 mg/ml. Aliquot la proteína dializada en volúmenes de 10–15 l en tubos pequeños, congelación de flash en nitrógeno líquido, y almacenar a -80 oC.
6. Experimentos FRET de una sola molécula
NOTA: Los experimentos smFRET se realizan en una configuración TIRF basada en objetivos personalizados(Figura 1C)descrita anteriormente31.
- Experimentos FRET de un solo color
- Aplique una gota de aceite de inmersión sobre el objetivo TIRF 100x. Establezca el EMCCD en una ganancia adecuada para optimizar la señal en segundo plano y evitar la saturación.
NOTA: No mire directamente en el rayo láser y use gafas protectoras al alinear el láser. - Coloque la celda de flujo cuidadosamente en el soporte de la muestra. Elevar gradualmente el objetivo mediante un ajuste grueso hasta que el aceite toque el cubreobjetos.
- Encienda el láser verde (532 nm). Cambie al modo de ajuste fino del objetivo. Dirija la emisión al puerto de la cámara para observar la imagen en el monitor.
- Ajuste la altura del objetivo hasta que la superficie funcionalizada del cubreobjetos se enfoque y se pueda observar en el monitor.
NOTA: La adquisición de imágenes por parte de EMCCD desencadena la excitación láser a través del filtro acostoóptico ajustable (AOTF) para evitar el fotoblanqueo de muestras cuando no se están adquiriendo imágenes. - Compruebe que el fondo de la superficie funcionalizada de los cubreobjetos no exceda de pocos puntos antes de fluir en el HJ con etiqueta fluorescente.
- Diluir el sustrato de material aproximadamente 1000 veces en tampón TE100(Tabla 1) a una concentración final de 1–5 nM. Pipetear de 0,2 a 0,5 ml del sustrato diluido en 120 ml del tampón de imágenes con SSS en un tubo de 0,5 ml.
- Conecte la salida de la celda de flujo a la bomba de la jeringa. Inserte el tubo de entrada de la célula de flujo en un tubo de 1,5 ml y accione la bomba de la jeringa a un caudal de 30 a 50 ml/min para extraer la solución del tubo.
- Compruebe con frecuencia la superficie en busca de una buena cobertura (100–300 de sustrato homogéneo y bien espaciado) mediante imágenes breves con el láser verde.
- Si la cobertura de la superficie todavía no es suficiente, espere unos minutos a que el HJ etiquetado fluorescentemente de la solución se asiente en la superficie o repita el paso de flujo.
- Flujo de otros 120 l de tampón de imagen(Tabla 1) a 30–50 l/min para lavar el HJ con etiqueta fluorescente sin consolidar. Luego deje que la célula de flujo se sitúe durante 5 minutos para permitir que el OSS agote el oxígeno disuelto. El fotoblanqueo de los fluoróforos debe ser mínimo al inicio de la toma de imágenes.
- Establezca el tiempo de exposición (60 ms), el tiempo de ciclo se establecerá automáticamente por software en función de la velocidad de transferencia de datos (104 ms) y especifique el número deseado de ciclos o fotogramas (400 euros). La emisión del donante (Cy3) y del aceptador (Alexa Fluor 647) se divide en dos canales de color por un dispositivo divisor de imagen.
- Encuentra un área adecuada en la superficie, enfoca la imagen ajustando la altura del objetivo y graba y guarda la película en formato TIFF de 16 bits.
- Muévase a una nueva área.
NOTA: Muévase siempre en una sola dirección (es decir, de la salida a la entrada) para evitar la toma de imágenes de la misma área dos veces. - Prepare 1, 2, 5, 10, 25, 50, 75 y 100 nM GEN1 en 120 ml de tampón de imágenes de uno en uno. Flujo de la solución a un caudal de 30-50 l/min.
NOTA: Si la medición requerida se realiza en estado estacionario como en la unión de HJ por GEN1 o la isomerización del HJ libre, espere 3-5 minutos después de que el flujo se detenga para grabar la película. Adquirir de tres a cuatro películas de nuevas áreas para cada concentración gen1. - Si la medición se realiza bajo flujo continuo como en el escote de HJ por GEN1, entonces comience a registrar 5-10 s antes de la entrada de GEN1 en la celda de flujo. Repita la medición moviéndose a un nuevo canal en la celda de flujo de seis canales.
- Al final, utilice una diapositiva de cordón fluorescente fija para asignar las partículas del donante y del aceptador entre sí en el dispositivo de división de imágenes.
- Añadir 0,2 ml de perlas fluorescentes de 1 m de diámetro en 500 ml de tris de 1 M (pH a 8,0) para permitir que las perlas se peguen a la superficie.
- Corte un cuadrado (18 mm x 18 mm) dentro de una pieza de 22 mm x 22 mm de un sello adhesivo de doble cara. Despegue y pegue la pieza en el centro de un portaobjetos de cuarzo de 76 mm x 25 mm.
- Colocar 50 sl de la solución de perlas diluidas y dejar actuar durante 5-10 minutos para asentarse. Coloque un cubreobjetos de 22 mm x 22 mm en la parte superior de la pieza cuadrada. Solución de perlas de exceso de secado con tejido, luego sellar la cámara por pegamento epoxi.
- Adquiera 100 fotogramas de la diapositiva de cuentas a un tiempo de exposición de 60 ms.
ADVERTENCIA: Reduzca la potencia del láser y la ganancia de EMCCD al mínimo para evitar la saturación del detector. - Instale el paquete de software (por ejemplo, TwoTones) y abra las películas en el mismo como se indica en el manual de usuario36. Seleccione las posiciones de las cuentas individuales en los canales del donante y del aceptador. Genere una matriz de transformación como se describe en el manual.
NOTA: Este software utiliza la matriz de transformación para que coincida con las posiciones de las partículas en los canales del donante y del aceptador y corregir cualquier desalineación leve en el dispositivo de división de imágenes. - Vaya a Archivo, pulse Cargar película, seleccione el archivo de película y pulse Abrir. En el menú de archivos, presione Cargar TFORM y seleccione la matriz de transformación generada a partir de la diapositiva de cuentas. Ajuste el umbral para los canales del donante y del aceptador hasta que no se incluyan falsos positivos.
- En el menú Filtro de canal, elija la opción D&&A para seleccionar las partículas etiquetadas con el donante y el aceptador. Compruebe el campo Límite de vecino más cercano para excluir las moléculas que están muy cerca unas de otras. Compruebe la elíptica máxima para excluir moléculas muy excéntricas y compruebe los límites de anchura para excluir moléculas muy anchas o muy estrechas.
- Escriba plotHistCW como se indica en el manual De dos tonos para construir histogramas.
NOTA: El programa calcula la eficiencia "aparente" de FRET dividiendo la emisión del aceptador entre las emisiones totales del donante y del aceptador. Twotones utiliza 100 intervalos para evitar la distribución de los estados de las moléculas contra la eficiencia fret. - Escriba plotTimetraceCW como se indica en el manual de dos tonos para generar las trazas de tiempo para cada molécula.
NOTA: VbFRET37 puede analizar más a fondo los seguimientos de tiempo para identificar diferentes estados de FRET, sus respectivos tiempos de permanencia y tasas de transición entre diferentes estados.
- Aplique una gota de aceite de inmersión sobre el objetivo TIRF 100x. Establezca el EMCCD en una ganancia adecuada para optimizar la señal en segundo plano y evitar la saturación.
- Experimentos de excitación alterna de dos colores FRET (ALEX)
- Grabar una película compuesta de fotogramas consecutivos de emisiones de donantes y aceptadores por excitaciones directas con los láseres verdes y rojos, respectivamente, cada uno de 80 ms de duración.
- Abre las películas ALEX adquiridas en Twotones. Establezca el umbral de detección adecuado en 300 euros para los tres canales: emisión del donante debido a la excitación del donante (DexDem); emisión del aceptador debido a la excitación del donante (DexAem); emisión de aceptadores debido a excitación directa (AexAem).
- Aplique el filtro de canal DexDem&&DexAem&&AexAem para seleccionar las partículas que tienen tanto donante como aceptador. Enlace las partículas de 200 a 300 en los tres canales.
- Utilice el código plotHistALEX MATLAB para generar histogramas ALEX. Ajuste diferentes picos en los histogramas a funciones gaussianas y determine el porcentaje de cada población del área bajo la curva utilizando el software Origin38.
NOTA: Los picos del ensayo vinculante corresponden al complejo GEN1-HJ enlazado, mientras que en el HJ libre, los picos representan los isómeros de intercambio. - Utilice el código plotTimetraceALEX MATLAB para generar un seguimiento temporal para cada molécula que muestre la emisión de los donantes por excitación directa y las emisiones de los aceptadores debido a la FRET y la excitación directa.
NOTA: ALEX traza el tiempo de forma independiente para mostrar las emisiones tanto del donante como del aceptador, pero con una resolución temporal más baja que el FRET de un solo color. Al igual que el FRET de un solo color, los rastros de tiempo ALEX pueden ser analizados por vbFRET para identificar diferentes estados de FRET y sus respectivos tiempos de permanencia. - Determinar la constante de disociación ajustando los porcentajes de la población enlazada frente a la concentración de GEN1 a una función hiperbólica.
- TRANSTES de un solo color de lapso de tiempo
- Ajuste el tiempo de exposición del láser verde a 60 ms y el tiempo de ciclo a 624 ms o inferior, dependiendo de la velocidad de la dinámica observada.
- Establezca el caudal en 110 l/min en un canal de la celda de flujo de seis canales. Inicie la grabación brevemente antes de la entrada de GEN1 dentro de la celda de flujo.
NOTA: El flujo continuo conduce a un fotoblanqueo rápido de los fluoróforos, por lo que la sincronización del inicio de la imagen y la entrada de proteínas maximiza el número de eventos capturados. La lectura óptima de la bomba de jeringa depende del volumen muerto y del tubo exacto utilizado para ensamblar la celda de flujo; en nuestro caso es de 25 ol. - Adquiera una película de 125 fotogramas para un tiempo total de adquisición de 78 s. Al final de la grabación, exponga la muestra al láser rojo durante 50 fotogramas cada uno con un tiempo de exposición de 25 ms para sondear el aceptador.
NOTA: Este método prolonga la ventana de observación en el experimento de escisión a través de la disminución del número de ciclos de excitación. Los parámetros cinéticos como kon y koff de la dimerización se derivan ajustando la distribución a un modelo biexponencial30,38.
7. Ensayos de cambio de movilidad electroforética (EMSA)
- En un volumen total de 50 l, incubar la concentración deseada de GEN1 con 50 pM de HJ con la etiqueta Cy5 a RT durante 30 minutos en búfer de unión EMSA (Tabla 1).
- Cargue las muestras en 8 cm x 8 cm de gel Tris-borate-EDTA al 6%. Ejecute el gel con 100 V durante 1 h + 20 min en 1 búfer TBE en RT.
- Determinar el porcentaje de sustrato consolidado a una concentración de GEN1 a partir de su contribución relativa a la intensidad total de fluorescencia del carril respectivo.
NOTA: GEN1 monómero-HJ (banda I) se identifica por el acuerdo de su tamaño con la unión picomolar del monómero GEN1 al HJ21,30. GEN1-dimer-HJ se asigna a la banda II debido a la unión escalonada del monómero GEN1 al HJ21,23. - Calcule las constantes de enlace aparentes Kd-monomer-app-EMSA y Kd-dimer-app-EMSA utilizando la ecuación:

Dónde: Max es la concentración en la que las especies respectivas alcanzaron su máxima unión (monómero o dímero); n es el coeficiente de Hill; K d-app-EMSA es la constante de unión aparente de las especies respectivas, que denota la concentración de GEN1 en la que está presente el medio máximo de monómero o dímero.
Resultados
Sesgo del conformador e isomerización del HJ
La isomerización de HJ ha sido ampliamente investigada por FRET a través del etiquetado de dos brazos adyacentes de la unión17,18,39. El donante (Cy3) y el aceptador (Alexa Fluor 647) se colocan en los dos brazos vecinos, R (hebra 2) y X (hebra 3), respectivamente(Figura 2A). Los isómeros X apilados fueron asignados por sus dos hebras continuas [es decir, Iso(1,3) o Iso(2,4)]. El histograma ALEX FRET de la etiqueta adyacente X0 muestra dos picos que corresponden al intercambio de iso(1,3) más abundante (E 0,75) y Iso menos abundante(2,4) (E .0.40) (Figura 2B).
EL FRET de un solo color se utiliza para adquirir trazas de tiempo para registrar los rápidos cambios de conformación en el HJ libre con alta resolución temporal a través de la reducción del área utilizada de la cámara EMCCD2. Un seguimiento de tiempo FRET de un solo color representativo de la unión X0 muestra las transiciones entre isómeros FRET altos y bajos(Figura 2B). Las tasas de isomerización kIso(1,3)-Iso(2,4) y kIso(2,4)-Iso(1,3) obtenidas de los histogramas de tiempo de permanencia de Iso(1,3) e Iso(2,4)(Figura 2C) son coherentes con las notificadas anteriormente17.
SMFRET demuestra una distorsión activa del HJ por GEN1
HJ se somete a una reorganización estructural al vincularse a GEN122. Por lo tanto, el espaciado entre el donante y el aceptador es similar tanto en Iso(1,3) como en Iso(2,4)(Figura 3A). Los ensayos de unión smFRET se llevaron a cabo en presencia de Ca2+ para evitar el escote del HJ. LOS histogramas FRET de la unión de etiqueta adyacente X0 a diferentes concentraciones de GEN1 fueron adquiridos por ALEX(Figura 3B). El histograma se ajusta a dos funciones gaussianas: una correspondiente al iso FRET alto libre(1,3), y la otra correspondiente a la población GEN1-HJ enlazada después de restar la contribución del Iso(2,4) del pico bajo fret.
Al saturar la concentración de GEN1, el histograma FRET de X0 sólo tiene un único pico FRET bajo correspondiente a GEN1 vinculado a cualquiera de los isómeros del HJ según lo predicho por el modelo22. La constante de disociación de monómeros aparentes (Kd-monomero-app) se determina a partir del ajuste hiperbólico de los porcentajes de población ligada a GEN1 en función de la concentración de GEN1 (Figura 3C). La etiqueta adyacente nk-X0 representa una versión de rrutas por separado HJ que imita el producto después de la primera reacción de incisión. Debido al alivio de la deformación unitaria por el nick simulado, nk-X0 es una estructura no isomeroizante40 como se puede ver desde el pico de sustrato único en E 0,40, a diferencia de X0 (Figura 3D frente a figura 3B). La estructura del complejo GEN1-nk-X0 es similar a la del complejo GEN1-X0, como lo indica la similitud en las eficiencias de FRET (E 0,25 para nk-X0 y 0,32 para X0)(Figura 3D frente a. Figura 3B). La unión fuerte del monómero GEN1 a nk-X0 se demuestra con el valor de laaplicación de monómero d K inferior 40 veces menor que el de X0 (Figura 3E frente a la figura 3C). Esta estrecha unión puede actuar como un mecanismo de salvaguardia contra la resolución incompleta del HJ en el improbable caso de disociación del dimer GEN1 o de uno de sus monómeros.
Unión escalonada del monómero GEN1 al HJ
La unión del monómero GEN1 al HJ seguido de la formación de dimer es una característica única para la resolvasa eucariota HJ resolvasa GEN1 en comparación con las resolvasas procariotas, que existen en forma dimerica en la solución21,23,41. EMSA de GEN1 a 50 pM X0 muestra la asociación escalonada de GEN1 en complejos de orden superior, como indican los números romanos en el panel superior(Figura 4A). La constante de disociación del monómero GEN1 determinada por EMSA (Kd-monomer-EMSA) coincide con la constante de disociación del ensayo de unión smFRET Kd-monomer-app (Figura 4A y Figura 3C , respectivamente). La cuantificación de la banda II se utiliza para calcular la constante de disociación de equilibrio del dimer GEN1 (Kd-dimer-EMSA). EMSA de GEN1 a 50 pM nk-X0 demuestra la unión de monómero prominente como lo indica el muy bajo Kd-monomero-app-EMSA que es 30 veces menor que el de X0, mientras que su Kd-dimer-EMSA es comparable a la de X0 (Figura 4B).
Otras pruebas de que el monómero GEN1 une y distorsiona el HJ es la observación de un número significativo de trazas de partículas no deslacadas con un estado estable de FRET bajo(Figura 4C)en presencia de Mg2+ a bajas concentraciones de GEN1. El número de estos rastros disminuyó al aumentar la concentración de GEN1. La resolución del HJ es impulsada por la estrecha unión del monómero GEN1, que apoya la formación de dimer. La unión monómero se observa en los rastros de tiempo del nk-X0 sin calibre en Mg2+,que se extiende hasta poco sin concentración nanomolar(Figura 4D). El monómero GEN1 se une firmemente para salvaguardar el nk-X0, asegurando finalmente la resolución completa a través de la formación de dimer.
Ensayo de resolución SMFRET del HJ
El término "escisión" en los ensayos smFRET se utiliza indistintamente con "resolución" del HJ, ya que en este ensayo sólo se detecta el lanzamiento del producto que sigue al segundo evento de escisión. Los eventos se registran mediante excitación de un solo color de lapso de tiempo para minimizar el fotoblanqueo del aceptador fotosensible durante el tiempo de adquisición de 1,3 min.
El esquema de la Figura 5A ilustra las incisiones de las hebras 1 y 3 de X0 Iso(1,3) después de la unión y distorsión por GEN1 de un X0 unido al vidrio funcionalizado. Tanto el donante como el aceptador entran en solución, lo que resulta en la pérdida de sus señales después de la resolución HJ. La primera y la segunda incisiones se desacoplan en nk-X0, lo que ejemplifica un prototipo para el HJ parcialmente resuelto. Tras la unión de GEN1, nk-X0 adopta una estructura similar a X0. La resolución procede de una sola incisión en la hebra 1, como se ilustra en la Figura 5B.
La salida simultánea del donante y del aceptador después de un estado estable de FRET bajo en las trazas de X0 resuelto se produjo sin la aparición de un FRET intermedio (E -0,40) indica que la resolución completa se produce dentro de la vida útil del GEN1-HJ complejo(Figura 5C). Por lo tanto, estos resultados sugieren que la resolución HJ se produce dentro de la vida útil compleja GEN1-HJ. La resolución de nk-X0 también procede después de la reorganización estructural y concluye por la salida del dúplex que lleva dos fluoróforos(Figura 5D)similares a X0.
Cinética de la dimerización GEN1 en el monómero GEN1 enlazado a HJ
El smFRET de lapso de tiempo mide antes delescote, que incluye principalmente el tiempo necesario para la formación de dimer y la resolución del HJ después de la distorsión por parte del monómero GEN1. Aplicando esta técnica, se proporcionan pruebas directas para apoyar la afirmación de que la formación de dimer es necesaria para la resolución de X0 y nk-X0,ya que la distribución dela escisión anterior es dependiente de la concentración GEN1.
La velocidad aparente de la resolución HJ (kapp) se define como la inversa de la media de la escisión antes de laescisión en la concentración gen1 respectiva. El término "aparente" se utiliza para describir la tasa de la resolución HJ, ya que no se puede excluir la posibilidad de que GEN1 permanezca vinculada al producto después de la resolución HJ.
Las funciones de densidad de probabilidad (PDF) de lasdistribuciones de x0 (Figura 6A)reflejan el tiempo de formación de dimer, que es más larga en concentraciones bajas de GEN1, y luego más cortas a concentraciones de GEN1 más altas. Las tasas de asociación y disociación para el dimer, kon-dimer y koff-dimer, respectivamente, se determinan a partir de un modelo biexponencial30. Además, los ARCHIVOS PDF de nk-X0 (Figura 6B)muestran una distribución similar a X0 que indica el requisito de formación de dimer.
La gráfica de laaplicación kfrente a la concentración de GEN1 se ha ajustado a una función hiperbólica. Las constantes de velocidad de catálisis aparentes (kMax-app) de X0 y nk-X0 son 0.107 a 0.011 s-1 y 0.231 a 0.036 s-1, respectivamente(Figura 6C). Las gráficas de kapp para x0 y nk-X0 uniones se cruzan a concentración de GEN1 5,6 nM debido a la más rápida kMax-app y más lento ken eldimer de la rascaen en comparación con la unión intacta.
En resumen, el ken-dimer relativamente rápido y lento koff-dimer conducen a la progresión de la reacción hacia adelante hacia la resolución HJ una vez que se forma el dimer. La fuerte unión del monómero GEN1 a la unión nk-X0 constituye un mecanismo a prueba de fallos contra cualquier segunda escisión abortada poco probable o ayuda a recoger cualquier HJ incompletamente no resuelto dejado por las vías de resolución primaria en la célula.
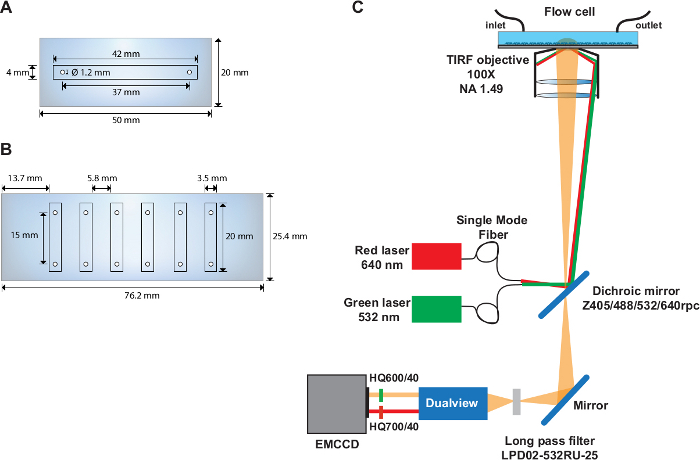
Figura 1: Células de flujo de un solo canal y múltiples canales y diseño de la configuración óptica.
(A) Esquema de la celda de flujo de un solo canal. (B) Esquema de la celda de flujo de seis canales. (C) Disposición de la configuración óptica que representa las fuentes de excitación, el objetivo TIRF, el espejo dicroico instalado dentro del cubo de filtro y los filtros de emisión utilizados en el dispositivo divisor de imagen. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Sesgo de conformador e isomerización del HJ observado por FRET.
(A) Isomerización de los conformadores HJ apilados por X de etiqueta adyacente que llevan el nombre de las dos hebras continuas. Las hebras están numeradas, mientras que los brazos se denotan con letras. Los sitios de incisión se muestran mediante flechas. Se indican las posiciones del donante (verde) y del aceptador (rojo) y el cambio de FRET tras la isomerización. (B) Panel derecho: Traza de tiempo FRET (negro) e traza FRET idealizada (rojo) de X0 a 50 mM Mg2+. Panel izquierdo: histograma FRET de X0 a 50 mM Mg2+. Las intensidades de fluorescencia del donante (verde) y del aceptador (rojo) se muestran a continuación. (C) Los histogramas de tiempo de permanencia de las etiquetas adyacentes X0 Iso(1,3) e Iso(2,4) se instalaron en funciones de un solo exponencial para determinar las tasas de isomerización. Las incertidumbres indican el intervalo de confianza del 95% del ajuste. Esta figura ha sido modificada de la literatura publicada anteriormente30.
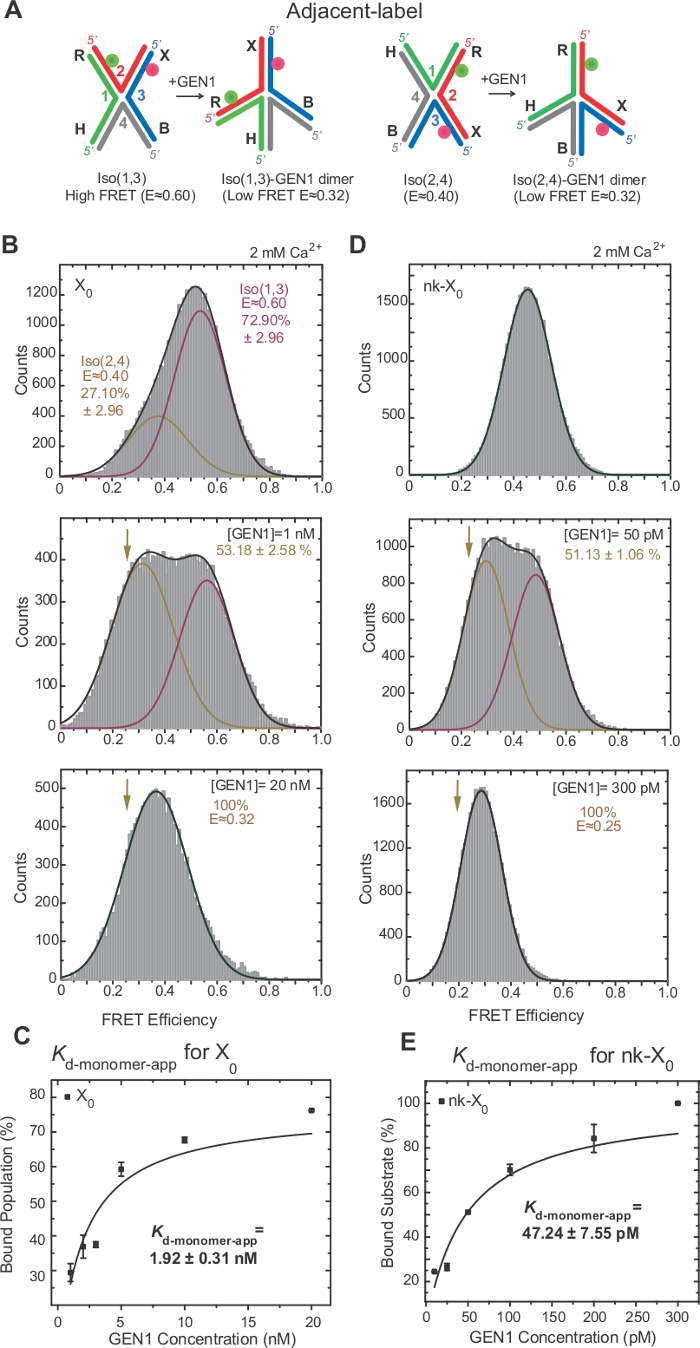
Figura 3: Distorsión activa del HJ por GEN1.
(A) Modificación estructural de la etiqueta adyacente HJ basada en el modelopropuesto 22. (B) el histograma ALEX FRET de la etiqueta adyacente X0 tiene un pico de FRET alto importante (E - 0,6) correspondiente a Iso(1,3) y un pico de FRET inferior (E a 0,4) para Iso(2,4). Todo el histograma se ajusta a dos funciones gaussianas: una correspondiente a la iso FRET alta libre(1,3), y la otra correspondiente a la población consolidada menos la contribución inicial de Iso(2,4) a la población total. (C) La constante aparente de disociación monomélica (Kd-monomero-app) se determina a partir de un ajuste hiperbólico de los porcentajes de poblaciones ligadas a GEN1 en función de la concentración de GEN1. (D) histogramas FRET de la etiqueta adyacente nk-X0 en diferentes concentraciones de GEN1. El área bajo el FRET bajo (E - 0,25) gaussiano corresponde al porcentaje de la población consolidada. (E) La aplicación Kd-monomero de nk-X0 se determina a partir del ajuste hiperbólico de la población enlazada a GEN1. Las barras de error representan las desviaciones estándar de dos o más experimentos. Esta figura ha sido modificada de la literatura publicada anteriormente30. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Unión escalonada de GEN1 al HJ.
(A) Ensayo de desplazamiento de movilidad electroforético (EMSA) de GEN1 a 50 pM X0. Panel superior: los números romanos indican el número de monómeros GEN1 en el complejo. Panel inferior: encuadernación del monómero GEN1 a X0. Las constantes de disociación aparentes se obtuvieron de un ajuste sigmoidal de la especie respectiva y representan el promedio de dos experimentos. (B) EMSA de GEN1 a 50 pM nk-X0 demuestra la unión de monómero prominente como se indica en el muy bajo Kd-monómero-app-EMSA. (C) TRAS trace de tiempo de la etiqueta adyacente enlazada pero sin dejar de dejar X0 en Mg2+. Se realizó una excitación de donantes durante 1,3 min, seguida de una excitación directa del aceptador (región rosa sombreada). (D) TRAS trace de tiempo de la etiqueta adyacente enlazada pero sin alarnada nk-X0 en Mg2+. Esta figura ha sido modificada de la literatura publicada anteriormente30. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Ensayo de resolución SMFRET del HJ.
(A) Esquema de la etiqueta adyacente X0 Iso(1,3) después de la distorsión por GEN1. El sustrato se une a la superficie funcionalizada a través del enlace biotina/avidina. La disociación de GEN1 después de las dos incisiones da como resultado la pérdida tanto del donante como del aceptador que entran en solución. (B) Esquema de la resolución de la etiqueta adyacente nk-X0 mediante el corte de la hebra 1. (C) Trazado temporal (negro) a 2 mM Mg2+ del escote de Iso(1,3). El inicio de la unión GEN1 forma un estado estable de FRET bajo hasta que la señal FRET se pierde abruptamente debido al escote. En consecuencia, el aumento del donante y la disminución de las intensidades de fluorescencia del aceptación tras la unión gen1 es seguido por la desaparición simultánea de la fluorescencia de ambos tintes tras el escote. (D) Del mismo modo, el seguimiento temporal de nk-X0 muestra un estado estable de FRET bajo GEN1 que se concluye por la pérdida abrupta de la señal FRET. Esta figura ha sido modificada de la literatura publicada anteriormente30. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Cinética de la dimización GEN1 en HJ enlazado a monómero GEN1.
(A) La gráfica de la función de densidad de probabilidad (PDF) de la distribución antes delescote de X0 ilustra su dependencia de la concentración de GEN1. Los tiempos de permanencia del estado de FRET bajo (antes de laescisión)en la concentración respectiva de GEN1 se obtuvieron a partir de dos o más experimentos y se utilizaron para obtener tasas medias (kapp). Las tarifas deaplicación kenumeradas se determinan a partir de la inversa de la mediade la escisión antes de la escisión en la concentración gen1 respectiva. Las tasas de asociación (kon-dimer) y disociación (koff-dimer) para la formación de dimer se calculan a partir de un modelo biexponencial38. Los errores representan SEM de kapp. (B) La gráfica PDF de las distribuciones antes de laescisión de las tarifas de laaplicación nk-X0 y k respectivas. (C) Gráfica de laaplicación k frente a la concentración GEN1 ajustada a una función hiperbólica para determinar la velocidad catalítica aparente (kMax-app). La gráfica de laaplicación k para X0 y nk-X0 ilustra laaplicación kinicial más rápida de X0, que luego es superada por nk-X0 por encima de [GEN1] 5,6 nM. Esta figura ha sido modificada de la literatura publicada anteriormente30. Haga clic aquí para ver una versión más grande de esta figura.
| Búfer | Compostion | ||
| Búfer de enlace | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 2 mM CaCl2, 1 mM DTT, 0,1% BSA y 5% (v/v) glicerol | ||
| Zona de influencia A | 20 mM Tris-HCl pH 8.0, 1 mM DTT y 300 mM NaCl | ||
| Buffer B | 20 mM Tris-HCl pH 8.0, 1 mM DTT y 100 mM NaCl | ||
| Buffer C | 20 mM Tris-HCl pH 8.0 y 1 mM DTT | ||
| Búfer de escote | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 2 mM MgCl2, 1 mM DTT, 0,1% BSA y 5% (v/v) glicerol | ||
| Búfer de enlace EMSA | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 1 mM DTT, 2 mM CaCl2, 0,1 mg/ml BSA, 5% (v/v) glicerol y 5 ng/l Poly-dI-dC | ||
| Búfer de imágenes (enlace) | 40 l (o)-6-Hidroxi-2,5,7,8-tetrametilquicrono-2-ácido carboxílico (4 m), 60 l de PCA (6 nM), PCD de 60 ml (60 nM) y 840 l de tampón de unión | ||
| Búfer de imágenes (escisión) | 40 l (o)-6-Hidroxi-2,5,7,8-tetrametilquicrono-2-ácido carboxílico (4 m), 60 ml de PCA (6 nM), 60 ml de PCD (60 nM) y 840 l de tampón de escisión | ||
| Buffer de Lysis | 20 mM Tris-HCl pH 8.0, 10 mM -mercaptoetanol, 300 mM NaCl y 2 mM PMSF | ||
| Búfer de almacenamiento PCD | 100 mM Tris-HCl pH 7,5, 1 mM EDTA, 50 mM KCl y 50% glicerol | ||
| buffer de almacenamiento | 20 mM Tris-HCl pH 8.0, 1 mM DTT, 0.1 mM EDTA, 100 mM NaCl y 10% glicerol | ||
| Buffer TBE | 89 mM Tris-HCl, 89 mM de ácido bórico y 2 mM EDTA | ||
| Búfer TE100 | 10 mM Tris.HCl pH 8.0 y 100 mM NaCl | ||
| Búfer Tris-EDTA | 50 mM Tris-HCl pH 8,0 y 1 mM EDTA pH 8,0 | ||
Tabla 1: La lista de tampones y sus composiciones utilizadas en este estudio.
| Oligo | Secuencia | ||
| X0-st1 | ACGCTGCCGAATTCTACCAGCCTTGCTAGTAGTAGATTTTGCCCACCTGCAGTCACCC | ||
| X0-st2 | GGGTGAACCTGCAGGTGGG/iCy3/AAAGATGTCCATCTGTTGTAATCGTCAAGCTTTATGCCGT | ||
| X0-st3 | ACGGCATAAAGCTTGACGA/iAF647-dT/TACAACAGATCATGGAGCTGTGTATCCGACTATCG | ||
| X0-st4 | 5'BiotinCGATAGTCGGATCCTCTAGACAGCTCCATGTAGCAAGGCACTGTAGAATTCGGCAGCGT | ||
| X0-Adj | X0-st1, X0-st2, X0-st3 & X0-st4 | ||
| X0In_st2 | GGGTGAACCTGCAGTGGGCAAAGATGTCCATCTGTTGTAATCGTCAAGCTTTATGCCGT | ||
| X0In_st4 | 5'BiotinCGATAGTCGGATCCTCTAGACAGCTCCATGTAGCAAGGCA/iCy3/TGGTAGAATTCGGCAGCGT | ||
| Nk-X0 | X0-st1, X0-st2, X0-nk3a, X0-nk3b & X0-st4 | ||
| X0-nk3a | ACGGCATAAAGCTTGACGA/iAF647-dT/TACAACAGATC | ||
| X0-nk3b | ATGGAGCTGTECTGATCCGACTATCG | ||
Tabla 2: Sustratos SMFRET y EMSA HJ. La lista de oligonucleótidos utilizados para la preparación de los HJs etiquetados fluorescentemente para smFRET y EMSA. Los oligos se obtuvieron comercialmente. Los oligos etiquetados fluorescentemente fueron purificados por HPLC y, cuando era posible, los oligos de 60 bp fueron purificados por PAGE.
Discusión
En este estudio, se implementaron diferentes técnicas de smFRET para determinar la cinética de la resolución HJ por GEN130. Se utilizaron enfoques similares de smFRET para seguir el requisito de conformación del ADN de doble aleta y el escote por la replicación del ADN y reparar la colgajo endonucleasa 142,43,44. Aquí, se discuten los pasos críticos en este protocolo. La reacción de silanización debe estar libre de cualquier rastro de humedad. La solución de pegilación se debe aplicar rápidamente en el vidrio silanizado una vez que se disuelva PEG para evitar la hidrólisis. En la celda de flujo multicanal, se debe eliminar cualquier aire atrapado en la hoja adhesiva para evitar fugas entre los canales vecinos. La solución de PCA debe estar recién preparada ya que se oxida con el tiempo. La adición de 10 N NaOH debe ser dropwise, con vórtice en el medio. El fondo de fluorescencia en el cubreobjetos debe ser mínimo antes de fluir el HJ con etiqueta fluorescente. Las imágenes en la célula de flujo se deben realizar en una dirección para evitar las imágenes en áreas blanqueadas. En los experimentos CON ALEX, se debe reducir la potencia del láser rojo para evitar el blanqueo rápido del aceptador. En los experimentos de lapso de tiempo, el tiempo de ciclo tiene que ser más corto que el evento más rápido.
smFRET es una técnica sensible que puede proporcionar valiosas perspectivas en tiempo real en reacciones biomoleculares. Sin embargo, este método tiene varios desafíos técnicos, entre los que se encuentra el logro de un cambio medible en FRET durante la reacción bioquímica. Esto es necesario para obtener entidades bien separadas en los histogramas y estados distinguibles en los seguimientos de tiempo. En muchos casos, smFRET requiere un diseño cuidadoso de los sustratos, la selección de los pares de fluoróforos y sus posiciones, y la amplificación de los cambios de FRET en el sustrato de ADN debido a los pequeños cambios estructurales en el sustrato45. Otro enfoque para realizar FRET es utilizar proteínas etiquetadas46. La ventana de observación en FRET está limitada por la estabilidad del aceptador como Cy5 o Alexa Fluor 647 que tiende a blanquear más rápidamente que el donante (Cy3 en este caso). Por lo tanto, FRET requiere una búsqueda continua de fluoróforos estables para extender la duración del experimento y los esfuerzos para desarrollar sistemas de barrido de oxígeno para prolongar la señal de fluorescencia y maximizar la relación señal-ruido47,48 .
Entre los consejos para la solución de problemas en smFRET está el equilibrio de los diversos parámetros involucrados en la imagen, como la potencia del láser, el tiempo de exposición, el tiempo de ciclo y el número de ciclos para maximizar la emisión de fluorescencia, prolongar la duración del experimento y lograr intervalos de muestreo adecuados para la dinámica enzimática. Los tiempos de observación más largos y los efectos mínimos del fotoblanqueo son esenciales para obtener distribuciones de tiempo de permanencia de alta fidelidad que representan la dinámica enzimática. ALEX genera mejores histogramas ya que este método está sujeto a menores contribuciones de partículas fotoblanqueadas en comparación con FRET de un solo color. Sin embargo, la resolución temporal en ALEX es menor que la de FRET de un solo color.
Por último, el énfasis de smFRET en la detección de cambios conformacionales/estructurales en moléculas individuales en tiempo real cierra la brecha entre las técnicas estructurales de alta resolución (es decir, cristalografía de rayos X, resonancia magnética nuclear, microscopía electrónica), que proporciona detalles estructurales de resolución atómica en condiciones estáticas y métodos a granel que producen el promedio de conjunto de una propiedad medible. En muchos aspectos, smFRET ha demostrado ser una técnica poderosa para estudiar sistemas biológicos en tiempo real.
Divulgaciones
Los autores no declaran intereses financieros en competencia.
Agradecimientos
Este trabajo fue apoyado por la Universidad De Ciencia y Tecnología Rey Abdullah a través de la financiación básica y el Premio de Investigación Competitiva (CRG3) a S. M. H.
Materiales
| Name | Company | Catalog Number | Comments |
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox) | Sigma-Aldrich | 238813 | |
| 0.1 M sodium bicarbonate buffer | Fisher | 144-55-8 | |
| 10 % Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6275BOX | |
| 100 X TIRF objective | Olympus | NAPO 1.49 | |
| 3,4-dihroxybenzoic acid (PCA) | Sigma-Aldrich | P5630 | |
| 3-aminopropyltriethoxysilane (APTES) | Sigma-Aldrich | 741442 | |
| 6% Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6265BOX | |
| Adhesive sheet | Grace bio-labs | SA-S-1L | |
| Benchtop refrigerated centrifuge | Eppendorf | Z605212 | |
| Biotin-PEG | Laysan Bio | Biotin-PEG-SVA 5000 | |
| Bovine Serum Albumin (BSA) | New England Biolabs | B9001S | |
| Calcium Chloride Dihydrate | Sigma-Aldrich | 31307 | |
| cation exchange column | GE healthcare | MonoS (4.6/100) | |
| Cell distruptor | Constant Cell Disruption System | TS5/40/CE/GA | |
| Coomassie Brilliant Blue | MP Biomedicals | 808274 | |
| Cy3 emission filter | Chroma | HQ600/40M-25 | |
| Cy5/Alexa Fluor 647 emission filter | Chroma | HQ700/40M-25 | |
| Dichroic for DV2 filter cube | Photometrics | 630dcxr-18x26 | |
| Dithiothreitol (DTT) | Thermo Scientific | R0861 | |
| Drill | Dremel | 200-1/21 | |
| Electronic cutter | Copam | CP-2500 | |
| EMCCD camera | Hamamatsu | C9100-13 | |
| Epoxy glue | Devcon | 14250 | |
| FPLC Aktapurifier UPC 10 | GE Healthcare | 28406268 | |
| GelQuant.NET software | biochemlabsolutions.com | Version 1.8.2 | |
| GEN1 entry vector | Harvard plasmid repository | HSCD00399935 | |
| Glycerol | Sigma Life Science | G5516 | |
| green laser (emission 532 nm) | Coherent | Compass 315M-100 | |
| Heparin column | GE healthcare | HiTrap Heparin column | |
| HEPES | BDH | BDH4162 | |
| Image splitter | Photometrics | Dualview (DV2) | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Inverted microscope | Olympus | IX81 | |
| Isopropyl-ß-D-thiogalactoside (IPTG) | Goldbio. | 12481C100 | |
| Laser scanner | GE healthcare | Typhoon Trio | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Long pass 532nm filter | Semrock | LPD02-532RU-25 | |
| Magnesium Chloride | Sigma Life Science | M8266 | |
| mPEG | Laysan Bio | mPEG-SVA 5000 | |
| Neutravidin | Pierce | 31000 | |
| Ni-NTA column | GE healthcare | HisTrap FF | |
| NuPAGE 10% Bis-Tris gels | Novex Life technologies | NP0301BOX | |
| NuPAGE 10% Bis-Tris Protein Gels | Thermo Fisher Scientific | NP0302PK2 | |
| Origin software | OriginLab Corporation | Version 8.5 | |
| Phenylmethylsulfonyl fluoride (PMSF) | Alexis Biochemicals | 270-184-G025 | |
| Phosphate-buffered saline | GIBCO | 14190 | |
| Polyethylene Tubing (I.D. 0.76 mm O.D. 1.22mm) | Fisher (Becton Dickinson) | 427416 | |
| Protocatechuate 3,4-dioxygenase (3,4-PCD) | Sigma-Aldrich | P8279-25UN | |
| Quad-band dichroic | Chroma Inc | Z405/488/532/640rpc | |
| red laser (emission 640 nm) | Coherent | Cube 640 100C | |
| Sodium Chloride | Fisher Chemical | S271 | |
| Sorvall RC-6 plus centrifuge | Thermo Fisher Scientific | 46910 | |
| Spectrophotometer | Thermo Fisher Scientific | Nanodrop 2000 | |
| Syringe pump | Harvard Apparatus | 70-3007 | |
| Teflon tweezers | Rubis | K35A | |
| Tris Base | Promega | H5135 | |
| Ultracentrifuge | Beckman Coulter | Optima L-90K | |
| Ultrafiltration membrane | Millipore | UFC90300 |
Referencias
- Moerner, W. E., Fromm, D. P. Methods of single-molecule fluorescence spectroscopy and microscopy. Review of Scientific Instruments. 74 (8), 3597-3619 (2003).
- Ha, T. Single-molecule fluorescence resonance energy transfer. Methods. 25 (1), 78-86 (2001).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47, 819-846 (1978).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nature Methods. 5 (6), 507-516 (2008).
- Walter, N. G., Huang, C. Y., Manzo, A. J., Sobhy, M. A. Do-it-yourself guide: how to use the modern single-molecule toolkit. Nature Methods. 5 (6), 475-489 (2008).
- Conchello, J. A., Lichtman, J. W. Optical sectioning microscopy. Nature Methods. 2 (12), 920-931 (2005).
- Axelrod, D. Total internal reflection fluorescence microscopy in cell biology. Methods in Enzymology. 361, 1-33 (2003).
- Kim, H. D., et al. Mg2+-dependent conformational change of RNA studied by fluorescence correlation and FRET on immobilized single molecules. Proceedings of the National Academy of Sciences of the United States of America. 99 (7), 4284-4289 (2002).
- Lee, T. H., et al. Measuring the folding transition time of single RNA molecules. Biophysical Journal. 92 (9), 3275-3283 (2007).
- Holliday, R. Mechanism for Gene Conversion in Fungi. Genetical Research. 5 (2), 282-304 (1964).
- West, S. C., et al. The Formation and Resolution of Holliday Junctions during the Recombinational Repair of DNA Damages. Journal of Cellular Biochemistry. , 269-269 (1995).
- Cox, M. M., et al. The importance of repairing stalled replication forks. Nature. 404 (6773), 37-41 (2000).
- West, S. C. Molecular views of recombination proteins and their control. Nature Reviews: Molecular Cell Biology. 4 (6), 435-445 (2003).
- Duckett, D. R., et al. The structure of the Holliday junction, and its resolution. Cell. 55 (1), 79-89 (1988).
- Clegg, R. M., et al. Fluorescence resonance energy transfer analysis of the structure of the four-way DNA junction. Biochemistry. 31 (20), 4846-4856 (1992).
- McKinney, S. A., Declais, A. C., Lilley, D. M., Ha, T. Structural dynamics of individual Holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2003).
- Joo, C., McKinney, S. A., Lilley, D. M., Ha, T. Exploring rare conformational species and ionic effects in DNA Holliday junctions using single-molecule spectroscopy. Journal of Molecular Biology. 341 (3), 739-751 (2004).
- Hyeon, C., Lee, J., Yoon, J., Hohng, S., Thirumalai, D. Hidden complexity in the isomerization dynamics of Holliday junctions. Nature Chemistry. 4 (11), 907-914 (2012).
- Ip, S. C., et al. Identification of Holliday junction resolvases from humans and yeast. Nature. 456 (7220), 357-361 (2008).
- Rass, U., et al. Mechanism of Holliday junction resolution by the human GEN1 protein. Genes & Development. 24 (14), 1559-1569 (2010).
- Liu, Y., et al. Crystal Structure of a Eukaryotic GEN1 Resolving Enzyme Bound to DNA. Cell Reports. 13 (11), 2565-2575 (2015).
- Chan, Y. W., West, S. GEN1 promotes Holliday junction resolution by a coordinated nick and counter-nick mechanism. Nucleic Acids Research. 43 (22), 10882-10892 (2015).
- van Gool, A. J., Hajibagheri, N. M., Stasiak, A., West, S. C. Assembly of the Escherichia coli RuvABC resolvasome directs the orientation of holliday junction resolution. Genes & Development. 13 (14), 1861-1870 (1999).
- Lee, S. H., et al. Human Holliday junction resolvase GEN1 uses a chromodomain for efficient DNA recognition and cleavage. eLife. 4, (2015).
- Chan, Y. W., West, S. C. Spatial control of the GEN1 Holliday junction resolvase ensures genome stability. Nature Communications. 5, 4844(2014).
- Liu, Y., Freeman, A. D., Declais, A. C., Lilley, D. M. J. A monovalent ion in the DNA binding interface of the eukaryotic junction-resolving enzyme GEN1. Nucleic Acids Research. 46 (20), 11089-11098 (2018).
- Zhou, R., et al. Junction resolving enzymes use multivalency to keep the Holliday junction dynamic. Nature Chemical Biology. 15 (3), 269-275 (2019).
- Bellendir, S. P., et al. Substrate preference of Gen endonucleases highlights the importance of branched structures as DNA damage repair intermediates. Nucleic Acids Research. 45 (9), 5333-5348 (2017).
- Sobhy, M. A., et al. Resolution of the Holliday junction recombination intermediate by human GEN1 at the single-molecule level. Nucleic Acids Research. 47 (4), 1935-1949 (2019).
- Sobhy, M. A., et al. Versatile single-molecule multi-color excitation and detection fluorescence set-up for studying biomolecular dynamics. Review of Scientific Instruments. 82 (11), 113702(2011).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Rashid, F., et al. Initial state of DNA-Dye complex sets the stage for protein induced fluorescence modulation. Nature Communications. 10 (1), 2104(2019).
- Sambrook, J., Russell, D. W. Standard ethanol precipitation of DNA in microcentrifuge tubes. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Holden, S. J., et al. Defining the limits of single-molecule FRET resolution in TIRF microscopy. Biophysical Journal. 99 (9), 3102-3111 (2010).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L. Jr, Wiggins, C. H. Learning rates and states from biophysical time series: a Bayesian approach to model selection and single-molecule FRET data. Biophysical Journal. 97 (12), 3196-3205 (2009).
- Kou, S. C., Cherayil, B. J., Min, W., English, B. P., Xie, X. S. Single-molecule Michaelis-Menten equations. Journal of Physical Chemistry B. 109 (41), 19068-19081 (2005).
- Clegg, R. M., Murchie, A. I., Lilley, D. M. The solution structure of the four-way DNA junction at low-salt conditions: a fluorescence resonance energy transfer analysis. Biophysical Journal. 66 (1), 99-109 (1994).
- Pohler, J. R., Duckett, D. R., Lilley, D. M. Structure of four-way DNA junctions containing a nick in one strand. Journal of Molecular Biology. 238 (1), 62-74 (1994).
- Fogg, J. M., Lilley, D. M. Ensuring productive resolution by the junction-resolving enzyme RuvC: large enhancement of the second-strand cleavage rate. Biochemistry. 39 (51), 16125-16134 (2000).
- Sobhy, M. A., Joudeh, L. I., Huang, X., Takahashi, M., Hamdan, S. M. Sequential and multistep substrate interrogation provides the scaffold for specificity in human flap endonuclease 1. Cell Reports. 3 (6), 1785-1794 (2013).
- Rashid, F., et al. Single-molecule FRET unveils induced-fit mechanism for substrate selectivity in flap endonuclease 1. eLife. 6, e21884(2017).
- Zaher, M. S., et al. Missed cleavage opportunities by FEN1 lead to Okazaki fragment maturation via the long-flap pathway. Nucleic Acids Research. 46 (6), 2956-2974 (2018).
- Didenko, V. V. DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. BioTechniques. 31 (5), 1106-1116 (2001).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados