Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Medición de distribuciones de cAMP tridimensionales en células vivas utilizando imágenes y análisis FRET hiperespectrales de 4 dimensiones (x, y, z y λ)
En este artículo
Resumen
Debido a la baja relación señal-ruido (SNR) inherente de los sensores basados en transferencia de energía de resonancia Fӧrster (FRET), la medición de señales cAMP ha sido un desafío, especialmente en tres dimensiones espaciales. Aquí, describimos una metodología de imagen y análisis FRET hiperespectral que permite la medición de la distribución de cAMP en tres dimensiones espaciales.
Resumen
El AMP cíclico es un segundo mensajero que está involucrado en una amplia gama de actividades celulares y fisiológicas. Varios estudios sugieren que las señales cAMP están compartimentadas, y que la compartimentación contribuye a la especificidad de la señalización dentro de la vía de señalización cAMP. El desarrollo de biosensores basados en la transferencia de energía de resonancia de Fӧrster (FRET) ha potenciado la capacidad de medir y visualizar señales cAMP en las células. Sin embargo, estas mediciones a menudo se limitan a dos dimensiones espaciales, lo que puede dar lugar a una mala interpretación de los datos. Hasta la fecha, solo ha habido mediciones muy limitadas de señales cAMP en tres dimensiones espaciales (x, y y z), debido a las limitaciones técnicas en el uso de sensores FRET que inherentemente exhiben una baja relación señal-ruido (SNR). Además, los enfoques tradicionales de imágenes basadas en filtros a menudo son ineficaces para la medición precisa de señales cAMP en regiones subcelulares localizadas debido a una variedad de factores, incluida la diafonía espectral, la intensidad de señal limitada y la autofluorescencia. Para superar estas limitaciones y permitir que los biosensores basados en FRET se utilicen con múltiples fluoróforos, hemos desarrollado enfoques hiperespectrales de imágenes y análisis de FRET que proporcionan especificidad espectral para calcular las eficiencias de FRET y la capacidad de separar espectralmente las señales FRET de la autofluorescencia de confusión y / o señales de etiquetas fluorescentes adicionales. Aquí, presentamos la metodología para implementar imágenes FRET hiperespectrales, así como la necesidad de construir una biblioteca espectral apropiada que no esté ni submuestreada ni sobremuestreada para realizar la desmezcla espectral. Si bien presentamos esta metodología para la medición de distribuciones tridimensionales de cAMP en células endoteliales microvasculares pulmonares (PMVEC), esta metodología podría usarse para estudiar distribuciones espaciales de cAMP en una variedad de tipos de células.
Introducción
El monofosfato de adenosina cíclico (cAMP) es un segundo mensajero involucrado en procesos celulares y fisiológicos clave, incluida la división celular, la afluencia de calcio, la transcripción de genes y la transducción de señales. Un creciente cuerpo de evidencia sugiere la existencia de compartimentos cAMP en la célula a través de los cuales se logra la especificidad de señalización1,2,3,4,5,6,7. Hasta hace poco, la compartimentación de cAMP se infería en función de distintos efectos fisiológicos o celulares inducidos por diferentes agonistas del receptor acoplado g8,9,10,11. Más recientemente, las sondas de imágenes de fluorescencia basadas en FRET han proporcionado nuevos enfoques para la medición directa y la observación de señales cAMP en una celda12,13,14.
La transferencia de energía de resonancia de Förster (FRET) es un fenómeno físico en el que la transferencia de energía se produce entre las moléculas donantes y aceptoras de una manera no radiativa cuando las moléculas están muy cerca15,16. Con el desarrollo de indicadores fluorescentes basados en FRET, este fenómeno físico se ha utilizado en aplicaciones biológicas para estudiar las interacciones proteína-proteína17,la colocalizaciónde proteínas 18,la señalización deCa+2 19,la expresióngénica 20,la división celular21 y la señalización de nucleótidos cíclicos. Los indicadores de cAMP basados en FRET generalmente consisten en un dominio de unión a cAMP, un fluoróforo de donante y un fluoróforoaceptor 22. Por ejemplo, el sensor H188 cAMP12,22 utilizado en esta metodología consiste en un dominio de unión cAMP obtenido de Epac, intercalado entre fluoróforos Turquesa (donante) y Venus (aceptor). En condiciones basales (sin consolidar), Turquesa y Venus están en una orientación tal que fret ocurre entre los fluoróforos. Al unirse el cAMP al dominio de unión, se produce un cambio conformacional tal que Turquesa y Venus se separan, lo que resulta en una disminución de FRET.
Los enfoques de imágenes basados en FRET ofrecen una herramienta prometedora para investigar y visualizar señales de cAMP dentro de una célula. Sin embargo, las técnicas actuales de imágenes microscópicas basadas en FRET a menudo solo tienen un éxito parcial en el logro de la intensidad de señal suficiente para medir FRET con claridad espacial subcelular. Esto se debe a varios factores, incluida la intensidad de señal limitada de muchos reporteros FRET, el alto nivel de precisión requerido para cuantificar con precisión los cambios en la eficiencia fret y la presencia de factores de confusión, como la autofluorescencia celular23,24. El resultado es a menudo una imagen FRET que está plagada de SNR débil, lo que hace que la visualización de los cambios subcelulares en FRET sea muy difícil. Además, la investigación de señales cAMP localizadas espacialmente se ha realizado casi exclusivamente en solo dos dimensiones espaciales y la distribución axial cAMP rara vez se ha considerado25. Esto es probable porque el SNR bajo impidió la capacidad de medir y visualizar gradientes cAMP en tres dimensiones espaciales. Para superar las limitaciones del uso de sensores FRET con SNR bajo, hemos implementado enfoques de imágenes y análisis hiperespectrales para medir FRET en células individuales25,26,27.
Los enfoques de imágenes hiperespectrales fueron desarrollados por la NASA para diferenciar los objetos terrestres presentes en las imágenessatelitales28,29. Desde entonces, estas técnicas se han traducido al campo de la microscopía de fluorescencia30,con varios sistemas comerciales de microscopio confocal que ofrecen detectores espectrales. En las imágenes de fluorescencia tradicionales (no espectrales), la muestra se excita utilizando un filtro de paso de banda o una línea láser, y la emisión se recoge utilizando un segundo filtro de paso de banda, a menudo seleccionado para que coincida con la longitud de onda de emisión máxima del fluoróforo (s). Por el contrario, los enfoques de imágenes hiperespectrales buscan muestrear un perfil espectral completo de la emisión de fluorescencia26,31,32 o la excitación33,34 a intervalos de longitud de onda específicos. En nuestros estudios previos, demostramos que los enfoques de imágenes y análisis hiperespectrales pueden ofrecer una mejor cuantificación de las señales FRET en las células en comparación con las técnicas tradicionales de imágenes FRET basadas en filtros26. Aquí, presentamos una metodología para realizar imágenes y análisis FRET hiperespectrales de 4 dimensiones (x, y, z y λ) para medir y visualizar distribuciones de cAMP en tres dimensiones espaciales. Estos enfoques han permitido la visualización de gradientes espaciales cAMP inducidos por agonistas en celdas individuales25. Curiosamente, dependiendo del agonista, los gradientes de cAMP pueden ser evidentes en las células. La metodología presentada aquí utiliza la desmezcla espectral de fondo no uniforme y autofluorescencia celular para mejorar la precisión de las mediciones FRET. Si bien esta metodología se demuestra en células endoteliales microvasculares pulmonares (PMVEC) utilizando un biosensor cAMP FRET, la metodología podría modificarse fácilmente para su uso con reporteros FRET alternativos o líneas celulares alternativas.
Protocolo
Este protocolo sigue los procedimientos aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad del Sur de Alabama.
1. Preparación de células, muestras y reactivos para imágenes
- Aislar las células endoteliales microvasculares pulmonares de rata (PMVEC) como se describió anteriormente35.
NOTA: Las células fueron aisladas y cultivadas por el Cell Culture Core de la Universidad del Sur de Alabama, Mobile, AL en platos de cultivo celular de 100 mm. - Sepepe PMVEC aislados en cubiertas de vidrio redondas de 25 mm y déjelos crecer en la incubadora a 37 ° C hasta que las células alcancen al menos el 80% de confluencia (al menos 24 horas).
NOTA: Las células y el tipo de célula pueden variar de un estudio a otro y, por lo tanto, se deben seguir procedimientos de cultivo celular específicos de la célula para sembrar y cultivar células. El protocolo de siembra y cultivo celular utilizado en estos estudios está disponible como información complementaria en el archivo denominado "Cultivo y Transfección de File_Cell Suplementario". - Transfecta PMVECs con un biosensor FRET e incuba durante 48 horas a 37 °C.
NOTA: El protocolo para transfectar PMVEC también se describe en el archivo de información suplementaria denominado "Cultivo y transfección de File_Cell suplementarios". - El día de la imagen, caliente el tampón de Tyrode a 37 ° C en un baño de agua.
NOTA: El búfer de Tyrode consta de 145 mM NaCl, 4 mM KCl, 10 mM HEPES, 10 mM glucosa, 1 mM MgCl2 y 1 mM CaCl2 - Monte una cubierta que contenga celdas transfectadas en una cámara de celda y asegure la parte superior con una junta de montaje para evitar fugas.
- Limpie la parte inferior de la funda con una delicada limpieza de tareas para limpiar cualquier exceso de medios o células adherentes.
- Agregue 800 μL de tampón de trabajo y 4 μL de etiqueta nuclear de 5 mM a la cámara celular y meca suavemente durante 5 a 10 segundos.
NOTA: Al agregar soluciones tampón o reactivos a los cocoláculos montados en la cámara celular, asegúrese de agregar la solución suavemente y al costado de la cámara celular para no desalojar las células adherentes. La adición de 4 μL de etiqueta nuclear de 5 mM a 800 μL de tampón hace que la concentración final de 25 μM de la etiqueta nuclear. Para células poco adherentes, como las células HEK293, mezcle primero la etiqueta nuclear y el tampón en un vial y luego agréguelos a los cofundidos montados en la cámara celular. Esto evitará levantar las células de la cubierta. - Cubra la cámara de la celda con papel de aluminio para proteger de la luz e incube durante 10 minutos a temperatura ambiente.
- Preparación del reactivo: Añadir 1 μL de forskolina de 50 mM a 199 μL de tampón. Esto producirá una concentración final de forskolina de 50 μM cuando se agrega a las células que se prepararon con 800 μL de tampón. También debe prepararse 1 μL de DMSO en 199 μL de tampón para ser utilizado como control del vehículo.
NOTA: En estos estudios, la forskolina se utiliza como un activador de la adenicilo ciclasa para estimular la producción de cAMP. Si se desea, esta metodología puede modificarse fácilmente para permitir el tratamiento con reactivos alternativos para estimular o inhibir la adenicilo ciclasa, fosfodiesterasas, etc.
2. Adquisición de imágenes
- Utilice un microscopio confocal equipado con un detector espectral.
NOTA: Todos los pasos de adquisición de imágenes descritos aquí se desarrollaron utilizando un sistema de microscopio Nikon A1R disponible comercialmente. Es posible que sea necesario ajustar estos pasos si se utiliza un microscopio espectral alternativo. Asegúrese de que todo el equipo esté encendido al menos 30 minutos antes del inicio del experimento para alcanzar condiciones de funcionamiento estables. - Seleccione el objetivo de inmersión en agua 60x y agregue una gota de agua al objetivo.
NOTA: Para imágenes de células vivas de alta resolución, se recomienda utilizar un objetivo de apertura numérica alta. Consulte la Lista de materiales para obtener información sobre el objetivo utilizado en estos estudios. - Coloque la cámara celular cargada (a partir del paso 1.7) en la etapa del microscopio.
- Seleccione el conjunto de filtros DFT (DAPI/FITC/TRITC) ajustando la perilla del filtro en el lado derecho del microscopio.
- Opere el microscopio en modo de campo ancho de fluorescencia utilizando los oculares para seleccionar un campo de visión que contenga células que expresen el sensor cAMP FRET.
NOTA: Asegúrese de que la intensidad media de la señal FRET en la longitud de onda máxima de emisión del donante o aceptor en la celda seleccionada sea de al menos 100 unidades de intensidad (U.A.) o al menos 4 veces la señal de referencia de una región sin células expresas. Esto se puede confirmar utilizando el visor de perfiles de espectro disponible en el software NIS Elements. Cuando se busca una célula con buena señal, es recomendable descartar células excesivamente brillantes (pueden estar comprometidas). - Abra el software NIS, cambie al modo confocal, desbloquee el botón de enclavamiento láser y haga clic en Live.
- Utilice la perilla de enfoque para centrarse en las celdas mirando la vista previa en la pantalla.
- Configure los ajustes del dispositivo, la adquisición y la pila z en el software, como se describe a continuación.
- Configuración de adquisición:
NOTA: La configuración de adquisición de la cámara y el dispositivo se puede aplicar utilizando una imagen adquirida previamente. Abra la imagen, haga clic con el botón derecho y seleccione Reutilizar configuración de cámara.- Abra el menú de configuración A1, marque las casillas correspondientes a las líneas láser de 405 nm y 561 nm, seleccione SD para el detector espectral, seleccione 10 para la resolución y 31 para los canales.
NOTA: El menú de configuración de A1 se muestra como un pequeño icono de engranaje en la esquina superior izquierda de la ventana configuración de A1 Plus. El láser de 405 nm se utiliza para la excitación del donante y el láser de 561 nm se utiliza para la excitación de la etiqueta nuclear. - Establezca el rango de longitud de onda (410 – 730 nm) seleccionando los valores de longitud de onda inicial y final.
- Haga clic en el icono de binning/skip en el menú de configuración de A1 y seleccione el cuadro numerado 15 y, a continuación, haga clic en Aceptar en el menú de configuración de A1.
NOTA: Esto es para eliminar el canal de longitud de onda que corresponde al láser de excitación de 561 nm (este es típicamente el canal de longitudde onda 15). Es importante no utilizar esta banda de longitud de onda para evitar una señal artificialmente baja, que puede crear un artefacto espectral. La señal es más baja en esta banda debido al dedo mecánico que cubre el elemento detector para protegerlo del daño del láser. - Establezca las intensidades del láser en 8% y 2% para los láseres de 405 nm y 561 nm, respectivamente, Si Hv (ganancia del detector) en 149 y un radio estenopeico de 2,4 unidades de disco aireado (AU).
NOTA: Es posible que las intensidades del láser deban ajustarse según la edad del instrumento y la condición de los láseres. Si se ajustan las intensidades del láser entre diferentes muestras o grupos experimentales, es importante mantener la misma proporción de intensidades del láser (por ejemplo, 8:2). Además, es importante seleccionar una intensidad de láser que no sea tan brillante como para crear un fotobleaching rápido. La ganancia del detector debe ajustarse para maximizar la intensidad de la señal y minimizar el ruido del detector. Para estos estudios, se utilizó una ganancia de 149. Se seleccionó un tamaño estenopeico de 2,4 UA como equilibrio entre la adquisición de imágenes con suficiente relación señal/ruido (SNR) y el mantenimiento de la sección óptica (confocalidad). Un aumento en el tamaño del agujero de alfiler aumenta el SNR pero disminuye la confocalidad. - Establezca la velocidad de escaneo en 0,25 fotogramas espectrales por segundo, seleccione el icono correspondiente a unidireccional para la dirección de escaneo, introduzca 4 para el recuento y establezca 1024 x 1024 para el tamaño de escaneo.
NOTA: Las señales FRET son débiles y, a menudo, se requiere una velocidad de escaneo lenta. Utilizando una velocidad de escaneo de 0.25, la adquisición de una pila z espectral se completa en ~ 3 minutos. La velocidad de escaneo se puede aumentar o disminuir dependiendo de los fluoróforos utilizados. Por ejemplo, para fluoróforos más brillantes como eGFP, se puede utilizar una velocidad de escaneo más rápida (2 fotogramas / segundo). El número ingresado bajo conteo corresponde a un valor promedio de fotograma de 4, lo que ayuda en la reducción de ruido durante la adquisición de imágenes. Para muestras muy estables y donde el tiempo no es una restricción, se pueden utilizar valores de promedio más altos (hasta 16) para obtener imágenes con SNR mejorado.
- Abra el menú de configuración A1, marque las casillas correspondientes a las líneas láser de 405 nm y 561 nm, seleccione SD para el detector espectral, seleccione 10 para la resolución y 31 para los canales.
- Defina los parámetros de adquisición de z-stack:
NOTA: Es posible que sea necesario ajustar los valores introducidos en los pasos 2.10 para adaptarse a los cambios en la unión o concentración de etiquetas fluorescentes, el tipo de etiqueta, el número de etiquetas utilizadas, la línea celular y otros cambios en la preparación de la muestra que pueden afectar la densidad del etiquetado celular y / o la autofluorescencia celular. Al ajustar los parámetros de adquisición, se debe tener cuidado para lograr un SNR suficiente al tiempo que se minimiza el fotobleaching. Además, al configurar un ensayo FRET espectral, se debe tener cuidado de garantizar que los parámetros funcionen bien en todos los grupos de tratamiento. Es aconsejable realizar un ensayo de cada grupo de tratamiento con la configuración de parámetros propuesta para garantizar que el SNR sea suficiente y que se minimice el fotobleaching.- Abra la ventana de adquisición de ND haciendo clic en ver → control de adquisición → adquisición de ND.
- Introduzca la ruta/destino y un nombre de archivo para guardar el archivo ND en la ventana emergente.
- Marque la casilla correspondiente a la serie z.
- Haga clic en live en la ventana Configuración de A1 Plus. Esto abrirá una ventana de visualización en vivo.
- Ajuste la perilla de enfoque en el microscopio para seleccionar la parte superior de la celda y haga clic en Arriba en la ventana de adquisición de ND para establecer la posición actual como la parte superior.
NOTA: Se sugiere enfocar ligeramente por encima de la parte superior de la celda para asegurarse de que toda la celda se muestree en la serie z. - Ajuste la perilla de enfoque en el microscopio para seleccionar la parte inferior de la celda y haga clic en Inferior en la ventana de adquisición de ND para establecer la posición actual como la parte inferior.
NOTA: Enfoque ligeramente por debajo de la parte inferior de la celda para asegurarse de que se muestrea toda la celda. - Introduzca 1 μm para el tamaño del paso, seleccione la parte superior inferior para la dirección z-scan y haga clic en ejecutar en la ventana Adquisición ND para adquirir una pila z.
NOTA: El tamaño del paso determina el número de cortes z que se adquirirán dependiendo de las ubicaciones superiores e inferiores (es decir, la distancia recorrida). Se seleccionó un tamaño de paso de 1 μm como compromiso entre la velocidad de imagen, el muestreo del eje Z y el fotobleaching. El uso del diámetro de agujero de alfiler confocal de 2,4 UA y el objetivo de inmersión en agua 60x dio como resultado un espesor de sección óptica de 1,73 μm. Por lo tanto, un tamaño de paso de 1 μm está ligeramente por debajo de los criterios de muestreo de Nyquist, pero este es un compromiso que se hizo para reducir el tiempo necesario para adquirir una pila z. Para muestras muy estables, para las cuales la velocidad no es crítica, se puede usar un paso de eje Z más pequeño y posiblemente un diámetro de agujero de alfiler confocal más pequeño para aumentar la resolución del eje z. La parte inferior superior debe producir resultados similares y se puede usar para evaluar cualquier efecto del fotobleaching que pueda ocurrir durante el escaneo z.
- Configure el sistema de enfoque perfecto (PFS) si está disponible:
NOTA: PFS permite que el sistema compense las fluctuaciones en la profundidad focal durante la adquisición de imágenes. Los siguientes pasos se pueden utilizar para configurar PFS, y estos pasos pueden variar ligeramente dependiendo de la versión de la Nikon A1R y la versión de NIS Elements utilizada.- Resalte el modo simétrico definido por el icono de rango en la ventana de adquisición de ND.
- Encienda el botón PFS en la cara frontal del microscopio (asegúrese de que la perilla del espejo dicroico ubicada en la sección debajo de la etapa de muestra esté "en").
- Redefina la parte superior (gire en sentido contrario a las agujas del reloj) y la parte inferior (gire en el sentido de las agujas del reloj) utilizando la perilla en la cara frontal del controlador de desplazamiento PFS.
- Defina una posición z relativa/profundidad z haciendo clic en 'relativo' en la ventana de adquisición de ND.
- Haga clic en la memoria en la cara frontal del microscopio para que el software memoriza la profundidad z relativa.
- Una vez completada la adquisición de la pila z, agregue suavemente el reactivo deseado (forskolina o control del vehículo) con una pipeta y espere 10 minutos.
NOTA: Agregue el reactivo muy suavemente para no perturbar las células o mover la posición de la cámara celular dentro de la etapa XY del microscopio; es útil verificar en una vista en vivo o imagen posterior que el campo de visión no se ha desplazado durante la adición del reactivo. El tiempo de espera de 10 minutos es para que el tratamiento con forskolina surta efecto. Si se utiliza un tratamiento alternativo, es posible que sea necesario ajustar el tiempo de espera. - Después de 10 minutos, cambie el nombre del archivo y haga clic en ejecutar en la ventana de adquisición de ND.
- Repita los pasos 2.11 – 2.13 como se describió anteriormente para al menos 5 cubiertas a fin de lograr resultados suficientes para el análisis estadístico (n = 5 para cada grupo de tratamiento – forskolina y control del vehículo).
- Prepare muestras y muestras en blanco para construir la biblioteca espectral y adquirir imágenes espectrales utilizando configuraciones de adquisición similares a las descritas en los pasos 2.9 y 2.10.
3. Análisis de imágenes
NOTA: Estas imágenes se utilizarán para construir una biblioteca espectral que contenga los espectros puros de todos los miembros finales individuales presentes en el estudio. Los miembros finales de la biblioteca espectral pueden variar de un estudio a otro si se utilizan diferentes fluoróforos. Un procedimiento detallado para construir la biblioteca espectral se proporciona en un archivo de información suplementaria llamado "Biblioteca de File_Spectral suplementaria". Aquí, describimos la exportación de datos a archivos .tiff, la desmezcla espectral lineal, las mediciones de eficiencia FRET, la reconstrucción tridimensional y la estimación de niveles de cAMP. El análisis de imágenes se puede realizar utilizando diferentes plataformas de análisis y programación de imágenes como ImageJ, Python, MATLAB o CellProfiler. En estos estudios, se utilizaron scripts de MATLAB.
- Exportar datos de imagen:
- Cree nuevas carpetas con el mismo nombre de archivo correspondiente a las imágenes espectrales z-stack adquiridas en los pasos 2.13 y 2.14.
NOTA: Los siguientes pasos descritos para exportar datos son específicos para NIS Elements AR versión 4.30.01. Estos pasos pueden variar ligeramente dependiendo de la versión del software. - Haga clic en Archivo, que abrirá una ventana de archivo. Busque y seleccione el archivo de imagen espectral adquirido en el paso 2.12 y haga clic en Abrir.
- Una vez que se cargue el archivo, haga clic en Archivo→ Importar/Exportar→ exportar documento ND.
- En la ventana emergente: busque y seleccione la carpeta creada en el paso 3.1.1, seleccione Formato de imagen etiquetado (TIF) para Tipo de archivo, luego seleccione Imagen mono para cada canal y Mantener profundidad de bits.
NOTA: El prefijo Archivo se generará previamente; cambie este valor por conveniencia. El orden del índice cambiará en función de los canales seleccionados y debe mostrar "z, c" para la indexación de acuerdo con la ubicación del segmento z primero y el número de banda de longitud de onda en segundo lugar. Asegúrese de que los cuadros correspondientes a Aplicar LUT o Insertar superposiciones o Usar nombres de punto no estén seleccionados. - Haga clic en Exportar para exportar los archivos tiff a una carpeta de destino como archivos tiff individuales.
- Repita los pasos 3.1.2 – 3.1.5 para exportar archivos de imagen espectral adquiridos en el paso 2.13.
- Cree nuevas carpetas con el mismo nombre de archivo correspondiente a las imágenes espectrales z-stack adquiridas en los pasos 2.13 y 2.14.
- Desmezcla espectral lineal:
- Abra el software de programación.
NOTA: El script de programación desarrollado a medida para desmezclar imágenes espectrales en bruto se proporciona en el sitio web de BioImaging y BioSystems de la Universidad del Sur de Alabama, en la pestaña Recursos (https://www.southalabama.edu/centers/bioimaging/resources.html). - Abra el archivo etiquetado como "Desmezcla lineal.m" y haga clic en el botón Ejecutar en la barra de herramientas del editor.
- Busque y seleccione la carpeta que contiene la secuencia de archivos *.tif exportada generada por el software NIS Elements.
- Haga clic en Aceptar para continuar, que abrirá una nueva ventana llamada Longitud de onda y Z-Slice.
- Copie el nombre de archivo del primer archivo (sin z-slice y número de canal) en la carpeta seleccionada en el paso 3.2.4 y péguelo en el primer paso del cuadro de diálogo con la etiqueta "Introduzca el nombre de la imagen".
- Ingrese el número de canales en el segundo paso etiquetado como "Ingrese el número de bandas de longitud de onda", el número de cortes z en el tercer paso etiquetado como "Ingrese el número de cortes Z" y haga clic en Aceptar.
NOTA: El número de bandas de longitudes de onda puede cambiar si se realizan cambios en la configuración de adquisición, como ajustar el rango de longitud de onda o el tamaño del paso de longitud de onda. El número de cortes Z también puede cambiar dependiendo de la altura de la celda. - Busque y seleccione el archivo de longitud de onda llamado "Wavelength.mat" en la ventana emergente etiquetada "Seleccione el archivo de información de longitud de onda" y haga clic en abrir.
- Busque y seleccione el archivo "Library.mat" en la nueva ventana emergente etiquetada "Seleccionar el archivo de biblioteca espectral", haga clic en abrir y espere hasta que finalice la desmezcla de los segmentos.
NOTA: El archivo Library.mat es un archivo que contiene espectros puros para cada fluoróforo de miembro final junto con autofluorescencia celular y firmas espectrales de fondo. En este caso, los fluoróforos de los miembros finales incluyen Turquesa, Venus y DRAQ5. Las firmas espectrales de fondo incluyen autofluorescencia celular o matricial, fluorescencia de recubre y difracción de la cubierta. El archivo Wavelength.mat es un archivo que contiene información del canal de longitud de onda utilizada para adquirir la imagen espectral. Un archivo de biblioteca de ejemplo y un archivo de longitud de onda están disponibles en el sitio web de Bioimaging and Biosystems (consulte la nota en 3.2.1). Para obtener más información sobre cómo generar archivos de biblioteca espectral y longitud de onda, consulte el archivo de información suplementaria denominado "Biblioteca de File_Spectral suplementaria". Las imágenes no mezcladas correspondientes a cada z-slice se guardarán en la carpeta llamada "Unmixed" creada durante el proceso de desmezcla dentro de la carpeta que se seleccionó en el paso 3.2.3.
- Abra el software de programación.
- Cálculo de la eficiencia de FRET:
- Abra el script de programación llamado "multiFRRCF.m" y haga clic en ejecutar.
NOTA: Este archivo de programación está disponible en el sitio web de Bioimaging and Biosystems de la Universidad del Sur de Alabama (consulte la nota en el paso 3.2.1). - Introduzca el número de ensayos experimentales que desea analizar en el cuadro de diálogo emergente denominado "cuántas carpetas desea volver a utilizar" y haga clic en Aceptar.
NOTA: Los datos de imagen de cada experimento deben guardarse en una carpeta de imágenes sin mezclar separada. Este paso simplemente permite que el código de análisis se repita en muchas carpetas como un paso que ahorra tiempo. - Busque y seleccione las carpetas sin mezclar y haga clic en Aceptar.
NOTA: El número de veces que se abre la ventana emergente "Buscar carpeta" depende del número especificado en el cuadro de diálogo "Cuántas carpetas se van a cambiar" en el paso anterior. Busque y seleccione las carpetas una tras otra. - En la nueva ventana emergente, ingrese la siguiente información en los cuadros respectivos: el factor de escala es 12.4, el umbral es 5.6, la frecuencia X, Y y Z son 5, 5 y 1 respectivamente, y el algoritmo de suavizado es gaussiano.
NOTA: El factor de escala es un valor en píxeles/μm y se utilizará para escalar el muestreo de dirección Z al de la dirección XY. El factor de escala se obtiene del tamaño del píxel de la imagen, que generalmente se proporciona como metadatos en la imagen para la mayoría de los sistemas de microscopio confocal. Por ejemplo, si la imagen se adquiere con un espaciado de 0,08 μm/píxeles, el factor de escala debe ser de 12,5 píxeles/μm. El valor de umbral se utilizará para establecer el umbral de las imágenes y generar una máscara binaria de la celda. Creamos una lista de valores óptimos basados en la intensidad de la imagen donante+aceptor. Utilice 4,5 como valor de umbral si la imagen tiene una intensidad brillante donante+aceptor y un fondo bajo, un valor entre 5,6 y 6,5 para las imágenes que solo tienen una intensidad moderada donante+aceptor y/o un fondo más alto, y un valor de 7,5 y superior para las imágenes que tienen una intensidad donante+aceptor inferior al fondo. El valor de frecuencia corresponde al intervalo, en número de píxeles, en el que se realiza el corte en los pasos siguientes. Por ejemplo, si la profundidad Z de la celda es de 17 μm con un tamaño de paso de 1 μm y se utiliza un factor de escala de 12,5 píxeles/μm en la dirección XY, entonces la profundidad del conjunto de datos de imagen en 3 dimensiones se volverá a muestrear a 212 píxeles (dirección Z). Según el valor de frecuencia Z introducido (por ejemplo, 1 píxel), el conjunto de datos de imagen en 3 dimensiones se volverá a dividir comenzando en la parte superior del conjunto de datos de imagen y luego moviéndose en incrementos de 1 píxel hacia abajo. Esto da como resultado 212 imágenes recortadas. Si se ingresara un intervalo de valor de frecuencia mayor para la frecuencia Z, se generarían menos imágenes recortadas. Las imágenes redimensionadas se guardan en pasos posteriores. - Haga clic en ejecutar y espere hasta que se realicen todas las mediciones fret y el reslicing.
Nota : se crea una carpeta independiente dentro del directorio primario en la que se guardan las imágenes de eficiencia FRET en escala de grises redimensionadas y las imágenes de eficiencia FRET coloreadas (un mapa de color aplicado). Por ejemplo, todas las imágenes FRET en escala de grises y mapa de colores redimensionadas en la dirección X (plano YZ) se guardan en una carpeta llamada "Resliced_XFRET". - Repita el análisis con configuraciones similares para todos los experimentos: antes y después de los tratamientos con forskolina y los controles del vehículo.
NOTA: Los pasos mencionados en la sección 3.3 describen los valores que se deben introducir para el script de programación de análisis FRET personalizado para generar datos de imagen FRET en 3 dimensiones. Sin embargo, este script ejecuta varias operaciones mientras se ejecuta, que incluyen: cargar datos de imagen, crear pilas de imágenes, suavizar, cálculos de eficiencia FRET, crear y aplicar una máscara de borde de celda, reconstrucción de imagen en 3 dimensiones, reslicing de imágenes tridimensionales a intervalos especificados (frecuencias), aplicación de un mapa de colores para visualizar los cambios fret y guardar los datos de imagen redimensionados en el mismo directorio. Se han incluido detalles adicionales como comentarios en el script del programa.
- Abra el script de programación llamado "multiFRRCF.m" y haga clic en ejecutar.
4. Mapeo de la eficiencia fret a los niveles de cAMP
- Abra el archivo de programación llamado 'Mapping_FRET Efficiency_to_cAMP_concentration.m' y haga clic en ejecutar en la ventana principal.
NOTA: El archivo está disponible en el sitio web de BioImaging y BioSystems (consulte la nota en 3.2.1). Este archivo lee imágenes de eficiencia FRET en escala de grises y las convierte a niveles cAMP basados en una curva característica. Esta curva característica utiliza una relación cAMP-a-FRET documentada en la literatura15,36 que se describe mediante la ecuación de Hill (la tercera ecuación que se muestra a continuación). Sin embargo, Kd de la sonda en células intactas es difícil de estimar y hemos asumido que es 1 μM en nuestros cálculos. Por lo tanto, los resultados se muestran en función de Kd. (es decir, [cAMP] = x* Kd). Las ecuaciones utilizadas para medir la eficiencia de FRET y mapear FRET a niveles de cAMP se muestran a continuación: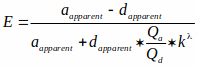
Donde E es la eficiencia FRET, y unaaparente y daparente son intensidades de píxeles no mezcladas de imágenes aceptoras y donantes, respectivamente.
Qa y Qd son rendimientos cuánticos de aceptor y donante. Nótese que Q a y Qd se cancelan cuando la ecuación para kλ se incorpora en la ecuación de eficiencia FRET, kλ es un factor de corrección: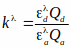
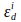 y
y 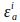 son coeficientes de extinción de donante y aceptor en la longitud de onda de excitación del donante, i (405nm).
son coeficientes de extinción de donante y aceptor en la longitud de onda de excitación del donante, i (405nm).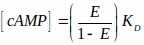
E es la eficiencia FRET y KD = constante de disociación = 1 μM. - Navegue y seleccione la primera imagen FRET en escala de grises (guardada en el paso 3.3.5) y haga clic en Aceptar.
- Abra las imágenes FRET/cAMP para inspeccionar la distribución de señales cAMP en tres dimensiones.
Resultados
Este protocolo describe el uso de imágenes y enfoques de análisis FRET hiperespectrales para medir gradientes de cAMP en tres dimensiones espaciales en células vivas. Hay varios pasos clave involucrados en la generación de estos resultados, para los cuales se requiere una atención cuidadosa al analizar y cuantificar los datos. Estos pasos clave incluyen la construcción de una biblioteca espectral apropiada, la desmezcla espectral de fondo, el umbral para identificar los bordes de las celdas y los cálculos de efici...
Discusión
El desarrollo de biosensores FRET ha permitido la medición y visualización de señales de nucleótidos cíclicos en células individuales, y existe una gran promesa para visualizar eventos de señalización subcelular13,22,37,38. Sin embargo, el uso de biosensores FRET presenta varias limitaciones, incluyendo las características de baja señal a ruido de muchos reporteros FRET basados en pro...
Divulgaciones
Los doctores Leavesley y Rich revelan interés financiero en una empresa de nueva creación, SpectraCyte, LLC, que se formó para comercializar tecnologías de imágenes espectrales. Sin embargo, todos los procedimientos descritos en este protocolo se llevaron a cabo utilizando productos disponibles comercialmente no asociados con SpectraCyte, LLC.
Agradecimientos
Los autores desean agradecer al Dr. Kees Jalink (Instituto del Cáncer de los Países Bajos y centro van Leeuwenhoek de Microscopía Avanzada, Ámsterdam, Países Bajos) por proporcionarnos el biosensor H188 cAMP FRET y a Kenny Trinh (Facultad de Ingeniería, Universidad del Sur de Alabama) por su ayuda técnica para reducir el tiempo necesario para ejecutar nuestros scripts de programación desarrollados a medida.
Los autores desean reconocer las fuentes de financiación: American Heart Association (16PRE27130004), National Science Foundation; (1725937) NIH, S100D020149, S10RR027535, R01HL058506, P01HL066299).
Materiales
| Name | Company | Catalog Number | Comments |
| Attofluor Cell Chamber | Invitrogen | A7816 | Attofluor contains steel cell chambers and a rubber O-ring. Cell chamber holds the coverslip and O-ring provides a lock in mechanism to hold the buffer in cell chamber with out leakage |
| Dimethyl Sulfoxide (DMSO) | Fisher Scientific | BP231-100 | Solvent used to prepare stock solution forskolin. |
| DRAQ5 Fluoroscent Probe Solution | Thermo Scientific | 62252 | Nuclear label |
| Dulbecco Modified Eagle Medium (DMEM) | Gibco | 11965-092 | Contains nutrients and growth factors for the cells to grow and divide in the culture dishes. |
| Fetal Bovine Serum (FBS) | Sigma | F6178 | Growth factor suppliment that is added to culture medium, DMEM |
| Forskolin | Sigma | F3917 | Adenyly cyclase activator. |
| H188 Cyclic AMP FRET biosensor | Netherland Cancer Institute, Dr. K. Jalink | Gift | Plasmid encoding Turquoise (donor fluorophore), Venus (acceptor fluorophore), and binding domain obtained from Epac. |
| Image J | image.net | Free download | Another image processing platform used to extact spectral information and image processing. |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used to measure illumination intensity of the laser line at different laser intensities (?). |
| Laminin Mouse Protein, Natural | Invitrogen | 23017-015 | Coverslips are coated with laminin and this helps in cell attachment, growth and motility of the cell. |
| Lipofectamine 3000 Transfection Kit | Invitrogen | L3000-015 | Transfection reagent used to transfect cells with H188 FRET biosensor |
| MATLAB | Mathworks | R2019a | Image processing operations (linear unmixing and FRET efficiency calculations) are performed by writing custom programs in MATLAB programming environment |
| Nikon A1R confocal microscope | Nikon Instruments | Nikon A1R | Spectral image acquisition is performed using confocal microscope. |
| Nikon Elements Software | Nikon Instruments | Software dongle | used to export and handle nd2 image files (multidimensional image files) that are aquired using Nikon A1R |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| PBS pH 7.4 (1X) | Gibco | 10010-023 | coomonly used buffer suring cell culture |
| Pulmonary Microvascular Endothelial Cells (PMVECs) | In house - Cell culture core, Univeristy of South Alabama | Isolated from Rat pulmonary microvasculature | PMVECs form inner lining of a blood vessel. |
| Penicillin-Streptomycin (10,000 U/ml) | Gibco | 15140-122 | antibiotics are added to culture medum to prevent contamination of the cells. |
| Pre-Cleaned Gold Seal Micro Slides | Clay Adams | 3010 | Microscope slides used for cell fixation |
| ProLong Diamond Antifade Mounting Media | Invitrogen | P36961 | If samples are fixed using antifade mountant, then the later protects fluoroscent dyes and chromophores from fading. |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral response of the light source (?) |
| Trypsin-EDTA (0.25%) | Gibco | 25200-056 | Digests the protein-protein bond between the cell and cell matrix and helps to disscociate and lift the cells during cell plating. |
| Tyrodes Buffer | Made in-house | Made in-house | Tyrodes buffer is used to make working solutions and to maintain cells in aqueous solution during image acquisition. |
| 6 Well Cell Culture Plate | Corning | 3506 | Laminin coated coverslips are placed in 6-well culture dish (one coverlisps/well). Cells along with medium is added into each well. |
| 25 mm Round Microscope Cover Slips | Fisher Scientific | 12545102 | Cells were grown on round glass coverslips |
| 60X Ojective | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | water immersion and commonly used objective for cells |
Referencias
- Corbin, J. D., Sugden, P. H., Lincoln, T. M., Keely, S. L. Compartmentalization of adenosine 3':5'-monophosphate and adenosine 3':5'-monophosphate-dependent protein kinase in heart tissue. The Journal of Biological Chemistry. 252, 3854-3861 (1977).
- Terrin, A., et al. PGE1 stimulation of HEK293 cells generates multiple contiguous domains with different [cAMP]: role of compartmentalized phosphodiesterases. The Journal of Cell Biology. 175, 441-451 (2006).
- Bacskai, B. J., et al. Spatially resolved dynamics of cAMP and protein kinase A subunits in Aplysia sensory neurons. Science. 260, 222-226 (1993).
- Iancu, R. V., Ramamurthy, G., Harvey, R. D. Spatial and temporal aspects of cAMP signalling in cardiac myocytes. Clinical and Experimental Pharmacology & Physiology. 35, 1343-1348 (2008).
- Brunton, L. L., Hayes, J. S., Mayer, S. E. Functional compartmentation of cyclic AMP and protein kinase in heart. Advances in Cyclic Nucleotide Research. 14, 391-397 (1981).
- Hohl, C. M., Li, Q. Compartmentation of cAMP in adult canine ventricular myocytes. Relation to single-cell free Ca2+ transients. Circulation Research. 69, 1369-1379 (1991).
- Rich, T. C., et al. A uniform extracellular stimulus triggers distinct cAMP signals in different compartments of a simple cell. Proceedings of the National Academy of Sciences of the United States of America. 98, 13049-13054 (2001).
- Sayner, S. L., Alexeyev, M., Dessauer, C. W., Stevens, T. Soluble adenylyl cyclase reveals the significance of cAMP compartmentation on pulmonary microvascular endothelial cell barrier. Circulation Research. 98, 675-681 (2006).
- Rich, T. C., Tse, T. E., Rohan, J. G., Schaack, J., Karpen, J. W. In vivo assessment of local phosphodiesterase activity using tailored cyclic nucleotide-gated channels as cAMP sensors. The Journal of General Physiology. 118, 63-78 (2001).
- Blackman, B. E., et al. PDE4D and PDE4B function in distinct subcellular compartments in mouse embryonic fibroblasts. Journal of Biological Chemistry. 286, 12590-12601 (2011).
- Sayner, S. L., et al. Paradoxical cAMP-induced lung endothelial hyperpermeability revealed by Pseudomonas aeruginosa ExoY. Circulation Research. 95, 196-203 (2004).
- Klarenbeek, J., Jalink, K. Detecting cAMP with an EPAC-based FRET sensor in single living cells. Methods in Molecular Biology. 1071, 49-58 (2014).
- Surdo, N. C., et al. FRET biosensor uncovers cAMP nano-domains at β-adrenergic targets that dictate precise tuning of cardiac contractility. Nature Communications. 8, 15031 (2017).
- Ponsioen, B., et al. Detecting cAMP-induced Epac activation by fluorescence resonance energy transfer: Epac as a novel cAMP indicator. EMBO Reports. 5, 1176-1180 (2004).
- Vogel, S. S., Thaler, C., Koushik, S. V. Fanciful FRET. Science's STKE. 2006, (2006).
- Clegg, R. M. The History of FRET: From conception through the labors of birth. Reviews in Fluorescence. 3, (2006).
- Giepmans, B. N. G., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Manzella-Lapeira, J., Brzostowski, J. A. Imaging protein-protein interactions by Förster resonance energy transfer (FRET) microscopy in live cells. Current Protocols in Protein Science. 93, 58 (2018).
- Cooper, D. M. F., Mons, N., Karpen, J. W. Adenylyl cyclases and the interaction between calcium and cAMP signalling. Nature. 374, 421-424 (1995).
- Sassone-Corsi, P. Coupling gene expression to cAMP signalling: role of CREB and CREM. The International Journal of Biochemistry & Cell Biology. 30, 27-38 (1998).
- Rebhun, L. I. Cyclic nucleotides, calcium, and cell division. International Review of Cytology. 49, 1-54 (1977).
- Klarenbeek, J., Goedhart, J., Van Batenburg, A., Groenewald, D., Jalink, K. Fourth-generation Epac-based FRET sensors for cAMP feature exceptional brightness, photostability and dynamic range: characterization of dedicated sensors for FLIM, for ratiometry and with high affinity. PLOS ONE. 10, 0122513 (2015).
- Leavesley, S. J., Rich, T. C. FRET: signals hidden within the noise. Cytometry Part A. 85, 918-920 (2014).
- Rich, T. C., Webb, K. J., Leavesley, S. J. Can we decipher the information content contained within cyclic nucleotide signals. The Journal of General Physiology. 143, 17-27 (2014).
- Annamdevula, N. S., et al. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Leavesley, S. J., Britain, A. L., Cichon, L. K., Nikolaev, V. O., Rich, T. C. Assessing FRET using spectral techniques. Cytometry Part A. 83, 898-912 (2013).
- Leavesley, S. J., Rich, T. C. Overcoming limitations of FRET measurements. Cytometry Part A. 89, 325-327 (2016).
- Fink, D. J. Monitoring Earcths Resources from Space. Technology Review. 75, 32-41 (1973).
- Goetz, A. F. H., Vane, G., Solomon, J. E., Rock, B. N. Imaging Spectrometry for Earth Remote Sensing. Science. 228, 1147-1153 (1985).
- Bücherl, C. A., Bader, A., Westphal, A. H., Laptenok, S. P., Borst, J. W. FRET-FLIM applications in plant systems. Protoplasma. 251, 383-394 (2014).
- Chen, Y., Mauldin, J. P., Day, R. N., Periasamy, A. Characterization of spectral FRET imaging microscopy for monitoring nuclear protein interactions. Journal of Microscopy. 228, 139-152 (2007).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Letters. 531, 245-249 (2002).
- Griswold, J. R., Annamdevula, N., Deal, J., Rich, T., Leavesley, S. Estimating FRET Efficiency using Excitation-Scanning Hyperspectral Imaging. Biophysical Journal. 112, 586 (2017).
- Favreau, P. F., et al. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19, 046010 (2014).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67, 139-151 (2004).
- Thaler, C., Koushik, S. V., Blank, P. S., Vogel, S. S. Quantitative multiphoton spectral imaging and its use for measuring resonance energy transfer. Biophysical Journal. 89, 2736-2749 (2005).
- Agarwal, S. R., et al. Compartmentalized cAMP signaling associated with lipid raft and non-raft membrane domains in adult ventricular myocytes. Frontiers in Pharmacology. 9, 332 (2018).
- Johnstone, T. B., Agarwal, S. R., Harvey, R. D., Ostrom, R. S. cAMP signaling compartmentation: Adenylyl cyclases as anchors of dynamic signaling complexes. Mol Pharmacol. , (2017).
- Zhang, J., Li, H., Chai, L., Zhang, L., Qu, J., Chen, T. Quantitative FRET measurement using emission-spectral unmixing with independent excitation crosstalk correction. Journal of Microscopy. 257, 104-116 (2015).
- Zhang, J., Lin, F., Chai, L., Wei, L., Chen, T. IIem-spFRET: improved Iem-spFRET method for robust FRET measurement. Journal of Biomedical Optics. 21, 105003 (2016).
- Levy, S., et al. SpRET: highly sensitive and reliable spectral measurement of absolute FRET efficiency. Microscopy and Microanalysis. 17, 176-190 (2011).
- West, S. J., et al. Hyperspectral Measurements Allow Separation of FRET Signals from Non-Uniform Background Fluorescence. Biophysical Journal. 112, 453 (2017).
- Annamdevula, N. S., et al. An approach for characterizing and comparing hyperspectral microscopy systems. Sensors. 13, 9267-9293 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados