このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
4次元(x、y、z、λ)ハイパースペクトルFRETイメージングと解析を用いた生体細胞における3次元cAMP分布の測定
要約
Fӧrster共鳴エネルギー伝達(FRET)ベースのセンサの信号対雑音比(SNR)が低いため、cAMP信号の測定は特に3つの空間次元において困難でした。ここでは、3つの空間次元でcAMP分布を測定できるハイパースペクトルFRETイメージングおよび解析方法論について述べる。
要約
サイクリックAMPは、細胞および生理活性の広い範囲に関与している第二のメッセンジャーです。いくつかの研究は、cAMP信号が区画化され、区画化がcAMPシグナル経路内のシグナル特異性に寄与することを示唆している。Fӧrster共鳴エネルギー伝達(FRET)ベースのバイオセンサーの開発は、細胞内のcAMP信号を測定し、可視化する能力を高めています。ただし、これらの測定値は、多くの場合、2 つの空間次元に限定され、データの誤解釈につながる可能性があります。これまで、3空間次元(x、y、z)でのcAMP信号の測定は非常に限られており、本質的に低信号対雑音比(SNR)を示すFRETセンサの使用に関する技術的な制限によりあります。さらに、従来のフィルタベースのイメージング手法は、スペクトルクロストーク、限定的な信号強度、自己蛍光などのさまざまな要因により、局在化した細胞下領域でcAMP信号を正確に測定するのには効果的でないことがよくあります。これらの制限を克服し、FRETベースのバイオセンサーを複数の蛍光ホルで使用できるようにするために、FRET効率を計算するためのスペクトル特異性と、FRET信号を同時交流自発および/または追加の蛍光標識からのシグナルからスペクトル的に分離する能力を提供するハイパースペクトルFRETイメージングおよび分析アプローチを開発しました。ここでは、ハイパースペクトルFRETイメージングを実装するための方法論と、スペクトルアンミックスを行うためにアンダーサンプリングもオーバーサンプリングもしない適切なスペクトルライブラリを構築する必要性を提示する。この方法論は、肺微小血管内皮細胞(PMVEC)における3次元cAMP分布の測定に用いるが、この方法論は、細胞タイプの範囲におけるcAMPの空間分布を研究するために使用することができる。
概要
環状アデノシン一リン酸(cAMP)は、細胞分裂、カルシウム流入、遺伝子転写、シグナル伝達を含む主要な細胞および生理学的プロセスに関与する第二のメッセンジャーである。証拠の成長体は、シグナル特異性が達成される細胞内のcAMPコンパートメントの存在を示唆しています1,2,3,4,5,6,7.最近まで、cAMP区画化は、異なるG結合受容体アゴニスト8、9、10、11によって誘発される明確な生理学的または細胞的効果に基づいて推測された。より最近では、FRETベース蛍光イメージングプローブは、細胞12、13、14におけるcAMP信号の直接測定および観察のための新しいアプローチを提供している。
フェルスター共鳴エネルギー移動(FRET)は、分子が近接15,16の場合にドナー分子とアクセプター分子間でエネルギー移動が起こる物理的現象である。FRETベース蛍光指標の開発に伴い、この物理現象は、タンパク質相互作用17、タンパク質共局在18、Ca+2シグナル伝達19、遺伝子発現20、細胞分裂21および環状ヌクレオチドシグナル伝達を研究する生物学的応用において使用されてきた。FRET ベースの cAMP 指標は、通常、cAMP 結合ドメイン、ドナーフルオロフォアおよびアクセプターフルオロフォア22から構成されます。例えば、この方法論で使用されるH188 cAMPセンサ12,22は、Epacから得られたcAMP結合ドメインから成り、ターコイズ(ドナー)と金星(アクセプター)フルオロフォアの間に挟まれる。基礎条件(非結合)では、ターコイズとヴィーナスは、蛍光色素間でFRETが発生するような向きである。結合ドメインにcAMPを結合すると、ターコイズとヴィーナスが離れて移動するような立体構造変化が起こり、FRETが減少する。
FRETベースのイメージングアプローチは、細胞内のcAMP信号を調査し、可視化するための有望なツールを提供します。しかし、現在のFRETベースの顕微鏡イメージング技術は、多くの場合、細胞内空間的明瞭度でFRETを測定するのに十分な信号強度を達成するのに部分的にしか成功していない。これは、多くのFRETレポーターの信号強度の制限、FRET効率の変化を正確に定量するために必要な高い精度、および細胞自家蛍光23、24などの交易因子の存在を含むいくつかの要因によるものである。その結果、多くの場合、弱いSNRに悩まされているFRET画像であり、FRETにおける細胞内変化の視覚化は非常に困難になります。さらに、空間的に局在したcAMP信号の調査は、ほぼ2つの空間次元でほぼ独占的に行われ、軸方向のcAMP分布はめったに25と考えられてはなされていない。これは、SNR が低いために cAMP 勾配を 3 つの空間次元で測定および視覚化する機能が妨げられたためです。低SNRを有するFRETセンサの使用の限界を克服するために、我々は、単一細胞25、26、27でFRETを測定するためのハイパースペクトルイメージングおよび分析アプローチを実施した。
ハイパースペクトルイメージングアプローチは、衛星画像28,29に存在する地上の物体を区別するためにNASAによって開発された。これらの技術は、その後、蛍光顕微鏡観察分野30に翻訳され、いくつかの商業的共焦点顕微鏡システムがスペクトル検出器を提供する。従来の(非スペクトル)蛍光イメージングでは、サンプルはバンドパスフィルタまたはレーザーラインを使用して励起され、発光は2番目のバンドパスフィルタを使用して収集され、フッ素フォロアのピーク発光波長に合わせて選択されることがよくあります。対照的に、ハイパースペクトルイメージングアプローチは、特定の波長間隔で蛍光発光26、31、32または励起33、34のいずれかの完全なスペクトルプロファイルをサンプリングすることを求める。我々のこれまでの研究では、ハイパースペクトルイメージングおよび解析アプローチは、従来のフィルタベースのFRETイメージング技術26と比較して、細胞内のFRET信号の定量化を改善できることを示した。ここでは、4次元(x、y、z、λ)ハイパースペクトルFRETイメージングおよび解析を行い、cAMP分布を3つの空間次元で測定および可視化する方法論を提示する。これらのアプローチは、単一細胞25におけるアゴニスト誘発cAMP空間勾配の可視化を可能にした。興味深いことに、アゴニストによっては、cAMP勾配が細胞内で明らかである可能性がある。ここで提示される方法論は、FRET測定の精度を向上させるために、不均一な背景および細胞自家蛍光のスペクトルの非混合を利用する。この方法論は、cAMP FRETバイオセンサーを用いた肺微小血管内皮細胞(PMVEC)で実証されているが、この方法論は、代替FRETレポーターまたは代替細胞株で使用するために容易に変更することができる。
プロトコル
このプロトコルは、南アラバマ大学の制度的動物の世話と使用委員会によって承認された手順に従います.
1. イメージング用の細胞、サンプル、試薬の準備
- ラット肺微小血管内皮細胞(PMVEC)を先に説明した35を単離する。
注:細胞は、100 mm細胞培養皿上のサウスアラバマ大学モバイル、ALの細胞培養コアによって単離され、培養されました。 - 25 mm丸ガラスカバーリップ上に分離したPMVECをシードし、細胞が少なくとも80%の合流性(少なくとも24時間)になるまで37°Cでインキュベーターで成長させます。
注:細胞と細胞の種類は研究によって異なる可能性があるため、細胞特異的な細胞培養手順は、細胞を播種および成長させるために従う必要があります。これらの研究で使用される細胞の播種および培養プロトコルは、「補足的なFile_Cell培養とトランスフェクション」という名前のファイルの補足情報として利用可能です。 - FRETバイオセンサーを用いたトランスフェクトPMVECを、37°Cで48時間インキュベートします。
注: PMVECs をトランスフェクトするプロトコルは、「補足的なFile_Cell文化とトランスフェクション」という補足情報ファイルにも記載されています。 - イメージングの日には、水浴中の37°Cにタイローデのバッファーを温めます。
注意: タイロードのバッファーは、145 mM NaCl、4 mM KCl、10 mM HEPES、10 mMグルコース、1 mM MgCl2および 1 mM CaCl2 から構成されています。 - トランスフェクションした細胞を含むカバースリップをセルチャンバーに取り付け、漏れを防ぐために取り付けガスケットで上部を固定します。
- 繊細な作業ワイプを使用してカバースリップの底を拭き取り、余分なメディアや接着細胞を洗浄します。
- 800 μL の作業バッファーと 4 μL の 5 mM 核ラベルをセルチャンバーに加え、5 ~ 10 秒間静かに揺れます。
注:細胞室に取り付けられたカバースリップにバッファーまたは試薬溶液を追加する場合は、接着細胞を外さないように、穏やかに細胞室の側面に溶液を追加してください。5 mMの核ラベルを800 μLのバッファに4 μL加えると、25 μM の最終濃度の核ラベルが作成されます。HEK293細胞のような緩やかに付着した細胞の場合、最初にバイアルに核ラベルとバッファを混ぜ、次に細胞室に取り付けられたカバースリップに加えます。これにより、カバースリップから細胞を持ち上げないようにします。 - セルチャンバーをアルミホイルで覆い、光から保護し、室温で10分間インキュベートします。
- 試薬の準備:199 μLのバッファーに50 mMフォルスコリンを1 μL加えます。これは800 μLのバッファーで調製された細胞に加えた場合50 μMのフォルスコリンの最終濃度を生成します。199 μL のバッファで 1 μL の DMSO を車両制御として使用できるように準備する必要があります。
注: これらの研究で, フォルスコリンは、cAMP の生産を刺激するアデニルシクラーゼ活性化剤として使用されます。.必要に応じて、この方法論は、アデニルシクラーゼ、ホスホジエステラーゼなどを刺激または阻害するための代替試薬による治療を容易に変更することができる。
2. 画像取得
- スペクトル検出器を搭載した共焦点顕微鏡を使用してください。
注:ここで概説されているすべての画像取得手順は、市販のニコンA1R顕微鏡システムを使用して開発されました。代替スペクトル顕微鏡を使用する場合、これらのステップを調整する必要があります。安定した動作条件に達するために、実験開始の少なくとも30分前にすべての機器の電源が入っていることを確認してください。 - 60倍の水浸漬目的を選択し、目的に水滴を追加します。
メモ: 高解像度のライブセルイメージングの場合は、高い開口の目的を使用することをお勧めします。これらの研究で使用される目的については、資料一覧を参照してください。 - 積まれた細胞チャンバー(ステップ1.7から)を顕微鏡のステージに置きます。
- 顕微鏡の右側にあるフィルターノブを調整して、DFT(DAPI/FITC/TRITC)フィルタセットを選択します。
- 眼球を使用して蛍光ワイドフィールドモードで顕微鏡を操作し、cAMP FRETセンサーを発現する細胞を含む視野を選択します。
注:選択したセルのドナーまたはアクセプター発光ピーク波長におけるFRET信号の平均強度が、発現細胞のない領域の少なくとも100の強度単位(A.U.)または少なくとも4倍のベースライン信号であることを確認してください。これは、NISエレメントソフトウェアで利用可能なスペクトルプロファイルビューアを使用して確認することができます。シグナルが良いセルを探す場合は、過度に明るいセルを破棄することをお勧めします(危険にさらされる可能性があります)。 - NISソフトウェアを開き、共焦点モードに切り替え、レーザーインターロックボタンのロックを解除し、 ライブをクリックします。
- フォーカスノブを使用して、画面上のプレビューを見てセルに焦点を当てます。
- 以下に説明するとおり、ソフトウェアでデバイス、取得、および Z スタックの設定を構成します。
- 取得設定:
注意:カメラとデバイスの取得設定は、以前に取得した画像を使用して適用することができます。イメージを開き、右クリックして [ カメラ設定を再利用]を選択します。- A1 設定メニューを開き、405 nm および 561 nm レーザーラインに対応するチェックボックスをオンにし、スペクトル検出器の SD を選択し、解像度として 10、チャンネルに 31 を選択します。
注:A1設定メニューは、A1プラス設定ウィンドウの左上隅に小さな歯車アイコンとして表示されます。405 nmレーザーはドナー励起に使用され、561 nmレーザーは核ラベル励起に使用される。 - 始点と終了波長の値を選択して、波長範囲(410~730 nm)を設定します。
- A1 設定メニューのビン分割/スキップアイコンをクリックし、15 の番号が付いたボックスを選択してから、A1 設定メニューで [OK] をクリックします。
注:これは、561 nmの励起レーザに対応する波長チャネルを除去することです(これは通常、15番目 の波長チャネルです)。この波長バンドを使用して、スペクトルアーティファクトを生み出す人工的に低い信号を避けることは重要です。このバンドでは、レーザー損傷から保護するために検出器要素をカバーする機械的な指のために、信号が低くなります。 - 405 nm および 561 nm レーザーのレーザー強度を 8% と 2% に設定し、それぞれ 149 の Si Hv (検出器ゲイン) と 2.4 風通しのディスクユニット (AU) のピンホール半径を設定します。
注:レーザー強度は、機器の年齢とレーザーの状態に応じて調整する必要があります。異なるサンプルまたは実験群間でレーザー強度を調整する場合、レーザー強度の同じ比率(例えば、8:2)を維持することが重要です。さらに、急速な光の漂白を作成するほど明るくないレーザー強度を選択することが重要です。検出器のゲインは、検出器のノイズを最小限に抑えながら信号強度を最大化するように調整する必要があります。これらの研究のために、149の利益が使用された。ピンホールサイズ2.4 AUは、十分な信号対雑音比(SNR)を持つ画像を取得することと、光断面(共焦点性)を維持することとのバランスとして選択されました。ピンホールサイズを大きくするとSNRは増加しますが、共焦点性は低下します。 - スキャン速度を 1 秒あたり 0.25 スペクトル フレームに設定し、スキャン方向の単方向に対応するアイコンを選択し、カウントに 4 と入力し、スキャン サイズに 1024 x 1024 を設定します。
注: FRET 信号は弱く、低速スキャン速度が必要な場合が多いです。0.25のスキャン速度を使用して、スペクトルzスタックの取得は約3分で完了する。スキャン速度は、使用される蛍光器に応じて増加または減少させることができます。例えば、eGFPのような明るい蛍光色素の場合、スキャン速度が速くなる(2フレーム/秒)が使用できます。カウントの下で入力された数は、フレーム平均値 4 に対応し、画像取得時のノイズ低減に役立ちます。非常に安定したサンプルで、時間が制約ではない場合、より高い平均値(最大16)を使用して、SNRが改善された画像を取得できます。
- A1 設定メニューを開き、405 nm および 561 nm レーザーラインに対応するチェックボックスをオンにし、スペクトル検出器の SD を選択し、解像度として 10、チャンネルに 31 を選択します。
- z スタック取得パラメータを定義します。
注:ステップ2.10で入力した値は、蛍光標識の結合または濃度、ラベルの種類、使用するラベルの数、細胞株、および細胞標識密度および/または細胞自家蛍光に影響を与える可能性のあるサンプル調製のその他の変化の変化に対応するために調整する必要があります。取得パラメータを調整する場合は、フォトブリーチを最小限に抑えながら、十分なSNRを達成するように注意する必要があります。さらに、スペクトルFRETアッセイを構成する際には、すべての治療群でパラメータがうまく機能するように注意する必要があります。SNRが十分であり、フォトブリーチが最小限に抑ことを確認するために、提案されたパラメータ設定を用いて各治療グループの試験を実行することをお勧めします。- ND 取得コントロールの 表示 をクリックして ND 取得 ウィンドウ →→開きます。
- パス/宛先とファイル名を入力して、NDファイルをポップアップウィンドウに保存します。
- z 系列に対応するボックスをオンにします。
- A1 Plus設定ウィンドウで ライブ をクリックします。ライブ表示ウィンドウが開きます。
- 顕微鏡のフォーカスノブを調整してセルの上部を選択し、ND取得ウィンドウで [上 ]をクリックして現在位置をトップに設定します。
注: セルの上部に少し焦点を合わせると、すべてのセルが z 系列でサンプリングされるようにすることをお勧めします。 - 顕微鏡のフォーカスノブを調整してセルの底部を選択し、ND取得ウィンドウの [下部 ]をクリックして現在位置を底部に設定します。
注: セルの下部に少し下にフォーカスを設定して、すべてのセルがサンプリングされるようにします。 - ステップサイズに1μmを入力し、z-スキャン方向の上底を選択し、NDアクイジションウィンドウで 実行 をクリックしてZスタックを取得します。
注: ステップサイズは、上と下の位置(つまり、移動距離)に応じて取得されるzスライスの数を決定します。1 μmのステップサイズは、撮像速度、Z軸サンプリング、およびフォトブリーチの間の妥協点として選択されました。2.4 AUの共焦点ピンホール径と60倍の水浸し目的を使用して、光学断面厚は1.73 μmでした。したがって、1 μmのステップサイズはナイキストのサンプリング基準をわずかに下回っていますが、これはZスタックの獲得に必要な時間を短縮するために行われた妥協点です。非常に安定したサンプルでは、速度が重要ではないため、Z軸のステップが小さくなり、場合によっては小さな共焦点ピンホール径を使用して Z 軸の分解能を高める場合があります。ボトムトップは同様の結果を得る必要があり、zスキャン中に発生する可能性のあるフォトブリーチの影響を評価するために使用できます。
- パーフェクト・フォーカス・システム (PFS) をセットアップします (可能な場合)。
注: PFS では、画像取得中に焦点深度の変動を補正することができます。次の手順は PFS のセットアップに使用することができ、これらの手順は、ニコン A1R のバージョンと使用する NIS 要素のバージョンによって若干異なる場合があります。- ND取得ウィンドウの範囲アイコンで定義された対称モードをハイライト表示します。
- 顕微鏡の前面にあるPFSボタンをオンにします(サンプルステージの下のセクションにある二色性ミラーノブが'in'であることを確認してください)。
- PFSオフセットコントローラの前面のノブを使用して、上面(反時計回りに回転)と下面(時計回りに回転)を再定義します。
- ND取得ウィンドウで「相対」をクリックして、相対Z位置/Z深度を定義します。
- ソフトウェアが相対的なZ深さを記憶するように、顕微鏡の前面のメモリをクリックします。
- zスタックの取得が完了したら、ピペットを使用して目的の試薬(フォルスコリンまたは車両制御)を静かに加え、10分間待ちます。
注:細胞を乱したり、顕微鏡XY段階内の細胞チャンバーの位置を移動しないように、試薬を非常に穏やかに追加します。後続のライブビューまたは画像で、試薬の添加中に視野がずれていないことを確認すると便利です。10分の待ち時間は、フォルスコリン治療が有効にすることです。代替治療を使用する場合は、待ち時間を調整する必要があります。 - 10 分後に、ファイル名を変更し、ND 取得ウィンドウで 実行 をクリックします。
- 統計分析に十分な結果を得るために、上記のステップ 2.11 ~ 2.13 を少なくとも 5 つのカバーリップに対して繰り返して、(各処理グループの n =5 - フォルスコリンと車両制御) を実行します。
- サンプルとサンプルブランクを準備してスペクトルライブラリを構築し、手順2.9と2.10で説明した同様の取得設定を使用してスペクトル画像を取得します。
3. 画像解析
注:これらの画像は、研究に存在するすべての個々のエンドメンバーの純粋なスペクトルを含むスペクトルライブラリを構築するために使用されます。スペクトルライブラリのエンドメンバーは、異なる蛍光ホルを使用する場合、研究によって異なる場合があります。スペクトルライブラリを構築するための詳細な手順は、「補足File_Spectralライブラリ」という名前の補足情報ファイルで提供されています。ここでは、.tiffファイルへのデータのエクスポート、線形スペクトル非混合、FRET効率測定、3次元再構成、およびcAMPレベル推定について述べています。画像解析は、ImageJ、Python、MATLAB、CellProfiler などの異なる画像解析およびプログラミング プラットフォームを使用して実行できます。これらの研究では、MATLABスクリプトが使用されました。
- イメージ データをエクスポートする:
- ステップ 2.13 および 2.14 で取得したスペクトル Z スタックイメージに対応する同じファイル名で新しいフォルダを作成します。
注: データをエクスポートする手順は、NIS 要素 AR バージョン 4.30.01 に固有の手順です。これらの手順は、ソフトウェアのバージョンによって若干異なる場合があります。 - [ ファイル] をクリックすると、ファイル ウィンドウが開きます。ステップ 2.12 で取得したスペクトル画像ファイルを参照して選択し、 開くをクリックします。
- ファイルが読み込まれたら、[ ファイル→ インポート/エクスポート→ ND ドキュメントのエクスポート] をクリックします。
- ポップアップウィンドウで、ステップ3.1.1で作成したフォルダを参照して選択し、[ファイルタイプ]にタグ付き画像フォーマット(TIF)を選択し、 各チャンネルのモノラル画像 と ビット深度を保持を選択します。
注: ファイルプレフィックスは事前に生成されます。便宜上、この値を変更します。インデックスの順序は、選択されているチャンネルによって変わり、zスライスの位置が最初に、波長バンド数秒に従ってインデックスを作成する場合は「z,c」と表示されます。[LUTを適用]または[オーバーレイを挿入]または[ポイント名を使用]に対応するボックスが選択解除されていることを確認します。 - [ エクスポート ] をクリックして、tiff ファイルを個別の tiff ファイルとしてエクスポート先フォルダにエクスポートします。
- ステップ 2.1.2 ~ 3.1.5 を繰り返して、ステップ 2.13 で取得したスペクトル画像ファイルをエクスポートします。
- ステップ 2.13 および 2.14 で取得したスペクトル Z スタックイメージに対応する同じファイル名で新しいフォルダを作成します。
- 線形スペクトルの混合解除:
- プログラミング ソフトウェアを開きます。
注:生のスペクトル画像をアンミックスするカスタム開発プログラミングスクリプトは、サウスアラバマ大学BioImagingとBioSystemsのウェブサイトで、リソースタブ(https://www.southalabama.edu/centers/bioimaging/resources.html)の下に提供されています。 - 「リニアアンミックス.m」というラベルの付いたファイルを開き、エディタツールバーの 実行 ボタンをクリックします。
- NIS Elements ソフトウェアによって生成されたエクスポートされた *.tif ファイル シーケンスを含むフォルダーを参照して選択します。
- [OK] をクリックして続行すると、新しいウィンドウが[波長] と [Z スライス]と呼ばれるウィンドウが開きます。
- ステップ 3.2.4 で選択したフォルダの最初のファイル(Z スライスとチャンネル番号を除く)のファイル名をコピーし、ダイアログボックスの最初のステップに「イメージ名を入力してください」と貼り付けます。
- 「波長バンドの数を入力してください」とラベル付けされた2番目のステップのチャンネル数を入力し、「Zスライスの数を入力してください」とラベル付けされた3番目のステップのZスライスの数を入力し 、[OK]をクリックします。
注: 波長範囲や波長ステップサイズの調整など、取得設定を変更すると、波長バンド数が変わることがあります。Z スライスの数もセルの高さに応じて変わる場合があります。 - 「波長情報ファイルを選択」というラベルの付いたポップアップウィンドウで「波長.mat」という波長ファイルを参照して選択し、 開くをクリックします。
- 「スペクトルライブラリファイルを選択」というラベルの付いた新しいポップアップウィンドウで「Library.mat」ファイルを参照して選択し、 開く をクリックしてスライスのアンミックスが完了するまで待ちます。
注:Library.matファイルは、細胞自家蛍光およびバックグラウンドスペクトルシグネチャと一緒に各エンドメンバーフルオロフォアのための純粋なスペクトルを含むファイルです。この場合、エンドメンバーの蛍光色素はターコイズ、ヴィーナス、DRAQ5を含む。背景スペクトルシグネチャには、細胞またはマトリックス自己蛍光、カバースリップ蛍光、カバースリップ回折などがあります。波長.matファイルは、スペクトル画像を取得するために使用される波長チャネル情報を含むファイルです。サンプルライブラリファイルと波長ファイルは、バイオイメージングとバイオシステムズのウェブサイトで入手できます(3.2.1の下のノートを参照)。スペクトルライブラリと波長ファイルの生成方法については、「補足File_Spectralライブラリ」という補足情報ファイルを参照してください。各 z スライスに対応する混合されていないイメージは、ステップ 3.2.3 で選択したフォルダー内の非混合プロセス中に作成された「Unmixed」というフォルダーに保存されます。
- プログラミング ソフトウェアを開きます。
- フレット効率計算:
- "multiFRRCF.m" というプログラム スクリプトを開き、[ 実行] をクリックします。
注:このプログラミングファイルは、サウスアラバマ大学バイオイメージングとバイオシステムズのウェブサイトから入手できます(ステップ3.2.1の下のメモを参照)。 - 「再スライスするフォルダの数」というポップアップダイアログボックスに、分析する実験試験の数を入力し 、[OK]をクリックします。
注: 各実験の画像データは、別の混合されていない画像フォルダに保存する必要があります。この手順では、時間を節約する手順として、分析コードが多数のフォルダをループ処理できます。 - 混在していないフォルダを参照して選択し 、[OK]をクリックします。
注: 「フォルダの参照」ポップアップウィンドウが開く回数は、前の手順で[再スライスするフォルダの数]ダイアログ ボックスに入力した数によって異なります。フォルダを参照して選択します。 - 新しいポップアップウィンドウで、それぞれのボックスに次の情報を入力します:スケーリング係数は12.4、しきい値は5.6、X、Y、Z周波数はそれぞれ5、5、および1であり、平滑化アルゴリズムはガウスです。
注: スケーリング係数はピクセル/μm の値で、Z 方向サンプリングを XY 方向の値にスケーリングするために使用されます。拡大縮小係数は、画像ピクセルサイズから取得され、通常はほとんどの共焦点顕微鏡システムの画像内でメタデータとして提供されます。たとえば、0.08 μm/ピクセル間隔で画像を取得する場合、スケーリング係数は 12.5 ピクセル/μm にする必要があります。しきい値は、画像をしきい値にし、セルのバイナリマスクを生成するために使用されます。画像ドナー+アクサクサの強度に基づいて、最適値のリストを作成しました。画像の明るいドナー+アクセクサ強度と背景が低い場合はしきい値として4.5を使用し、中程度のドナー+アクセクサ強度および/または高い背景を持つ画像の場合は5.6~6.5の値、背景よりも低いドナー+アクセクサ強度を持つ画像の場合は7.5以上の値を使用します。周波数の値は、スライスが後続のステップで実行される間隔 (ピクセル数) に対応します。たとえば、セルの Z 深度が 1 μm のステップ サイズで 17 μm、XY 方向に 12.5 ピクセル/μm のスケーリング係数が使用されている場合、3 次元画像データセットの深さは 212 ピクセル (Z 方向) でリサンプリングされます。入力された Z 周波数値 (1 ピクセルなど) に基づいて、3 次元イメージ データ セットは、イメージ データセットの先頭から再スライスされ、1 ピクセル下に移動します。これにより、212 の再スライスされたイメージが作成されます。[Z 周波数] に周波数値の間隔を大きくする場合は、再スライスされたイメージの数が少なくなります。再スライスされたイメージは、以降の手順で保存されます。 - 実行 を クリックし、すべてのFRET測定と複製が実行されるまで待ちます。
注: 別のフォルダは、グレースケール FRET 効率イメージを再スライスし、色付き (カラーマップが適用) FRET 効率イメージを保存する親ディレクトリ内に作成されます。たとえば、X 方向(YZ 平面)で再スライスされたすべてのグレースケールおよびカラーマップ FRET イメージは、"Resliced_XFRET" というフォルダに保存されます。 - フォルスコリン処理と車両制御の前後のすべての実験に対して、同様の設定で分析を繰り返します。
注: セクション 3.3 で説明するステップでは、3 次元の FRET 画像データを生成するカスタム FRET 解析プログラミング スクリプトに入力する値について説明します。ただし、このスクリプトは、イメージ データの読み込み、イメージ スタックの作成、スムージング、FRET 効率の計算、セルの境界マスクの作成と適用、3 次元イメージの再構築、指定した間隔 (周波数) での 3 次元イメージの再利用、FRET の変更を視覚化するためのカラーマップの適用、再スライスされたイメージ データの同じディレクトリへの保存など、いくつかの操作を実行します。プログラム スクリプトには、コメントとして追加の詳細が含まれています。
- "multiFRRCF.m" というプログラム スクリプトを開き、[ 実行] をクリックします。
4. FRET効率をcAMPレベルにマッピング
- 'Mapping_FRET Efficiency_to_cAMP_concentration.m'という名前のプログラミングファイルを開き、メインウィンドウで 実行 をクリックします。
注:ファイルは、バイオイメージングとバイオシステムのウェブサイトで入手できます(3.2.1の下の注意を参照)。このファイルは、グレースケールのFRET効率画像を読み取り、特性曲線に基づいてcAMPレベルに変換します。この特徴的な曲線は、ヒル方程式(以下に示す第3の式)で記述されている文献15、36に記載されているcAMP-fret関係を使用する。しかし、無傷の細胞内のプローブのKdは推定が難しく、計算では1μMと仮定しています。したがって、結果は Kdの関数として表示されます。(つまり、[cAMP] = x* Kd)。FRET 効率を測定し、FRET を cAMP レベルにマッピングするために使用される式を以下に示します。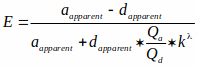
ここでEはFRET効率であり、明らかなとdは、それぞれアクセクターとドナー画像の混合ピクセル強度です。
QaおよびQdは、アクサクサおよびドナーの量子収量である。なお、Kλの式がFRET効率式に組み込まれる場合、QaおよびQdはキャンセルされ、kλは補正係数です。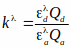
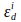
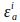 ドナー励起波長でドナーとアクセクサの絶滅係数である、i(405nm)。
ドナー励起波長でドナーとアクセクサの絶滅係数である、i(405nm)。 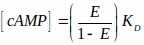
EはFRET効率であり、KD=解離定数=1μMである。 - 最初のグレースケールのFRETイメージ(ステップ3.3.5で保存)をナビゲートして選択し、[OK]をクリックします。
- FRET/cAMP画像を開いて、cAMP信号の分布を3次元で検査します。
結果
このプロトコルは、ハイパースペクトルFRETイメージングの使用と、生細胞における3つの空間次元におけるcAMP勾配を測定するための分析アプローチについて説明する。これらの結果の生成には、データの分析と定量を行う際に細心の注意が必要な重要なステップがいくつかあります。これらの主要なステップには、適切なスペクトルライブラリの構築、バックグラウンドスペクトルの非混合?...
ディスカッション
FRETバイオセンサーの開発により、単一細胞における環状ヌクレオチドシグナルの測定と可視化が可能となり、細胞内シグナル伝達事象13,22,37,38を可視化する大きな約束がある。しかし、FRETバイオセンサーの使用は、多くの蛍光タンパク質ベースのFRETレポーターの低いシグナル間特性およびFRETレ?...
開示事項
リーブズリー博士とリッチ博士は、スペクトルイメージング技術を商業化するために設立された新興企業SpectraCyte,LLCに対する金銭的関心を開示しています。しかし、このプロトコルに記載されているすべての手順は、SpectraCyte, LLCに関連しない市販の製品を使用して実施した。
謝辞
著者らは、H188 cAMP FRETバイオセンサーとケニー・トリン(南アラバマ大学工学部)が独自に開発したプログラミングスクリプトを実行するのに要する時間を短縮するための技術的支援を提供したキース・ジャリンク博士(オランダ癌研究所とファン・レーウェンフック先端顕微鏡センター、アムステルダム、オランダ)を認めたいと考えています。
著者らは、資金源を認めたい:アメリカ心臓協会(16PRE27130004)、国立科学財団。(1725937) NIH, S100D020149, S10RR027535, R01HL058506, P01HL066299).
資料
| Name | Company | Catalog Number | Comments |
| Attofluor Cell Chamber | Invitrogen | A7816 | Attofluor contains steel cell chambers and a rubber O-ring. Cell chamber holds the coverslip and O-ring provides a lock in mechanism to hold the buffer in cell chamber with out leakage |
| Dimethyl Sulfoxide (DMSO) | Fisher Scientific | BP231-100 | Solvent used to prepare stock solution forskolin. |
| DRAQ5 Fluoroscent Probe Solution | Thermo Scientific | 62252 | Nuclear label |
| Dulbecco Modified Eagle Medium (DMEM) | Gibco | 11965-092 | Contains nutrients and growth factors for the cells to grow and divide in the culture dishes. |
| Fetal Bovine Serum (FBS) | Sigma | F6178 | Growth factor suppliment that is added to culture medium, DMEM |
| Forskolin | Sigma | F3917 | Adenyly cyclase activator. |
| H188 Cyclic AMP FRET biosensor | Netherland Cancer Institute, Dr. K. Jalink | Gift | Plasmid encoding Turquoise (donor fluorophore), Venus (acceptor fluorophore), and binding domain obtained from Epac. |
| Image J | image.net | Free download | Another image processing platform used to extact spectral information and image processing. |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used to measure illumination intensity of the laser line at different laser intensities (?). |
| Laminin Mouse Protein, Natural | Invitrogen | 23017-015 | Coverslips are coated with laminin and this helps in cell attachment, growth and motility of the cell. |
| Lipofectamine 3000 Transfection Kit | Invitrogen | L3000-015 | Transfection reagent used to transfect cells with H188 FRET biosensor |
| MATLAB | Mathworks | R2019a | Image processing operations (linear unmixing and FRET efficiency calculations) are performed by writing custom programs in MATLAB programming environment |
| Nikon A1R confocal microscope | Nikon Instruments | Nikon A1R | Spectral image acquisition is performed using confocal microscope. |
| Nikon Elements Software | Nikon Instruments | Software dongle | used to export and handle nd2 image files (multidimensional image files) that are aquired using Nikon A1R |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| PBS pH 7.4 (1X) | Gibco | 10010-023 | coomonly used buffer suring cell culture |
| Pulmonary Microvascular Endothelial Cells (PMVECs) | In house - Cell culture core, Univeristy of South Alabama | Isolated from Rat pulmonary microvasculature | PMVECs form inner lining of a blood vessel. |
| Penicillin-Streptomycin (10,000 U/ml) | Gibco | 15140-122 | antibiotics are added to culture medum to prevent contamination of the cells. |
| Pre-Cleaned Gold Seal Micro Slides | Clay Adams | 3010 | Microscope slides used for cell fixation |
| ProLong Diamond Antifade Mounting Media | Invitrogen | P36961 | If samples are fixed using antifade mountant, then the later protects fluoroscent dyes and chromophores from fading. |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral response of the light source (?) |
| Trypsin-EDTA (0.25%) | Gibco | 25200-056 | Digests the protein-protein bond between the cell and cell matrix and helps to disscociate and lift the cells during cell plating. |
| Tyrodes Buffer | Made in-house | Made in-house | Tyrodes buffer is used to make working solutions and to maintain cells in aqueous solution during image acquisition. |
| 6 Well Cell Culture Plate | Corning | 3506 | Laminin coated coverslips are placed in 6-well culture dish (one coverlisps/well). Cells along with medium is added into each well. |
| 25 mm Round Microscope Cover Slips | Fisher Scientific | 12545102 | Cells were grown on round glass coverslips |
| 60X Ojective | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | water immersion and commonly used objective for cells |
参考文献
- Corbin, J. D., Sugden, P. H., Lincoln, T. M., Keely, S. L. Compartmentalization of adenosine 3':5'-monophosphate and adenosine 3':5'-monophosphate-dependent protein kinase in heart tissue. The Journal of Biological Chemistry. 252, 3854-3861 (1977).
- Terrin, A., et al. PGE1 stimulation of HEK293 cells generates multiple contiguous domains with different [cAMP]: role of compartmentalized phosphodiesterases. The Journal of Cell Biology. 175, 441-451 (2006).
- Bacskai, B. J., et al. Spatially resolved dynamics of cAMP and protein kinase A subunits in Aplysia sensory neurons. Science. 260, 222-226 (1993).
- Iancu, R. V., Ramamurthy, G., Harvey, R. D. Spatial and temporal aspects of cAMP signalling in cardiac myocytes. Clinical and Experimental Pharmacology & Physiology. 35, 1343-1348 (2008).
- Brunton, L. L., Hayes, J. S., Mayer, S. E. Functional compartmentation of cyclic AMP and protein kinase in heart. Advances in Cyclic Nucleotide Research. 14, 391-397 (1981).
- Hohl, C. M., Li, Q. Compartmentation of cAMP in adult canine ventricular myocytes. Relation to single-cell free Ca2+ transients. Circulation Research. 69, 1369-1379 (1991).
- Rich, T. C., et al. A uniform extracellular stimulus triggers distinct cAMP signals in different compartments of a simple cell. Proceedings of the National Academy of Sciences of the United States of America. 98, 13049-13054 (2001).
- Sayner, S. L., Alexeyev, M., Dessauer, C. W., Stevens, T. Soluble adenylyl cyclase reveals the significance of cAMP compartmentation on pulmonary microvascular endothelial cell barrier. Circulation Research. 98, 675-681 (2006).
- Rich, T. C., Tse, T. E., Rohan, J. G., Schaack, J., Karpen, J. W. In vivo assessment of local phosphodiesterase activity using tailored cyclic nucleotide-gated channels as cAMP sensors. The Journal of General Physiology. 118, 63-78 (2001).
- Blackman, B. E., et al. PDE4D and PDE4B function in distinct subcellular compartments in mouse embryonic fibroblasts. Journal of Biological Chemistry. 286, 12590-12601 (2011).
- Sayner, S. L., et al. Paradoxical cAMP-induced lung endothelial hyperpermeability revealed by Pseudomonas aeruginosa ExoY. Circulation Research. 95, 196-203 (2004).
- Klarenbeek, J., Jalink, K. Detecting cAMP with an EPAC-based FRET sensor in single living cells. Methods in Molecular Biology. 1071, 49-58 (2014).
- Surdo, N. C., et al. FRET biosensor uncovers cAMP nano-domains at β-adrenergic targets that dictate precise tuning of cardiac contractility. Nature Communications. 8, 15031 (2017).
- Ponsioen, B., et al. Detecting cAMP-induced Epac activation by fluorescence resonance energy transfer: Epac as a novel cAMP indicator. EMBO Reports. 5, 1176-1180 (2004).
- Vogel, S. S., Thaler, C., Koushik, S. V. Fanciful FRET. Science's STKE. 2006, (2006).
- Clegg, R. M. The History of FRET: From conception through the labors of birth. Reviews in Fluorescence. 3, (2006).
- Giepmans, B. N. G., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Manzella-Lapeira, J., Brzostowski, J. A. Imaging protein-protein interactions by Förster resonance energy transfer (FRET) microscopy in live cells. Current Protocols in Protein Science. 93, 58 (2018).
- Cooper, D. M. F., Mons, N., Karpen, J. W. Adenylyl cyclases and the interaction between calcium and cAMP signalling. Nature. 374, 421-424 (1995).
- Sassone-Corsi, P. Coupling gene expression to cAMP signalling: role of CREB and CREM. The International Journal of Biochemistry & Cell Biology. 30, 27-38 (1998).
- Rebhun, L. I. Cyclic nucleotides, calcium, and cell division. International Review of Cytology. 49, 1-54 (1977).
- Klarenbeek, J., Goedhart, J., Van Batenburg, A., Groenewald, D., Jalink, K. Fourth-generation Epac-based FRET sensors for cAMP feature exceptional brightness, photostability and dynamic range: characterization of dedicated sensors for FLIM, for ratiometry and with high affinity. PLOS ONE. 10, 0122513 (2015).
- Leavesley, S. J., Rich, T. C. FRET: signals hidden within the noise. Cytometry Part A. 85, 918-920 (2014).
- Rich, T. C., Webb, K. J., Leavesley, S. J. Can we decipher the information content contained within cyclic nucleotide signals. The Journal of General Physiology. 143, 17-27 (2014).
- Annamdevula, N. S., et al. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Leavesley, S. J., Britain, A. L., Cichon, L. K., Nikolaev, V. O., Rich, T. C. Assessing FRET using spectral techniques. Cytometry Part A. 83, 898-912 (2013).
- Leavesley, S. J., Rich, T. C. Overcoming limitations of FRET measurements. Cytometry Part A. 89, 325-327 (2016).
- Fink, D. J. Monitoring Earcths Resources from Space. Technology Review. 75, 32-41 (1973).
- Goetz, A. F. H., Vane, G., Solomon, J. E., Rock, B. N. Imaging Spectrometry for Earth Remote Sensing. Science. 228, 1147-1153 (1985).
- Bücherl, C. A., Bader, A., Westphal, A. H., Laptenok, S. P., Borst, J. W. FRET-FLIM applications in plant systems. Protoplasma. 251, 383-394 (2014).
- Chen, Y., Mauldin, J. P., Day, R. N., Periasamy, A. Characterization of spectral FRET imaging microscopy for monitoring nuclear protein interactions. Journal of Microscopy. 228, 139-152 (2007).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Letters. 531, 245-249 (2002).
- Griswold, J. R., Annamdevula, N., Deal, J., Rich, T., Leavesley, S. Estimating FRET Efficiency using Excitation-Scanning Hyperspectral Imaging. Biophysical Journal. 112, 586 (2017).
- Favreau, P. F., et al. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19, 046010 (2014).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67, 139-151 (2004).
- Thaler, C., Koushik, S. V., Blank, P. S., Vogel, S. S. Quantitative multiphoton spectral imaging and its use for measuring resonance energy transfer. Biophysical Journal. 89, 2736-2749 (2005).
- Agarwal, S. R., et al. Compartmentalized cAMP signaling associated with lipid raft and non-raft membrane domains in adult ventricular myocytes. Frontiers in Pharmacology. 9, 332 (2018).
- Johnstone, T. B., Agarwal, S. R., Harvey, R. D., Ostrom, R. S. cAMP signaling compartmentation: Adenylyl cyclases as anchors of dynamic signaling complexes. Mol Pharmacol. , (2017).
- Zhang, J., Li, H., Chai, L., Zhang, L., Qu, J., Chen, T. Quantitative FRET measurement using emission-spectral unmixing with independent excitation crosstalk correction. Journal of Microscopy. 257, 104-116 (2015).
- Zhang, J., Lin, F., Chai, L., Wei, L., Chen, T. IIem-spFRET: improved Iem-spFRET method for robust FRET measurement. Journal of Biomedical Optics. 21, 105003 (2016).
- Levy, S., et al. SpRET: highly sensitive and reliable spectral measurement of absolute FRET efficiency. Microscopy and Microanalysis. 17, 176-190 (2011).
- West, S. J., et al. Hyperspectral Measurements Allow Separation of FRET Signals from Non-Uniform Background Fluorescence. Biophysical Journal. 112, 453 (2017).
- Annamdevula, N. S., et al. An approach for characterizing and comparing hyperspectral microscopy systems. Sensors. 13, 9267-9293 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved