JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
4차원(x, y, z 및 λ) 하이퍼스펙트럼 FRET 이미징 및 분석을 사용하여 살아있는 세포에서 3차원 cAMP 분포 측정
요약
Fónster 공명 에너지 전송 (FRET) 기반 센서의 내재 된 낮은 신호 대 잡음 비율 (SNR)으로 인해, 특히 세 공간 차원에서 cAMP 신호의 측정이 도전하고있다. 여기서는 3개의 공간 차원에서 cAMP 분포를 측정할 수 있는 하이퍼스펙트럼 FRET 이미징 및 분석 방법론을 설명합니다.
초록
순환 AMP는 세포 및 생리 활동의 넓은 범위에 관여하는 두 번째 메신저입니다. 몇몇 연구 결과는 cAMP 신호가 구획화되고, 그 구획화가 cAMP 신호 통로 내의 신호 특이성에 기여한다는 것을 건의합니다. F렌스터 공명 에너지 전달(FRET) 기반 바이오 센서의 개발은 세포내 cAMP 신호를 측정하고 시각화하는 능력을 발전시켰습니다. 그러나 이러한 측정은 종종 두 개의 공간 차원으로 제한되어 데이터를 잘못 해석할 수 있습니다. 현재까지, 본질적으로 낮은 신호 대 잡음 비(SNR)를 나타내는 FRET 센서를 사용하는 기술적 한계로 인해 3개의 공간 차원(x, y 및 z)으로 cAMP 신호의 측정이 매우 제한적이었습니다. 또한, 전통적인 필터 기반 이미징 접근법은 스펙트럼 크로스토크, 제한된 신호 강도 및 자동 형광을 포함한 다양한 요인으로 인해 국소화 된 세포 이형 지역에서 cAMP 신호를 정확하게 측정하는 데 효과가 없습니다. 이러한 한계를 극복하고 FRET 기반 바이오 센서를 여러 형광과 함께 사용할 수 있도록 FRET 기반 의 바이오 센서를 여러 형광과 함께 사용할 수 있도록 FRET 효율을 계산하기 위한 스펙트럼 특이성 및 추가 형광 라벨에서 접건 성 자동 형광 및/또는 신호를 반사적으로 분리하는 기능을 제공하는 하이퍼 스펙트럼 FRET 이미징 및 분석 접근 방식을 개발했습니다. 여기서는 하이퍼스펙트럼 FRET 이미징을 구현하기 위한 방법론과 스펙트럼 미혼을 수행하기 위해 샘플링되거나 과샘플링되지 않은 적절한 스펙트럼 라이브러리를 구성할 필요성을 제시합니다. 우리는 폐 미세 혈관 내피 세포 (PMVEC)에서 3 차원 cAMP 분포의 측정을위한이 방법론을 제시하는 동안,이 방법론은 세포 유형의 범위에서 cAMP의 공간 분포를 연구하는 데 사용할 수 있습니다.
서문
순환 아데노신 모노인산염(cAMP)은 세포 분열, 칼슘 유입, 유전자 전사 및 신호 변환을 포함한 주요 세포 및 생리적 과정에 관여하는 두 번째 메신저입니다. 증거의 성장 몸은 신호 특이성을 달성하는 세포를 통해 cAMP 구획의 존재를 제안1,2,3,4,5,6,7. 최근까지, cAMP 구획화는 다른 G-결합 수용체작용제에의해 유도된 뚜렷한 생리적 또는 세포 효과에 기초하여 유추되었다8,9,10,11. 최근에는 FRET 기반 형광 이미징 프로브가 세포12,13,14에서cAMP 신호를 직접 측정 및 관찰하기 위한 새로운접근법을제공하고 있다.
Förster 공명 에너지 전달(FRET)은 분자가15,16에근접할 때 비방사적 방식으로 기증자와 수용자 분자 간에 에너지 전달이 발생하는 물리적 현상이다. FRET 기반 형광 지표의 발달과 함께, 이러한 물리적 현상은 단백질-단백질 상호 작용17,단백질 공동 국소화18,Ca+2 신호19,유전자 발현20,세포 부문21 및 순환 뉴클레오티드 신호화를 연구하기 위해 생물학적 응용 분야에서 사용되었습니다. FRET 기반 cAMP 지표는 일반적으로 cAMP 결합 도메인, 기증자 불소 호및 수용자불소호레(22)로구성됩니다. 예를 들어, 이 방법론에 사용되는 H188 cAMP센서(12)22는 에팍으로부터 얻은 cAMP 결합 도메인으로 구성되며, 청록색(기증자)과 금성(수용자) 형광 사이에 끼어 있다. 기저 조건(언바운드)에서 청록색과 금성은 형광류 사이에 FRET가 발생하는 방향으로 있습니다. 바인딩 도메인에 대한 cAMP의 바인딩시, 청록색과 금성이 떨어져 이동하도록 형성 변화가 발생하여 FRET가 감소합니다.
FRET 기반 이미징 접근 방식은 셀 내에서 cAMP 신호를 조사하고 시각화하기 위한 유망한 도구를 제공합니다. 그러나, 현재 FRET 기반 현미경 이미징 기술은 종종 세포 전 세포 공간 선명도로 FRET를 측정하기에 충분한 신호 강도를 달성하는 데 부분적으로만 성공적입니다. 이는 많은 FRET 기자의 제한된 신호 강도, FRET 효율의 변화를 정확하게 정량화하는 데 필요한 높은 수준의 정밀도, 세포 자동불화(23)및24와같은 혼란스러운 요인의 존재를 포함한 여러 가지 요인에 기인한다. 결과는 종종 약한 SNR에 의해 괴롭히는 FRET 이미지, FRET에 있는 세포 극변경의 시각화를 아주 어렵게 만듭니다. 또한, 공간적으로 국소화된 cAMP 신호에 대한 조사는 거의 2개의 공간 차원에서만 거의 독점적으로 수행되었으며 축 cAMP 분포는거의 25로간주되지 않습니다. 이는 SNR이 cAMP 그라데이션을 세 가지 공간 차원으로 측정하고 시각화하는 기능을 방해했기 때문일 수 있습니다. 낮은 SNR을 가진 FRET 센서를 사용하는 한계를 극복하기 위하여는, 우리는 단하나세포25,26,27에서FRET를 측정하기 위하여 하이퍼 스펙트럼 화상 진찰 및 분석 접근법을 구현했습니다.
하이퍼스펙트럼 이미징 접근법은 NASA가 위성 이미지28,29에존재하는 지상파 물체를 차별화하기 위해 개발되었다. 이러한 기술은 이후 형광 현미경 필드(30)로번역되었으며, 스펙트럼 검출기를 제공하는 여러 상용 공초점 현미경 시스템이 있습니다. 기존의 (비 스펙트럼) 형광 이미징에서, 샘플은 밴드 패스 필터 또는 레이저 라인을 사용하여 흥분하고, 방출은 종종 형광의 피크 방출 파장에 맞게 선택된 제2 밴드 패스 필터를 사용하여 수집된다. 대조적으로, hyperspectral 화상 진찰 접근은 특정 파장 간격에 형광 방출26,31,32 또는 여기33,34의 완전한 스펙트럼 단면도를 샘플링하는 것을 추구합니다. 우리의 이전 연구에서, 우리는 Hyperspectral 화상 진찰 및 분석 접근이 전통적인 필터 기지를 둔 FRET 화상 진찰 기술(26)에비교될 때 세포에 있는 FRET 신호의 향상한 정량화를 제공할 수 있다는 것을 보여주었습니다. 여기서는 4차원(x, y, z 및 λ) 하이퍼스펙트럼 FRET 이미징 및 분석을 수행하여 3개의 공간 차원에서 cAMP 분포를 측정하고 시각화하는 방법론을 제시합니다. 이러한 접근법은 단일세포(25)에서작용제 유도 된 cAMP 공간 그라데이션의 시각화를 허용하였다. 흥미롭게도, 고뇌에 따라, cAMP 그라데이션은 세포에서 명백할 수 있습니다. 여기에 제시된 방법론은 FRET 측정의 정확도를 향상시키기 위해 비 균일한 배경 및 세포 자동 형광의 스펙트럼 미혼을 사용합니다. 이 방법론은 cAMP FRET 바이오 센서를 사용하여 폐 미세 혈관 내피 세포 (PMVEC)에서 입증되지만, 방법론은 대체 FRET 기자 또는 대체 세포주와 함께 사용하기 위해 쉽게 수정 될 수 있습니다.
프로토콜
이 프로토콜은 사우스 앨라배마 대학 기관 동물 관리 및 사용위원회가 승인 한 절차를 따릅니다.
1. 이미징을 위한 세포, 샘플 및 시약 준비
- 이전에설명된 바와 같이 쥐 폐 내피 세포(PMVEC)를 분리한다.
참고: 세포는 100mm 세포 배양 접시에 사우스 앨라배마 대학, 모바일, AL에서 세포 배양 코어에 의해 분리되고 배양되었습니다. - 25mm 원형 유리 커버립에 시드 절연 PMVECs는 세포가 적어도 80 %의 합류 (적어도 24 시간)에 도달 할 때까지 37 ° C에서 인큐베이터에서 성장하게한다.
참고: 세포 와 세포 유형 연구에서 다양 한 수 있습니다 및 따라서 세포 특정 세포 배양 절차는 씨앗과 성장 세포에 따라야 한다. 이러한 연구에 사용되는 세포 시드 및 배양 프로토콜은 "보충 File_Cell 문화 및 트랜스페션"이라는 파일의 보충 정보로 사용할 수 있습니다. - FRET 바이오 센서를 장착하고 37°C에서 48시간 동안 배양할 수 있는 Transfect PMVECs.
참고: PMVEC를 횡단하는 프로토콜은 "보충 File_Cell 문화 및 트랜스페션"이라는 보충 정보 파일에도 설명되어 있습니다. - 이미징 당일, 티로드의 버퍼를 수조에서 37°C로 따뜻하게 합니다.
참고: 티로드의 버퍼는 145mM NaCl, 4mM KCl, 10m HEPES, 10mM 포도당, 1mM MgCl2 및 1 mM CaCl2로 구성됩니다. - 전감염된 세포를 포함하는 커버슬립을 세포 챔버에 장착하고 누출을 방지하기 위해 장착 개스킷으로 상단을 고정합니다.
- 커버슬립의 바닥을 섬세한 작업 닦아 여분의 미디어 나 부착 셀을 청소하십시오.
- 작업 버퍼 800 μL과 5mM 핵 라벨4μL을 셀 챔버에 넣고 5-10초 동안 부드럽게 흔들십시오.
참고: 세포 챔버에 장착된 커버립에 버퍼 또는 시약 솔루션을 추가할 때, 부착 세포를 빼내지 않도록 용액을 셀 챔버 의 측면에 부드럽게 추가해야 합니다. 완충제의 800 μL에 5mM 핵 라벨의 4 μL을 추가하면 25 μM최종 핵 라벨 농도가 됩니다. HEK293 세포와 같은 느슨하게 부착된 세포의 경우 핵 라벨과 버퍼를 먼저 유리병에 혼합한 다음 세포챔버에 장착된 커버립에 추가합니다. 이렇게 하면 커버슬립에서 셀을 들어 올리는 것을 방지할 수 있습니다. - 알루미늄 호일로 셀 챔버를 덮고 실온에서 10 분 동안 빛으로부터 보호하고 배양하십시오.
- 시약 준비: 버퍼의 199 μL에 50 mM 산림의 1 μL을 추가합니다. 이것은 완충의 800 μL로 제조된 세포에 추가할 때 50 μM의 산림의 최종 농도를 생성합니다. 199μL의 DMSO 1 μL 버퍼도 차량 제어로 사용할 수 있도록 준비해야 합니다.
참고: 이러한 연구에서, 산림은 cAMP 생산을 자극 하는 아데닐시클라제 활성제로 사용 됩니다. 원하는 경우, 이 방법론은 아데닐시클라아제, 인포디스세라아제 등을 자극하거나 억제하기 위한 대체 시약으로 치료를 쉽게 할 수 있도록 쉽게 수정할 수 있다.
2. 이미지 수집
- 스펙트럼 검출기가 장착된 공초점 현미경을 사용하십시오.
참고: 여기에 설명된 모든 이미지 수집 단계는 시판되는 Nikon A1R 현미경 시스템을 사용하여 개발되었습니다. 이러한 단계는 대체 스펙트럼 현미경을 사용하는 경우 조정해야 할 수 있습니다. 안정적인 작동 조건에 도달할 수 있도록 실험 시작 최소 30분 전에 모든 장비를 켜야 합니다. - 60x 물 침수 목표를 선택하고 목표에 물 한 방울을 추가합니다.
참고: 고해상도 라이브 셀 이미징의 경우 높은 수치 조리개 목표를 사용하는 것이 좋습니다. 이러한 연구에서 사용되는 목적에 대한 정보는 자료 목록을 참조하십시오. - 적재된 셀 챔버(1.7단계에서)를 현미경 단계에 놓습니다.
- 현미경의 오른쪽에 있는 필터 노브를 튜닝하여 설정된 DFT(DAPI/FITC/TRITC) 필터를 선택합니다.
- 안구를 이용하여 형광 광야 모드에서 현미경을 조작하여 cAMP FRET 센서를 발현하는 세포를 포함하는 시야를 선택한다.
참고: 선택한 셀의 기증자 또는 수용자 방출 피크 파장의 FRET 신호의 평균 강도가 적어도 100 강도 단위(A.U.) 또는 발현 세포가 없는 영역의 기준선 신호의 4배 이상인지 확인합니다. NIS Elements 소프트웨어에서 사용할 수 있는 스펙트럼 프로파일 뷰어를 사용하여 확인할 수 있습니다. 좋은 신호와 세포를 찾고 있을 때, 지나치게 밝은 세포를 폐기 하는 것이 좋습니다 (그들은 손상 될 수 있습니다). - NIS 소프트웨어를 열고, 공초점 모드로 전환, 레이저 연동 버튼의 잠금을 해제하고 라이브를 클릭합니다.
- 초점 노브를 사용하여 화면의 미리 보기를 보고 셀에 초점을 맞춥니다.
- 아래 설명과 같이 소프트웨어에서 장치, 수집 및 z 스택 설정을 구성합니다.
- 획득 설정:
참고: 이전에 획득한 이미지를 사용하여 카메라 및 장치 획득 설정을 적용할 수 있습니다. 이미지를 열고 마우스 오른쪽 단추로 클릭하고 카메라 설정을 다시 사용합니다.- A1 설정 메뉴를 열고 405 nm 및 561 nm 레이저 라인에 해당하는 상자를 선택하고 스펙트럼 검출기에 대한 SD를 선택하고 해상도를 위해 10 개, 채널용 31을 선택합니다.
참고: A1 설정 메뉴는 A1 Plus 설정 창의 왼쪽 상단 모서리에 작은 기어 아이콘으로 표시됩니다. 405 nm 레이저는 기증자 여기에 사용되며 561 nm 레이저는 핵 라벨 여기에 사용됩니다. - 시작 및 끝 파장 값을 선택하여 파장 범위(410 ~730nm)를 설정합니다.
- A1 설정 메뉴에서 비닝/건너뛰기 아이콘을 클릭하고 번호가 15인 상자를 선택한 다음 A1 설정 메뉴에서 확인을 클릭합니다.
참고: 이것은 561 nm 흥분 레이저에 해당하는 파장 채널을 제거하는 것입니다 (이것은 일반적으로 15파장 채널입니다). 스펙트럼 아티팩트를 만들 수 있는 인위적으로 낮은 신호를 피하기 위해 이 파장 밴드를 사용하지 않는 것이 중요합니다. 레이저 손상으로부터 보호하기 위해 검출기 요소를 덮는 기계식 손가락으로 인해 이 대역에서 신호가 낮습니다. - 레이저 강도를 각각 405nm 및 561 nm 레이저에 대해 8%와 2%로 설정하고, Si Hv(검출기 게인)는 각각 149개, 2.4개의 통풍 디스크 유닛(AU)의 핀홀 반경을 설정한다.
참고: 레이저 강도는 기기의 나이와 레이저의 상태에 따라 조정해야 할 수 있습니다. 다른 시료 또는 실험 그룹 간의 레이저 강도를 조정하는 경우 동일한 비율의 레이저 강도(예: 8:2)를 유지하는 것이 중요합니다. 또한, 빠른 광표백을 생성하기 위해 너무 밝지 않은 레이저 강도를 선택하는 것이 중요하다. 검출기 게인은 감지기 소음을 최소화하면서 신호 강도를 최대화하도록 조정해야 합니다. 이러한 연구를 위해, 의 이득 149 사용 되었다. 2.4 AU의 핀홀 크기는 충분한 신호 대 노이즈 비(SNR)를 가진 이미지를 획득하고 광학 단면(공초점)을 유지하는 것 사이의 균형으로 선택되었습니다. 핀홀 크기가 증가하면 SNR이 증가하지만 공초점성이 줄어듭니다. - 스캔 속도를 초당 0.25 스펙트럼 프레임으로 설정하고, 검색 방향을 위해 단방향에 해당하는 아이콘을 선택하고, 카운트에 대해 4를 입력하고, 스캔 크기에 대해 1024 x 1024를 설정합니다.
참고: FRET 신호가 약하며 스캔 속도가 느린 경우가 많습니다. 0.25의 스캔 속도를 사용하여 스펙트럼 z 스택의 획득이 ~3분 만에 완료됩니다. 사용되는 형광에 따라 스캔 속도가 증가하거나 감소할 수 있습니다. 예를 들어 eGFP와 같은 밝은 형광류의 경우 더 빠른 스캔 속도(2프레임/초)를 사용할 수 있습니다. 카운트 아래에 입력된 숫자는 프레임 평균 값 4에 해당하므로 이미지 수집 시 노이즈를 줄이는 데 도움이 됩니다. 매우 안정적인 샘플과 시간이 제약 조건이 아닌 경우 더 높은 평균 값(최대 16개)을 사용하여 향상된 SNR을 사용하여 이미지를 얻을 수 있습니다.
- A1 설정 메뉴를 열고 405 nm 및 561 nm 레이저 라인에 해당하는 상자를 선택하고 스펙트럼 검출기에 대한 SD를 선택하고 해상도를 위해 10 개, 채널용 31을 선택합니다.
- z 스택 획득 매개 변수 정의:
참고: 2.10 단계에서 입력된 값은 형광 라벨 결합 또는 농도, 라벨 유형, 사용된 라벨 수, 세포주 및 세포 표시 밀도 및/또는 세포 자동 형광에 영향을 줄 수 있는 샘플 준비의 기타 변화를 수용하기 위해 조정해야 할 수 있습니다. 획득 매개 변수를 조정할 때광 표백을 최소화하면서 충분한 SNR을 달성하기 위해 주의를 기울여야 합니다. 또한 스펙트럼 FRET 분석기를 구성할 때 매개 변수가 모든 치료 그룹에서 잘 작동하는지 확인하기 위해 주의를 기울여야합니다. SNR이 충분하고 광표백이 최소화되도록 제안 된 매개 변수 설정으로 각 치료 그룹의 평가판을 실행하는 것이 좋습니다.- ND 인수 → → 인수 제어 보기를 클릭하여 ND 획득 창을 엽니다.
- 팝업 창에 ND 파일을 저장하려면 경로/대상 및 파일 이름을 입력합니다.
- z 계열에해당하는 확인란을 선택합니다.
- A1 플러스 설정 창에서 라이브를 클릭합니다. 이렇게 하면 라이브 보기 창이 열립니다.
- 현미경의 초점 노브를 조정하여 셀의 상단을 선택하고 ND 획득 창에서 위를 클릭하여 현재 위치를 맨 위로 설정합니다.
참고: 모든 셀이 z 계열에서 샘플링되도록 셀 상단 위에 약간 초점을 맞추는 것이 좋습니다. - 현미경의 초점 노브를 조정하여 셀의 바닥을 선택하고 ND 획득 창의 아래쪽을 클릭하여 현재 위치를 바닥으로 설정합니다.
참고: 셀 하단 아래에 약간 초점을 맞추어 모든 셀이 샘플링되도록 합니다. - 단계 크기에 대해 1 μm을 입력하고 z 스캔 방향의 위쪽 아래쪽을 선택하고 ND 획득 창에서 실행을 클릭하여 z 스택을 획득합니다.
참고: 단계 크기는 위쪽 및 하단 위치(예: 이동 거리)에 따라 획득되는 z 슬라이스 수를 결정합니다. 1 μm 스텝 크기는 이미징 속도, z축 샘플링 및 광표백 사이의 손상으로 선택되었습니다. 공초점 핀홀 직경 2.4 AU와 60배 물 침수 목표를 사용하여 광학 단면 두께는 1.73 μm이 되었다. 따라서 1 μm 스텝 크기는 나이퀴스트 샘플링 기준보다 약간 낮지만 z 스택을 획득하는 데 필요한 시간을 줄이기 위해 만들어진 타협입니다. 속도가 중요하지 않은 매우 안정적인 샘플의 경우, 더 작은 z축 단계와 더 작은 공초점 핀홀 직경을 사용하여 z축 해상도를 높일 수 있습니다. 맨 아래 상단은 유사한 결과를 산출해야하며 z 스캔 중에 발생할 수있는 광표백의 효과를 평가하는 데 사용할 수 있습니다.
- 가능한 경우 완벽한 초점 시스템(PFS)을 설정합니다.
참고: PFS를 통해 시스템은 이미지 수집 중 초점 깊이의 변동을 보정할 수 있습니다. 다음 단계는 PFS를 설정하는 데 사용될 수 있으며, 이러한 단계는 니콘 A1R 버전과 사용된 NIS 요소 버전에 따라 약간 다를 수 있습니다.- ND 획득 창의 범위 아이콘에 의해 정의된 대칭 모드를 강조 표시합니다.
- 현미경의 전면에 있는 PFS 버튼을 켭니다(샘플 스테이지 아래 섹션에 있는 이색 거울 손잡이가 '인'인지 확인하십시오).
- PFS 오프셋 컨트롤러의 전면에 노브를 사용하여 위쪽(시계 반대 방향으로 회전) 및 아래쪽(시계 방향으로 회전)을 재정의합니다.
- ND 획득 창에서 '상대적'을 클릭하여 상대 z 위치/z 깊이를 정의합니다.
- 소프트웨어가 상대 z 깊이를 기억할 수 있도록 현미경의 전면에 메모리를 클릭합니다.
- z 스택 획득이 완료되면 파이펫을 사용하여 원하는 시약(forskolin 또는 차량 제어)을 부드럽게 추가하고 10분 동안 기다립니다.
참고: 세포를 방해하거나 현미경 XY 단계 내에서 세포 실의 위치를 이동하지 않도록 시약을 매우 부드럽게 추가하십시오. 후속 라이브 뷰 또는 이미지에서 시약 추가 중에 시야가 이동되지 않았는지 확인하는 것이 좋습니다. 10분 대기 시간은 산림 치료가 적용됩니다. 대체 치료를 사용하는 경우 대기 시간을 조정해야 할 수 있습니다. - 10분 후 파일 이름을 변경하고 ND 획득 창에서 실행을 클릭합니다.
- 반복 단계 2.11 – 2.13 적어도 에 대해 위에서 설명 한 대로 5 커버립 통계 분석에 대한 충분한 결과를 달성하기 위해 (각 치료 그룹에 대한 n = 5 – 산림 및 차량 제어).
- 샘플 및 샘플 공백을 준비하여 스펙트럼 라이브러리를 구성하고 2.9 단계 및 2.10 단계에 설명된 것과 유사한 획득 설정을 사용하여 스펙트럼 이미지를 수집합니다.
3. 이미지 분석
참고: 이러한 이미지는 연구에 존재하는 모든 개별 엔멤버의 순수한 스펙트럼을 포함하는 스펙트럼 라이브러리를 구성하는 데 사용됩니다. 스펙트럼 라이브러리의 엔멤버는 다른 형광을 사용하는 경우 연구마다 다를 수 있습니다. 스펙트럼 라이브러리를 구성하는 자세한 절차는 "보충 File_Spectral 라이브러리"라는 추가 정보 파일에 제공됩니다. 여기에서는 .tiff 파일, 선형 스펙트럼 해제, FRET 효율 측정, 3차원 재구성 및 cAMP 수준 추정으로 데이터를 내보내는 것을 설명합니다. 이미지 분석은 ImageJ, 파이썬, MATLAB 또는 CellProfiler와 같은 다양한 이미지 분석 및 프로그래밍 플랫폼을 사용하여 수행될 수 있습니다. 이러한 연구에서는 MATLAB 스크립트가 사용되었습니다.
- 이미지 데이터 내보내기:
- 2.13 및 2.14 단계에서 획득한 스펙트럼 z 스택 이미지에 해당하는 동일한 파일 이름으로 새 폴더를 만듭니다.
참고: 데이터를 내보내기 위해 설명된 다음 단계는 NIS Elements AR 버전 4.30.01에 따라 다릅니다. 이러한 단계는 소프트웨어 버전에 따라 약간 다를 수 있습니다. - 파일 창을 여는 파일을클릭합니다. 2.12 단계에서 획득한 스펙트럼 이미지 파일을 찾아보고 선택하고 열기를 클릭합니다.
- 파일이 로드되면 가져오기/내보내기→ → 파일을 클릭합니다.
- 팝업 창에서: 3.1.1 단계에서 생성된 폴더를 찾아서 선택하고, 파일 유형에 대해 태그가 지정된 이미지 형식(TIF)을 선택한 다음 각 채널에 대한 모노 이미지를 선택하고 비트 깊이를 유지합니다.
참고: 파일 접두사는 미리 생성됩니다. 편의를 위해 이 값을 변경합니다. 인덱스 순서는 선택한 채널에 따라 변경되며 z 슬라이스 위치에 따라 인덱싱을 위한 "z, c"를 먼저 표시해야 하며 파장 대역 번호는 두 번째입니다. LUT 또는 삽입 오버레이 또는 점 이름 사용에 해당하는 상자가 선택되지 않았는지 확인합니다. - 티프 파일을 개별 tiff 파일로 대상 폴더로 내보내려면 내보내기를 클릭합니다.
- 2.13 단계에서 획득한 스펙트럼 이미지 파일을 내보내기 위해 3.1.2 - 3.1.5 단계를 반복합니다.
- 2.13 및 2.14 단계에서 획득한 스펙트럼 z 스택 이미지에 해당하는 동일한 파일 이름으로 새 폴더를 만듭니다.
- 선형 스펙트럼 혼합 해제:
- 프로그래밍 소프트웨어를 엽니다.
참고: 원시 스펙트럼 이미지를 혼합해제하기 위해 사용자 정의 개발 프로그래밍 스크립트는 자원 탭 (https://www.southalabama.edu/centers/bioimaging/resources.html)에서 사우스 앨라배마 바이오 이미징 및 바이오 시스템 즈 웹 사이트에 제공됩니다. - "선형 언믹싱.m"이라고 표시된 파일을 열고 편집기 도구 모음에서 실행 단추를 클릭합니다.
- NIS Elements 소프트웨어에서 생성된 내보낸 *.tif 파일 시퀀스가 포함된 폴더를 찾아보고 선택합니다.
- 계속하려면 확인을 클릭하여 파장 및 Z-Slice라는새 창을 엽니다.
- 3.2.4 단계에서 선택한 폴더에 첫 번째 파일 이름(z-slice 및 채널 번호 없음)을 복사하여 "이미지 이름 입력"이라고 표시된 대화 상자의 첫 번째 단계에 붙여 넣습니다.
- "파장 대역 수를 입력"이라고 표시된 두 번째 단계에서 채널 수를 입력하고 "Z 슬라이스 수를 입력"이라고 표시된 세 번째 단계에서 z 슬라이스 수를 입력하고 확인을 클릭합니다.
참고: 파장 범위 또는 파장 스텝 크기를 조정하는 등 획득 설정이 변경되면 파장 대역 수가 변경될 수 있습니다. Z 슬라이스의 수는 셀의 높이에 따라 변경될 수도 있습니다. - "파장 정보 파일 선택"이라고 표시된 팝업 창에서 "파장.mat"이라는 파장 파일을 찾아보고 선택하고 엽니다.
- "스펙트럼 라이브러리 파일 선택"이라는 새 팝업 창에서 "Library.mat" 파일을 찾아보고 선택하고, 열기를 클릭하고 슬라이스의 혼합 해제가 완료될 때까지 기다립니다.
참고: Library.mat 파일은 셀 자동 불피및 배경 스펙트럼 서명과 함께 각 최종 멤버 형광에 대한 순수한 스펙트럼을 포함하는 파일입니다. 이 경우, 최종 구성원 형광은 청록색, 금성 및 DRAQ5를 포함한다. 배경 스펙트럼 시그니처에는 셀룰러 또는 매트릭스 자동 형광, 커버 슬립 형광 및 커버 슬립 회절이 포함됩니다. wavelength.mat 파일은 스펙트럼 이미지를 획득하는 데 사용되는 파장 채널 정보를 포함하는 파일입니다. 예를 들어 라이브러리 파일 및 파장 파일은 Bioimaging 및 Biosystems 웹 사이트에서 사용할 수 있습니다(3.2.1 미만의 참고 사항 참조). 스펙트럼 라이브러리 및 파장 파일을 생성하는 방법에 대한 자세한 내용은 "보충 File_Spectral 라이브러리"라는 추가 정보 파일을 참조하십시오. 각 z-슬라이스에 해당하는 혼합되지 않은 이미지는 3.2.3 단계에서 선택한 폴더 내의 혼합 해제 프로세스 중에 생성된 "혼합 되지 않은"폴더에 저장됩니다.
- 프로그래밍 소프트웨어를 엽니다.
- FRET 효율성 계산:
- "multiFRRCF.m"라는 프로그래밍 스크립트를 열고 실행을 클릭합니다.
참고: 이 프로그래밍 파일은 사우스 앨라배마 대학교 바이오 이미징 및 바이오 시스템즈 웹 사이트에서 사용할 수 있습니다(3.2.1단계 아래 참고 사항 참조). - "다시 슬라이스할 폴더 수"라는 팝업 대화 상자에서 분석할 실험 시험 수를 입력하고 확인을 클릭합니다.
참고: 각 실험의 이미지 데이터는 별도의 혼합되지 않은 이미지 폴더에 저장해야 합니다. 이 단계를 사용하면 분석 코드가 시간 절약 단계로 많은 폴더를 반복할 수 있습니다. - 혼합되지 않은 폴더를 찾아서 선택하고 확인을 클릭합니다.
참고: "폴더 탐색" 팝업 창이 열리는 횟수는 이전 단계에서 "다시 슬라이스할 폴더 수" 대화 상자에 입력된 수에 따라 달라집니다. 폴더를 차례로 찾아보고 선택합니다. - 새로운 팝업 창에서, 각 상자에 다음 정보를 입력: 배율 계수는 12.4, 임계값은 5.6, X, Y 및 Z 주파수는 각각 5, 5, 및 1이며, 스무딩 알고리즘은 가우시안이다.
참고: 배율 조정 계수는 픽셀/μm의 값이며 Z 방향 샘플링을 XY 방향의 값으로 조정하는 데 사용됩니다. 배율 조정 계수는 이미지 픽셀 크기에서 얻어지며, 이는 일반적으로 대부분의 공초점 현미경 시스템에 대한 이미지의 메타데이터로 제공됩니다. 예를 들어 이미지가 0.08 μm/픽셀 간격으로 획득되는 경우 배율 조정 계수는 12.5픽셀/μm이어야 합니다. 임계값은 이미지를 임계값으로 표시하고 셀의 이진 마스크를 생성하는 데 사용됩니다. 이미지 기증자+수락자 강도에 따라 최적의 값 목록을 만들었습니다. 이미지가 밝은 기증자 +수용자 강도와 낮은 배경이있는 경우 4.5을 임계 값으로 사용, 중간 기증자 + 수락자 강도 및 / 또는 더 높은 배경을 갖는 이미지에 대한 5.6 에서 6.5 사이의 값, 그리고 7.5 이상의 값은 배경보다 낮은 기증자 + 수락자 강도를 갖는 이미지의 경우. 주파수 값은 후속 단계에서 슬라이스가 수행되는 픽셀 수의 간격에 해당합니다. 예를 들어, 셀의 Z 깊이가 1μm 스텝 크기와 12.5 픽셀/μm의 스케일링 계수가 XY 방향으로 사용되는 경우, 3차원 이미지 데이터 세트의 깊이는 212픽셀(Z 방향)에서 다시 샘플링됩니다. 입력된 Z 주파수 값(예: 1픽셀)에 따라 3차원 이미지 데이터 세트는 이미지 데이터 세트의 맨 위에서 다시 슬라이스한 다음 1픽셀 아래쪽으로 이동합니다. 이로 인해 212개의 다시 슬라이스된 이미지가 생성됩니다. Z 주파수에 대해 더 큰 주파수 값 간격을 입력하면 다시 슬라이스된 이미지가 생성됩니다. 다시 슬라이스된 이미지는 후속 단계에서 저장됩니다. - 모든 FRET 측정 및 재칭이 수행될 때까지 실행을 클릭하고 기다립니다.
참고: 회색 스케일 FRET 효율 이미지와 색상(컬러맵 적용) FRET 효율 이미지가 저장되는 부모 디렉토리 내에서 별도의 폴더가 만들어집니다. 예를 들어 X 방향(YZ 평면)으로 다시 슬라이스된 모든 회색 스케일 및 컬러맵 FRET 이미지는 "Resliced_XFRET"이라는 폴더에 저장됩니다. - 모든 실험에 대해 유사한 설정으로 분석을 반복하십시오 - 포스콜린 치료 및 차량 제어 전후.
참고: 섹션 3.3에서 언급된 단계는 사용자 지정 FRET 분석 프로그래밍 스크립트에 입력할 값을 설명하여 3차원 FRET 이미지 데이터를 생성합니다. 그러나 이 스크립트는 이미지 데이터 로드, 이미지 스택 생성, 스무딩, FRET 효율성 계산, 셀 테두리 마스크 작성 및 적용, 3차원 이미지 재구성, 지정된 간격(주파수)으로 3차원 이미지 재구성, FRET 변경 시각화를 위한 컬러맵 적용, 동일한 디렉터에 다시 슬라이스된 이미지 데이터 저장 등 여러 작업을 실행합니다. 추가 세부 정보는 프로그램 스크립트의 주석으로 포함되었습니다.
- "multiFRRCF.m"라는 프로그래밍 스크립트를 열고 실행을 클릭합니다.
4. CAMP 레벨에 FRET 효율 매핑
- 'Mapping_FRET Efficiency_to_cAMP_concentration.m'이라는 프로그래밍 파일을 열고 주 창에서 실행을 클릭합니다.
참고: 이 파일은 바이오이미징 및 바이오시스템즈 웹사이트에서 확인할 수 있습니다(3.2.1 미만 의 참고 사항 참조). 이 파일은 회색 축척 FRET 효율 이미지를 읽고 특성 곡선을 기반으로 cAMP 수준으로 변환합니다. 이 특성 곡선은 힐 방정식(아래 그림세 방정식)에 의해 설명되는 문헌15,36에 문서화된 cAMP-to-FRET 관계를 사용합니다. 그러나, 그대로 셀에 있는 프로브의 Kd는 추정하기 어렵고 우리는 우리의 계산에 있는 1 μM로 가정했습니다. 따라서 결과는 Kd의함수로 표시됩니다. (즉, [cAMP] = x * Kd). FRET 효율을 측정하고 FRET를 cAMP 수준으로 매핑하는 데 사용되는 방정식은 다음과 같습니다.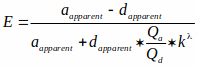
여기서 E는 FRET 효율이며, 명백하고 명백한것은 각각 수락자와 기증자 이미지의 혼합되지 않은 픽셀 강도입니다.
Qa 및 Qd는 수용자와 기증자의 양자 수율입니다. Kλ에 대한 방정식이 FRET 효율 방정식에 통합될 때 Qa 및 Qd가 취소되며, kλ는 보정 계수입니다.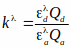
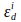
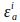 기증자의 소멸 계수및 기증자 의 수용자 인경우, i (405nm).
기증자의 소멸 계수및 기증자 의 수용자 인경우, i (405nm).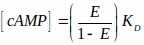
E는 FRET 효율및 KD = 해리 상수 = 1 μM입니다. - 첫 번째 회색 축척 FRET 이미지를 탐색하고 선택하고(3.3.5 단계에 저장됨)를 선택하고 확인을 클릭합니다.
- FRET/cAMP 이미지를 열어 3차원으로 cAMP 신호 분포를 검사합니다.
결과
이 프로토콜은 살아있는 세포에 있는 3개의 공간 차원에서 cAMP 그라데이션을 측정하기 위하여 hyperspectral FRET 화상 진찰 및 분석 접근의 사용을 기술합니다. 이러한 결과를 생성하는 데 관련된 몇 가지 주요 단계가 있으며, 데이터를 분석하고 정량화하는 동안 주의 깊게 주의를 기울여야 합니다. 이러한 주요 단계에는 적절한 스펙트럼 라이브러리 구성, 백그라운드 스펙트럼 해제, 셀 테두리 식별을...
토론
FRET 바이오센서의 개발은 단일 세포에서 순환 뉴클레오티드 신호의 측정 및 시각화를 허용하고, 세포 외 신호 이벤트13,22,37,38을시각화하기위한 큰 약속이있다. 그러나 FRET 바이오 센서의 사용은 많은 형광 단백질 계 FRET 리포터의 낮은 신호-잡음 특성 및 FRET 기자의 약한 트랜스퍼션 또는 발현 효율을 ...
공개
리프슬리 박사와 리치 박사는 스펙트럼 이미징 기술을 상용화하기 위해 설립된 스타트업 회사인 SpectraCyte, LLC에 대한 재정적 관심을 공개합니다. 그러나 이 프로토콜에 설명된 모든 절차는 SPECtraCyte, LLC와 관련이 없는 상업적으로 이용 가능한 제품을 사용하여 수행되었습니다.
감사의 말
저자는 H188 cAMP FRET 바이오 센서와 케니 트린 (사우스 앨라배마 대학)을 제공하는 데 도움이 우리의 사용자 정의 개발 프로그래밍 스크립트를 실행하는 데 걸린 시간을 줄이기 위해 우리에게 제공하는 박사 Kees Jalink (네덜란드 암 연구소 및 고급 현미경 검사법을위한 반 Leeuwenhoek 센터) 인정하고 싶습니다.
저자는 자금 출처를 인정하고 싶습니다: 미국 심장 협회 (16PRE2713004), 국립 과학 재단; (1725937) NIH, S100D020149, S10RR027535, R01HL058506, P01HL0666299).
자료
| Name | Company | Catalog Number | Comments |
| Attofluor Cell Chamber | Invitrogen | A7816 | Attofluor contains steel cell chambers and a rubber O-ring. Cell chamber holds the coverslip and O-ring provides a lock in mechanism to hold the buffer in cell chamber with out leakage |
| Dimethyl Sulfoxide (DMSO) | Fisher Scientific | BP231-100 | Solvent used to prepare stock solution forskolin. |
| DRAQ5 Fluoroscent Probe Solution | Thermo Scientific | 62252 | Nuclear label |
| Dulbecco Modified Eagle Medium (DMEM) | Gibco | 11965-092 | Contains nutrients and growth factors for the cells to grow and divide in the culture dishes. |
| Fetal Bovine Serum (FBS) | Sigma | F6178 | Growth factor suppliment that is added to culture medium, DMEM |
| Forskolin | Sigma | F3917 | Adenyly cyclase activator. |
| H188 Cyclic AMP FRET biosensor | Netherland Cancer Institute, Dr. K. Jalink | Gift | Plasmid encoding Turquoise (donor fluorophore), Venus (acceptor fluorophore), and binding domain obtained from Epac. |
| Image J | image.net | Free download | Another image processing platform used to extact spectral information and image processing. |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used to measure illumination intensity of the laser line at different laser intensities (?). |
| Laminin Mouse Protein, Natural | Invitrogen | 23017-015 | Coverslips are coated with laminin and this helps in cell attachment, growth and motility of the cell. |
| Lipofectamine 3000 Transfection Kit | Invitrogen | L3000-015 | Transfection reagent used to transfect cells with H188 FRET biosensor |
| MATLAB | Mathworks | R2019a | Image processing operations (linear unmixing and FRET efficiency calculations) are performed by writing custom programs in MATLAB programming environment |
| Nikon A1R confocal microscope | Nikon Instruments | Nikon A1R | Spectral image acquisition is performed using confocal microscope. |
| Nikon Elements Software | Nikon Instruments | Software dongle | used to export and handle nd2 image files (multidimensional image files) that are aquired using Nikon A1R |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| PBS pH 7.4 (1X) | Gibco | 10010-023 | coomonly used buffer suring cell culture |
| Pulmonary Microvascular Endothelial Cells (PMVECs) | In house - Cell culture core, Univeristy of South Alabama | Isolated from Rat pulmonary microvasculature | PMVECs form inner lining of a blood vessel. |
| Penicillin-Streptomycin (10,000 U/ml) | Gibco | 15140-122 | antibiotics are added to culture medum to prevent contamination of the cells. |
| Pre-Cleaned Gold Seal Micro Slides | Clay Adams | 3010 | Microscope slides used for cell fixation |
| ProLong Diamond Antifade Mounting Media | Invitrogen | P36961 | If samples are fixed using antifade mountant, then the later protects fluoroscent dyes and chromophores from fading. |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral response of the light source (?) |
| Trypsin-EDTA (0.25%) | Gibco | 25200-056 | Digests the protein-protein bond between the cell and cell matrix and helps to disscociate and lift the cells during cell plating. |
| Tyrodes Buffer | Made in-house | Made in-house | Tyrodes buffer is used to make working solutions and to maintain cells in aqueous solution during image acquisition. |
| 6 Well Cell Culture Plate | Corning | 3506 | Laminin coated coverslips are placed in 6-well culture dish (one coverlisps/well). Cells along with medium is added into each well. |
| 25 mm Round Microscope Cover Slips | Fisher Scientific | 12545102 | Cells were grown on round glass coverslips |
| 60X Ojective | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | water immersion and commonly used objective for cells |
참고문헌
- Corbin, J. D., Sugden, P. H., Lincoln, T. M., Keely, S. L. Compartmentalization of adenosine 3':5'-monophosphate and adenosine 3':5'-monophosphate-dependent protein kinase in heart tissue. The Journal of Biological Chemistry. 252, 3854-3861 (1977).
- Terrin, A., et al. PGE1 stimulation of HEK293 cells generates multiple contiguous domains with different [cAMP]: role of compartmentalized phosphodiesterases. The Journal of Cell Biology. 175, 441-451 (2006).
- Bacskai, B. J., et al. Spatially resolved dynamics of cAMP and protein kinase A subunits in Aplysia sensory neurons. Science. 260, 222-226 (1993).
- Iancu, R. V., Ramamurthy, G., Harvey, R. D. Spatial and temporal aspects of cAMP signalling in cardiac myocytes. Clinical and Experimental Pharmacology & Physiology. 35, 1343-1348 (2008).
- Brunton, L. L., Hayes, J. S., Mayer, S. E. Functional compartmentation of cyclic AMP and protein kinase in heart. Advances in Cyclic Nucleotide Research. 14, 391-397 (1981).
- Hohl, C. M., Li, Q. Compartmentation of cAMP in adult canine ventricular myocytes. Relation to single-cell free Ca2+ transients. Circulation Research. 69, 1369-1379 (1991).
- Rich, T. C., et al. A uniform extracellular stimulus triggers distinct cAMP signals in different compartments of a simple cell. Proceedings of the National Academy of Sciences of the United States of America. 98, 13049-13054 (2001).
- Sayner, S. L., Alexeyev, M., Dessauer, C. W., Stevens, T. Soluble adenylyl cyclase reveals the significance of cAMP compartmentation on pulmonary microvascular endothelial cell barrier. Circulation Research. 98, 675-681 (2006).
- Rich, T. C., Tse, T. E., Rohan, J. G., Schaack, J., Karpen, J. W. In vivo assessment of local phosphodiesterase activity using tailored cyclic nucleotide-gated channels as cAMP sensors. The Journal of General Physiology. 118, 63-78 (2001).
- Blackman, B. E., et al. PDE4D and PDE4B function in distinct subcellular compartments in mouse embryonic fibroblasts. Journal of Biological Chemistry. 286, 12590-12601 (2011).
- Sayner, S. L., et al. Paradoxical cAMP-induced lung endothelial hyperpermeability revealed by Pseudomonas aeruginosa ExoY. Circulation Research. 95, 196-203 (2004).
- Klarenbeek, J., Jalink, K. Detecting cAMP with an EPAC-based FRET sensor in single living cells. Methods in Molecular Biology. 1071, 49-58 (2014).
- Surdo, N. C., et al. FRET biosensor uncovers cAMP nano-domains at β-adrenergic targets that dictate precise tuning of cardiac contractility. Nature Communications. 8, 15031 (2017).
- Ponsioen, B., et al. Detecting cAMP-induced Epac activation by fluorescence resonance energy transfer: Epac as a novel cAMP indicator. EMBO Reports. 5, 1176-1180 (2004).
- Vogel, S. S., Thaler, C., Koushik, S. V. Fanciful FRET. Science's STKE. 2006, (2006).
- Clegg, R. M. The History of FRET: From conception through the labors of birth. Reviews in Fluorescence. 3, (2006).
- Giepmans, B. N. G., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Manzella-Lapeira, J., Brzostowski, J. A. Imaging protein-protein interactions by Förster resonance energy transfer (FRET) microscopy in live cells. Current Protocols in Protein Science. 93, 58 (2018).
- Cooper, D. M. F., Mons, N., Karpen, J. W. Adenylyl cyclases and the interaction between calcium and cAMP signalling. Nature. 374, 421-424 (1995).
- Sassone-Corsi, P. Coupling gene expression to cAMP signalling: role of CREB and CREM. The International Journal of Biochemistry & Cell Biology. 30, 27-38 (1998).
- Rebhun, L. I. Cyclic nucleotides, calcium, and cell division. International Review of Cytology. 49, 1-54 (1977).
- Klarenbeek, J., Goedhart, J., Van Batenburg, A., Groenewald, D., Jalink, K. Fourth-generation Epac-based FRET sensors for cAMP feature exceptional brightness, photostability and dynamic range: characterization of dedicated sensors for FLIM, for ratiometry and with high affinity. PLOS ONE. 10, 0122513 (2015).
- Leavesley, S. J., Rich, T. C. FRET: signals hidden within the noise. Cytometry Part A. 85, 918-920 (2014).
- Rich, T. C., Webb, K. J., Leavesley, S. J. Can we decipher the information content contained within cyclic nucleotide signals. The Journal of General Physiology. 143, 17-27 (2014).
- Annamdevula, N. S., et al. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Leavesley, S. J., Britain, A. L., Cichon, L. K., Nikolaev, V. O., Rich, T. C. Assessing FRET using spectral techniques. Cytometry Part A. 83, 898-912 (2013).
- Leavesley, S. J., Rich, T. C. Overcoming limitations of FRET measurements. Cytometry Part A. 89, 325-327 (2016).
- Fink, D. J. Monitoring Earcths Resources from Space. Technology Review. 75, 32-41 (1973).
- Goetz, A. F. H., Vane, G., Solomon, J. E., Rock, B. N. Imaging Spectrometry for Earth Remote Sensing. Science. 228, 1147-1153 (1985).
- Bücherl, C. A., Bader, A., Westphal, A. H., Laptenok, S. P., Borst, J. W. FRET-FLIM applications in plant systems. Protoplasma. 251, 383-394 (2014).
- Chen, Y., Mauldin, J. P., Day, R. N., Periasamy, A. Characterization of spectral FRET imaging microscopy for monitoring nuclear protein interactions. Journal of Microscopy. 228, 139-152 (2007).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Letters. 531, 245-249 (2002).
- Griswold, J. R., Annamdevula, N., Deal, J., Rich, T., Leavesley, S. Estimating FRET Efficiency using Excitation-Scanning Hyperspectral Imaging. Biophysical Journal. 112, 586 (2017).
- Favreau, P. F., et al. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19, 046010 (2014).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67, 139-151 (2004).
- Thaler, C., Koushik, S. V., Blank, P. S., Vogel, S. S. Quantitative multiphoton spectral imaging and its use for measuring resonance energy transfer. Biophysical Journal. 89, 2736-2749 (2005).
- Agarwal, S. R., et al. Compartmentalized cAMP signaling associated with lipid raft and non-raft membrane domains in adult ventricular myocytes. Frontiers in Pharmacology. 9, 332 (2018).
- Johnstone, T. B., Agarwal, S. R., Harvey, R. D., Ostrom, R. S. cAMP signaling compartmentation: Adenylyl cyclases as anchors of dynamic signaling complexes. Mol Pharmacol. , (2017).
- Zhang, J., Li, H., Chai, L., Zhang, L., Qu, J., Chen, T. Quantitative FRET measurement using emission-spectral unmixing with independent excitation crosstalk correction. Journal of Microscopy. 257, 104-116 (2015).
- Zhang, J., Lin, F., Chai, L., Wei, L., Chen, T. IIem-spFRET: improved Iem-spFRET method for robust FRET measurement. Journal of Biomedical Optics. 21, 105003 (2016).
- Levy, S., et al. SpRET: highly sensitive and reliable spectral measurement of absolute FRET efficiency. Microscopy and Microanalysis. 17, 176-190 (2011).
- West, S. J., et al. Hyperspectral Measurements Allow Separation of FRET Signals from Non-Uniform Background Fluorescence. Biophysical Journal. 112, 453 (2017).
- Annamdevula, N. S., et al. An approach for characterizing and comparing hyperspectral microscopy systems. Sensors. 13, 9267-9293 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유