Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Visualización y cuantificación de la señalización TGFβ/BMP/SMAD bajo diferentes condiciones de tensión por cizallamiento de fluidos mediante ensayo de ligadura de proximidad
En este artículo
Resumen
Aquí, establecemos un protocolo para visualizar y analizar simultáneamente múltiples complejos SMAD utilizando el ensayo de ligadura de proximidad (PLA) en células endoteliales expuestas a condiciones patológicas y fisiológicas de estrés por cizallamiento de fluidos.
Resumen
La señalización del factor de crecimiento transformador β (TGFβ)/proteína morfogenética ósea (BMP) está estrechamente regulada y equilibrada durante el desarrollo y la homeostasis del sistema de vasculatura Por lo tanto, la desregulación en esta vía de señalización da como resultado patologías vasculares graves, como hipertensión arterial pulmonar, telangiectasia hemorrágica hereditaria y aterosclerosis. Las células endoteliales (CE), como la capa más interna de los vasos sanguíneos, están constantemente expuestas al estrés por cizallamiento de líquidos (SS). Se ha demostrado que los patrones anormales de SS de líquido mejoran la señalización TGFβ / BMP, que, junto con otros estímulos, inducen aterogénesis. En relación con esto, se encontró que la ateroprofona, SS laminar baja, mejora la señalización TGFβ / BMP, mientras que la SS ateroprotectora y laminar alta disminuye esta señalización. Para analizar de manera eficiente la activación de estas vías, diseñamos un flujo de trabajo para investigar la formación de complejos de factores de transcripción en condiciones de SS laminar baja y SS laminar alta utilizando un sistema de bomba neumática disponible comercialmente y un ensayo de ligadura de proximidad (PLA).
La señalización activa de TGFβ/BMP requiere la formación de complejos SMAD triméricos que consisten en dos SMAD reguladores (R-SMAD); SMAD2/3 y SMAD1/5/8 para la señalización TGFβ y BMP, respectivamente) con un mediador común SMAD (co-SMAD; SMAD4). Utilizando PLA dirigido a diferentes subunidades del complejo SMAD trimérico, es decir, R-SMAD /co-SMAD o R-SMAD/R-SMAD, la formación de complejos activos de factor de transcripción SMAD se puede medir cuantitativa y espacialmente utilizando microscopía de fluorescencia.
El uso de diapositivas de flujo con 6 pequeños canales paralelos, que se pueden conectar en serie, permite la investigación de la formación del complejo del factor de transcripción y la inclusión de los controles necesarios.
El flujo de trabajo explicado aquí se puede adaptar fácilmente para estudios dirigidos a la proximidad de los SMAD a otros factores de transcripción o a complejos de factores de transcripción distintos de los SMAD, en diferentes condiciones de SS fluidos. El flujo de trabajo presentado aquí muestra una forma rápida y efectiva de estudiar la señalización fluida inducida por SS TGFβ / BMP en ECs, tanto cuantitativa como espacialmente.
Introducción
Las proteínas de la superfamilia de factores de crecimiento transformantes beta (TGFβ) son citoquinas pleiotrópicas con una variedad de miembros, incluyendo TGFβs, proteínas morfogenéticas óseas (BMP) y activinas1,2. La unión al ligando induce la formación de oligómeros receptores que conducen a la fosforilación y, por lo tanto, a la activación del SMAD regulador citosólico (R-SMAD). Dependiendo de la subfamilia de ligandos, se activan diferentes R-SMADs1,2. Mientras que TGFβs y Activinas inducen principalmente la fosforilación de SMAD2/3, los BMP inducen la fosforilación de SMAD1/5/8. Sin embargo, se acumulan evidencias de que los BMP y TGFβs/Activinas también activan los R-SMAD de la otra subfamilia respectiva, en un proceso denominado como 'señalización lateral'3,4,5,6,7,8 y que existen complejos SMAD mixtos formados por ambos, SMAD1/5 y SMAD2/3, miembros3,9 . Dos R-SMAD activados posteriormente forman complejos triméricos con el mediador común SMAD4. Estos complejos de factores de transcripción son capaces de translocarse al núcleo y regular la transcripción de genes diana. Los SMADs pueden interactuar con una variedad de diferentes co-activadores transcripcionales y co-represores, lo que lleva a la diversificación de las posibilidades de regular los genes diana10. La desregulación de la señalización SMAD tiene graves implicaciones en una variedad de enfermedades. En línea con esto, la señalización desequilibrada de TGFβ/BMP puede conducir a patologías vasculares graves, como hipertensión arterial pulmonar, telangiectasia hemorrágica hereditaria o aterosclerosis3,11,12,13,14.
Las células endoteliales (CE) forman la capa más interna de los vasos sanguíneos y, por lo tanto, están expuestas al estrés cortante (SS), una fuerza de fricción ejercida por el flujo viscoso de la sangre. Curiosamente, las CE que residen en las partes de la vasculatura, que están expuestas a altos niveles de SS laminar uniforme, se mantienen en un estado homeostático y quiescente. Por el contrario, las CE que experimentan SS bajas y no uniformes, por ejemplo, en bifurcaciones o en la menor curvatura del arco aórtico, son proliferativas y activan vías inflamatorias15. A su vez, los sitios de CE disfuncionales son propensos a desarrollar aterosclerosis. Curiosamente, las CE en estas áreas de ateróprona muestran niveles aberrantemente altos de SMAD2/3 y SMAD1/516 activados,17,18. En este contexto, se encontró que la señalización mejorada de TGFβ/BMP era un evento temprano en el desarrollo de lesiones ateroscleróticas19 y se encontró que la interferencia con la señalización de BMP reduce notablemente la inflamación vascular, la formación de ateroma y la calcificación asociada20.
El Ensayo de Ligadura de Proximidad (PLA) es una técnica bioquímica para estudiar in situ las interacciones proteína-proteína21,22. Se basa en la especificidad de anticuerpos de diferentes especies que pueden unirse a proteínas diana de interés, lo que permite una detección altamente específica de interacciones de proteínas endógenas a nivel de una sola célula. Aquí, los anticuerpos primarios tienen que unirse a su epítopo objetivo a una distancia de menos de 40 nm para permitir la detección23. Por lo tanto, el PLA es muy beneficioso sobre los enfoques tradicionales de coinmunoprecipitación, donde se necesitan varios millones de células para detectar interacciones endógenas de proteínas. En el PLA, los anticuerpos secundarios específicos de la especie, unidos covalentemente a fragmentos de ADN (denominados sondas Plus y Minus), se unen a los anticuerpos primarios y si las proteínas de interés interactúan, las sondas Plus y Minus se acercan mucho. El ADN se liga en el siguiente paso y la amplificación del círculo rodante del ADN circular es posible. Durante la amplificación, los oligonucleótidos complementarios marcados fluorescentemente se unen al ADN sintetizado, lo que permite que estas interacciones proteicas se visualicen mediante microscopía de fluorescencia convencional.
El protocolo descrito aquí permite a los científicos comparar cuantitativamente el número de complejos de transcripción SMAD activos en condiciones de SS ateroprotectoras y ateroprofónicas in vitro utilizando PLA. SS se genera a través de un sistema de bomba neumática programable que es capaz de generar flujo unidireccional laminar de niveles definidos y permite aumentos escalonados de los caudales. Este método permite la detección de interacciones entre SMAD1/5 o SMAD2/3 con SMAD4, así como complejos mixtos-R-SMAD. Se puede ampliar fácilmente para analizar las interacciones de los SMAD con los co-reguladores transcripcionales o con complejos de factores de transcripción distintos de los SMAD. La Figura 1 muestra los principales pasos del protocolo que se presentan a continuación.
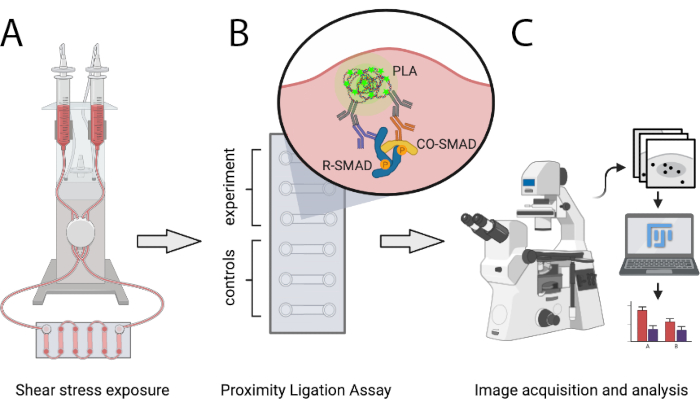
Figura 1: Representación esquemática del protocolo descrito. (A) Las células sembradas en portaobjetos de 6 canales se exponen a la tensión de cizallamiento con un sistema de bomba neumática. (B) Las células fijas se utilizan para experimentos de PLA o para condiciones de control. (C) Las imágenes de los experimentos de PLA se adquieren con un microscopio de fluorescencia y se analizan utilizando el software de análisis ImageJ. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Cultivo celular y exposición al estrés por cizallamiento de fluidos
NOTA: Las CE de vena umbilical humana (HUVEC) se utilizaron como ejemplo para estudiar la interacción inducida por SS de los SMAD. El protocolo que se describe a continuación se puede aplicar a cada tipo de célula sensible a SS.
- Cubra el portaobjetos de 6 canales con gelatina de piel porcina al 0,1% en PBS durante 30 min a 37 °C.
- HUVEC de semillas en portaobjetos pre-recubiertos de 6 canales a una densidad de 2,5 x 106 células por ml en 30 μL de medio completo M199.
NOTA: Para obtener más información sobre cómo sembrar celdas en la diapositiva de flujo, consulte la referencia24. - Deje que las células se adhieran durante 1 h a 37 °C en una incubadora humidificada.
- Añadir 60 μL de medio completo M199 precalentado a cada uno de los reservorios.
- Cultivo durante 2 días, con un intercambio medio suave una vez al día, a 37 °C en una incubadora humidificada.
- Aspire los reservorios completamente, agregue 120 μL de medio completo M199 precalentado en uno de los reservorios y aspire desde el otro lado.
- Añadir 60 μL de medio completo M199 precalentado a ambos reservorios.
- Ensamble e inicie la configuración de flujo como se detalla en la referencia25.
- Montar tubos en unidades fluídicas. Aquí, se utilizan tubos de silicona con un diámetro de 0,8 mm y 1,6 mm para aplicar esfuerzos de corte de 1 dina/cm2 y 30 dinas/cm2, respectivamente.
NOTA: El material y la longitud del tubo deben permanecer constantes, ya que los cambios podrían influir en la tensión de corte resultante. En general, se pueden utilizar otras combinaciones de sistemas de bomba y tuberías, siempre que se conozca la tensión de cizallamiento resultante y la bomba cree un flujo laminar constante. - Llene los depósitos con una cantidad adecuada de medio completo M199 precalentado (mínimo 10 ml).
- Conecte las unidades fluídicas con el tubo al sistema de bombeo y realice un pre-funcionamiento sin celdas para equilibrar el medio y eliminar el aire restante25.
- Conecte en serie los canales individuales de la diapositiva de 6 canales entre sí mediante tubos de conexión serie. El primer y el último canal de la corredera se conectarán a la tubería ensamblada en 1.6.1 (consulte la Figura 1A para un esquema). Tenga cuidado de no introducir aire en el sistema, ya que esto podría dañar gravemente las células. Puede encontrarse más información sobre la conexión serie en la referencia26.
- Para la exposición de las células a altos niveles de esfuerzo cortante (>20 dyn/cm2), utilice una fase de rampa, es decir, aumente la tensión de cizallamiento por pasos con las fases de adaptación. Los pasos se pueden establecer en incrementos de 5 dinas/cm2 por 30 min.
- Montar tubos en unidades fluídicas. Aquí, se utilizan tubos de silicona con un diámetro de 0,8 mm y 1,6 mm para aplicar esfuerzos de corte de 1 dina/cm2 y 30 dinas/cm2, respectivamente.
2. Fijación
- Separe las correderas de las bombas después de la exposición al fluido SS. Use abrazaderas en el tubo al separarse, para evitar el derrame medio en la incubadora.
- El flujo de transferencia inmediata se desliza sobre hielo, mientras que el tubo restante se separa secuencialmente. Al retirar el tubo de los depósitos, el depósito del otro lado debe mantenerse cerrado con un dedo para evitar atrapar burbujas de aire en el canal. Esto podría interferir con los pasos de fijación.
- Manteniendo las células en hielo, aspire el medio cuidadosamente desde los reservorios, pero no desde el canal donde residen las células. Posteriormente, lavar las muestras con PBS estéril en frío (4 °C) con tres veces el volumen del canal (90 μL). Agregue PBS en un reservorio y aspire cuidadosamente desde el otro reservorio. Repita este paso en cada uno de los 6 canales por diapositiva.
NOTA: Para todas las etapas de lavado e incubación, la solución respectiva se agrega en uno de los reservorios, lo que conduce a un intercambio de soluciones en el canal. Para permitir la sustitución completa de soluciones en el canal, el exceso de solución se aspira desde el otro reservorio. La solución en la parte superior de las células en el canal no se elimina. Las células no deben secarse en ningún momento. Por lo tanto, es importante lavar con cuidado sin ninguna inserción de burbujas de aire en los portaobjetos. - Fije las células agregando 90 μL de solución tamponada de PFA al 4% en el mismo reservorio donde se agregó el PBS de antemano y aspire de manera similar el líquido del otro reservorio. Repita este paso en cada canal de cada diapositiva. Después de la adición de la solución de PFA, transfiera las muestras del hielo a temperatura ambiente (RT) e incube durante 20 minutos.
PRECAUCIÓN: El PFA es tóxico y debe manipularse con cuidado. Use guantes y trabaje bajo una campana de humos. - Lave las celdas 3x con PBS (RT) agregándolas en un depósito y aspirando cuidadosamente desde el otro depósito. Vacíe solo los depósitos, asegurándose de no secar el canal. Repita este paso para cada uno de los 6 canales por diapositiva.
- Apague la fijación de PFA agregando 90 μL de cloruro de amonio ambiental de 50 mM en PBS en uno de los reservorios. Aspirar el exceso de solución del otro reservorio. Repita el proceso para cada canal de la diapositiva. Incubar las muestras durante 10 min en RT.
- Lavar como se describe en el paso 2.5.
NOTA: En este punto, las muestras pueden almacenarse a 4 °C durante la noche, o el protocolo puede continuarse inmediatamente con el bloqueo y la incubación primaria de anticuerpos (consulte el paso 3).
3. Bloqueo e incubación primaria de anticuerpos
- Para permeabilizar las células, agregue 90 μL de Triton-X-100 al 0,3% en PBS en un reservorio vacío y aspire desde el otro reservorio para cada canal. Incubar durante 10 min en RT.
- Lavar como se describe en el paso 2.5.
- Añadir 90 μL de solución estéril de bloqueo de PLA en un reservorio de un canal y aspirar desde el otro lado. Repita este paso para cada canal. Bloque durante 1 h a 37 °C en una cámara humidificada.
- Para hacer una cámara humidificada, use un plato de 10 cm con tejido húmedo sellado con película de cera y coloque el plato en la incubadora. Alternativamente, se pueden utilizar otros formatos de cámara de humedad que proporcionan una atmósfera húmeda.
NOTA: Alternativamente, se puede usar una solución de bloqueo de fabricación propia (por ejemplo, 3% (p/v) de BSA en PBS, filtrada estéril).
- Para hacer una cámara humidificada, use un plato de 10 cm con tejido húmedo sellado con película de cera y coloque el plato en la incubadora. Alternativamente, se pueden utilizar otros formatos de cámara de humedad que proporcionan una atmósfera húmeda.
- Preparar anticuerpos primarios (1:100) en el diluyente de anticuerpos PLA. Preparar 30 μL de la solución por canal. Agregue ambos anticuerpos primarios simultáneamente y vórtice.
NOTA: Alternativamente, se puede usar un diluyente de anticuerpos de fabricación propia (por ejemplo, 1% (p/v) de BSA en PBS). Los anticuerpos utilizados aquí son combinaciones de SMAD1-SMAD2/3, SMAD2/3-SMAD4 y fosfo-SMAD1/5-SMAD4. La información detallada se puede encontrar en la Tabla de Materiales. - Antes de la aplicación de anticuerpos primarios, aspire la solución bloqueadora de los reservorios y, también, con cuidado desde el canal. Pipetear 30 μL de la solución de anticuerpos primario inmediatamente en el canal vacío inclinando el canal mientras se agrega la solución.
NOTA: Realice la eliminación de la solución de bloqueo y la adición de la solución de anticuerpos canal por canal para garantizar que las células no se sequen en el medio. - Incubar muestras con los anticuerpos primarios durante la noche en cámaras humidificadas a 4 °C.
NOTA: La incubación también se puede realizar durante 1 h a temperatura ambiente, si se desea continuar con los siguientes pasos en el mismo día.
4. Incubación de la sonda PLA
NOTA: Para todos los pasos de la sección 4.1-7.3, los tampones de lavado A y B se almacenan a 4 °C y deben calentarse a RT antes del uso.
- Diluya las sondas pla (+)-ratón y (-)-conejo a 1:5 en solución de diluyente de anticuerpos PLA (o BSA al 1%). Preparar 30 μL por canal.
- Lave las muestras 2x durante 5 min utilizando 90 μL del tampón de lavado A en RT agregándolo en uno de los depósitos y aspirando cuidadosamente desde el otro depósito. Repita este paso para cada uno de los 6 canales por diapositiva.
- Aspire el tampón de lavado A con cuidado y agregue 30 μL de solución de sonda de PLA (preparada en la etapa 4.1), similar a la adición de anticuerpos primarios en la etapa 3.5.
- Incubar muestras durante 1 h a 37 °C en una cámara humidificada.
5. Ligadura
- Lave las muestras 2x durante 5 min utilizando 90 μL del tampón de lavado A en RT, como se describe en 4.2.
- Prepare una dilución 1:5 del tampón de ligadura en agua desionizada. Use este tampón para diluir la enzima ligasa a 1:40 (en hielo). Utilice 30 μL por canal.
- Aspire completamente el tampón de lavado A y agregue la solución de ligadura como se describe en 3.5.
- Incubar muestras durante 30 min a 37 °C en una cámara humidificada.
6. Amplificación
- Lavar las muestras 2x durante 2 min utilizando 90 μL de tampón de lavado A en RT, como se describe en 4.2.
- Prepare el tampón de amplificación diluyéndolo 1:5 en agua desionizada y úselo para diluir la enzima polimerasa a 1:80 (en hielo). Proteger de la luz. Preparar 30 μL por canal.
- Aspire completamente el tampón de lavado A e inmediatamente agregue la solución de amplificación preparada en el canal vacío, como se describe en 3.5. Incubar muestras durante 100 min a 37 °C en una cámara humidificada.
7. Montaje
- Lave las muestras 2x durante 10 min utilizando 90 μL de Wash Buffer B en RT como se describe en 4.2. Añadir DAPI (1:500) a partir de 1 mg/ml de solución madre (en agua desionizada) en el primer lavado para teñir los núcleos. No seque el canal.
- Diluya el tampón de lavado B en agua desionizada (1:10) y lave 1x con 90 μL de solución B tampón 0.1x como se describe en 4.2.
- Aspire completamente el tampón de lavado B e inmediatamente agregue 2-3 gotas del medio de montaje líquido en un depósito. Distribúyalo en el canal inclinando la diapositiva. Guarde las muestras a 4 °C en un ambiente humidificado hasta la obtención de imágenes.
8. Adquisición de imágenes
- Adquirir imágenes usando un microscopio de fluorescencia. Asegúrese de que los filtros respectivos que se ajustan a las sondas pla fluorescentes estén disponibles.
NOTA: Es beneficioso hacer uso de un microscopio confocal, si es posible, ya que las manchas plasmáticas obtenidas están más definidas. Esto también admite un mayor procesamiento de imágenes y análisis de datos.
9. Análisis y cuantificación de imágenes utilizando ImageJ/FIJI
- Procese las imágenes exportadas (.tiff) con un programa de procesamiento de imágenes, como ImageJ27.
NOTA: Todos los scripts utilizados dentro de este estudio y que son necesarios para el conteo automático de eventos celulares, nucleares y todos los eventos PLA (por celda) se pueden encontrar en un repositorio de GitHub: https://github.com/Habacef/Proximity-Ligation-Assay-analysis. Realizar análisis estadísticos utilizando cualquier programa o herramienta adecuada.
Resultados
Anteriormente hemos utilizado PLA para detectar interacciones de diferentes proteínas SMAD3 y analizado cambios inducidos por estrés cortante en la fosforilación de SMAD28.
Aquí, ambos métodos se combinaron con el protocolo descrito anteriormente. Los HUVEC fueron sometidos a un esfuerzo cortante de 1 dina/cm2 y 30 dinas/cm2 y analizados para detectar interacciones de factores de transcripción SMAD. Mostramos que, en c...
Discusión
El protocolo basado en PLA descrito aquí ofrece una forma eficiente de determinar la proximidad de dos proteínas (por ejemplo, su interacción directa) en CE expuestas a tensión cortante con resolución cuantitativa y espacial. Mediante el uso de portaobjetos de flujo con múltiples canales paralelos, se pueden examinar varias interacciones de proteínas diferentes al mismo tiempo en células en condiciones mecánicas idénticas. Por el contrario, los sistemas de cámara de flujo personalizados a menudo hacen uso de u...
Divulgaciones
Los autores declaran no tener conflicto de intereses.
Agradecimientos
Agradecemos a la Dra. Maria Reichenbach y al Dr. Christian Hiepen por su apoyo en el sistema de configuración de flujo y a Eleanor Fox y Yunyun Xiao por leer críticamente el manuscrito. P-L.M. fue financiado por la escuela internacional de investigación Max Planck IMPRS-Biology and Computation (IMPRS-BAC). PK recibió financiación de la DFG-SFB1444. La Figura 1 se creó utilizando BioRender.
Materiales
| Name | Company | Catalog Number | Comments |
| µ-Slide VI 0.4 | ibidi | 80606 | 6-channel slide |
| Ammonium Chloride | Carl Roth | K298.1 | Quenching |
| Bovine Serum Albumin | Carl Roth | 8076.4 | Blocking |
| DAPI | Sigma Aldrich/ Merck | D9542 | Stain DNA/Nuclei |
| DPBS | PAN Biotech | P04-53500 | PBS |
| Duolink In Situ Detection Reagents Green | Sigma Aldrich/ Merck | DUO92014 | PLA kit containing Ligase, ligation buffer, polymerase and amplification buffer (with green labeled oligonucleotides) |
| Duolink In Situ PLA Probe Anti-Mouse MINUS | Sigma Aldrich/ Merck | DUO92004 | MINUS probe |
| Duolink In Situ PLA Probe Anti-Rabbit PLUS | Sigma Aldrich/ Merck | DUO92002 | PLUS probe |
| Duolink In Situ Wash Buffers, Fluorescence | Sigma Aldrich/ Merck | DUO82049 | PLA wash buffers A and B |
| Endothelial Cell Growth Supplement | Corning | supplement for medium (ECGS) | |
| Fetal calf Serum | supplement for medium | ||
| FIJI | Image Analysis software | ||
| Formaldehyde solution 4% buffered | KLINIPATH/VWR | VWRK4186.BO1 | PFA |
| Full medium | M199 basal medium +20 % FCS +1 % P/S + 2 nM L-Glu + 25 µg/mL Hep + 50 µg/mL ECGS | ||
| Gelatin from porcine skin, Type A | Sigma Aldrich | G2500 | Use 0.1% in PBS for coating of flow channels |
| GraphPad Prism v.7 | GarphPad | Statistical Program used for the Plots and statistical calculations | |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H4784-250MG | supplement for medium (Hep) |
| HUVECs | |||
| ibidi Mounting Medium | ibidi | 50001 | Liquid mounting medium |
| ibidi Pump System | ibidi | 10902 | pneumatic pump |
| Leica TCS SP8 | Leica | confocal microscope | |
| L-Glutamin 200mM | PAN Biotech | P04-80100 | supplement for medium (L-Glu) |
| Medium 199 | Sigma Aldrich | M2154 | Base medium |
| mouse anti- SMAD1 Antibody | Abcam | ab53745 | Suited for PLA |
| mouse anti- SMAD2/3 Antibody | BD Bioscience | 610843 | Not suited for PLA in combination with CST 9515 |
| mousee anti- SMAD4 Antibody | Sanata Cruz Biotechnology | sc-7966 | Suited for PLA |
| Penicillin 10.000U/ml /Streptomycin 10mg/ml | PAN Biotech | P06-07100 | supplement for medium (P/S) |
| Perfusion Set WHITE | ibidi | 10963 | Tubings used for 1 dyn/cm2 |
| Perfusion Set YELLOW and GREEN | ibidi | 10964 | Tubings used for 30 dyn/cm2 |
| rabbit anti- phospho SMAD1/5 Antibody | Cell Signaling Technologies | 9516 | Suited for PLA |
| rabbit anti- SMAD2/3 XP Antibody | Cell Signaling Technologies | 8685 | Suited for PLA |
| rabbit anti- SMAD4 Antibody | Cell Signaling Technologies | 9515 | Not suited for PLA in combination with BD 610843 |
| Serial Connector for µ-Slides | ibidi | 10830 | serial connection tubes |
| Triton X-100 | Carl Roth | 6683.1 | Permeabilization |
Referencias
- Yadin, D., Knaus, P., Mueller, T. D. Structural insights into BMP receptors: Specificity, activation and inhibition. Cytokine and Growth Factor Reviews. 27, 13-34 (2016).
- Sieber, C., Kopf, J., Hiepen, C., Knaus, P. Recent advances in BMP receptor signaling. Cytokine and Growth Factor Reviews. 20 (5-6), 343-355 (2009).
- Hiepen, C., et al. BMPR2 acts as a gatekeeper to protect endothelial cells from increased TGFβ responses and altered cell mechanics. PLoS Biology. 17 (12), 3000557 (2019).
- Hildebrandt, S., et al. ActivinA induced SMAD1/5 Signaling in an iPSC derived EC model of Fibrodysplasia Ossificans Progressiva (FOP) can be rescued by the drug candidate saracatinib. Stem Cell Reviews and Reports. , (2021).
- Goumans, M. J., et al. Balancing the activation state of the endothelium via two distinct TGF-beta type I receptors. The EMBO Journal. 21 (7), 1743-1753 (2002).
- Goumans, M. J., et al. Activin receptor-like kinase (ALK)1 is an antagonistic mediator of lateral TGFbeta/ALK5 signaling. Molecular Cell. 12 (4), 817-828 (2003).
- Daly, A. C., Randall, R. A., Hill, C. S. Transforming growth factor beta-induced Smad1/5 phosphorylation in epithelial cells is mediated by novel receptor complexes and is essential for anchorage-independent growth. Molecular and Cellular Biology. 28 (22), 6889-6902 (2008).
- Ramachandran, A., et al. TGF-β uses a novel mode of receptor activation to phosphorylate SMAD1/5 and induce epithelial-to-mesenchymal transition. eLife. 7, 31756 (2018).
- Flanders, K. C., et al. Brightfield proximity ligation assay reveals both canonical and mixed transforming growth factor-β/bone morphogenetic protein Smad signaling complexes in tissue sections. The Journal of Histochemistry and Cytochemistry : The Official Journal of The Histochemistry Society. 62 (12), 846-863 (2014).
- Miyazono, K., Maeda, S., Imamura, T., Dijke, P. T., Heldin, C. -. H. . Smad Signal Transduction: Smads in Proliferation, Differentiation and Disease. , 277-293 (2006).
- Goumans, M. J., Zwijsen, A., Ten Dijke, P., Bailly, S. Bone morphogenetic proteins in vascular homeostasis and disease. Cold Spring Harbor Perspectives in Biology. 10 (2), 031989 (2018).
- Cai, J., Pardali, E., Sánchez-Duffhues, G., ten Dijke, P. BMP signaling in vascular diseases. FEBS Letters. 586 (14), 1993-2002 (2012).
- Cunha, S. I., Magnusson, P. U., Dejana, E., Lampugnani, M. G. Deregulated TGF-β/BMP signaling in vascular malformations. Circulation research. 121 (8), 981-999 (2017).
- MacCarrick, G., et al. Loeys-Dietz syndrome: a primer for diagnosis and management. Genetics in Medicine : An Official Journal of the American College of Medical Genetics. 16 (8), 576-587 (2014).
- Baeyens, N., Bandyopadhyay, C., Coon, B. G., Yun, S., Schwartz, M. A. Endothelial fluid shear stress sensing in vascular health and disease. The Journal of Clinical Investigation. 126 (3), 821-828 (2016).
- Min, E., et al. Activation of Smad 2/3 signaling by low shear stress mediates artery inward remodeling. bioRxiv. , 691980 (2019).
- Zhou, J., et al. BMP receptor-integrin interaction mediates responses of vascular endothelial Smad1/5 and proliferation to disturbed flow. Journal of Thrombosis and Haemostasis. 11 (4), 741-755 (2013).
- Zhou, J., et al. Force-specific activation of Smad1/5 regulates vascular endothelial cell cycle progression in response to disturbed flow. Proceedings of the National Academy of Sciences of the United States of America. 109 (20), 7770-7775 (2012).
- van Dijk, R. A., et al. Visualizing TGF-β and BMP signaling in human atherosclerosis: A histological evaluation based on Smad activation. Histology and Histopathology. 27 (3), 387-396 (2012).
- Derwall, M., et al. Inhibition of bone morphogenetic protein signaling reduces vascular calcification and atherosclerosis. Arteriosclerosis, Thrombosis, and Vascular Biology. 32 (3), 613-622 (2012).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nature Biotechnology. 20 (5), 473-477 (2002).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nature Methods. 3 (12), 995-1000 (2006).
- Alam, M. S. Proximity Ligation Assay (PLA). Current Protocols in Immunology. 123 (1), 58 (2018).
- Application Note 03: Growing Cells in µ-Channels. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN03_Growing_cells.pdf (2012)
- Application Note 13: HUVECs under perfusion. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN13_HUVECs_under_perfusion.pdf (2019)
- ibidi. Application Note 31: Instructions µ-Slide VI 0.4. ibidi. , (2013).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Reichenbach, M., et al. Differential impact of fluid shear stress and YAP/TAZ on BMP/TGF-β induced osteogenic target genes. Advanced Biology. 5 (2), 2000051 (2021).
- Hiepen, C., Mendez, P. L., Knaus, P. It takes two to tango: Endothelial TGFβ/BMP signaling crosstalk with mechanobiology. Cells. 9 (9), 1965 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados