Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Visualisierung und Quantifizierung der TGFβ/BMP/SMAD-Signalisierung unter verschiedenen Fluidscherspannungsbedingungen mittels Proximity-Ligation-Assay
In diesem Artikel
Zusammenfassung
Hier etablieren wir ein Protokoll zur gleichzeitigen Visualisierung und Analyse mehrerer SMAD-Komplexe mittels Proximity Ligation Assay (PLA) in Endothelzellen, die pathologischen und physiologischen Flüssigkeitsscherstressbedingungen ausgesetzt sind.
Zusammenfassung
Transforming Growth Factor β (TGFβ)/Bone Morphogenetic Protein (BMP) Signalisierung ist während der Entwicklung und Homöostase des Gefäßsystems streng reguliert und ausgeglichen Daher führt eine Deregulation in diesem Signalweg zu schweren vaskulären Pathologien wie pulmonaler Arterienhypertonie, hereditärer hämorrhagischer Teleangiektasie und Atherosklerose. Endothelzellen (ECs), als innerste Schicht der Blutgefäße, sind ständig Flüssigkeitsscherstress (SS) ausgesetzt. Es wurde gezeigt, dass abnormale Muster von Fluid-SS die TGFβ / BMP-Signalgebung verbessern, die zusammen mit anderen Reizen eine Atherogenese induzieren. In diesem Zusammenhang wurde festgestellt, dass atheroprones, niedriges laminares SS die TGFβ / BMP-Signalisierung verbessert, während atheroprotektives, hochlaminares SS diese Signalisierung verringert. Um die Aktivierung dieser Signalwege effizient zu analysieren, haben wir einen Workflow entwickelt, um die Bildung von Transkriptionsfaktorkomplexen unter niedrigen laminaren SS- und hohen laminaren SS-Bedingungen unter Verwendung eines kommerziell erhältlichen pneumatischen Pumpensystems und eines Proximity Ligation Assay (PLA) zu untersuchen.
Aktive TGFβ/BMP-Signalisierung erfordert die Bildung von trimeren SMAD-Komplexen, die aus zwei regulatorischen SMADs (R-SMAD) bestehen; SMAD2/3 bzw. SMAD1/5/8 für TGFβ bzw. BMP Signalisierung) mit einem gemeinsamen Mediator SMAD (Co-SMAD; SMAD4). Mit Hilfe von PLA, das auf verschiedene Untereinheiten des trimeren SMAD-Komplexes abzielt, d.h. entweder R-SMAD/co-SMAD oder R-SMAD/R-SMAD, kann die Bildung aktiver SMAD-Transkriptionsfaktorkomplexe quantitativ und räumlich mittels Fluoreszenzmikroskopie gemessen werden.
Die Verwendung von Strömungsschienen mit 6 kleinen parallelen Kanälen, die in Reihe geschaltet werden können, ermöglicht die Untersuchung der Komplexbildung des Transkriptionsfaktors und die Einbeziehung notwendiger Kontrollen.
Der hier erläuterte Workflow kann leicht für Studien angepasst werden, die auf die Nähe von SMADs zu anderen Transkriptionsfaktoren oder zu Transkriptionsfaktorkomplexen außer SMADs unter verschiedenen flüssigen SS-Bedingungen abzielen. Der hier vorgestellte Workflow zeigt eine schnelle und effektive Möglichkeit, die fluid-SS-induzierte TGFβ/BMP-Signalisierung in ECs sowohl quantitativ als auch räumlich zu untersuchen.
Einleitung
Proteine der Superfamilie der transformierenden Wachstumsfaktoren beta (TGFβ) sind pleiotrope Zytokine mit einer Vielzahl von Mitgliedern, darunter TGFβs, knochenmorphogenetische Proteine (BMPs) und Activine1,2. Die Ligandenbindung induziert die Bildung von Rezeptoroligomeren, die zur Phosphorylierung und damit zur Aktivierung der zytosolischen regulatorischen SMAD (R-SMAD) führen. Je nach Unterfamilie der Liganden werden unterschiedliche R-SMADs aktiviert1,2. Während TGFβs und Activine hauptsächlich die Phosphorylierung von SMAD2/3 induzieren, induzieren BMPs die SMAD1/5/8-Phosphorylierung. Es gibt jedoch immer mehr Hinweise darauf, dass BMPs und TGFβs/Activine auch R-SMADs der jeweiligen anderen Unterfamilie in einem als "laterale Signalisierung" bezeichneten Prozess aktivieren3,4,5,6,7,8 und dass es gemischte SMAD-Komplexe gibt, die sowohl aus SMAD1/5 als auch AUS SMAD2/3 bestehen, Mitglieder3,9 . Zwei aktivierte R-SMADs bilden anschließend trimerische Komplexe mit dem gemeinsamen Mediator SMAD4. Diese Transkriptionsfaktorkomplexe sind dann in der Lage, in den Zellkern zu translozieren und die Transkription von Zielgenen zu regulieren. SMADs können mit einer Vielzahl verschiedener transkriptioneller Koaktivatoren und Co-Repressoren interagieren, was zur Diversifizierung der Möglichkeiten zur Regulierung von Zielgenen führt10. Die Deregulierung der SMAD-Signalgebung hat schwerwiegende Auswirkungen auf eine Vielzahl von Krankheiten. Dementsprechend kann eine unausgewogene TGFβ/BMP-Signalgebung zu schweren vaskulären Pathologien wie pulmonaler Arterienhypertonie, hereditärer hämorrhagischer Teleangiektasie oder Atherosklerose führen3,11,12,13,14.
Endothelzellen (ECs) bilden die innerste Schicht der Blutgefäße und sind daher Scherstress (SS) ausgesetzt, einer Reibungskraft, die durch den viskosen Blutfluss ausgeübt wird. Interessanterweise werden ECs, die sich in den Teilen des Gefäßsystems befinden, die einem hohen Maß an einheitlichem, laminarem SS ausgesetzt sind, in einem homöostatischen und ruhenden Zustand gehalten. Im Gegensatz dazu sind ECs, die eine niedrige, ungleichmäßige SS erfahren, z. B. bei Verzweigungen oder der geringeren Krümmung des Aortenbogens, proliferativ und aktivieren Entzündungswege15. Im Gegenzug neigen Stellen von dysfunktionalen ECs dazu, Atherosklerose zu entwickeln. Interessanterweise zeigen ECs in diesen atheropronen Bereichen ungewöhnlich hohe Konzentrationen von aktiviertem SMAD2/3 und SMAD1/516,17,18. In diesem Zusammenhang wurde festgestellt, dass eine verbesserte TGFβ/BMP-Signalisierung ein frühes Ereignis in der Entwicklung atherosklerotischer Läsionen19 ist und eine Interferenz mit der BMP-Signalgebung die vaskuläre Entzündung, die Atherombildung und die damit verbundene Verkalkung deutlich reduziert20.
Proximity Ligation Assay (PLA) ist eine biochemische Technik zur Untersuchung von Protein-Protein-Interaktionen in situ21,22. Es beruht auf der Spezifität von Antikörpern verschiedener Spezies, die Zielproteine von Interesse binden können, was den hochspezifischen Nachweis endogener Proteininteraktionen auf Einzelzellebene ermöglicht. Dabei müssen primäre Antikörper in einem Abstand von weniger als 40 nm an ihr Zielepitop binden, um den Nachweis zu ermöglichen23. Daher ist PLA gegenüber herkömmlichen Co-Immunpräzipitationsansätzen, bei denen mehrere Millionen Zellen benötigt werden, um endogene Proteininteraktionen nachzuweisen, von großem Vorteil. Bei PLA binden speziesspezifische sekundäre Antikörper, kovalent verknüpft mit DNA-Fragmenten (sogenannte Plus- und Minus-Sonden), die primären Antikörper und wenn die proteininteressierten Proteine interagieren, kommen Plus- und Minus-Sonden in unmittelbarer Nähe. Die DNA wird im folgenden Schritt ligiert und die Rolling Circle Amplification der zirkulären DNA wird ermöglicht. Während der Amplifikation binden fluoreszenzmarkierte komplementäre Oligonukleotide an die synthetisierte DNA, so dass diese Proteininteraktionen durch konventionelle Fluoreszenzmikroskopie sichtbar gemacht werden können.
Das hier beschriebene Protokoll ermöglicht es Wissenschaftlern, die Anzahl der aktiven SMAD-Transkriptionskomplexe unter atheroprotektiven und atheropronen SS-Bedingungen in vitro unter Verwendung von PLA quantitativ zu vergleichen. SS wird über ein programmierbares pneumatisches Pumpensystem erzeugt, das in der Lage ist, laminaren unidirektionalen Durchfluss definierter Füllstände zu erzeugen und schrittweise Erhöhungen der Durchflussraten zu ermöglichen. Diese Methode ermöglicht den Nachweis von Wechselwirkungen zwischen SMAD1/5 oder SMAD2/3 mit SMAD4 sowie von Mixed-R-SMAD-Komplexen. Es kann leicht erweitert werden, um Wechselwirkungen von SMADs mit transkriptionellen Co-Regulatoren oder auf andere Transkriptionsfaktorkomplexe als SMADs zu analysieren. Abbildung 1 zeigt die wichtigsten Schritte des unten dargestellten Protokolls.
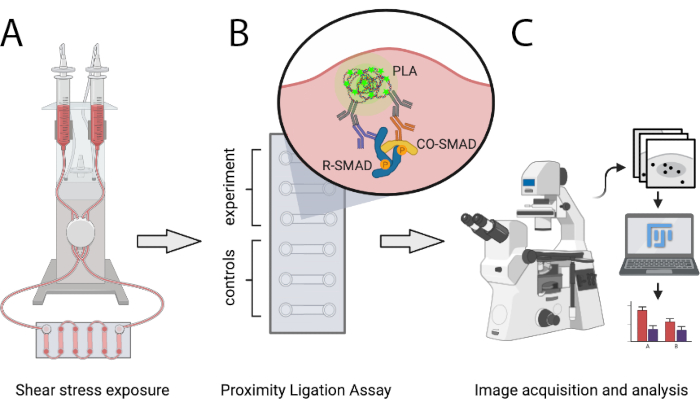
Abbildung 1: Schematische Darstellung des beschriebenen Protokolls. (A) Zellen, die in 6-Kanal-Objektträgern ausgesät sind, werden mit einem pneumatischen Pumpensystem einer Schubbeanspruchung ausgesetzt. (B) Feste Zellen werden für PLA-Experimente oder für Kontrollbedingungen verwendet. (C) Bilder von PLA-Experimenten werden mit einem Fluoreszenzmikroskop aufgenommen und mit der Analysesoftware ImageJ analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Exposition gegenüber Zellkultur- und Flüssigkeitsscherstress
HINWEIS: Menschliche Nabelschnurvenen-ECs (HUVECs) wurden als Beispiel verwendet, um die SS-induzierte Interaktion von SMADs zu untersuchen. Das unten beschriebene Protokoll kann auf jeden SS-responsiven Zelltyp angewendet werden.
- 6-Kanal-Objektträger mit 0,1% Schweinehautgelatine in PBS für 30 min bei 37 °C bestreichen.
- Seed HUVECs in vorbeschichteten 6-Kanal-Objektträgern mit einer Dichte von 2,5 x 106 Zellen pro ml in 30 μL M199 Vollmedium.
HINWEIS: Weitere Informationen zum Seeding von Zellen im Flow Slide finden Sie unter reference24. - Lassen Sie die Zellen 1 h bei 37 °C in einem befeuchteten Inkubator haften.
- Jedem der Reservoirs werden 60 μL vorgewärmtes M199-Vollmedium zugegeben.
- Kultur für 2 Tage, mit einem sanften Mittleren Austausch einmal täglich, bei 37 ° C in einem befeuchteten Inkubator.
- Die Reservoirs vollständig absaugen, 120 μL vorgewärmtes M199-Vollmedium in eines der Reservoirs geben und von der anderen Seite absaugen.
- Geben Sie 60 μL vorgewärmtes M199-Vollmedium zu beiden Reservoirs hinzu.
- Montieren und starten Sie die Strömungseinrichtung wie in der Referenz25 beschrieben.
- Montieren Sie Schläuche auf fluidischen Einheiten. Dabei werden Silikonschläuche mit einem Durchmesser von 0,8 mm und 1,6 mm zur Aufbringung von Scherspannungen von 1 dyn/cm2 bzw. 30 dyn/cm2 eingesetzt.
HINWEIS: Das Material und die Schlauchlänge sollten konstant bleiben, da Änderungen die resultierende Schubspannung beeinflussen können. Im Allgemeinen können andere Kombinationen von Pumpensystemen und Schläuchen verwendet werden, solange die resultierende Scherspannung bekannt ist und die Pumpe eine stetige laminare Strömung erzeugt. - Füllen Sie die Reservoirs mit einer angemessenen Menge vorgewärmtem M199-Vollmedium (mindestens 10 ml).
- Schließen Sie fluidische Einheiten mit dem Schlauch an das Pumpensystem an und führen Sie einen Vorlauf ohne Zellen durch, um das Medium auszugleichen und verbleibende Luft zu entfernen25.
- Verbinden Sie die einzelnen Kanäle auf dem 6-Kanal-Dia seriell über serielle Anschlussschläuche miteinander. Der erste und der letzte Kanal auf dem Schlitten werden mit dem in 1.6.1 montierten Schlauch verbunden (siehe Abbildung 1A für ein Schema). Achten Sie darauf, keine Luft in das System einzubringen, da dies die Zellen stark schädigen könnte. Weitere Informationen zur seriellen Verbindung finden Sie in reference26.
- Für die Exposition von Zellen gegenüber hoher Scherspannung (>20 dyn/cm2) verwenden Sie eine Rampenphase, d.h. erhöhen Sie die Schubspannung schrittweise mit Anpassungsphasen. Schritte können in Schritten von 5 Dyn/cm2 pro 30 min eingestellt werden.
- Montieren Sie Schläuche auf fluidischen Einheiten. Dabei werden Silikonschläuche mit einem Durchmesser von 0,8 mm und 1,6 mm zur Aufbringung von Scherspannungen von 1 dyn/cm2 bzw. 30 dyn/cm2 eingesetzt.
2. Fixierung
- Trennen Sie die Schlitten nach der SS-Exposition der Flüssigkeit von den Pumpen. Verwenden Sie beim Abnehmen Klemmen am Schlauch, um das Verschütten des Mediums im Inkubator zu vermeiden.
- Übertragen Sie sofort Strömungsrutschen auf Eis, während der verbleibende Schlauch nacheinander gelöst wird. Wenn Sie den Schlauch aus den Behältern entfernen, sollte der Behälter auf der anderen Seite mit einem Finger verschlossen gehalten werden, um luftblasen im Kanal nicht einzufangen. Dies kann die Fixierungsschritte beeinträchtigen.
- Halten Sie die Zellen auf Eis und saugen Sie das Medium vorsichtig aus den Reservoirs ab, aber nicht aus dem Kanal, in dem sich die Zellen befinden. Anschließend werden die Proben mit kaltem sterilem PBS (4 °C) mit dem dreifachen Kanalvolumen (90 μL) gewaschen. Fügen Sie PBS in einem Reservoir hinzu und aspirieren Sie vorsichtig aus dem anderen Reservoir. Wiederholen Sie diesen Schritt in jedem der 6 Kanäle pro Folie.
HINWEIS: Für alle Wasch- und Inkubationsschritte wird die jeweilige Lösung in einen der Behälter gegeben, was zu einem Austausch von Lösungen im Kanal führt. Um eine vollständige Substitution von Lösungen im Kanal zu ermöglichen, wird die überschüssige Lösung dann aus dem anderen Reservoir abgesaugt. Lösung auf der Oberseite der Zellen im Kanal wird nicht entfernt. Zellen sollten zu keiner Zeit trocknen. Daher ist es wichtig, sorgfältig zu waschen, ohne dass Luftblasen in die Objektträger eingeführt werden. - Fixieren Sie die Zellen, indem Sie 90 μL gepufferte 4% PFA-Lösung in dasselbe Reservoir geben, in dem zuvor das PBS zugegeben wurde, und saugen Sie die Flüssigkeit in ähnlicher Weise aus dem anderen Reservoir ab. Wiederholen Sie diesen Schritt in jedem Kanal in jeder Folie. Nach Zugabe der PFA-Lösung die Proben von Eis auf Raumtemperatur (RT) übertragen und 20 min inkubieren.
ACHTUNG: PFA ist giftig und sollte vorsichtig behandelt werden. Verwenden Sie Handschuhe und arbeiten Sie unter einem Abzug. - Waschen Sie die Zellen 3x mit PBS (RT), indem Sie sie in ein Reservoir geben und vorsichtig aus dem anderen Reservoir absaugen. Leeren Sie nur die Reservoirs, um sicherzustellen, dass der Kanal nicht austrocknet. Wiederholen Sie diesen Schritt für jeden der 6 Kanäle pro Folie.
- Löschen Sie die PFA-Fixierung durch Zugabe von 90 μL 50 mM Ammoniumchlorid in PBS in einem der Reservoirs. Aspirieren Sie überschüssige Lösung aus dem anderen Reservoir. Wiederholen Sie den Vorgang für jeden Kanal in der Folie. Inkubieren Sie die Proben für 10 min bei RT.
- Waschen Sie wie in Schritt 2.5 beschrieben.
HINWEIS: Zu diesem Zeitpunkt können die Proben über Nacht bei 4 °C gelagert werden, oder das Protokoll kann sofort mit Blockierung und primärer Antikörperinkubation fortgesetzt werden (siehe Schritt 3).
3. Blockierung und primäre Antikörperinkubation
- Um die Zellen zu permeabilisieren, fügen Sie 90 μL 0,3% Triton-X-100 in PBS in einem entleerten Reservoir hinzu und aspirieren Sie aus dem anderen Reservoir für jeden Kanal. Inkubieren Sie für 10 min bei RT.
- Waschen Sie wie in Schritt 2.5 beschrieben.
- 90 μL sterile PLA-Blockierlösung in ein Reservoir eines Kanals geben und von der anderen Seite absaugen. Wiederholen Sie diesen Schritt für jeden Kanal. Block für 1 h bei 37 °C in einer befeuchteten Kammer.
- Um eine befeuchtete Kammer herzustellen, verwenden Sie eine 10 cm große Schale mit nassem Gewebe, das mit Wachsfilm versiegelt ist, und legen Sie die Schale in den Inkubator. Alternativ können auch andere Feuchtekammerformate verwendet werden, die für eine feuchte Atmosphäre sorgen.
HINWEIS: Alternativ kann selbstgemachte Blockierlösung verwendet werden (z. B. 3% (w/v) BSA in PBS, steril gefiltert).
- Um eine befeuchtete Kammer herzustellen, verwenden Sie eine 10 cm große Schale mit nassem Gewebe, das mit Wachsfilm versiegelt ist, und legen Sie die Schale in den Inkubator. Alternativ können auch andere Feuchtekammerformate verwendet werden, die für eine feuchte Atmosphäre sorgen.
- Primäre Antikörper (1:100) in PLA-Antikörperverdünner herstellen. 30 μL der Lösung pro Kanal zubereiten. Fügen Sie beide primären Antikörper gleichzeitig hinzu und wirbeln Sie.
HINWEIS: Alternativ kann ein selbst hergestelltes Antikörperverdünnungsmittel verwendet werden (z. B. 1% (w/v) BSA in PBS). Die hier verwendeten Antikörper sind Kombinationen von SMAD1-SMAD2/3, SMAD2/3-SMAD4 und Phospho-SMAD1/5-SMAD4. Detaillierte Informationen finden Sie in der Materialtabelle. - Vor der Anwendung von primären Antikörpern die blockierende Lösung aus den Reservoirs und auch vorsichtig aus dem Kanal absaugen. 30 μL der primären Antikörperlösung werden sofort in den leeren Kanal pipettiert, indem der Kanal unter Zugabe der Lösung gekippt wird.
HINWEIS: Führen Sie die Entfernung der Blockierenden Lösung und die Zugabe der Antikörperlösung Kanal für Kanal durch, um sicherzustellen, dass die Zellen zwischendurch nicht austrocknen. - Inkubieren Sie Proben mit den primären Antikörpern über Nacht in befeuchteten Kammern bei 4 °C.
HINWEIS: Die Inkubation kann auch für 1 h bei Raumtemperatur durchgeführt werden, wenn Sie die folgenden Schritte am selben Tag fortsetzen möchten.
4. Pla-Sonden-Inkubation
HINWEIS: Für alle Schritte in Abschnitt 4.1-7.3 werden die Waschpuffer A und B bei 4 °C gelagert und müssen vor der Verwendung auf RT erwärmt werden.
- Verdünnung der PLA-Sonden (+)-Maus und (-)-Kaninchen auf 1:5 in PLA-Antikörperverdünnungslösung (oder 1% BSA). Bereiten Sie 30 μL pro Kanal vor.
- Proben 2x für 5 min mit 90 μL des Waschpuffers A bei RT waschen, indem sie in einen der Behälter gegeben und vorsichtig aus dem anderen Reservoir abgesaugt werden. Wiederholen Sie diesen Schritt für jeden der 6 Kanäle pro Folie.
- Den Waschpuffer A vorsichtig absaugen und 30 μL PLA-Sondenlösung (hergestellt in Schritt 4.1) hinzufügen, ähnlich der Zugabe von primären Antikörpern in Schritt 3.5.
- Proben 1 h bei 37 °C in einer befeuchteten Kammer inkubieren.
5. Ligatur
- Waschen Sie Proben 2x für 5 min unter Verwendung von 90 μL des Waschpuffers A bei RT, wie in 4.2 beschrieben.
- Bereiten Sie eine 1:5-Verdünnung des Ligaturpuffers in entionisiertem Wasser vor. Verwenden Sie diesen Puffer, um das Ligaseenzym auf 1:40 (auf Eis) zu verdünnen. Verwenden Sie 30 μL pro Kanal.
- Den Waschpuffer A vollständig absaugen und die Ligationslösung wie in 3.5 beschrieben hinzufügen.
- Proben 30 min bei 37 °C in einer befeuchteten Kammer inkubieren.
6. Verstärkung
- Waschen Sie Proben 2x für 2 min unter Verwendung von 90 μL Waschpuffer A bei RT, wie in 4.2 beschrieben.
- Bereiten Sie den Amplifikationspuffer vor, indem Sie ihn 1:5 in deionisiertem Wasser verdünnen und damit das Polymerase-Enzym auf 1:80 (auf Eis) verdünnen. Vor Licht schützen. Bereiten Sie 30 μL pro Kanal vor.
- Den Waschpuffer A vollständig absaugen und die vorbereitete Amplifikationslösung sofort in den leeren Kanal geben, wie in 3.5 beschrieben. Proben 100 min bei 37 °C in einer befeuchteten Kammer inkubieren.
7. Montage
- Waschen Sie Proben 2x für 10 min unter Verwendung von 90 μL Wash Buffer B bei RT wie in 4.2 beschrieben. DAPI (1:500) aus 1 mg/ml Stammlösung (in entionisiertem Wasser) in der ersten Wäsche zugeben, um die Kerne zu färben. Trocknen Sie den Kanal nicht.
- Der Waschpuffer B wird in entionisiertem Wasser (1:10) verdünnt und 1x mit 90 μL 0,1x Puffer B-Lösung wie in 4.2 beschrieben gewaschen.
- Den Waschpuffer B vollständig absaugen und sofort 2-3 Tropfen des flüssigen Montagemediums in ein Reservoir geben. Verteilen Sie es im Kanal, indem Sie die Folie neigen. Lagern Sie die Proben bei 4 °C in einer befeuchteten Umgebung bis zur Bildgebung.
8. Bilderfassung
- Erfassen Sie Bilder mit einem Fluoreszenzmikroskop. Stellen Sie sicher, dass die entsprechenden Filter für die fluoreszierenden PLA-Sonden verfügbar sind.
HINWEIS: Es ist vorteilhaft, wenn möglich ein konfokales Mikroskop zu verwenden, da die erhaltenen PLA-Flecken definierter sind. Dies unterstützt auch die weitere Bildverarbeitung und Datenanalyse.
9. Bildanalyse und Quantifizierung mit ImageJ/FIJI
- Verarbeiten Sie exportierte Bilder (.tiff) mit einem Bildverarbeitungsprogramm wie ImageJ27.
HINWEIS: Alle Skripte, die in dieser Studie verwendet werden und für die automatische Zählung von zellulären, nuklearen und allen PLA-Ereignissen (pro Zelle) erforderlich sind, finden Sie in einem GitHub-Repository: https://github.com/Habacef/Proximity-Ligation-Assay-analysis. Führen Sie statistische Analysen mit einem geeigneten Programm oder Tool durch.
Ergebnisse
Wir haben pla zuvor verwendet, um Wechselwirkungen verschiedener SMAD-Proteine3 zu detektieren und scherspannungsinduzierte Veränderungen in der SMAD-Phosphorylierung zu analysieren28.
Hier wurden beide Methoden mit dem oben beschriebenen Protokoll kombiniert. HUVECs wurden einer Scherspannung von 1 Dyn/cm2 und 30 Dyn/cm2 ausgesetzt und auf Wechselwirkungen von SMAD-Transkriptionsfaktoren analysiert. Wir zeigen, dass im Ver...
Diskussion
Das hier beschriebene PLA-basierte Protokoll bietet eine effiziente Möglichkeit, die Nähe von zwei Proteinen (z. B. deren direkte Interaktion) in ECs, die Scherspannung ausgesetzt sind, mit quantitativer und räumlicher Auflösung zu bestimmen. Durch den Einsatz von Strömungsobjektträgern mit mehreren parallelen Kanälen können mehrere verschiedene Proteininteraktionen gleichzeitig in Zellen unter identischen mechanischen Bedingungen untersucht werden. Im Gegensatz dazu verwenden speziell angefertigte Durchflusskamm...
Offenlegungen
Die Autoren erklären keinen Interessenkonflikt.
Danksagungen
Wir danken Dr. Maria Reichenbach und Dr. Christian Hiepen für ihre Unterstützung beim Flow-Setup-System und Eleanor Fox und Yunyun Xiao für die kritische Lektüre des Manuskripts. P-L.M. wurde von der internationalen Max Planck Research School IMPRS-Biology and Computation (IMPRS-BAC) gefördert. PK wurde vom DFG-SFB1444 gefördert. Abbildung 1 wurde mit BioRender erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| µ-Slide VI 0.4 | ibidi | 80606 | 6-channel slide |
| Ammonium Chloride | Carl Roth | K298.1 | Quenching |
| Bovine Serum Albumin | Carl Roth | 8076.4 | Blocking |
| DAPI | Sigma Aldrich/ Merck | D9542 | Stain DNA/Nuclei |
| DPBS | PAN Biotech | P04-53500 | PBS |
| Duolink In Situ Detection Reagents Green | Sigma Aldrich/ Merck | DUO92014 | PLA kit containing Ligase, ligation buffer, polymerase and amplification buffer (with green labeled oligonucleotides) |
| Duolink In Situ PLA Probe Anti-Mouse MINUS | Sigma Aldrich/ Merck | DUO92004 | MINUS probe |
| Duolink In Situ PLA Probe Anti-Rabbit PLUS | Sigma Aldrich/ Merck | DUO92002 | PLUS probe |
| Duolink In Situ Wash Buffers, Fluorescence | Sigma Aldrich/ Merck | DUO82049 | PLA wash buffers A and B |
| Endothelial Cell Growth Supplement | Corning | supplement for medium (ECGS) | |
| Fetal calf Serum | supplement for medium | ||
| FIJI | Image Analysis software | ||
| Formaldehyde solution 4% buffered | KLINIPATH/VWR | VWRK4186.BO1 | PFA |
| Full medium | M199 basal medium +20 % FCS +1 % P/S + 2 nM L-Glu + 25 µg/mL Hep + 50 µg/mL ECGS | ||
| Gelatin from porcine skin, Type A | Sigma Aldrich | G2500 | Use 0.1% in PBS for coating of flow channels |
| GraphPad Prism v.7 | GarphPad | Statistical Program used for the Plots and statistical calculations | |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H4784-250MG | supplement for medium (Hep) |
| HUVECs | |||
| ibidi Mounting Medium | ibidi | 50001 | Liquid mounting medium |
| ibidi Pump System | ibidi | 10902 | pneumatic pump |
| Leica TCS SP8 | Leica | confocal microscope | |
| L-Glutamin 200mM | PAN Biotech | P04-80100 | supplement for medium (L-Glu) |
| Medium 199 | Sigma Aldrich | M2154 | Base medium |
| mouse anti- SMAD1 Antibody | Abcam | ab53745 | Suited for PLA |
| mouse anti- SMAD2/3 Antibody | BD Bioscience | 610843 | Not suited for PLA in combination with CST 9515 |
| mousee anti- SMAD4 Antibody | Sanata Cruz Biotechnology | sc-7966 | Suited for PLA |
| Penicillin 10.000U/ml /Streptomycin 10mg/ml | PAN Biotech | P06-07100 | supplement for medium (P/S) |
| Perfusion Set WHITE | ibidi | 10963 | Tubings used for 1 dyn/cm2 |
| Perfusion Set YELLOW and GREEN | ibidi | 10964 | Tubings used for 30 dyn/cm2 |
| rabbit anti- phospho SMAD1/5 Antibody | Cell Signaling Technologies | 9516 | Suited for PLA |
| rabbit anti- SMAD2/3 XP Antibody | Cell Signaling Technologies | 8685 | Suited for PLA |
| rabbit anti- SMAD4 Antibody | Cell Signaling Technologies | 9515 | Not suited for PLA in combination with BD 610843 |
| Serial Connector for µ-Slides | ibidi | 10830 | serial connection tubes |
| Triton X-100 | Carl Roth | 6683.1 | Permeabilization |
Referenzen
- Yadin, D., Knaus, P., Mueller, T. D. Structural insights into BMP receptors: Specificity, activation and inhibition. Cytokine and Growth Factor Reviews. 27, 13-34 (2016).
- Sieber, C., Kopf, J., Hiepen, C., Knaus, P. Recent advances in BMP receptor signaling. Cytokine and Growth Factor Reviews. 20 (5-6), 343-355 (2009).
- Hiepen, C., et al. BMPR2 acts as a gatekeeper to protect endothelial cells from increased TGFβ responses and altered cell mechanics. PLoS Biology. 17 (12), 3000557 (2019).
- Hildebrandt, S., et al. ActivinA induced SMAD1/5 Signaling in an iPSC derived EC model of Fibrodysplasia Ossificans Progressiva (FOP) can be rescued by the drug candidate saracatinib. Stem Cell Reviews and Reports. , (2021).
- Goumans, M. J., et al. Balancing the activation state of the endothelium via two distinct TGF-beta type I receptors. The EMBO Journal. 21 (7), 1743-1753 (2002).
- Goumans, M. J., et al. Activin receptor-like kinase (ALK)1 is an antagonistic mediator of lateral TGFbeta/ALK5 signaling. Molecular Cell. 12 (4), 817-828 (2003).
- Daly, A. C., Randall, R. A., Hill, C. S. Transforming growth factor beta-induced Smad1/5 phosphorylation in epithelial cells is mediated by novel receptor complexes and is essential for anchorage-independent growth. Molecular and Cellular Biology. 28 (22), 6889-6902 (2008).
- Ramachandran, A., et al. TGF-β uses a novel mode of receptor activation to phosphorylate SMAD1/5 and induce epithelial-to-mesenchymal transition. eLife. 7, 31756 (2018).
- Flanders, K. C., et al. Brightfield proximity ligation assay reveals both canonical and mixed transforming growth factor-β/bone morphogenetic protein Smad signaling complexes in tissue sections. The Journal of Histochemistry and Cytochemistry : The Official Journal of The Histochemistry Society. 62 (12), 846-863 (2014).
- Miyazono, K., Maeda, S., Imamura, T., Dijke, P. T., Heldin, C. -. H. . Smad Signal Transduction: Smads in Proliferation, Differentiation and Disease. , 277-293 (2006).
- Goumans, M. J., Zwijsen, A., Ten Dijke, P., Bailly, S. Bone morphogenetic proteins in vascular homeostasis and disease. Cold Spring Harbor Perspectives in Biology. 10 (2), 031989 (2018).
- Cai, J., Pardali, E., Sánchez-Duffhues, G., ten Dijke, P. BMP signaling in vascular diseases. FEBS Letters. 586 (14), 1993-2002 (2012).
- Cunha, S. I., Magnusson, P. U., Dejana, E., Lampugnani, M. G. Deregulated TGF-β/BMP signaling in vascular malformations. Circulation research. 121 (8), 981-999 (2017).
- MacCarrick, G., et al. Loeys-Dietz syndrome: a primer for diagnosis and management. Genetics in Medicine : An Official Journal of the American College of Medical Genetics. 16 (8), 576-587 (2014).
- Baeyens, N., Bandyopadhyay, C., Coon, B. G., Yun, S., Schwartz, M. A. Endothelial fluid shear stress sensing in vascular health and disease. The Journal of Clinical Investigation. 126 (3), 821-828 (2016).
- Min, E., et al. Activation of Smad 2/3 signaling by low shear stress mediates artery inward remodeling. bioRxiv. , 691980 (2019).
- Zhou, J., et al. BMP receptor-integrin interaction mediates responses of vascular endothelial Smad1/5 and proliferation to disturbed flow. Journal of Thrombosis and Haemostasis. 11 (4), 741-755 (2013).
- Zhou, J., et al. Force-specific activation of Smad1/5 regulates vascular endothelial cell cycle progression in response to disturbed flow. Proceedings of the National Academy of Sciences of the United States of America. 109 (20), 7770-7775 (2012).
- van Dijk, R. A., et al. Visualizing TGF-β and BMP signaling in human atherosclerosis: A histological evaluation based on Smad activation. Histology and Histopathology. 27 (3), 387-396 (2012).
- Derwall, M., et al. Inhibition of bone morphogenetic protein signaling reduces vascular calcification and atherosclerosis. Arteriosclerosis, Thrombosis, and Vascular Biology. 32 (3), 613-622 (2012).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nature Biotechnology. 20 (5), 473-477 (2002).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nature Methods. 3 (12), 995-1000 (2006).
- Alam, M. S. Proximity Ligation Assay (PLA). Current Protocols in Immunology. 123 (1), 58 (2018).
- Application Note 03: Growing Cells in µ-Channels. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN03_Growing_cells.pdf (2012)
- Application Note 13: HUVECs under perfusion. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN13_HUVECs_under_perfusion.pdf (2019)
- ibidi. Application Note 31: Instructions µ-Slide VI 0.4. ibidi. , (2013).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Reichenbach, M., et al. Differential impact of fluid shear stress and YAP/TAZ on BMP/TGF-β induced osteogenic target genes. Advanced Biology. 5 (2), 2000051 (2021).
- Hiepen, C., Mendez, P. L., Knaus, P. It takes two to tango: Endothelial TGFβ/BMP signaling crosstalk with mechanobiology. Cells. 9 (9), 1965 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten