Method Article
Limpieza selectiva de nematodos silvestres de Caenorhabditis para enriquecer las bacterias del microbioma intestinal
En este artículo
Resumen
Los nematodos de Caenorhabditis silvestre están asociados con muchos microbios, a menudo en la luz intestinal o infectando el intestino. Este protocolo detalla un método para enriquecer microbios inculturables que colonizan el intestino, aprovechando la resistencia de la cutícula dauer.
Resumen
Caenorhabditis elegans (C. elegans) ha demostrado ser un excelente modelo para estudiar las interacciones huésped-microbioma y el microbioma, especialmente en el contexto de los intestinos. Recientemente, el muestreo ecológico de nematodos silvestres de Caenorhabditis ha descubierto una amplia gama de microbios asociados, incluidas bacterias, virus, hongos y microsporidios. Muchos de estos microbios tienen fenotipos interesantes de colonización o infección que justifican un mayor estudio, pero a menudo son inculturables. Este protocolo presenta un método para enriquecer los microbios intestinales deseados en C. elegans y nematodos relacionados y reducir la presencia de los muchos microbios contaminantes que se adhieren a la cutícula. Este protocolo implica forzar a los animales a la etapa de desarrollo y usar una serie de lavados de antibióticos y detergentes para eliminar la contaminación externa. Como los animales dauer tienen cambios fisiológicos que protegen a los nematodos de las duras condiciones ambientales, cualquier microbio intestinal estará protegido de estas condiciones. Pero, para que el enriquecimiento funcione, el microbio de interés debe mantenerse cuando los animales se convierten en dauers. Cuando los animales abandonan la etapa de dauer, se propagan individualmente en líneas individuales. Las poblaciones de F1 se seleccionan para los microbios deseados o los fenotipos de infección y contra la contaminación visible. Estos métodos permitirán a los investigadores enriquecer microbios inculturables en la luz intestinal, que forman parte del microbioma natural de C. elegans y patógenos intestinales intracelulares. Estos microbios pueden ser estudiados para la colonización o fenotipos de infección y sus efectos sobre la aptitud del huésped.
Introducción
El organismo modelo genético C. elegans es un excelente sistema in vivo para estudiar las interacciones huésped-microbio1,2. Tienen una fisiología relativamente simple en comparación con otros animales, sin embargo, gran parte de su biología celular es fundamentalmente similar a la de los mamíferos, lo que los convierte en un buen modelo para la investigación biológica1,3,4. Además, son microscópicos, fáciles de mantener y permanecen transparentes durante su corta vida útil. Estas propiedades permiten estudios rápidos de los mecanismos que rigen las interacciones huésped-microbio y visualización de la infección in vivo y colonización de los huéspedes genéticamente flexibles5,6. Finalmente, C. elegans responde rápidamente a las infecciones bacterianas, fúngicas y virales, lo que las convierte en un excelente modelo para estudiar las interacciones huésped-microbio y el microbioma intestinal7,8,9.
Un aumento en el muestreo de C. elegans silvestres y otros nematodos ha permitido investigar la ecología de los nematodos de vida libre y la variación genética natural10,11. Al mismo tiempo, el muestreo también ha aumentado el descubrimiento de patógenos biológicos y microbios naturales que interactúan con C. elegans12,13,14,15, lo que lleva al establecimiento de muchos sistemas modelo huésped-microbio que estudian las interacciones con virus, bacterias, microsporidios, oomicetos u hongos16,17,18,19,20 . Típicamente, la C. elegans silvestre se encuentra en tallos y frutos podridos, a menudo en climas más templados, y en su mayoría se autorreproducen21. Cuando estas muestras se llevan al laboratorio, los nematodos silvestres se aíslan en poblaciones clonales, llevando una serie de microbiota asociada. Al descubrir nuevos microbios de interés en los nematodos caenorhabditis, los animales a menudo son examinados directamente para detectar infecciones o colonización por microscopía utilizando fenotipos fácilmente visualizables. Por ejemplo, la infección viral puede visualizarse como una desintegración de las estructuras intestinales, y las etapas microsporidios se pueden ver dentro de las células huésped como esporas o merontes14,22. Cuando se descubre un microbio de interés para futuras investigaciones, debe separarse de los otros microbios contaminantes que se encuentran en los nematodos silvestres para que pueda estudiarse de forma aislada. En muchos casos, el microbio de interés no se puede cultivar in vitro, por lo que es esencial enriquecer el microbio en el nematodo huésped.
Por ejemplo, este protocolo describe un aislado silvestre de C. tropicalis que contiene una bacteria que coloniza dentro de la luz intestinal de los nematodos, adhiriéndose a las células epiteliales intestinales de manera direccional. Fenotípicamente, la bacteria crece perpendicularmente a lo largo de los lados internos de la luz intestinal, dándole una apariencia de cerdas, visualizada en un microscopio Normarski estándar en todas las etapas del animal, incluida la etapa dauer. La placa del medio de crecimiento de nematodos (NGM) en la que se cultivó esta cepa silvestre de C. tropicalis contenía contaminación visible con otros microbios. Este protocolo fue desarrollado para reducir el crecimiento microbiano contaminante adicional en las placas para estudiar esta bacteria adherente desconocida. Los nematodos fueron forzados a entrar en la etapa de dauer para proteger las bacterias en el lumen, y luego se limpiaron con una serie de lavados. Posteriormente, se identificaron las especies bacterianas desconocidas mediante disección de los intestinos y amplificación por PCR del ADN ribosómico 16S para su secuenciación.
En general, este protocolo puede enriquecer potencialmente cualquier microbio de interés asociado con un nematodo capturado en la naturaleza. Después, los investigadores identificarán el microbio objetivo, visualizarán fenotipos de infección o colonización in vivo a través de microscopía y estudiarán los efectos sobre la aptitud del huésped u otros aspectos de las interacciones huésped-microbio. El aislamiento y la investigación de nuevas especies microbianas que interactúan con los nematodos Caenorhabditis pueden descubrir los mecanismos genéticos de la inmunidad del huésped y nuevos paradigmas de las interacciones huésped-microbio relevantes para la patogénesis microbiana y los estudios del microbioma.
Protocolo
1. Inducción de la formación de dauer para nematodos silvestres en placas NGM
- Cultive la cepa silvestre caenorhabditis en placas NGM estándar con OP50-1 E. coli como fuente de alimento después de obtener los gusanos con un microbio de interés incultivable. Incubar los nematodos a 20 °C.
NOTA: La temperatura estándar para cultivar nematodos Caenorhabditis está entre 15-25 ° C, pero debe determinarse empíricamente para evitar la pérdida del microbio de interés. - Matar de hambre el plato de animales a 20 ° C durante 4-5 días hasta que se consuma todo el OP50-1 y la mayoría haya alcanzado la etapa de dauer.
NOTA: Los Dauers son larvas de larga vida que se desarrollan debido a la ausencia de nutrición y tienen cutículas protectoras.
2. Lavado de los nematodos
- Agregue 5 ml de medios de sales mínimas M9 (42 mM Na2HPO4, 22 mM KH2PO4, 8.6 mM NaCl, 19 mM NH4Cl) a una placa de 6 cm de gusanos hambrientos.
- Usando una pipeta y una bombilla de vidrio estéril, pipetee el M9 y los gusanos de la placa y transfiéralos a un tubo de centrífuga estéril de 15 ml.
- Usando una centrífuga clínica, gire hacia abajo los gusanos en el tubo a 1000 x g durante 30 s a temperatura ambiente.
- Usando una pipeta estéril de 15 ml, retire y deseche el sobrenadante M9 por encima de la bolita de gusanos. No moleste a los gusanos vivos en la parte inferior del tubo dejando aproximadamente 50 μL de M9 por encima de los gusanos.
- Agregue 10 ml de M9 + 0.05% de Triton X-100 al mismo tubo de centrífuga y apriete adecuadamente la tapa del tubo.
- Incubar el tubo en un nutator durante 20 min a temperatura ambiente para eliminar los microbios externos. Retire el tubo del nutator y gire hacia abajo los gusanos a 1000 x g durante 30 s.
- Usando una pipeta estéril, retire y deseche el sobrenadante M9 por encima de la bolita de gusanos. No moleste a los gusanos vivos en la parte inferior del tubo dejando aproximadamente 50 μL de M9 por encima de los gusanos.
- Siga y repita los pasos 2.5-2.8 tres veces más.
3. Desinfección de los nematodos
- Preparar una solución antibiótica y SDS en 10 ml de tampón M9 añadiendo 0,25% de SDS (250 μL de 10% de SDS), 50 μg/ml de carbenicilina, 25 μg/ml de kanamicina, 12,5 μg/ml de tetraciclina, 100 μg/ml de gentamicina, 50 μg/ml de estreptomicina, 37,5 μg/ml de cloranfenicol y 200 μg/ml de cefotaxima (ver Tabla de materiales).
- Incubar el tubo que contiene nematodos en la solución de antibiótico y SDS en un nutator a temperatura ambiente durante la noche.
NOTA: Todos los gusanos y embriones que no son dauer morirán, pero muchos de los gusanos dauer sobrevivirán a este proceso de limpieza.
4. Extracción del antibiótico y la solución de SDS
- Gire hacia abajo los gusanos en el tubo a 1000 x g durante 1 minuto a temperatura ambiente. Con una pipeta estéril, retire el sobrenadante del tubo de la centrífuga sin molestar a los gusanos en la parte inferior del tubo.
- Añadir 10 mL de M9 + 0,05% Tritón X-100 y apretar adecuadamente la tapa del tubo. Incubar el tubo en un nutator durante 20 min a temperatura ambiente.
- Retire el tubo del nutator y gire hacia abajo los gusanos a 1000 x g durante 1 min. Siga y repita los pasos 4.2-4.3 tres veces.
- Después del último lavado, deje el pellet de gusano sin molestar en la parte inferior en 100 μL de la solución y deseche el resto.
5. Propagar una cepa de nematodo limpia
- Usando una pipeta de vidrio estéril, transfiera 100 μL del sobrenadante y el pellet al centro de una placa NGM de 10 cm sembrada con OP50-1. Deje que la placa se seque sin molestar el líquido en el centro.
- Permita que los dauers se arrastren fuera del centro y a través del césped OP50-1 durante 5-10 minutos.
- Recoja cuidadosamente un solo dauer y cuéntelo en una placa con semillas ngm de 6 cm. En total, recoja al menos 10 dauers y enchapándolos en placas NGM individuales de 6 cm sembradas con OP50-1 (10 placas en total).
- Incubar las placas durante 4-5 días a 20 °C hasta que los dauers hayan crecido y la siguiente generación (F1) haya alcanzado la etapa adulta. Verifique visualmente si hay contaminación en todas las placas, vista como crecimiento microbiano no OP50-1.
- Para cada placa limpia, verifique la propagación del microbio de interés a través de Normarski o microscopía fluorescente, como la hibridación fluorescente in situ (FISH)12, dependiendo del fenotipo de interés.
6. Disección intestinal e identificación por PCR de especies microbianas
- Cultive animales a 20 ° C durante 3-4 días para permitirles morir de hambre y reducir la cantidad de bacterias OP50-1. Agregue 5 ml de medios M9 a la placa de gusanos hambrientos.
- Usando una pipeta y una bombilla de vidrio estéril, pipetee los gusanos M9 + de la placa y transfiéralos a un tubo de centrífuga estéril de 15 ml.
- Usando una centrífuga clínica, gire hacia abajo los gusanos en el tubo a 1000 x g durante 30 s a temperatura ambiente.
- Usando una pipeta estéril de 15 ml, retire el sobrenadante del tubo de la centrífuga sin molestar a los gusanos vivos en la parte inferior del tubo.
- Agregue 10 ml de M9 + 0.05% Triton X-100 e incube el tubo en un nutator durante 20 minutos para eliminar los microbios externos. Repita cuatro veces para lavar los gusanos.
- Después del último lavado, con una punta de pipeta estéril, retire el sobrenadante sin molestar el gránulo en la parte inferior en 100 μL de la solución.
- Usando una punta de pipeta estéril, transfiera M9 y los gusanos a una placa NGM sin semilla y deje que la placa se seque mientras los gusanos se arrastran durante 20 minutos para ayudar a eliminar OP50-1 de la cutícula y el intestino.
- Agregue 250 μL de M9 a la placa seca y use una pipeta de vidrio para transferir gusanos M9 + a una nueva placa NGM sin semilla. Una vez más, deje que la placa se seque y deje que los gusanos se arrastren durante 20 minutos.
- Agregue 250 μL de M9 a la placa y transfiera 100 μL de los gusanos M9 + a un vidrio de reloj limpio.
- Usando dos agujas de jeringa estériles de 26 G, decapita los nematodos sosteniendo el gusano con una aguja y usando la otra aguja para cortar el gusano. Después de la decapitación, el intestino (granular) y la gónada (transparente) del cuerpo saldrán naturalmente del cuerpo.
- Corte un pedazo del intestino expuesto sosteniendo el intestino con una aguja y cortándolo con la otra.
NOTA: Si es posible, verifique que el microbio de interés todavía esté presente en los intestinos evertidos utilizando la microscopía Normarski, FISH o inmunohistoquímica23. - Transfiera un solo intestino diseccionado a un tubo de PCR de 0,5 ml que contenga 10 μL de agua estéril. Repetir para un total de al menos 5 tubos de PCR que contengan intestinos de diferentes animales.
- Congelar los tubos de PCR a -80 °C durante un mínimo de 5 min. Retire los tubos de PCR del congelador y descongele las muestras.
- Pipetear el líquido hacia arriba y hacia abajo varias veces para separar las bacterias del intestino.
- Realizar PCR utilizando pares de cebadores universales contra la secuencia de ADN de la pequeña subunidad ribosómica de bacterias, levaduras o microsporidios, dependiendo del tipo de microbio sospechoso.
NOTA: Por ejemplo, en la Tabla 1 se presenta un protocolo de muestra que utiliza cebadores bacterianos universales 16S. - Purifique el amplicón utilizando un kit de limpieza y concentración de ADN basado en filtro (consulte la Tabla de materiales) y realice la secuenciación de Sanger.
Resultados
Una cepa silvestre de C. tropicalis (JU1848) fue aislada de frutos de palmeras podridas en el Bosque de Nouragues de la Guayana Francesa (Figura 1A)24. Se encontró que esta cepa tiene microbios delgados que colonizan la luz del intestino de manera direccional (Figura 1B). Este microbio se transfirió fácilmente a la cepa N2 de C. elegans a través del cocultivo con la cepa JU1848, donde colonizó la luz del intestino de manera similar.
La propagación de JU1848 en placas NGM estándar sembradas con E. coli OP50-1 durante múltiples generaciones resultó continuamente en contaminación visible, vista como varias colonias oscuras de mucoides dentro y fuera del césped OP50-1 (Figura 2A). Una placa de JU1848 salvaje fue hambrienta para obligar a los animales a entrar en dauer y limpiada como se describe. Los animales de dauer individual que sobrevivieron a la limpieza fueron chapados en placas NGM individuales de 6 cm sembradas con OP50-1 y se les permitió crecer durante 4 días a 20 ° C. Se observaron múltiples placas de progenie F1 sin contaminación microbiana visible (Figura 2B). Se verificó que la progenie F1 todavía contenía bacterias adheridas en la luz del intestino (ver más abajo).
Los animales limpios JU1848 fueron lavados y decapitados para aislar las piezas intestinales como se describe en el protocolo (pasos 6.1-6.12). Las bacterias adheridas en la luz del intestino diseccionado se verificaron mediante microscopía nomarski (Figura 3). Se sospechaba que el microbio en el lumen de JU1848 era una bacteria, por lo que los intestinos disecados se utilizaron como plantilla para pcr utilizando cebadores bacterianos universales 16S, 27F y 1492R. De un total de seis intestinos disecados individuales, los productos de PCR se secuenciaron a través de Sanger, y los cromatógrafos limpios mostraron que las seis secuencias eran idénticas. Sobre la base de estas secuencias, esta bacteria fue identificada como una nueva especie en la clase Alphaproteobacteria, pero no pudo clasificarse en un orden o género conocido (Archivo Complementario 1).

Figura 1: Bacterias adheridas que colonizan la luz de un C. tropicalis silvestre. (A) Imagen de muestra de campo de frutos podridos de palmeras Euterpe sp. (Familia: Arecaceae) en el bosque de Nouragues de la Guayana Francesa. (B) Imagen de Nomarski de la cepa JU1848 vista con miles de bacterias largas y delgadas que forman una apariencia de cepillo en la luz (lu) del intestino del huésped (in). Las capas bacterianas (ba) que recubren el intestino están indicadas con brackets ([). Haga clic aquí para ver una versión más grande de esta figura.
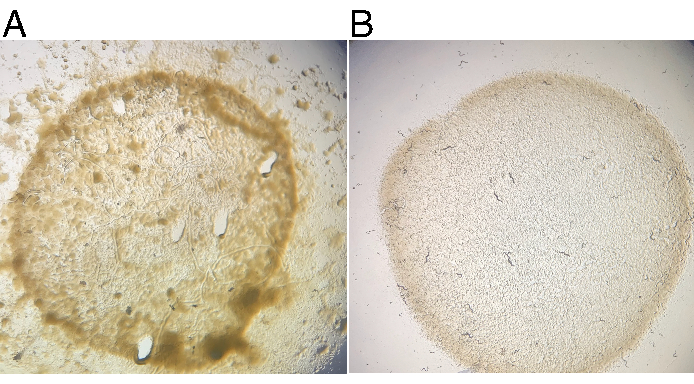
Figura 2: El crecimiento microbiano contaminante se pierde después de la limpieza de nematodos. (A) La cepa silvestre JU1848 propaga un crecimiento microbiano notable en placas NGM estándar de 6 cm sembradas con bacterias E. coli OP50-1. (B) Después de la limpieza, una placa de progenie F1 de un solo dauer no muestra contaminación microbiana visible después de 4 días de incubación a 20 ° C. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Las bacterias adheridas se ven en la luz del intestino disecado. Imagen de Nomarski de un animal JU1848 limpio que fue decapitado para que los intestinos se derramaran. Las bacterias colonizadoras (ba) están indicadas con un corchete ([) y se ven en la luz (lu) de un pedazo del intestino (in) que está fuera del cuerpo del nematodo (nb). Haga clic aquí para ver una versión más grande de esta figura.
| Reactivo | Concentración | Importe |
| Imprimación 27F (5'-AGAGTTTGATCMTGGCTCAG-3') | 20 metros | 2,5 μL |
| Imprimación 1492R (5'-GGTTACCTTGTTACGACTT-3') | 20 metros | 2,5 μL |
| Intestino diseccionado en agua | N/A | 3 μL |
| Búfer de PCR 10x | 10x | 5 μL |
| dNTP | 10 mM | 1 μL |
| Taq Polimerasa | 5 U/μL | 0,5 μL |
| Agua | N/A | 35,5 μL |
Tabla 1: Muestra del protocolo de PCR utilizando cebadores bacterianos universales e intestino disecado.
Archivo complementario 1: Alineación MUSCULAR de secuencias bacterianas de ADNr 16S derivadas de PCR de seis intestinos JU1848 disecados. Haga clic aquí para descargar este archivo.
Discusión
Este protocolo describe el aislamiento y la identificación de microbios de nematodos Caenorhabditis aislados en la naturaleza mediante una serie de procedimientos de limpieza. Numerosos microbios están asociados con nematodos aislados en la naturaleza, y algunos de ellos tienen fenotipos emocionantes que se pueden usar para futuros estudios en interacciones huésped-microbio e inmunidad innata. Se han aislado muchos microbiomas cultivables y bacterias patógenas de nematodos silvestres de Caenorhabditis utilizando técnicas estándar para el crecimiento bacteriano in vitro25,26. Sin embargo, no todos los microbios se pueden cultivar in vitro, y se hace necesario enriquecerlos en nematodos silvestres. Algunos microbios tienen una etapa de esporas resistentes, como los microsporidios, y se pueden usar altas concentraciones de SDS para matar la mayoría de las bacterias y hongos, lo que permite un enriquecimiento específico de las esporas12. Este protocolo presenta un método para enriquecer microbios intestinales incultivables que no son resistentes a la SDS y al tratamiento con antibióticos.
La técnica aquí presentada aprovecha la resistencia ambiental observada en los animales dauer debido a cambios fisiológicos como el fortalecimiento de la cutícula, la supresión del bombeo faríngeo y el recubrimiento de la boca con un tapón bucal27. Un paso crítico en este protocolo es la incubación durante la noche con varios antibióticos y 0.25% SDS. Este paso se utiliza para matar todos los microbios externos mientras se dejan intactos los microbios internos. Si bien se ha demostrado que C. elegans dauers sobrevive a concentraciones de SDS tan altas como 10% durante 30 min27, este protocolo utiliza una incubación moderada pero prolongada no solo para matar microbios sino también para exponer aún más a las bacterias a los antibióticos. Además, una concentración moderada de SDS puede ayudar a garantizar que los dauers de otras especies de Caenorhabditis sobrevivan, ya que la exposición de C. tropicalis al 1% de SDS durante la noche resultó en la muerte de todos los animales dauer. Si todos los dauers mueren, entonces la concentración de SDS y / o la duración de la exposición a SDS deben reducirse. Por el contrario, si las placas de generación F1 todavía tienen contaminación visible después de la limpieza, se debe aumentar la concentración de SDS y el tiempo de incubación.
Otro paso crítico es el aislamiento de animales de dauer individual después de la limpieza. Este paso es crucial ya que no todos los animales están limpios después de la SDS y el tratamiento con antibióticos. Por lo tanto, los animales se colocan en el centro de una placa NGM de 10 cm con OP50-1 y se les permite arrastrarse radialmente hacia afuera. A menudo es mejor elegir más animales distales, ya que el rastreo prolongado a través de OP50-1 parece ayudar a eliminar cualquier posible microbio sobreviviente unido a la cutícula. Sin embargo, esto conduce a una limitación del protocolo, ya que será más difícil enriquecer para un microbio de interés si no está presente en la población a una frecuencia alta. Aquí, la Alfaproteobacteria adherente estaba presente en el 90%-95% de la población; por lo tanto, la mayoría de las placas limpias tenían la bacteria del microbioma. Sin embargo, si un microbio de interés está presente a una frecuencia mucho menor en la población, puede ser necesario examinar muchas más placas F1 .
Este protocolo probablemente podría usarse para aislar cualquier número de microbios no cultivables de interés que se encuentran en los nematodos silvestres. Sin embargo, el microbio debe estar en un tejido protegido por la cutícula dauer, capaz de sobrevivir en animales dauer, y tener un fenotipo observable en el huésped. Como tal, esta técnica se puede utilizar para enriquecer otras bacterias del microbioma en la luz intestinal además de las especies de Alphaproteobacteria descritas aquí, incluidas las bacterias que no se adhieren. Asimismo, se utilizó el protocolo para enriquecer una bacteria intracelular facultativa, Bordetella atropi, que infecta al nematodo Oscheius tipulae28. Después del enriquecimiento, se encontró que B. atropi forma colonias en placas NGM, lo que demuestra que se puede descubrir que un microbio de interés es cultivable in vitro una vez que se eliminan los contaminantes de crecimiento más rápido. Esta técnica probablemente funcionaría para microspridios y virus, incluido el virus de Orsay, dada esta capacidad de enriquecer una bacteria intracelular. Sin embargo, estos microbios deben ser capaces de sobrevivir a la transición dentro y fuera de dauer.
Es importante recordar que, si bien este protocolo se puede realizar en un laboratorio de Nivel 1 de Bioseguridad, se debe mantener una técnica estéril para evitar una mayor contaminación microbiana. El protocolo se puede cambiar de acuerdo con las necesidades del investigador, incluidos los tipos / concentraciones de antibióticos, el porcentaje de SDS y / o la adición de antifúngicos como la nistatina. A menudo, el número de microbios contaminantes que se encuentran en un nematodo aislado en la naturaleza puede variar drásticamente. Aquí, la pérdida aparente del crecimiento de E. coli no OP50-1 en placas NGM se utilizó como lectura para una cepa de nematodo limpia. Pero, puede haber poblaciones no cultivables de microbios contaminantes presentes, por lo que es esencial realizar un método de metagenómica como la secuenciación del amplicón 16S rRNA para ver el alcance de la contaminación26. Una vez que se limpia la cepa del gusano, se puede congelar y almacenar para futuros estudios. En general, este protocolo permite a los investigadores enriquecer microbios inculturables en nematodos silvestres, lo que les permite estudiar los efectos sobre la aptitud del huésped, caracterizar fenotipos de colonización o infección y aprovechar las herramientas genéticas para comprender los mecanismos subyacentes a las interacciones huésped-microbio.
Divulgaciones
Los autores declaran que no hay conflictos de intereses.
Agradecimientos
Gracias al Dr. Christian Braendle y a la Estación de Campo Nouragues del Centre Nationale de la Recherche Scientifique (CNRS).
Materiales
| Name | Company | Catalog Number | Comments |
| Agarose | Fisher Scientific | BP1356 | |
| 10% SDS | Invitrogen | AM9822 | |
| BD PrecisionGlide Needle - 26 G | Fisher Scientific | 305115 | |
| Carbenicillin | Millipore-Sigma | C1389-1G | |
| Cefotaxime | Millipore-Sigma | C7039-500mg | |
| Chloramphenicol | Millipore-Sigma | C0378-25G | |
| DNA Clean and Concentrator Kit | Zymo Research | 11-303C | |
| DreamTaq Polymerase | Fisher Scientific | EP0711 | |
| Gentamycin | Millipore-Sigma | G1264-250mg | |
| Kanamycin | Millipore-Sigma | K1876-1G | |
| KH2PO4 | Fisher Scientific | P-286 | |
| NaCl | Fisher Scientific | S-671 | |
| NH4Cl | Fisher Scientific | A-661 | |
| Streptomycin | Millipore-Sigma | S6501-50G | |
| Tetracyclin | Millipore-Sigma | T7660-5G | |
| Triton X-100 | Fisher Scientific | BP-151 | |
| Watch glasses | VWR | 470144-850 |
Referencias
- Balla, K. M., Troemel, E. R. Caenorhabditis elegans as a model for intracellular pathogen infection. Cellular Microbiology. 15, 1313-1322 (2013).
- Pukkila-Worley, R., Ausubel, F. M. Immune defense mechanisms in the Caenorhabditis elegans intestinal epithelium. Current Opinion in Immunology. 24, 3-9 (2012).
- Bossinger, O., Fukushige, T., Claeys, M., Borgonie, G., McGhee, J. D. The apical disposition of the Caenorhabditis elegans intestinal terminal web is maintained by LET-413. Developmental Biology. 268, 448-456 (2004).
- Dimov, I., Maduro, M. F. The C. elegans intestine: organogenesis, digestion, and physiology. Cell and Tissue Research. 377, 383-396 (2019).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Szumowski, S. C., Botts, M. R., Popovich, J. J., Smelkinson, M. G., Troemel, E. R. The small GTPase RAB-11 directs polarized exocytosis of the intracellular pathogen N. parisii for fecal-oral transmission from C. elegans. Proceedings of the National Academy of Sciences of the United States of America. 111, 8215-8220 (2014).
- Bakowski, M. A., et al. Ubiquitin-mediated response to microsporidia and virus infection in C. elegans. PLoS Pathogens. 10, 1004200 (2014).
- Sowa, J. N., et al. The Caenorhabditis elegans RIG-I Homolog DRH-1 Mediates the Intracellular Pathogen Response upon Viral Infection. Journal of Virology. 94, 01173 (2020).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15, 833-838 (2014).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10, 59 (2012).
- Lee, D., et al. Balancing selection maintains hyper-divergent haplotypes in Caenorhabditis elegans. Nature Ecology and Evolution. 5, 794-807 (2021).
- Luallen, R. J., et al. Discovery of a natural microsporidian pathogen with a broad tissue tropism in Caenorhabditis elegans. PLOS Pathogens. 12, 1005724 (2016).
- Osman, G. A., et al. Natural infection of C. elegans by an oomycete reveals a new pathogen-specific immune response. Current Biology. 28, 640-648 (2018).
- Felix, M. A., et al. Natural and experimental infection of Caenorhabditis nematodes by novel viruses related to nodaviruses. PLoS Biology. 9, 1000586 (2011).
- Zhang, G., et al. A large collection of novel nematode-infecting microsporidia and their diverse interactions with Caenorhabditis elegans and other related nematodes. PLoS Pathogens. 12, 1006093 (2016).
- Félix, M. -. A., Wang, D. Natural viruses of Caenorhabditis nematodes. Annual Review Genetics. 53, 313-326 (2019).
- Grover, M., Barkoulas, M. C. elegans as a new tractable host to study infections by animal pathogenic oomycetes. PLoS Pathogens. 17, 1009316 (2021).
- Bakowski, M. A., Luallen, R. J., Troemel, E. R. Microsporidia Infections in Caenorhabditis Elegans and Other Nematodes. Microsporidia: Pathogens of Opportunity: First Edition. , (2014).
- Taffoni, C., Pujol, N. Mechanisms of innate immunity in C. elegans epidermis. Tissue Barriers. 3, 1078432 (2015).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10, 3025-3039 (2020).
- Frézal, L., Félix, M. -. A. C. elegans outside the Petri dish. eLife. 4, 05849 (2015).
- Troemel, E. R., Félix, M. -. A., Whiteman, N. K., Barrière, A., Ausubel, F. M. Microsporidia are natural intracellular parasites of the nematode Caenorhabditis elegans. PLoS Biology. 6, 2736-2752 (2008).
- Luallen, R. J., Bakowski, M. A., Troemel, E. R. Characterization of microsporidia-induced developmental arrest and a transmembrane leucine-rich repeat protein in Caenorhabditis elegans. PLoS One. 10, 0124065 (2015).
- Félix, M. -. A., et al. Species richness, distribution and genetic diversity of Caenorhabditis nematodes in a remote tropical rainforest. BMC Evolutionary Biology. 13, 10 (2013).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113, 3941-3949 (2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10, 1998-2009 (2016).
- Androwski, R. J., Flatt, K. M., Schroeder, N. E. Phenotypic plasticity and remodeling in the stress-induced C. elegans dauer. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 278 (2017).
- Tran, T. D., Ali, M. A., Lee, D., Félix, M. -. A., Luallen, R. J. Bacterial filamentation is an in vivo mechanism for cell-to-cell spreading. bioRxiv. , (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados