Method Article
Pulizia selettiva dei nematodi caenorhabiditi selvatici per arricchire i batteri del microbioma intestinale
In questo articolo
Riepilogo
I nematodi caenorhabditis selvatici sono associati a molti microbi, spesso nel lume intestinale o infettando l'intestino. Questo protocollo descrive un metodo per arricchire i microbi incolturabili che colonizzano l'intestino, sfruttando la resistenza della cuticola dauer.
Abstract
Caenorhabditis elegans (C. elegans) ha dimostrato di essere un modello eccellente per studiare le interazioni ospite-microbo e il microbioma, specialmente nel contesto dell'intestino. Recentemente, il campionamento ecologico dei nematodi selvatici Caenorhabditis ha scoperto una vasta gamma di microbi associati, tra cui batteri, virus, funghi e microsporidi. Molti di questi microbi hanno fenotipi di colonizzazione o infezione interessanti che meritano ulteriori studi, ma sono spesso incolpevoli. Questo protocollo presenta un metodo per arricchire i microbi intestinali desiderati in C. elegans e nematodi correlati e ridurre la presenza dei molti microbi contaminanti che aderiscono alla cuticola. Questo protocollo prevede di costringere gli animali nella fase di sviluppo e di utilizzare una serie di lavaggi antibiotici e detergenti per rimuovere la contaminazione esterna. Poiché gli animali dauer hanno cambiamenti fisiologici che proteggono i nematodi da condizioni ambientali difficili, tutti i microbi intestinali saranno protetti da queste condizioni. Ma, affinché l'arricchimento funzioni, il microbo di interesse deve essere mantenuto quando gli animali si sviluppano in dauers. Quando gli animali lasciano lo stadio dauer, vengono propagati singolarmente in singole linee. Le popolazioni F1 vengono quindi selezionate per i microbi desiderati o i fenotipi di infezione e contro la contaminazione visibile. Questi metodi consentiranno ai ricercatori di arricchire microbi incolturabili nel lume intestinale, che costituiscono parte del microbioma naturale di C. elegans e patogeni intestinali intracellulari. Questi microbi possono quindi essere studiati per la colonizzazione o i fenotipi di infezione e i loro effetti sulla forma fisica dell'ospite.
Introduzione
L'organismo modello genetico C. elegans è un eccellente sistema in vivo per studiare le interazioni ospite-microbo1,2. Hanno una fisiologia relativamente semplice rispetto ad altri animali, tuttavia, gran parte della loro biologia cellulare è fondamentalmente simile ai mammiferi, rendendoli un buon modello per la ricerca biologica1,3,4. Inoltre, sono microscopici, facili da mantenere e rimangono trasparenti per tutta la loro breve durata. Queste proprietà consentono studi rapidi sui meccanismi che governano le interazioni ospite-microbo e la visualizzazione dell'infezione in vivo e della colonizzazione degli ospiti geneticamente flessibili5,6. Infine, C. elegans risponde rapidamente alle infezioni batteriche, fungine e virali, rendendole un modello eccellente per studiare le interazioni ospite-microbo e il microbioma intestinale7,8,9.
Un aumento del campionamento di C. elegans selvatici e altri nematodi ha permesso la ricerca sull'ecologia dei nematodi viventi liberi e sulla variazione genetica naturale10,11. Allo stesso tempo, il campionamento ha anche aumentato la scoperta di patogeni biologici naturali e microbi che interagiscono con C. elegans12,13,14,15, portando alla creazione di molti sistemi modello ospite-microbo che studiano le interazioni con virus, batteri, microsporidi, oomiceti o funghi16,17,18,19,20 . Tipicamente, C. elegans selvatico si trova in steli e frutti in decomposizione, spesso in climi più temperati, e per lo più si riproducono da soli21. Quando questi campioni vengono portati in laboratorio, i nematodi selvatici vengono isolati in popolazioni clonali, trasportando una serie di microbiota associati. Quando si scoprono nuovi microbi di interesse per i nematodi Caenorhabditis, gli animali vengono spesso sottoposti a screening diretto per l'infezione o la colonizzazione al microscopio utilizzando fenotipi facilmente visualizzabili. Ad esempio, l'infezione virale può essere visualizzata come una disintegrazione delle strutture intestinali e gli stadi microsporidici possono essere visti all'interno delle cellule ospiti come spore o meroni 14,22. Quando un microbo di interesse viene scoperto per indagini future, deve essere separato dagli altri microbi contaminanti trovati nei nematodi selvatici in modo che possa essere studiato isolatamente. In molti casi, il microbo di interesse non può essere coltivato in vitro, rendendo essenziale arricchire il microbo nel nematode ospite.
Ad esempio, questo protocollo descrive un isolato selvatico di C. tropicalis contenente un batterio che colonizza all'interno del lume intestinale dei nematodi, aderendo alle cellule epiteliali intestinali in modo direzionale. Fenotipicamente, il batterio cresce perpendicolarmente lungo i lati interni del lume intestinale, dandogli un aspetto simile a una setola, visualizzato su un microscopio Normarski standard in tutte le fasi dell'animale, incluso lo stadio dauer. La placca del mezzo di crescita dei nematodi (NGM) su cui è stato coltivato questo ceppo selvatico di C. tropicalis conteneva una contaminazione visibile con altri microbi. Questo protocollo è stato sviluppato per ridurre ulteriormente la crescita microbica contaminante sulle piastre per studiare questo batterio aderente sconosciuto. I nematodi sono stati costretti allo stadio dauer per proteggere i batteri nel lume e quindi puliti utilizzando una serie di lavaggi. Successivamente, la specie batterica sconosciuta è stata identificata dalla dissezione dell'intestino e dall'amplificazione PCR del DNA ribosomiale 16S per il sequenziamento.
Nel complesso, questo protocollo può potenzialmente arricchire qualsiasi microbo di interesse associato a un nematode catturato in natura. Successivamente, i ricercatori identificheranno il microbo bersaglio, visualizzeranno i fenotipi di infezione o colonizzazione in vivo tramite microscopia e studieranno gli effetti sulla forma fisica dell'ospite o altri aspetti delle interazioni ospite-microbo. L'isolamento e lo studio di nuove specie microbiche che interagiscono con i nematodi caenorhabditis possono scoprire i meccanismi genetici dell'immunità dell'ospite e nuovi paradigmi di interazioni ospite-microbo rilevanti per la patogenesi microbica e gli studi sul microbioma.
Protocollo
1. Indurre la formazione di dauer per nematodi selvatici su piastre NGM
- Coltiva il ceppo selvatico Caenorhabditis su piastre NGM standard con OP50-1 E. coli come fonte di cibo dopo aver ottenuto i vermi con un microbo incolturabile di interesse. Incubare i nematodi a 20 °C.
NOTA: La temperatura standard per coltivare i nematodi caenorhabditis è compresa tra 15-25 °C, ma deve essere determinata empiricamente per prevenire la perdita del microbo di interesse. - Affamare la piastra di animali a 20 °C per 4-5 giorni fino a quando tutto l'OP50-1 non viene consumato e la maggior parte ha raggiunto lo stadio dauer.
NOTA: I Dauers sono larve longeve che si sviluppano a causa dell'assenza di nutrizione e hanno cuticole protettive.
2. Lavare i nematodi
- Aggiungere 5 mL di mezzi salici minimi M9 (42 mM Na2HPO4, 22 mM KH2PO4, 8,6 mM NaCl, 19 mM NH4Cl) a una piastra di 6 cm di vermi affamati.
- Utilizzando una pipetta e una lampadina di vetro sterili, pipettare l'M9 e i vermi dalla piastra e trasferirli in un tubo centrifugo sterile da 15 ml.
- Utilizzando una centrifuga clinica, far girare i vermi nel tubo a 1000 x g per 30 s a temperatura ambiente.
- Utilizzando una pipetta sterile da 15 ml, rimuovere e scartare il surnatante M9 sopra il pellet di vermi. Non disturbare i vermi vivi sul fondo del tubo lasciando circa 50 μL di M9 sopra i vermi.
- Aggiungere 10 ml di M9 + 0,05% di Triton X-100 allo stesso tubo della centrifuga e stringere adeguatamente il cappuccio del tubo.
- Incubare il tubo su un nutatore per 20 minuti a temperatura ambiente per rimuovere i microbi esterni. Rimuovere il tubo dal nutator e far girare i vermi a 1000 x g per 30 s.
- Utilizzando una pipetta sterile, rimuovere e scartare il surnatante M9 sopra il pellet di vermi. Non disturbare i vermi vivi sul fondo del tubo lasciando circa 50 μL di M9 sopra i vermi.
- Seguire e ripetere i passaggi 2.5-2.8 altre tre volte.
3. Disinfettare i nematodi
- Preparare una soluzione antibiotica e SDS in 10 mL di tampone M9 aggiungendo 0,25% di SDS (250 μL del 10% SDS), 50 μg/mL di carbenicillina, 25 μg/mL di kanamicina, 12,5 μg/mL di tetraciclina, 100 μg/mL di gentamicina, 50 μg/mL di streptomicina, 37,5 μg/mL di cloramfenicolo e 200 μg/mL di cefotaxima (vedere Tabella dei materiali).
- Incubare il tubo contenente nematodi nella soluzione antibiotica e SDS su un nutatore a temperatura ambiente durante la notte.
NOTA: Tutti i vermi e gli embrioni non-dauer moriranno, ma molti dei vermi dauer sopravviveranno a questo processo di pulizia.
4. Rimozione dell'antibiotico e della soluzione SDS
- Girare i vermi nel tubo a 1000 x g per 1 minuto a temperatura ambiente. Utilizzando una pipetta sterile, rimuovere il surnatante dal tubo della centrifuga senza disturbare i vermi sul fondo del tubo.
- Aggiungere 10 ml di M9 + 0,05% Triton X-100 e stringere adeguatamente il cappuccio del tubo. Incubare il tubo su un nutatore per 20 minuti a temperatura ambiente.
- Rimuovere il tubo dal nutator e far girare i vermi a 1000 x g per 1 minuto. Seguire e ripetere i passaggi 4.2-4.3 tre volte.
- Dopo l'ultimo lavaggio, lasciare indisturbato il pellet di verme sul fondo in 100 μL della soluzione e scartare il resto.
5. Propagare un ceppo di nematodi pulito
- Utilizzando una pipetta di vetro sterile, trasferire 100 μL del surnatante e del pellet al centro di una piastra NGM da 10 cm seminata con OP50-1. Lasciare asciugare la piastra senza disturbare il liquido al centro.
- Lascia che i dauers strisciano fuori dal centro e attraverso il prato OP50-1 per 5-10 min.
- Prelevare con cura un singolo dauer e placcarlo su un piatto seminato NGM da 6 cm. In totale, scegli almeno 10 dauer e placcali in singole piastre NGM da 6 cm seminate con OP50-1 (10 piastre totali).
- Incubare le piastre per 4-5 giorni a 20 ° C fino a quando i dauers sono cresciuti e la generazione successiva (F1) ha raggiunto lo stadio adulto. Controllare visivamente la contaminazione su tutte le piastre, vista come crescita microbica non OP50-1.
- Per ogni piastra pulita, verificare la propagazione del microbo di interesse tramite Normarski o microscopia fluorescente , come l'ibridazione fluorescente in situ (FISH)12, a seconda del fenotipo di interesse.
6. Dissezione intestinale e identificazione PCR di specie microbiche
- Coltiva gli animali a 20 °C per 3-4 giorni per consentire loro di morire di fame e ridurre la quantità di batteri OP50-1. Aggiungere 5 ml di mezzi M9 alla piastra di vermi affamati.
- Utilizzando una pipetta di vetro sterile e una lampadina, pipettare i vermi M9 + dalla piastra e trasferirli in un tubo centrifugo sterile da 15 ml.
- Utilizzando una centrifuga clinica, far girare i vermi nel tubo a 1000 x g per 30 s a temperatura ambiente.
- Utilizzando una pipetta sterile da 15 ml, rimuovere il surnatante dal tubo della centrifuga senza disturbare i vermi vivi sul fondo del tubo.
- Aggiungere 10 ml di M9 + 0,05% Triton X-100 e incubare il tubo su un nutatore per 20 minuti per rimuovere i microbi esterni. Ripeti quattro volte per lavare i vermi.
- Dopo l'ultimo lavaggio, utilizzando una punta di pipetta sterile, rimuovere il surnatante senza disturbare il pellet sul fondo in 100 μL della soluzione.
- Utilizzando una punta di pipetta sterile, trasferire M9 e i vermi su una piastra NGM non seminata e lasciare asciugare la piastra mentre i vermi strisciano per 20 minuti per aiutare a rimuovere OP50-1 dalla cuticola e dall'intestino.
- Aggiungere 250 μL di M9 alla piastra essiccata e utilizzare una pipetta di vetro per trasferire i vermi M9 + su una nuova piastra NGM non seminata. Ancora una volta, lascia asciugare quel piatto e lascia che i vermi strisciano per 20 minuti.
- Aggiungere 250 μL di M9 alla piastra e trasferire 100 μL dei vermi M9 + in un vetro dell'orologio pulito.
- Usando due aghi sterili da 26 G, decapitare i nematodi tenendo il verme verso il basso con un ago e usando l'altro ago per tagliare il verme. Dopo la decapitazione, l'intestino (granulare) e la gonade (trasparente) dal corpo usciranno naturalmente dal corpo.
- Tagliare un pezzo dell'intestino esposto tenendo premuto l'intestino con un ago e tagliandolo con l'altro.
NOTA: Se possibile, verificare che il microbo di interesse sia ancora presente nell'intestino estroflesso utilizzando la microscopia Normarski, la FISH o l'immunoistochimica23. - Trasferire un singolo intestino sezionato in un tubo PCR da 0,5 ml contenente 10 μL di acqua sterile. Ripetere per un totale di almeno 5 tubi PCR contenenti intestini di animali diversi.
- Congelare i tubi PCR a -80 °C per un minimo di 5 min. Rimuovere i tubi PCR dal congelatore e scongelare i campioni.
- Pipettare il liquido su e giù un paio di volte per separare i batteri dall'intestino.
- Condurre la PCR utilizzando coppie di primer universali contro la sequenza di DNA della piccola subunità ribosomiale di batteri, lieviti o microsporidi, a seconda del tipo sospetto di microbo.
NOTA: Ad esempio, un protocollo di esempio che utilizza primer batterici universali 16S è presentato nella Tabella 1. - Purificare l'amplicon utilizzando un kit di pulizia e concentrazione del DNA basato su filtro (vedere Tabella dei materiali) ed eseguire il sequenziamento Sanger.
Risultati
Un ceppo selvatico di C. tropicalis (JU1848) è stato isolato dai frutti marci delle palme nella foresta di Nouragues nella Guyana francese (Figura 1A)24. Questo ceppo è stato trovato per avere microbi sottili che colonizzano il lume dell'intestino in modo direzionale (Figura 1B). Questo microbo è stato facilmente trasferito al ceppo N2 di C. elegans tramite co-coltura con il ceppo JU1848, dove ha colonizzato il lume dell'intestino in modo simile.
La propagazione di JU1848 su piastre NGM standard seminate con E. coli OP50-1 su più generazioni ha continuamente provocato una contaminazione visibile, vista come varie colonie scure di mucoidi dentro e fuori il prato OP50-1 (Figura 2A). Un piatto di JU1848 selvatico è stato affamato per costringere gli animali a dauer e pulito come descritto. Gli animali dauer singoli sopravvissuti alla pulizia sono stati placcati su singole piastre NGM da 6 cm seminate con OP50-1 e lasciate crescere per 4 giorni a 20 °C. Sono state osservate più piastre di progenie F1 senza contaminazione microbica visibile (Figura 2B). La progenie F1 è stata verificata per contenere ancora batteri aderenti nel lume dell'intestino (vedi sotto).
Gli animali PULITI JU1848 sono stati lavati e decapitati per isolare i pezzi intestinali come descritto nel protocollo (passaggi 6.1-6.12). I batteri aderenti nel lume dell'intestino sezionato sono stati verificati tramite microscopia Nomarski (Figura 3). Il microbo nel lume di JU1848 è stato sospettato di essere un batterio, quindi l'intestino sezionato è stato utilizzato come modello per la PCR utilizzando primer batterici universali 16S, 27F e 1492R. Da un totale di sei singoli intestini sezionati, i prodotti PCR sono stati sequenziati tramite Sanger e cromatografi puliti hanno mostrato che tutte e sei le sequenze erano identiche. Sulla base di queste sequenze, questo batterio è stato identificato come una nuova specie nella classe Alphaproteobacteria ma non ha potuto essere classificato in un ordine o genere noto (File supplementare 1).

Figura 1: Batteri aderenti che colonizzano il lume di un C. tropicalis selvatico. (A) Immagine campione di campo di frutti di palma marci di Euterpe sp. (Famiglia: Arecaceae) nella foresta di Nouragues nella Guyana francese. (B) Immagine di Nomarski del ceppo JU1848 visto con migliaia di batteri lunghi e sottili che formano un aspetto simile a un pennello nel lume (lu) dell'intestino ospite (in). Gli strati batterici (ba) che rivestono l'intestino sono indicati con parentesi ([). Fare clic qui per visualizzare una versione più grande di questa figura.
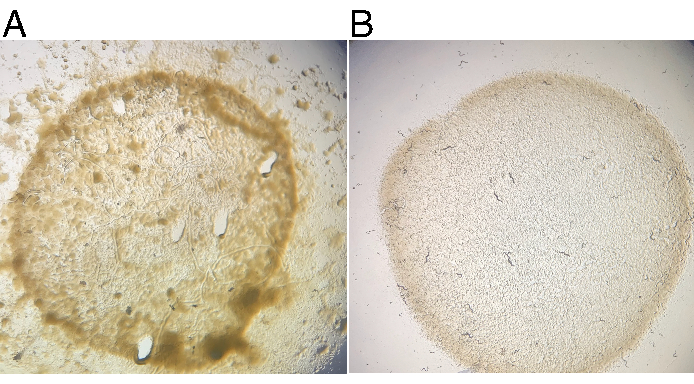
Figura 2: La contaminazione della crescita microbica viene persa dopo la pulizia dei nematodi. (A) Il ceppo selvatico JU1848 propaga una notevole crescita microbica su piastre NGM standard da 6 cm seminate con batteri E. coli OP50-1. (B) Dopo la pulizia, una piastra di progenie F1 di un singolo dauer non mostra alcuna contaminazione microbica visibile dopo 4 giorni di incubazione a 20 ° C. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: I batteri aderenti sono visti nel lume dell'intestino sezionato. Immagine nomarski di un animale JU1848 pulito che è stato decapitato in modo che l'intestino fuoriuscisca. I batteri colonizzanti (ba) sono indicati con una parentesi ([) e sono visti nel lume (lu) di un pezzo dell'intestino (in) che si trova al di fuori del corpo del nematode (nb). Fare clic qui per visualizzare una versione più grande di questa figura.
| Reagente | Concentrazione | Importo |
| Primer 27F (5'-AGAGTTTGATCMTGGCTCAG-3') | 20 mM | 2,5 μL |
| Primer 1492R (5'-GGTTACCTTGTTACGACTT-3') | 20 mM | 2,5 μL |
| Intestino sezionato in acqua | N/D | 3 μL |
| Buffer PCR 10x | 10x | 5 μL |
| dNTP | 10 mM | 1 μL |
| Taq Polimerasi | 5 U/μL | 0,5 μL |
| Acqua | N/D | 35,5 μL |
Tabella 1: Protocollo PCR campione che utilizza primer batterici universali e intestino sezionato.
File supplementare 1: Allineamento MUSCOLARE di sequenze batteriche di rDNA 16S derivate dalla PCR di sei intestini JU1848 sezionati. Clicca qui per scaricare questo File.
Discussione
Questo protocollo descrive l'isolamento e l'identificazione dei microbi dai nematodi caenorhabditis isolati in natura utilizzando una serie di procedure di pulizia. Numerosi microbi sono associati a nematodi isolati in natura e alcuni di essi hanno fenotipi eccitanti che possono essere utilizzati per studi futuri sulle interazioni ospite-microbo e sull'immunità innata. Molti microbiomi coltivabili e batteri patogeni sono stati isolati dai nematodi selvatici caenorhabditis utilizzando tecniche standard per la crescita batterica in vitro25,26. Tuttavia, non tutti i microbi possono essere coltivati in vitro e diventa necessario arricchirli in nematodi selvatici. Alcuni microbi hanno uno stadio di spore resistente, come i microsporidi, e alte concentrazioni di SDS possono essere utilizzate per uccidere la maggior parte dei batteri e dei funghi, consentendo un arricchimento specifico delle spore12. Questo protocollo presenta un metodo per arricchire i microbi intestinali incolti che non sono resistenti alla SDS e al trattamento antibiotico.
La tecnica qui presentata sfrutta la resistenza ambientale osservata negli animali dauer a causa di cambiamenti fisiologici come il rafforzamento della cuticola, la soppressione del pompaggio faringeo e la copertura della bocca con un tappo buccale27. Un passo critico in questo protocollo è l'incubazione notturna con vari antibiotici e 0,25% SDS. Questo passaggio viene utilizzato per uccidere tutti i microbi esterni lasciando intatti i microbi interni. Mentre C. elegans dauers ha dimostrato di sopravvivere a concentrazioni di SDS fino al 10% per 30 min27, questo protocollo utilizza un'incubazione moderata ma prolungata non solo per uccidere i microbi, ma esporre ulteriormente i batteri agli antibiotici. Inoltre, una moderata concentrazione di SDS può aiutare a garantire che i dauer di altre specie di Caenorhabditis sopravvivano, poiché l'esposizione di C. tropicalis all'1% di SDS durante la notte ha provocato la morte di tutti gli animali dauer. Se tutti i dauer muoiono, la concentrazione di SDS e/o la durata dell'esposizione alla SDS devono essere ridotte. Al contrario, se le piastre di generazione F1 hanno ancora una contaminazione visibile dopo la pulizia, la concentrazione di SDS e il tempo di incubazione dovrebbero essere aumentati.
Un altro passo critico è l'isolamento degli animali single dauer dopo la pulizia. Questo passaggio è fondamentale in quanto non tutti gli animali sono puliti dopo la SDS e il trattamento antibiotico. Pertanto, gli animali sono posti al centro di una piastra NGM di 10 cm con OP50-1 e lasciati strisciare radialmente verso l'esterno. Spesso è meglio scegliere più animali distali, poiché il strisciare esteso attraverso OP50-1 sembra aiutare a rimuovere eventuali microbi sopravvissuti attaccati alla cuticola. Tuttavia, questo porta a una limitazione del protocollo, in quanto sarà più difficile arricchire per un microbo di interesse se non è presente nella popolazione ad alta frequenza. Qui, gli alfaproteobatteri aderenti erano presenti nel 90%-95% della popolazione; pertanto, la maggior parte delle piastre pulite aveva il batterio del microbioma. Tuttavia, se un microbo di interesse è presente a una frequenza molto più bassa nella popolazione, potrebbe essere necessario schermare molte più piastre F1 .
Questo protocollo potrebbe probabilmente essere utilizzato per isolare qualsiasi numero di microbi non coltivabili di interesse trovati nei nematodi selvatici. Tuttavia, il microbo deve trovarsi in un tessuto protetto dalla cuticola dauer, in grado di sopravvivere negli animali dauer, e avere un fenotipo osservabile nell'ospite. Come tale, questa tecnica può essere utilizzata per arricchire altri batteri del microbioma nel lume intestinale oltre alle specie di alfaproteobatteri qui descritte, compresi i batteri che non aderiscono. Inoltre, il protocollo è stato utilizzato per arricchire un batterio intracellulare facoltativo, Bordetella atropi, che infetta il nematode Oscheius tipulae28. Dopo l'arricchimento, È stato scoperto che B. atropi forma colonie su piastre NGM, dimostrando che un microbo di interesse può essere scoperto essere coltivabile in vitro una volta rimossi i contaminanti a crescita più rapida. Questa tecnica probabilmente funzionerebbe per microsporidi e virus, incluso il virus Orsay, data questa capacità di arricchire un batterio intracellulare. Tuttavia, questi microbi devono essere in grado di sopravvivere alla transizione dentro e fuori da dauer.
È importante ricordare che mentre questo protocollo può essere eseguito in un laboratorio di livello di biosicurezza 1, è necessario mantenere una tecnica sterile per tutto il tempo per prevenire un'ulteriore contaminazione microbica. Il protocollo può essere modificato in base alle esigenze del ricercatore, compresi i tipi / concentrazioni di antibiotici, la percentuale di SDS e / o l'aggiunta di antifungini come la nistatina. Spesso, il numero di microbi contaminanti trovati in un nematode isolato in natura può variare notevolmente. Qui, l'apparente perdita di crescita di E. coli non-OP50-1 su piastre NGM è stata utilizzata come lettura per un ceppo di nematodi pulito. Ma potrebbero essere presenti popolazioni non coltivabili di microbi contaminanti, quindi è essenziale condurre un metodo di metagenomica come il sequenziamento dell'amplicone dell'rRNA 16S per vedere l'entità della contaminazione26. Una volta che il ceppo di verme viene pulito, può essere congelato e conservato per studi futuri. Nel complesso, questo protocollo consente ai ricercatori di arricchire microbi incolturabili nei nematodi selvatici, consentendo loro di studiare gli effetti sulla forma fisica dell'ospite, caratterizzare i fenotipi di colonizzazione o infezione e sfruttare gli strumenti genetici per comprendere i meccanismi alla base delle interazioni ospite-microbo.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Grazie al Dr. Christian Braendle e al Centre Nationale de la Recherche Scientifique (CNRS) Nouragues Field Station.
Materiali
| Name | Company | Catalog Number | Comments |
| Agarose | Fisher Scientific | BP1356 | |
| 10% SDS | Invitrogen | AM9822 | |
| BD PrecisionGlide Needle - 26 G | Fisher Scientific | 305115 | |
| Carbenicillin | Millipore-Sigma | C1389-1G | |
| Cefotaxime | Millipore-Sigma | C7039-500mg | |
| Chloramphenicol | Millipore-Sigma | C0378-25G | |
| DNA Clean and Concentrator Kit | Zymo Research | 11-303C | |
| DreamTaq Polymerase | Fisher Scientific | EP0711 | |
| Gentamycin | Millipore-Sigma | G1264-250mg | |
| Kanamycin | Millipore-Sigma | K1876-1G | |
| KH2PO4 | Fisher Scientific | P-286 | |
| NaCl | Fisher Scientific | S-671 | |
| NH4Cl | Fisher Scientific | A-661 | |
| Streptomycin | Millipore-Sigma | S6501-50G | |
| Tetracyclin | Millipore-Sigma | T7660-5G | |
| Triton X-100 | Fisher Scientific | BP-151 | |
| Watch glasses | VWR | 470144-850 |
Riferimenti
- Balla, K. M., Troemel, E. R. Caenorhabditis elegans as a model for intracellular pathogen infection. Cellular Microbiology. 15, 1313-1322 (2013).
- Pukkila-Worley, R., Ausubel, F. M. Immune defense mechanisms in the Caenorhabditis elegans intestinal epithelium. Current Opinion in Immunology. 24, 3-9 (2012).
- Bossinger, O., Fukushige, T., Claeys, M., Borgonie, G., McGhee, J. D. The apical disposition of the Caenorhabditis elegans intestinal terminal web is maintained by LET-413. Developmental Biology. 268, 448-456 (2004).
- Dimov, I., Maduro, M. F. The C. elegans intestine: organogenesis, digestion, and physiology. Cell and Tissue Research. 377, 383-396 (2019).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Szumowski, S. C., Botts, M. R., Popovich, J. J., Smelkinson, M. G., Troemel, E. R. The small GTPase RAB-11 directs polarized exocytosis of the intracellular pathogen N. parisii for fecal-oral transmission from C. elegans. Proceedings of the National Academy of Sciences of the United States of America. 111, 8215-8220 (2014).
- Bakowski, M. A., et al. Ubiquitin-mediated response to microsporidia and virus infection in C. elegans. PLoS Pathogens. 10, 1004200 (2014).
- Sowa, J. N., et al. The Caenorhabditis elegans RIG-I Homolog DRH-1 Mediates the Intracellular Pathogen Response upon Viral Infection. Journal of Virology. 94, 01173 (2020).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15, 833-838 (2014).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10, 59 (2012).
- Lee, D., et al. Balancing selection maintains hyper-divergent haplotypes in Caenorhabditis elegans. Nature Ecology and Evolution. 5, 794-807 (2021).
- Luallen, R. J., et al. Discovery of a natural microsporidian pathogen with a broad tissue tropism in Caenorhabditis elegans. PLOS Pathogens. 12, 1005724 (2016).
- Osman, G. A., et al. Natural infection of C. elegans by an oomycete reveals a new pathogen-specific immune response. Current Biology. 28, 640-648 (2018).
- Felix, M. A., et al. Natural and experimental infection of Caenorhabditis nematodes by novel viruses related to nodaviruses. PLoS Biology. 9, 1000586 (2011).
- Zhang, G., et al. A large collection of novel nematode-infecting microsporidia and their diverse interactions with Caenorhabditis elegans and other related nematodes. PLoS Pathogens. 12, 1006093 (2016).
- Félix, M. -. A., Wang, D. Natural viruses of Caenorhabditis nematodes. Annual Review Genetics. 53, 313-326 (2019).
- Grover, M., Barkoulas, M. C. elegans as a new tractable host to study infections by animal pathogenic oomycetes. PLoS Pathogens. 17, 1009316 (2021).
- Bakowski, M. A., Luallen, R. J., Troemel, E. R. Microsporidia Infections in Caenorhabditis Elegans and Other Nematodes. Microsporidia: Pathogens of Opportunity: First Edition. , (2014).
- Taffoni, C., Pujol, N. Mechanisms of innate immunity in C. elegans epidermis. Tissue Barriers. 3, 1078432 (2015).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10, 3025-3039 (2020).
- Frézal, L., Félix, M. -. A. C. elegans outside the Petri dish. eLife. 4, 05849 (2015).
- Troemel, E. R., Félix, M. -. A., Whiteman, N. K., Barrière, A., Ausubel, F. M. Microsporidia are natural intracellular parasites of the nematode Caenorhabditis elegans. PLoS Biology. 6, 2736-2752 (2008).
- Luallen, R. J., Bakowski, M. A., Troemel, E. R. Characterization of microsporidia-induced developmental arrest and a transmembrane leucine-rich repeat protein in Caenorhabditis elegans. PLoS One. 10, 0124065 (2015).
- Félix, M. -. A., et al. Species richness, distribution and genetic diversity of Caenorhabditis nematodes in a remote tropical rainforest. BMC Evolutionary Biology. 13, 10 (2013).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113, 3941-3949 (2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10, 1998-2009 (2016).
- Androwski, R. J., Flatt, K. M., Schroeder, N. E. Phenotypic plasticity and remodeling in the stress-induced C. elegans dauer. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 278 (2017).
- Tran, T. D., Ali, M. A., Lee, D., Félix, M. -. A., Luallen, R. J. Bacterial filamentation is an in vivo mechanism for cell-to-cell spreading. bioRxiv. , (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon