Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un ensayo celular basado en reporteros para monitorear la eficiencia del empalme
En este artículo
Resumen
Este protocolo describe un ensayo de reportero de minigenes para monitorear el impacto de las mutaciones del sitio de empalme 5'en el empalme y desarrolla snRNA supresor U1 para el rescate de la inhibición del empalme inducida por la mutación. Las construcciones de snRNA U1 reportero y supresor se expresan en células HeLa, y el empalme se analiza por extensión de cebador o RT-PCR.
Resumen
Durante la expresión génica, el paso vital del empalme pre-ARNm implica el reconocimiento preciso de los sitios de empalme y el ensamblaje eficiente de los complejos espliceosómicos para unir exones y eliminar los intrones antes de la exportación citoplasmática del ARNm maduro. La eficiencia del empalme puede verse alterada por la presencia de mutaciones en los sitios de empalme, la influencia de los factores de empalme de acción trans o la actividad de la terapéutica. Aquí, describimos el protocolo para un ensayo celular que se puede aplicar para monitorear la eficiencia de empalme de cualquier exón dado. El ensayo utiliza un plásmido adaptable codificado 3-exon/2-intron minigene reporter, que puede expresarse en células de mamíferos por transfección transitoria. Después de la transfección, se aísla el ARN celular total, y la eficiencia del empalme de exones en el ARNm reportero se determina mediante la extensión del cebador o la reacción en cadena semicuantitativa de la polimerasa con transcriptasa inversa (RT-PCR). Describimos cómo se puede determinar el impacto de las mutaciones del sitio de empalme 5′ asociadas a la enfermedad introduciéndolas en el reportero; y cómo la supresión de estas mutaciones se puede lograr mediante la coinfección con el ARN nuclear pequeño (snRNA) U1 que lleva mutaciones compensatorias en su región de 5′ que se fusiona con los sitios de empalme 5′ en las uniones exón-intrón en pre-ARNm. Por lo tanto, el reportero se puede utilizar para el diseño de partículas terapéuticas de U1 para mejorar el reconocimiento de los sitios de empalme mutantes de 5 '. La inserción de sitios reguladores de acción cis, como secuencias de potenciador de empalme o silenciador, en el informador también se puede utilizar para examinar el papel de U1 snRNP en la regulación mediada por un factor de empalme alternativo específico. Finalmente, las células que expresan al reportero se pueden incubar con moléculas pequeñas para determinar el efecto de la terapéutica potencial en el empalme constitutivo de pre-ARNm o en exones que transportan sitios de empalme mutantes de 5 '. En general, el ensayo de reportero se puede aplicar para monitorear la eficiencia del empalme en una variedad de condiciones para estudiar los mecanismos fundamentales de empalme y las enfermedades asociadas al empalme.
Introducción
El empalme pre-ARNm es un paso de procesamiento esencial que elimina los intrones no codificantes y liga con precisión los exones codificantes para formar ARNm maduro. El reconocimiento de secuencias de consenso en las uniones exón-intrón, denominadas sitio de empalme de 5′ y sitio de empalme de 3′, por componentes de la maquinaria de empalme inicia el proceso de empalme. La ribonucleoproteína nuclear pequeña U1 (snRNP) reconoce el sitio de empalme 5′ por emparejamiento de bases del snRNA U1 con el pre-ARNm1. Las mutaciones heredadas genéticamente que alteran las secuencias del sitio de empalme 5′ se asocian con muchas enfermedades2,3. Se predice que la pérdida de emparejamiento base de SnRNA U1 con los sitios de empalme mutantes de 5'causa un empalme aberrante, que puede comprometer la traducción de la transcripción afectada. Un enfoque terapéutico potencial para corregir los defectos de empalme implica la supresión de mutaciones por snRNA U1 modificado que transporta cambios de nucleótidos compensatorios en su región de 5′que se basa con el sitio de empalme de 5'. Se ha encontrado que estos snRNAs U1 modificados, también conocidos como snRNAs U1 específicos del exón, son efectivos para revertir los defectos de empalme, lo que resulta en una mayor expresión de proteínas del ARNm rescatado4,5,6,7,8.
Aquí, describimos el ensayo de complementación U1 snRNP, un ensayo de empalme celular basado en reporteros que permite la evaluación del efecto de las mutaciones de 5′-ss en el empalme de un exón y también se puede usar para el desarrollo de snRNAs U1 modificados para permitir el rescate de la inclusión de exones. También proporcionamos protocolos para el monitoreo de las transcripciones de reportero empalmadas por extensión de imprimación y RT-PCR, y para determinar la expresión de snRNAs U1 modificados por extensión de imprimación y RT-qPCR.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Reactivos y tampones
NOTA: Toda la esterilización con filtros de vacío debe realizarse con una membrana de polietersulfona (PES) de 0,2 μm en un gabinete de bioseguridad.
- Prepare agua sin RNasa agregando 1.0 ml de dietilpirocarbonato (DEPC) a 1.0 L de agua desionizada, mezcle durante al menos 1 hora a temperatura ambiente (RT), autoclave dos veces y luego enfríe a RT antes de usar.
- Prepare el Medio Águila Modificada (DMEM) de Dulbecco mezclando un paquete de polvo de DMEM (13.4 g), 3.7 g de bicarbonato de sodio, 100 ml de suero fetal bovino (FBS), penicilina y estreptomicina a ~ 800 ml de agua desionizada estéril. La concentración final de penicilina y estreptomicina en DMEM debe ser de 50 U/mL y 50 μg/mL, respectivamente. Ajuste el pH a 7.4 y luego suba el volumen a 1.0 L con agua desionizada estéril. Esterilizar por filtración y conservar a 4 °C.
- Preparar 10x solución salina tamponada con fosfato (10x PBS) añadiendo 25,6 g de hidrógeno disódico heptahidratado (Na2HPO4· 7H2O), 2 g de fosfato de dihidrógeno de potasio (KH2PO4), 2 g de cloruro de potasio (KCl) y 80 g de cloruro de sodio (NaCl) a 800 ml de agua desionizada. Mezclar para disolver y enrasar el volumen hasta 1.0 L. Esterilizar por filtración y almacenar a 4 °C.
- Prepare 0.5 M de ácido tetraacético de etileno diamina (EDTA) disolviendo 186.1 g de Na2•EDTA•2H2O en ~ 800 mL de agua desionizada. Ajuste el pH a 8.0 y luego suba el volumen a 1.0 L con agua desionizada estéril. Esterilizar por filtración y conservar a 4 °C.
- Prepare 1 solución de tripsina-EDTA mezclando 100 ml de 10 veces tripsina (2,5%), 2 ml de 0,5 m de EDTA y agregue 1 ml de PBS hasta 1,0 L. Esterilizar por filtración y alícuota en tubos cónicos de 50 ml. Conservar a 4 °C durante 1-2 semanas o congelar a -20 °C para uso a largo plazo.
- Preparar 2x colorante de carga de ADN/ARN formamida mezclando 14,4 mL de formamida y 0,6 mL de 0,5 M EDTA para un volumen final de 15 mL. Añadir bromofenol azul y xileno en polvo de cianol a una concentración final de 0,02% y conservar a 4 °C.
NOTA: La formamida es tóxica y corrosiva. Lea las hojas de datos de seguridad de materiales para obtener recomendaciones de seguridad adicionales. - Prepare 5x Tampón Tris/Borato/EDTA (5x TBE) mezclando 54.0 g de base tris, 27.5 g de ácido bórico y 20 mL de 0.5 M EDTA en ~800 mL de agua desionizada. Mezclar para disolver y hacer el volumen a 1.0 L con agua desionizada.
- Prepare la solución de electroforesis en gel de urea-poliacrilamida (urea-PAGE) mezclando 200 ml de 5x TBE, 250 mL de 40% 19: 1 bis / acrilamida y 450.5 g de urea. Luego agregue agua desionizada hasta 1.0 L. Mezcle hasta que los ingredientes estén completamente disueltos, luego esterilice por filtración y almacene a 4 ° C en una botella de vidrio ámbar.
NOTA: El bis/acrilamida es tóxico. Lea las hojas de datos de seguridad de materiales para conocer los procedimientos de seguridad adicionales. - Preparar persulfato de amonio al 10% (APS) disolviendo 1 g de APS en 10 mL de agua desionizada y almacenar a 4 °C.
2. Cotransfección de células HeLa con el reportero y plásmidos SnRNA U1
NOTA: La transfección de células Hela debe realizarse en condiciones estériles en un gabinete de seguridad biológica. La superficie exterior de todos los materiales debe rociarse con etanol al 70% antes de introducirse en el gabinete de seguridad biológica.
- Mantenga las células Hela en DMEM en una incubadora de 37 °C con un 5% de CO2 pasando cada 2-3 días cuando las células son aproximadamente 80-90% confluentes.
- Para las células HeLa de paso, aspire el medio gastado y luego incube las células con 3 ml de tripsina al 0,25% que contenga 1 mM de EDTA a 37 °C durante 3 min.
- Después de la incubación, agregue 7 ml de DMEM fresco. Transfiera la suspensión celular a un tubo de 10 ml, centrífuga a 1.000 x g durante 5 min.
- Resuspenda el pellet celular en 10 ml de DMEM fresco, y luego coloque las células en una nueva placa de cultivo de tejidos al 20% de confluencia.
- Para transfecciones transitorias, cuente las células Hela con un portaobjetos limpio del hemocitómetro y prepare una suspensión con una densidad de 2,5 x 105 células/ml.
- Sembrar 1,0 ml de 2,5 x 105 células/ml de suspensión de células Hela en cada pocillo de una placa de 12 pocillos e incubar durante la noche a 37 °C.
- Al día siguiente, aspire el medio gastado y agregue 0.8 ml de DMEM fresco con suero.
- Prepare la Solución I agregando 0,2 μg de plásmido reportero Dup51 o Dup51p, 1,8 μg de pcDNA, pNS6U1 o plásmido pNS6U1-5a, y 100 μL de medio de transfección en un nuevo tubo de microcentrífuga de 1,5 ml.
- Preparar una mezcla maestra de la Solución II para todas las muestras a transfectar añadiendo 100 μL de medio de transfección y 4,0 μL de reactivo de transfección por muestra.
- Prepare la mezcla de transfección añadiendo 100 μL de Solución II en cada tubo de microcentrífuga que contenga la Solución I.
- Vórtice las mezclas de transfección durante 15 s, centrífuga en una microcentrífuga de mesa a 3.000 x g durante 10 s en RT, y luego incube en RT durante 5 min.
- Agregue los 200 μL de la mezcla de transfección en un pozo de la placa celular HeLa de 12 pocillos para lograr un volumen final de 1.0 mL por pozo e incube la placa a 37 ° C durante 48 horas.
- Después de la incubación, extraiga el ARN de las células HeLa transfectadas con tiocianato de guanidina y solución de fenol disponibles comercialmente.
NOTA: Este reactivo contiene fenol y este paso debe realizarse en una campana extractora de humos. Se recomienda el uso de agua tratada con DEPC para la resuspensión del ARN extraído.- Aspire el medio gastado y agregue 500 μL del reactivo en cada pozo. Incubar en RT durante 5 min.
- Homogeneizar mediante pipeteos hacia arriba y hacia abajo. Luego transfiera la solución a un nuevo tubo de microcentrífuga de 1,5 ml.
- Añadir 100 μL de cloroformo y vórtice durante 15 s.
- Centrifugadora a 12.000 x g durante 15 min en RT.
- Transfiera 200 μL de la capa acuosa superior que contiene arnés a un nuevo tubo de microcentrífuga de 1,5 ml.
- Añadir 2 μg de glucógeno y 200 μL de isopropanol a cada muestra de ARN. Mezclar invirtiendo los tubos.
- Recoger el precipitado de ARN por centrifugación a 12.000 x g durante 10 min a 4 °C.
- Retire y deseche el sobrenadante sin perturbar el gránulo de ARN.
- Lave el pellet dos veces agregando 1.0 ml de etanol al 70%, invirtiendo los tubos y centrifugando como se describe en el Paso 2.13.7.
- Seque al aire el pellet durante ~ 10-20 min en RT y resuspenda el ARN en 10-20 μL de agua libre de RNasa.
- Determine la concentración de ARN midiendo la absorbancia a 260 nm como lo describen Desjardins y Conklin9.
- Proceda con la extensión del cebador o almacene muestras de ARN a -20 °C. El ARN aislado se puede almacenar a -20 °C durante 6-12 meses.
3. Etiquetado 32P de oligonucleótidos
NOTA: Los pasos que impliquen el uso de oligonucleótidos marcados con 32P-ATP y 32P deben realizarse detrás de un escudo acrílico por personas capacitadas con la aprobación de entidades institucionales autorizadas. El protocolo descrito a continuación se puede utilizar para el etiquetado de oligonucleótidos, Dup3r y U17-26-R, y marcadores para urea-PAGE. Se recomienda el uso de agua tratada con DEPC para la resuspensión de oligonucleótidos y perlas de exclusión de tamaño.
- A un tubo de microcentrífuga de 1,5 ml, agregue oligonucleótido, polinucleótido quinasa T4 (T4 PNK), tampón T4 PNK y 32P-ATP como se describe en la Tabla 1. Agregue 32P-ATP por última vez a la mezcla; esto es importante.
NOTA: Para la adición de soluciones radiactivas, se recomienda el uso de puntas de filtro. - Incubar en un baño de agua a 37 °C durante 30 min.
- Mientras se incuban las reacciones de etiquetado, resuspender las perlas de exclusión de tamaño con un peso molecular de 25 kDa cortado mediante un vórtice suave durante ~ 10 s.
NOTA: Las perlas de exclusión de tamaño deben prepararse de acuerdo con las instrucciones del fabricante y almacenarse como una suspensión al 50% en etanol al 25% a 4 °C. - Prepare las columnas transfiriendo 600 μL de las perlas resuspendidas a una mini columna desechable colocada en un tubo de recolección de 1,5 ml y devolviendo el stock de perlas a 4 °C.
- Centrifugar a 2.000 x g durante 1 min en RT y desechar el flujo.
- Lave las perlas agregando 300 μL de agua libre de RNasa a la columna.
- Repita los pasos 3.5. y 3.6. dos veces y transfiera la mini-columna a un nuevo tubo centrífuga de 1,5 ml.
- Agregue la mezcla de reacción de quinasa a la columna de perlas de exclusión de tamaño y a la centrífuga a 5000 x g durante 1 min en RT.
- Recoja y guarde el flujo, que tiene el oligonucleótido marcado con 32P, y deseche todas las puntas y tubos en una caja de desechos de acrílico.
- Añadir 20 μL de agua libre de RNasa para diluir el oligonucleótido marcado con 32P a una concentración final de 2,5 μM.
NOTA: Diluya los marcadores etiquetados según sea necesario para la carga en geles de urea-PAGE. - Guarde el oligonucleótido etiquetado en una caja de microtubos acrílicos a -20 °C o proceda con el análisis de extensión de imprimación.
4. Análisis de las transcripciones del reportero empalmado por extensión de imprimación
NOTA: Se recomienda limpiar las superficies y el equipo con un reactivo inactivador RNasa antes de su uso.
- Agregue 2.0 μg de ARN extraído de las células Hela en tubos de microcentrífuga separados de 200 μL y agregue agua libre de RNasa para completar el volumen a 6.55 μL.
- Prepare Master Mix I con el 32P-Dup3r y dNTP diluidos como se muestra en la Tabla 2 y agregue 0.9 μL de la mezcla a cada tubo de PCR que contenga muestras de ARN.
- Incubar los tubos, primero a 65 °C durante 5 min y luego en hielo durante 1 min.
- Prepare Master Mix II con 5x First Strand Buffer, ditiotreitol (TDT), inhibidor de la RNasa y transcriptasa inversa como se muestra en la Tabla 2.
- Añadir 2,55 μL de la mezcla a cada tubo que contenga ARN y Master Mix 1; el volumen total de la reacción debe ser de 10 μL. Mantenga los tubos en RT durante 10 min.
- Transfiera los tubos a un baño seco o a un termociclador e incube, primero a 50 °C durante 60 min y luego a 70 °C durante 15 min.
- Después de la incubación, agregue 10 μL de colorante de carga de ARN de formamida 2x a cada muestra y guárdelo en una caja acrílica a -20 ° C o proceda con la separación de fragmentos por urea-PAGE utilizando un gel de 14 cm de largo y la visualización de la imagen del gel como se describe a continuación en el Paso 8.
- Realice un escaneo densitométrico de la imagen del gel con un software de análisis de imágenes y use las intensidades de banda de los productos incluidos y omitidos para calcular el porcentaje de inclusión del exón 2 como se muestra a continuación.

5. Análisis de las transcripciones del reportero empalmado mediante RT-PCR fluorescente
NOTA: El análisis rt-PCR que se describe a continuación utiliza hexámeros aleatorios para la síntesis de ADNc y una combinación de oligonucleótidos Dup8f y Dup3r marcados con Cy5 para la amplificación por PCR de los productos empalmados.
- Para la síntesis de ADNc, agregue 2.0 μg de ARN extraído de las células Hela transfectadas en tubos de microcentrífuga separados de 200 μL y agregue agua libre de RNasa para completar el volumen a 11.0 μL.
- Prepare Master Mix I, que contenga hexámeros aleatorios y dNTP como se muestra en la Tabla 3 y agregue 2.0 μL de la mezcla a cada muestra. Incubar, primero a 65 °C durante 5 min y luego en hielo durante 1 min.
- Prepare Master Mix II, que contiene First Strand Buffer, inhibidor de RNasa, TDT y transcriptasa inversa como se muestra en la Tabla 3 y agregue 7.0 μL de la mezcla a cada tubo que contenga ARN y Master Mix I.
- Mantenga los tubos en RT durante 10 min, y luego incube a 50 °C durante 60 min y 70 °C durante 15 min.
NOTA: Las reacciones completadas de ADNc pueden almacenarse a -20 °C. - Para la PCR, transfiera 1,0 μL (100 ng/μL) de cada muestra de ADNc a nuevos tubos de PCR.
- Prepare una mezcla maestra que consista en Dup8f, Cy5-Dup3r, dNTPs, tampón Taq, Taq polimerasa y agua como se describe en la Tabla 4 y agregue 11.5 μL de la mezcla a cada tubo que contenga ADNc.
- Realizar PCR utilizando un termociclador con un paso de desnaturalización inicial a 94 °C durante 3 min; seguido de 20 ciclos de desnaturalización (94 °C durante 30 s), recocido (65 °C durante 30 s) y extensión (72 °C durante 15 s), y un paso de terminación a 72 °C durante 5 min.
- Añadir 12,5 μL de colorante de carga de ADN de formamida 2x a cada tubo y calentar a 95 °C durante 5 min.
- Guarde la reacción de PCR a -20 °C o proceda con urea-PAGE como se describe a continuación en el paso 8.1-8.4.
- Después de la electroforesis, retire las placas de vidrio del aparato de electroforesis y escanee con un generador de imágenes de fluorescencia para visualizar el gel.
- Realice un escaneo densitométrico de la imagen del gel y use la intensidad de la banda de los productos incluidos y omitidos para calcular el porcentaje de inclusión del exón 2 como se describe en el Paso 4.8.
6. Análisis de la expresión de snRNA variante U1 por extensión de cebador
- Agregue 2.0 μg de ARN extraído de las células Hela en tubos de microcentrífuga separados de 200 μL y agregue agua libre de RNasa para compensar el volumen a 4.325 μL y luego agregue dATP, como se muestra en la Tabla 5.
- Agregue 10,000 CPM del oligonucleótido 32P-U17-26-R a cada tubo.
NOTA: Para preparar una solución de 10.000 cpm/μL de 32P-U17-26-R (a partir del Paso 3), diluya el oligonucleótido marcado (dilución 1:20), determine cpm en 1,0 μL utilizando un contador de centelleo y diluya aún más con agua desionizada para preparar una solución de 10.000 cpm/μL en un nuevo tubo de microcentrífuga de 1,5 ml. - Incubar a 65 °C durante 5 min, y luego en hielo durante 1 min.
- Prepare una mezcla maestra con 5x First Strand Buffer, inhibidor de la RNasa, TDT y transcriptasa inversa como se muestra en la Tabla 5 y agregue 1.8 μL de la mezcla a cada muestra.
- Incubar, primero a RT durante 10 min y luego a 42 °C durante 10 min.
- Después de la incubación, agregue 10 μL de 2x colorante de carga de ARN formamida en cada muestra y guárdelo en una caja acrílica a -20 °C o proceda a la separación de fragmentos por urea-PAGE utilizando un gel de 38 cm de largo (consulte el Paso 8).
7. Análisis de la expresión de snRNA variante U1 por RT-qPCR
- Diluir el stock de ADNc preparado como se describe anteriormente en los pasos 5.1 a 5.4 a una concentración de 0,2 ng/μL.
- Pipetear 5,0 μL de ADNc diluido en pocillos individuales de una placa qPCR de 96 pocillos por triplicado. Agregue agua desionizada en lugar de ADNc para el control sin plantilla (NTC).
- Prepare dos mezclas de imprimación separadas que consistan en las imprimaciones hacia adelante y hacia atrás para los ARNSR U1 y U2, y agua como se muestra en la Tabla 6.
NOTA: Las secuencias para cebadores específicos de snRNA U1 y U2 se proporcionan en la Tabla 7. - Agregue 5.0 μL de la mezcla de imprimación de ARNs SnRNA U1 y U2 a los pocillos de muestra y NTC.
- Agregue 10.0 μL de mezcla de PCR en tiempo real a cada pozo.
- Selle las placas con una película óptica, luego centrífuga a 1.000 x g durante 2 minutos en RT para recoger las reacciones al fondo de los pozos.
- Realice qPCR con un paso de desnaturalización inicial a 95 °C durante 10 min, seguido de 40 ciclos de un protocolo de 2 pasos que consiste en desnaturalización (95 °C durante 15 s) y recocido/extensión (62 °C durante 60 s) mientras se recopilan los valores del ciclo de cuantificación de umbral (Cq) de los amplicones objetivo.
- Finalice la reacción qPCR comprobando un solo pico en la curva de disociación para las reacciones de snRNA U1 y U2.
- A partir de los valores de Cq, calcule delta Cq (ΔCq) para los snRNAs U1 y U2 en comparación con el control pcDNA.
- Determine la expresión de snRNA variante U1 como cantidad relativa (RQ) de U1 en comparación con U2 utilizando el valor de ΔΔCq como se muestra a continuación para todas las muestras.

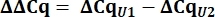
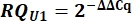
8. Configuración y funcionamiento de los geles Urea-PAGE
NOTA: El montaje de placas de vidrio y el aparato de funcionamiento en gel deben realizarse de acuerdo con las instrucciones del fabricante. La fundición del gel 10% urea-PAGE se puede realizar según un protocolo previamente descrito por Summer et al.10. A continuación se describen los pasos que implican la preparación de marcadores y muestras, y el funcionamiento y la visualización de geles. Opcionalmente, para evitar que el gel se pegue a las placas de vidrio, la superficie interna se puede recubrir con una solución de silicona agregando 1 ml de la solución a la superficie y extendiéndose uniformemente sobre toda la superficie con tejido. Una vez secas, las placas deben lavarse con agua desionizada y secarse de nuevo.
PRECAUCIÓN: La acrilamida no polimerizada es neurotóxica y debe manipularse con las protecciones recomendadas en la hoja de datos de seguridad del material.
- Prepare las muestras y marcadores de extensión de imprimación calentando a 95 °C durante 5 min y luego centrifugando en una microcentrífuga de mesa a 3.000 x g durante 5 s a RT.
- Antes de cargar los marcadores y las muestras, enjuague los pozos con 1x tampón TBE para eliminar la urea asentada.
- Cargue 10 μL / muestra / pozo y ejecute el gel a 300-500V durante 2-3 horas o hasta que el cianol de xileno llegue al fondo.
NOTA: Se pueden cargar alrededor de 1,000 cpm del marcador etiquetado con 32P. - Después de la electroforesis, retire las placas de vidrio del aparato de electroforesis.
- Separe cuidadosamente las dos placas para que el gel quede plano sobre cualquiera de las superficies de vidrio y transfiera el gel a un papel de filtro y cúbralo con una envoltura de plástico.
- Seque al vacío el gel en el papel de filtro a 80 °C durante 30 min con un secador de gel.
- Coloque el gel seco en un cassette de imágenes de fósforo y manténgalo en RT durante la noche.
NOTA: La pantalla de fósforo debe borrarse con una caja de luz antes de su uso. - Para visualizar la imagen del gel, retire la pantalla y escanee con un generador de imágenes de fósforo.
Access restricted. Please log in or start a trial to view this content.
Resultados
El reportero de empalme Dup51, un minigén de tres exones-dos intrón, se derivó del gen humano β-globina y ha sido descrito previamente (Figura 1A)11,12 . Creamos un reportero mutante, Dup51p, introduciendo mutaciones en el sitio de empalme 5' asociadas al síndrome de Usher que ocurren en el exón 3 del gen de la protocadherina 15 (PCDH15)13. La secuencia del sitio de empalme 5' en la unión exón...
Access restricted. Please log in or start a trial to view this content.
Discusión
El ensayo se puede adaptar para el análisis de empalme en líneas celulares distintas de HeLa, sin embargo, es posible que sea necesario optimizar los factores que afectan la eficiencia de la transfección, como la confluencia celular y la cantidad de ADN. La relación de constructo reportero-U1 es otro parámetro crítico que puede necesitar ser determinado dependiendo de los niveles de expresión observados en otros tipos de células. La calidad del ARN extraído es crítica para el análisis de empalme; por lo tanto,...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por fondos a S.S. de los Institutos Nacionales de Salud (R21CA170786 y R01GM127464) y la Sociedad Americana del Cáncer (la Subvención de Investigación Institucional 74-001-34-IRG) y a S.S. y W.M. del Programa de Asociación de Investigación del Valle (P1-4009 y VRP77). El contenido es responsabilidad exclusiva de los autores y no representa necesariamente las opiniones oficiales de los Institutos Nacionales de Salud.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Reagent Grade Deionized Water | ThermoFisher Scientific | 23-751628 | |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758-25ML | |
| Dulbecco's Modified Eagle Medium (DMEM) powder packet | Gibco | 12100-046 | |
| Sodium Bicarbonate | ThermoFisher Scientific | S233-500 | |
| Fetal Bovine Serum (FBS), Australian Source, Heat Inactivated | Omega Scientific | FB-22 | |
| Penicillin-Streptomycin (P/S) | Sigma-Aldrich | P4458-100ML | |
| Sodium Hydroxide, Standard Solution 1.0N | Sigma-Aldrich | S2567-16A | |
| Hydrochloric Acid, Certified ACS Plus, 36.5 to 38.0% | ThermoFisher Scientific | A144-500 | |
| Disposable PES Bottle Top Filters | ThermoFisher Scientific | FB12566510 | |
| EDTA Disodium Salt Dihydrate | Amresco | 0105-2.5KG | |
| 2.5% Trypsin (10x), no phenol red | ThermoFisher Scientific | 15090046 | |
| Sodium Chloride | Fisher Bioreagent | BP358-212 | |
| Potassium Chloride | Fisher Bioreagent | BP366-1 | |
| Disodium Hydrogen Phosphate Heptahydrate | Fisher Bioreagent | BP332-1 | |
| Potassium Dihydrogen Phosphate | Fisher Bioreagent | BP362-1 | |
| Transfection medium - Opti-MEM™ I Reduced Serum Medium, no phenol red | ThermoFisher Scientific | 11058021 | |
| Transfection Reagent - Lipofectamine™ 2000 | ThermoFisher Scientific | 13778150 | |
| TRIzol™ Reagent | ThermoFisher Scientific | 15596018 | |
| Chloroform (Approx. 0.75% Ethanol as Preservative/Molecular Biology) | ThermoFisher Scientific | BP1145-1 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2818-4 | |
| Isopropanol, Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2618-212 | |
| Glycogen (5 mg/ml) | ThermoFisher Scientific | AM9510 | |
| Direct-zol RNA Miniprep Kit | Zymo Research | R2052 | |
| ATP, [γ-32P]- 6000Ci/mmol 150mCi/ml Lead, 1 mCi | PerkinElmer | NEG035C001MC | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| Size exclusion beands - Sephadex® G-25 | Sigma-Aldrich | G2580-10G | |
| Size exclusion mini columns | USA Scientific | 1415-0600 | |
| pBR322 DNA-MspI Digest | New England Biolabs | N3032S | |
| Low Molecular Weight Marker, 10-100 nt | Affymetrix | 76410 100 UL | |
| Rnase inactivating reagents - RNaseZAP™ | Sigma-Aldrich | R2020-250ML | |
| dNTP Mix (10 mM ea) | ThermoFisher Scientific | 18427013 | |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | ThermoFisher Scientific | 10777019 | |
| Reverse Transcriptase - M-MLV Reverse Transcriptase | ThermoFisher Scientific | 28025013 | used for primer extension |
| Taq DNA Polymerase | ThermoFisher Scientific | 10342020 | |
| Random Hexamers (50 µM) | ThermoFisher Scientific | N8080127 | |
| Real time PCR mix - SYBR™ Select Master Mix | ThermoFisher Scientific | 4472903 | |
| SuperScript™ III Reverse Transcriptase | ThermoFisher Scientific | 18080093 | used for cDNA preparation |
| Dithiothreitol (DTT) | ThermoFisher Scientific | 18080093 | |
| 5X First-Strand Buffer | ThermoFisher Scientific | 18080093 | |
| Formamide (≥99.5%) | ThermoFisher Scientific | BP228-100 | Review Material Safety Data Sheets |
| Bromophenol Blue sodium salt | Sigma-Aldrich | 114405-5G | |
| Xylene Cyanol FF | Sigma-Aldrich | 2650-17-1 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | ThermoFisher Scientific | BP152-5 | |
| Boric Acid (Crystalline/Electrophoresis) | ThermoFisher Scientific | BP168-500 | |
| Acrylamide: Bis-Acrylamide 19:1 (40% Solution/Electrophoresis) | ThermoFisher Scientific | BP1406-1 | Review Material Safety Data Sheets |
| Urea (Colorless-to-White Crystals or Crystalline Powder/Mol. Biol.) | ThermoFisher Scientific | BP169-212 | |
| Ammonium peroxodisulphate (APS) ≥98%, Pro-Pure, Proteomics Grade | VWR | M133-25G | |
| Sigmacote | Sigma-Aldrich | SL2-100ML | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) ≥99%, Ultrapure | VWR | 0761-25ML | Review Material Safety Data Sheets |
| Adjustable Slab Gel Systems, Expedeon | VWR | ASG-400 | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 14.5cm | VWR | NGP-125NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 22.0cm | VWR | NGP-200NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 38.7cm | VWR | NGP-400NR | |
| GE Storage Phosphor Screens | Sigma-Aldrich | GE28-9564 | |
| Typhoon™ FLA 7000 Biomolecular Imager | GE Healthcare | 28-9610-73 AB | |
| Beckman Coulter LS6500 Liquid Scintillation Counter | GMI | 8043-30-1194 | |
| C1000 Touch Thermal Cycler | ThermoFisher Scientific | ||
| QuantStudio 6 Flex Real-Time PCR Systems | ThermoFisher Scientific |
Referencias
- Zhuang, Y., Weiner, A. M. A compensatory base change in U1 snRNA suppresses a 5' splice site mutation. Cell. 46 (6), 827-835 (1986).
- Scotti, M. M., Swanson, M. S. RNA mis-splicing in disease. Nature Review Genetics. 17 (1), 19-32 (2016).
- Ward, A. J., Cooper, T. A. The pathobiology of splicing. Journal of Pathology. 220 (2), 152-163 (2010).
- Scalet, D., et al. Disease-causing variants of the conserved +2T of 5' splice sites can be rescued by engineered U1snRNAs. Human Mutatation. 40 (1), 48-52 (2019).
- Yamazaki, N., et al. Use of modified U1 small nuclear RNA for rescue from exon 7 skipping caused by 5'-splice site mutation of human cathepsin A gene. Gene. 677, 41-48 (2018).
- Yanaizu, M., Sakai, K., Tosaki, Y., Kino, Y., Satoh, J. I. Small nuclear RNA-mediated modulation of splicing reveals a therapeutic strategy for a TREM2 mutation and its post-transcriptional regulation. Science Reports. 8 (1), 6937(2018).
- Balestra, D., et al. Splicing mutations impairing CDKL5 expression and activity can be efficiently rescued by U1snRNA-based therapy. International Journal of Molecular Sciences. 20 (17), 20174130(2019).
- Donadon, I., et al. Exon-specific U1 snRNAs improve ELP1 exon 20 definition and rescue ELP1 protein expression in a familial dysautonomia mouse model. Human Molecular Genetics. 27 (14), 2466-2476 (2018).
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565(2010).
- Summer, H., Gramer, R., Droge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). Journal of Visualized Experiments. (32), e1485(2009).
- Dominski, Z., Kole, R. Selection of splice sites in pre-mRNAs with short internal exons. Molecular Cell Biology. 11 (12), 6075-6083 (1991).
- Amir-Ahmady, B., Boutz, P. L., Markovtsov, V., Phillips, M. L., Black, D. L. Exon repression by polypyrimidine tract binding protein. RNA. 11 (5), 699-716 (2005).
- Le Guedard-Mereuze, S., et al. Sequence contexts that determine the pathogenicity of base substitutions at position +3 of donor splice-sites. Human Mutation. 30 (9), 1329-1339 (2009).
- Sharma, S., Wongpalee, S. P., Vashisht, A., Wohlschlegel, J. A., Black, D. L. Stem-loop 4 of U1 snRNA is essential for splicing and interacts with the U2 snRNP-specific SF3A1 protein during spliceosome assembly. Genes and Development. 28 (22), 2518-2531 (2014).
- Steitz, J. A., et al. Functions of the abundant U-snRNPs. Structure and function of major and minor small nuclear ribonucleoprotein particles. , Springer Berlin Heidelberg. Berlin, Heidelberg. 115-154 (1988).
- Fortes, P., et al. Inhibiting expression of specific genes in mammalian cells with 5' end-mutated U1 small nuclear RNAs targeted to terminal exons of pre-mRNA. Proceedings of the National Academy of Sciences U.S.A. 100 (14), 8264-8269 (2003).
- Roca, X., et al. Widespread recognition of 5' splice sites by noncanonical base-pairing to U1 snRNA involving bulged nucleotides. Genes and Development. 26 (10), 1098-1109 (2012).
- Roca, X., Krainer, A. R. Recognition of atypical 5' splice sites by shifted base-pairing to U1 snRNA. Nature Structural Molecular Biology. 16 (2), 176-182 (2009).
- Taladriz-Sender, A., Campbell, E., Burley, G. A. Splice-switching small molecules: A new therapeutic approach to modulate gene expression. Methods. 167, 134-142 (2019).
- Hamid, F. M., Makeyev, E. V. A mechanism underlying position-specific regulation of alternative splicing. Nucleic Acids Research. 45 (21), 12455-12468 (2017).
- Martelly, W., et al. Synergistic roles for human U1 snRNA stem-loops in pre-mRNA splicing. RNA Biology. , 1-18 (2021).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados