A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בדיקה סלולרית מבוססת כתב לניטור יעילות חיבור
In This Article
Summary
פרוטוקול זה מתאר בדיקה עיתונאית minigene כדי לפקח על ההשפעה של מוטציות באתר 5-splice באתר על חיבור ומפתח מדכא U1 snRNA להצלת עיכוב חיבור המושרה מוטציה. הכתב והמדכא U1 snRNA מבנים באים לידי ביטוי בתאי HeLa, ו splicing מנותח על ידי הרחבת פריימר או RT-PCR.
Abstract
במהלך ביטוי הגנים, השלב החיוני של חיבור טרום mRNA כרוך בזיהוי מדויק של אתרי חיבור והרכבה יעילה של מתחמים spliceosomal להצטרף exons ולהסיר introns לפני ייצוא cytoplasmic של mRNA בוגר. יעילות חיבור יכולה להשתנות על ידי נוכחות של מוטציות באתרי חיבור, ההשפעה של גורמי חיבור טרנס-פעולה, או את הפעילות של טיפולית. כאן, אנו מתארים את הפרוטוקול לבדיקה סלולרית שניתן ליישם לניטור יעילות החיבור של כל אקסון נתון. הבדיקה משתמשת בפלסמיד אדפטיבי המקודד 3-exon/2-intron minigene כתב, אשר יכול לבוא לידי ביטוי בתאי יונקים על ידי טרנספקטיבה חולפת. לאחר ההעברה, הרנ"א התאי הכולל מבודד, והיעילות של חיבור אקסון ב-mRNA הכתב נקבעת על ידי הרחבת פריימר או תגובת שרשרת חצי כמותית הפוכה תמלול-פולימראז (RT-PCR). אנו מתארים כיצד ניתן לקבוע כיצד ניתן לקבוע את ההשפעה של מחלות הקשורות 5′ מוטציות באתר splice-site על ידי הצגתם בכתב; וכיצד ניתן להשיג את הדיכוי של מוטציות אלה על ידי שיתוף טרנספקטיה עם U1 קטן גרעיני RNA (snRNA) לבנות נושא מוטציות מפצות באזור 5 ′ שלה כי basepairs עם אתרי 5 ′-splice בצמתים exon-intron ב pre-mRNAs. לכן, הכתב יכול לשמש לעיצוב של חלקיקי U1 טיפולית כדי לשפר את ההכרה של מוטציה 5′ splice-sites. החדרת אתרים רגולטוריים הפועלים cis- פועל, כגון חיבור משפר או רצפי משתיק קול, לתוך הכתב יכול לשמש גם כדי לבחון את התפקיד של U1 snRNP ברגולציה בתיווך על ידי גורם חיבור חלופי ספציפי. לבסוף, כתב המבטא תאים יכול להיות דגירה עם מולקולות קטנות כדי לקבוע את ההשפעה של טיפוליים פוטנציאליים על חיבור לפני mRNA המכונן או על exons נושא אתרי מוטציה 5 ′ splice. בסך הכל, בדיקת הכתב ניתן ליישם כדי לפקח על יעילות splicing במגוון רחב של תנאים כדי ללמוד מנגנונים חיבור בסיסיים ומחלות הקשורות splicing.
Introduction
חיבור טרום-mRNA הוא שלב עיבוד חיוני שמסיר אינטרונים שאינם מקודדים ובדיוק ליגטות exons קידוד כדי ליצור mRNA בוגר. הכרה ברצפי קונצנזוס בצמתים אקסון-אינטרון, המכונים אתר 5′-splice ואתר 3′-splice, על ידי רכיבים של מכונות החיבור יוזמת את תהליך החיבור. ריבונוקלאופרוטאין גרעיני קטן U1 (snRNP) מזהה את האתר 5′-splice על ידי זיווג בסיס של SnRNA U1 לקדם mRNA1. מוטציות תורשתיות גנטית שמשנות 5′-splice רצפי אתר קשורים למחלות רבות2,3. זה צפוי כי אובדן basepairing של U1 snRNA עם אתרי מוטציה 5′-splice גורם חיבור חריג, אשר יכול לסכן את התרגום של התמליל המושפע. גישה טיפולית פוטנציאלית לתיקון פגמי החיבור כרוכה בדיכוי מוטציות על ידי U1 snRNA שונה הנושא שינויים נוקלאוטידים מפצים באזור 5′שלו כי basepairs עם אתר 5′-splice. כזה שונה U1 snRNAs, המכונה גם exon ספציפי U1 snRNAs, נמצאו יעילים בהיפוך פגמים חיבור, וכתוצאה מכך ביטוי חלבון מוגבר מן mRNA4,5,5,6,7,8.
כאן, אנו מתארים את בדיקת ההשלמה של U1 snRNP, בדיקת חיבור סלולרי המבוססת על כתב המאפשרת הערכה של ההשפעה של מוטציות 5′-ss על חיבור של אקסון וניתן להשתמש בה גם לפיתוח של U1 snRNAs שונה כדי לאפשר את ההצלה של הכללת exon. אנו מספקים גם פרוטוקולים לניטור תמלילי הכתבים המשולבים על ידי הרחבת פריימר ו- RT-PCR, ולקביעת הביטוי של U1 snRNAs שונה על ידי הרחבת פריימר ו- RT-qPCR.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. ריאגנטים ומאגרים
הערה: כל עיקור באמצעות מסנני ואקום צריך להתבצע עם 0.2 מיקרומטר פוליאתרסלפון (PES) קרום בארון בטיחות ביולוגית.
- הכן מים ללא RNase על ידי הוספת 1.0 מ"ל של diethylpyrocarbonate (DEPC) ל 1.0 L של מים deionized, לערבב לפחות 1 שעה בטמפרטורת החדר (RT), autoclave פעמיים, ולאחר מכן לצנן ל RT לפני השימוש.
- הכינו את מדיום הנשר המתוקן (DMEM) של Dulbecco על ידי ערבוב חבילה אחת של אבקת DMEM (13.4 גרם), 3.7 גרם נתרן ביקרבונט, 100 מ"ל של סרום בקר עוברי (FBS), פניצילין וסטרפטומיצין לכ-800 מ"ל של מים שעברו דה-יוניזציה סטריליים. הריכוז הסופי של פניצילין וסטרפטומיצין ב- DMEM צריך להיות 50 U / mL ו 50 מיקרוגרם / מ"ל, בהתאמה. כווננו את רמת החומציות ל-7.4 ולאחר מכן הגדילו את הנפח ל-1.0 ליטר עם מים סטריליים שעברו דה-יוניזציה. מעוקרים על ידי סינון ומאחסנים בטמפרטורה של 4 מעלות צלזיוס.
- הכינו תמיסת מלח עם אגירת פוספט 10x (10x PBS) על ידי הוספת 25.6 גרם של דיסודיום מימן הפטהידרט (Na2HPO4· 7H2O), 2 גרם של אשלגן דיהידרוגן פוספט (KH2PO4), 2 גרם של אשלגן כלורי (KCl), ו 80 גרם של נתרן כלורי (NaCl) עד 800 מ"ל של מים deionized. מערבבים כדי להתמוסס ולהרכיב את הנפח ל 1.0 L. לעקר על ידי סינון ולאחסן ב 4 °C (60 °F).
- הכן 0.5 M אתילן דיאמין טטרה-חומצה אצטית (EDTA) על ידי המסת 186.1 גרם של Na2 •EDTA•2H2O לתוך ~ 800 מ"ל של מים deionized. התאימו את רמת החומציות ל-8.0 ולאחר מכן הגדילו את הנפח ל-1.0 ליטר עם מים סטריליים שעברו דה-יוניזציה. מעוקרים על ידי סינון ומאחסנים בטמפרטורה של 4 מעלות צלזיוס.
- הכן פתרון טריפסין-EDTA 1x על ידי ערבוב 100 מ"ל של 10x טריפסין (2.5%), 2 מ"ל של 0.5 M EDTA, ולהוסיף 1x PBS עד 1.0 L. עיקור על ידי סינון aliquot לתוך צינורות חרוטיים 50 מ"ל. יש לאחסן בטמפרטורה של 4 °C (64 °F) למשך 1-2 שבועות או להקפיא בטמפרטורה של -20 °C (60 °F) לשימוש ארוך טווח.
- הכן 2x פורממיד DNA / RNA טעינת צבע על ידי ערבוב 14.4 מ"ל של פורממיד ו 0.6 מ"ל של 0.5 M EDTA עבור נפח סופי של 15 מ"ל. מוסיפים אבקת ברומופנול כחול וקסילן ציאנול לריכוז סופי של 0.02% ומאחסנים ב-4 °C (60 °F).
הערה: פורממיד רעיל ומאכל. קרא את גליונות נתוני הבטיחות החומריים לקבלת המלצות בטיחות נוספות. - הכן 5x טריס / Borate / EDTA חוצץ (5x TBE) על ידי ערבוב 54.0 גרם של בסיס tris, 27.5 גרם של חומצה בורית, ו 20 מ"ל של 0.5 M EDTA לתוך ~ 800 מ"ל של מים deionized. מערבבים כדי להתמוסס ולהרכיב את הנפח ל 1.0 L עם מים deionized.
- הכינו פתרון אלקטרופורזה של ג'ל אוריאה-פוליאקרילמיד (אוריאה-PAGE) על ידי ערבוב של 200 מ"ל של 5x TBE, 250 מ"ל של 40% 19:1 ביס/אקרילאמיד, ו-450.5 גרם אוריאה. לאחר מכן מוסיפים מים deionized עד 1.0 L. לערבב עד החומרים מומסים לחלוטין, ולאחר מכן לעקר על ידי סינון, ולאחסן ב 4 °C (50 °F) בבקבוק זכוכית ענבר.
הערה: ביס/אקרילאמיד רעיל. קרא את גליונות נתוני הבטיחות החומריים לקבלת נהלי בטיחות נוספים. - הכן 10% אמוניום persulfate (APS) על ידי המסת 1 גרם של APS ב 10 מ"ל של מים deionized ולאחסן ב 4 °C (60 °F).
2. Cotransfection של תאי HeLa עם הכתב ו- U1 snRNA plasmids
הערה: ההעברה של תאי הלה חייבת להתבצע בתנאים סטריליים בארון בטיחות ביולוגי. יש לרסס את המשטח החיצוני של כל החומרים ב-70% אתנול לפני הכניסה לארון הבטיחות הביולוגי.
- לשמור על תאי הלה ב- DMEM בחממה של 37 °C עם 5% CO2 על ידי מעבר כל 2-3 ימים כאשר התאים הם כ 80-90% מפגש.
- עבור עובר תאי HeLa, לשאוף בינוני בילה ולאחר מכן לדגור תאים עם 3 מ"ל של 0.25% טריפסין המכיל 1 מ"מ EDTA ב 37 °C (37 °F) במשך 3 דקות.
- לאחר הדגירה, הוסיפו 7 מ"ל של DMEM טרי. מעבירים את מתלי התא לצינור 10 מ"ל, צנטריפוגה ב 1,000 x g במשך 5 דקות.
- Resuspend גלולה התא ב 10 מ"ל של DMEM טרי, ולאחר מכן צלחת התאים על צלחת תרבית רקמות חדשה ב 20% מפגש.
- עבור ארעיות חולפות, לספור תאי הלה עם שקופית hemocytometer נקי ולהכין השעיה עם צפיפות של 2.5 x 105 תאים / מ"ל.
- זרע 1.0 מ"ל של 2.5 x 105 תאים / מ"ל השעיית תא הלה לתוך כל באר של צלחת 12-well ולדגור לילה ב 37 °C (57 °F).
- למחרת, שאפו את המדיום המבוזבז והוסיפו 0.8 מ"ל של DMEM טרי עם סרום.
- הכן פתרון I על ידי הוספת 0.2 מיקרוגרם של Dup51 או Dup51p כתב plasmid, 1.8 מיקרוגרם של pcDNA, pNS6U1, או pNS6U1-5a plasmid, ו 100 μL של מדיום טרנספקטיון לתוך צינור מיקרוצנטריפוגה חדש 1.5 מ"ל.
- הכן תערובת ראשית של פתרון II עבור כל הדגימות להיות transfected על ידי הוספת 100 μL של בינוני transfection ו 4.0 μL של ריאגנט טרנספקטיה לכל מדגם.
- הכן את תערובת השינוע על ידי הוספת 100 μL של פתרון II לתוך כל צינור microcentrifuge המכיל פתרון I.
- מערבולת הטרנספקטיון מתערבבת במשך 15 שניות, צנטריפוגה במיקרוצנטריפוגה על השולחן ב 3,000 x גרם עבור 10 s ב RT, ולאחר מכן דגירה ב RT במשך 5 דקות.
- הוסף את כל 200 μL של תערובת transfection לתוך באר אחת של צלחת תא HeLa 12-well כדי להשיג נפח סופי של 1.0 מ"ל לבאר ודגרו את הצלחת ב 37 °C (60 °F) במשך 48 שעות.
- לאחר הדגירה, לחלץ RNA מתאי HeLa שהושפעו transfected עם גוואנידין תיוציאנט זמין מסחרית תמיסת פנול.
הערה: ריאגנט זה מכיל פנול וצעד זה צריך להתבצע ברדס אדים. השימוש במים שטופלו DEPC מומלץ לחידוש של RNA שחולץ.- שאפו את המדיום המבוזבז והוסיפו 500 μL של ריאגנט לכל באר. דגירה ב RT במשך 5 דקות.
- הומוגניזציה על ידי צנרת למעלה ולמטה. לאחר מכן העבירו את הפתרון לצינור מיקרוצנטריפוגה חדש של 1.5 מ"ל.
- הוסף 100 μL של כלורופורם ומערבולת עבור 15 s.
- צנטריפוגה ב 12,000 x g במשך 15 דקות ב RT.
- העבר 200 μL של RNA המכיל, שכבה מימית עליונה לצינור מיקרוצנטריפוגה חדש 1.5 מ"ל.
- הוסף 2 מיקרוגרם של גליקוגן ו 200 μL של איזופרופנול לכל דגימת RNA. מערבבים על ידי היפוך הצינורות.
- לאסוף את משקעי RNA על ידי צנטריפוגה ב 12,000 x g במשך 10 דקות ב 4 °C (4 °F).
- הסר והשלך את supernatant מבלי להפריע גלולה RNA.
- לשטוף את הכדור פעמיים על ידי הוספת 1.0 מ"ל של 70% אתנול, היפוך הצינורות, צנטריפוגה כמתואר בשלב 2.13.7.
- אוויר לייבש את הכדור במשך ~ 10-20 דקות ב RT resuspend את RNA ב 10-20 μL של מים ללא RNase.
- לקבוע ריכוז RNA על ידי מדידת ספיגה ב 260 ננומטר כפי שתואר על ידי Desjardins ו Conklin9.
- המשך עם הרחבת פריימר או לאחסן דגימות RNA ב -20 °C (60 °F). ניתן לאחסן רנ"א מבודד בטמפרטורה של -20 °C (6-12 חודשים).
3. 32P-תיוג של oligonucleotides
הערה: שלבים הכרוכים בשימוש באוליגונוקלאוטידים בעלי תווית 32P-ATP ו- 32P חייבים להתבצע מאחורי מגן אקריל על ידי אנשים מיומנים באישור גורמים מוסדיים מורשים. הפרוטוקול המתואר להלן יכול לשמש לתיוג של oligonucleotides, Dup3r ו U17-26-R, וסמנים עבור urea-PAGE. השימוש במים שטופלו DEPC מומלץ עבור resuspension של oligonucleotides וחרוזים אי הכללת גודל.
- לצינור מיקרוצנטריפוגה של 1.5 מ"ל, הוסף אוליגונוקלאוטיד, T4 פולינוקלאוטיד קינאז (T4 PNK), מאגר T4 PNK ו- 32P-ATP כמתואר בטבלה 1. הוסף 32P-ATP אחרון לתערובת; זה חשוב.
הערה: לקבלת פתרונות רדיואקטיביים, מומלץ להשתמש בעצות סינון. - דגירה באמבט מים בטמפרטורה של 37 מעלות צלזיוס למשך 30 דקות.
- בעוד תגובות התיוג הן דגירה, resuspend חרוזי אי הכללת גודל עם משקל מולקולרי 25 kDa מנותק על ידי מערבולת בעדינות עבור ~ 10 שניות.
הערה: חרוזי אי הכללת הגודל צריכים להיות מוכנים על פי הוראות היצרן ומאוחסנים כהשעיה של 50% ב 25% אתנול ב 4 °C (4 °F). - הכן עמודות על ידי העברת 600 μL של החרוזים resuspended לעמודה מיני חד פעמית להציב בצינור איסוף 1.5 מ"ל והחזרת מלאי חרוזים ל 4 °C (600 °F).
- צנטריפוגה ב 2,000 x g במשך 1 דקות ב RT ולהשליך את הזרימה דרך.
- לשטוף את החרוזים על ידי הוספת 300 μL של מים ללא RNase לעמודה.
- חזור על שלבים 3.5. ו-3.6. פעמיים והעבירו את המיני-טור לצינור צנטריפוגות חדש של 1.5 מ"ל.
- הוסף את תערובת התגובה קינאז לעמודת חרוזים גודל אי הכללה וצנטריפוגה ב 5000 x g במשך 1 דקה ב RT.
- לאסוף ולשמור את הזרימה דרך, אשר יש oligonucleotide 32P שכותרתו, ולהשליך את כל הטיפים והצינורות לתוך תיבת פסולת אקרילי.
- הוסף 20 μL של מים ללא RNase כדי לדלל oligonucleotide 32P שכותרתו לריכוז סופי של 2.5 מיקרומטר.
הערה: יש לדלל את הסמנים המסומנים לפי הצורך לטעינה על ג'לים מסוג אוריאה-PAGE. - אחסן את האוליגונוקלאוטיד המסומן בקופסת מיקרו-טיוב אקרילית בטמפרטורה של -20 °C (60 °F) או המשך בניתוח הרחבת פריימר.
4. ניתוח תמלילי הכתבים המשולבים על ידי הרחבת פריימר
הערה: מומלץ לנקות משטחים וציוד עם ריאגנט השבתת RNase לפני השימוש.
- הוסף 2.0 מיקרוגרם של RNA המופק מתאי הלה לצינורות מיקרוצנטריפוגה נפרדים של 200 μL והוסף מים ללא RNase כדי לפצות את הנפח ל 6.55 μL.
- הכן את Master Mix I עם 32P-Dup3r ו- dNTPs המדוללים כפי שמוצג בטבלה 2 והוסף 0.9 μL של התערובת לכל צינור PCR המכיל דגימות RNA.
- לדגור על הצינורות, תחילה ב 65 °C (65 °F) במשך 5 דקות ולאחר מכן על קרח במשך 1 דקות.
- הכן תערובת ראשית II עם מאגר גדיל ראשון 5x, dithiothreitol (DTT), מעכב RNase, ותמלול הפוך כפי שמוצג בטבלה 2.
- הוסף 2.55 μL של התערובת לכל צינור המכיל RNA ומאסטר לערבב 1; הנפח הכולל של התגובה צריך להיות 10 μL. שמור את הצינורות ב RT במשך 10 דקות.
- מעבירים את הצינורות לאמבטיה יבשה או למחזור תרמי ודגרה, תחילה בטמפרטורה של 50 מעלות צלזיוס למשך 60 דקות ולאחר מכן ב-70 מעלות צלזיוס למשך 15 דקות.
- לאחר הדגירה, הוסיפו 10 μL של 2x formamide RNA טעינה צבע לכל מדגם ולאחסן בתיבה אקרילי ב -20 °C (20 °F) או להמשיך עם הפרדת שברים על ידי urea-PAGE באמצעות ג'ל ארוך 14 ס"מ הדמיה של תמונת ג'ל כמתואר להלן בשלב 8.
- בצע סריקה densitometric של תמונת הג'ל עם תוכנת ניתוח תמונה ולהשתמש בעוצמות הרצועה של מוצרים כלולים ודילוג כדי לחשב את אחוז ההכללה exon 2 כפי שמוצג להלן.

5. ניתוח תמלילי הכתבים המשולבים על ידי RT-PCR פלואורסצנטי
הערה: ניתוח RT-PCR המתואר להלן משתמש משושים אקראיים עבור סינתזת cDNA ושילוב של Dup8f ללא תווית ו Cy5 תווית Dup3r oligonucleotides להגברת PCR של המוצרים המשולבים.
- עבור סינתזה cDNA, להוסיף 2.0 מיקרוגרם של RNA שחולצו מתאי Hela transfected לתוך צינורות מיקרוצנטריפוגה נפרדים 200 μL ולהוסיף מים ללא RNase כדי לפצות את הנפח ל 11.0 μL.
- הכן את Master Mix I, המכיל כישופים אקראיים ו- dNTP כפי שמוצג בטבלה 3 והוסף 2.0 μL של התערובת לכל דגימה. דגירה, תחילה ב 65 °C (65 °F) במשך 5 דקות ואז על קרח במשך 1 דקות.
- הכינו את Master Mix II, המכיל את מאגר הגדיל הראשון, מעכב RNase, DTT ותמלול הפוך כפי שמוצג בטבלה 3 והוסיפו 7.0 μL של התערובת לכל צינור המכיל RNA ומאסטר מיקס I.
- שמור את הצינורות ב RT במשך 10 דקות, ולאחר מכן לדגור ב 50 °C (50 °F) במשך 60 דקות ו 70 °C (70 °F) במשך 15 דקות.
הערה: תגובות cDNA שהושלמו עשויות להיות מאוחסנות בטמפרטורה של -20 °C (60 °F). - עבור PCR, העבר 1.0 μL (100 ng/ μL) של כל דגימת cDNA לצינורות PCR חדשים.
- הכינו תערובת ראשית המורכבת מ- Dup8f, Cy5-Dup3r, dNTPs, מאגר טאק, טאק פולימראז ומים כמתואר בטבלה 4 והוסיפו 11.5 μL של התערובת לכל צינור המכיל cDNA.
- בצע PCR באמצעות רוכב אופניים תרמי עם שלב denaturation ראשוני ב 94 °C (5 °F) במשך 3 דקות; ואחריו 20 מחזורים של denaturation (94 °C (94 °C (30 °C ( 30 °F ) עבור 30 s), והרחבה (72 °C (72 °C (72 °F עבור 15 s), וצעד סיום ב 72 °C ((72 °F) במשך 5 דקות.
- הוסף 12.5 μL של 2x פורממיד צבע טעינת DNA פורממיד לכל צינור וחום ב 95 °C (5 דקות).
- אחסן את תגובת ה- PCR בטמפרטורה של -20 °C (60 °F) או המשך עם אוריאה-PAGE כמתואר להלן בשלב 8.1-8.4.
- לאחר אלקטרופורזה, הסר את לוחות הזכוכית ממנגנון האלקטרופורזה וסרוק באמצעות הדמיית פלואורסצנטיות כדי לדמיין את הג'ל.
- בצע סריקה densitometric של תמונת הג'ל והשתמש בעוצמת הרצועה של המוצרים הכלולים והמוצרים שהדילג עליהם כדי לחשב את אחוזי ההכללה של אקסון 2 כמתואר בשלב 4.8.
6. ניתוח של ביטוי SnRNA U1 משתנה על ידי הרחבת פריימר
- הוסף 2.0 מיקרוגרם של RNA המופק מתאי הלה לצינורות מיקרוצנטריפוגות נפרדים של 200 μL והוסף מים נטולי RNase כדי להפוך את הנפח ל- 4.325 μL ולאחר מכן להוסיף dATP, כפי שמוצג בטבלה 5.
- הוסף 10,000 עלות לדקה של 32P-U17-26-R oligonucleotide לכל צינור.
הערה: כדי להכין פתרון של 10,000 cpm/μL של 32P-U17-26-R (משלב 3), לדלל את האוליגונוקלאוטיד המסומן (דילול של 1:20), לקבוע cpm ב- 1.0 μL באמצעות מונה נוצץ, ולדלל עוד יותר עם מים deionized כדי להכין פתרון של 10,000 cpm / μL בצינור מיקרו-צנטריפוגה חדש של 1.5 מ"ל. - דגירה בטמפרטורה של 65 מעלות צלזיוס למשך 5 דקות, ולאחר מכן על קרח למשך דקה אחת.
- הכינו תערובת ראשית עם מאגר גדילים ראשון 5x, מעכב RNase, DTT ותמלול הפוך כפי שמוצג בטבלה 5 והוסיפו 1.8 μL של התערובת לכל דגימה.
- דגירה, תחילה ב RT במשך 10 דקות ולאחר מכן ב 42 °C (50 °F) במשך 10 דקות.
- לאחר הדגירה, הוסיפו 10 μL של 2x formamide RNA טעינה צבע לתוך כל מדגם ולאחסן בתיבה אקרילי ב -20 °C (60 °F) או להמשיך עם הפרדת שברים על ידי urea-PAGE באמצעות ג'ל ארוך 38 ס"מ (ראה שלב 8).
7. ניתוח של ביטוי snRNA U1 משתנה על ידי RT-qPCR
- לדלל את מלאי cDNA מוכן כמתואר לעיל בשלבים 5.1 - 5.4 לריכוז של 0.2 ng / μL.
- Pipette 5.0 μL של cDNA מדולל לתוך בארות בודדות של צלחת qPCR 96-well בשלושה עותקים. הוסף מים שעברו דה-יוניזציה במקום cDNA עבור פקד ללא תבנית (NTC).
- הכן שני תערובות פריימר נפרדות המורכבות מהפריימרים הקדמיים וההפוכים עבור U1 ו- U2 snRNAs, ומים כפי שמוצג בטבלה 6.
הערה: רצפים עבור U1 ו- U2 snRNA ספציפיים פריימרים מסופקים בטבלה 7. - הוסף 5.0 μL של U1 ו U2 snRNA פריימר לערבב מדגם בארות NTC.
- הוסף 10.0 μL של תערובת PCR בזמן אמת לכל באר.
- לאטום את הצלחות עם סרט אופטי, ולאחר מכן צנטריפוגה ב 1,000 x g במשך 2 דקות ב RT כדי לאסוף את התגובות לתחתית הבארות.
- בצע qPCR עם שלב denaturation ראשוני ב 95 °C (95 °C ( 95 ° C ) במשך 10 דקות, ואחריו 40 מחזורים של פרוטוקול 2 שלבים המורכב denaturation (95 °C ( 95 °C עבור 15 s) וחישול / הרחבה (62 °C (62 °C CC עבור 60 s) ערכים בעת איסוף מחזור כימות הסף (Cq) ערכים של משרעות היעד.
- סיים את תגובת qPCR על-ידי בדיקת שיא יחיד בעקומת הניתוק לתגובות SnRNA U1 ו- U2.
- מערכי Cq, חשב את דלתא Cq (ΔCq) עבור U1 ו- U2 snRNAs בהשוואה לפקד pcDNA.
- קבע את ביטוי snRNA של משתנה U1 ככמות יחסית (RQ) של U1 בהשוואה ל- U2 באמצעות ערך ΔΔCq כפי שמוצג להלן עבור כל הדגימות.

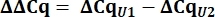
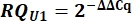
8. התקנה והפעלה של ג'לים Urea-PAGE
הערה: הרכבה של צלחות זכוכית ואת מנגנון הפעלת ג'ל צריך להתבצע על פי הוראות היצרן. הליהוק של ג'ל 10% אוריאה-PAGE יכול להתבצע על פי פרוטוקול שתואר בעבר על ידי Summer et al.10. צעדים הכוללים הכנת סמנים ודגימות, ושל ריצה ויזואליזציה של ג'לים מתוארים להלן. לחלופין, כדי למנוע מהג'ל להידבק ללוחות זכוכית, המשטח הפנימי עשוי להיות מצופה בתמיסת סיליקון על ידי הוספת 1 מ"ל של הפתרון על פני השטח והתפשטות שווה על פני השטח כולו עם רקמות. לאחר הייבוש, יש לשטוף את הצלחות במים deionized ויבש שוב.
אזהרה: אקרילאמיד לא פולימרי הוא רעלן עצבי ויש לטפל בו עם הגנות המומלצות בגיליון נתוני הבטיחות החומרית.
- הכן את דגימות הרחבת פריימר וסמנים על ידי חימום ב 95 °C (5 °F) במשך 5 דקות ולאחר מכן צנטריפוגה ב microcentrifuge שולחן ב 3,000 x גרם עבור 5 s ב RT.
- לפני טעינת הסמנים והדגימות, לשטוף את הבארות עם מאגר TBE 1x כדי להסיר את אוריאה מיושב.
- טען 10 μL / מדגם / טוב ולהפעיל את הג'ל ב 300-500V במשך 2-3 שעות או עד קסילן ציאנול מגיע לתחתית.
הערה: ניתן לטעון כ- 1,000 סל"ד של הסמן בעל התווית 32P. - לאחר אלקטרופורזה, להסיר את לוחות הזכוכית ממנגנון אלקטרופורזה.
- מפרידים בזהירות את שתי הצלחות כך שהג'ל מונח שטוח על משטח זכוכית ומעבירים את הג'ל לנייר סינון ומכסים אותו בניילון נצמד.
- יש לייבש את הג'ל על נייר הסינון בטמפרטורה של 80 מעלות צלזיוס למשך 30 דקות באמצעות מייבש ג'ל.
- מניחים את הג'ל היבש בקלטת הדמיה זרחנית ושומרים על RT למשך הלילה.
הערה: יש למחוק את מסך הזרחן באמצעות תיבת אור לפני השימוש. - כדי לדמיין את תמונת הג'ל, הסירו את המסך וסרקו באמצעות תמונה זרחנית.
Access restricted. Please log in or start a trial to view this content.
תוצאות
כתב ההסתבכות Dup51, מיניגן תלת-שני אקסון-2, נגזר מהגן האנושי β-גלובין ומתואר בעבר (איור 1A)11,12 . יצרנו כתב מוטציה, Dup51p, על ידי החדרת תסמונת אשר הקשורים 5-splice מוטציות באתר המתרחשות exon 3 של protocadherin 15 (PCDH15) gene13. רצף האתרים של 5-splice בצומת א?...
Access restricted. Please log in or start a trial to view this content.
Discussion
הבדיקה יכולה להיות מותאמת לניתוח חיבור בשורות תאים שאינם HeLa, עם זאת, גורמים המשפיעים על יעילות טרנספקטיה, כגון מפגש תאים וכמות של DNA ייתכן שיהיה צורך אופטימיזציה. יחס מבנה הכתב ל- U1 הוא פרמטר קריטי נוסף שייתכן שיהיה צורך לקבוע בהתאם לרמות הביטוי שנצפו בסוגי תאים אחרים. איכות הרנ"א המופק היא ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי כספים ל- S.S. מהמכונים הלאומיים לבריאות (R21CA170786 ו- R01GM127464) ולאגודה האמריקאית לסרטן (מענק המחקר המוסדי 74-001-34-משמרות המהפכה) ול- S.S. ו- W.M מתוכנית שותפות המחקר בעמק (P1-4009 ו- VRP77). התוכן הוא באחריות המחברים בלבד ואינו מייצג בהכרח את הדעות הרשמיות של המכונים הלאומיים לבריאות.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Reagent Grade Deionized Water | ThermoFisher Scientific | 23-751628 | |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758-25ML | |
| Dulbecco's Modified Eagle Medium (DMEM) powder packet | Gibco | 12100-046 | |
| Sodium Bicarbonate | ThermoFisher Scientific | S233-500 | |
| Fetal Bovine Serum (FBS), Australian Source, Heat Inactivated | Omega Scientific | FB-22 | |
| Penicillin-Streptomycin (P/S) | Sigma-Aldrich | P4458-100ML | |
| Sodium Hydroxide, Standard Solution 1.0N | Sigma-Aldrich | S2567-16A | |
| Hydrochloric Acid, Certified ACS Plus, 36.5 to 38.0% | ThermoFisher Scientific | A144-500 | |
| Disposable PES Bottle Top Filters | ThermoFisher Scientific | FB12566510 | |
| EDTA Disodium Salt Dihydrate | Amresco | 0105-2.5KG | |
| 2.5% Trypsin (10x), no phenol red | ThermoFisher Scientific | 15090046 | |
| Sodium Chloride | Fisher Bioreagent | BP358-212 | |
| Potassium Chloride | Fisher Bioreagent | BP366-1 | |
| Disodium Hydrogen Phosphate Heptahydrate | Fisher Bioreagent | BP332-1 | |
| Potassium Dihydrogen Phosphate | Fisher Bioreagent | BP362-1 | |
| Transfection medium - Opti-MEM™ I Reduced Serum Medium, no phenol red | ThermoFisher Scientific | 11058021 | |
| Transfection Reagent - Lipofectamine™ 2000 | ThermoFisher Scientific | 13778150 | |
| TRIzol™ Reagent | ThermoFisher Scientific | 15596018 | |
| Chloroform (Approx. 0.75% Ethanol as Preservative/Molecular Biology) | ThermoFisher Scientific | BP1145-1 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2818-4 | |
| Isopropanol, Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2618-212 | |
| Glycogen (5 mg/ml) | ThermoFisher Scientific | AM9510 | |
| Direct-zol RNA Miniprep Kit | Zymo Research | R2052 | |
| ATP, [γ-32P]- 6000Ci/mmol 150mCi/ml Lead, 1 mCi | PerkinElmer | NEG035C001MC | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| Size exclusion beands - Sephadex® G-25 | Sigma-Aldrich | G2580-10G | |
| Size exclusion mini columns | USA Scientific | 1415-0600 | |
| pBR322 DNA-MspI Digest | New England Biolabs | N3032S | |
| Low Molecular Weight Marker, 10-100 nt | Affymetrix | 76410 100 UL | |
| Rnase inactivating reagents - RNaseZAP™ | Sigma-Aldrich | R2020-250ML | |
| dNTP Mix (10 mM ea) | ThermoFisher Scientific | 18427013 | |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | ThermoFisher Scientific | 10777019 | |
| Reverse Transcriptase - M-MLV Reverse Transcriptase | ThermoFisher Scientific | 28025013 | used for primer extension |
| Taq DNA Polymerase | ThermoFisher Scientific | 10342020 | |
| Random Hexamers (50 µM) | ThermoFisher Scientific | N8080127 | |
| Real time PCR mix - SYBR™ Select Master Mix | ThermoFisher Scientific | 4472903 | |
| SuperScript™ III Reverse Transcriptase | ThermoFisher Scientific | 18080093 | used for cDNA preparation |
| Dithiothreitol (DTT) | ThermoFisher Scientific | 18080093 | |
| 5X First-Strand Buffer | ThermoFisher Scientific | 18080093 | |
| Formamide (≥99.5%) | ThermoFisher Scientific | BP228-100 | Review Material Safety Data Sheets |
| Bromophenol Blue sodium salt | Sigma-Aldrich | 114405-5G | |
| Xylene Cyanol FF | Sigma-Aldrich | 2650-17-1 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | ThermoFisher Scientific | BP152-5 | |
| Boric Acid (Crystalline/Electrophoresis) | ThermoFisher Scientific | BP168-500 | |
| Acrylamide: Bis-Acrylamide 19:1 (40% Solution/Electrophoresis) | ThermoFisher Scientific | BP1406-1 | Review Material Safety Data Sheets |
| Urea (Colorless-to-White Crystals or Crystalline Powder/Mol. Biol.) | ThermoFisher Scientific | BP169-212 | |
| Ammonium peroxodisulphate (APS) ≥98%, Pro-Pure, Proteomics Grade | VWR | M133-25G | |
| Sigmacote | Sigma-Aldrich | SL2-100ML | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) ≥99%, Ultrapure | VWR | 0761-25ML | Review Material Safety Data Sheets |
| Adjustable Slab Gel Systems, Expedeon | VWR | ASG-400 | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 14.5cm | VWR | NGP-125NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 22.0cm | VWR | NGP-200NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 38.7cm | VWR | NGP-400NR | |
| GE Storage Phosphor Screens | Sigma-Aldrich | GE28-9564 | |
| Typhoon™ FLA 7000 Biomolecular Imager | GE Healthcare | 28-9610-73 AB | |
| Beckman Coulter LS6500 Liquid Scintillation Counter | GMI | 8043-30-1194 | |
| C1000 Touch Thermal Cycler | ThermoFisher Scientific | ||
| QuantStudio 6 Flex Real-Time PCR Systems | ThermoFisher Scientific |
References
- Zhuang, Y., Weiner, A. M. A compensatory base change in U1 snRNA suppresses a 5' splice site mutation. Cell. 46 (6), 827-835 (1986).
- Scotti, M. M., Swanson, M. S. RNA mis-splicing in disease. Nature Review Genetics. 17 (1), 19-32 (2016).
- Ward, A. J., Cooper, T. A. The pathobiology of splicing. Journal of Pathology. 220 (2), 152-163 (2010).
- Scalet, D., et al. Disease-causing variants of the conserved +2T of 5' splice sites can be rescued by engineered U1snRNAs. Human Mutatation. 40 (1), 48-52 (2019).
- Yamazaki, N., et al. Use of modified U1 small nuclear RNA for rescue from exon 7 skipping caused by 5'-splice site mutation of human cathepsin A gene. Gene. 677, 41-48 (2018).
- Yanaizu, M., Sakai, K., Tosaki, Y., Kino, Y., Satoh, J. I. Small nuclear RNA-mediated modulation of splicing reveals a therapeutic strategy for a TREM2 mutation and its post-transcriptional regulation. Science Reports. 8 (1), 6937(2018).
- Balestra, D., et al. Splicing mutations impairing CDKL5 expression and activity can be efficiently rescued by U1snRNA-based therapy. International Journal of Molecular Sciences. 20 (17), 20174130(2019).
- Donadon, I., et al. Exon-specific U1 snRNAs improve ELP1 exon 20 definition and rescue ELP1 protein expression in a familial dysautonomia mouse model. Human Molecular Genetics. 27 (14), 2466-2476 (2018).
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565(2010).
- Summer, H., Gramer, R., Droge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). Journal of Visualized Experiments. (32), e1485(2009).
- Dominski, Z., Kole, R. Selection of splice sites in pre-mRNAs with short internal exons. Molecular Cell Biology. 11 (12), 6075-6083 (1991).
- Amir-Ahmady, B., Boutz, P. L., Markovtsov, V., Phillips, M. L., Black, D. L. Exon repression by polypyrimidine tract binding protein. RNA. 11 (5), 699-716 (2005).
- Le Guedard-Mereuze, S., et al. Sequence contexts that determine the pathogenicity of base substitutions at position +3 of donor splice-sites. Human Mutation. 30 (9), 1329-1339 (2009).
- Sharma, S., Wongpalee, S. P., Vashisht, A., Wohlschlegel, J. A., Black, D. L. Stem-loop 4 of U1 snRNA is essential for splicing and interacts with the U2 snRNP-specific SF3A1 protein during spliceosome assembly. Genes and Development. 28 (22), 2518-2531 (2014).
- Steitz, J. A., et al. Functions of the abundant U-snRNPs. Structure and function of major and minor small nuclear ribonucleoprotein particles. , Springer Berlin Heidelberg. Berlin, Heidelberg. 115-154 (1988).
- Fortes, P., et al. Inhibiting expression of specific genes in mammalian cells with 5' end-mutated U1 small nuclear RNAs targeted to terminal exons of pre-mRNA. Proceedings of the National Academy of Sciences U.S.A. 100 (14), 8264-8269 (2003).
- Roca, X., et al. Widespread recognition of 5' splice sites by noncanonical base-pairing to U1 snRNA involving bulged nucleotides. Genes and Development. 26 (10), 1098-1109 (2012).
- Roca, X., Krainer, A. R. Recognition of atypical 5' splice sites by shifted base-pairing to U1 snRNA. Nature Structural Molecular Biology. 16 (2), 176-182 (2009).
- Taladriz-Sender, A., Campbell, E., Burley, G. A. Splice-switching small molecules: A new therapeutic approach to modulate gene expression. Methods. 167, 134-142 (2019).
- Hamid, F. M., Makeyev, E. V. A mechanism underlying position-specific regulation of alternative splicing. Nucleic Acids Research. 45 (21), 12455-12468 (2017).
- Martelly, W., et al. Synergistic roles for human U1 snRNA stem-loops in pre-mRNA splicing. RNA Biology. , 1-18 (2021).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved