Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Expresión bacteriana y purificación de la metaloproteinasa-3 de matriz humana mediante cromatografía de afinidad
* Estos autores han contribuido por igual
En este artículo
Resumen
La purificación, diálisis y activación de His-tag se emplean para aumentar los rendimientos de la expresión de proteínas del dominio catalítico soluble y activo metaloproteinasa-3 en bacterias. Las fracciones de proteínas se analizan a través de geles SDS-PAGE.
Resumen
Las metaloproteinasas de matriz (MMP) pertenecen a la familia de las proteasas de metzincina con funciones centrales en la degradación y remodelación de la matriz extracelular (ECM), así como interacciones con varios factores de crecimiento y citoquinas. La sobreexpresión de MMP específicos es responsable en varias enfermedades como el cáncer, las enfermedades neurodegenerativas y las enfermedades cardiovasculares. Los MMP han sido el centro de atención recientemente como objetivos para desarrollar terapias que puedan tratar enfermedades correlacionadas con la sobreexpresión de MMP.
Para estudiar el mecanismo MMP en solución, se necesitan métodos de expresión y purificación de proteínas recombinantes más fáciles y robustos para la producción de MMP activos y solubles. Sin embargo, el dominio catalítico de la mayoría de las MMP no se puede expresar en Escherichia coli (E. coli) en forma soluble debido a la falta de maquinaria posttraduccional, mientras que los sistemas de expresión de mamíferos suelen ser costosos y tienen rendimientos más bajos. Los cuerpos de inclusión MMP deben someterse al tedioso y laborioso proceso de purificación y repliega extensos, reduciendo significativamente el rendimiento de las MMP en la conformación nativa. Este artículo presenta un protocolo que utiliza células Rosetta2(DE3)pLysS (en adelante, R2DP) para producir el dominio catalítico de la metaloproteinasa-3 de la matriz (MMP-3cd), que contiene una etiqueta His-terminal N seguida de pro-dominio (Hisx6-pro-MMP-3cd) para su uso en la purificación de afinidad. Las células R2DP mejoran la expresión de proteínas eucariotas a través de un plásmido resistente al cloranfenicol que contiene codones normalmente raros en los sistemas de expresión bacteriana. En comparación con la línea celular tradicional de elección para la expresión de proteínas recombinantes, BL21 (DE3), la purificación con esta nueva cepa mejoró el rendimiento de Hisx6-pro-MMP-3cd purificado. Tras la activación y desalinización, el dominio pro se divide junto con la etiqueta His-tag N-terminal, proporcionando MMP-3cd activo para uso inmediato en innumerables aplicaciones in vitro . Este método no requiere equipos costosos o proteínas de fusión complejas y describe la producción rápida de MMP humanos recombinantes en bacterias.
Introducción
La mayoría de las proteínas eucariotas complejas sufren elaboradas modificaciones posttraduccionales después de la expresión, requiriendo un plegamiento de proteínas altamente asistido y cofactores para ser funcionales1. La producción de grandes cantidades de proteína humana soluble en un huésped bacteriano sigue siendo un desafío importante debido a los altos costos y la falta de métodos robustos de expresión y purificación, incluso para experimentos de laboratorio a menor escala2,3. Las MMP, endopeptidasas humanas con gran peso molecular, generalmente se expresan como cuerpos de inclusión insolubles cuando se expresan en E. coli. La extracción de MMP humanas solubles a menudo conduce a un laborioso y lento proceso de solubilización y repliega4.
Las MMP tienen funciones críticas tanto en procesos fisiológicos como patógenos. Las MMP humanas son una familia de 23 endopeptidasas de zinc, categorizadas por estructura y especificidad de sustrato, y expresadas diferencialmente a pesar de un dominio catalítico altamente conservado5,6. Las MMP se secretan como zimogenas inactivas, reguladas a través de la activación posttraduccional y sus inhibidores endógenos, inhibidores tisulares de metaloproteinasas (TIMP)7,8,9,10. Aunque inicialmente se reconocieron por su papel en el recambio de ECM, las MMP también se han implicado en el desarrollo, la morfogénesis, la reparación de tejidos y la remodelación8. La desregulación de las MMP se ha relacionado notablemente con el cáncer junto con las enfermedades neurodegenerativas, cardiovasculares y fibróticas, entre otras enfermedades5,7.
El desarrollo de métodos robustos de producción de MMP a gran escala es fundamental para garantizar el éxito de futuros estudios de mecanismos de MMP a través de ensayos bioquímicos y basados en células. Varias MMP se han expresado previamente en bacterias11, incluidas las MMP marcadas con Hisx6, sin alterar la actividad de MMP12,13,14,15. Sin embargo, estos métodos incluyen pasos tediosos y largos que pueden ser difíciles de replicar.
Las células de mamíferos también se pueden utilizar para expresar muchas proteínas humanas diferentes al tiempo que garantizan las modificaciones posttraduccionales adecuadas16. Aunque el sistema de expresión de mamíferos es una opción ideal para producir proteínas humanas recombinantes con modificaciones post-traduccionales adecuadas, las principales desventajas de este método son los bajos rendimientos iniciales, los costosos medios de crecimiento y reactivos, los largos plazos para alcanzar líneas de expresión estables y el riesgo de contaminación con otras especies como hongos o bacterias2,11 . Además, la producción de MMP en líneas celulares de mamíferos produce impurezas a partir de proteínas celulares asociadas como TIMPs o fibronectinas11. A diferencia del lento crecimiento celular observado en las células de mamíferos, el sistema de expresión bacteriana ofrece una producción de proteínas a gran escala en un corto período de tiempo junto con medios y requisitos de crecimiento más simples. Sin embargo, debido a la falta de otras proteínas celulares asociadas (es decir, TIMP) en los sistemas de expresión bacteriana, las MMP activas a concentraciones más altas están sujetas a degradación a través de la autoproteólisis, lo que resulta en un rendimiento deficiente de MMP17.
Este artículo describe un método detallado para la expresión bacteriana, purificación y activación de Hisx6-pro-MMP-3cd recombinante utilizando E. coli como huésped de expresión debido a su asequibilidad, simplicidad y éxito en la producción de mayores rendimientos de MMPs2,3,18. Dado que E. coli carece de la maquinaria de plegamiento de proteínas y el procesamiento posttraduccional requerido para las MMP recombinantes y otras proteínas complejas, muchas cepas de E. coli han sido diseñadas para superar estas limitaciones, haciendo de E. coli un huésped más adecuado para la expresión de MMP-3cd humano recombinante,19,20 . Por ejemplo, la cepa R2DP utilizada en este estudio mejora la expresión eucariota al suministrar un plásmido resistente al cloranfenicol que contiene codones raramente utilizados en E. coli.
Como se describe en este protocolo, después de la sobreexpresión de cuerpos de inclusión relativamente puros del vector pET-3a (Figura 1) en células R2DP, se extraen y desnaturalizan las proteínas del dominio catalítico Hisx6-pro-MMP-3 (MMP-3cd)4. Hisx6-pro-MMP-3cd3,19 se purificó mediante cromatografía de etiqueta de afinidad. Tras el repliegue y la diálisis, el pro-MMP-3cd (zymogen) fue activado por acetato de 4-aminofenilmercúrico (APMA), y el análisis SDS-PAGE se utiliza para evaluar los rendimientos y la necesidad de una mayor purificación5,21. Este protocolo describe la expresión, purificación y activación de MMP-3cd soluble como ejemplo. Sin embargo, también se puede utilizar como guía para la expresión de otras MMP y proteasas humanas con expresión similar, y mecanismos de activación (Figura 2). Para otras proteínas distintas de MMP-3cd, se recomienda al lector que determine las composiciones tampón óptimas y los métodos para su proteína objetivo antes de intentar este protocolo.
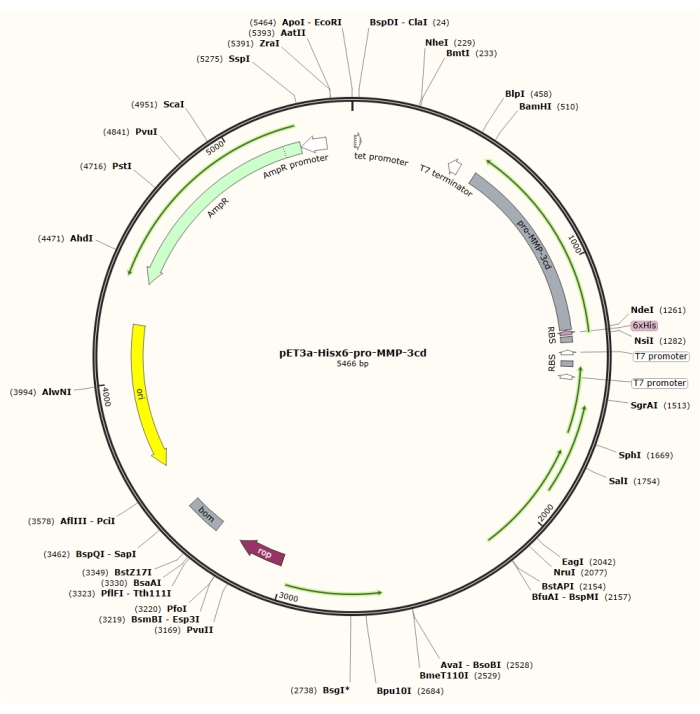
Figura 1: Mapa plásmido del plásmido pET-3a-Hisx6-pro-MMP-3cd. El vector pET-3a incluye un gen de resistencia a la ampicilina. Una secuencia de etiquetas Hisx6 N-terminal se clona en el vector basado en pET-3a, incluyendo pro-MMP-3cd, para producir la construcción pET-3a-Hisx6-pro-MMP-3cd bajo control del promotor T7 entre los sitios de restricción BamHI y NdeI. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Expresión bacteriana del plásmido pro-MMP-3cd, purificación, repliegue y activación. 1.1: El plásmido pET-3a-Hisx6-pro-MMP-3cd se transformó en células BL21(DE3) o R2DP. 1.2: La expresión de la proteína Pro-MMP-3cd se indujo mediante IPTG. 1.3: La lisis química y la sonicación se utilizan para extraer proteínas Hisx6-pro-MMP-3cd que son principalmente insolubles y se encuentran en los cuerpos de inclusión. La urea se utilizó para desnaturalizar y solubilizar proteínas de los cuerpos de inclusión. 2.1. La proteína Hisx6-pro-MMP-3cd desnaturalizada se purificó mediante cromatografía por afinidad. 3. El Hisx6-pro-MMP-3cd eluido se replegó lentamente durante la diálisis a través de la eliminación gradual de la urea del tampón. 4. Finalmente, la proteína MMP-3cd replegada se activó utilizando APMA mediante la eliminación del dominio propéptido N-terminal. APMA se elimina más tarde de la solución a través de la desalinización. Los números corresponden a las secciones del protocolo que describen estos pasos. Abreviaturas: MMP-3cd = Dominio catalítico de la metaloproteinasa-3 de la matriz; APMA = acetato de 4-aminofenilmercúrico. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Expresión MMP
- Clonación y transformación de pET-3a-Hisx6-pro-MMP-3cd en células R2DP
- Digerir el plásmido pET-3a (ver la Tabla de Materiales) con enzimas de restricción NdeI y BamHI en Digest Buffer (ver la Tabla de Materiales). En un volumen total de reacción de 40 μL, agregue 4 μL de Digest Buffer, 33 μL de plásmido de 100 ng / μL y 1.5 μL de cada enzima de restricción y permita que la reacción continúe durante ~ 2 h hasta su finalización a 37 ° C.
- Realice una reacción de PCR en la secuencia MMP-3cd para insertar una etiqueta His-terminal N. Utilice 25 μL de mezcla de PCR (véase la Tabla de materiales), 2,5 μL de cebadores de 10 μM (Figura suplementaria S1) y 1,25 μL de la secuencia de inserción de 100 ng/μL. Añadir agua estéril a un volumen de reacción final de 50 μL.
- Ejecute el producto de PCR y el vector digerido en un gel de agarosa al 1%. Purifique las bandas de gel utilizando un kit de recuperación de gel (consulte la Tabla de materiales) según el protocolo del fabricante.
- Clonar el producto de PCR amplificado en el vector digerido entre los sitios de restricción NdeI y BamHI utilizando DNA Assembly Mix (ver la Tabla de Materiales). Utilice herramientas en línea para determinar el volumen requerido del vector de inserción y corte para un volumen de reacción total de 15 μL.
- Descongelar una alícuota de 50 μL de células de alta eficiencia de transformación (ver la Tabla de Materiales) en hielo hasta que se descongele. Medio de crecimiento SOC precalentado (consulte la Tabla de materiales) a 37 °C y placas lb-ampicilina (LB Amp) (consulte la Tabla de materiales).
- Añadir 1-2 μL de la reacción de ensamblaje pET-3a-Hisx6-pro-MMP-3cd a la alícuota de 50 μL. Incubar en hielo durante 30 min.
- Choque térmico de las células incubando a 42 °C durante 30 s. Incubar sobre hielo durante 2 min.
- Añadir 950 μL de medio de crecimiento SOC a cada mezcla transformadora. Agitar durante 1 h a 250 rpm y 37 °C.
- Placa 100 μL de los transformadores en placas LB Amp e incubar durante la noche a 37 °C.
- Inocular cada colonia aislada en 10 mL de LB Amp medio. Agitar durante la noche a 250 rpm y 37 °C.
- Extraer ADN plásmido según el protocolo del fabricante para el kit miniprep (consulte la Tabla de materiales). Confirme la secuencia de la construcción utilizando cebadores T7 hacia adelante y hacia atrás (Figura suplementaria S1).
NOTA: El ADN de construcción pET-3a-Hisx6-pro-MMP-3cd se puede almacenar a -20 °C. Cuando esté listo, proceda con la transformación en celdas R2DP. - Descongele una alícuota de 20 μL de células R2DP (ver la Tabla de Materiales) en hielo durante 2-5 min. Medio de crecimiento SOC precalentado a temperatura ambiente y placas LB Amp CamR a 37 °C (consulte la Tabla de Materiales).
- Añadir 1 μL de pET-3a-Hisx6-pro-MMP-3cd de 100 ng/μL confirmada por secuencia a la alícuota de 50 μL. Revuelva suavemente para mezclar y devuelva el tubo al hielo.
- Incubar el tubo en hielo durante 5 min.
- Choque térmico de las células incubando a 42 °C durante exactamente 30 s. No agitar.
- Coloque las celdas en hielo durante 2 min.
- Añadir 80 μL de medio SOC a temperatura ambiente a la mezcla transformadora. Agitar durante 1 h a 250 rpm y 37 °C.
- Coloque los transformadores en placas LB Amp CamR e incube durante la noche a 37 °C.
- Crecimiento e inducción
- Inocular una colonia única y aislada de transformador R2DP pET-3a-Hisx6-pro-MMP-3cd de una placa LB Amp CamR en 10 mL de medios LB Amp CamR a 37 °C. Agitar a 250 rpm durante la noche (~16 h). Ahorre alícuotas de cada cultivo y prepare existencias de glicerol al 40% (v / v) (consulte la Tabla de materiales) si lo desea.
- Por cultivo nocturno, inocular un matraz de 1 L que contenga 500 ml de medio LB Amp CamR a una densidad óptica a 600 nm (OD600) de 0,05-0,1.
NOTA: Esto debería devolver las células al crecimiento logarítmico. - Mida el OD600 en varios puntos de tiempo, generalmente durante 3-4 h, hasta que caiga entre 0.4 y 0.6.
- Antes de la inducción, alícuota una fracción de cultivo en un tubo de microfuge de 1,5 ml (consulte la Tabla de materiales) y etiquételo como Fracción no inducida. Guárdelo a -80 °C para el análisis del gel. Si no está ejecutando un gel SDS-PAGE, omita este paso y continúe con el paso 1.2.5.
- Inducir los cultivos a una concentración final de 1 mM utilizando 1 M de stock de isopropil-ß-D-tiogalactopiranósido (IPTG) (ver la Tabla de Materiales). Continúe incubando en el agitador de 37 °C durante 3-4 h adicionales.
NOTA: Durante la expresión, el lector debe determinar el óptimo OD600 en el momento de la inducción y la concentración de IPTG. Si el rendimiento disminuye sustancialmente después de la purificación, la concentración de imidazol en los tampones de purificación puede requerir un ajuste, o el pellet celular puede necesitar ser sonicado aún más. - Antes de centrifugar los cultivos, alícuota una fracción de cultivo en un segundo tubo de microfuge de 1,5 ml y etiquételo Fracción inducida. Guárdelo a -80 °C para el análisis del gel. Si no está ejecutando un gel SDS-PAGE, omita este paso y continúe con el paso 1.2.7.
- Centrifugar el cultivo celular en botellas cónicas de 250 ml (ver la Tabla de Materiales) a máxima velocidad y 4 °C durante 10 min.
- Repita el paso 1.2.7 hasta que los cultivos estén completamente peletizados.
NOTA: PAUSA: Los pellets celulares se pueden congelar a -80 °C y descongelar más tarde para su posterior procesamiento. De lo contrario, omita este paso y continúe con el paso 1.3.1.
- Extracción y solubilización de cuerpos de inclusión
NOTA: Prepare 10 M de urea fresca, preferiblemente no antes de un día de anticipación, revolviendo bien hasta que se disuelva por completo. No calentar ni autoclave urea; guárdelo a temperatura ambiente.- Vuelva a suspender el pellet (a partir del paso 1.2.8) en tampón de lisis (consulte la Tabla de materiales). Por gramo de pellet, añadir 3 mL de tampón de lisis y resuspender por vórtice o pipeteo. Agitar durante la noche a 4 °C.
- Añadir 1,25 ml de desoxicolato de sodio al 10% (p/v) (véase la Tabla de Materiales) por 1 L de cultivo. Agitar a temperatura ambiente durante 30 min a 150 rpm.
- Añadir 10 μL de DNasa I (véase la Tabla de Materiales) por 1 L de cultivo. Agitar a temperatura ambiente durante 30 min a 150 rpm.
- Centrifugar durante 10 min a 13.000 × g y 4 °C.
- Reserve una fracción de Lysed MMP para el análisis de gel. Guárdelo a -80 °C. Si no realiza el análisis de gel, continúe con el paso 1.3.6.
NOTA: Después de la centrifugación, el pellet puede ser fibroso y no empaquetado compactamente, por lo que es arriesgado desechar el sobrenadante. Si este es el caso, omita el paso 1.3.6 y continúe con el paso 1.3.7. - Deseche el sobrenadante de las muestras centrifugadas.
NOTA: El protocolo se puede pausar en este punto y los gránulos celulares se congelan a -80 °C y se descongelan más tarde. De lo contrario, omita este paso y continúe con el paso 1.3.7. - Resuspend el pellet en cultivo de 100 mL/L de Inclusion Body Buffer (ver la Tabla de Materiales) mediante pipeteo hacia arriba y hacia abajo.
- Durante la sonicación, mantenga las muestras en hielo para evitar el sobrecalentamiento. Sonicar cada muestra durante 6 ciclos de 15 s, salida 5 y 50% de pulso. Permita períodos de descanso de 15 s para enfriar entre ciclos.
NOTA: Si es necesario, transfiera las muestras a tubos cónicos de 50 ml para una centrifugación adicional (consulte la Tabla de materiales). Centrifugar durante 10 min a 13.000 × g y 4 °C. - Reserve una fracción de Sonicated MMP para el análisis de gel. Guárdelo a -80 °C. Si no realiza el análisis de gel, continúe con el paso 1.3.11.
- Revisa el pellet. Si está fibroso, repita los pasos 1.3.8-1.3.10. Si el pellet es compacto, deseche el sobrenadante y proceda al paso 1.3.12.
NOTA: La sonicación en Inclusion Body Buffer se puede repetir para recuperar más proteína de los restos celulares lisados. Sin embargo, demasiada sonicación puede causar cizallamiento, lo que perjudica el rendimiento de MMP. El protocolo se puede pausar en esta etapa, y los gránulos celulares se pueden congelar a -80 ° C y descongelar más tarde. - Resuspenda cada pellet de un cultivo de 1 L en 5 mL de Tampón de Solubilización (ver la Tabla de Materiales) mediante pipeteo. Incubar durante al menos 30 min en hielo para permitir que las proteínas se solubilicen.
- Reserve una fracción de MMP solubilizado para el análisis de gel. Guárdelo a -80 °C. Si no realiza el análisis de gel, continúe con el paso 1.3.14.
- Centrifugar las células durante 10 min a 13.000 × g y 4 °C. NO DESECHAR EL SOBRENADANTE.
- Si se forma/queda un gránulo después de la centrifugación, vierta el sobrenadante en un tubo cónico separado de 50 ml. Resuspend el pellet en otros 5 mL de Tampón de Solubilización (por 1 L de cultivo) mediante pipeteo hacia arriba y hacia abajo.
- Centrifugar durante 10 min a 13.000 × g y 4 °C. NO DESECHAR EL SOBRENADANTE.
- Repita los pasos 1.3.13 y 1.3.14 hasta que se forme un poco o ningún gránulo después de la centrifugación o solo quede precipitado gris. Agrupa a los sobrenadantes. Deseche o almacene el pellet a -80 °C para una sonicación adicional.
2. Purificación y repliegue MMP
- Purificación de afinidad His-tag (HT)
- Según el protocolo del fabricante, llene una columna de flujo por gravedad (consulte la Tabla de materiales) con resina de Ni-NTA bien mezclada (consulte la Tabla de materiales). Permita que la resina se asiente y se separe del tampón de almacenamiento de tal manera que se forme una línea distinta entre las dos capas.
NOTA: Nunca permita que la resina se seque, ya que el aire penetrará en la resina y dañará el rendimiento de la proteína. Entre usos, realizar el procedimiento de regeneración de resina descrito en la sección 2.2. - Permita que el búfer de almacenamiento se drene. Llene la columna con dos volúmenes de lecho de resina de HT Equilibration Buffer.
- Drene el búfer de equilibrio HT y deseche. A medida que la columna se está drenando, centrífuga el extracto de proteína a 13.000 × g durante 1 min y esterilice con un filtro de 0,22 μm (consulte la Tabla de materiales).
- Cambie el contenedor de residuos por un tubo cónico de 50 ml etiquetado como HT Flowthrough. Agregue el extracto de proteína preparado a la columna.
- Vuelva a aplicar el flujo para maximizar el enlace.
- Reserve una fracción de la fracción flowthrough para el análisis de gel. Guárdelo a -80 °C. Si no realiza el análisis de gel, continúe con el paso 2.1.7.
- Lave inmediatamente la resina con 15 ml de tampón de lavado HT (consulte la Tabla de materiales). Recoja el flujo en tubos cónicos de 15 ml (consulte la Tabla de materiales) etiquetados como HT Wash.
NOTA: Los valores de absorbancia a 280 nm (A280) se obtuvieron mediante espectrofotometría y se utilizaron junto con el peso molecular y el coeficiente de extinción, ε, para estimar las concentraciones de proteínas. Para Hisx6-pro-MMP-3cd desnaturalizado, el peso molecular es de 29.86 kDa, y ε es de 34.38 M-1 cm-1. - Blanking contra HT Wash Buffer, mida y registre el A280. Repita los pasos 2.1.7 y 2.1.8 con fracciones de lavado adicionales. Una vez que el A280 se acerque a la línea de base y se hayan minimizado las impurezas, continúe con el paso 2.1.9.
- Reserve una fracción de fracción de lavado para el análisis del gel. Repita para varias fracciones de lavado. Conservar las fracciones a -80 °C. Si no realiza el análisis de gel, continúe con el paso 2.1.10.
- Eluya inmediatamente las proteínas marcadas con His agregando 5 ml de HT Elution Buffer (consulte la Tabla de materiales). Recoja el flujo como fracciones de 0.5-1 ml en tubos de microfuge etiquetados como HT Elution.
- Reserve una fracción de fracción de elución para el análisis de gel. Repita para múltiples fracciones de elución. Conservar las fracciones a -80 °C. Si no realiza el análisis de gel, continúe con el paso 2.1.12.
- Si el A280 es >0,3 mg/ml, diluya la fracción con HT Equilibration Buffer (consulte la Tabla de Materiales).
NOTA: La fracción eluida debe diluirse a un A280 de 0,3 mg/ml o menos para evitar precipitaciones durante la diálisis. El protocolo se puede pausar aquí y las fracciones agrupadas se congelan a -80 ° C y se descongelan más tarde. De lo contrario, omita este paso y continúe con el paso 2.2.1.
- Según el protocolo del fabricante, llene una columna de flujo por gravedad (consulte la Tabla de materiales) con resina de Ni-NTA bien mezclada (consulte la Tabla de materiales). Permita que la resina se asiente y se separe del tampón de almacenamiento de tal manera que se forme una línea distinta entre las dos capas.
- Regeneración de resina
- Lave la resina con diez volúmenes de lecho de resina de HT Regeneration Buffer (consulte la Tabla de materiales) y diez volúmenes de lecho de resina de agua estéril.
- Almacene la resina como una suspensión al 50% en etanol al 20% (v/v) en agua.
3. Repliegue de proteínas
NOTA: Para volúmenes más pequeños, los casetes de diálisis se pueden usar con un menor riesgo de pérdida de muestra. Se requieren tubos de diálisis si se utilizan volúmenes más grandes (consulte la Tabla de materiales).
- Diálisis
NOTA: La concentración óptima de proteínas para la diálisis es de ~0.3 mg/ml. Si se produce una precipitación significativa durante la diálisis, reduzca el gradiente de concentración de urea entre cada diálisis utilizando un método de diálisis gradual y agregue más pasos intermedios (por ejemplo, de 6 M a 5 M, y luego de 5 M a 4 M, en lugar de omitir la etapa de 5 M). Como los ciclos de congelación-descongelación dañan la estructura celular y proteica, es vital minimizar las pausas en el protocolo.- Según el protocolo del fabricante, use la cantidad adecuada de tubos de diálisis de acuerdo con el volumen de muestras de fracción de elución .
- Sumergir las fracciones de MMP eluidas en tubos de diálisis en 1 L de Diálisis Buffer 1 (ver la Tabla de Materiales). Revuelva el tubo y su contenido en un agitador magnético durante no menos de 8 h a 4 °C.
- Transfiera a 1 L de tampón de diálisis 2 (consulte la Tabla de materiales). Revuelva el tubo y su contenido en un agitador magnético durante no menos de 8 h a 4 °C.
- Transfiera a 1 L de tampón de diálisis 3 (consulte la Tabla de materiales). Revuelva el tubo y su contenido en un agitador magnético durante no menos de 8 h a 4 °C.
- Transfiera la muestra a nuevos tubos cónicos de 50 ml y etiquételos como MMP dializado.
- Examine el tubo en busca de precipitado. Si se ha formado precipitado, centrífuga la muestra durante 1 min a 13.000 × g y 4 °C.
- Transfiera el sobrenadante a nuevos tubos cónicos de 15 ml y etiquételos como MMP plegado.
- Reserve una fracción para el análisis de gel y etiquétela como MMP plegado. Guárdelo a -80 °C. Si no realiza el análisis de gel, continúe con el paso 3.1.9.
NOTA: Si los rendimientos son bajos, el precipitado se puede disolver en HT Equilibration Buffer y los pasos de la sección 3.1 se repiten con tubos de diálisis. Si no se va a realizar un análisis de gel o si el protocolo debe detenerse aquí, congele las muestras a -80 °C y descongele más tarde. Si los rendimientos están en el rango deseado, continúe con el paso 3.2.1.
- Reconcentración
NOTA: Se espera que los coeficientes de extinción para Hisx6-pro-MMP-3cd replegado y desnaturalizado sean los mismos; por lo tanto, los cálculos de A280 no se ven afectados.- Reconcentrar la muestra hasta 0,5 mg/ml. Utilice una celda agitada de 400 ml (consulte la Tabla de materiales) para concentrar la muestra a 15 ml. Para evitar la formación de espuma, use un tubo de reconcentración de 50 ml para concentrarse aún más si es necesario.
NOTA: Si se forma un precipitado, se puede peletizar y disolver en HT Equilibration Buffer. A continuación, repita las secciones 3.1 y 3.2.1. De lo contrario, continúe con el paso 3.2.2. - Reserve una fracción para el análisis de gel y etiquétela como MMP concentrado.
NOTA: El protocolo se puede pausar aquí y las muestras se congelan a -80 °C y se descongelan más tarde.
- Reconcentrar la muestra hasta 0,5 mg/ml. Utilice una celda agitada de 400 ml (consulte la Tabla de materiales) para concentrar la muestra a 15 ml. Para evitar la formación de espuma, use un tubo de reconcentración de 50 ml para concentrarse aún más si es necesario.
4. Activación
- Activación del acetato de 4-aminofenilmercúrico (APMA)
NOTA: APMA es altamente tóxico. Haga una solución madre fresca de 20 mM APMA antes de la activación, y siempre trabaje bajo una campana de humos cuando use APMA. Deseche los residuos de APMA en su contenedor.- Por cada 1 ml de alícuota de MMP (1 mg/ml), añadir 50 μL de 20 mM de APMA (véase la Tabla de materiales) para alcanzar una concentración final de APMA de 1 mM. Incubar durante la noche a 37 °C.
- Si se forma un precipitado, centrímelo a máxima velocidad durante 10 min a 4 °C. Guarde el sobrenadante en un tubo de microfuge de 1,5 ml con la etiqueta MMP activado. Deseche el precipitado en un recipiente marcado para los residuos de APMA.
- Reservar una fracción para el análisis de gel y etiquetarlo como MMP activado.
NOTA: El protocolo se puede pausar aquí y las muestras se congelan a -80 °C y se descongelan más tarde. Si no realiza el análisis de gel, continúe con el paso 4.2.1. Después de la activación, el peso molecular y el coeficiente de extinción de MMP-3cd son 19,40 kDa y 28,42 M-1 cm-1, respectivamente.
- Desalación
- Elimine APMA de la muestra MMP-3cd activada con una columna de desalinización de 2 ml (consulte la Tabla de materiales), siguiendo el protocolo del fabricante.
- Reservar una fracción para el análisis de gel y etiquetarlo como MMP desalinizado. Guárdelo a -80 °C. Si no realiza el análisis de gel, vaya a la sección 4.3 con las muestras restantes.
NOTA: El protocolo se puede pausar aquí y las muestras se congelan a -80 °C y se descongelan más tarde.
- Ejecución de los geles SDS-PAGE
- Ejecute todas las fracciones de proteínas en geles SDS-PAGE: Fracción no inducida, Fracción inducida, MMP lisado, MMP sonicado, MMP solubilizado, Fracción de flujo, Fracción de lavado, Fracción de elución, MMP replegado, MMP concentrado, MMP activado y MMP desalinizado.
- Almacenamiento a largo plazo de MMP-3
- Añadir surfactante no iónico al 0,05% (v/v) (véase la Tabla de Materiales) a las muestras deSaladas de MMP-3cd y almacenarlas a -80 °C.
Resultados
Al ejecutar muestras en SDS-PAGE, debido a que la proteína se expresa en forma de cuerpos de inclusión insolubles, las fracciones lisadas y sonicadas deben contener poco o ningún extracto de Hisx6-pro-MMP-3cd, ya que la proteína aún no se ha resuelto en urea. La Figura 3 compara las fracciones de elución de purificación his-tag de Hisx6-pro-MMP-3cd de células BL21(DE3) y células R2DP. Las fracciones de elución se agruparon por separado para las células BL21(DE3) y R2DP antes de la...
Discusión
La producción a gran escala de MMP solubles, humanos y recombinantes sigue siendo una tarea desafiante. Las células de mamíferos pueden expresar MMP funcionales a altos costos y largos tiempos de espera, mientras que E. coli produce rápidamente altas cantidades de cuerpos de inclusión MMP que deben ser purificados y replegados11,16. Las células R2DP aumentan significativamente el rendimiento de los cuerpos de inclusión MMP, lo que permite un proce...
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos.
Agradecimientos
Evette Radisky y Alexandra Hockla en la Clínica Mayo en Jacksonville, Florida, por proporcionar el plásmido pET-3a-pro-MMP-3cd como la plantilla para clonar el gen Hisx6pro-MMP-3cd, y sus comentarios, junto con el Dr. Paul Hartley del Centro de Genómica de Nevada en la Universidad de Nevada, Reno, para la secuenciación del ADN. Los autores también desean agradecer a Cassandra Hergenrader por ayudar con parte de la expresión de proteínas. M.R.-S. desea agradecer la subvención NIH-P20 GM103650-COBRE Integrative Neuroscience y el PREMIO UNR R&D mICRO SEED Grant Award.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm sterile filter | Sigma Aldrich | SLGP033RS | Used to remove some contaminants from the protein extract before purification, and prevent the Ni-NTA column from clogging |
| 1 L Erlenmeyer flasks | Thermo Fisher Scientific | S76106F | n/a |
| 1 L glass bottles | Thermo Fisher Scientific | 06-414-1D | n/a |
| 1.5 mL microfuge tubes | Thermo Fisher Scientific | 02-682-002 | n/a |
| 15 mL conical tubes | Thermo Fisher Scientific | 339650 | n/a |
| 18 G, 1-in. beveled needle | Amazon | B07S7VBHM2 | Used in combination with the dialysis casette |
| 2 mL desalting column | Thermo Fisher Scientific | 89890 | Removes APMA following activation |
| 2-(N-Morpholino)ethanesulfonic acid (MES) | Thermo Fisher Scientific | AAA1610422 | n/a |
| 250 mL conical bottle cushions | Thermo Fisher Scientific | 05-538-53A | Stabilize conical bottles during large-volume centrifugation |
| 250 mL conical bottles | Thermo Fisher Scientific | 05-538-53 | n/a |
| 400 mL stirred cell | Sigma Aldrich | UFSC40001 | Re-concentrates a much larger volume than the centrifugal filter unit. Rosetta2(DE3)pLysS cells produce high volumes of protein that may exceed the 15 mL limit of the centrifugal filter unit |
| 4-aminophenylmercuric acetate (APMA) | Sigma Aldrich | A9563-5G | Activates MMP-3 by cleaving the propeptide |
| 5 mL syringe | Thermo Fisher Scientific | NC0829167 | Used in combination with the dialysis casette |
| 50 mL conical tubes | Thermo Fisher Scientific | 339650 | Used for storage in many purification steps |
| 50 mL re-concentration tube | Sigma Aldrich | UFC901024D | Used for re-concentrating protein samples after dialysis or removing contaminants |
| Agar | Thermo Fisher Scientific | BP1423-500 | Buffer ingredient that solidifies autoclaved LB media upon cooling |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | Antibiotic used with pET3a vector; used at 100 µg/mL in LB media |
| BamHI | NEB | R3136S | Restriction enzyme to be used with the pET3a vector |
| Calcium chloride (CaCl2) | Thermo Fisher Scientific | 600-30-23 | The calcium ion stabilizes MMP structure |
| Cell spreaders | Thermo Fisher Scientific | 50-189-7544 | Can be used to spread cells across a petri dish after transformation |
| Chloramphenicol | Thermo Fisher Scientific | 22-055-125GM | Antibiotic used with pET3a vector; used at 34 µg/mL in LB media |
| Dialysis Buffer 1 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 4 M Urea. |
| Dialysis Buffer 2 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 2 M Urea. |
| Dialysis Buffer 3 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2 , 1 µM ZnCl2. |
| Dialysis clips | Thermo Fisher Scientific | 68011 | Used in combination with snakeskin dialysis tubing |
| Dialysis tubing | Thermo Fisher Scientific | 88243 | Alternative dialysis method that holds much larger sample volumes, but with higher risk of sample loss |
| Digest buffer | NEB | B7204S | Buffer used in digesting the pET3a vector |
| Disposable cuvettes | Thermo Fisher Scientific | 21-200-257 | Used to measure the bacterial culture OD during growth and expression |
| Dithiothreitol (DTT) | Thermo Fisher Scientific | D107125G | Assists with protein denaturation by reducing any disulfide bonds |
| DNA assembly mix | NEB | E2621S | Used to ligate the Hisx6-pro-MMP-3cd PCR product and digested pET3a vector |
| DNase I | NEB | M0303S | Endonuclease for degrading unfavorable DNA contaminants that could later affect protein purification |
| Ethanol | Thermo Fisher Scientific | A995-4 | n/a |
| Ethylenediaminetetraacetic acid (EDTA) | Thermo Fisher Scientific | J15694-AE | Used in denaturation. Prevents oxidation and subsequent formation of disulfide bonds |
| Gel recovery kit | Promega | A9281 | Isolates and purifies DNA from agarose gels |
| Glycerol | Thermo Fisher Scientific | G33-500 | Used for making glycerol stocks, which are frozen at -80 °C |
| Gravity flow column | BioRad | 7321010 | Used for Ni-NTA purification of recombinantly His-tagged proteins |
| High-transformation efficiency cells | NEB | C2987 | High-transformation efficiency cells with greater chance of success for cloning the N-terminal His-tag into the pET3a-pro-MMP-3cd construct |
| HT Elution Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 250 mM imidazole. Adjust pH to 7.4 |
| HT Equilibration Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea. Adjust pH to 7.4 |
| HT Regeneration Buffer | n/a | n/a | 20 mM MES, 0.1 M NaCl. Adjust pH to 5.0 |
| HT Wash Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 25 mM imidazole. Adjust pH to 7.4 |
| Hydrochloric acid (HCl) | Thermo Fisher Scientific | A144C-212 | Used to pH buffers |
| Imidazole | Thermo Fisher Scientific | AAA1022122 | Mimics the histidine side group. Used to separate non-specifically binding proteins from the his-tagged target protein |
| Inclusion Body Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 5 mM DTT, 2% v/v Triton X 100, 0.5 M Urea. Adjust pH to 8.0 |
| Isopropyl-ß-D-thiogalactopyranoside (IPTG) | Thermo Fisher Scientific | FERR0392 | A reagent that induces target gene expression in pET3a. Make 0.5 mL 1 M aliquots, filter sterilize and store in -20 °C |
| LB Amp CamR media | n/a | n/a | To be poured into a sterible 1 L bottle or 1 L flask. For 1 L, add 25 g LB Broth. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Amp CamR plates | n/a | n/a | To be poured into sterile petri dishes. Pour until the petri dish lid is completely covered. 1 L of media yields 40-60 plates. For 1 L: 25 g LB Broth, 16 g Agar. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Broth | Thermo Fisher Scientific | BP1426-2 | Pre-mixed with tryptone, yeast extract, and sodium chloride |
| Lysis Buffer | n/a | n/a | 50 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 0.133 g/mL lysozyme, 0.49% v/v Triton X-100. Adjust pH to 8.0 |
| Lysozyme | MP Biomedicals | 195303 | Used in protein extraction. Enzyme that lyses bacterial cell walls |
| Miniprep kit | Promega | A1330 | If successful, extracts the pET3a-pro-MMP-3cd construct from transformants |
| NdeI | NEB | R0111S | Restriction enzyme to be used with the pET3a vector |
| Ni-NTA resin | Thermo Fisher Scientific | PI88221 | Used to bind recombinant his-tagged proteins. This strong interaction can be displaced with higher concentrations of imidazole |
| Nonionic surfactant | Thermo Fisher Scientific | PI28316 | Storage detergent for preventing MMP aggregation. Minimizes interactions between hydrophobic residues on the MMP surface and water molecules, without disrupting catalytic activity. |
| PCR mix | NEB | M0492S | A PCR reagent for inserting an N-terminal his-tag into the pET3a-pro-MMP-3cd vector |
| pET plasmid | Addgene | n/a | The pET3a vector offers ampicillin resistance, inducible expression of a target gene, and sequencing with T7 primers |
| Petri dishes | VWR | 25384-342 | Used for plating transformants on LB agar media |
| R2DP cells | Novagen | 714033 | BL21 derivatives with enhanced expression of eukaryotic proteins. Contain tRNAs of codons found to be rare in e. coli |
| SOC growth media | NEB | B9020S | Non-selective growth media for rapid growth during transformation |
| Sodium chloride (NaCl) | Thermo Fisher Scientific | BP358-1 | Used in buffers and helps with protein stability |
| Sodium deoxycholate | Thermo Fisher Scientific | PI89905 | Detergent used in protein extraction. Lyses cell walls |
| Solubilization Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 10 mM DTT, 6 M Urea. Adjust pH to 8.0 |
| Tris base | Thermo Fisher Scientific | BP152-1 | Common buffer used in the physiological pH range. Temperature-sensitive |
| Triton X-100 | Thermo Fisher Scientific | M1122980101 | Detergent used for cell lysis |
| Urea | Thermo Fisher Scientific | AAJ75826A7 | First chaotropic agent for disrupting protein secondary structure |
| Zinc chloride (ZnCl2) | Thermo Fisher Scientific | AAA162810E | Stabilizes MMP structure. The zinc ion is found in the catalytic site of MMP-3 |
Referencias
- Portolano, N., et al. Recombinant protein expression for structural biology in HEK 293F suspension cells: A novel and accessible approach. Journal of Visualized Experiments: JoVE. (92), e51897 (2014).
- Subedi, G. P., Johnson, R. W., Moniz, H. A., Moremen, K. W., Barb, A. High yield expression of recombinant human proteins with the transient transfection of HEK293 cells in suspension. Journal of Visualized Experiments: JoVE. (106), e53568 (2015).
- Nilvebrant, J., Alm, T., Hober, S. Orthogonal protein purification facilitated by a small bispecific affinity tag. Journal of Visualized Experiments: JoVE. (59), e3370 (2012).
- Yang, Z., et al. Highly efficient production of soluble proteins from insoluble inclusion bodies by a two-step-denaturing and refolding method. PLoS One. 6 (7), 22981 (2011).
- Hu, X., Beeton, C. Detection of functional matrix metalloproteinases by zymography. Journal of Visualized Experiments: JoVE. (45), e2445 (2010).
- Radisky, E. S., Raeeszadeh-Sarmazdeh, M., Radisky, D. C. Therapeutic potential of matrix metalloproteinase inhibition in breast cancer. Journal of Cellular Biochemistry. 118 (11), 3531-3548 (2017).
- Raeeszadeh-Sarmazdeh, M., Do, L. D., Hritz, B. G. Metalloproteinases and their inhibitors: Potential for the development of new therapeutics. Cells. 9 (5), 1313 (2020).
- Nagase, H., Visse, R., Murphy, G. Structure and function of matrix metalloproteinases and TIMPs. Cardiovascular Research. 69 (3), 562-573 (2006).
- Raeeszadeh-Sarmazdeh, M., et al. Directed evolution of the metalloproteinase inhibitor TIMP-1 reveals that its N- and C-terminal domains cooperate in matrix metalloproteinase recognition. Journal of Biological Chemistry. 294 (24), 9476-9488 (2019).
- Batra, J., et al. Matrix metalloproteinase-10 (MMP-10) interaction with tissue inhibitors of metalloproteinases TIMP-1 and TIMP-2. Journal of Biological Chemistry. 287 (19), 15935-15946 (2012).
- Singh, K. K., Jain, R., Ramanan, H., Saini, D. K., Galea, C. A. Expression and purification of matrix metalloproteinases in Escherichia coli. Matrix Metalloproteases. , 3-16 (2017).
- Manka, S. W., et al. Structural insights into triple-helical collagen cleavage by matrix metalloproteinase 1. Proceedings of the National Academy of Sciences of the United States of America. 109 (31), 12461-12466 (2012).
- Gomis-Ruth, F. X., et al. Mechanism of inhibition of the human matrix metalloproteinase stromelysin-1 by TIMP-1. Nature. 389 (6646), 77-81 (1997).
- Shirian, J., et al. Converting a broad matrix metalloproteinase family inhibitor into a specific inhibitor of MMP-9 and MMP-14. FEBS Letters. 592 (7), 1122-1134 (2018).
- Li, C., et al. Purification of recombinant histidine-tagged catalytic domain of MMP-13 in one step using affinity column and renaturation of it with histidine tag. Journal of Liquid Chromatography & Related Technologies. 37 (15), 2118-2130 (2014).
- Aydin, H., Azimi, F. C., Cook, J. D., Lee, J. E. A convenient and general expression platform for the production of secreted proteins from human cells. Journal of Visualized Experiments: JoVE. (65), e4041 (2012).
- McNiff, M. L., Haynes, E. P., Dixit, N., Gao, F. P., Laurence, J. S. Thioredoxin fusion construct enables high-yield production of soluble, active matrix metalloproteinase-8 (MMP-8) in Escherichia coli. Protein Expression and Purification. 122, 64-71 (2016).
- Maity, R., et al. GST-His purification: A two-step affinity purification protocol yielding full-length purified proteins. Journal of Visualized Experiments: JoVE. (80), e50320 (2013).
- Stefan, A., Ceccarelli, A., Conte, E., Montón Silva, A., Hochkoeppler, A. The multifaceted benefits of protein co-expression in Escherichia coli. Journal of Visualized Experiments: JoVE. (96), e52431 (2015).
- Yadavalli, R., Sam-Yellowe, T. HeLa based cell free expression systems for expression of Plasmodium rhoptry proteins. Journal of Visualized Experiments: JoVE. (100), e52772 (2015).
- Zeytuni, N., Zarivach, R. Purification of the M. magneticum strain AMB-1 magnetosome associated protein MamAΔ41. Journal of Visualized Experiments: JoVE. (37), e1844 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados