Method Article
Análisis automatizado de fenotipos intracelulares de salmonela usando ImageJ
En este artículo
Resumen
La salmonela invade y se replica dentro de las células epiteliales intestinales tanto en las vacuolas específicas de Salmonella como libre en el citosol (hiperreplicación). Aquí se describe un protocolo basado en microscopía de fluorescencia de alto rendimiento para cuantificar los fenotipos intracelulares de Salmonella mediante dos análisis de imágenes complementarios a través de ImageJ, alcanzando una resolución y puntuación de una sola célula.
Resumen
La salmonela es un patógeno entérico capaz de invadir el epitelio intestinal y replicarse en enterocitos, tanto dentro de las vacuolas específicas de Salmonella como libres en el citosol (hiperreplicación citosólica). Estos diferentes fenotipos de replicación intracelular conducen a diferentes vías de patogénesis, es decir, la hiperreplicación citosólica induce la muerte celular inflamatoria y la extrusión en la luz intestinal, mientras que la replicación vacuolar conduce a la penetración transepitelio y la propagación sistémica. Se hizo un esfuerzo significativo para crear herramientas de microscopía para estudiar el comportamiento de Salmonella dentro de las células invadidas, como el plásmido reportero de fluorescencia pCHAR-Duo que permite la discriminación entre bacterias vacuolares y citosólicas mediante la expresión diferencial de mCherry y GFP. Sin embargo, los fenotipos intracelulares a menudo se puntúan manualmente, un procedimiento que requiere mucho tiempo y que limita el análisis a un pequeño número de muestras y células. Para superar estas limitaciones, se desarrollaron dos análisis de imágenes complementarios y automatizados utilizando ImageJ, un software de análisis de imágenes disponible gratuitamente. En el protocolo de alto rendimiento, las células epiteliales se infectaron con Salmonella portadora de pCHAR-Duo utilizando placas de 96 pocillos. Las imágenes se realizaron utilizando un microscopio de fluorescencia automatizado. Luego, se aplicaron dos métodos de análisis de imágenes para medir el comportamiento intracelular de Salmonella en diferentes niveles de detalle. El primer método mide la carga bacteriana intracelular global y el grado de hiperreplicación citosólica. Es rápido y permite la puntuación de un gran número de células y muestras, por lo que es adecuado para ensayos de alto rendimiento, como experimentos de detección. El segundo método realiza análisis de células individuales para determinar el porcentaje de células infectadas, la carga vacuolar media de Salmonella y la tasa de hiperreplicación citosólica, lo que brinda mayores detalles sobre el comportamiento de Salmonella dentro de las células epiteliales. Los protocolos se pueden realizar mediante scripts de ImageJ diseñados específicamente para ejecutar automáticamente análisis por lotes de los pasos principales de la interacción Salmonella-enterocitos.
Introducción
La salmonela es el agente bacteriano notificado con mayor frecuencia que causa brotes de enfermedades transmitidas por los alimentos en la Unión Europea1. La principal manifestación patológica de la infección por Salmonella es la enteritis, que es el resultado del comportamiento del patógeno en el intestino después de la ingestión y la consecuente respuesta inflamatoria local2. Sin embargo, la Salmonella también puede diseminarse a sitios extraintestinales y causar infección sistémica, especialmente en individuos inmunocomprometidos. El tipo de interacción entre Salmonella y el epitelio intestinal condiciona el resultado de la infección. Una vez en la luz intestinal, la Salmonella invade y se replica dentro de las células epiteliales intestinales. A nivel intracelular, Salmonella puede presentar dos fenotipos de replicación diferentes, la hiperreplicación citosólica y la replicación lenta intravacuolar dentro de las vacuolas que contienen Salmonella (SCV). La hiperreplicación citosólica induce la muerte de la célula huésped inflamatoria y la extrusión de Salmonella en la luz intestinal3; La replicación vacuolar conduce a una penetración transepitelial y diseminación sistémica4. Por lo tanto, el grado de invasión y replicación vacuolar vs. citosólica influye en el curso de la infección.
El género Salmonella es muy diverso, incluyendo miles de serotipos con diferentes rangos de huéspedes y habilidades para causar enfermedades. Por ejemplo, S. Typhimurium se define como un serovar generalista, porque infecta múltiples huéspedes no relacionados, y representa una de las principales causas de salmonelosis humana. De manera diferente, S. Derby se considera un serovar adaptado a los cerdos, ya que está aislado principalmente de los cerdos, pero también se informa en los cinco primeros de los serovares responsables de la infección humana1. Sin embargo, el conocimiento sobre el comportamiento bacteriano dentro de las células epiteliales se limita esencialmente al estudio de unas pocas cepas de referencia, como S. Typhimurium SL1344, que no representan la vasta diversidad natural de la patogenicidad de Salmonella . La caracterización de la interacción de diferentes cepas de Salmonella con las células epiteliales contribuiría a comprender su diferente patogenicidad. Por esta razón, se desarrolló un protocolo basado en microscopía de fluorescencia de alto rendimiento para analizar el comportamiento intracelular de un gran número de cepas de una manera rápida y en gran medida automatizada. En este protocolo, la infección de las células epiteliales se realizó en placas de imágenes de 96 pocillos y la adquisición de imágenes se realizó utilizando un microscopio de fluorescencia automatizado. El plásmido pCHAR-Duo fue utilizado para observar los fenotipos de invasión y replicación de Salmonella dentro de las células epiteliales a través de microscopía fluorescente5. Este plásmido lleva el gen que codifica el reportero fluorescente rojo mCherry, expresado constitutivamente por todas las células bacterianas transformadas, y el gen que codifica el reportero fluorescente verde GFP, cuya expresión es activada por la glucosa-6-fosfato presente exclusivamente en el citosol de las células eucariotas y ausente en los SCV. Por lo tanto, el plásmido permite la discriminación entre bacterias vacuolares y citosólicas mediante la expresión diferencial de mCherry y GFP reporteros.
Las bacterias vacuolares y citosólicas en las imágenes de microscopía se cuantifican comúnmente mediante puntuación manual6, pero este es un método que requiere mucho tiempo y limita el análisis a un pequeño número de muestras. Por lo tanto, se desarrollaron dos análisis de imágenes complementarios y automatizados, el análisis de área y el análisis de una sola célula, utilizando ImageJ7, un software de análisis de imágenes disponible gratuitamente. El análisis de área mide la carga bacteriana intracelular general y el grado de hiperreplicación citosólica mediante el uso de datos de áreas ocupadas por células epiteliales, Salmonella roja y verde en cada imagen microscópica adquirida. Este método se puede aplicar a imágenes adquiridas con bajo aumento; Por lo tanto, permite puntuar un alto número de células epiteliales con pocas imágenes, acortando el tiempo de adquisición. El análisis de células individuales utiliza la segmentación celular para determinar el porcentaje de células infectadas, la carga vacuolar media y el porcentaje de células infectadas que experimentan hiperreplicación citosólica con resolución de una sola célula.
En este protocolo, todos los pasos del análisis de imágenes se describen en detalle para que se realicen manualmente, pero el mismo análisis puede ser automatizado por nuestros scripts ImageJ diseñados específicamente. Estos scripts también permiten ejecutar análisis por lotes para analizar automáticamente múltiples imágenes y así acelerar la ejecución del método.
Protocolo
1. Infección de células epiteliales con Salmonella portadora de plásmido reportero pCHAR-Duo
NOTA: Se recomienda una pipeta multicanal.
- Cubra las placas de imágenes de 96 pocillos con paredes negras y fondo de vidrio plano con colágeno justo antes de usarlas.
- Diluir ácido acético glacial (17,4 M) en agua desmineralizada estéril bajo una campana química para obtener una solución de ácido acético de 20 mM. En condiciones estériles, filtrar la solución a través de un filtro de jeringa con un tamaño de poro de 0,2 μm. Dejar la solución en una rejilla a 0-4 °C durante 5 min.
- Diluir 3 mg/ml de colágeno a 50 μg/ml en una solución de ácido acético de 20 mM preenfriada. Mezclar invirtiendo 10 veces, y luego dispensar 30 μL de solución de colágeno por pocillo (5 μg de colágeno/cm2), manteniendo la solución en una rejilla de 0-4 °C para evitar la gelificación del colágeno.
- Asegúrese de que los fondos del pozo estén completamente cubiertos con colágeno y luego deje la placa bajo flujo laminar durante 1 h a temperatura ambiente (RT).
- Retire suavemente la solución y realice tres lavados con 30 μL de solución salina tamponada con fosfato (PBS). Utilice una pipeta multicanal de 100 μL o 50 μL para evitar el desprendimiento de colágeno.
- Cultivo de células epiteliales INT407 en placas de imagen recubiertas de colágeno 20-24 h antes de la infección.
- Cultivar rutinariamente células INT407 en matraces de 25 cm2 en Medio Esencial Mínimo con 10% de suero fetal bovino (FBS), en adelante definido Medio de Cultivo (CM), suplementado con penicilina 100 U/mL y estreptomicina 100 μg/mL (Pen/Strep).
- Lavar dos veces los matraces INT407 con 5 ml de PBS y, a continuación, separar las células con 1 ml de solución de tripsina-EDTA durante 3-5 min a 37 °C en atmósfera humidificada deCO2 al 5% 20-24 h antes de la infección. Contar las células y preparar una suspensión de 3 x 105 células/ml en el CM.
- Dispensar 100 μL de suspensión celular/pocillo en placas de imagen recubiertas de colágeno para obtener una confluencia del 100%. Incubar a 37 °C en atmósfera humidificada deCO2 al 5% durante 1 h para facilitar la adhesión celular. A continuación, añadir 100 μL de CM e incubar a 37 °C en atmósfera humidificada deCO2 al 5% durante 20-24 h hasta la infección (paso 1.4).
NOTA: Para obtener imágenes de bacterias intracelulares, las cepas de Salmonella se transforman con el plásmido reportero pCHAR-Duo que amablemente fue proporcionado por la Dra. Olivia Steele-Mortimer. Las cepas probadas aquí fueron seleccionadas para representar la diversidad fenotípica cubierta por el protocolo y validar los análisis de imagen en la sección 4. Vea los resultados representativos para obtener más detalles sobre las cepas utilizadas en este estudio.
- Preparar cultivos en fase estacionaria de cepas de Salmonella que transporten el plásmido reportero pCHAR-Duo.
- Muestrear el caldo de glicerol de Salmonella con la punta de una pipeta de 10 μL e inocularlo en 1 ml de caldo Luria Bertani (10 g de triptona, 5 g de extracto de levadura y 10 g de cloruro de sodio por litro) suplementado con ampicilina 100 μg/ml.
- Incubar el inóculo estáticamente a 37 °C durante 20 h antes de la infección para alcanzar la fase estacionaria de crecimiento, correspondiente a ~1 x 109 Unidades formadoras de colonias (UFC)/mL8.
- Infectar las células epiteliales INT407 con Salmonella.
NOTA: Las infecciones se realizan por triplicado.- Lave suavemente las células INT407 con 200 μL de PBS/pocillo.
- Preparar el inóculo de Salmonella diluyendo el cultivo de bacterias durante la noche en CM para obtener el número deseado de UFC por célula epitelial, definida como la multiplicidad de infección (MOI). Aquí se utilizó MOI 100.
- Inocular 200 μL/pocillo y, a continuación, cubrir la placa con una membrana de sellado transpirable e incubar a 37 °C en atmósfera humidificada deCO2 al 5% durante 1 h. La inoculación se considera como el tiempo cero de la infección.
- Retire el inóculo y lave suavemente con 200 μL de PBS/pocillo, y luego agregue 200 μL de CM con gentamicina 100 μg/mL por pocillo. Cubra la placa con una membrana de sellado transpirable y luego incubar a 37 °C en atmósfera humidificada deCO2 al 5% durante 1 h.
- Retirar el CM con gentamicina 100 μg/ml y lavar suavemente con 200 μL de PBS/pocillo. Añadir 200 μL de CM con gentamicina 10 μg/ml por pocillo, cubrir la placa con una membrana de sellado transpirable y, a continuación, incubar a 37 °C en atmósfera humidificada deCO2 al 5% durante 8 h.
2. Fijación de la muestra y tinción de células epiteliales
NOTA: Mantenga las muestras protegidas de la exposición directa a la luz. Los volúmenes están indicados para pozos de una placa de 96 pocillos. Se requiere la optimización del volumen para diferentes placas o soportes de cultivo celular.
- A las 8 h después de la infección, retirar la CM y lavar suavemente tres veces con 200 μL/pocillo de PBS. Retire PBS invirtiendo la placa en papel absorbente; Evite aspirar.
- Fijar las monocapas infectadas con 100 μL/pocillo de paraformaldehído (PFA) al 4% en PBS durante 20 min a RT. Eliminar PFA 4% y lavar tres veces con 200 μL/pocillo de PBS. La placa se puede almacenar durante un máximo de 16-24 h a 4 °C. Continúe con el siguiente paso.
- Tinción de células epiteliales.
NOTA: La tinción de ADN DAPI (4′,6-diamidino-2-fenilindol) (paso 2.3.1) se utiliza para el análisis de área para teñir núcleos solamente, y la tinción de detección de alto contenido (HCS) (paso 2.3.2) se utiliza para el análisis de células individuales para teñir toda la célula epitelial. Se pueden usar otras tinciones celulares, pero se requiere la optimización de la adquisición y el análisis de imágenes.- Análisis de área: dispensar 100 μL/pocillo de DAPI (solución de 300 nM en PBS) e incubar 5 min a RT protegido de la luz.
- Análisis unicelular: permeabilizar con 100 μL/pocillo de tritón 0.1x en PBS durante 15 min a RT. Lavar tres veces con PBS y dispensar 100 μL/pocillo de tinción HCS diluida 1:2000 en PBS. Incubar durante 30 min a RT protegido de la luz.
- Retirar la solución de tinción y lavar tres veces con 200 μL/pocillo de PBS. Agregue 50 μL/pocillo de PBS para la adquisición automatizada de imágenes.
3. Adquisición de imágenes con un microscopio de fluorescencia automatizado
NOTA: Aquí, la baja ampliación (objetivo de 10x/0,3 NA, 1 μm/píxel) en el paso 3.1.1 para el análisis de área y la gran ampliación (objetivo de 40x/0,75 NA, 0,255 μm/píxel) en el paso 3.1.2 para el análisis de celda única se utilizan en el protocolo de adquisición. Se pueden utilizar otros aumentos, pero se requiere la optimización de la adquisición y el análisis de imágenes. Si el microscopio disponible lo permite, adquiera imágenes como regiones de mosaico (TR), un "mosaico" de campos contiguos llamados mosaicos, para registrar grandes áreas de muestra. Establezca el enfoque automático en el centro de un TR en lugar de establecer uno para cada mosaico, para reducir la exposición de la muestra durante el procedimiento de enfoque automático.
- Utilice el enfoque automático de software en el canal de tinción celular (canal azul para tinción HCS y tinción nuclear DAPI) como posición z de referencia. Adquirir imágenes en canal de tinción celular (465 nm, células o núcleos) y en canales reporteros pCHAR-Duo mCherry (610 nm, Salmonelas intracelulares) y GFP (509 nm, Salmonellae hiperreplicante citosólica).
- Análisis de área: Imagen ≥104 celdas/réplica técnica (es decir, se recomiendan al menos tres TR de cuatro baldosas/pocillo correspondientes a una réplica técnica) con un aumento de 10x.
NOTA: Un solo plano z es suficiente para obtener imágenes tanto de células que contienen vacuolar como de células que contienen Salmonellae hiperreplicante citosólica con un aumento de 10x. - Análisis de una sola celda: Imagen ≥1.000 células/réplica técnica (es decir, se recomiendan al menos dos TR de 16 baldosas/pocillo correspondientes a una réplica técnica) con un aumento de 40x. Se requieren múltiples planos z para obtener imágenes tanto de las células que contienen vacuolar como de las células que contienen Salmonella hiperreplicante citosólica. Defina la pila z óptima (se indica una pila z de 3,85 μm con un intervalo de 0,55 μm para obtener ocho planos z para obtener imágenes de células INT407 infectadas con Salmonellae hiperreplicantes citosólicas a 40x). Cada plano z se identificará en lo sucesivo como n/8, con n entre 1 (correspondiente al plano z inferior) y 8 (correspondiente al plano z superior).
- Análisis de área: Imagen ≥104 celdas/réplica técnica (es decir, se recomiendan al menos tres TR de cuatro baldosas/pocillo correspondientes a una réplica técnica) con un aumento de 10x.
- El resultado de cada adquisición es un archivo denominado Acquisition.czi que incluye imágenes de todos los campos, canales y planos z. Si se adquirieron TR, fusione los mosaicos para obtener una imagen general de cada TR utilizando un único archivo Acquisition.czi como entrada para el método Stitching.
4. Análisis de imágenes usando ImageJ
NOTA: Los pasos 4.1 y 4.2 están diseñados específicamente para el experimento y la adquisición descritos anteriormente. Otros entornos experimentales podrían requerir la optimización del análisis. Se describe el análisis de un único archivo Acquisition.czi. Para el análisis por lotes, busque el script de análisis de celda única y el script de análisis de área como archivos complementarios (archivo complementario 1 y archivo complementario 2). Las etiquetas utilizadas en los scripts y comandos de ImageJ están en negrita en las secciones siguientes.
- Análisis de área
- Abra el archivo Acquisition.czi en ImageJ, denominado AcquisitionTitle en el script: Archivo > Abra > AcquisitionTitle.czi. Se abrirá una ventana que muestra todos los canales. Los canales se indican como c:1-3/3 dependiendo de la orden de adquisición. Aquí, c:1/3 corresponde al azul (DAPI), c:2/3 a mCherry ( Salmonella intracelular) y c:3/3 a GFP ( Salmonellae hiperreplicante citosólica).
- Reduzca el ruido aleatorio para preparar las imágenes para la medición del área en los pasos 4.1.6 y 4.1.7.
- Duplique la ventana AcquisitionTitle mediante Image > Duplicate > Aceptar para obtener la ventana AcquisitionTitle1 .
- Aplique desenfoque gaussiano a AcquisitionTitle1 mediante Filtro > proceso > Desenfoque gaussiano. Deje el valor Sigma predeterminado como 2 y haga clic en Aceptar. Se abrirá una ventana de Process Stack? , haga clic en Sí para procesar todos los canales.
- Reste el AcquisitionTitle1 filtrado gaussiano del archivo original AcquisitionTitle por proceso > Calculadora de imágenes: seleccione AcquisitionTitle como Image1, elija la operación Restar en la lista desplegable y, a continuación, seleccione AcquisitionTitle1 como Image2. Haga clic en Aceptar. Se abrirá una ventana de Pila de procesos? Haga clic en Sí para procesar las tres imágenes (canales) para obtener la ventana ResultsOfAcquisitionTitle.
- Divida canales a través de Imagen > Color > Canales divididos. Cada imagen de canal ahora se muestra en una ventana separada, automáticamente nombrada por ImageJ como C-ResultsOfAcquisitionTitle. Aquí C1-ResultsOfAcquisitionTitle corresponde a DAPI, C2-ResultsOfAcquisitionTitle a mCherry (Salmonella intracelular) y C3-ResultsOfAcquisitionTitle a GFP (Salmonella hiperreplicante citosólica).
- Procese la imagen C1-ResultsOfAcquisitionTitle para medir el área ocupada por los núcleos de células epiteliales.
- Navegue hasta Proceso > Suave para homogeneizar el nucléolo que aparece como agujeros dentro del área de los núcleos.
- Umbralice la imagen C1-ResultsOfAcquisitionTitle para excluir el fondo mediante Image > Ajustar > umbral: marque Fondo oscuro y seleccione Rojo en la lista desplegable. Establezca el umbral automático Triángulo y, a continuación, utilice el control deslizante superior para establecer el valor de umbral mínimo cuando los núcleos aparezcan en rojo y el fondo aparezca en negro (Umbral = 100, aplicado en este protocolo).
NOTA: Los umbrales automáticos descritos en los pasos 4.1.4.2, 4.1.7, 4.2.3.6.1 y 4.2.4.3 se sugieren como guía para que el usuario defina manualmente el umbral de mejor ajuste para núcleos/células epiteliales y bacterias, respectivamente. El valor del umbral automático será reemplazado en el script por el umbral manual definido. El umbral manual definido se aplicará a todo el análisis por lotes.
- Elija lasmedidas de interés mediante Analizar > establecer medición: Compruebe Límite a umbral para limitar la medición a píxeles umbral. Área de verificación, correspondiente al área total ocupada por píxeles umbrales (es decir, núcleos en C1-ResultsOfAcquisitionTitle, Salmonellae intracelular en C2-ResultsOfAcquisitionTitle, Salmonellae hiperreplicante citosólica en C3- ResultsOfAcquisitionTitle). Marque Mostrar etiqueta para registrar el título y el canal de adquisición para cada imagen en la tabla de resultados.
- Mida el área ocupada por los núcleos usando Analizar > Medir. Las mediciones se registran en la tabla de resultados que se abre automáticamente.
- Proceso C2-ResultsOfAcquisitionTitle y C3-ResultsOfAcquisitionTitle para medir el área ocupada por Salmonellae intracelular general y Salmonellae hiperrreplicante citosólica, respectivamente. Siga los pasos 4.1.4-4.1.6 con algunas modificaciones: omita el paso de suavizado en el paso 4.1.4.1 y use el umbral automático de Otsu en lugar del Triángulo en el paso 4.1.4.2 como guía para establecer el valor de umbral mínimo (control deslizante superior) cuando las Salmonellae aparecen en rojo y el fondo aparece negro (Umbral = 200 sugerido).
- Guarde la tabla de resultados mediante Archivo > Guardar como > Todos los archivos.
- Abra la tabla de resultados en una hoja de cálculo para calcular las proporciones de área para cada archivo AcquisitionTitle analizado:
- Calcular la razón de infección: dividir el área ocupada por Salmonella intracelular total (aquí canal rojo, C2-) por el área ocupada por núcleos de células epiteliales (aquí DAPI, C1-).
- Calcular la relación de hiperreplicación: dividir el área ocupada por Salmonellae hiperreplicante citosólica (canal verde, C3-) por el área ocupada por Salmonellae intracelular total (canal rojo, C2-).
- Análisis unicelular
- Abra el archivo Acquisition.czi en ImageJ, denominado AcquisitionTitle en el script: File Menu > Abra > Acquisition.czi. Se abrirá una ventana que muestra las imágenes de todos los canales y todos los planos z.
- Divida los canales por Imagen > Color > Canales divididos. Los canales ahora se muestran en ventanas separadas, automáticamente nombradas por ImageJ como C-AcquisitionTitle. Aquí, la ventana C1-AcquisitionTitle corresponde a las células epiteliales (azul), la ventana C2-AcquisitionTitle a las Salmonellae intracelulares (mCherry) y la ventana C3-AcquisitionTitle al canal de Salmonellae hiperreplicante citosólico (GFP). Cada ventana C-AcquisitionTitle incluye todos los z-planes adquiridos.
- Procese el C1-AcquisitionTitle ventana, correspondiente a las células epiteliales, para segmentar las células.
- Elija la imagen del plano z que desea utilizar para la segmentación de celdas. Seleccione la ventana C1-AcquisitionTitle y use Image > Duplicate: escriba el número del plano z elegido (es decir, 1 para seleccionar z 1/8), escriba C1Zplane en el cuadro Título, desmarque Duplicate Stack y luego haga clic en OK para duplicar solo la imagen del plano z seleccionada. Se abrirá una ventana llamada C1Zplane que muestra la imagen del plano z seleccionada.
- Procese la imagen C1Zplane obtenida para mejorar el contraste de la imagen. Proceso abierto > Mejorar contraste: ajuste los píxeles saturados (es decir, aquí se usa el 1%) y marque Normalizar. Luego, haga clic en Aceptar para aplicar la técnica de mejora de contraste para obtener una imagen C1Zplane normalizada por contraste.
- Procese la imagen C1Zplane normalizada con contraste para segmentar las células epiteliales. Utilice Procesar > Buscar máximos para abrir el menú FindMaxima : primero, marque la Selección de punto de vista previa para establecer la tolerancia al ruido con el fin de atribuir solo un punto máximo a cada celda epitelial. Marcar Excluir máximos de arista. Seleccione el tipo de salida Partículas segmentadas y haga clic en Aceptar para obtener C1ZplaneSegmented, una nueva imagen binaria similar a una máscara que muestra cada partícula segmentada por punto máximo marcado.
NOTA: El algoritmo Find Maxima ImageJ se utiliza para segmentar celdas. Los puntos máximos (picos de intensidad de píxeles) se detectan en toda la imagen, potencialmente correspondientes a las celdas. Se establece un umbral de ruido (tolerancia al ruido) y se analiza el área contigua alrededor de los puntos máximos para crear una imagen binaria similar a una máscara que define cada partícula segmentada por punto máximo, por lo tanto, cada celda. - Procesar C1ZplaneImagen segmentada para crear una máscara de celdas segmentadas. Utilice Analizar > Analizar partículas: seleccione la opción Mostrar máscaras y Excluir en bordes. Ajuste el rango de área de partículas segmentadas para incluir en la máscara, correspondiente al parámetro Size , para excluir objetos segmentados erróneamente, como grupos de celdas y fracciones de celda. Para puntuar el área de objetos segmentados erróneamente, use la herramienta Seguimiento de varita en la barra de herramientas: seleccione partícula con la varita y, a continuación, abra Analizar > medida para medir el área de objetos erróneos. Establezca el intervalo de tamaño (rango sugerido 250-1700 píxeles2) y haga clic en Aceptar para obtener la máscara binaria MaskofC1ZplaneSegmented .
- De forma predeterminada, la máscara binaria MaskOfC1ZplaneSegmented tiene una LUT invertida. Usar LUT > Invertir LUT en la barra de herramientas.
- Procese la máscara binaria MaskOfC1ZplaneSegmented para corregir la segmentación de celdas:
- Imagen C1Zplane normalizada por contraste de umbral obtenida en el paso 4.2.3.2. Usar imagen > ajustar > umbral: marque Fondo oscuro. Establezca la configuración Umbral automático predeterminado . Elija la opción Rojo y ajuste el valor de corte mínimo (barra superior) hasta que las celdas aparezcan completamente rojas, dejando el fondo oscuro (Umbral = 8,000 aplicado en este protocolo). Luego, haga clic en Aplicar para convertir la imagen C1Zplane normalizada por contraste en una imagen binaria con celdas en blanco y el fondo en negro.
- Corregir la segmentación de celdas en MaskOfC1ZplaneMáscara binaria segmentada . Use la calculadora de > imágenes de proceso: seleccione MaskOfC1ZplaneSegmentado como Imagen1, elija la operación Y en la lista desplegable y, a continuación, seleccione el C1Zplane normalizado por contraste con umbral como Imagen2. Haga clic en Aceptar. La imagen de salida es nombrada automáticamente por ImageJ como Resultados de Máscara de C1Zplane Segmentado.
- Resultados del proceso de la máscara de C1Zplane segmentado para etiquetar cada celda de un solo segmento como una región de interés (ROI). Utilice Analizar > Analizar partículas. Ajuste el tamaño como en el paso 4.2.3.4 y, a continuación, seleccione la opción No mostrar nada. Marque Agregar al administrador para agregar todas las partículas (celdas segmentadas) a la herramienta ROI Manager. Además, marque Excluir en bordes. Haga clic en Aceptar. El menú del administrador de ROI mostrará una lista de todas las partículas (celdas segmentadas), definidas como ROI, etiquetadas de forma única con sus respectivas coordenadas y y x.
- Guarde los datos de ROI como ROI-cells-AcquisitionTitle.zip a través del menú ROI Manager haciendo clic en Más > Guardar. ROI-cells-AcquisitionTitle.zip se abrirá en el paso 4.2.4.6 para el análisis de celda única.
- Procese el C2-AcquisitionTitle ventana (aquí canal rojo), correspondiente a intracelular Salmonellae, para medir el número de células infectadas y la carga vacuolar intracelular.
- Reste el canal verde (C3-) del canal rojo (C2-) para eliminar los píxeles desenfocados de las Salmonellae hiperreplicantes citosólicas.
- Utilice Process > Image Calculator. Seleccione C2-Acquisitiontitle como Image1, elija la operación Restar en la lista desplegable y, a continuación, seleccione C3-Acquisitiontitle como Image2. Marque Crear nueva ventana y haga clic en Aceptar.
- Aplique resta a toda la pila z haciendo clic en Sí en la pila de procesos? ventana. La ventana de salida se denomina ResultsOfC2AcquisitionTitle en el script.
- Resultados del procesoOfC2AcquisitionTitle para reducir el ruido aleatorio.
- Duplique la ventana ResultsOfC2AcquisitionTitle haciendo clic en Menú Imagen > Duplicar. Marque Pila duplicada y presione OK para obtener la ventana ResultsOfC2AcquisitionTitle1 .
- Aplique Desenfoque gaussiano a la ventana ResultsOfC2AcquisitionTitle1 mediante Filtro > proceso > Desenfoque gaussiano. Deje el valor Sigma como 2 o personalícelo (es decir, aquí se usó Sigma: 4). Haga clic en Aceptar y aplique el desenfoque gaussiano a toda la pila z haciendo clic en ¿Sí en la ventana Pila de proceso?
- Reste el ResultsOfC2AcquisitionTitle1 filtrado gaussiano a ResultsOfC2AcquisitionTitle haciendo clic en Procesar > Calculadora de imágenes. Seleccione ResultsOfC2AcquisitionTitle como Image1, elija Operation Subrest en la lista desplegable y, a continuación, seleccione ResultsOfC2AcquisitionTitle1 como Image2. Haga clic en Aceptar. Aplique resta a toda la pila z haciendo clic en ¿Sí en la ventana Pila de procesos? para obtener una ventana nombrada automáticamente por ImageJ como Resultados de Resultados de C2-AcquisitionTitle.
- Procesar los resultados de los resultados de C2-AcquisitionTitle para separar Salmonellae del fondo. Usar imagen > ajustar > umbral: marque Fondo oscuro y establezca el umbral automático de Otsu . Ajuste el valor de corte mínimo (barra superior) hasta que las salmonelas aparezcan completamente rojas, dejando un fondo oscuro (umbral = 100 aplicado en este protocolo). Haga clic en Aplicar y se abrirá la ventana Convertir a binario : marque Fondo negro y luego haga clic en Aceptar. La pila z completa ahora se convierte en imágenes binarias.
- Seleccione el plano Z medio, correspondiente al plano de enfoque vacuolar de Salmonellae, en los resultados de la ventana binaria Resultados de C2-AcquisitionTitle del paso 4.2.4.3: Imagen > Pilas > Establecer corte y escriba el número del plano z de elección (por ejemplo, aquí se usa z: 4/8). Haga clic en Aceptar.
- Establezca las mediciones para registrar cada ROI (celda). Utilice Analizar > establecer medición: comprobar área, correspondiente al área total de cada ROI. Marque Fracción de área y Límite a umbral para registrar solo la fracción de área ocupada por píxeles umbrales (salmonelas intracelulares) para cada ROI. Marque Display Label para etiquetar cada ROI con el título de la imagen, el canal, el plano z y las coordenadas x-y en la tabla de resultados.
- Procese el plano z elegido de Salmonellae intracelular para grabar sellos, Área y % de área ocupada por Salmonellae para cada célula (ROI): abra el archivo ROI-cells-AcquisitionTitle.zip, guardado en el paso 4.2.3.8, por Archivo > Abra y haga clic en Medir en el menú del Administrador de ROI. El resultado es una tabla que informa las medidas seleccionadas.
- Guarde la tabla de resultados mediante Archivo > Guardar como en la ventana de resultados.
- Abrir ResultTable en una hoja de cálculo: etiqueta El área y el % de área ocupada por Salmonellae intracelular se indican para cada ROI, correspondiente a una celda contorneada identificada de forma única con coordenadas x e y.
- Mida el número de células infectadas correspondientes a ROI que muestran un %Área > 0 ocupada por píxeles umbral, correspondiente a Salmonella (es decir, aquí se considera un %Área > 0,2% para identificar las células infectadas). Luego, calcule el porcentaje de células infectadas en el total de células.
- Medir la carga vacuolar de Salmonella/célula calculando el %Área media ocupada por Salmonelas vacuolares para todas las células infectadas.
- Reste el canal verde (C3-) del canal rojo (C2-) para eliminar los píxeles desenfocados de las Salmonellae hiperreplicantes citosólicas.
- Proceso de canal verde (C3-AcquisitionTitle) para medir el número de células que muestran Salmonella hiperrreplicante citosólica. Siga los pasos 4.2.4.2 a 4.2.4.9, pero con algunas modificaciones.
- Trabajar en los planos z superiores (aquí z: 7/8), ya que las células que muestran Salmonellae hiperreplicantes citosólicas sobresalen de la monocapa; por lo tanto, están enfocados en un plano diferente en comparación con las células sin Salmonella hiper-replicantes.
- Mida el número de células que contienen Salmonellae hiperreplicante citosólica (verde) mediante la puntuación de células (ROI) que muestran un alto % de área ocupada por Salmonellae (por ejemplo, se considera un %Área > 20% para identificar células que contienen Salmonella hiperreplicante citosólica). Luego, calcule el porcentaje de células que contienen Salmonellae hiperreplicante citosólica sobre el total de células infectadas puntuadas en el paso 4.2.4.9.
Resultados
Infección de células epiteliales con cepas de Salmonella
Este protocolo fue desarrollado para analizar la invasión celular y la replicación citosólica (Figura 1A) vs carga vacuolar (Figura 1B) de Salmonella dentro de las células epiteliales. El protocolo fue validado mediante el uso de las tres siguientes cepas de Salmonella, S. Typhimurium SL1344 cepa de referencia (S. Tm), S. Derby ER1175 wildtype (S. Derby wt) y el mutante isogénico de S. Derby ER1175 sin gen sipA (S. Derby ΔsipA). La cepa S. Derby ER1175 se aisló de cerdos y pertenece a la colección de vigilancia IZSLER de aislados de Salmonella. Las cepas fueron seleccionadas para representar la diversidad fenotípica cubierta por el protocolo. En particular, estas cepas fueron elegidas porque ya se sabía que su comportamiento dentro de las células epiteliales difería en términos de invasión o replicación9; por lo tanto, fueron controles valiosos para probar si el protocolo permitía distinguir cuantitativamente las diferencias en los fenotipos intracelulares de Salmonella. En particular, S. Tm y S. Derby wt se incluyeron porque habíamos demostrado previamente que S. Tm tiene mayor eficiencia de invasión y replicación intracelular que S. Derby wt9 mientras que S. Derby ΔsipA se añadió como una cepa con hiperreplicación ya que el efector de virulencia SipA juega un papel crucial en el inicio de la hiperreplicación10,11. Anteriormente se informó para S. Tm que la replicación citosólica comienza 4 h después de la invasión, y luego la población citosólica se hiper-replica rápidamente para llenar la célula epitelial a las 8 h11,12. Consistentemente, la infección de 8 h de duración fue adecuada para observar y cuantificar el fenotipo de hiperreplicación citosólica también en S. Derby wt y S. Derby ΔsipA.
Análisis de área
El análisis de área en el paso 4.1 proporciona una medición de la colonización global de células epiteliales por Salmonella (razón de infección), junto con una medición de la hiperreplicación (relación de hiperreplicación). En el flujo de trabajo de análisis de área descrito en la figura 2, el ruido aleatorio se reduce y, a continuación, los canales se dividen y procesan de forma independiente. Se establece un umbral para cada canal con el fin de excluir el fondo de la medición del área. Luego, el área ocupada por píxeles umbrales se mide para cada canal. El resultado del análisis de área es una tabla que informa, para cada archivo de adquisición, la extensión de las áreas ocupadas por núcleos de células epiteliales (canal azul), Salmonellae intracelular que expresa mCherry (canal rojo) y Salmonellae hiperreplicante citosólica que expresa GFP (canal verde) junto con mCherry. La razón de infección se calcula dividiendo el área ocupada por Salmonellae que expresa mCherry por el área ocupada por los núcleos de la célula huésped. Los resultados de las cepas probadas mostraron que S. Tm muestra una relación de infección significativamente mayor en comparación con ambos S. Cepas derby, como se esperaba (Figura 3A). Por lo tanto, estos resultados demuestran la eficacia del análisis de área en la detección de diferencias en la capacidad de las cepas de Salmonella para colonizar las células epiteliales. La relación de hiperreplicación de Salmonella se mide dividiendo el área ocupada por Salmonellae que expresa GFP por el área ocupada por Salmonella intracelular que expresa mCherry. De acuerdo con el papel de SipA en la determinación de la hiper-replicación, una relación de hiper-replicación significativamente reducida para S. La cepa Derby ΔsipA se midió en comparación con S. Derby wt, (Figura 3B). No se observó diferencia de hiperreplicación entre S. Tm y S. Derby wt. En general, el análisis de área reveló efectivamente diferentes niveles de hiperreplicación entre las cepas ensayadas.
Análisis unicelular
El análisis unicelular permite cuantificar la invasión de Salmonella y la carga vacuolar frente a la replicación citosólica dentro de las células epiteliales con resolución unicelular. Como se describe en el paso 4.2, para cada archivo de adquisición, los canales azul, rojo y verde se procesaron de forma independiente (Figura 4). En concreto, el canal azul, correspondiente a las células epiteliales, se procesó en el paso 4.2.3 para obtener la segmentación celular. Luego, las celdas segmentadas se agregaron al Administrador de regiones de interés (ROI) para obtener una lista de ROI, correspondiente a todas las celdas segmentadas etiquetadas de forma única con coordenadas y-x. Para eliminar los píxeles desenfocados de Salmonellae hiper-replicantes que expresan GFP, los píxeles del canal verde se restaron de los del canal rojo. La tabla de salida del análisis de células individuales informa, para cada ROI, el área y el porcentaje del área de ROI ocupada por Salmonellae que expresa solo el reportero constitutivo mCherry o el reportero sensible al citosol GFP. El porcentaje del área de la célula ocupada por Salmonellae que solo expresa mCherry se procesó en el paso 4.2.4 para calcular el porcentaje de células infectadas y la carga vacuolar media (Figura 5A). El análisis de la carga vacuolar mostró que el S. Las cepas derby generan una carga vacuolar media significativamente menor que S. Tm (Figura 5B). Por el contrario, solo se observó una ligera y no significativa reducción del porcentaje de células infectadas en S. Cepas Derby comparadas con S. Tm (Figura 5A). El porcentaje del área de la célula ocupada por Salmonellae que expresan GFP se procesó en el paso 4.2.5 para obtener la tasa de hiperreplicación. No se observaron diferencias entre S. Tm y S. Derby wt, mientras que S. Derby ΔsipA mostró una disminución significativa de la hiperreplicación, consistente con el resultado del análisis de área (Figura 5C) y el papel de SipA en la inducción de la hiperreplicación.
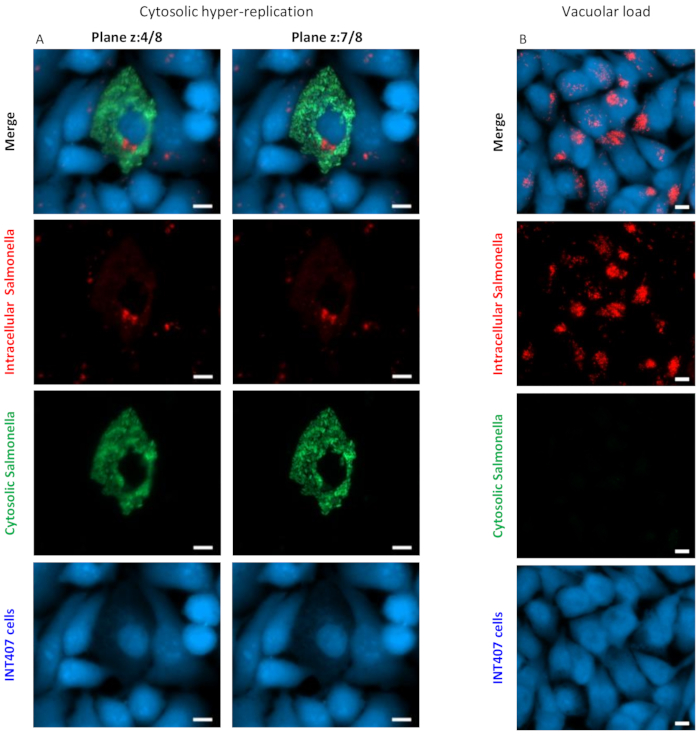
Figura 1: Hiperreplicación citosólica de Salmonella y carga vacuolar. Se muestran imágenes representativas de (A) la hiperreplicación de Salmonella y (B) la carga vacuolar adquirida a gran aumento (40x). El Panel B muestra los diferentes planos de enfoque de las células que contienen Salmonellae hiper-replicantes (z:7/8), en comparación con las Salmonellae no hiper-replicantes (z:4/8). Las barras de escala blanca son de 10 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Flujo de trabajo del análisis de área. El flujo de trabajo del análisis de área se muestra para una adquisición representativa de bajo aumento (10x) de células INT407 infectadas durante 8 h con Salmonella que transportan el plásmido pCHAR-Duo (MOI 100). Primero, se reduce el ruido aleatorio y luego los canales se dividen en C1, C2 y C3 y se procesan de forma independiente. Se establece un umbral para cada canal con el fin de excluir el fondo del área de medición. Luego, el área ocupada por los píxeles umbrales solamente, correspondientes a los núcleos celulares en C1, todas las salmonelas intracelulares en C2 y las salmonelas citosólicas en C3, se mide para cada canal. Las imágenes analizadas aquí son el resultado de cuatro baldosas adquiridas con un aumento de 10x fusionadas mediante costuras. Las barras de escala blanca son de 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
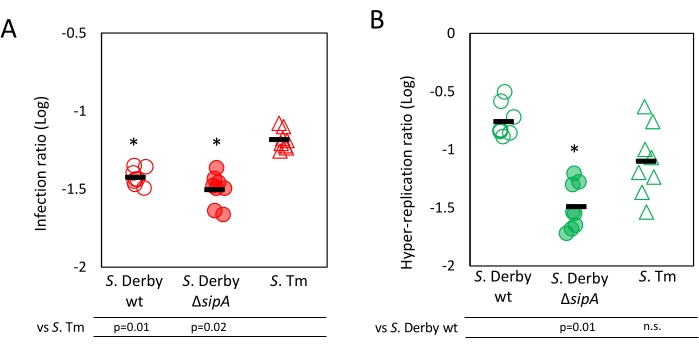
Figura 3: Resultados del análisis de área. Se muestran los resultados del análisis de área. (A) La razón de infección se calcula dividiendo el área ocupada por Salmonella que expresa mCherry (canal C2), que representa todas las bacterias intracelulares, por el área ocupada por los núcleos de la célula huésped (canal C1). (B) La relación de hiperreplicación se midió dividiendo el área ocupada por Salmonellae que expresan GFP (canal C3), que representa solo bacterias hiperreplicantes citosólicas, por el área ocupada por todas las salmonelas intracelulares que expresan mCherry. Cada punto representa una réplica. El análisis se realizó en tres réplicas biológicas, cada una probada por triplicado. Las líneas negras indican los valores medios. La significancia se calculó mediante una prueba t de dos colas, y se informan los valores de p . Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Flujo de trabajo del análisis de una sola célula. El flujo de trabajo del análisis de células individuales se muestra para una adquisición representativa de alto aumento (40x) de células INT407 infectadas durante 8 h con Salmonella que transportan el plásmido pCHAR-Duo (MOI 100). Primero, los canales se dividen en C1, C2 y C3 y se procesan de forma independiente. El canal azul (C1), correspondiente a las células epiteliales, se procesa para obtener la segmentación celular, y luego cada célula segmentada se define como una Región de Interés (ROI) etiquetada de forma única con coordenadas y-x. El canal rojo (C2) se procesa restando el canal verde (C3) para eliminar las salmonelas hiperreplicantes citosólicas desenfocadas, dejando todas las salmonelas vacuolares que expresan mCherry solamente, y luego se redujo el ruido aleatorio, y el umbral se establece para excluir el fondo de las mediciones. Finalmente, se mide el área ocupada por Salmonellae vacuolar para cada ROI. El canal verde (C3), correspondiente a las salmonelas que expresan GFP citosólicas totales, se procesa de manera similar. Las imágenes analizadas aquí son el resultado de 16 azulejos fusionados mediante costura. Las barras de escala blanca son de 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
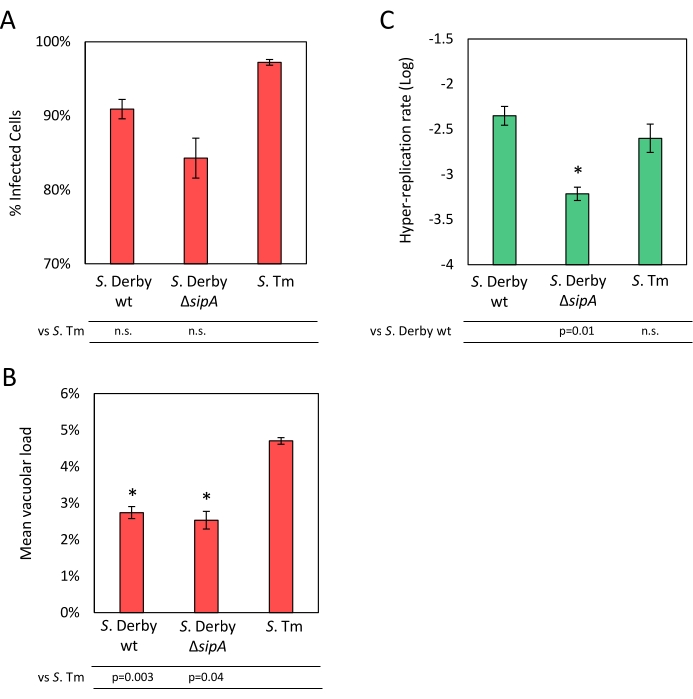
Figura 5: Resultados del análisis unicelular. Se muestran los resultados del análisis de células individuales. (A) El porcentaje de células infectadas se obtiene dividiendo el número de células con un porcentaje de área ocupada por Salmonella que solo expresa mCherry> 0.2 por el número total de células. (B) La carga vacuolar media se obtuvo calculando el área porcentual media ocupada por Salmonellae que solo expresa mCherry en las células infectadas. (C) La tasa de hiperreplicación se calculó dividiendo el número de células con un porcentaje de área ocupada por Salmonella que expresa GFP≥20% por el número total de células infectadas. Se informan datos de tres a cuatro réplicas biológicas probadas por triplicado. Las barras indican el error estándar de medición. La significancia se calculó mediante una prueba t de dos colas, y se informan los valores de p . Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: Script de análisis de área. Haga clic aquí para descargar este archivo.
Archivo complementario 2: Script de análisis de celda única. Haga clic aquí para descargar este archivo.
Discusión
La forma en que Salmonella coloniza las células epiteliales intestinales influye en el resultado de la infección. Tras la invasión, la hiperreplicación citosólica induce inflamación del intestino3, mientras que la replicación vacuolar puede conducir a diseminación sistémica4. Las cepas de Salmonella pueden variar en su capacidad para invadir y replicarse dentro de las células epitelialesintestinales 9. De hecho, Salmonella es un género extremadamente diverso que comprende más de 2.500 serovares, que tienen diferentes habilidades para causar enfermedades. Además, la Salmonella es la causa más frecuente de brotes transmitidos por los alimentos en la Unión Europea, lo que indica una gran propagación en la población humana1. Por lo tanto, la disponibilidad de métodos confiables y de alto rendimiento para la cuantificación de los fenotipos intracelulares de Salmonella es de gran importancia para evaluar las diferencias en la virulencia entre un gran número de cepas de Salmonella y, en última instancia, permitir una evaluación precisa y específica del riesgo de este patógeno.
El protocolo descrito aquí mide el comportamiento de Salmonella dentro de las células epiteliales de una manera rápida y automatizada. La infección de las células epiteliales se realiza en microplacas de imágenes de 96 pocillos y se utiliza un microscopio de fluorescencia automatizado para la adquisición de imágenes, lo que hace que el protocolo sea adecuado para aplicaciones de alto rendimiento. Este protocolo aprovecha el plásmido pCHAR-Duo, que permite distinguir Salmonellae vacuolar de citosólica a través de la expresión diferencial de dos reporteros fluorescentes5. Los fenotipos intracelulares de Salmonella a menudo se analizan mediante puntuación manual, un procedimiento que consume mucho tiempo y que no es adecuado para el análisis de un gran número de cepas y cultivos celulares por cepa y es propenso a errores del operador y variación entre operadores. Para superar estas limitaciones, se desarrollaron dos análisis de imagen complementarios y automatizados, el análisis de área y el análisis de una sola célula. ImageJ, un software de libre acceso, se utilizó para desarrollar los dos análisis. Para acelerar la ejecución del protocolo, los scripts de ImageJ para el análisis por lotes de múltiples archivos de adquisición sin intervención del operador se proporcionan como archivos complementarios.
El análisis de área fue diseñado para cuantificar, en pocos pasos, la colonización global de células epiteliales (razón de infección) y el nivel de hiperreplicación (relación de hiperreplicación) a través de la medición de las áreas específicamente ocupadas por los núcleos de células epiteliales y por Salmonellae que expresan mCherry o mCherry junto con GFP. El análisis de área se aplica a imágenes adquiridas a bajo aumento, donde tanto las Salmonellae vacuolares como las hiperrreplicantes están enfocadas en el mismo plano z, y se muestra un gran número de células epiteliales por campo microscópico, reduciendo el tamaño y el número de archivos de adquisición. El análisis de área permite un análisis automatizado, rápido y computacionalmente ligero de un gran número de muestras, lo que lo hace adecuado para ensayos de alto rendimiento, como experimentos de detección.
El análisis unicelular fue diseñado para cuantificar fenotipos de Salmonella dentro de células epiteliales con resolución unicelular, obtenidos a través de la segmentación celular y la medición del área celular y el porcentaje de área ocupada por Salmonellae hiperreplicante vacuolar y citosólica. La colonización global de células epiteliales caracterizadas a través del análisis de área se desglosa aquí en tres parámetros cuantitativos, el porcentaje de células infectadas, la carga vacuolar media y la tasa de hiperreplicación, lo que permite evaluar y cuantificar la contribución de cada fenotipo a la colonización global, complementando así los resultados del análisis de área. Los mayores detalles ofrecidos por el análisis de una sola celda tienen el costo de una adquisición y análisis de imágenes más lentos. De hecho, para lograr una resolución de una sola celda, la adquisición de imágenes se realiza con un gran aumento. Esto implica que se necesitan múltiples planos z para observar Salmonellae vacuolar y citosólica en foco y que se necesita la adquisición de un gran número de campos por muestra para puntuar un alto número de células epiteliales, extendiendo así el tiempo de adquisición en comparación con el análisis de área. Además, el análisis de varios archivos de adquisición grandes es computacionalmente exigente, por lo que requiere una estación de trabajo adecuada (se utilizó una estación de trabajo de 6 núcleos y 32 GB de RAM). Por lo tanto, el análisis de células individuales se puede utilizar como un método independiente en condiciones de rendimiento limitado o como un método de segundo nivel acoplado al análisis de área para obtener una comprensión más profunda de los fenotipos de Salmonella dentro de las células epiteliales.
Los dos análisis complementarios se validaron mediante el uso de S. Tm, S. Derby wt, y S. Derby ΔsipA, elegido porque su comportamiento dentro de las células epiteliales ya era conocido por diferir en términos de invasión o replicación 9,10. Los resultados de los análisis de área y unicelulares muestran que el protocolo permitió distinguir cuantitativamente las diferencias en los fenotipos intracelulares de Salmonella de acuerdo con las características de las cepas probadas. Además, estos resultados demuestran que el análisis unicelular permite cuantificar la contribución de cada fenotipo intracelular (invasión, carga vacuolar y replicación citosólica) a la colonización global puntuada mediante el análisis de área.
Este protocolo se aplicó aquí para estudiar la patogenicidad in vitro de diferentes cepas de Salmonella, pero puede tener otras aplicaciones como el estudio de mutantes aleatorios para identificar genes implicados en la invasión y/o replicación dentro de las células epiteliales. Además, el protocolo se puede adaptar para analizar el comportamiento de Salmonella dentro de otras líneas celulares que no sean células INT407. También se puede utilizar como punto de partida para desarrollar métodos similares para estudiar la interacción célula-patógeno de otros microorganismos intracelulares.
Divulgaciones
Los autores declaran que no tienen intereses contrapuestos.
Agradecimientos
Olivia Steele-Mortimer por compartir el plásmido pCHAR-Duo. Este trabajo fue financiado por el Ministerio de Salud italiano, subvenciones PRC2019014 y PRC2021004.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well imaging microplate | Eppendorf | 30741030 | Cell culture and infection |
| Ampicillin | Sigma-Aldrich | 59349 | Bacteria culture |
| Axio observer inverted microscope | ZEISS | Automated fluorescence microscope | |
| Axiocam 305 Mono | ZEISS | Microscope Camera | |
| Breathable sealing membrane | Sigma-Aldrich | Z380059 | Infection assay |
| Colibrì 5/7 | ZEISS | Led light source | |
| Collagen I rat tail | Life Technologies | A1048301 | Collagen coating |
| DAPI | Invitrogen | D3571 | Cell staining |
| Fetal bovine serum | Gibco | 10099-141 | Cell culture and infection |
| Gentamicin | Sigma-Aldrich | G12664 | Infection assay |
| Glacial acetic acid | Carlo Erba | 401391 | Collagen coating |
| HCS CellMask Blue | Invitrogen | H32720 | Cell staining |
| ImageJ | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation (LOCI, University of Wisconsin) | Image processing software | |
| Minimum Essential Eagle's Medium | Sigma-Aldrich | M5650-500ML | Cell culture and infection |
| Paraformaldehyde 4% | Invitrogen | FB002 | Cell fixation |
| Potassium chloride | PanReac AppliChem | 131494.1211 | PBS preparation |
| Potassium phosphate monobasic | Sigma-Aldrich | 60220 | PBS preparation |
| Sodium Chloride | PanReac AppliChem | 131659.1214 | Bacteria culture and PBS preparation |
| Sodium phosphate Dibasic | Sigma-Aldrich | 71640 | PBS preparation |
| Tissue Culture Flask 25 cm2 plug seal screw cap | Euroclone | ET7025 | Cell culture |
| Triton X-100 | Biorad | 1610407 | Cell staining |
| Trypsin-EDTA (0.25%) | Gibco | 25200056 | Cell culture and infection |
| Tryptone | Oxoid | LP0042B | Bacteria culture |
| Yeast extract | Biolife | 4122202 | Bacteria culture |
Referencias
- European Food Safety Authority & European Centre for Disease Prevention and Control. The European Union One Health 2020 Zoonoses Report. EFSA Journal. 19 (12), 6971 (2021).
- Fattinger, S. A., Sellin, M. E., Hardt, W. D. Salmonella effector driven invasion of the gut epithelium: breaking in and setting the house on fire. Current Opinion in Microbiology. 64, 9-18 (2021).
- Knodler, L. A., et al. Dissemination of invasive Salmonella via bacterial-induced extrusion of mucosal epithelia. Proceedings of the National Academy of Sciences of the United States of America. 107 (41), 17733-17738 (2010).
- Fulde, M., et al. Salmonella SPI2 T3SS mediates transcytosis in polarized enterocytes in vivo. Cell Host & Microbe. , (2019).
- Cooper, K. G., Chong, A., Starr, T., Finn, C. E., Steele-Mortimer, O. Predictable, tunable protein production in Salmonella for studying host-pathogen interactions. Frontiers in Cellular and Infection Microbiology. 7, 475 (2017).
- Klein, J. A., Grenz, J. R., Slauch, J. M., Knodler, L. A. Controlled activity of the Salmonella invasion-associated injectisome reveals its intracellular role in the cytosolic population. mBio. 8 (6), 01931 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Ibarra, J. A., et al. Induction of Salmonella pathogenicity island 1 under different growth conditions can affect Salmonella-host cell interactions in vitro. Microbiology. 156 (4), 1120-1133 (2010).
- Tambassi, M., et al. Mutation of hilD in a Salmonella Derby lineage linked to swine adaptation and reduced risk to human health. Scientific Reports. 10 (1), 21539 (2020).
- Finn, C. E., Chong, A., Cooper, K. G., Starr, T., Steele-Mortimer, O. A second wave of Salmonella T3SS1 activity prolongs the lifespan of infected epithelial cells. PLoS Pathogens. 13 (4), 1006354 (2017).
- Knodler, L. A., Nair, V., Steele-Mortimer, O. Quantitative assessment of cytosolic Salmonella in epithelial cells. PLoS One. 9 (1), 84681 (2014).
- Chong, A., Starr, T., Finn, C. E., Steele-Mortimer, O. A role for the Salmonella Type III Secretion System 1 in bacterial adaptation to the cytosol of epithelial cells. Molecular Microbiology. 112 (4), 1270-1283 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados