Method Article
Analisi automatizzata dei fenotipi intracellulari di Salmonella con ImageJ
In questo articolo
Riepilogo
La Salmonella invade e si replica all'interno delle cellule epiteliali intestinali sia nei vacuoli specifici della Salmonella che libera nel citosol (iperreplicazione). Un protocollo basato sulla microscopia a fluorescenza ad alta produttività è descritto qui per quantificare i fenotipi intracellulari di Salmonella mediante due analisi di immagini complementari attraverso ImageJ, raggiungendo la risoluzione e il punteggio di una singola cellula.
Abstract
La Salmonella è un patogeno enterico in grado di invadere l'epitelio intestinale e replicarsi negli enterociti, sia all'interno dei vacuoli specifici della Salmonella che liberi nel citosol (iperreplicazione citosolica). Questi diversi fenotipi di replicazione intracellulare guidano verso diverse vie di patogenesi, cioè l'iperreplicazione citosolica induce la morte e l'estrusione delle cellule infiammatorie nel lume intestinale, mentre la replicazione vacuolare porta alla penetrazione transepiteliale e alla diffusione sistemica. È stato fatto uno sforzo significativo per creare strumenti di microscopia per studiare il comportamento della Salmonella all'interno delle cellule invase, come il plasmide reporter di fluorescenza pCHAR-Duo che consente la discriminazione tra batteri vacuolari e citosolici mediante espressione differenziale di mCherry e GFP. Tuttavia, i fenotipi intracellulari sono spesso valutati manualmente, una procedura che richiede tempo e che limita l'analisi a un piccolo numero di campioni e cellule. Per superare questi limiti, sono state sviluppate due analisi delle immagini complementari e automatizzate utilizzando ImageJ, un software di analisi delle immagini disponibile gratuitamente. Nel protocollo ad alto rendimento, le cellule epiteliali sono state infettate da Salmonella trasportando pCHAR-Duo utilizzando piastre a 96 pozzetti. L'imaging è stato eseguito utilizzando un microscopio a fluorescenza automatizzato. Quindi, sono stati applicati due metodi di analisi delle immagini per misurare il comportamento intracellulare di Salmonella a diversi livelli di dettaglio. Il primo metodo misura la carica batterica intracellulare complessiva e l'estensione dell'iperreplicazione citosolica. È veloce e consente il punteggio di un numero elevato di cellule e campioni, rendendolo adatto per saggi ad alto rendimento come esperimenti di screening. Il secondo metodo esegue l'analisi di singole cellule per determinare la percentuale di cellule infette, il carico vacuolare medio di Salmonella e il tasso di iperreplicazione citosolica fornendo maggiori dettagli sul comportamento di Salmonella all'interno delle cellule epiteliali. I protocolli possono essere eseguiti da script ImageJ appositamente progettati per eseguire automaticamente analisi batch delle fasi principali dell'interazione Salmonella-enterociti.
Introduzione
La Salmonella è l'agente batterico più frequentemente segnalato che causa focolai di malattie di origine alimentare nell'Unione europea1. La principale manifestazione patologica dell'infezione da Salmonella è l'enterite, che è il risultato del comportamento patogeno nell'intestino dopo l'ingestione e della conseguente risposta infiammatoria locale2. Tuttavia, la Salmonella può anche diffondersi in siti extra-intestinali e causare infezioni sistemiche, specialmente in individui immunocompromessi. Il tipo di interazione tra Salmonella ed epitelio intestinale condiziona l'esito dell'infezione. Una volta nel lume intestinale, la Salmonella invade e si replica all'interno delle cellule epiteliali intestinali. A livello intracellulare, la Salmonella può presentare due diversi fenotipi di replicazione, l'iperreplicazione citosolica e la replicazione lenta intravacuolare all'interno dei vacuoli contenenti Salmonella (SCV). L'iperreplicazione citosolica induce la morte infiammatoria delle cellule ospiti e l'estrusione di Salmonella nel lume intestinale3; La replicazione vacuolare porta ad una penetrazione trans-epiteliale e alla diffusione sistemica4. Pertanto, l'estensione dell'invasione e la replicazione vacuolare rispetto a quella citosolica influenzano il decorso dell'infezione.
Il genere Salmonella è molto vario, tra cui migliaia di sierotipi con diversi intervalli di ospiti e capacità di causare malattie. Ad esempio, S. Il Typhimurium è definito come un sierotipo generalista, perché infetta più ospiti non correlati e rappresenta una delle principali cause di salmonellosi umana. Diversamente, S. Il derby è considerato un sierotipo adattato ai suini, poiché è per lo più isolato dai suini, ma è anche segnalato tra i primi cinque sierotipi responsabili dell'infezione umana1. Tuttavia, la conoscenza del comportamento batterico all'interno delle cellule epiteliali è essenzialmente limitata allo studio di alcuni ceppi di riferimento, come S. Typhimurium SL1344, che non rappresentano la vasta diversità naturale della patogenicità della Salmonella . La caratterizzazione dell'interazione di diversi ceppi di Salmonella con cellule epiteliali contribuirebbe a comprendere la loro diversa patogenicità. Per questo motivo, è stato sviluppato un protocollo basato sulla microscopia a fluorescenza ad alta produttività per analizzare il comportamento intracellulare di un gran numero di ceppi in modo rapido e ampiamente automatizzato. In questo protocollo, l'infezione delle cellule epiteliali è stata eseguita in piastre di imaging a 96 pozzetti e l'acquisizione delle immagini è stata effettuata utilizzando un microscopio a fluorescenza automatizzato. Il plasmide pCHAR-Duo è stato utilizzato per osservare i fenotipi di invasione e replicazione della Salmonella all'interno delle cellule epiteliali attraverso la microscopia fluorescente5. Questo plasmide veicola il gene che codifica per il reporter fluorescente rosso mCherry, costitutivamente espresso da tutte le cellule batteriche trasformate, e il gene codificante per il reporter fluorescente verde GFP, la cui espressione è attivata dal glucosio-6-fosfato presente esclusivamente nel citosol delle cellule eucariotiche e assente negli SCV. Pertanto, il plasmide consente la discriminazione tra batteri vacuolari e citosolici mediante l'espressione differenziale di mCherry e dei reporter GFP.
I batteri vacuolari e citosolici sulle immagini al microscopio sono comunemente quantificati dal punteggio manuale6, ma questo è un metodo che richiede tempo e limita l'analisi a un piccolo numero di campioni. Pertanto, sono state sviluppate due analisi di immagini complementari e automatizzate: l'analisi ad area e l'analisi a cella singola, utilizzando ImageJ7, un software di analisi delle immagini disponibile gratuitamente. L'analisi dell'area misura la carica batterica intracellulare complessiva e l'estensione dell'iperreplicazione citosolica utilizzando i dati delle aree occupate da cellule epiteliali, Salmonelle rosse e verdi in ciascuna immagine di microscopia acquisita. Questo metodo può essere applicato a immagini acquisite a basso ingrandimento; Pertanto, consente di segnare un numero elevato di cellule epiteliali con poche immagini, riducendo i tempi di acquisizione. L'analisi a singola cellula utilizza la segmentazione cellulare per determinare la percentuale di cellule infette, il carico vacuolare medio e la percentuale di cellule infette sottoposte a iperreplicazione citosolica con risoluzione a singola cellula.
In questo protocollo, tutte le fasi dell'analisi delle immagini sono descritte in dettaglio per essere eseguite manualmente, ma la stessa analisi può essere automatizzata dai nostri script ImageJ appositamente progettati. Questi script consentono inoltre di eseguire analisi batch per analizzare automaticamente più immagini e velocizzare così l'esecuzione del metodo.
Protocollo
1. Infezione delle cellule epiteliali con Salmonella trasportatrice del plasmide reporter pCHAR-Duo
NOTA: si consiglia una pipetta multicanale.
- Rivestire le lastre di imaging a 96 pozzetti con pareti nere e fondo di vetro piatto con collagene subito prima dell'uso.
- Diluire l'acido acetico glaciale (17,4 M) in acqua demineralizzata sterile sotto una cappa chimica per ottenere una soluzione di acido acetico di 20 mM. In condizioni sterili, filtrare la soluzione attraverso un filtro a siringa con una dimensione dei pori di 0,2 μm. Lasciare la soluzione in una griglia a 0-4 °C per 5 min.
- Diluire 3 mg/mL di collagene a 50 μg/mL in una soluzione di acido acetico 20 mM preraffreddata. Mescolare capovolgendo 10 volte, quindi erogare 30 μL di soluzione di collagene per pozzetto (5 μg di collagene/cm2), mantenendo la soluzione in una griglia a 0-4 °C per evitare gelificare il collagene.
- Assicurarsi che i fondi del pozzo siano completamente ricoperti di collagene, quindi lasciare la piastra sotto flusso laminare per 1 ora a temperatura ambiente (RT).
- Rimuovere delicatamente la soluzione ed eseguire tre lavaggi con 30 μL di soluzione salina tamponata fosfato (PBS). Utilizzare una pipetta multicanale da 100 μL o 50 μL per evitare il distacco del collagene.
- Coltura di cellule epiteliali INT407 in piastre di imaging rivestite di collagene 20-24 ore prima dell'infezione.
- Coltura abituale di cellule INT407 in palloni da 25 cm2 in Minimum Essential Medium con il 10% di siero fetale bovino (FBS), di seguito definito terreno di coltura (CM), integrato con penicillina 100 U/mL e streptomicina 100 μg/mL (Pen/Strep).
- Lavare due volte i palloni INT407 con 5 mL di PBS, quindi staccare le cellule con 1 mL di soluzione di tripsina-EDTA per 3-5 minuti a 37 °C in atmosfera umidificata al 5% di CO2 20-24 ore prima dell'infezione. Contare le cellule e preparare una sospensione 3 x 105 celle / ml nel CM.
- Erogare 100 μL di sospensione/pozzetto cellulare in piastre di imaging rivestite di collagene per ottenere il 100% di confluenza. Incubare a 37 °C in atmosfera umidificata al 5% di CO2 per 1 h per facilitare l'adesione cellulare. Quindi, aggiungere 100 μL di CM e incubare a 37 °C in atmosfera umidificata al 5% di CO2 per 20-24 ore fino all'infezione (fase 1.4).
NOTA: Per visualizzare i batteri intracellulari, i ceppi di Salmonella vengono trasformati con il plasmide reporter pCHAR-Duo che è stato gentilmente fornito dalla dottoressa Olivia Steele-Mortimer. I ceppi qui testati sono stati selezionati per rappresentare la diversità fenotipica coperta dal protocollo e convalidare le analisi delle immagini nella sezione 4. Vedere i risultati rappresentativi per maggiori dettagli sui ceppi utilizzati in questo studio.
- Preparare colture in fase stazionaria di ceppi di Salmonella portatori del plasmide reporter pCHAR-Duo.
- Prelevare il campione del brodo di glicerolo di Salmonella con la punta di una pipetta da 10 μL e inocularlo in 1 mL di brodo Luria Bertani (10 g di triptone, 5 g di estratto di lievito e 10 g di cloruro di sodio per litro) integrato con ampicillina 100 μg/ml.
- Incubare l'inoculo staticamente a 37 °C per 20 ore prima dell'infezione per raggiungere la fase stazionaria di crescita, corrispondente a ~1 x 109 Colony Forming Units (CFU)/mL8.
- Infettare le cellule epiteliali INT407 con Salmonella.
NOTA: Le infezioni vengono eseguite in triplice copia.- Lavare delicatamente le celle INT407 con 200 μL di PBS/pozzetto.
- Preparare l'inoculo di Salmonella diluendo la coltura notturna dei batteri in CM per ottenere il numero desiderato di CFU per cellula epiteliale, definita come molteplicità di infezione (MOI). Qui è stato utilizzato MOI 100.
- Inoculare 200 μL/pozzetto, quindi coprire la piastra con una membrana sigillante traspirante e incubare a 37 °C in atmosfera umidificata al 5% di CO2 per 1 ora. L'inoculazione è considerata come il tempo zero dell'infezione.
- Rimuovere l'inoculo e lavare delicatamente con 200 μL di PBS/pozzetto, quindi aggiungere 200 μL di CM con gentamicina 100 μg/mL per pozzetto. Coprire la piastra con una membrana sigillante traspirante, quindi incubare a 37 °C in atmosfera umidificata al 5% di CO2 per 1 ora.
- Rimuovere il CM con gentamicina 100 μg/mL e lavare delicatamente con 200 μL di PBS/pozzetto. Aggiungere 200 μL di CM con gentamicina 10 μg/mL per pozzetto, coprire la piastra con una membrana sigillante traspirante e quindi incubare a 37 °C in atmosfera umidificata al 5% di CO2 per 8 ore.
2. Fissazione del campione e colorazione delle cellule epiteliali
NOTA: mantenere i campioni protetti dall'esposizione diretta alla luce. I volumi sono indicati per pozzi di una piastra da 96 pozzetti. L'ottimizzazione del volume è necessaria per diverse piastre o supporti di coltura cellulare.
- A 8 ore dopo l'infezione, rimuovere CM e lavare delicatamente tre volte con 200 μL/pozzetto di PBS. Rimuovere il PBS capovolgendo la piastra su carta assorbente; Evitare di aspirare.
- Fissare i monostrati infetti con 100 μL/pozzetto di paraformaldeide (PFA) al 4% in PBS per 20 minuti a RT. Rimuovere PFA 4% e lavare tre volte con 200 μL/pozzetto di PBS. La piastra può essere conservata per un massimo di 16-24 h a 4 °C. Procedere con il passaggio successivo.
- Colorare le cellule epiteliali.
NOTA: la colorazione del DNA DAPI (4′,6-diamidino-2-fenilindolo) (fase 2.3.1) viene utilizzata solo per l'analisi dell'area per colorare i nuclei e la colorazione High Content Screening (HCS) (fase 2.3.2) viene utilizzata per l'analisi a singola cellula per colorare l'intera cellula epiteliale. È possibile utilizzare altre colorazioni cellulari, ma è necessaria l'ottimizzazione dell'acquisizione e dell'analisi delle immagini.- Analisi dell'area: erogare 100 μL/pozzetto di DAPI (soluzione 300 nM in PBS) e incubare 5 minuti a RT al riparo dalla luce.
- Analisi a singola cellula: permeabilizzare con 100 μL/pozzetto di tritone 0,1x in PBS per 15 minuti a RT. Lavare tre volte con PBS ed erogare 100 μL/pozzetto di colorazione HCS diluita 1:2000 in PBS. Incubare per 30 minuti a RT al riparo dalla luce.
- Rimuovere la soluzione colorante e lavare tre volte con 200 μL/pozzetto di PBS. Aggiungere 50 μL/pozzetto di PBS per l'acquisizione automatica delle immagini.
3. Acquisizione di immagini con un microscopio a fluorescenza automatizzato
NOTA: Qui, nel protocollo di acquisizione vengono utilizzati bassi ingrandimenti (obiettivo 10x/0,3 NA, 1 μm/pixel) nel passaggio 3.1.1 per l'analisi dell'area e un ingrandimento elevato (obiettivo 40x/0,75 NA, 0,255 μm/pixel) nel passaggio 3.1.2 per l'analisi a cella singola. È possibile utilizzare altri ingrandimenti, ma è necessaria l'ottimizzazione dell'acquisizione e dell'analisi delle immagini. Se consentito dal microscopio disponibile, acquisire immagini come Tile Regions (TR), un "mosaico" di campi contigui chiamati tessere, per registrare grandi aree campione. Impostate la messa a fuoco automatica al centro di un TR anziché impostarne una per ogni riquadro, per ridurre l'esposizione del campione durante la procedura di messa a fuoco automatica.
- Utilizzare l'autofocus software nel canale di colorazione della cella (canale blu per la colorazione HCS e la colorazione nucleare DAPI) come posizione z di riferimento. Acquisire immagini nel canale di colorazione cellulare (465 nm, cellule o nuclei) e nei canali reporter pCHAR-Duo mCherry (610 nm, Salmonelle intracellulari) e GFP (509 nm, Salmonelle iperreplicanti citosoliche).
- Analisi dell'area: Immagine ≥104 celle/replica tecnica (cioè si raccomandano almeno tre TR di quattro tessere/pozzetto corrispondenti a una replica tecnica) con ingrandimento 10x.
NOTA: Un singolo piano z è sufficiente per visualizzare sia le cellule contenenti vacuolari che le cellule contenenti Salmonelle iperreplicanti citosoliche con un ingrandimento 10x. - Analisi a cella singola: immagine ≥1.000 celle/replica tecnica (cioè almeno due TR di 16 tessere/pozzetto corrispondenti a una replica tecnica) con ingrandimento 40x. Sono necessari più piani z per visualizzare sia le cellule contenenti vacuolari che le cellule contenenti Salmonelle iperreplicanti citosoliche. Definire lo z-stack ottimale (3,85 μm z-stack con intervallo di 0,55 μm per ottenere otto piani z è indicato per visualizzare le cellule INT407 infettate da Salmonelle iperreplicanti citosoliche a 40x). Ogni piano z è qui di seguito identificato come n/8, con n compreso tra 1 (corrispondente al piano z inferiore) e 8 (corrispondente al piano z superiore).
- Analisi dell'area: Immagine ≥104 celle/replica tecnica (cioè si raccomandano almeno tre TR di quattro tessere/pozzetto corrispondenti a una replica tecnica) con ingrandimento 10x.
- L'output di ogni acquisizione è un file denominato Acquisition.czi che include immagini di tutti i campi, canali e piani z. Se sono stati acquisiti TR, unire i riquadri per ottenere un'immagine complessiva di ogni TR utilizzando un singolo file Acquisition.czi come input per il metodo Stitching.
4. Analisi delle immagini con ImageJ
NOTA: i passaggi 4.1 e 4.2 sono progettati specificamente per l'esperimento e l'acquisizione descritti sopra. Altre impostazioni sperimentali potrebbero richiedere l'ottimizzazione dell'analisi. Viene descritta l'analisi di un singolo file Acquisition.czi. Per l'analisi batch, individuare lo script di analisi a cella singola e lo script di analisi dell'area come file supplementari (file supplementare 1 e file supplementare 2). Le etichette utilizzate negli script e nei comandi ImageJ sono in grassetto nelle sezioni seguenti.
- Analisi dell'area
- Aprire il file Acquisition.czi in ImageJ, denominato AcquisitionTitle nello script: File > Open > AcquisitionTitle.czi. Si aprirà una finestra che mostra tutti i canali. I canali sono indicati come c:1-3/3 a seconda dell'ordine di acquisizione. Qui, c:1/3 corrisponde al blu (DAPI), c:2/3 a mCherry (Salmonelle intracellulari) e c:3/3 a GFP ( Salmonelle iperreplicanti citosoliche).
- Ridurre il rumore casuale per preparare le immagini per la misurazione dell'area nei punti 4.1.6 e 4.1.7.
- Duplicare la finestra AcquisitionTitle utilizzando Image > Duplicate > OK per ottenere la finestra AcquisitionTitle1 .
- Applicare la sfocatura gaussiana a AcquisitionTitle1 tramite Process > Filter > Sfocatura gaussiana. Lasciare il valore Sigma predefinito come 2 e fare clic su OK. Si aprirà una finestra Process Stack? , fare clic su Sì per elaborare tutti i canali.
- Sottrarre l'oggetto AcquisitionTitle1 filtrato gaussiana dal file originale AcquisitionTitle per processo > Image Calculator: selezionare AcquisitionTitle come Image1, scegliere l'operazione Subtract nell'elenco a discesa, quindi selezionare AcquisitionTitle1 come Image2. Fare clic su OK. Si aprirà una finestra Process Stack?. Fare clic su Sì per elaborare tutte e tre le immagini (canali) per ottenere la finestra ResultsOfAcquisitionTitle.
- Dividi i canali tramite Image > Color > Dividi canali. Ogni immagine del canale viene ora visualizzata in una finestra separata, denominata automaticamente da ImageJ come C-ResultsOfAcquisitionTitle. Qui C1-ResultsOfAcquisitionTitle corrisponde a DAPI, C2-ResultsOfAcquisitionTitle a mCherry (Salmonelle intracellulari) e C3-ResultsOfAcquisitionTitle a GFP ( Salmonelle iperreplicanti citosoliche).
- Elaborare l'immagine C1-ResultsOfAcquisitionTitle per misurare l'area occupata dai nuclei delle cellule epiteliali.
- Passare a Elabora > liscio per omogeneizzare il nucleolo che appare come fori all'interno dell'area dei nuclei.
- Sospendere l'immagine C1-ResultsOfAcquisitionTitle per escludere lo sfondo utilizzando Image > Adjust > Threshold: seleziona Sfondo scuro e seleziona Rosso nell'elenco a discesa. Impostare la soglia automatica del triangolo , quindi utilizzare il dispositivo di scorrimento superiore per impostare il valore di soglia minima quando i nuclei appaiono rossi e lo sfondo appare nero (soglia = 100, applicato in questo protocollo).
NOTA: Le autosoglie descritte nei passaggi 4.1.4.2, 4.1.7, 4.2.3.6.1 e 4.2.4.3 sono suggerite come guida per l'utente per definire manualmente la soglia più adatta per nuclei/cellule epiteliali e batteri. Il valore della soglia automatica verrà sovrascritto nello script dalla soglia manuale definita. La soglia manuale definita verrà applicata all'intera analisi batch.
- Scegliere i misuratoridi interesse utilizzando Analizza > Imposta misurazione: selezionare Limite a soglia per limitare la misurazione ai pixel con soglia. Area di controllo, corrispondente all'area totale occupata dai pixel sogliati (ad esempio, nuclei in C1-ResultsOfAcquisitionTitle, Salmonelle intracellulari in C2-ResultsOfAcquisitionTitle, Salmonelle iperreplicanti citosoliche in C3- ResultsOfAcquisitionTitle). Selezionare Visualizza etichetta per registrare il titolo e il canale di acquisizione per ogni immagine nella tabella dei risultati.
- Misurare l'area occupata dai nuclei utilizzando Analizza > Misura. Le misurazioni vengono registrate nella tabella dei risultati che si apre automaticamente.
- Elaborare C2-ResultsOfAcquisitionTitle e C3-ResultsOfAcquisitionTitle per misurare l'area occupata rispettivamente dalle Salmonelle intracellulari complessive e dalle Salmonelle iperreplicanti citosoliche. Seguire i passaggi 4.1.4-4.1.6 con alcune modifiche: saltare il passaggio di livellamento nel punto 4.1.4.1 e utilizzare la soglia automatica Otsu anziché il triangolo al punto 4.1.4.2 come guida per impostare il valore minimo di soglia (cursore superiore) quando le salmonelle appaiono rosse e lo sfondo appare nero (soglia = 200 suggerita).
- Salvare la tabella dei risultati utilizzando File > Salva con nome > Tutti i file.
- Aprire la tabella dei risultati in un foglio di calcolo per calcolare i rapporti di area per ciascun file AcquisitionTitle analizzato:
- Calcolare il rapporto di infezione: dividere l'area occupata dalle Salmonelle intracellulari totali (qui canale rosso, C2-) per l'area occupata dai nuclei delle cellule epiteliali (qui DAPI, C1-).
- Calcolare il rapporto di iperreplicazione: dividere l'area occupata dalle Salmonelle iperreplicanti citosoliche (canale verde, C3-) per l'area occupata dalle Salmonelle intracellulari totali (canale rosso, C2-).
- Analisi a cella singola
- Aprire il file Acquisition.czi in ImageJ, denominato AcquisitionTitle nello script: Menu File > Apri > Acquisition.czi. Si aprirà una finestra che mostra le immagini di tutti i canali e di tutti i piani z.
- Dividi i canali per immagine > colore > dividi canali. I canali sono ora visualizzati in finestre separate, denominate automaticamente da ImageJ come C-AcquisitionTitle. Qui, la finestra C1-AcquisitionTitle corrisponde alle cellule epiteliali (blu), la finestra C2-AcquisitionTitle alle Salmonelle intracellulari (mCherry) e la finestra C3-AcquisitionTitle al canale citosolico iperreplicante delle Salmonelle (GFP). Ogni finestra di C-AcquisitionTitle include tutti gli z-plane acquisiti.
- Elaborare il C1-AcquisitionTitle finestra, corrispondente alle cellule epiteliali, alle cellule segmentate.
- Scegliere l'immagine del piano z da utilizzare per la segmentazione delle celle. Selezionare la finestra C1-AcquisitionTitle e utilizzare Image > Duplicate: scrivere il numero del piano z scelto (cioè 1 per selezionare z 1/8), scrivere C1Zplane nella casella Titolo, deselezionare Duplicate Stack, quindi fare clic su OK per duplicare solo l'immagine del piano z selezionata. Si aprirà una finestra chiamata C1Zplane che mostra l'immagine del piano z selezionata.
- Elaborare l'immagine C1Zplane ottenuta per migliorare il contrasto dell'immagine. Apri processo > Migliora contrasto: regola i pixel saturi (cioè, qui viene utilizzato l'1%) e seleziona Normalizza. Quindi, fare clic su OK per applicare la tecnica di miglioramento del contrasto per ottenere un'immagine C1Zplane normalizzata dal contrasto.
- Elaborare l'immagine C1Zplane normalizzata con contrasto per segmentare le cellule epiteliali. Utilizzare Process > Find Maxima per aprire il menu FindMaxima : per prima cosa, selezionare Preview Point Selection per impostare Noise Tolerance in modo da attribuire un solo punto maxima ad ogni singola cellula epiteliale. Contrassegnare Escludi limite massimo spigolo. Selezionare il tipo di output Segmented Particles e fare clic su OK per ottenere C1ZplaneSegmented, una nuova immagine binaria simile a una maschera che mostra ogni particella segmentata per punto massimo contrassegnato.
NOTA: l'algoritmo Find Maxima ImageJ viene utilizzato per segmentare le celle. I punti massimi (picchi di intensità dei pixel) vengono rilevati in tutta l'immagine, potenzialmente corrispondenti alle celle. Viene impostata una soglia di rumore (tolleranza al rumore) e l'area contigua attorno ai punti massimi viene analizzata per creare un'immagine binaria simile a una maschera che definisce ogni particella segmentata per punto massimo, quindi ogni cella. - Elabora C1ZplaneSegmented image per creare una maschera di celle segmentate. Usa Analizza > Analizza particelle: seleziona l'opzione Mostra maschere ed Escludi sui bordi. Regolare l'intervallo di aree delle particelle segmentate da includere nella maschera, corrispondente al parametro Dimensione , per escludere oggetti segmentati erroneamente come cluster di celle e frazioni di celle. Per assegnare un punteggio all'area degli oggetti segmentati erroneamente, utilizzare lo strumento Ricalco bacchetta nella barra degli strumenti: selezionare particella con la bacchetta, quindi aprire Analizza > misura per misurare l'area degli oggetti errati. Impostare l'intervallo Size (intervallo suggerito 250-1700 pixel2) e cliccare su OK per ottenere la maschera binaria MaskofC1ZplaneSegmented .
- Per impostazione predefinita, la maschera binaria MaskOfC1ZplaneSegmented dispone di una LUT invertente. Utilizzare LUT > Inverti LUT nella barra degli strumenti.
- Elaborare la maschera binaria MaskOfC1ZplaneSegmented per correggere la segmentazione delle celle:
- Immagine C1Zplane normalizzata con contrasto di soglia ottenuta al punto 4.2.3.2. Usa immagine > regola > soglia: seleziona Sfondo scuro. Impostare l'impostazione Soglia automatica predefinita . Scegli l'opzione Rosso e regola il valore limite minimo (barra superiore) fino a quando le celle appaiono completamente rosse, lasciando uno sfondo scuro (Soglia = 8.000 applicato in questo protocollo). Quindi, fai clic su Applica per convertire l'immagine C1Zplane normalizzata dal contrasto in un'immagine binaria con celle in bianco e lo sfondo in nero.
- Correggere la segmentazione delle celle in MaskOfC1ZplaneSegmented binary mask. Usa Process > Image Calculator: seleziona MaskOfC1ZplaneSegmented as Image1, scegli l'operazione AND nell'elenco a discesa, quindi seleziona C1Zplane normalizzato con soglia di contrasto come Image2. Fare clic su OK. L'immagine di output viene automaticamente denominata da ImageJ come Risultati della maschera di C1Zplane segmentata.
- Risultati del processo della maschera di C1Zplane Segmentato per etichettare ogni singola cella segmentata come regione di interesse (ROI). Utilizzare Analizza > Analizza particelle. Regolare le dimensioni come al punto 4.2.3.4, quindi selezionare l'opzione Mostra nulla. Seleziona Aggiungi a Manager per aggiungere tutte le particelle (celle segmentate) allo strumento ROI Manager. Inoltre, selezionate Escludi sui bordi. Fare clic su OK. Il menu ROI Manager mostrerà un elenco di tutte le particelle (celle segmentate), definite come ROI , etichettate in modo univoco con le rispettive coordinate y e x.
- Salva i dati ROI come ROI-cells-AcquisitionTitle.zip attraverso il menu ROI Manager facendo clic su Altro > Salva. ROI-cells-AcquisitionTitle.zip verrà aperto nel passaggio 4.2.4.6 per l'analisi a cella singola.
- Elaborare il C2-AcquisitionTitle finestra (qui canale rosso), corrispondente a intracellulare Salmonellae, per misurare il numero di cellule infette e la carica vacuolare intracellulare.
- Sottrarre il canale verde (C3-) dal canale rosso (C2-) per rimuovere i pixel fuori fuoco delle Salmonelle iperreplicanti citosoliche.
- Utilizzare Process > Image Calculator. Selezionare C2-Acquisitiontitle come Image1, scegliere l'operazione Subtract nell'elenco a discesa e quindi selezionare C3-Acquisitiontitle come Image2. Selezionare Crea nuova finestra e fare clic su OK.
- Applicare la sottrazione all'intero z-stack facendo clic su Sì nello stack di processo? finestra. La finestra di output è denominata ResultsOfC2AcquisitionTitle nello script.
- Process ResultsOfC2AcquisitionTitle per ridurre il rumore casuale.
- Duplicare la finestra ResultsOfC2AcquisitionTitle facendo clic sul menu Immagine > Duplica. Selezionare Duplicate Stack e premere OK per ottenere la finestra ResultsOfC2AcquisitionTitle1 .
- Applicare la sfocatura gaussiana alla finestra ResultsOfC2AcquisitionTitle1 tramite Process > Filter > Sfocatura gaussiana. Lasciare il valore Sigma come 2 o personalizzarlo (ad esempio, qui è stato utilizzato Sigma: 4). Fate clic su OK e applicate la sfocatura gaussiana all'intero z-stack facendo clic su Sì nella finestra Stack di processi? .
- Sottrarre l'ResultsOfC2AcquisitionTitle1 filtrato gaussiano a ResultsOfC2AcquisitionTitle facendo clic su Process > Image Calculator. Selezionare ResultsOfC2AcquisitionTitle come Image1, scegliere Operation Subtract nell'elenco a discesa e quindi selezionare ResultsOfC2AcquisitionTitle1 come Image2. Fare clic su OK. Applicare la sottrazione all'intero z-stack facendo clic su Sì nella finestra Process Stack? per ottenere una finestra denominata automaticamente da ImageJ come Risultati dei risultati di C2-AcquisitionTitle.
- Elaborare i risultati dei risultati di C2-AcquisitionTitle per separare le salmonelle dallo sfondo. Usa immagine > Regola > soglia: seleziona Sfondo scuro e imposta la soglia automatica Otsu . Regolare il valore limite minimo (barra superiore) fino a quando le Salmonelle appaiono completamente rosse, lasciando sfondo scuro (soglia = 100 applicata in questo protocollo). Fare clic su Applica e si aprirà la finestra Converti in binario : selezionare Sfondo nero, quindi fare clic su OK. Lo z-stack completo viene ora convertito in immagini binarie.
- Selezionare il piano Z centrale, corrispondente al piano di messa a fuoco vacuolare di Salmonelle, nei risultati della finestra binaria Risultati di C2-AcquisitionTitle dal passaggio 4.2.4.3: Image > Stacks > Set Slice e scrivere il numero del piano z scelto (ad esempio, qui viene utilizzato z: 4/8). Fare clic su OK.
- Impostare le misurazioni da registrare per ogni ROI (cella). Usa Analizza > imposta misurazione: controlla Area, corrispondente all'area totale di ciascun ROI. Controllare Area Fraction e Limit to Threshold per registrare solo la frazione di Area occupata da pixel sospesi (Salmonelle intracellulari) per ogni ROI. Controlla su Display Label per etichettare ogni singolo ROI con il titolo dell'immagine, il canale, il piano z e le coordinate x-y nella tabella dei risultati.
- Elaborare il piano z scelto di Salmonelle intracellulari per registrare etichette, Area e %Area occupata da Salmonelle per ogni singola cellula (ROI): aprire il file ROI-cells-AcquisitionTitle.zip, salvato al passaggio 4.2.3.8, da File > Apri e fai clic su Misura nel menu ROI Manager. L'output è una tabella che riporta le misurazioni selezionate.
- Salvare la tabella dei risultati utilizzando File > Salva con nome nella finestra dei risultati.
- Apri ResultTable in un foglio di calcolo: l'etichetta Area e %Area occupata da Salmonelle intracellulari sono indicate per ogni ROI, corrispondenti a una cella sagomata identificata in modo univoco con coordinate x e y.
- Misurare il numero di cellule infette corrispondenti ai ROI mostrando una %Area > 0 occupata da pixel soglia, corrispondenti a Salmonelle (cioè, qui si considera una %Area > 0,2% per identificare le cellule infette). Quindi, calcola la percentuale di cellule infette sulle celle totali.
- Misurare la carica vacuolare di Salmonella/cellula calcolando la % media di Area occupata dalle Salmonelle vacuolari per tutte le cellule infette.
- Sottrarre il canale verde (C3-) dal canale rosso (C2-) per rimuovere i pixel fuori fuoco delle Salmonelle iperreplicanti citosoliche.
- Elaborare il canale verde (C3-AcquisitionTitle) per misurare il numero di cellule che mostrano Salmonelle iperreplicanti citosoliche. Seguire dai passaggi da 4.2.4.2 a 4.2.4.9, ma con alcune modifiche.
- Lavoro sui piani z superiori (qui z:7/8), poiché le cellule che mostrano Salmonelle iperreplicanti citosoliche sporgono dal monostrato; pertanto, sono a fuoco su un piano diverso rispetto alle cellule senza Salmonelle iperreplicanti.
- Misurare il numero di cellule contenenti Salmonelle iperreplicanti citosoliche (verde) mediante cellule di punteggio (ROI) che mostrano un'elevata %Area occupata da Salmonelle (ad esempio, si ritiene che una %Area > 20% identifichi cellule contenenti Salmonelle iperreplicanti citosoliche). Quindi, calcolare la percentuale di cellule contenenti Salmonelle iperreplicanti citosoliche sul totale delle cellule infette valutate nella fase 4.2.4.9.
Risultati
Infezione delle cellule epiteliali con ceppi di Salmonella
Questo protocollo è stato sviluppato per analizzare l'invasione cellulare e la replicazione citosolica (Figura 1A) vs carico vacuolare (Figura 1B) di Salmonella all'interno delle cellule epiteliali. Il protocollo è stato convalidato utilizzando i tre seguenti ceppi di Salmonella, S. Ceppo di riferimento Typhimurium SL1344 (S. Tm), S. Derby ER1175 wildtype (S. Derby wt) e il mutante isogeno di S. Derby ER1175 senza gene sipA (S. Derby ΔsipA). Il ceppo S. Derby ER1175 è stato isolato dai suini e appartiene alla collezione di sorveglianza IZSLER di isolati di Salmonella. I ceppi sono stati selezionati per rappresentare la diversità fenotipica oggetto del protocollo. In particolare, questi ceppi sono stati scelti perché il loro comportamento all'interno delle cellule epiteliali era già noto per differire in termini di invasione o replicazione9; pertanto, sono stati controlli preziosi per verificare se il protocollo consentisse di distinguere quantitativamente le differenze nei fenotipi intracellulari di Salmonella. In particolare, S. Tm e S. I derby sono stati inclusi perché avevamo precedentemente dimostrato che S. La Tm ha una maggiore efficienza di invasione e replicazione intracellulare rispetto a S. Derby wt9 mentre S. Derby ΔsipA è stato aggiunto come ceppo alterato da iper-replicazione poiché l'effettore di virulenza SipA svolge un ruolo cruciale nell'insorgenza dell'iperreplicazione10,11. In precedenza era stato segnalato per S. Tm che la replicazione citosolica inizia 4 ore dopo l'invasione, e poi la popolazione citosolica rapidamente iper-replica per riempire la cellula epiteliale entro 8 h11,12. Coerentemente, l'infezione lunga 8 ore è stata adatta per osservare e quantificare il fenotipo dell'iperreplicazione citosolica anche in S. Derby wt e S. Derby ΔsipA.
Analisi dell'area
L'analisi dell'area nella fase 4.1 fornisce una misurazione della colonizzazione complessiva delle cellule epiteliali da parte di Salmonella (rapporto di infezione), insieme a una misurazione dell'iper-replicazione (rapporto di iper-replicazione). Nel flusso di lavoro di analisi dell'area descritto nella Figura 2, il rumore casuale viene ridotto e quindi i canali vengono suddivisi ed elaborati in modo indipendente. Viene impostata una soglia per ciascun canale al fine di escludere lo sfondo dalla misurazione dell'area. Quindi, l'area occupata dai pixel con soglia viene misurata per ciascun canale. L'output dell'analisi di area è una tabella che riporta, per ogni file di acquisizione, l'estensione delle aree occupate dai nuclei delle cellule epiteliali (canale blu), dalle Salmonelle intracellulari che esprimono mCherry (canale rosso) e dalle Salmonelle iperreplicanti citosoliche che esprimono GFP (canale verde) insieme a mCherry. Il rapporto di infezione viene calcolato dividendo l'area occupata dalle Salmonelle che esprimono mCherry per l'area occupata dai nuclei delle cellule ospiti. I risultati dei ceppi testati hanno mostrato che S. Tm mostra un rapporto di infezione significativamente più alto rispetto a entrambe le S. Ceppi derby, come previsto (Figura 3A). Pertanto, questi risultati dimostrano l'efficacia dell'analisi dell'area nel rilevare differenze nella capacità dei ceppi di Salmonella di colonizzare le cellule epiteliali. Il rapporto di iperreplicazione di Salmonella viene misurato dividendo l'area occupata dalle Salmonelle che esprimono GFP per l'area occupata dalle Salmonelle intracellulari che esprimono mCherry. Coerentemente con il ruolo di SipA nel determinare l'iper-replicazione, un rapporto di iper-replicazione significativamente ridotto per S. Il ceppo Derby ΔsipA è stato misurato rispetto a S. Derby wt, (Figura 3B). Non è stata osservata alcuna differenza di iperreplicazione tra S. Tm e S. Derby wt. Nel complesso, l'analisi dell'area ha rivelato efficacemente diversi livelli di iper-replicazione tra i ceppi analizzati.
Analisi a cella singola
L'analisi a singola cellula consente di quantificare l'invasione di Salmonella e il carico vacuolare rispetto alla replicazione citosolica all'interno delle cellule epiteliali con risoluzione a singola cellula. Come descritto nel passaggio 4.2, per ogni file di acquisizione, i canali blu, rosso e verde sono stati elaborati in modo indipendente (Figura 4). In particolare, il canale blu, corrispondente alle cellule epiteliali, è stato elaborato nella fase 4.2.3 per ottenere la segmentazione cellulare. Quindi, le celle segmentate sono state aggiunte al gestore della regione di interesse (ROI) per ottenere un elenco di ROI, corrispondenti a tutte le celle segmentate etichettate in modo univoco con coordinate y-x. Al fine di rimuovere i pixel fuori fuoco delle Salmonelle iperreplicanti che esprimono GFP, i pixel del canale verde sono stati sottratti da quelli del canale rosso. La tabella di output dell'analisi a singola cellula riporta, per ciascun ROI, l'area e la percentuale dell'area del ROI occupata dalle Salmonelle che esprimono solo il reporter costitutivo mCherry o il reporter GFP cytosol-responsive. La percentuale dell'area della cellula occupata da Salmonelle che esprimono solo mCherry è stata elaborata nella fase 4.2.4 per calcolare la percentuale di cellule infette e il carico vacuolare medio (Figura 5A). L'analisi della carica vacuolare ha mostrato che il S. I ceppi derby generano un carico vacuolare medio significativamente inferiore a S. Tm (Figura 5B). Al contrario, solo una leggera e non significativa riduzione della percentuale di cellule infette è stata osservata in S. Ceppi Derby rispetto a S. Tm (Figura 5A). La percentuale dell'area della cellula occupata da Salmonelle che esprimono GFP è stata elaborata nella fase 4.2.5 per ottenere il tasso di iperreplicazione. Non è stata osservata alcuna differenza tra S. Tm e S. Derby wt, mentre S. Derby ΔsipA ha mostrato una significativa diminuzione dell'iperreplicazione, coerente con il risultato dell'analisi dell'area (Figura 5C) e il ruolo di SipA nell'indurre iper-replicazione.
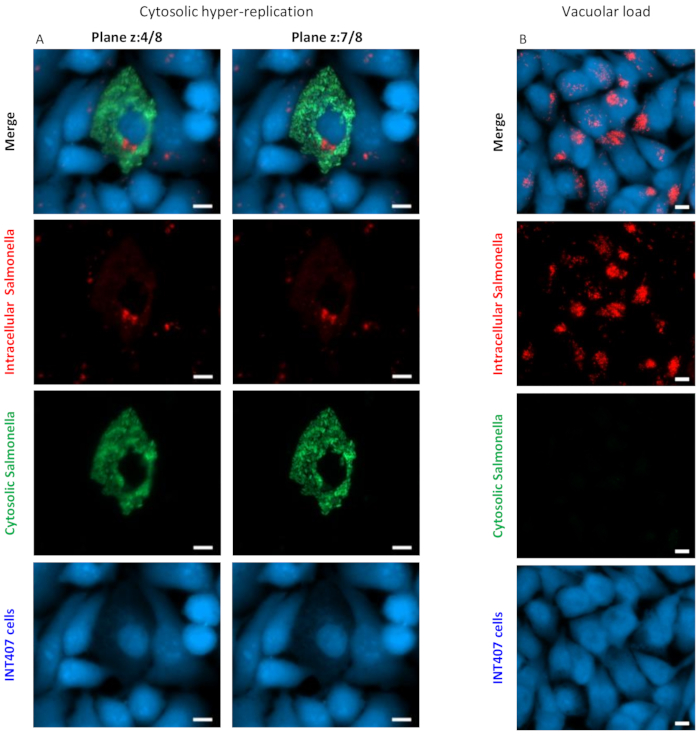
Figura 1: Iperreplicazione citosolica di Salmonella e carico vacuolare. Vengono mostrate immagini rappresentative di (A) l'iperreplicazione di Salmonella e (B) la carica vacuolare acquisita ad alto ingrandimento (40x). Il pannello B mostra i diversi piani di messa a fuoco delle cellule contenenti Salmonelle iperreplicanti (z:7/8), rispetto alle Salmonelle non iperreplicanti (z:4/8). Le barre della scala bianca sono 10 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Flusso di lavoro dell'analisi dell'area. Il flusso di lavoro dell'analisi dell'area è mostrato per un'acquisizione rappresentativa a basso ingrandimento (10x) di cellule INT407 infettate per 8 ore con Salmonella che trasporta il plasmide pCHAR-Duo (MOI 100). In primo luogo, il rumore casuale viene ridotto, quindi i canali vengono suddivisi in C1, C2 e C3 ed elaborati in modo indipendente. Per ogni canale viene impostata una soglia al fine di escludere lo sfondo dall'area di misurazione. Quindi, l'area occupata solo dai pixel sogliati - corrispondenti ai nuclei cellulari in C1, tutte le Salmonelle intracellulari in C2 e le Salmonelle citosoliche in C3 - viene misurata per ciascun canale. Le immagini qui analizzate sono i risultati di quattro tessere acquisite a ingrandimento 10x fuse insieme mediante cucitura. Le barre della scala bianca sono 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
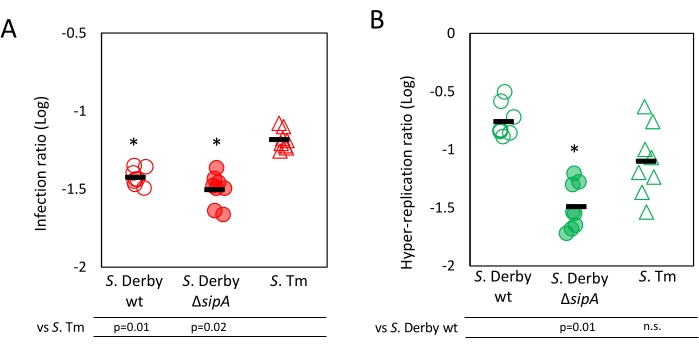
Figura 3: Risultati dell'analisi dell'area. Vengono mostrati i risultati dell'analisi dell'area. (A) Il rapporto di infezione è calcolato dividendo l'area occupata dalle Salmonelle che esprimono mCherry (canale C2), che rappresenta tutti i batteri intracellulari, per l'area occupata dai nuclei delle cellule ospiti (canale C1). (B) Il rapporto di iperreplicazione è stato misurato dividendo l'area occupata dalle Salmonelle che esprimono GFP (canale C3), che rappresentano solo i batteri iperreplicanti citosolici, per l'area occupata da tutte le Salmonelle intracellulari che esprimono mCherry. Ogni punto rappresenta una replica. L'analisi è stata condotta su tre repliche biologiche, ciascuna testata in triplice copia. Le linee nere indicano i valori medi. La significatività è stata calcolata utilizzando un test t a due code e sono riportati i valori p. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 4: Flusso di lavoro dell'analisi a cella singola. Il flusso di lavoro dell'analisi a singola cellula è mostrato per un'acquisizione rappresentativa ad alto ingrandimento (40x) di cellule INT407 infettate per 8 ore con Salmonella che trasporta il plasmide pCHAR-Duo (MOI 100). Innanzitutto, i canali vengono suddivisi in C1, C2 e C3 ed elaborati in modo indipendente. Il canale blu (C1), corrispondente alle cellule epiteliali, viene elaborato per ottenere la segmentazione cellulare, quindi ogni cellula segmentata viene definita come una regione di interesse (ROI) etichettata in modo univoco con coordinate y-x. Il canale rosso (C2) viene elaborato sottraendo il canale verde (C3) per rimuovere le Salmonelle iperreplicanti citosoliche fuori fuoco, lasciando tutte le Salmonelle vacuolari che esprimono solo mCherry, e quindi il rumore casuale è stato ridotto e la soglia è impostata per escludere lo sfondo dalle misurazioni. Infine, viene misurata l'area occupata da Salmonelle vacuolari per ogni ROI. Il canale verde (C3), corrispondente alle Salmonelle citosoliche che esprimono GFP totali, viene elaborato in modo simile. Le immagini qui analizzate sono i risultati di 16 piastrelle fuse insieme da cuciture. Le barre della scala bianca sono 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
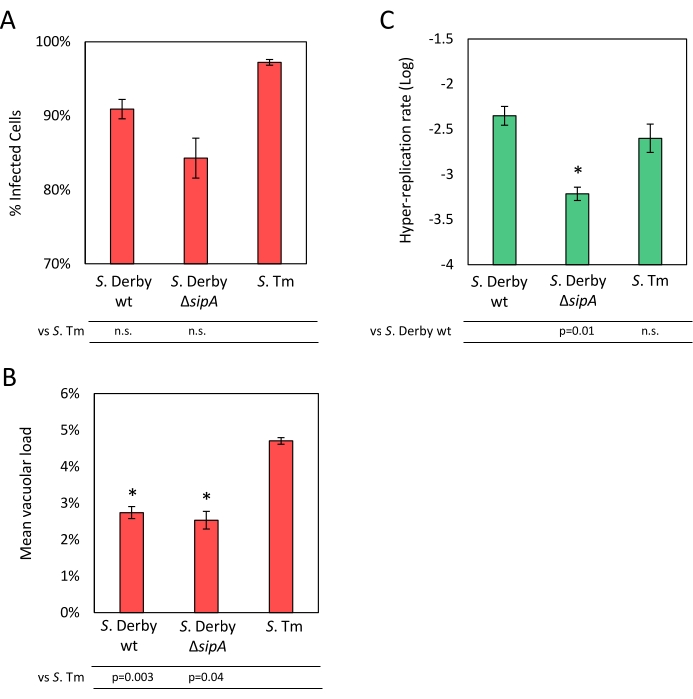
Figura 5: Risultati dell'analisi a cella singola. Vengono mostrati i risultati dell'analisi a cella singola. (A) La percentuale di cellule infette si ottiene dividendo il numero di cellule con una percentuale di superficie occupata da Salmonelle che esprimono solo ciliegie> 0,2 per il numero totale di cellule. (B) Il carico vacuolare medio è stato ottenuto calcolando l'area percentuale media occupata dalle salmonelle che esprimono solo ciliegie nelle cellule infette. (C) Il tasso di iperreplicazione è stato calcolato dividendo il numero di cellule con una percentuale di area occupata da Salmonelle che esprimono GFP≥20% per il numero totale di cellule infette. Sono riportati dati da tre a quattro repliche biologiche testate in triplice copia. Le barre indicano l'errore standard di misurazione. La significatività è stata calcolata utilizzando un test t a due code e sono riportati i valori p . Fare clic qui per visualizzare una versione ingrandita di questa figura.
File supplementare 1: script di analisi dell'area. Clicca qui per scaricare questo file.
File supplementare 2: script di analisi a cella singola. Clicca qui per scaricare questo file.
Discussione
Il modo in cui la Salmonella colonizza le cellule epiteliali intestinali influenza l'esito dell'infezione. Dopo l'invasione, l'iperreplicazione citosolica induce l'infiammazione dell'intestino3, mentre la replicazione vacuolare può portare alla diffusione sistemica4. I ceppi di Salmonella possono variare nella loro capacità di invadere e replicarsi all'interno delle cellule epiteliali intestinali9. In effetti, Salmonella è un genere estremamente diversificato che comprende più di 2.500 sierotipi, che hanno diverse capacità di causare malattie. Inoltre, la Salmonella è la causa più frequentemente segnalata di focolai di tossinfezione alimentare nell'Unione europea, indicando una grande diffusione nella popolazione umana1. Pertanto, la disponibilità di metodi affidabili e ad alto rendimento per la quantificazione dei fenotipi intracellulari di Salmonella è di grande importanza per valutare le differenze di virulenza tra un gran numero di ceppi di Salmonella e, in ultima analisi, consentire una valutazione accurata e specifica del rischio di questo patogeno.
Il protocollo qui descritto misura il comportamento della Salmonella all'interno delle cellule epiteliali in modo rapido e automatizzato. L'infezione delle cellule epiteliali viene eseguita in micropiastre di imaging a 96 pozzetti e un microscopio a fluorescenza automatizzato viene utilizzato per l'acquisizione delle immagini, rendendo il protocollo adatto per applicazioni ad alto rendimento. Questo protocollo sfrutta il plasmide pCHAR-Duo, che permette di distinguere le Salmonelle vacuolari da quelle citosoliche attraverso l'espressione differenziale di due reporter fluorescenti5. I fenotipi intracellulari di Salmonella sono spesso analizzati mediante punteggio manuale, una procedura dispendiosa in termini di tempo che non è adatta per l'analisi di un gran numero di ceppi e colture cellulari per ceppo e soggetta a errori dell'operatore e variazioni tra operatori. Per superare questi limiti, sono state sviluppate due analisi di immagini complementari e automatizzate, l'analisi dell'area e l'analisi a cella singola. ImageJ, un software liberamente disponibile, è stato utilizzato per sviluppare le due analisi. Al fine di accelerare l'esecuzione del protocollo, gli script ImageJ per l'analisi batch di più file di acquisizione senza intervento da parte dell'operatore vengono forniti come file supplementari.
L'analisi dell'area è stata progettata per quantificare, in pochi passaggi, la colonizzazione complessiva delle cellule epiteliali (infection ratio) e il livello di iper-replicazione (hyper-replication ratio) attraverso la misurazione delle aree specificamente occupate dai nuclei delle cellule epiteliali e dalle Salmonelle che esprimono mCherry o mCherry insieme a GFP. L'analisi dell'area viene applicata alle immagini acquisite a basso ingrandimento, dove sia le Salmonelle vacuolari che quelle iperreplicanti sono a fuoco nello stesso piano z e un gran numero di cellule epiteliali viene visualizzato per campo microscopico, riducendo le dimensioni e il numero di file di acquisizione. L'analisi dell'area consente un'analisi automatizzata, veloce e computazionalmente leggera di un gran numero di campioni, rendendola adatta per saggi ad alto rendimento come esperimenti di screening.
L'analisi unicellulare è stata progettata per quantificare i fenotipi di Salmonella all'interno di cellule epiteliali con risoluzione monocellulare, ottenuta attraverso la segmentazione cellulare e la misurazione dell'area cellulare e della percentuale di area occupata da Salmonelle iperreplicanti vacuolari e citosoliche. La colonizzazione complessiva delle cellule epiteliali caratterizzata attraverso l'analisi dell'area è qui suddivisa in tre parametri quantitativi, la percentuale di cellule infette, il carico vacuolare medio e il tasso di iperreplicazione, consentendo di valutare e quantificare il contributo di ciascun fenotipo alla colonizzazione complessiva, completando quindi i risultati dell'analisi dell'area. I maggiori dettagli offerti dall'analisi a cella singola vanno a scapito di un'acquisizione e di un'analisi delle immagini più lente. Infatti, al fine di ottenere una risoluzione a cella singola, l'acquisizione delle immagini viene eseguita ad alto ingrandimento. Ciò implica che sono necessari più piani z per osservare le salmonelle vacuolari e citosoliche a fuoco e che l'acquisizione di un gran numero di campi per campione è necessaria per segnare un numero elevato di cellule epiteliali, estendendo così il tempo di acquisizione rispetto all'analisi dell'area. Inoltre, l'analisi di diversi file di acquisizione di grandi dimensioni è impegnativa dal punto di vista computazionale, richiedendo quindi una workstation adatta (è stata utilizzata una workstation a 6 core e 32 GB di RAM). Pertanto, l'analisi a singola cellula può essere utilizzata come stand-alone in condizioni di produttività limitata o come metodo di secondo livello accoppiato all'analisi dell'area per ottenere una comprensione più profonda dei fenotipi di Salmonella all'interno delle cellule epiteliali.
Le due analisi complementari sono state convalidate utilizzando S. Tm, S. Derby wt, e S. Derby ΔsipA, scelto perché il loro comportamento all'interno delle cellule epiteliali era già noto per differire in termini di invasione o replicazione 9,10. I risultati delle analisi di area e di singole cellule mostrano che il protocollo ha permesso di distinguere quantitativamente le differenze nei fenotipi intracellulari di Salmonella in base alle caratteristiche dei ceppi testati. Inoltre, questi risultati dimostrano che l'analisi a singola cellula consente di quantificare il contributo di ciascun fenotipo intracellulare (invasione, carico vacuolare e replicazione citosolica) alla colonizzazione complessiva valutata utilizzando l'analisi dell'area.
Questo protocollo è stato applicato qui per studiare la patogenicità in vitro di diversi ceppi di Salmonella, ma può avere altre applicazioni come lo studio di mutanti casuali per identificare geni coinvolti nell'invasione e / o nella replicazione all'interno delle cellule epiteliali. Inoltre, il protocollo può essere adattato per analizzare il comportamento della Salmonella all'interno di altre linee cellulari rispetto alle cellule INT407. Può anche essere usato come punto di partenza per sviluppare metodi simili per studiare l'interazione cellula-patogeno di altri microrganismi intracellulari.
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti.
Riconoscimenti
Gli autori desiderano ringraziare la dottoressa Olivia Steele-Mortimer per aver condiviso il plasmide pCHAR-Duo. Questo lavoro è stato finanziato dal Ministero della Salute italiano, sovvenzioni PRC2019014 e PRC2021004.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well imaging microplate | Eppendorf | 30741030 | Cell culture and infection |
| Ampicillin | Sigma-Aldrich | 59349 | Bacteria culture |
| Axio observer inverted microscope | ZEISS | Automated fluorescence microscope | |
| Axiocam 305 Mono | ZEISS | Microscope Camera | |
| Breathable sealing membrane | Sigma-Aldrich | Z380059 | Infection assay |
| Colibrì 5/7 | ZEISS | Led light source | |
| Collagen I rat tail | Life Technologies | A1048301 | Collagen coating |
| DAPI | Invitrogen | D3571 | Cell staining |
| Fetal bovine serum | Gibco | 10099-141 | Cell culture and infection |
| Gentamicin | Sigma-Aldrich | G12664 | Infection assay |
| Glacial acetic acid | Carlo Erba | 401391 | Collagen coating |
| HCS CellMask Blue | Invitrogen | H32720 | Cell staining |
| ImageJ | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation (LOCI, University of Wisconsin) | Image processing software | |
| Minimum Essential Eagle's Medium | Sigma-Aldrich | M5650-500ML | Cell culture and infection |
| Paraformaldehyde 4% | Invitrogen | FB002 | Cell fixation |
| Potassium chloride | PanReac AppliChem | 131494.1211 | PBS preparation |
| Potassium phosphate monobasic | Sigma-Aldrich | 60220 | PBS preparation |
| Sodium Chloride | PanReac AppliChem | 131659.1214 | Bacteria culture and PBS preparation |
| Sodium phosphate Dibasic | Sigma-Aldrich | 71640 | PBS preparation |
| Tissue Culture Flask 25 cm2 plug seal screw cap | Euroclone | ET7025 | Cell culture |
| Triton X-100 | Biorad | 1610407 | Cell staining |
| Trypsin-EDTA (0.25%) | Gibco | 25200056 | Cell culture and infection |
| Tryptone | Oxoid | LP0042B | Bacteria culture |
| Yeast extract | Biolife | 4122202 | Bacteria culture |
Riferimenti
- European Food Safety Authority & European Centre for Disease Prevention and Control. The European Union One Health 2020 Zoonoses Report. EFSA Journal. 19 (12), 6971 (2021).
- Fattinger, S. A., Sellin, M. E., Hardt, W. D. Salmonella effector driven invasion of the gut epithelium: breaking in and setting the house on fire. Current Opinion in Microbiology. 64, 9-18 (2021).
- Knodler, L. A., et al. Dissemination of invasive Salmonella via bacterial-induced extrusion of mucosal epithelia. Proceedings of the National Academy of Sciences of the United States of America. 107 (41), 17733-17738 (2010).
- Fulde, M., et al. Salmonella SPI2 T3SS mediates transcytosis in polarized enterocytes in vivo. Cell Host & Microbe. , (2019).
- Cooper, K. G., Chong, A., Starr, T., Finn, C. E., Steele-Mortimer, O. Predictable, tunable protein production in Salmonella for studying host-pathogen interactions. Frontiers in Cellular and Infection Microbiology. 7, 475 (2017).
- Klein, J. A., Grenz, J. R., Slauch, J. M., Knodler, L. A. Controlled activity of the Salmonella invasion-associated injectisome reveals its intracellular role in the cytosolic population. mBio. 8 (6), 01931 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Ibarra, J. A., et al. Induction of Salmonella pathogenicity island 1 under different growth conditions can affect Salmonella-host cell interactions in vitro. Microbiology. 156 (4), 1120-1133 (2010).
- Tambassi, M., et al. Mutation of hilD in a Salmonella Derby lineage linked to swine adaptation and reduced risk to human health. Scientific Reports. 10 (1), 21539 (2020).
- Finn, C. E., Chong, A., Cooper, K. G., Starr, T., Steele-Mortimer, O. A second wave of Salmonella T3SS1 activity prolongs the lifespan of infected epithelial cells. PLoS Pathogens. 13 (4), 1006354 (2017).
- Knodler, L. A., Nair, V., Steele-Mortimer, O. Quantitative assessment of cytosolic Salmonella in epithelial cells. PLoS One. 9 (1), 84681 (2014).
- Chong, A., Starr, T., Finn, C. E., Steele-Mortimer, O. A role for the Salmonella Type III Secretion System 1 in bacterial adaptation to the cytosol of epithelial cells. Molecular Microbiology. 112 (4), 1270-1283 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon