Method Article
Análise Automatizada de Fenótipos Intracelulares de Salmonella utilizando ImageJ
Neste Artigo
Resumo
A salmonela invade e se replica no interior das células epiteliais intestinais tanto em vacúolos específicos de Salmonella quanto livre no citosol (hiper-replicação). Um protocolo baseado em microscopia de fluorescência de alto rendimento é descrito aqui para quantificar os fenótipos intracelulares de Salmonella por meio de duas análises de imagem complementares através do ImageJ, alcançando resolução e pontuação de célula única.
Resumo
A salmonela é um patógeno entérico capaz de invadir o epitélio intestinal e se replicar em enterócitos, tanto dentro de vacúolos específicos de Salmonella quanto livre no citosol (hiperreplicação citosólica). Esses diferentes fenótipos de replicação intracelular conduzem a diferentes vias de patogênese, ou seja, a hiperreplicação citosólica induz a morte celular inflamatória e a extrusão para o lúmen intestinal, enquanto a replicação vacuolar leva à penetração transepitélio e à disseminação sistêmica. Esforços significativos foram feitos para criar ferramentas de microscopia para estudar o comportamento de Salmonella dentro de células invadidas, como o plasmídeo repórter de fluorescência pCHAR-Duo que permite a discriminação entre bactérias vacuolares e citosólicas pela expressão diferencial de mCherry e GFP. No entanto, os fenótipos intracelulares são frequentemente pontuados manualmente, um procedimento demorado que limita a análise a um pequeno número de amostras e células. Para superar essas limitações, duas análises de imagem complementares e automatizadas foram desenvolvidas usando o ImageJ, um software de análise de imagens disponível gratuitamente. No protocolo de alto rendimento, células epiteliais foram infectadas com Salmonella carregando pCHAR-Duo usando placas de 96 poços. A imagem foi realizada em microscópio automatizado de fluorescência. Em seguida, dois métodos de análise de imagem foram aplicados para medir o comportamento intracelular de Salmonella em diferentes níveis de detalhe. O primeiro método mede a carga bacteriana intracelular global e a extensão da hiperreplicação citosólica. É rápido e permite a pontuação de um alto número de células e amostras, tornando-o adequado para ensaios de alto rendimento, como experimentos de triagem. O segundo método realiza análise de célula única para determinar a porcentagem de células infectadas, a carga vacuolar média de Salmonella e a taxa de hiperreplicação citosólica , fornecendo maiores detalhes sobre o comportamento da Salmonella dentro das células epiteliais. Os protocolos podem ser executados por scripts ImageJ projetados especificamente para executar automaticamente análises em lote das principais etapas da interação Salmonella-enterócito.
Introdução
A salmonela é o agente bacteriano mais frequentemente notificado que provoca surtos de doenças transmitidas por alimentos na União Europeia1. A principal manifestação patológica da infecção por Salmonella é a enterite, que é o resultado do comportamento do patógeno no intestino após a ingestão e a consequente resposta inflamatória local2. No entanto, a Salmonella também pode se disseminar para locais extraintestinais e causar infecção sistêmica, especialmente em indivíduos imunocomprometidos. O tipo de interação entre Salmonella e o epitélio intestinal condiciona o resultado da infecção. Uma vez no lúmen intestinal, a Salmonella invade e se replica dentro das células epiteliais intestinais. No nível intracelular, a Salmonella pode apresentar dois fenótipos de replicação diferentes, a hiperreplicação citosólica e a replicação lenta intravacuolar dentro de vacúolos contendo Salmonella (SCVs). A hiperreplicação citosólica induz a morte inflamatória das células hospedeiras e a extrusão de Salmonella para o lúmen intestinal3; a replicação vacuolar leva a uma penetração trans-epitélio e disseminação sistêmica4. Portanto, a extensão da invasão e da replicação vacuolar versus citosólica influencia o curso da infecção.
O gênero Salmonella é muito diversificado, incluindo milhares de sorotipos com diferentes faixas de hospedeiros e habilidades para causar doenças. Por exemplo, S. O typhimurium é definido como um sorovar generalista, porque infecta vários hospedeiros não relacionados e representa uma das principais causas de salmonelose humana. Diferentemente, S. O derby é considerado um sorovar adaptado aos suínos, pois é isolado principalmente dos suínos, mas também é relatado nos cinco principais dos sorovares responsáveis pela infecção humana1. No entanto, o conhecimento sobre o comportamento bacteriano no interior das células epiteliais é essencialmente limitado ao estudo de algumas cepas de referência, como S. Typhimurium SL1344, que não representam a vasta diversidade natural da patogenicidade da Salmonella . Caracterizar a interação de diferentes cepas de Salmonella com células epiteliais contribuiria para a compreensão de suas diferentes patogenicidades. Por esta razão, um protocolo baseado em microscopia de fluorescência de alto rendimento foi desenvolvido para analisar o comportamento intracelular de um grande número de cepas de forma rápida e amplamente automatizada. Neste protocolo, a infecção de células epiteliais foi realizada em placas de imagem de 96 poços e a aquisição de imagens foi feita usando um microscópio de fluorescência automatizado. O plasmídeo pCHAR-Duo foi utilizado para observar os fenótipos de invasão e replicação de Salmonella no interior das células epiteliais através de microscopia fluorescente5. Este plasmídeo carrega o gene que codifica o repórter fluorescente vermelho mCherry, expresso constitutivamente por todas as células bacterianas transformadas, e o gene que codifica o repórter fluorescente verde GFP, cuja expressão é ativada pela glicose-6-fosfato presente exclusivamente no citosol das células eucarióticas e ausente nos SCVs. Portanto, o plasmídeo permite a discriminação entre bactérias vacuolares e citosólicas pela expressão diferencial de repórteres mCherry e GFP.
As bactérias vacuolares e citosólicas nas imagens microscópicas são comumente quantificadas pela pontuação manual6, mas este é um método demorado que limita a análise a um pequeno número de amostras. Portanto, duas análises de imagem complementares e automatizadas foram desenvolvidas - análise de área e análise de célula única - usando o ImageJ7, um software de análise de imagem disponível gratuitamente. A análise de área mede a carga bacteriana intracelular global e a extensão da hiperreplicação citosólica usando dados de áreas ocupadas por células epiteliais, salmonelas vermelhas e verdes em cada imagem de microscopia adquirida. Este método pode ser aplicado a imagens adquiridas em baixa ampliação; portanto, permite pontuar um elevado número de células epiteliais com poucas imagens, encurtando o tempo de aquisição. A análise de célula única usa segmentação celular para determinar a porcentagem de células infectadas, a carga vacuolar média e a porcentagem de células infectadas submetidas à hiperreplicação citosólica com resolução de célula única.
Neste protocolo, todas as etapas da análise de imagem são descritas em detalhes para serem realizadas manualmente, mas a mesma análise pode ser automatizada por nossos scripts ImageJ projetados especificamente. Esses scripts também permitem executar análises em lote para analisar automaticamente várias imagens e, assim, acelerar a execução do método.
Protocolo
1. Infecção de células epiteliais com Salmonella portadora de plasmídeo repórter pCHAR-Duo
NOTA: Recomenda-se uma pipeta multicanal.
- Revesti placas de imagem de 96 poços com paredes pretas e fundo plano de vidro com colágeno logo antes do uso.
- Diluir o ácido acético glacial (17,4 M) em água desmineralizada estéril sob um exaustor químico para obter uma solução de ácido acético de 20 mM. Em condições estéreis, filtrar a solução através de um filtro de seringa com um tamanho de poro de 0,2 μm. Deixe a solução num rack de 0-4 °C durante 5 minutos.
- Diluir 3 mg/ml de colagénio em 50 μg/ml em solução de ácido acético 20 mM pré-arrefecida. Misture invertendo 10 vezes e, em seguida, dispense 30 μL de solução de colágeno por poço (5 μg de colágeno/cm2), mantendo a solução em um rack de 0-4 °C para evitar a gelificação do colágeno.
- Certifique-se de que os fundos do poço estejam completamente cobertos com colágeno e, em seguida, deixe a placa sob fluxo laminar por 1 h à temperatura ambiente (RT).
- Remova suavemente a solução e realize três lavagens com 30 μL de solução salina tamponada com fosfato (PBS). Use uma pipeta multicanal de 100 μL ou 50 μL para evitar o descolamento de colágeno.
- Cultura de células epiteliais INT407 em placas de imagem revestidas de colágeno 20-24 h antes da infecção.
- Cultivar rotineiramente células INT407 em frascos de25 cm 2 em Meio Essencial Mínimo com 10% de soro fetal bovino (FBS), doravante meio de cultura (CM), doravante definido, suplementado com penicilina 100 U/mL e estreptomicina 100 μg/mL (Pen/Strep).
- Lavar duas vezes os frascos de INT407 com 5 ml de PBS e, em seguida, separar as células com 1 ml de solução de tripsina-EDTA durante 3-5 min a 37 °C em atmosfera de CO2 a 5% humidificada 20-24 h antes da infecção. Conte as células e prepare uma suspensão de 3 x 105 células/ml no MC.
- Dispensar 100 μL de suspensão/poço celular em placas de imagem revestidas de colágeno para obter 100% de confluência. Incubar a 37 °C em atmosfera de CO2 a 5% umidificada durante 1 h para facilitar a adesão celular. Em seguida, adicionar 100 μL de MC e incubar a 37 °C em atmosfera de CO2 a 5% umidificada por 20-24 h até a infecção (etapa 1.4).
NOTA: Para obter imagens de bactérias intracelulares, as cepas de Salmonella são transformadas com o plasmídeo repórter pCHAR-Duo que foi gentilmente fornecido pela Dra. Olivia Steele-Mortimer. As cepas aqui testadas foram selecionadas para representar a diversidade fenotípica coberta pelo protocolo e validar as análises de imagem na seção 4. Veja os resultados representativos para mais detalhes sobre as cepas utilizadas neste estudo.
- Preparar culturas de fase estacionária de cepas de Salmonella carregando o plasmídeo repórter pCHAR-Duo.
- Colher amostras do caldo de Salmonella glycerol com a ponta de uma pipeta de 10 μL e inocular-o em 1 ml de caldo de Luria Bertani (10 g de triptona, 5 g de extrato de levedura e 10 g de cloreto de sódio por litro) suplementado com ampicilina 100 μg/ml.
- Incubar o inóculo estaticamente a 37 °C por 20 h antes da infecção para atingir a fase estacionária de crescimento, correspondendo a ~1 x 109 Unidades Formadoras de Colônias (UFC)/mL8.
- Infectar células epiteliais INT407 com Salmonella.
NOTA: As infecções são realizadas em triplicados.- Lave suavemente as células INT407 com 200 μL de PBS/poço.
- Preparar inóculo de Salmonella diluindo bactérias durante a cultura em MC para obter o número desejado de UFCs por célula epitelial, definida como a multiplicidade de infecção (MOI). Aqui foi utilizado o MOI 100.
- Inocular 200 μL/poço e, em seguida, cobrir a placa com uma membrana de vedação respirável e incubar a 37 °C em atmosfera umidificada a 5% de CO2 durante 1 h. A inoculação é considerada como tempo zero da infecção.
- Retire o inóculo e lave suavemente com 200 μL de PBS/poço e, em seguida, adicione 200 μL de CM com gentamicina 100 μg/mL por poço. Cobrir a placa com uma membrana de vedação respirável e, em seguida, incubar a 37 °C em atmosfera de CO2 a 5% humidificada durante 1 h.
- Retire o MC com gentamicina 100 μg/mL e lave suavemente com 200 μL de PBS/poço. Adicionar 200 μL de MC com gentamicina a 10 μg/mL por poço, cobrir a placa com uma membrana de vedação respirável e, em seguida, incubar a 37 °C em atmosfera umidificada a 5% de CO2 por 8 h.
2. Fixação da amostra e coloração das células epiteliais
NOTA: Mantenha as amostras protegidas da exposição direta à luz. Os volumes são indicados para poços de uma placa de 96 poços. A otimização de volume é necessária para diferentes placas ou suportes de cultura de células.
- Às 8 h pós-infecção, remova a MC e lave suavemente três vezes com 200 μL/poço de PBS. Remova o PBS invertendo a placa em papel absorvente; evitar aspirar.
- Fixar as monocamadas infectadas com 100 μL/poço de paraformaldeído (PFA) a 4% em PBS por 20 min no RT. Retirar o PFA 4% e lavar três vezes com 200 μL/poço de PBS. A placa pode ser armazenada por um período máximo de 16-24 h a 4 °C. Prossiga com a próxima etapa.
- Células epiteliais coradas.
NOTA: A coloração de ADPI (4′,6-diamidino-2-fenilindol) (etapa 2.3.1) é usada para a análise de área para manchar apenas núcleos, e a coloração de High Content Screening (HCS) (etapa 2.3.2) é usada para a análise de célula única para manchar toda a célula epitelial. Outras manchas celulares podem ser usadas, mas a otimização da aquisição e análise de imagens é necessária.- Análise de área: dispensar 100 μL/poço de DAPI (solução de 300 nM em PBS) e incubar 5 min no RT protegido da luz.
- Análise unicelular: permeabilizar com 100 μL/poço de tritão 0,1x em PBS por 15 min no RT. Lavar três vezes com PBS e dispensar 100 μL/poço de HCS corante diluído 1:2000 em PBS. Incubar por 30 min no RT protegido da luz.
- Retire a solução de coloração e lave três vezes com 200 μL/poço de PBS. Adicione 50 μL/poço de PBS para aquisição automatizada de imagens.
3. Aquisição de imagens com microscópio de fluorescência automatizado
NOTA: Aqui, baixa ampliação (10x/0,3 NA, objetiva de 1 μm/pixel) na etapa 3.1.1 para a análise de área e alta ampliação (40x/0,75 NA, objetiva de 0,255 μm/pixel) na etapa 3.1.2 para a análise de célula única são usadas no protocolo de aquisição. Outras ampliações podem ser usadas, mas a otimização da aquisição e análise de imagens é necessária. Se permitido pelo microscópio disponível, adquira imagens como Tile Regions (TRs), um "mosaico" de campos contíguos chamados telhas, para registrar grandes áreas de amostra. Defina o foco automático no centro de um TR em vez de definir um para cada bloco, para reduzir a exposição da amostra durante o procedimento de foco automático.
- Use o foco automático do software no canal de coloração da célula (canal azul para coloração HCS e coloração nuclear DAPI) como a posição z de referência. Adquirir imagens no canal de coloração celular (465 nm, células ou núcleos) e nos canais repórteres pCHAR-Duo mCherry (610 nm, Salmonelas intracelulares) e GFP (509 nm, Salmonelas citosólicas hiper-replicantes).
- Análise de área: Imagem ≥104 células/replicação técnica (ou seja, recomenda-se pelo menos três TRs de quatro telhas/poço correspondentes a uma réplica técnica) com ampliação de 10x.
NOTA: Um único plano z é suficiente para a imagem de ambas as células contendo vacuolar e células contendo Salmonellae citosólica hiper-replicante com ampliação de 10x. - Análise de célula única: Imagem ≥1.000 células/replicação técnica (ou seja, pelo menos duas TRs de 16 telhas/poço correspondentes a uma replicação técnica são recomendadas) com ampliação de 40x. Múltiplos planos z são necessários para a imagem de células contendo vacuolar e células contendo Salmonelas citosólicas hiper-replicantes. Defina a pilha z ideal (3,85 μm z-stack com intervalo de 0,55 μm para obter oito planos z é indicado para a imagem de células INT407 infectadas com Salmonellae citosólica hiper-replicante a 40x). Cada plano z é doravante identificado como n/8, com n entre 1 (correspondente ao plano z inferior) e 8 (correspondente ao plano z superior).
- Análise de área: Imagem ≥104 células/replicação técnica (ou seja, recomenda-se pelo menos três TRs de quatro telhas/poço correspondentes a uma réplica técnica) com ampliação de 10x.
- A saída de cada aquisição é um arquivo chamado Acquisition.czi que inclui imagens de todos os campos, canais e planos z. Se os TRs foram adquiridos, funda os blocos para obter uma imagem geral de cada TR usando um único arquivo Acquisition.czi como entrada para o método Stitching.
4. Análise de imagem usando ImageJ
NOTA: As etapas 4.1 e 4.2 são projetadas especificamente para o experimento e a aquisição descritos acima. Outras configurações experimentais podem exigir otimização da análise. A análise de um único arquivo Acquisition.czi é descrita. Para a análise em lote, localize o script de análise de célula única e o script de análise de área como arquivos suplementares (Arquivo Suplementar 1 e Arquivo Suplementar 2). Os rótulos usados nos scripts e comandos ImageJ estão em negrito nas seções abaixo.
- Análise de área
- Abra o arquivo Acquisition.czi no ImageJ, nomeado como AcquisitionTitle no script: File > Open > AcquisitionTitle.czi. Uma janela mostrando todos os canais será aberta. Os canais são indicados como c:1-3/3 dependendo da ordem de aquisição. Aqui, c:1/3 corresponde ao azul (DAPI), c:2/3 a mCherry (Salmonellae intracelular) e c:3/3 à GFP (Salmonellae citosólica hiper-replicante).
- Reduza o ruído aleatório para preparar imagens para medição de área nas etapas 4.1.6 e 4.1.7.
- Duplique a janela AcquisitionTitle usando Image > Duplicate > OK para obter a janela AcquisitionTitle1 .
- Aplique o desfoque gaussiano a AcquisitionTitle1 por meio do filtro de > de processo > desfoque gaussiano. Deixe o valor Sigma padrão como 2 e clique em OK. Uma janela Process Stack? será aberta, clique em Sim para processar todos os canais.
- Subtraia o AcquisitionTitle1 filtrado por gaussian do arquivo original AcquisitionTitle by Process > Image Calculator: selecione AcquisitionTitle como Image1, escolha a operação Subtract na lista suspensa e selecione AcquisitionTitle1 como Image2. Clique em OK. Uma janela Process Stack? será aberta. Clique em Sim para processar todas as três imagens (canais) para obter a janela ResultsOfAcquisitionTitle.
- Canais divididos através de Canais de Imagem > Cor > Divididos. Cada imagem de canal agora é mostrada em uma janela separada, automaticamente nomeada pelo ImageJ como C-ResultsOfAcquisitionTitle. Aqui C1-ResultsOfAcquisitionTitle corresponde a DAPI, C2-ResultsOfAcquisitionTitle a mCherry (Salmonellae intracelular) e C3-ResultsOfAcquisitionTitle a GFP (Salmonellae citosólica hiper-replicante).
- Processe a imagem C1-ResultsOfAcquisitionTitle para medir a área ocupada pelos núcleos das células epiteliais.
- Navegue até Processar > Suave para homogeneizar o nucléolo que aparece como buracos dentro da área dos núcleos.
- Limite a imagem C1-ResultsOfAcquisitionTitle para excluir o plano de fundo usando Imagem > Ajustar Limite de >: marque Plano de Fundo Escuro e selecione Vermelho na lista suspensa. Defina o limite automático do Triângulo e use o controle deslizante superior para definir o valor mínimo de limite quando os núcleos aparecerem vermelhos e o plano de fundo aparecer preto (Limiar = 100, aplicado neste protocolo).
NOTA: Os limiares automáticos descritos nas etapas 4.1.4.2, 4.1.7, 4.2.3.6.1 e 4.2.4.3 são sugeridos como um guia para o usuário definir o melhor limiar de ajuste para núcleos/células epiteliais e bactérias manualmente, respectivamente. O valor do limite automático será substituído no script pelo limite manual definido. O limite manual definido será aplicado a toda a análise do lote.
- Escolha osmedidores de interesse usando Analisar > Definir Medição: Marcar Limite ao Limite para limitar a medição a pixels com limite. Área de verificação, correspondente à área total ocupada por pixels limiares (ou seja, núcleos em C1-ResultsOfAcquisitionTitle, Salmonellae intracelular em C2-ResultsOfAcquisitionTitle, Salmonellae citosólica hiper-replicante em C3- ResultsOfAcquisitionTitle). Marque Display Label para registrar o título e o canal de aquisição de cada imagem na tabela de resultados.
- Meça a área ocupada pelos núcleos usando Analisar > Medir. As medições são registradas na tabela de resultados que é aberta automaticamente.
- Processo C2-ResultsOfAcquisitionTitle e C3-ResultsOfAcquisitionTitle para medir a área ocupada por Salmonelas intracelulares globais e Salmonellae hiper-replicantes citosólicas, respectivamente. Siga as etapas 4.1.4-4.1.6 com algumas modificações: pule a etapa de suavização na etapa 4.1.4.1 e use o limite automático Otsu em vez do Triângulo na etapa 4.1.4.2 como um guia para definir o valor mínimo do limite (controle deslizante superior) quando as Salmonellae aparecerem vermelhas e o fundo aparecer preto (Limiar = 200 sugerido).
- Salve a tabela Resultado usando Arquivo > Salvar como > Todos os Arquivos.
- Abra a tabela Resultado em uma planilha para calcular as proporções de área para cada arquivo AcquisitionTitle analisado:
- Calcule a razão de infecção: divida a área ocupada por Salmonellae intracelular total (aqui canal vermelho, C2-) pela área ocupada por núcleos de células epiteliais (aqui DAPI, C1-).
- Calcular a razão de hiper-replicação: dividir a área ocupada por Salmonellae citosólica hiper-replicante (canal verde, C3-) pela área ocupada por Salmonellae intracelular total (canal vermelho, C2-).
- Análise de célula única
- Abra o arquivo Acquisition.czi no ImageJ, nomeado como AcquisitionTitle no script: File Menu > Open > Acquisition.czi. Uma janela mostrando as imagens de todos os canais e todos os planos z será aberta.
- Divida os canais por imagem > cor > canais divididos. Os canais agora são mostrados em janelas separadas, automaticamente nomeadas pelo ImageJ como C-AcquisitionTitle. Aqui, a janela C1-AcquisitionTitle corresponde às células epiteliais (azul), a janela C2-AcquisitionTitle às Salmonellae intracelulares (mCherry) e a janela C3-AcquisitionTitle ao canal de Salmonellae citosólico hiper-replicante (GFP). Cada janela C-AcquisitionTitle inclui todos os z-planes adquiridos.
- Processe o C1-AcquisitionTitle janela, correspondente a células epiteliais, a células segmentadas.
- Escolha a imagem do plano z a ser usada para segmentação de célula. Selecione a janela C1-AcquisitionTitle e use Imagem > Duplicar: escreva o número do plano z escolhido (ou seja, 1 para selecionar z 1/8), escreva C1Zplane na caixa Título, desmarque Pilha Duplicada e clique em OK para duplicar somente a imagem do plano z selecionada. Uma janela chamada C1Zplane mostrando a imagem do plano z selecionada será aberta.
- Processe a imagem C1Zplane obtida para melhorar o contraste da imagem. Processo aberto > Melhorar contraste: ajuste os pixels saturados (ou seja, aqui 1% é usado) e marque Normalizar. Em seguida, clique em OK para aplicar a técnica de realce de contraste para obter uma imagem C1Zplane normalizada por contraste.
- Processe a imagem C1Zplane normalizada por contraste para segmentar as células epiteliais. Use Processo > Localizar Maxima para abrir o menu FindMaxima : primeiro, sinalize a Seleção de Ponto de Visualização para definir Tolerância ao Ruído para atribuir apenas um ponto máximo a cada célula epitelial. Sinalizar Excluir Edge Maxima. Selecione o tipo de saída Partículas Segmentadas e clique em OK para obter C1ZplaneSegmented, uma nova imagem binária semelhante a uma máscara mostrando cada partícula segmentada por ponto máximo marcado.
Observação : O algoritmo Find Maxima ImageJ é usado para segmentar células. Pontos máximos (picos de intensidade de pixel) são detectados em toda a imagem, potencialmente correspondendo a células. Um limiar de ruído (tolerância ao ruído) é definido, e a área contígua em torno de pontos máximos é analisada para criar uma imagem binária semelhante a uma máscara definindo cada partícula segmentada por ponto máximo, portanto, cada célula. - Processe a imagem C1ZplaneSegmented para criar uma máscara de células segmentadas. Use Analisar > Analisar partículas: selecione a opção Mostrar máscaras e Excluir nas bordas. Ajuste o intervalo de área de partículas segmentadas a ser incluído na máscara, correspondente ao parâmetro Size , para excluir objetos segmentados incorretamente, como aglomerados de células e frações de células. Para pontuar a área de objetos segmentados erroneamente, use a ferramenta Rastreamento de varinha na barra de ferramentas: selecione partícula com a varinha e, em seguida, abra Analisar > medida para medir a área de objetos errôneos. Defina o intervalo de tamanho (intervalo sugerido de 250 a 1700 pixels2) e clique em OK para obter a máscara binária MaskofC1ZplaneSegmented .
- Por padrão, a máscara binária MaskOfC1ZplaneSegmented tem uma LUT invertida. Use LUT > Inverter LUT na barra de ferramentas.
- Processe a máscara binária MaskOfC1ZplaneSegmented para corrigir a segmentação da célula:
- Imagem C1Zplane normalizada por contraste limiar obtida no passo 4.2.3.2. Use Imagem > Ajustar > Limite: marque Fundo Escuro. Defina a configuração de limite automático padrão . Escolha a opção Vermelho e ajuste o valor de corte mínimo (barra superior) até que as células apareçam completamente vermelhas, deixando o fundo escuro (Limiar = 8.000 aplicado neste protocolo). Em seguida, clique em Aplicar para converter a imagem C1Zplane normalizada por contraste em uma imagem binária com células em branco e o fundo em preto.
- Corrigir a segmentação de célula em MaskOfC1ZplaneSegmented máscara binária. Use Processo > Calculadora de Imagens: selecione MaskOfC1ZplaneSegmented como Image1, escolha a operação E na lista suspensa e, em seguida, selecione o C1Zplane normalizado por contraste com limite como Image2. Clique em OK. A imagem de saída é automaticamente nomeada pelo ImageJ como Resultados da Máscara do C1Zplane Segmentado.
- Resultados do processo da máscara de C1Zplane segmentado para rotular cada célula segmentada como uma região de interesse (ROI). Use Analisar > Analisar partículas. Ajuste o tamanho como na etapa 4.2.3.4 e, em seguida, selecione a opção Mostrar nada. Marque Adicionar ao Gerenciador para adicionar todas as partículas (células segmentadas) à ferramenta Gerenciador de ROI. Além disso, marque Excluir em Bordas. Clique em OK. O ROI Manager Menu mostrará uma lista de todas as partículas (células segmentadas), definidas como ROIs, exclusivamente rotuladas com suas respectivas coordenadas y e x.
- Salve os dados de ROIs como ROI-cells-AcquisitionTitle.zip por meio do menu do ROI Manager clicando em Mais > Salvar. ROI-cells-AcquisitionTitle.zip será aberto na etapa 4.2.4.6 para a análise de célula única.
- Processe o C2-AcquisitionTitle janela (aqui canal vermelho), correspondente a intracelular Salmonellae, para medir o número de células infectadas e a carga vacuolar intracelular.
- Subtraia o canal verde (C3-) do canal vermelho (C2-) para remover pixels fora de foco das Salmonellae hiper-replicantes citosólicas.
- Use a Calculadora de Imagens de > de Processo. Selecione C2-Acquisitiontitle como Image1, escolha a operação Subtract na lista suspensa e selecione C3-Acquisitiontitle como Image2. Marque Criar nova janela e clique em OK.
- Aplicar subtração a toda a pilha z clicando em Sim na pilha de processos? janela. A janela de saída é chamada ResultsOfC2AcquisitionTitle no script.
- Process ResultsOfC2AcquisitionTitle para reduzir o ruído aleatório.
- Duplique a janela ResultsOfC2AcquisitionTitle clicando no menu Imagem > Duplicar. Marque Pilha duplicada e pressione OK para obter a janela ResultsOfC2AcquisitionTitle1 .
- Aplique o Desfoque Gaussiano à janela ResultsOfC2AcquisitionTitle1 por meio do Filtro de > de Processo > Desfoque Gaussiano. Deixe o valor Sigma como 2 ou personalize-o (ou seja, aqui Sigma: 4 foi usado). Clique em OK e aplique o Gaussian Blur a toda a pilha z clicando em Sim na janela Pilha de Processo ?.
- Subtraia o Gaussian filtered ResultsOfC2AcquisitionTitle1 para ResultsOfC2AcquisitionTitle clicando em Process > Image Calculator. Selecione ResultsOfC2AcquisitionTitle como Image1, escolha a Operação Subtrair na lista suspensa e selecione ResultsOfC2AcquisitionTitle1 como Image2. Clique em OK. Aplique a subtração a toda a pilha z clicando em Sim na janela Pilha de processos? para obter uma janela automaticamente nomeada pelo ImageJ como Resultados dos Resultados do C2-AcquisitionTitle.
- Processar os resultados dos resultados do C2-AcquisitionTitle para separar as salmonelas do plano de fundo. Use Imagem > Ajustar Limite de >: marque Plano de fundo escuro e defina o limite automático do Otsu . Ajuste o valor de corte mínimo (barra superior) até que as Salmonellae apareçam completamente vermelhas, deixando o fundo escuro (Limiar = 100 aplicado neste protocolo). Clique em Aplicar e a janela Converter em binário será aberta: marque Fundo preto e, em seguida, clique em OK. A pilha z completa agora é convertida em imagens binárias.
- Selecione o plano Z médio, correspondente ao plano de foco vacuolar de Salmonellae, nos resultados da janela binária Resultados do C2-AcquisitionTitle da etapa 4.2.4.3: Image > Stacks > Set Slice e escreva o número do plano z de escolha (por exemplo, aqui z: 4/8 é usado). Clique em OK.
- Defina as medições a serem registradas para cada ROI (célula). Use Analisar > Definir Medição: verifique a área, correspondente à área total de cada ROI. Marque Fração de Área e Limite ao Limite para registrar apenas a fração de Área ocupada por pixels com limite ( Salmonellae intracelular) para cada ROI. Verifique o Display Label para rotular cada ROI com o título da imagem, o canal, o plano z e as coordenadas x-y na tabela de resultados.
- Processe o plano z escolhido de Salmonellae intracelular para gravadoras, Área e %Área ocupada por Salmonellae para cada célula (ROI): abra o arquivo ROI-cells-AcquisitionTitle.zip, salvo na etapa 4.2.3.8, por Arquivo > Abrir e clique em Medir no menu do Gerenciador de ROI. A saída é uma tabela que relata as medidas selecionadas.
- Salve a tabela de resultados usando Arquivo > Salvar como na janela de resultados.
- Abra a Tabela de Resultados em uma planilha: a área do rótulo e a área ocupada por salmonelas intracelulares são indicadas para cada ROI, correspondendo a uma célula contornada identificada exclusivamente com coordenadas x e y.
- Meça o número de células infectadas correspondentes a ROIs mostrando uma %Área > 0 ocupada por pixels limiares, correspondendo a Salmonellae (ou seja, aqui uma %Área > 0,2% é considerada para identificar células infectadas). Em seguida, calcule a porcentagem de células infectadas no total de células.
- Medir a carga vacuolar de Salmonella/célula calculando a %Área média ocupada por Salmonellae vacuolar para todas as células infectadas.
- Subtraia o canal verde (C3-) do canal vermelho (C2-) para remover pixels fora de foco das Salmonellae hiper-replicantes citosólicas.
- Canal verde de processo (C3-AcquisitionTitle) para medir o número de células que mostram Salmonellae citosólica hiper-replicante. Siga as etapas 4.2.4.2 a 4.2.4.9, mas com algumas modificações.
- Trabalho nos planos z superiores (aqui z:7/8), uma vez que as células que apresentam Salmonellae citosólica hiper-replicante se projetam da monocamada; portanto, eles estão em foco em um plano diferente em comparação com as células sem hiper-replicantes Salmonellae.
- Meça o número de células contendo Salmonellae citosólica hiper-replicante (verde) por meio da pontuação de células (ROIs) mostrando alta %Área ocupada por Salmonellae (por exemplo, uma %Área > 20% é considerada para identificar células contendo Salmonellae citosólica hiper-replicante). Em seguida, calcule a porcentagem de células contendo Salmonellae citosólica hiper-replicante no total de células infectadas pontuadas na etapa 4.2.4.9.
Resultados
Infecção de células epiteliais com cepas de Salmonella
Este protocolo foi desenvolvido para analisar a invasão celular e a replicação citosólica (Figura 1A) vs carga vacuolar (Figura 1B) de Salmonella no interior das células epiteliais. O protocolo foi validado utilizando-se as três seguintes cepas de Salmonella, S. Typhimurium SL1344 cepa de referência (S. Tm), S. Derby ER1175 wildtype (S. Derby wt) e o mutante isogênico de S. Derby ER1175 sem gene sipA (S. Derby ΔsipA). A cepa S. Derby ER1175 foi isolada de suínos e pertence à coleção de vigilância IZSLER de isolados de Salmonella. As cepas foram selecionadas para representar a diversidade fenotípica coberta pelo protocolo. Em particular, essas cepas foram escolhidas porque seu comportamento dentro das células epiteliais já era conhecido por diferir em termos de invasão ou replicação9; portanto, foram controles valiosos para testar se o protocolo permitiu distinguir quantitativamente diferenças nos fenótipos intracelulares de Salmonella. Em particular, S. Tm e S. O Derby wt foi incluído porque já tínhamos demonstrado que S. Tm tem maior invasão e eficiência de replicação intracelular do que S. Derby wt9 enquanto S. Derby ΔsipA foi adicionado como uma cepa de hiper-replicação prejudicada, uma vez que o efetor de virulência SipA desempenha um papel crucial no início da hiper-replicação10,11. Foi relatado anteriormente para S. Tm que a replicação citosólica inicia-se 4 h após a invasão, e então a população citosólica rapidamente hiper-replica-se para preencher a célula epitelial por 8 h11,12. Consistentemente, a infecção de 8 h de duração foi adequada para observar e quantificar o fenótipo de hiperreplicação citosólica também em S. Derby wt e S. Derby ΔsipA.
Análise de área
A análise de área na etapa 4.1 fornece uma medida da colonização geral de células epiteliais por Salmonella (razão de infecção), juntamente com uma medida da hiper-replicação (razão de hiper-replicação). No fluxo de trabalho de análise de área descrito na Figura 2, o ruído aleatório é reduzido e, em seguida, os canais são divididos e processados de forma independente. Um limite é definido para cada canal, a fim de excluir o plano de fundo da medição de área. Em seguida, a área ocupada por pixels com limite é medida para cada canal. O resultado da análise de área é uma tabela relatando, para cada arquivo de aquisição, a extensão das áreas ocupadas por núcleos de células epiteliais (canal azul), Salmonelas intracelulares que expressam mCherry (canal vermelho) e Salmonelas citosólicas hiper-replicantes que expressam GFP (canal verde) junto com mCherry. A razão de infecção é calculada dividindo a área ocupada por Salmonellae que expressam mCherry pela área ocupada pelos núcleos das células hospedeiras. Os resultados das cepas testadas mostraram que S. Tm apresenta uma taxa de infecção significativamente maior em comparação com ambos os S. Cepas de Derby, como esperado (Figura 3A). Portanto, esses resultados demonstram a eficácia da análise de área na detecção de diferenças na capacidade de cepas de Salmonella de colonizar células epiteliais. A razão de hiperreplicação de Salmonella é medida dividindo-se a área ocupada por Salmonellae que expressam GFP pela área ocupada por Salmonellae intracelular que expressam mCherry. Consistentemente com o papel do SipA na determinação da hiperreplicação, uma taxa de hiperreplicação significativamente reduzida para S. A deformação de Derby ΔsipA foi medida em comparação com S. Derby wt, (Figura 3B). Não foi observada diferença de hiper-replicação entre S. Tm e S. Derby wt. No geral, a análise de área efetivamente revelou diferentes níveis de hiper-replicação entre as cepas ensaiadas.
Análise de célula única
A análise unicelular permite quantificar a invasão de Salmonella e a carga vacuolar versus replicação citosólica dentro de células epiteliais com resolução de célula única. Conforme descrito na etapa 4.2, para cada arquivo de aquisição, os canais azul, vermelho e verde foram processados de forma independente (Figura 4). Especificamente, o canal azul, correspondente às células epiteliais, foi processado na etapa 4.2.3 para obtenção da segmentação celular. Em seguida, as células segmentadas foram adicionadas ao Gerente de Região de Interesse (ROI) para obter uma lista de ROIs, correspondendo a todas as células segmentadas exclusivamente marcadas com coordenadas y-x. A fim de remover pixels fora de foco de Salmonellae hiper-replicantes que expressam GFP, os pixels do canal verde foram subtraídos daqueles do canal vermelho. A tabela de saída da análise de célula única relata, para cada ROI, a área e a porcentagem da área do ROI ocupada por Salmonellae expressando apenas o repórter constitutivo mCherry ou o repórter responsivo ao citosol GFP. A porcentagem da área da célula ocupada por Salmonellae que apenas expressam mCherry foi processada na etapa 4.2.4 para calcular a porcentagem de células infectadas e a carga vacuolar média (Figura 5A). A análise da carga vacuolar mostrou que o S. As cepas de Derby geram uma carga vacuolar média significativamente menor que a S. Tm (Figura 5B). Por outro lado, apenas uma ligeira e não significativa redução da percentagem de células infectadas foi observada em S. Derby degraus em comparação com S. Tm (Figura 5A). A porcentagem da área da célula ocupada por Salmonellae expressoras de GFP foi processada na etapa 4.2.5 para obter a taxa de hiperreplicação. Não foi observada diferença entre S. Tm e S. Derby wt, enquanto S. Derby ΔsipA apresentou uma diminuição significativa da hiper-replicação, consistente com o resultado da análise de área (Figura 5C) e o papel do SipA na indução da hiper-replicação.
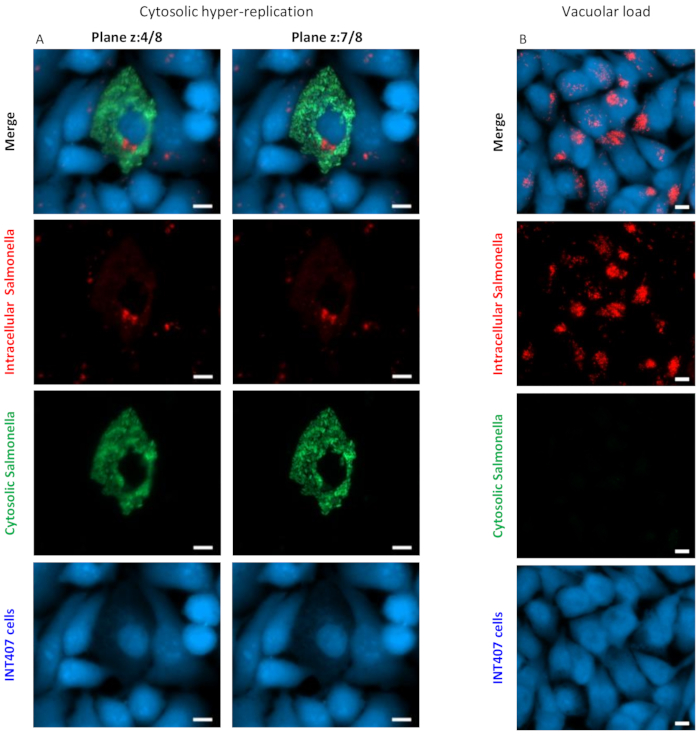
Figura 1: Hiperreplicação citosólica de Salmonella e carga vacuolar. Imagens representativas de (A) a hiper-replicação de Salmonella e (B) a carga vacuolar adquirida em alta ampliação (40x) são mostradas. O Painel B mostra os diferentes planos de foco das células contendo Salmonellae hiper-replicantes (z:7/8), em comparação com Salmonellae não hiper-replicantes (z:4/8). As barras de escala branca são de 10 μm. Clique aqui para ver uma versão maior desta figura.

Figura 2: Fluxo de trabalho da análise da área. O fluxo de trabalho da análise de área é mostrado para uma aquisição representativa de baixa ampliação (10x) de células INT407 infectadas por 8 h com Salmonella carregando o plasmídeo pCHAR-Duo (MOI 100). Primeiro, o ruído aleatório é reduzido e, em seguida, os canais são divididos em C1, C2 e C3 e processados de forma independente. Um limite é definido para cada canal, a fim de excluir o plano de fundo da área de medição. Em seguida, a área ocupada pelos pixels limiares correspondentes apenas aos núcleos celulares em C1, todas as Salmonelas intracelulares em C2 e Salmonelas citosólicas em C3 – é medida para cada canal. As imagens aqui analisadas são os resultados de quatro azulejos adquiridos com ampliação de 10x fundidos por costura. As barras de escala branca são de 100 μm. Clique aqui para ver uma versão maior desta figura.
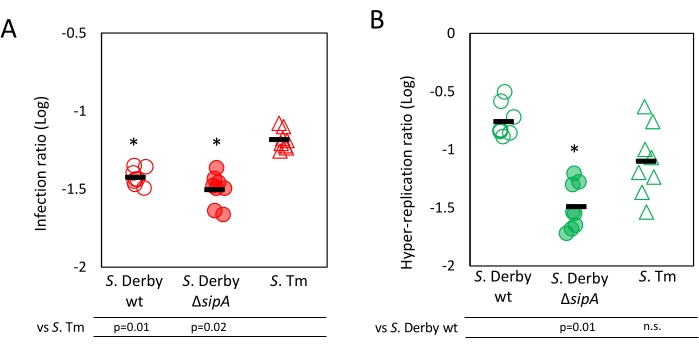
Figura 3: Resultados da análise da área. Os resultados da análise de área são mostrados. (A) A razão de infecção é calculada dividindo-se a área ocupada por Salmonellae que expressam mCherry (canal C2), representando todas as bactérias intracelulares, pela área ocupada pelos núcleos das células hospedeiras (canal C1). (B) A razão de hiperreplicação foi medida dividindo-se a área ocupada por Salmonellae expressoras de GFP (canal C3), representando apenas bactérias citosólicas hiper-replicantes, pela área ocupada por todas as Salmonellae intracelulares que expressam mCherry. Cada ponto representa uma replicação. A análise foi realizada em três repetições biológicas, cada uma testada em triplicata. As linhas pretas indicam os valores médios. A significância foi calculada por meio do teste t bicaudal, e os valores de p são relatados. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 4: Fluxo de trabalho da análise de célula única. O fluxo de trabalho da análise unicelular é mostrado para uma aquisição representativa de alta ampliação (40x) de células INT407 infectadas por 8 h com Salmonella carregando o plasmídeo pCHAR-Duo (MOI 100). Primeiro, os canais são divididos em C1, C2 e C3 e processados de forma independente. O canal azul (C1), correspondente às células epiteliais, é processado para obter a segmentação celular e, em seguida, cada célula segmentada é definida como uma Região de Interesse (ROI) exclusivamente marcada com coordenadas y-x. O canal vermelho (C2) é processado subtraindo o canal verde (C3) para remover Salmonellae citosólica hiper-replicante fora de foco, deixando todas as Salmonellae vacuolares expressando apenas mCherry e, em seguida, o ruído aleatório foi reduzido, e o limiar é definido para excluir o fundo das medições. Finalmente, a área ocupada por Salmonellae vacuolar para cada ROI é medida. O canal verde (C3), correspondente às Salmonellae que expressam GFP citosólica total, é processado de forma semelhante. As imagens aqui analisadas são os resultados de 16 azulejos fundidos por costura. As barras de escala branca são de 100 μm. Clique aqui para ver uma versão maior desta figura.
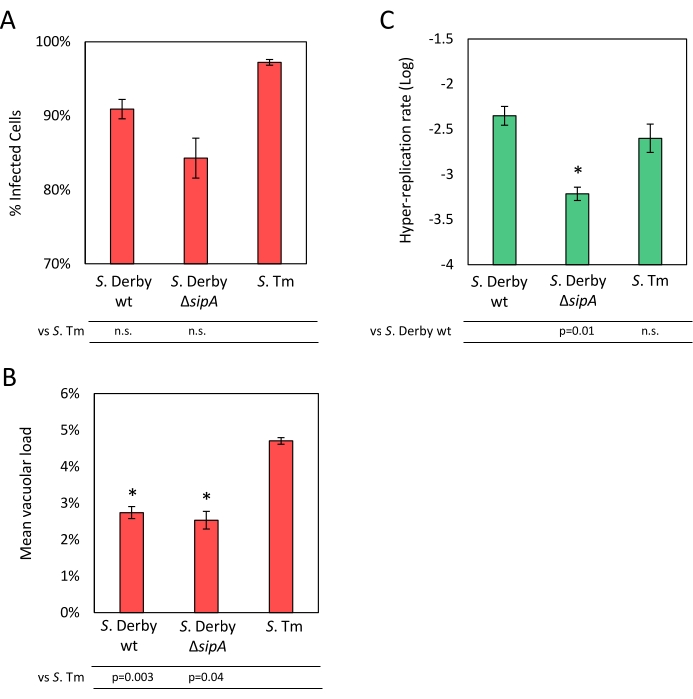
Figura 5: Resultados da análise unicelular. Os resultados da análise de célula única são mostrados. (A) A percentagem de células infectadas é obtida dividindo o número de células com uma percentagem de área ocupada por Salmonellae que apenas mCherry> 0,2 pelo número total de células. (B) A carga vacuolar média foi obtida calculando-se a porcentagem média de área ocupada por mCherry-only expressando Salmonellae nas células infectadas. (C) A taxa de hiperreplicação foi calculada dividindo-se o número de células com uma porcentagem de área ocupada por Salmonellae que expressam GFP≥20% pelo número total de células infectadas. Dados de três a quatro replicações biológicas testadas em triplicado são relatados. As barras indicam o erro padrão de medição. A significância foi calculada por meio do teste t bicaudal, e os valores de p são relatados. Por favor, clique aqui para ver uma versão maior desta figura.
Arquivo Suplementar 1: Script de análise de área. Clique aqui para baixar este arquivo.
Arquivo suplementar 2: Script de análise de célula única. Clique aqui para baixar este arquivo.
Discussão
A forma como a Salmonella coloniza as células epiteliais intestinais, influencia o desfecho da infecção. Após a invasão, a hiperreplicação citosólica induz inflamação do intestino3, enquanto a replicação vacuolar pode levar à disseminação sistêmica4. As cepas de salmonela podem variar em sua capacidade de invadir e replicar dentro das células epiteliais intestinais9. De fato, Salmonella é um gênero extremamente diversificado, compreendendo mais de 2.500 sorovares, que têm diferentes habilidades para causar doenças. Além disso, a salmonela é a causa mais frequentemente referida de surtos de origem alimentar na União Europeia, indicando uma grande propagação na população humana1. Portanto, a disponibilidade de métodos confiáveis e de alto rendimento para a quantificação dos fenótipos intracelulares de Salmonella é de grande importância para avaliar as diferenças de virulência entre um grande número de cepas de Salmonella e, finalmente, permitir uma avaliação de risco precisa e específica da cepa desse patógeno.
O protocolo aqui descrito mede o comportamento de Salmonella dentro das células epiteliais de forma rápida e automatizada. A infecção de células epiteliais é realizada em microplacas de imagem de 96 poços e um microscópio de fluorescência automatizado é usado para aquisição de imagens, tornando o protocolo adequado para aplicações de alto rendimento. Este protocolo aproveita o plasmídeo pCHAR-Duo, que permite distinguir as Salmonelas vacuolares das citosólicas através da expressão diferencial de dois repórteres fluorescentes5. Os fenótipos intracelulares de Salmonella são frequentemente analisados por pontuação manual, um procedimento demorado que é inadequado para a análise de um grande número de cepas e culturas de células por cepa e propenso a erros do operador e variação interoperador. Para superar essas limitações, foram desenvolvidas duas análises de imagem complementares e automatizadas, a análise de área e a análise de célula única. O ImageJ, um software disponível gratuitamente, foi utilizado para desenvolver as duas análises. Para acelerar a execução do protocolo, os scripts ImageJ para análise em lote de vários arquivos de aquisição sem intervenção do operador são fornecidos como arquivos suplementares.
A análise de área foi projetada para quantificar, em poucas etapas, a colonização global de células epiteliais (razão de infecção) e o nível de hiper-replicação (razão de hiper-replicação) através da medição das áreas especificamente ocupadas pelos núcleos de células epiteliais e por Salmonellae expressando mCherry ou mCherry juntamente com GFP. A análise de área é aplicada a imagens adquiridas em baixa ampliação, onde tanto as Salmonellae vacuolares quanto as hiper-replicantes estão em foco no mesmo plano z, e um grande número de células epiteliais é exibido por campo microscópico, reduzindo o tamanho e o número de arquivos de aquisição. A análise de área permite uma análise automatizada, rápida e computacionalmente leve de um grande número de amostras, tornando-a adequada para ensaios de alto rendimento, como experimentos de triagem.
A análise unicelular foi desenhada para quantificar fenótipos de Salmonella no interior de células epiteliais com resolução unicelular, obtidas através da segmentação celular e medição da área celular e da porcentagem de área ocupada por Salmonelas hiper-replicantes vacuolares e citosólicas. A colonização global das células epiteliais caracterizada por meio da análise de área é aqui dividida em três parâmetros quantitativos, a porcentagem de células infectadas, a carga vacuolar média e a taxa de hiper-replicação, permitindo avaliar e quantificar a contribuição de cada fenótipo para a colonização global, complementando, portanto, os resultados da análise de área. Os maiores detalhes oferecidos pela análise de célula única vêm ao custo de aquisição e análise de imagem mais lentas. De fato, para alcançar a resolução de célula única, a aquisição de imagens é realizada em alta ampliação. Isso implica que múltiplos planos z são necessários para observar Salmonellae vacuolar e citosólica em foco e que a aquisição de um grande número de campos por amostra é necessária para pontuar um alto número de células epiteliais, estendendo assim o tempo de aquisição em comparação com a análise de área. Além disso, a análise de vários grandes arquivos de aquisição é computacionalmente exigente, exigindo assim uma estação de trabalho adequada (uma estação de trabalho de 6 núcleos e 32 GB de RAM foi usada). Portanto, a análise de célula única pode ser usada como autônoma em condições de rendimento limitado ou como um método de segundo nível acoplado à análise de área para obter uma compreensão mais profunda dos fenótipos de Salmonella dentro das células epiteliais.
As duas análises complementares foram validadas por meio de S. Tm, S. Derby wt, e S. Derby ΔsipA, escolhido porque seu comportamento dentro das células epiteliais já era conhecido por diferir em termos de invasão ou replicação 9,10. Os resultados das análises de área e unicelular mostram que o protocolo permitiu distinguir quantitativamente diferenças nos fenótipos intracelulares de Salmonella de acordo com as características das cepas testadas. Além disso, esses resultados demonstram que a análise unicelular permite quantificar a contribuição de cada fenótipo intracelular (invasão, carga vacuolar e replicação citosólica) para a colonização global pontuada por meio da análise de área.
Este protocolo foi aplicado aqui para estudar a patogenicidade in vitro de diferentes cepas de Salmonella, mas pode ter outras aplicações, como o estudo de mutantes aleatórios para identificar genes envolvidos na invasão e/ou replicação dentro de células epiteliais. Além disso, o protocolo pode ser adaptado para analisar o comportamento de Salmonella dentro de outras linhagens celulares que não as células INT407. Também pode ser usado como ponto de partida para desenvolver métodos semelhantes para estudar a interação célula-patógeno de outros microrganismos intracelulares.
Divulgações
Os autores declaram que não têm interesses concorrentes.
Agradecimentos
Os autores gostariam de agradecer à Dra. Olivia Steele-Mortimer por compartilhar o plasmídeo pCHAR-Duo. Este trabalho foi financiado pelo Ministério da Saúde italiano, concede as bolsas PRC2019014 e PRC2021004.
Materiais
| Name | Company | Catalog Number | Comments |
| 96-well imaging microplate | Eppendorf | 30741030 | Cell culture and infection |
| Ampicillin | Sigma-Aldrich | 59349 | Bacteria culture |
| Axio observer inverted microscope | ZEISS | Automated fluorescence microscope | |
| Axiocam 305 Mono | ZEISS | Microscope Camera | |
| Breathable sealing membrane | Sigma-Aldrich | Z380059 | Infection assay |
| Colibrì 5/7 | ZEISS | Led light source | |
| Collagen I rat tail | Life Technologies | A1048301 | Collagen coating |
| DAPI | Invitrogen | D3571 | Cell staining |
| Fetal bovine serum | Gibco | 10099-141 | Cell culture and infection |
| Gentamicin | Sigma-Aldrich | G12664 | Infection assay |
| Glacial acetic acid | Carlo Erba | 401391 | Collagen coating |
| HCS CellMask Blue | Invitrogen | H32720 | Cell staining |
| ImageJ | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation (LOCI, University of Wisconsin) | Image processing software | |
| Minimum Essential Eagle's Medium | Sigma-Aldrich | M5650-500ML | Cell culture and infection |
| Paraformaldehyde 4% | Invitrogen | FB002 | Cell fixation |
| Potassium chloride | PanReac AppliChem | 131494.1211 | PBS preparation |
| Potassium phosphate monobasic | Sigma-Aldrich | 60220 | PBS preparation |
| Sodium Chloride | PanReac AppliChem | 131659.1214 | Bacteria culture and PBS preparation |
| Sodium phosphate Dibasic | Sigma-Aldrich | 71640 | PBS preparation |
| Tissue Culture Flask 25 cm2 plug seal screw cap | Euroclone | ET7025 | Cell culture |
| Triton X-100 | Biorad | 1610407 | Cell staining |
| Trypsin-EDTA (0.25%) | Gibco | 25200056 | Cell culture and infection |
| Tryptone | Oxoid | LP0042B | Bacteria culture |
| Yeast extract | Biolife | 4122202 | Bacteria culture |
Referências
- European Food Safety Authority & European Centre for Disease Prevention and Control. The European Union One Health 2020 Zoonoses Report. EFSA Journal. 19 (12), 6971 (2021).
- Fattinger, S. A., Sellin, M. E., Hardt, W. D. Salmonella effector driven invasion of the gut epithelium: breaking in and setting the house on fire. Current Opinion in Microbiology. 64, 9-18 (2021).
- Knodler, L. A., et al. Dissemination of invasive Salmonella via bacterial-induced extrusion of mucosal epithelia. Proceedings of the National Academy of Sciences of the United States of America. 107 (41), 17733-17738 (2010).
- Fulde, M., et al. Salmonella SPI2 T3SS mediates transcytosis in polarized enterocytes in vivo. Cell Host & Microbe. , (2019).
- Cooper, K. G., Chong, A., Starr, T., Finn, C. E., Steele-Mortimer, O. Predictable, tunable protein production in Salmonella for studying host-pathogen interactions. Frontiers in Cellular and Infection Microbiology. 7, 475 (2017).
- Klein, J. A., Grenz, J. R., Slauch, J. M., Knodler, L. A. Controlled activity of the Salmonella invasion-associated injectisome reveals its intracellular role in the cytosolic population. mBio. 8 (6), 01931 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Ibarra, J. A., et al. Induction of Salmonella pathogenicity island 1 under different growth conditions can affect Salmonella-host cell interactions in vitro. Microbiology. 156 (4), 1120-1133 (2010).
- Tambassi, M., et al. Mutation of hilD in a Salmonella Derby lineage linked to swine adaptation and reduced risk to human health. Scientific Reports. 10 (1), 21539 (2020).
- Finn, C. E., Chong, A., Cooper, K. G., Starr, T., Steele-Mortimer, O. A second wave of Salmonella T3SS1 activity prolongs the lifespan of infected epithelial cells. PLoS Pathogens. 13 (4), 1006354 (2017).
- Knodler, L. A., Nair, V., Steele-Mortimer, O. Quantitative assessment of cytosolic Salmonella in epithelial cells. PLoS One. 9 (1), 84681 (2014).
- Chong, A., Starr, T., Finn, C. E., Steele-Mortimer, O. A role for the Salmonella Type III Secretion System 1 in bacterial adaptation to the cytosol of epithelial cells. Molecular Microbiology. 112 (4), 1270-1283 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados