Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aislamiento de vectores de terapia génica de próxima generación a través de ingeniería, códigos de barras y detección de variantes de cápside del virus adenoasociado (AAV)
En este artículo
Resumen
Generación de la biblioteca de visualización de péptidos AAV y posterior validación a través del código de barras de candidatos con propiedades novedosas para la creación de AAV de próxima generación.
Resumen
Los vectores de entrega de genes derivados del virus adenoasociado (AAV) son una de las herramientas más prometedoras para el tratamiento de enfermedades genéticas, evidenciada por el fomento de datos clínicos y la aprobación de varias terapias génicas AAV. Dos razones principales para el éxito de los vectores AAV son (i) el aislamiento previo de varios serotipos virales naturales con propiedades distintas, y (ii) el posterior establecimiento de tecnologías poderosas para su ingeniería molecular y reutilización en alto rendimiento. Impulsando aún más el potencial de estas técnicas se han implementado recientemente estrategias para codificar cápsides AAV seleccionadas a nivel de ADN y ARN, lo que permite su estratificación in vivo completa y paralela en todos los órganos principales y tipos de células en un solo animal. Aquí, presentamos una tubería básica que abarca este conjunto de vías complementarias, utilizando la visualización de péptidos AAV para representar el diverso arsenal de tecnologías de ingeniería de cápside disponibles. En consecuencia, primero describimos los pasos fundamentales para la generación de una biblioteca de visualización de péptidos AAV para la selección in vivo de candidatos con las propiedades deseadas, seguido de una demostración de cómo codificar las variantes de cápside más interesantes para el cribado secundario in vivo. A continuación, ejemplificamos la metodología para la creación de bibliotecas para la secuenciación de próxima generación (NGS), incluida la amplificación de códigos de barras y la ligadura de adaptadores, antes de concluir con una descripción general de los pasos más críticos durante el análisis de datos NGS. Como los protocolos informados aquí son versátiles y adaptables, los investigadores pueden aprovecharlos fácilmente para enriquecer las variantes óptimas de la cápside AAV en su modelo de enfermedad favorito y para aplicaciones de terapia génica.
Introducción
La terapia de transferencia de genes es la introducción de material genético en las células para reparar, reemplazar o alterar el material genético celular para prevenir, tratar, curar o mejorar la enfermedad. La transferencia de genes, tanto in vivo como ex vivo, se basa en diferentes sistemas de administración, no virales y virales. Los virus han evolucionado naturalmente para transducir eficientemente sus células diana y pueden ser utilizados como vectores de entrega. Entre los diferentes tipos de vectores virales empleados en la terapia génica, los virus adenoasociados se han utilizado cada vez más, debido a su falta de patogenicidad, seguridad, baja inmunogenicidad y, lo que es más importante, su capacidad para mantener la expresión no integradora a largo plazo 1,2,3. La terapia génica AAV ha producido logros considerables en la última década; tres terapias han sido aprobadas por la Agencia Europea de Medicamentos y la Administración de Alimentos y Medicamentos de los Estados Unidos para su uso en humanos 3,4. También se están realizando varios ensayos clínicos para tratar una variedad de enfermedades, como la hemofilia, las enfermedades musculares, cardíacas y neurológicas, como se revisó en otra parte3. A pesar de décadas de avances, el campo de la terapia génica ha experimentado una serie de reveses en los últimos años4, sobre todo muertes en ensayos clínicos5 que se han suspendido debido a las toxicidades limitantes de la dosis, particularmente para tejidos que son masivos, como los músculos, o difíciles de alcanzar, como el cerebro6.
Los vectores AAV que se utilizan actualmente en ensayos clínicos pertenecen a los serotipos naturales con algunas excepciones1. La ingeniería AAV ofrece la oportunidad de desarrollar vectores con una especificidad y eficiencia superior de órganos o células. En las últimas dos décadas, se han aplicado con éxito varios enfoques, como la visualización de péptidos, el intercambio de bucles, la mezcla de ADN de la cápside, la PCR propensa a errores y el diseño dirigido, para generar variantes individuales de AAV o bibliotecas de las mismas con diversas propiedades7. Estos se someten a múltiples rondas de evolución dirigida para seleccionar las variantes dentro de ellos con las propiedades deseadas, como se revisa en otra parte 1,3. De todas las estrategias de evolución de la cápside, las bibliotecas AAV de visualización de péptidos han sido las más utilizadas, debido a algunas propiedades únicas: son relativamente fáciles de generar y pueden lograr una alta diversidad y una secuenciación de alto rendimiento, lo que permite seguir su evolución.
Las primeras bibliotecas AAV de inserción de péptidos exitosas se describieron hace casi 20 años. En uno de los primeros, Perabo et al.8 construyeron una biblioteca de cápsides AAV2 modificadas, en la que se insertó un conjunto de oligonucleótidos generados aleatoriamente en un plásmido en una posición que corresponde al aminoácido 587 de la proteína de la cápside VP1, en el eje triple que sobresale de la cápside. Utilizando la coinfección por adenovirus, la biblioteca AAV evolucionó a través de múltiples rondas de selección, y se demostró que las variantes finales redirigidas eran capaces de transducir líneas celulares refractarias al AAV2 parental8. Poco después, Müller et al.9 introdujeron el sistema de dos pasos para la producción bibliotecaria, una mejora significativa del protocolo. Inicialmente, la biblioteca de plásmidos, junto con un plásmido auxiliar adenoviral, se utilizan para producir una biblioteca AAV que contiene cápsides quiméricas. Esta biblioteca de lanzadera AAV se utiliza para infectar células a baja multiplicidad de infección (MOI), con el objetivo de introducir un genoma viral por célula. La coinfección con adenovirus asegura la producción de AAV con un genoma y una cápside9 coincidentes. Aproximadamente una década después, Dalkara10 utilizó la evolución dirigida in vivo para crear la variante de 7m8. Esta variante tiene una inserción de 10 aminoácidos (LALGETTRPA), tres de los cuales actúan como enlazadores, y se dirige eficientemente a la retina externa después de la inyección intravítrea10. Esta cápside diseñada es una historia de éxito excepcional, ya que es una de las pocas cápsides diseñadas para llegar a la clínica hasta ahora11.
El campo experimentó un segundo impulso con la introducción de técnicas de secuenciación de próxima generación (NGS). Dos publicaciones de Adachi et al.12 en 2014 y de Marsic et al.13 en 2015, mostraron el poder de NGS para rastrear la distribución de bibliotecas de cápside AAV con código de barras con alta precisión. Unos años más tarde, la NGS de las regiones con código de barras se adaptó a la región de inserción de péptidos para seguir la evolución de la cápside. Körbelin et al.14 realizaron un cribado guiado por NGS para identificar una cápside basada en AAV2 dirigida a los pulmones. El análisis NGS ayudó a calcular tres puntajes de calificación: el puntaje de enriquecimiento entre las rondas de selección, el puntaje de especificidad general para determinar la especificidad del tejido y, finalmente, el puntaje combinado14. El laboratorio Gradinaru15 publicó el sistema de evolución dirigida AAV (CREATE) basado en la recombinación de Cre en el mismo año, que facilita una selección específica del tipo de célula. En este sistema, la biblioteca de cápside lleva un interruptor invertible Cre, ya que la señal polyA está flanqueada por dos sitios loxP. Luego, la biblioteca AAV se inyecta en ratones Cre, donde la señal de poliA se invierte solo en células Cre+, proporcionando la plantilla para la unión de un cebador de PCR inversa con el cebador directo dentro del gen de la cápside. Este rescate de PCR altamente específico permitió la identificación del AAV-PHP. Variante B que puede atravesar la barrera hematoencefálica15. Este sistema evolucionó aún más en M-CREATE (Multiplexed-CREATE), en el que NGS y la generación de bibliotecas sintéticas se integraron en la tubería16.
Una versión mejorada basada en ARN de este sistema del laboratorio17 de Maguire, iTransduce, permite la selección a nivel de ADN de cápsides que transducen funcionalmente células y expresan sus genomas. El genoma viral de la biblioteca de visualización de péptidos comprende un gen Cre bajo el control de un promotor ubicuo y el gen de la cápside bajo el control del promotor p41. La biblioteca se inyecta en ratones que tienen un casete loxP-STOP-loxP aguas arriba de tdTomato. Las células transducidas con variantes AAV que expresan el genoma viral y, por lo tanto, Cre expresan tdTomato y, en combinación con marcadores celulares, pueden clasificarse y seleccionarse17. Del mismo modo, Nonnenmacher et al.18 y Tabebordbar et al.19 colocaron la biblioteca de genes de la cápside bajo el control de promotores específicos de tejido. Después de la inyección en diferentes modelos animales, se utilizó ARN viral para aislar las variantes de la cápside.
Un enfoque alternativo es usar códigos de barras para etiquetar las bibliotecas de cápside. El laboratorio20 de Björklund utilizó este enfoque para las bibliotecas de cápside de inserción de péptidos de códigos de barras y desarrolló la evolución racional del vector AAV con código de barras (BRAVE). En un plásmido, el casete Rep2Cap se clona junto a un transgén marcado con código de barras que expresa repeticiones terminales invertidas (ITR) y que expresa proteínas fluorescentes amarillas (YFP). Utilizando sitios loxP entre el final de la tapa y el comienzo del código de barras, una recombinación de Cre in vitro genera un fragmento lo suficientemente pequeño para NGS, lo que permite la asociación de la inserción del péptido con el código de barras único (tabla de búsqueda, LUT). La producción de AAV se realiza utilizando la biblioteca de plásmidos y los códigos de barras expresados en el ARNm se examinan después de la aplicación in vivo , nuevamente con NGS20. Cuando las bibliotecas de cápside comprenden variantes de todo el gen de la cápside (es decir, bibliotecas barajadas), se debe utilizar la secuenciación de lectura larga. Varios grupos han utilizado códigos de barras para etiquetar estas diversas bibliotecas, lo que permite que NGS tenga una mayor profundidad de lectura. El laboratorio Kay21 etiquetó bibliotecas barajadas de cápside muy diversas con códigos de barras aguas abajo de la señal de poliA de la tapa . En un primer paso, se generó una biblioteca de plásmidos con código de barras, y la biblioteca de genes de la cápside barajada se clonó en ella. Luego se utilizó una combinación de MiSeq (lectura corta, mayor profundidad de lectura) y PacBio (lectura larga, menor profundidad de lectura), así como la secuenciación de Sanger para generar su LUT21. En 2019, Ogden y sus colegas del laboratorio Church22 delinearon la aptitud de la cápside AAV2 para múltiples funciones utilizando bibliotecas que tenían mutaciones, inserciones y eliminaciones de un solo punto en cada posición, lo que finalmente permitió el diseño guiado por máquina. Para la generación de la biblioteca, se sintetizaron fragmentos más pequeños del gen de la cápside, se marcaron con un código de barras, se secuenciaron de próxima generación y luego se clonaron en el gen completo de la cápside. Los datos de NGS se utilizaron para generar una LUT. Luego, la biblioteca se examinó utilizando solo los códigos de barras y la secuenciación de lectura corta, lo que a su vez permite una mayor profundidad de lectura22.
Las bibliotecas con código de barras se han utilizado predominantemente para examinar un conjunto de variantes conocidas, naturales y de ingeniería después de varias rondas de selección de bibliotecas de cápside o independientes de un estudio de evolución de la cápside. La ventaja de tales bibliotecas es la oportunidad de examinar múltiples cápsides, al tiempo que reduce el número de animales y minimiza la variación entre los animales. Los primeros estudios que introdujeron esta tecnología en el campo de AAV se publicaron hace casi una década. El laboratorio Nakai 12 etiquetó 191 mutantes de alanina doble que cubrían los aminoácidos 356 a 736 en el VP1 de AAV9 con un par de códigos de barras de12 nucleótidos. Utilizando NGS, la biblioteca fue examinada in vivo para la unión a galactosa y otras propiedades12. Marsic y sus colegas delinearon la biodistribución de las variantes de AAV utilizando también un análisis de doble barra 1 año después13. Un estudio más reciente en primates no humanos comparó la biodistribución en el sistema nervioso central de 29 cápsides utilizando diferentes vías de entrega23. Nuestro laboratorio ha publicado recientemente pantallas de biblioteca AAV con código de barras de 183 variantes que incluían AAV naturales y de ingeniería. Estas pantallas a nivel de ADN y ARN condujeron a la identificación de una variante AAV24 altamente miotrópica en ratones, así como otras que muestran una alta especificidad de tipo celular en el cerebro del ratón25.
Aquí, describimos la metodología utilizada en este trabajo y la ampliamos para incluir la detección de las bibliotecas de visualización de péptidos AAV. Esto comprende la generación de bibliotecas de visualización de péptidos AAV2, un método de PCR digital de gotas (dd-PCR) para la cuantificación y, finalmente, una tubería NGS para analizar las variantes de AAV, basada en parte en el trabajo de Weinmann y colegas24. Finalmente, se proporciona una descripción de la generación de bibliotecas AAV con código de barras y la canalización NGS utilizada en la misma publicación.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. AAV2 random 7-mer peptide display library preparation
NOTA: Para la preparación de una biblioteca de visualización de péptidos aleatorios AAV2, sintetice los oligonucleótidos degenerados como ADN monocatenario, conviértalo en ADN bicatenario, digiera, liga al plásmido aceptor y electroporate.
- Diseño de oligonucleótidos degenerados
- Ordene los oligonucleótidos degenerados y evite el sesgo del codón. En el oligonucleótido 5' CAGTCGGCCAG AG W GGC(X01)7 GCCCAGGCGGCTGACGAG 3', X01 corresponde a 20 codones, cada uno codificando uno de los 20 aminoácidos. La W puede ser A o T, produciendo los codones AGA o AGT, que codifican los aminoácidos arginina (R) o serina (S).
- Solicite el cebador de amplificación: 5' CTCGTCAGCCGCCTGG 3' (consulte la Figura 1 para obtener más detalles). Esto produce el siguiente inserto proteico: R/S GX 7. La diversidad teórica se calcula de la siguiente manera: 1 x 2 x 207 = 2,56 x 109 variantes únicas.
NOTA: Cabe señalar que esta diversidad puede estar restringida por la eficiencia de la transformación.
- Síntesis de segunda cadena
- Resuspender ambos oligonucleótidos (oligonucleótidos degenerados y cebador de amplificación) a una concentración final de 100 μM con tampón TE.
- Para la reacción de PCR, establezca una reacción de 50 μL con 1 μL de cada cebador, 10 μL del tampón, 1.5 μL de DMSO, 0.5 μL de dNTP (10 mM), 0.5 μL de polimerasa II de arranque en caliente de alta fidelidad y 35.5 μL de agua libre de nucleasa.
- Transfiera la reacción a un termociclador y ejecute una etapa de preincubación durante 10 s a 98 °C, seguida de tres ciclos de 10 s a 98 °C, 30 s a 59 °C y 10 s a 72 °C, luego 5 min a 72 °C y un paso final de enfriamiento.
- Purificar la reacción usando un kit de eliminación de nucleótidos y eluir en 100 μL de agua libre de nucleasas.
- Confirme la eficiencia de la síntesis de la segunda cadena mediante el análisis en un bioanalizador (ver Figura 2). Analice el tamaño y la pureza del inserto bicatenario cargando 1 μL de la reacción a un chip microfluídico de un kit de reactivos DNA 1000 de acuerdo con las instrucciones del fabricante. Este kit está optimizado para medir el tamaño y la concentración de fragmentos de ADN bicatenario de 25-1.000 bps.
- Resuspender ambos oligonucleótidos (oligonucleótidos degenerados y cebador de amplificación) a una concentración final de 100 μM con tampón TE.
- Digestión del inserto y del vector plásmido
- Digiera 85 μL del inserto purificado con 10 μL de tampón 10x y 5 μL de enzima BglI en un volumen de reacción final de 100 μL (ver Figura 1 para más detalles). Incubar a 37 °C durante la noche. Purificar usando un kit de eliminación de nucleótidos, eluye en 50 μL de agua libre de nucleasa y cuantifique usando el tipo "Oligo ADN" en un espectrofotómetro.
- Digerir 10 μg de un plásmido AAV (pRep2Cap2_PIS)26 competente para la replicación (genoma viral flanqueado por ITR) con 20 μL de tampón 10x y 10 μL de enzima SfiI en un volumen de reacción final de 200 μL (ver Figura 1 para más detalles). Incubar a 50 °C durante la noche. Purifice el vector en un gel de agarosa al 1% utilizando el kit de extracción en gel, seguido de un paso de purificación adicional con un kit purificador de ADN. Cuantificar la concentración en un espectrofotómetro.
- Ligadura del inserto al vector
- Ligate 955 ng de vector plásmido con 45 ng de inserto con 2 μL de tampón y 2 μL de ligasa en una reacción de ligadura de 20 μL. Incubar a 16 °C durante la noche, seguido de 10 minutos a 70 °C para inactivar la ligasa por calor.
- Transformación, cálculo de complejidad y preparación de bibliotecas de plásmidos
- Purificar la reacción con un kit purificador de ADN siguiendo las instrucciones del fabricante. Eluya la reacción en aproximadamente el 80% del volumen inicial de agua libre de nucleasas y guárdela en hielo para su posterior transformación.
- Transformar células electrocompetentes: descongelar un vial de células electrocompetentes en hielo durante 10 min. Luego agregue 1-2 μL de la reacción de ligadura purificada a 30 μL (un vial) de células electrocompetentes y mezcle golpeando suavemente. A continuación, pipete cuidadosamente la mezcla de células / ADN a una cubeta de electroporación de espacio de 1 mm preenfriada sin introducir burbujas de aire.
- Electroporar usando los siguientes ajustes: 1800 V, 600 Ω y 10 μF. Dentro de los 10 s del pulso de electroporación, añadir 970 μL de medios de recuperación precalentados (provistos de las celdas electrocompetentes) a la cubeta y mezclar por pipeteo. Por último, transfiera las células a un tubo de microcentrífuga e incube durante 1 h a 37 °C a 250 rpm. Para lograr una diversidad deseada, realice 10-100 reacciones y, después de la incubación, agrupe todas las reacciones en un tubo.
- Calcule la diversidad diluyendo 10 μL de las transformaciones agrupadas 10, 100 o 1,000 veces en PBS y extienda 100 μL en placas de agar nutriente que contengan el antibiótico apropiado (75 mg / ml de ampicilina). Incubar las placas de agar durante la noche a 37 °C y luego contar las colonias en las placas de agar.
- Calcule la diversidad teórica de la siguiente manera:
Diversidad máxima teórica = 10 x factor de dilución x número de colonias x número de reacciones de electroporación.
NOTA: Para confirmar la calidad de la biblioteca, secuencie al menos 20 colonias mediante la secuenciación de Sanger. La mayoría de los clones deben contener un inserto, y todos deben ser únicos. - Inocular 400-1.000 ml de medio LB que contenga el antibiótico apropiado con el resto de las transformaciones agrupadas e incubar durante la noche a 37 °C, 180 rpm.
- Preparación de la biblioteca de plásmidos
- A partir del cultivo nocturno, prepare un stock de glicerol (mezcle volúmenes iguales de cultivo bacteriano y solución de glicerol al 50% en agua libre de nucleasa y congele a -80 ° C) y purifique la biblioteca de plásmidos utilizando un kit maxi plásmido.
- Producción de la biblioteca viral AAV
- Prepare la biblioteca viral como se describió anteriormente27. Transfectar la biblioteca de plásmidos (pRep2Cap2_PI, inserto peptídico) junto con un plásmido adeno-ayudante a células HEK293T utilizando un reactivo de transfección como la polietilenimina (PEI).
- Recoger las células después de 3 días y someterlas a tres ciclos de congelación-descongelación. Purificar el lisado viral utilizando ultracentrifugación de gradiente de cloruro de cesio, seguido de intercambio de tampón a PBS, y finalmente concentrar las partículas virales.
- Titulación del vector AAV mediante dd-PCR
- Diluir en serie 2 μL de la cepa vectorial AAV en 198 μL de agua libre de nucleasas para obtener una dilución final 1:106 . Mezclar bien cada vez con una pipeta de 200 μL. Agregue un control sin plantilla (NTC) como control negativo.
NOTA: Se pueden ensayar diluciones adicionales más bajas o más altas (1:105-1:107). - Prepare una mezcla de imprimación y sonda 20x. Agregue 3,6 μL de cada uno de los cebadores de 100 μM (avance y retroceso, Rep2 e ITR), 1 μL de cada una de las sondas dd-PCR de 100 μM (Rep2 e ITR) y 3,6 μL de agua libre de nucleasa a un tubo de centrífuga de 1,5 ml.
NOTA: La biblioteca AAV se mide utilizando un conjunto de sondas de cebador dirigidas a transgenes (Rep2) detectado con una sonda marcada con FAM, y un conjunto de sondas de cebador dirigido a ITR detectado con una sonda marcada con HEX. - Prepare una reacción de PCR de 22 μL agregando 5,5 μL de muestra, 1,1 μL de mezcla de cebad-sonda 20x, 11 μL de supermezcla dd-PCR para sondas (sin dUTP) y 4,4 μL de agua libre de nucleasa. Esto produce concentraciones de 900 nM y 250 nM para los cebadores y la sonda, respectivamente.
- Genere las gotas utilizando un generador de gotas, transfiera la reacción a una placa de 96 pocillos, coloque la placa en un termociclador y ejecute un paso de desnaturalización durante 10 minutos a 94 °C, seguido de 40 ciclos de 30 s a 94 °C y 1 min a 58 °C. A continuación, inactive térmicamente la polimerasa durante 10 minutos a 98 °C y agregue un paso final de enfriamiento. Lea las reacciones en un lector de gotas y proceda al análisis28.
- Abra el archivo de placa dd-PCR guardado utilizando el software de análisis. Utilice la herramienta de umbral de la ficha Amplitud 1D (amplitud de fluorescencia frente a número de evento) para separar las gotas negativas y positivas de cada canal, utilizando el NTC como guía, y exporte los datos a un archivo csv.
- Para calcular la concentración del vector, primero calcule el factor de corrección CF utilizando la fórmula:
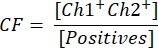
La FQ determina la proporción de gotitas positivas para el transgén [Positivos] que son positivas tanto para el transgén como para el ITR [Ch1+ Ch2+], para asegurar la detección de partículas vectoriales funcionales. La concentración vectorial final c ahora se puede calcular usando la siguiente ecuación: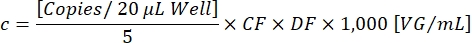
DF es el factor de dilución (1:105-1:107 como se determinó anteriormente). Las copias por reacción de 20 μL/pocillo corresponden a 5 μL de la muestra diluida. El factor 1.000 corrige la escala a VG/mL (genoma viral/mL). Un resultado ejemplar de valoración se demuestra en la Tabla 1 y la Figura 3.
- Diluir en serie 2 μL de la cepa vectorial AAV en 198 μL de agua libre de nucleasas para obtener una dilución final 1:106 . Mezclar bien cada vez con una pipeta de 200 μL. Agregue un control sin plantilla (NTC) como control negativo.
- Análisis de la biblioteca viral AAV por NGS
- Amplíe el fragmento de inserción del péptido de 96 nucleótidos configurando una reacción de PCR de 20 μL utilizando un kit de polimerasa de lectura de pruebas (2x; ver Figura 4). Añadir 1 μL de AAV que contenga 1 x 108 vg, 0,5 μL de cada uno de los 100 μM de cebador (NGS_forward y NGS_reverse), y 10 μL de la mezcla enzimática a la reacción. Ajustar el volumen final a 20 μL con agua libre de nucleasas.
- Transfiera la reacción a un termociclador y ejecute un paso de desnaturalización durante 3 min a 98 °C, seguido de 30-35 ciclos de 10 s a 98 °C, 10 s a 59 °C y 20 s a 72 °C, seguidos de 5 min a 72 °C y un paso final de enfriamiento.
- Purificar las muestras utilizando un kit de purificación por PCR. Cuantifique la concentración en un espectrofotómetro y ejecute un gel de agarosa al 3% para verificar la pureza y el tamaño del fragmento.
- Procesar los fragmentos de PCR utilizando el sistema de biblioteca para muestras de baja complejidad kit de acuerdo con las instrucciones del fabricante para la preparación de una biblioteca NGS. Realice la reacción de reparación final con 30 ng de fragmento de PCR, seguida de ligadura del adaptador y amplificación por PCR durante 10 ciclos. Utilice el kit de purificación por PCR para la purificación de las reacciones.
- Procesar los productos finales en un Bioanalizador para verificar el tamaño y la pureza, utilizando un kit de reactivos de ADN de acuerdo con las instrucciones del fabricante.
- Cuantifique los amplicones usando un fluorómetro y agruparlos. Cuantificar la biblioteca NGS final agrupada nuevamente en un fluorómetro (de acuerdo con las instrucciones del fabricante) y verificar la calidad en un Bioanalizador.
- Secuencie las bibliotecas NGS en un modo de extremo único (SE), utilizando un kit de salida alta de 75 ciclos, con una longitud de lectura de 84 y un índice de 1 de 8.
NOTA: La secuenciación de los ejemplos de este artículo se realizó en las instalaciones de GeneCore del EMBL Heidelberg (http://www.genecore.embl.de/). - Analice los datos de secuenciación NGS con Python 3 y biopython. Los archivos se pueden encontrar en https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (alternativamente en https://doi.org/10.5281/zenodo.7032215). El análisis NGS se compone de dos pasos.
- En el primer paso, busque en los archivos de secuencia secuencias que satisfagan ciertos criterios (presencia de secuencias de reconocimiento que flanquean el sitio de inserción) (consulte la Figura 4, paso 1.9.8.5.). Esto se hace utilizando un script (Script # 1) y un archivo de configuración que proporciona la información necesaria. Una vez que se identifica la secuencia correcta, el programa extrae y almacena la secuencia en el archivo de salida, que es un archivo txt con el mismo nombre que el archivo de secuenciación.
- El segundo paso es el análisis de los archivos de salida. Las secuencias en la biblioteca comienzan con cualquiera de los seis nucleótidos (AGWggc, W = A / T) en el inserto de nueve aminoácidos. Sobre la base de esta secuencia inicial, se traduce el péptido. Esto genera los archivos de salida que contienen las variantes peptídicas (PV).
- Prepare dos carpetas: Script y Data. En la carpeta Datos, copie los archivos comprimidos gzip resultantes de la secuenciación. En la carpeta Script, copie los siguientes archivos, archivo Python: Script#1_DetectionExtraction_JoVE_Py3.py; Archivo Python: Script#2_PV_extraction_and_ranking_Py3.py; Archivo de configuración: Barcode_Script_JoVE.conf; y archivo de tabla de búsqueda (LUT): Zuordnung.txt.
- Antes de ejecutar los scripts, edite los siguientes archivos en la carpeta Script. Abra el archivo "Zuordnung.txt" y agregue dos columnas separadas por tabulaciones, los nombres de los archivos gzip (columna 1) y el nombre final deseado (columna 2; valores separados por tabulaciones).
NOTA: Los archivos txt de ejemplo se encuentran en la carpeta de GitHub "PV_analysis_script". Los archivos proporcionados en la carpeta GitHub se preparan para el análisis de tres datos de ejemplo de la biblioteca anterior: xaa.txt.gz, xab.txt.gz y xac.txt.gz. También se proporcionan los archivos de salida. - Cambie las siguientes variables en el archivo de configuración "Barcode_Script_JoVE.conf":
my_dir = "~/Datos/"
filename_sample_file = "~/Script/Zuordnung.txt"
Las variables específicas de la secuencia: BCV_size = 27, BCVizquierdo = TCCAGGGCCAG, BCVderecho = GCCCAGG, BCVloc = 30,margen BCV = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC y BCVloc_revcomp = 41 (consulte la Figura 4 para obtener más detalles). - Utilice el siguiente comando para llamar a la detección y extracción de secuencias de variantes:
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
NOTA: La salida son archivos txt con las secuencias de ADN extraídas y su número de lecturas. El encabezado de este archivo contiene datos estadísticos (es decir, el número total de lecturas y las lecturas extraídas). Estos datos se transfieren a los siguientes archivos. Estos datos txt son los archivos de entrada para el Script # 2, en el que se traducen, clasifican y analizan las secuencias de ADN. - Realice la extracción y el análisis fotovoltaicos utilizando el siguiente comando:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - Analice los archivos de salida de texto del Script#2. Los archivos de salida del Script # 2 se nombran utilizando la segunda columna de la LUT en "Zuordnung.txt" con extensiones basadas en el tipo de análisis.
NOTA: Asegúrese de que los tres archivos de salida contengan datos estadísticos en las primeras filas ("# de lecturas PV válidas", "# de lecturas PV no válidas" y "# de lecturas PV únicas"), una primera columna con el índice de cada secuencia de ADN de los archivos txt de entrada (salida del script # 1) y las siguientes columnas: (1) "... analyzed_all.csv": "Muestra:" (secuencia de ADN), "#" (número de lecturas), "Frw o Rev" (lectura directa o inversa) y "PVs" (secuencia peptídica traducida). Las secuencias no válidas tienen "NA" y "no válido" en las dos últimas columnas. (2) "... analyzed_validSeq.csv": igual que el archivo anterior, filtrado por secuencias válidas. (3) "... analyzed_PV.csv": "PVs" (secuencia peptídica traducida), "#" (número de lecturas) y "count" (los recuentos frw y rev en los archivos anteriores se fusionan y el recuento recibe 1 o 2). - Visualice los archivos de salida utilizando el software disponible según las necesidades del usuario.
2. Selección aleatoria de la biblioteca de visualización de péptidos de 7 mer AAV2
- Utilice la biblioteca AAV después de la cuantificación y el control de calidad (sección 1) para la evolución dirigida en un modelo de elección para seleccionar iterativamente candidatos con propiedades deseadas (Ver Figura 5)16,18,21.
NOTA: Estos candidatos se utilizan para la generación de una biblioteca con código de barras como se describe a continuación en la sección 3.
3. Preparación y análisis de la biblioteca de cápside AAV con código de barras
NOTA: Tras la identificación de un conjunto de cápsides AAV potencialmente específicas y eficientes en la pantalla de visualización de péptidos, verifique la funcionalidad de las secuencias peptídicas identificadas y compárelas con un conjunto de variantes de cápside AAV de referencia comúnmente utilizadas o bien descritas. Para ello, la secuencia de la cápside se inserta en una construcción auxiliar Rep/Cap sin ITR.
- Producción de biblioteca AAV con código de barras
- Realizar la producción de AAV recombinante para cada variante de cápside utilizando el sistema de tres plásmidos, como se describió anteriormente24.
NOTA: Para distinguir las diferentes variantes de cápside, el plásmido transgénico reportero flanqueado por ITR alberga un código de barras único de 15 nucleótidos de longitud. El código de barras se encuentra en la 3' UTR (región no traducida) entre la proteína fluorescente amarilla mejorada (EYFP) y la señal poliA (ver Figura 6A). La expresión de EYFP es impulsada por un fuerte promotor ubicuo de citomegalovirus (CMV) que proporciona niveles suficientes de transcripciones de ARN. - Diseñar códigos de barras de 15 nucleótidos de longitud con homopolímeros de menos de tres nucleótidos, contenido de GC de <65%29 y una distancia de Hamming superior a cuatro nucleótidos24.
- Producir cada cápside por separado en combinación con un plásmido transgénico que lleva un código de barras único. De esta manera, cada variante de cápside se etiqueta con un código de barras distinto que permite su seguimiento específico (consulte la Figura 6B).
- Realizar la producción de AAV recombinante para cada variante de cápside utilizando el sistema de tres plásmidos, como se describió anteriormente24.
- Titulación del vector AAV mediante dd-PCR
- Realice la valoración AAV como se describió anteriormente en la sección 1.8, reemplazando el par de cebadores Rep2 por el par de cebadores YFP.
- Cuantifique las producciones individuales de AAV y agrupe cantidades iguales de cada producción para generar la biblioteca final con código de barras.
- Cuantifique la biblioteca final nuevamente para verificar la concentración final y la calidad (ver Figura 7).
- Aplicación in vivo de biblioteca AAV con código de barras
- Aplique la biblioteca AAV con código de barras sistémicamente al sistema modelo de elección (por ejemplo, sistémicamente en ratones24).
- Recolectar tejidos ON y OFF (es decir, hígado, pulmón, corazón, diafragma, músculo liso, duodeno, páncreas, colon, bíceps, ovarios, estómago, oído interno, riñón, aorta abdominal, aorta torácica, cerebro, grasa marrón y blanca y bazo) o tipos de células según el experimento. Congelarlos a -80 °C, extraer el ADN/ARN y aplicar el análisis de cuantificación NGS, como se describe en la siguiente sección.
- Extracción de ADN/ARN
- Extraiga el ADN y el ARN de los tejidos de interés utilizando el Mini Kit de ADN/ARN.
- Coloque una pequeña porción del tejido de interés (1 mm3, aproximadamente 5 mg) en un tubo de reacción de 2 ml.
- Agregue 350 μL de tampón de lisis mezclado con β-mercaptoetanol (1%) y perlas de acero de 5 mm al tejido (manipule muestras con β-mercaptoetanol debajo de una campana extractora).
- Homogeneizar el tejido en un tejidoLyser durante 45 s a 40 Hz.
- Añadir 10 μL de proteinasa K (10 mg/ml) e incubar durante 15 min a 55 °C mientras agitamos a 400 rpm.
- Centrifugar a 20.000 x g durante 3 min a temperatura ambiente, recoger el sobrenadante, y proceder con el protocolo del fabricante del kit de ADN/ARN.
- Divida el paso de lavado en dos pasos con 350 μL de tampón de lavado en cada paso. Entre estos pasos de lavado, digiera el ADN remanente en la columna con DNasa I libre de RNasa. Agregue 80 μL de la solución de DNasa I, preparada de acuerdo con las instrucciones del fabricante, en la columna e incube a temperatura ambiente durante 15 min.
- Eluya el ARN/ADN de la columna con agua libre de nucleasas. Almacenar el ARN aislado a -80 °C y el ADNg a -20 °C.
- Síntesis de ADNc
- Someter las muestras de ARN a otra ronda de tratamiento con DNasa I de 15-30 minutos (para la eliminación completa del ADN contaminante de las muestras de ARN) antes de la reacción de transcripción inversa. Agregue 1 μL de la solución de DNasa I, 4 μL de tampón (provisto con el kit) y agua libre de nucleasas a un volumen final de 40 μL a 212 ng de ARN. Incubar durante 30 min a temperatura ambiente e inactivar el calor a 70 °C durante 10 min.
- Sintetice cDNA, utilizando 150 ng de ARN utilizando un kit de acuerdo con las instrucciones del fabricante. Incluir controles sin la transcriptasa inversa, para asegurar la ausencia de ADN viral contaminante de la muestra. El ADNc se almacena a -20 °C.
NOTA: La cantidad de ARN de entrada para una transcripción inversa óptima puede variar según el tipo de tejido y la eficiencia de transducción esperada en el tejido respectivo.
- Análisis de la biblioteca viral AAV (in-vivo) por NGS
- Para lograr una alta profundidad de secuenciación a bajo costo, realice NGS a través de la secuenciación Illumina como se describió anteriormente (sección 1.9). Amplíe la secuencia de códigos de barras y, a continuación, vincule los adaptadores de secuenciación al amplicón.
- Debido a la corta longitud de lectura y la ligadura de los adaptadores de secuenciación en ambos lados del amplicón, al diseñar, verifique que el amplicón sea lo suficientemente pequeño como para garantizar la presencia de la secuencia de código de barras dentro de la lectura NGS. Para la secuenciación de los códigos de barras dentro de los genomas virales y las transcripciones virales, el amplicón de PCR está diseñado para tener una longitud de 113 pb (ver Figura 8).
- Amplíe la región con código de barras con los cebadores BC-seq hacia adelante y BC-seq hacia atrás. Prepare la siguiente reacción de PCR: 0,5 μL de ADN polimerasa de alta fidelidad, 10 μL de tampón 5x, 0,25 μL de cada cebador de 100 μM (BC-seq fw/BC-seq rv) y 1 μl de 10 mM dNTPs. Use 25 ng del ADNc o ADN/reacción como plantilla y ajuste el volumen final a 50 μL con agua libre de nucleasas.
- Prepare la mezcla maestra de PCR bajo una campana de PCR limpia para evitar la contaminación. Utilice las siguientes condiciones de ciclismo: 30 s a 98 °C, seguido de 40 ciclos a 98 °C durante 10 s y 72 °C durante 20 s, y un paso final de 5 minutos a 72 °C.
- Incluir controles de PCR para confirmar la ausencia de ADN contaminante en la mezcla maestra de PCR. Para las muestras de ADNc, incluya los controles sin transcriptasa inversa. Por último, incluya un ejemplo con la biblioteca de entrada AAV. Esta información se utilizará para generar el archivo Normalization_Variant.txt utilizado en el análisis.
- Verificar el tamaño del fragmento de PCR de cada muestra mediante electroforesis en gel antes de la purificación por PCR. Esto último se logra mediante el uso de perlas magnéticas disponibles comercialmente o sistemas de purificación de ADN basados en columnas (ver Tabla de materiales).
- Preparar la biblioteca NGS utilizando el sistema de biblioteca para muestras de baja complejidad de acuerdo con las instrucciones del fabricante, como se describió anteriormente en la sección 1.9.
- Determinar la concentración de ADN a través del kit dsDNA HS y analizar la calidad de la biblioteca como se describió anteriormente (sección 1.9.6), seguido de la agrupación. Cuantifique la biblioteca agrupada en un fluorómetro y evalúe la calidad en un bioanalizador.
- Realizar la secuenciación NGS como se explica en la sección 1.9.7.
- Cuantificar por qPCR el número de copias del transgén (genomas virales) y el gen de mantenimiento para evaluar la distribución de la biblioteca agrupada entre tejidos u órganos en el ADN.
- Configure una reacción qPCR de 30 μL de la siguiente manera, para determinar el número de copias de EYFP (transgén) y GAPDH (gliceraldehído 3-fosfato deshidrogenasa, gen de mantenimiento):
- Prepare una mezcla de cebador/sonda de 60x para AELP (1,5 μM YFP_fw, 1,5 μM YFP_rv y 0,6 μM YFP_probe; consulte la Tabla de materiales). Use la mezcla de cebador/sonda GAPDH (consulte la Tabla de materiales) para determinar el número de copias del gen del ama de llaves. Configura la reacción en hielo.
- Prepare una mezcla maestra de PCR (15 μL, consulte la Tabla de materiales) y agregue una mezcla de cebador/sonda 60x (0,5 μL) para todas las muestras y patrones (para calcular el número de copias de los estándares, use el siguiente enlace: http://cels.uri.edu/gsc/cndna.html). Configura la reacción en hielo.
- Transfiera 15,5 μL de la mezcla maestra a una placa de 96 pocillos y agregue 14,5 μL de muestra (75 ng de concentración total de ADN) o estándar al pocillo respectivo. Selle la placa de 96 pocillos con papel de aluminio, vórtice y gire brevemente.
- Transfiera 10 μL de cada muestra a una placa de 384 pocillos por duplicado. Sellar la placa con papel de aluminio y girar a 800 x g durante 5 min a 4 °C.
- Incubar la mezcla de reacción en un termociclador utilizando una temperatura inicial de 50 °C durante 2 min, seguida de un paso de activación inicial de 10 min a 95 °C. Realizar 40 ciclos de desnaturalización a 95 °C durante 15 s y recocido/extensión a 60 °C durante 1 min24.
- Para obtener el número de genomas diploides (dg), use el número de copia GAPDH y divida por dos. Luego, tome el valor del número de copias de EYFP y divídalo por el número de dg, lo que resulta en genomas vectoriales por genoma diploide (vg / dg). Utilice este valor para generar el archivo Normalization_Organ.txt para el análisis bioinformático.
- Realizar el análisis de los datos de secuenciación NGS como Weinmann et al.24, utilizando código personalizado en Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). El flujo de trabajo comprende la detección de secuencias de códigos de barras guiadas por secuencias de flanqueo, su longitud y ubicación (Script # 1_BarcodeDetection.py), así como el análisis del enriquecimiento y distribución de códigos de barras en el conjunto de tejidos (Script # 2_BarcodeAnalysis.py).
- Detecte códigos de barras y asígnelos a variantes AAV. Coloque los datos de secuenciación como archivos fastq archivados en un directorio (por ejemplo, "Data_to_analyze"). El archivo de datos de secuenciación para la biblioteca de entrada se incluye en este directorio y se utiliza sólo para calcular las proporciones de cápside en la biblioteca de entrada.
- Antes de ejecutar el script, cree dos archivos de texto delimitados por tabulaciones: el archivo de variantes de cápside (consulte el archivo de ejemplo "Variantes.txt") con las secuencias de códigos de barras asignadas a los nombres de variantes de cápside AAV, y el archivo de contaminación (consulte "Contaminaciones.txt") con secuencias de códigos de barras que provienen de una posible contaminación (otros códigos de barras disponibles en el laboratorio, que contribuyen a la contaminación).
- Finalmente, edite el archivo de configuración "Barcode_Script.conf" para incluir la siguiente información: ruta a la carpeta con datos de secuenciación (por ejemplo, "Data_to_analyze"), secuencia de regiones flanqueantes de los códigos de barras, su posición y tamaño de ventana para la detección de códigos de barras (similar a 1.9.8.5, consulte la Figura 8).
- Utilice el siguiente comando para solicitar la detección de códigos de barras con rutas de acceso proporcionadas a Script#1_BarcodeDetection.py y archivos de configuración:
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
NOTA: El resultado de la ejecución de Script # 1_BarcodeDetection.py son archivos de texto con recuentos de lectura por variante de cápside, así como el número total de lecturas recuperadas de los datos sin procesar. - Evalúe la distribución de cápsides AAV con código de barras entre tejidos u órganos, ejecutando Script # 2_BarcodeAnalysis.py junto con los siguientes archivos txt:
- En el archivo "Zuordnung.txt", asigne el nombre a cada archivo txt obtenido de la ejecución de detección de código de barras a un nombre de tejido/órgano: nombres de archivos txt en la primera columna y nombres de tejidos/órganos correspondientes en asignación delimitada por tabulaciones.
NOTA: Para un ejemplo, compruebe en la carpeta "Ejemplo" (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Cabe destacar que el nombre del tejido/órgano puede incluir caracteres que definan la medición de ADNc o ADNg y el número de réplicas biológicas (M1, M2, etc.). - Cree un archivo de texto "órganos.txt" con la lista de nombres para órganos ON y OFF-target, que correspondan a los nombres dados en el archivo de asignación "Zuordnung.txt" (consulte la carpeta "Ejemplo": https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- Cree archivos de texto delimitados por tabulaciones "Normalization_Organ.txt" y "Normalization_Variant.txt" con valores normalizados para todas las variantes de cápside y todos los órganos/tejidos. En la primera columna del fichero "Normalization_Organ.txt", escriban los nombres dados para cada órgano (como en el fichero de asignación "Zuordnung.txt") y en la segunda columna los valores de normalización para los tejidos correspondientes, generados en la sección 3.6.11.
- Rellene la primera columna del archivo "Normalization_Variant.txt" con la lista de nombres de cápside y la segunda columna con los valores normalizados de los recuentos de lectura para cada cápside en la biblioteca agrupada (la normalización se puede calcular en función del archivo de salida txt para la biblioteca de entrada resultante del primer script).
- Edite el archivo de configuración especificando las rutas completas a todos los archivos adicionales mencionados anteriormente. Ejecute el script #2_BarcodeAnalysis.py como:
>python3 /Script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
NOTA: El script de análisis de código de barras genera varios archivos: archivos de texto con valores de concentración relativa (RC) de la distribución de la cápside dentro de diferentes tejidos basados en múltiples pasos de normalización descritos anteriormente, y el archivo de hoja de cálculo que combina datos de archivos de texto en datos de matriz combinados. Este último se puede utilizar para el análisis y la visualización de clústeres. - Visualice los datos y realice análisis de conglomerados de los datos de la matriz para distinguir las propiedades de la cápside y evaluar sus similitudes en función de los perfiles RC entre tejidos. Utilice el script adicional PCA_heatmap_plot. R colocado en el repositorio:
>Rscript --vainilla ~/PCA. R ~/concentración relativa.xls
NOTA: El script toma archivos de concentración relativa.xls como entrada y genera dos gráficos de mapa de calor de clúster jerárquico y análisis de componentes principales (PCA). - Para modificar gráficos (ejes de mapa de calor, componentes principales de PCA) o parámetros png (color, tamaño, etiquetado), abra el script R y siga las instrucciones proporcionadas en las secciones comentadas.
- En el archivo "Zuordnung.txt", asigne el nombre a cada archivo txt obtenido de la ejecución de detección de código de barras a un nombre de tejido/órgano: nombres de archivos txt en la primera columna y nombres de tejidos/órganos correspondientes en asignación delimitada por tabulaciones.
Access restricted. Please log in or start a trial to view this content.
Resultados
Generación de una biblioteca de visualización de péptidos AAV2. Como primer paso hacia la selección de AAV diseñados, se describe la generación de una biblioteca de plásmidos. El inserto peptídico se produce mediante el uso de cebadores degenerados. Reducir la combinación de codones en aquellos de 64 a 20 tiene las ventajas de eliminar los codones de parada y facilitar el análisis NGS, al reducir la diversidad de bibliotecas en el ADN pero no en el nivel de proteína. El inserto de oligonucle?...
Access restricted. Please log in or start a trial to view this content.
Discusión
En este protocolo, se describen los pasos necesarios para la ingeniería de la cápside AAV de visualización de péptidos y para la detección de bibliotecas AAV con código de barras, así como para el análisis bioinformático de la composición de la biblioteca y el rendimiento de la cápside. Este protocolo se centra en los pasos que facilitan el análisis bioinformático de este tipo de bibliotecas, porque la mayoría de los laboratorios de virología están rezagados en las habilidades de programación para que co...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
D.G. es cofundador de AaviGen GmbH. D.G. y K.R. son inventores en una solicitud de patente pendiente relacionada con la generación de variantes de cápside AAV que evaden el sistema inmunológico. El resto de los autores no tienen nada que revelar.
Agradecimientos
D.G. agradece enormemente el apoyo de la Fundación Alemana de Investigación (DFG) a través de los Centros de Investigación Colaborativa DFG SFB1129 (Projektnummer 240245660) y TRR179 (Projektnummer 272983813), así como del Centro Alemán para la Investigación de Infecciones (DZIF, BMBF; TTU-VIH 04.819).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
Referencias
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621(2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756(2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075(2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041(2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432(2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819(2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899(2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272(2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200(2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825(2021).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados