Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Выделение векторов генной терапии следующего поколения с помощью инженерии, штрих-кодирования и скрининга вариантов капсида аденоассоциированного вируса (AAV)
В этой статье
Резюме
Генерация библиотеки пептидных дисплеев AAV и последующая проверка с помощью штрих-кодирования кандидатов с новыми свойствами для создания AAV следующего поколения.
Аннотация
Векторы доставки генов, полученные из аденоассоциированного вируса (AAV), являются одним из наиболее перспективных инструментов для лечения генетических заболеваний, о чем свидетельствуют обнадеживающие клинические данные и одобрение нескольких генных терапий AAV. Двумя основными причинами успеха векторов AAV являются (i) предварительное выделение различных встречающихся в природе вирусных серотипов с различными свойствами и (ii) последующее создание мощных технологий для их молекулярной инженерии и перепрофилирования с высокой пропускной способностью. Дальнейшим повышением потенциала этих методов являются недавно реализованные стратегии штрих-кодирования выбранных капсидов AAV на уровне ДНК и РНК, что позволяет проводить их всестороннюю и параллельную стратификацию in vivo во всех основных органах и типах клеток одного животного. Здесь мы представляем базовый конвейер, охватывающий этот набор дополнительных возможностей, используя пептидный дисплей AAV для представления разнообразного арсенала доступных технологий инженерии капсида. Соответственно, сначала мы описываем ключевые шаги по созданию библиотеки пептидных дисплеев AAV для отбора кандидатов in vivo с желаемыми свойствами, а затем демонстрируем, как штрих-кодировать наиболее интересные варианты капсида для вторичного скрининга in vivo . Далее мы проиллюстрируем методологию создания библиотек для секвенирования следующего поколения (NGS), включая амплификацию штрих-кода и лигирование адаптеров, прежде чем завершить обзор наиболее важных шагов во время анализа данных NGS. Поскольку протоколы, представленные здесь, универсальны и адаптируемы, исследователи могут легко использовать их для обогащения оптимальных вариантов капсида AAV в своей любимой модели заболевания и для применения в генной терапии.
Введение
Терапия переноса генов — это введение генетического материала в клетки для восстановления, замены или изменения клеточного генетического материала для предотвращения, лечения, лечения или улучшения заболевания. Перенос генов, как in vivo, так и ex vivo, зависит от различных систем доставки, невирусных и вирусных. Вирусы эволюционировали естественным образом, чтобы эффективно трансдуцировать свои клетки-мишени и могут использоваться в качестве векторов доставки. Среди различных типов вирусных векторов, используемых в генной терапии, все чаще используются аденоассоциированные вирусы из-за их недостаточной патогенности, безопасности, низкой иммуногенности и, что наиболее важно, их способности поддерживать долгосрочную, неинтегрирующую экспрессию 1,2,3. За последнее десятилетие генная терапия AAV принесла значительные достижения; три метода лечения были одобрены Европейским агентством по лекарственным средствам и Управлением по санитарному надзору за качеством пищевых продуктов и медикаментов США для использования у людей 3,4. Также проводится несколько клинических испытаний для лечения различных заболеваний, таких как гемофилия, мышечные, сердечные и неврологические заболевания, как рассмотрено в другом месте3. Несмотря на десятилетия прогресса, в последние годы область генной терапии пережила ряд неудач4, наиболее важным из которых были смерти в клиническихиспытаниях 5, которые были приостановлены из-за токсичности, ограничивающей дозу, особенно для тканей, которые являются массивными, такими как мышцы, или труднодоступными, такими как мозг6.
Векторы AAV, используемые в настоящее время в клинических испытаниях, относятся к естественным серотипам, за некоторыми исключениями:1. Инженерия AAV дает возможность разрабатывать векторы с превосходной органной или клеточной специфичностью и эффективностью. За последние два десятилетия было успешно применено несколько подходов, таких как пептидный дисплей, замена петли, перетасовка капсидной ДНК, подверженная ошибкам ПЦР и целевой дизайн, для создания отдельных вариантов AAV или их библиотек с различными свойствами7. Затем они подвергаются нескольким раундам направленной эволюции для выбора вариантов внутри них с желаемыми свойствами, как рассмотрено в другом месте 1,3. Из всех стратегий эволюции капсидов наиболее широко используются библиотеки AAV пептидных дисплеев благодаря некоторым уникальным свойствам: их относительно легко генерировать, и они могут достигать высокого разнообразия и высокой пропускной способности секвенирования, что позволяет отслеживать их эволюцию.
Первые успешные пептидные библиотеки AAV были описаны почти 20 лет назад. В одном из первых Perabo et al.8 построили библиотеку модифицированных капсидов AAV2, в которой пул случайно сгенерированных олигонуклеотидов был вставлен в плазмиду в положении, соответствующем аминокислоте 587 капсидного белка VP1, в тройной оси, выступающей из капсида. Используя коинфекцию аденовируса, библиотека AAV была разработана путем нескольких раундов отбора, и было показано, что окончательные ретаргетированные варианты способны трансдуцировать клеточные линии, рефрактерные к родительскому AAV28. Вскоре после этого Мюллер и др.9 представили двухступенчатую систему библиотечного производства, что значительно улучшило протокол. Первоначально библиотека плазмид вместе с аденовирусной вспомогательной плазмидой используется для получения библиотеки AAV, содержащей химерные капсиды. Эта челночная библиотека AAV используется для заражения клеток с низкой кратностью инфекции (MOI) с целью введения одного вирусного генома на клетку. Коинфекция аденовирусом обеспечивает выработку AAV с совпадающим геномом и капсидом9. Примерно десять лет спустя Dalkara10 использовала направленную эволюцию in vivo для создания варианта 7m8. Этот вариант имеет вставку 10 аминокислот (LALGETTRPA), три из которых действуют как линкеры и эффективно нацелены на внешнюю сетчатку после интравитреальной инъекции10. Этот инженерный капсид является исключительной историей успеха, так как это один из немногих инженерных капсидов, попавших в клинику на данный момент11.
Эта область пережила второй импульс с внедрением методов секвенирования следующего поколения (NGS). Две публикации от Adachi et al.12 в 2014 году и от Marsic et al.13 в 2015 году продемонстрировали возможности NGS для отслеживания распределения библиотек капсидов AAV со штрих-кодом с высокой точностью. Несколько лет спустя NGS областей со штрих-кодом был адаптирован к области введения пептидов, чтобы проследить эволюцию капсида. Körbelin et al.14 провели скрининг под контролем NGS для выявления капсида на основе AAV2, нацеленного на легкие. Анализ NGS помог рассчитать три рейтинговых балла: балл обогащения между отборочными раундами, общий балл специфичности для определения тканевой специфичности и, наконец, комбинированный балл14. В том же году лабораторияGradinaru 15 опубликовала систему целевой эволюции AAV (CREATE) на основе Cre-рекомбинации, которая облегчает отбор по типу клеток. В этой системе капсидная библиотека несет Cre-инвертируемый переключатель, так как сигнал polyA окружен двумя сайтами loxP. Затем библиотеку AAV вводят мышам Cre, где сигнал polyA инвертируется только в клетках Cre+, обеспечивая матрицу для связывания праймера обратной ПЦР с прямым праймером в гене капсида. Это высокоспецифическое спасение ПЦР позволило идентифицировать AAV-PHP. Вариант B, который может пересекать гематоэнцефалический барьер15. Эта система получила дальнейшее развитие в M-CREATE (Multiplexed-CREATE), в которой NGS и генерация синтетических библиотек были интегрированы в конвейер16.
Улучшенная версия этой системы на основе РНК из лабораторииМагуайра 17, iTransduce, позволяет отбирать на уровне ДНК капсиды, которые функционально трансдуцируют клетки и экспрессируют их геномы. Вирусный геном библиотеки пептидного дисплея включает ген Cre под контролем вездесущего промотора и ген капсида под контролем промотора p41. Библиотека вводится мышам, у которых есть кассета loxP-STOP-loxP перед tdTomato. Клетки, трансдуцированные вариантами AAV, которые экспрессируют вирусный геном и, следовательно, Cre экспрессируют tdTomato и в сочетании с клеточными маркерами могут быть отсортированы и отобраны17. Аналогичным образом, Nonnenmacher et al.18 и Tabebordbar et al.19 поместили библиотеку генов капсида под контроль тканеспецифических промоторов. После инъекции на разных животных моделях вирусная РНК использовалась для выделения вариантов капсида.
Альтернативным подходом является использование штрих-кодирования для маркировки библиотек капсидов. Лаборатория20 Бьорклунда использовала этот подход к библиотекам капсидов для вставки пептидов штрих-кода и разработала рациональную эволюцию вектора AAV со штрих-кодом (BRAVE). В одной плазмиде кассета Rep2Cap клонируется рядом с трансгеном с инвертированным концевым повтором (ITR), экспрессирующим желтый флуоресцентный белок (YFP), помеченным штрих-кодом. Используя сайты loxP между концом колпачка и началом штрих-кода, рекомбинация Cre in vitro генерирует фрагмент, достаточно маленький для NGS, тем самым позволяя ассоциировать вставку пептида с уникальным штрих-кодом (справочная таблица, LUT). Производство AAV осуществляется с использованием библиотеки плазмид, а штрих-коды, экспрессированные в мРНК, проверяются после применения in vivo , опять же с помощью NGS20. Когда библиотеки капсида содержат варианты всего гена капсида (т.е. перетасованные библиотеки), необходимо использовать секвенирование с длинным чтением. Несколько групп использовали штрих-коды для маркировки этих разнообразных библиотек, что позволяет NGS иметь более высокую глубину считывания. ЛабораторияКея 21 пометила очень разнообразные перетасованные капсидные библиотеки штрих-кодами после сигнала cap polyA. На первом этапе была сгенерирована библиотека плазмид со штрих-кодом, и в нее была клонирована перетасованная библиотека генов капсида. Затем для генерации LUT21 была использована комбинация MiSeq (короткое чтение, более высокая глубина чтения) и PacBio (длинное чтение, меньшая глубина чтения), а также секвенирование Сэнгера. В 2019 году Огден и его коллеги из лабораторииЧерча 22 определили пригодность капсида AAV2 для нескольких функций, используя библиотеки, которые имели одноточечные мутации, вставки и делеции в каждой позиции, что в конечном итоге позволило использовать машинно-управляемое проектирование. Для генерации библиотеки были синтезированы меньшие фрагменты гена капсида, помечены штрих-кодом, секвенированы следующего поколения, а затем клонированы в ген полного капсида. Данные NGS были использованы для создания LUT. Затем библиотека была проверена с использованием только штрих-кодов и короткой последовательности считывания, что, в свою очередь, обеспечивает более высокую глубину считывания22.
Библиотеки со штрих-кодом в основном использовались для скрининга пула известных, естественных и инженерных вариантов после нескольких раундов отбора библиотек капсида или независимо от исследования эволюции капсида. Преимуществом таких библиотек является возможность скрининга нескольких капсидов, при этом сокращая количество животных и сводя к минимуму различия между животными. Первые исследования, которые представили эту технологию в области AAV, были опубликованы почти десять лет назад. Лаборатория Накаи 12 пометила 191 двойного аланинового мутанта, охватывающего аминокислоты с 356 по 736 на VP1 из AAV9 парой 12-нуклеотидных штрих-кодов. С помощью NGS библиотека была проверена in vivo на связывание галактозы и другие свойства12. Марсик и его коллеги очертили биораспределение вариантов AAV, используя также анализ с двойным шнуром 1 год спустя13. В более позднем исследовании, проведенном на нечеловекообразных приматах, сравнивалось биораспределение в центральной нервной системе 29 капсидов с использованием различных путей доставки23. Наша лаборатория недавно опубликовала библиотечные экраны AAV со штрих-кодом из 183 вариантов, которые включали натуральные и инженерные AAV. Эти скрининги на уровне ДНК и РНК привели к идентификации высокомиотропного вариантаAAV 24 у мышей, а также других, демонстрирующих высокую специфичность клеточного типа в мозгемыши 25.
Здесь мы описываем методологию, используемую в этой работе, и расширяем ее, включая скрининг библиотек пептидных дисплеев AAV. Это включает в себя создание библиотек пептидных дисплеев AAV2, метод цифровой капельной ПЦР (dd-PCR) для количественной оценки и, наконец, конвейер NGS для анализа вариантов AAV, частично основанный на работе Вайнманна и его коллег24. Наконец, приводится описание генерации библиотек AAV со штрих-кодом и конвейера NGS, используемых в той же публикации.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Подготовка библиотеки случайных 7-мерных пептидных дисплеев AAV2
ПРИМЕЧАНИЕ: Для получения библиотеки дисплея случайных пептидов AAV2 синтезируйте вырожденные олигонуклеотиды в виде одноцепочечной ДНК, преобразуйте ее в двухцепочечную ДНК, расщепляйте, связывайте с акцепторной плазмидой и электропорат.
- Конструирование вырожденных олигонуклеотидов
- Упорядочивайте вырожденные олигонуклеотиды и избегайте смещения кодонов. В олигонуклеотиде 5' CAGTCGGCCAG AG W GGC (X01)7 GCCCAGGCGGCTGACGAG 3', X01 соответствует 20 кодонам, каждый из которых кодирует одну из 20 аминокислот. W может быть A или T, продуцируя кодоны AGA или AGT, которые кодируют аминокислоты аргинин (R) или серин (S).
- Закажите праймер усиления: 5' CTCGTCAGCCGCCTGG 3' (подробнее см. Рисунок 1 ). При этом образуется следующая белковая вставка: R / S GX 7. Теоретическое разнообразие рассчитывается следующим образом: 1 х 2 х 207 = 2,56 х 109 уникальных вариантов.
ПРИМЕЧАНИЕ: Следует отметить, что это разнообразие может быть ограничено эффективностью трансформации.
- Синтез второй цепи
- Ресуспендируют оба олигонуклеотида (вырожденные олигонуклеотиды и праймер амплификации) до конечной концентрации 100 мкМ с буфером TE.
- Для реакции ПЦР установите реакцию 50 мкл с 1 мкл каждого праймера, 10 мкл буфера, 1,5 мкл ДМСО, 0,5 мкл dNTP (10 мМ), 0,5 мкл высокоточной полимеразы горячего старта II и 35,5 мкл воды, не содержащей нуклеаз.
- Перенесите реакцию в термоциклер и запустите стадию предварительной инкубации в течение 10 с при 98 °C, затем три цикла по 10 с при 98 °C, 30 с при 59 °C и 10 с при 72 °C, затем 5 мин при 72 °C и заключительную стадию охлаждения.
- Очистите реакцию с помощью набора для удаления нуклеотидов и разбавьте 100 мкл воды, не содержащей нуклеаз.
- Подтвердите эффективность синтеза второй цепи анализом на биоанализаторе (см. рис. 2). Проанализируйте размер и чистоту двухцепочечной вставки, загрузив 1 мкл реакции в микрофлюидный чип из набора реагентов DNA 1000 в соответствии с инструкциями производителя. Этот набор оптимизирован для измерения размера и концентрации двухцепочечных фрагментов ДНК в диапазоне от 25 до 1000 бит в секунду.
- Ресуспендируют оба олигонуклеотида (вырожденные олигонуклеотиды и праймер амплификации) до конечной концентрации 100 мкМ с буфером TE.
- Расщепление вставки и плазмидного вектора
- Расщепите 85 мкл очищенной вставки с 10 мкл 10-кратного буфера и 5 мкл фермента BglI в конечном реакционном объеме 100 мкл (подробнее см. Рисунок 1 ). Инкубировать при температуре 37 °C в течение ночи. Очистите с помощью набора для удаления нуклеотидов, растворите в 50 мкл воды, не содержащей нуклеаз, и количественно определите с помощью типа «олиго-ДНК» в спектрофотометре.
- Расщепите 10 мкг репликационно-компетентной плазмиды AAV (pRep2Cap2_PIS)26 (вирусный геном с фланкированным ITR) с 20 мкл 10-кратного буфера и 10 мкл фермента SfiI в конечном реакционном объеме 200 мкл (подробнее см. Рисунок 1 ). Инкубировать при температуре 50 °C в течение ночи. Очистите вектор на 1% агарозном геле с помощью набора для экстракции геля с последующим дополнительным этапом очистки с использованием набора для очистки ДНК. Количественно определите концентрацию в спектрофотометре.
- Перевязка вставки в вектор
- Лигат 955 нг плазмидного вектора с 45 нг вставки с 2 мкл буфера и 2 мкл лигазы в реакции лигирования 20 мкл. Инкубировать при 16 ° C в течение ночи, а затем 10 минут при 70 ° C для инактивации лигазы при нагревании.
- Преобразование, расчет сложности и подготовка библиотеки плазмид
- Очистите реакцию с помощью набора для очистки ДНК, следуя инструкциям производителя. Растворяют реакцию примерно в 80% исходного объема безнуклеазной воды и хранят на льду для последующего превращения.
- Трансформация электрокомпетентных ячеек: разморозьте один флакон электрокомпетентных ячеек на льду в течение 10 минут. Затем добавьте 1-2 мкл очищенной реакции лигирования в 30 мкл (один флакон) электрокомпетентных клеток и перемешайте, осторожно постукивая. Затем осторожно нанесите смесь клеток/ДНК пипеткой в предварительно охлажденную кювету для электропорации с зазором 1 мм без введения пузырьков воздуха.
- Электропорация с использованием следующих настроек: 1800 В, 600 Ω и 10 мкФ. В течение 10 с после импульса электропорации добавьте в кювету 970 мкл предварительно нагретой восстановительной среды (поставляемой с электрокомпетентными ячейками) и перемешайте пипеткой. Наконец, перенесите клетки в микроцентрифужную пробирку и инкубируйте в течение 1 ч при 37 ° C при 250 об/мин. Чтобы добиться желаемого разнообразия, выполните 10-100 реакций, а после инкубации объедините все реакции в одну пробирку.
- Рассчитайте разнообразие, разбавив 10 мкл объединенных превращений в 10, 100 или 1000 раз в PBS и распределив 100 мкл на пластинах с питательным агаром, содержащих соответствующий антибиотик (75 мг/мл ампициллина). Инкубируйте агаровые пластины в течение ночи при 37 ° C, а затем подсчитайте колонии на агаровых пластинах.
- Рассчитайте теоретическое разнообразие следующим образом:
Теоретическое максимальное разнообразие = 10 х коэффициент разбавления х количество колоний х число реакций электропорации.
ПРИМЕЧАНИЕ: Чтобы подтвердить качество библиотеки, секвенируйте не менее 20 колоний с помощью секвенирования Сэнгера. Большинство клонов должны содержать вставку, и все они должны быть уникальными. - Инокулируют 400-1000 мл среды LB, содержащей соответствующий антибиотик, с остальными объединенными превращениями и инкубируют в течение ночи при 37 ° C, 180 об/мин.
- Подготовка плазмидной библиотеки
- Из ночной культуры приготовьте глицериновый бульон (смешайте равные объемы бактериальной культуры и 50% раствора глицерина в воде без нуклеаз и заморозьте при -80 ° C) и очистите библиотеку плазмид с помощью набора плазмид макси.
- Производство вирусной библиотеки AAV
- Подготовьте вирусную библиотеку, как описаноранее 27. Трансфицируйте плазмидную библиотеку (pRep2Cap2_PI, пептидную вставку) вместе с аденохелперной плазмидой в клетки HEK293T с помощью трансфекционного реагента, такого как полиэтиленимин (PEI).
- Соберите клетки через 3 дня и подвергните их трем циклам замораживания-оттаивания. Очистите вирусный лизат с помощью ультрацентрифугирования с градиентом хлорида цезия с последующим буферным обменом на PBS и, наконец, сконцентрируйте вирусные частицы.
- Векторное титрование AAV с использованием dd-PCR
- Последовательно разбавляют 2 мкл векторного запаса AAV в 198 мкл воды, не содержащей нуклеаз, для получения окончательного разбавления 1:106 . Каждый раз тщательно перемешивайте с помощью пипетки объемом 200 мкл. Добавьте один элемент управления без шаблона (NTC) в качестве отрицательного элемента управления.
ПРИМЕЧАНИЕ: Могут быть проанализированы дополнительные более низкие или более высокие разведения (1:105-1:107). - Приготовьте 20-кратную смесь грунтовки и зонда. Добавьте по 3,6 мкл каждого из 100 мкМ праймеров (прямой и обратный, Rep2 и ITR), по 1 мкл каждого из 100 мкМ зондов dd-PCR (Rep2 и ITR) и 3,6 мкл безнуклеазной воды в центрифужную пробирку объемом 1,5 мл.
ПРИМЕЧАНИЕ: Библиотека AAV измеряется с использованием набора праймер-зондов, нацеленных на трансгены (Rep2), обнаруженного с помощью зонда, меченного FAM, и набора праймер-зондов, нацеленного на ITR, обнаруженного с помощью зонда, меченного HEX. - Приготовьте реакцию ПЦР 22 мкл, добавив 5,5 мкл образца, 1,1 мкл 20-кратной смеси праймер-зонд, 11 мкл суперсмеси dd-PCR для зондов (без dUTP) и 4,4 мкл воды, не содержащей нуклеаз. Это дает концентрации 900 нМ и 250 нМ для праймеров и зонда соответственно.
- Сгенерируйте капли с помощью генератора капель, перенесите реакцию на 96-луночную пластину, поместите пластину в термоциклер и запустите стадию денатурации в течение 10 минут при 94 ° C, а затем 40 циклов по 30 с при 94 ° C и 1 минуту при 58 ° C. Затем термоинактивируйте полимеразу в течение 10 мин при 98 ° C и добавьте последнюю стадию охлаждения. Прочтите реакции в считывателе капель и приступайте к анализу28.
- Откройте сохраненный файл планшета dd-PCR с помощью программного обеспечения для анализа. Используйте инструмент «Порог» на вкладке «Амплитуда 1D» (амплитуда флуоресценции в зависимости от номера события), чтобы разделить отрицательные и положительные капли для каждого канала, используя NTC в качестве ориентира, и экспортируйте данные в CSV-файл.
- Чтобы рассчитать векторную концентрацию, сначала рассчитайте поправочный коэффициент CF по формуле:
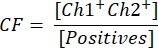
CF определяет долю капель, положительных для трансгена [Positives], которые являются положительными как для трансгена, так и для ITR [Ch1+ Ch2+], чтобы обеспечить обнаружение функциональных векторных частиц. Конечная векторная концентрация c теперь может быть рассчитана с помощью следующего уравнения: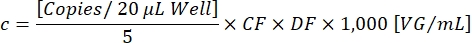
DF - коэффициент разбавления (1:105-1:107 , как определено ранее). Количество копий на реакцию 20 мкл соответствует 5 мкл разбавленного образца. Фактор 1,000 корректирует шкалу до VG / мл (вирусный геном / мл). Примерный результат титрования показан в таблице 1 и на рисунке 3.
- Последовательно разбавляют 2 мкл векторного запаса AAV в 198 мкл воды, не содержащей нуклеаз, для получения окончательного разбавления 1:106 . Каждый раз тщательно перемешивайте с помощью пипетки объемом 200 мкл. Добавьте один элемент управления без шаблона (NTC) в качестве отрицательного элемента управления.
- Анализ вирусной библиотеки AAV методом NGS
- Амплифицируйте фрагмент вставки 96-нуклеотидного пептида, настроив ПЦР-реакцию 20 мкл с использованием набора полимеразы для корректуры (2x; см. рис. 4). Добавьте в реакцию 1 мкл запаса AAV, содержащего 1 x 108 мкг, по 0,5 мкл каждого из 100 мкМ праймера (NGS_forward и NGS_reverse) и 10 мкл смеси ферментов. Отрегулируйте конечный объем до 20 мкл с водой, не содержащей нуклеаз.
- Перенесите реакцию в термоциклер и запустите стадию денатурации в течение 3 мин при 98 °C, затем 30-35 циклов по 10 с при 98 °C, 10 с при 59 °C и 20 с при 72 °C, затем 5 мин при 72 °C и заключительную стадию охлаждения.
- Очистите образцы с помощью набора для очистки ПЦР. Количественно определите концентрацию в спектрофотометре и запустите 3% агарозный гель для проверки чистоты и размера фрагмента.
- Обработайте фрагменты ПЦР с помощью библиотечной системы для набора образцов низкой сложности в соответствии с инструкциями производителя по подготовке библиотеки NGS. Проведите реакцию репарации конца с 30 нг фрагмента ПЦР с последующим лигированием адаптера и амплификацией ПЦР в течение 10 циклов. Используйте набор для очистки ПЦР для очистки реакций.
- Обработайте конечные продукты на биоанализаторе, чтобы проверить размер и чистоту, используя набор реагентов ДНК в соответствии с инструкциями производителя.
- Количественно оцените ампликоны с помощью флуорометра и объедините их. Снова количественно оцените окончательную объединенную библиотеку NGS на флуорометре (в соответствии с инструкциями производителя) и проверьте качество на биоанализаторе.
- Секвенируйте библиотеки NGS в одностороннем (SE) режиме, используя 75-тактный комплект с высокой производительностью, с длиной чтения 84 и индексом 1 из 8.
ПРИМЕЧАНИЕ: Секвенирование примеров в этой статье было выполнено на установке GeneCore EMBL Heidelberg (http://www.genecore.embl.de/). - Анализируйте данные секвенирования NGS с помощью Python 3 и biopython. Файлы можно найти по адресу https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (в качестве альтернативы по адресу https://doi.org/10.5281/zenodo.7032215). Анализ NGS состоит из двух этапов.
- На первом шаге выполните поиск в файлах последовательностей последовательностей, удовлетворяющих определенным критериям (наличие последовательностей распознавания, расположенных по бокам от места вставки) (см. рис. 4, этап 1.9.8.5.). Это делается с помощью скрипта (Script#1) и файла конфигурации, который предоставляет необходимую информацию. После того, как правильная последовательность определена, программа извлекает и сохраняет последовательность в выходном файле, который представляет собой текстовый файл с тем же именем, что и файл виртуализации.
- Вторым шагом является анализ выходных файлов. Последовательности в библиотеке начинаются с любого из шести нуклеотидов (AGWggc, W = A / T) в девяти аминокислотных вставках. На основе этой стартовой последовательности транслируется пептид. При этом генерируются выходные файлы, содержащие варианты пептидов (PV).
- Подготовьте две папки: «Сценарий» и «Данные». Скопируйте сжатые gzip-файлы, полученные в результате виртуализации, в папку Data. В папку Script скопируйте следующие файлы, файл Python: Script#1_DetectionExtraction_JoVE_Py3.py; Файл Python: Script#2_PV_extraction_and_ranking_Py3.py; Конфигурационный файл: Barcode_Script_JoVE.conf; и файл таблицы подстановки (LUT): Zuordnung.txt.
- Перед запуском сценариев отредактируйте следующие файлы в папке «Сценарий». Откройте файл «Zuordnung.txt» и добавьте в два столбца, разделенных табуляцией, имена файлов gzip (столбец 1) и желаемое конечное имя (столбец 2; значения, разделенные табуляцией).
ПРИМЕЧАНИЕ: Примеры текстовых файлов находятся в папке GitHub "PV_analysis_script". Файлы, предоставленные в папке GitHub, подготовлены для анализа трех примеров данных из приведенной выше библиотеки: xaa.txt.gz, xab.txt.gz и xac.txt.gz. Выходные файлы также предоставляются. - Измените следующие переменные в конфигурационном файле "Barcode_Script_JoVE.conf":
my_dir = "~/Данные/"
filename_sample_file = "~/Script/Zuordnung.txt"
Переменные, зависящие от последовательности: BCV_size = 27,BCV слева = TCCAGGGCCAG, BCVсправа = GCCCAGG, BCVloc = 30,маржа BCV = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC и BCVloc_revcomp = 41 (подробнее см. Рисунок 4 ). - Используйте следующую команду для вызова обнаружения и извлечения последовательности вариантов:
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
ПРИМЕЧАНИЕ: На выходе получаются текстовые файлы с извлеченными последовательностями ДНК и их количеством прочтений. Заголовок этого файла содержит статистические данные (т.е. общее количество прочтений и извлеченных чтений). Эти данные переносятся в следующие файлы. Эти текстовые данные являются входными файлами для Script#2, в которых последовательности ДНК переводятся, ранжируются и анализируются. - Выполните извлечение и анализ PV с помощью следующей команды:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - Проанализируйте текстовые выходные файлы Script#2. Выходные файлы Script#2 именуются с использованием второго столбца LUT в "Zuordnung.txt" с расширениями, основанными на типе анализа.
ПРИМЕЧАНИЕ: Убедитесь, что три выходных файла содержат статистические данные в первых строках («# допустимых считываний PV», «# недопустимых считываний PV» и «# уникальных считываний PV»), первом столбце с индексом каждой последовательности ДНК из входных текстовых файлов (вывод сценария # 1) и следующих столбцах: (1) «... analyzed_all.csv": "Образец:" (последовательность ДНК), "#" (количество считываний), "Frw или Rev" (прямое или обратное считывание) и "PVs" (переведенная пептидная последовательность). Недопустимые последовательности имеют «NA» и «недопустимо» в последних двух столбцах. (2) "... analyzed_validSeq.csv": то же, что и предыдущий файл, отфильтрованный по допустимым последовательностям. (3) "... analyzed_PV.csv": "PVs" (переведенная пептидная последовательность), "#" (количество прочтений) и "count" (счетчики frw и rev в предыдущих файлах объединяются, и количество дается 1 или 2). - Визуализируйте выходные файлы с помощью доступного программного обеспечения в зависимости от потребностей пользователя.
2. Случайный выбор библиотеки 7-мерных пептидных дисплеев AAV2
- Используйте библиотеку AAV после количественной оценки и контроля качества (раздел 1) для направленной эволюции в выбранной модели для итеративного отбора кандидатов с желаемыми свойствами (см. рис. 5)16,18,21.
ПРИМЕЧАНИЕ: Эти кандидаты затем используются для создания библиотеки штрих-кодов, как описано ниже в разделе 3.
3. Подготовка и анализ библиотеки капсидов AAV со штрих-кодом
ПРИМЕЧАНИЕ: После идентификации набора потенциально специфических и эффективных капсидов AAV на экране пептидного дисплея проверьте функциональность идентифицированных пептидных последовательностей и сравните их с набором широко используемых или хорошо описанных эталонных вариантов капсида AAV. Для этого последовательность капсидов вставляется во вспомогательную конструкцию Rep/Cap без РМЭ.
- Изготовление библиотеки AAV со штрих-кодом
- Продуцируют рекомбинантный AAV для каждого варианта капсида с использованием трехплазмидной системы, как описано ранее24.
ПРИМЕЧАНИЕ: Чтобы различать различные варианты капсида, репортерная трансгенная плазмида, окруженная ITR, содержит уникальный штрих-код длиной 15 нуклеотидов. Штрих-код расположен в 3' UTR (нетранслируемой области) между усиленным желтым флуоресцентным белком (EYFP) и сигналом polyA (см. рис. 6A). Экспрессия EYFP обусловлена сильным вездесущим промотором цитомегаловируса (ЦМВ), который обеспечивает достаточный уровень транскриптов РНК. - Дизайн штрих-кодов длиной 15 нуклеотидов с гомополимерами менее трех нуклеотидов, содержанием GC <65%29 и расстоянием Хэмминга более четырех нуклеотидов24.
- Производят каждый капсид отдельно в сочетании с трансгенной плазмидой, несущей уникальный штрих-код. Таким образом, каждый вариант капсида помечается отдельным штрих-кодом, который позволяет отслеживать его (см. рис. 6B).
- Продуцируют рекомбинантный AAV для каждого варианта капсида с использованием трехплазмидной системы, как описано ранее24.
- Векторное титрование AAV с использованием dd-PCR
- Выполните титрование AAV, как описано ранее в разделе 1.8, заменив пару праймеров Rep2 парой праймеров YFP.
- Количественно оцените отдельные производства AAV и объедините равные количества каждого производства для создания окончательной библиотеки штрих-кодов.
- Еще раз количественно оцените окончательную библиотеку, чтобы проверить конечную концентрацию и качество (см. рис. 7).
- Библиотека AAV со штрих-кодом в приложении vivo
- Системно примените библиотеку AAV со штрих-кодом к выбранной модельной системе (например, системно у мышей24).
- Соберите ткани-мишени (например, печень, легкие, сердце, диафрагма, гладкие мышцы, двенадцатиперстная кишка, поджелудочная железа, толстая кишка, бицепсы, яичники, желудок, внутреннее ухо, почки, брюшная аорта, грудная аорта, мозг, бурый и белый жир и селезенка) или типы клеток на основе эксперимента. Заморозьте их при -80 °C, извлеките ДНК/РНК и примените количественный анализ NGS, как описано в следующем разделе.
- Экстракция ДНК/РНК
- Извлеките ДНК и РНК из интересующих тканей с помощью мини-набора ДНК/РНК.
- Поместите небольшой кусочек интересующей ткани (1 мм3, около 5 мг) в реакционную пробирку объемом 2 мл.
- Добавьте в ткань 350 мкл лизисного буфера, смешанного с β-меркаптоэтанолом (1%) и стальными шариками толщиной 5 мм (обрабатывайте образцы с β-меркаптоэтанолом под вытяжным шкафом).
- Гомогенизируйте ткань в салфеткеLyser в течение 45 с при 40 Гц.
- Добавьте 10 мкл протеиназы К (10 мг/мл) и инкубируйте в течение 15 мин при 55 ° C, встряхивая при 400 об/мин.
- Центрифугу при 20 000 x g в течение 3 мин при комнатной температуре, соберите надосадочную жидкость и следуйте протоколу производителя набора ДНК/РНК.
- Разделите этап стирки на два этапа с 350 мкл буфера для стирки на каждом этапе. В промежутках между этими этапами промывки расщепляют остаточную ДНК на колонке с помощью ДНКазы I, не содержащей РНКазы. Добавьте 80 мкл раствора ДНКазы I, приготовленного в соответствии с инструкцией производителя, в колонку и инкубируйте при комнатной температуре в течение 15 мин.
- Элютировать РНК/ДНК из колонки с безнуклеазной водой. Храните выделенную РНК при -80 ° C, а гДНК - при -20 ° C.
- Синтез кДНК
- Подвергните образцы РНК еще одному раунду обработки ДНКазой I в течение 15-30 минут (для полного удаления загрязняющей ДНК из образцов РНК) перед реакцией обратной транскрипции. Добавьте 1 мкл раствора ДНКазы I, 4 мкл буфера (входит в комплект) и воду без нуклеаз в конечный объем 40 мкл на 212 нг РНК. Инкубировать в течение 30 минут при комнатной температуре и инактивировать при температуре 70 °C в течение 10 минут.
- Синтезируйте кДНК, используя 150 нг РНК с помощью набора в соответствии с инструкциями производителя. Включите контрольную группу без обратной транскриптазы, чтобы убедиться в отсутствии загрязняющей вирусной ДНК в образце. КДНК хранится при -20 °C.
ПРИМЕЧАНИЕ: Количество входной РНК для оптимальной обратной транскрипции может варьироваться в зависимости от типа ткани и ожидаемой эффективности трансдукции в соответствующей ткани.
- Анализ вирусной библиотеки AAV (in-vivo) от NGS
- Чтобы достичь высокой глубины секвенирования при низких затратах, выполните NGS с помощью секвенирования Illumina, как описано ранее (раздел 1.9). Усильте последовательность штрих-кода, а затем соедините адаптеры последовательности с ампликоном.
- Из-за короткой длины считывания и лигирования адаптеров секвенирования с обеих сторон ампликона при проектировании убедитесь, что ампликон достаточно мал, чтобы обеспечить наличие последовательности штрих-кода в считывании NGS. Для секвенирования штрих-кодов в вирусных геномах и вирусных транскриптов ампликон ПЦР рассчитан на длину 113.н. (см. рис. 8).
- Усильте область штрих-кода с помощью праймеров BC-seq вперед и BC-seq назад. Приготовьте следующую реакцию ПЦР: 0,5 мкл ДНК-полимеразы высокой точности, 10 мкл 5-кратного буфера, 0,25 мкл каждого 100 мкМ праймера (BC-seq fw/BC-seq rv) и 1 мкл 10 мМ dNTP. Используйте 25 нг кДНК или ДНК/реакции в качестве матрицы и отрегулируйте конечный объем до 50 мкл с водой, не содержащей нуклеаз.
- Приготовьте мастер-микс для ПЦР под чистым колпаком для ПЦР, чтобы избежать заражения. Используйте следующие условия циклирования: 30 с при 98 °C, затем 40 циклов при 98 °C в течение 10 с и 72 °C в течение 20 с и заключительный 5-минутный шаг при 72 °C.
- Включите ПЦР-контроль, чтобы подтвердить отсутствие загрязняющей ДНК в мастер-миксе ПЦР. Для образцов кДНК включите контрольную группу без обратной транскриптазы. Наконец, включите пример с входной библиотекой AAV. Эта информация будет использована для создания файла Normalization_Variant.txt, используемого в анализе.
- Проверьте размер ПЦР-фрагмента каждого образца с помощью гель-электрофореза перед очисткой ПЦР. Последнее достигается за счет использования либо коммерчески доступных магнитных шариков, либо систем очистки ДНК на основе колонок (см. Таблицу материалов).
- Подготовьте библиотеку NGS с помощью библиотечной системы для образцов низкой сложности в соответствии с инструкциями производителя, как описано ранее в разделе 1.9.
- Определите концентрацию ДНК с помощью набора dsDNA HS и проанализируйте качество библиотеки, как описано ранее (раздел 1.9.6), с последующим объединением. Количественно оцените объединенную библиотеку на флуорометре и оцените качество на биоанализаторе.
- Выполните секвенирование NGS, как описано в разделе 1.9.7.
- Количественно определите с помощью кПЦР количество копий трансгена (вирусных геномов) и гена, чтобы оценить распределение объединенной библиотеки между тканями или органами на ДНК.
- Настройте реакцию 30 мкл кПЦР следующим образом, чтобы определить число копий EYFP (трансген) и GAPDH (глицеральдегид-3-фосфатдегидрогеназа, ген домашнего хозяйства):
- Приготовьте 60-кратную смесь праймера и зонда для EYFP (1,5 мкМ YFP_fw, 1,5 мкМ YFP_rv и 0,6 мкМ YFP_probe; см. Таблицу материалов). Используйте смесь праймера и зонда GAPDH (см. Таблицу материалов), чтобы определить число копий гена домработницы. Настройте реакцию на льду.
- Приготовьте мастер-смесь для ПЦР (15 мкл, см. Таблицу материалов) и добавьте 60-кратную смесь праймера/зонда (0,5 мкл) для всех образцов и стандартов (чтобы рассчитать количество копий стандартов, воспользуйтесь следующей ссылкой: http://cels.uri.edu/gsc/cndna.html). Настройте реакцию на льду.
- Перенесите 15,5 мкл мастер-смеси в 96-луночную пластину и добавьте 14,5 мкл образца (75 нг общей концентрации ДНК) или стандарта в соответствующую лунку. Запечатайте 96-луночную пластину фольгой, вихревом и ненадолго открутите.
- Перенесите 10 мкл каждого образца в 384-луночную пластину в двух экземплярах. Запечатайте пластину фольгой и отжимайте при 800 x g в течение 5 минут при 4 °C.
- Инкубируют реакционную смесь в термоциклере, используя начальную температуру 50 °C в течение 2 мин, после чего следует начальная стадия активации 10 мин при 95 °C. Выполните 40 циклов денатурации при 95 °C в течение 15 с и отжига/удлинения при 60 °C в течение 1 мин24.
- Чтобы получить количество диплоидных геномов (dg), используйте число копий GAPDH и разделите на два. Затем возьмите значение числа копий EYFP и разделите на количество dg, в результате чего получится векторные геномы на диплоидный геном (vg/dg). Используйте это значение для создания файла Normalization_Organ.txt для биоинформатического анализа.
- Выполните анализ данных секвенирования NGS, как Weinmann et al.24, используя пользовательский код в Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Рабочий процесс включает в себя обнаружение последовательностей штрих-кодов с учетом фланкирующих последовательностей, их длины и местоположения (Script#1_BarcodeDetection.py), а также анализ обогащения и распределения штрих-кода по набору тканей (Script#2_BarcodeAnalysis.py).
- Обнаружение штрих-кодов и их отнесение к вариантам AAV. Поместите данные виртуализации в виде архивных файлов fastq в один каталог (например, «Data_to_analyze»). Файл данных виртуализации для входной библиотеки включен в этот каталог и используется только для вычисления пропорций капсида во входной библиотеке.
- Перед выполнением сценария создайте два текстовых файла с разделителями-табуляциями: файл вариантов капсида (см. пример файла «Варианты.txt») с последовательностями штрих-кодов, назначенными именам вариантов капсида AAV, и файл загрязнения (см. «Загрязнения.txt») с последовательностями штрих-кодов, которые происходят от возможного загрязнения (другие штрих-коды, доступные в лаборатории, способствуют загрязнению).
- Наконец, отредактируйте конфигурационный файл "Barcode_Script.conf", включив в него следующую информацию: путь к папке с данными секвенирования (например, "Data_to_analyze"), последовательность фланкирующих областей штрих-кодов, их положение и размер окна для обнаружения штрих-кода (аналогично 1.9.8.5, см. рисунок 8).
- Используйте следующую команду для вызова обнаружения штрих-кода с предоставленными путями к Script#1_BarcodeDetection.py и файлам конфигурации:
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
ПРИМЕЧАНИЕ: Результатом выполнения Script#1_BarcodeDetection.py являются текстовые файлы с количеством прочтений для каждого варианта капсида, а также общим количеством прочтений, восстановленных из необработанных данных. - Оцените распределение капсидов AAV со штрих-кодом между тканями или органами, выполнив Script#2_BarcodeAnalysis.py вместе со следующими текстовыми файлами:
- В файле «Zuordnung.txt» присвойте имя каждому текстовому файлу, полученному в результате обнаружения штрих-кода, имени ткани/органа: имена текстовых файлов в первом столбце и соответствующие названия тканей/органов в назначении с разделителями-табуляциями.
ПРИМЕЧАНИЕ: Для примера проверьте папку «Пример» (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Следует отметить, что название ткани/органа может включать символы, определяющие измерение кДНК или гДНК и число биологической репликации (M1, M2 и т. Д.). - Создайте текстовый файл "organs.txt" со списком названий органов-мишеней ON- и OFF, которые соответствуют именам, приведенным в файле задания "Zuordnung.txt" (см. папку "Example": https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- Создавайте текстовые файлы с разделителями-табуляциями «Normalization_Organ.txt» и «Normalization_Variant.txt» с нормализованными значениями для всех вариантов капсида и всех органов/тканей. В первом столбце файла «Normalization_Organ.txt» напишите имена, данные для каждого органа (как в файле присвоения «Zuordnung.txt»), а во втором столбце значения нормализации для соответствующих тканей, сгенерированные в разделе 3.6.11.
- Заполните первый столбец файла «Normalization_Variant.txt» списком имен капсидов, а второй столбец — нормализованными значениями количества прочитанных операций для каждого капсида в библиотеке в пуле (нормализация может быть рассчитана на основе выходного файла txt для входной библиотеки, полученного в результате первого скрипта).
- Отредактируйте конфигурационный файл, указав полные пути ко всем дополнительным файлам, упомянутым выше. Выполните Script#2_BarcodeAnalysis.py как:
>python3 /script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
ПРИМЕЧАНИЕ: Сценарий анализа штрих-кода выводит несколько файлов: текстовые файлы со значениями относительной концентрации (RC) распределения капсида в различных тканях на основе нескольких этапов нормализации, описанных ранее, и файл электронной таблицы, который объединяет данные текстового файла в объединенные матричные данные. Последние могут быть использованы для кластерного анализа и визуализации. - Визуализируйте данные и выполняйте кластерный анализ матричных данных, чтобы различать свойства капсида и оценивать их сходство на основе RC-профилей в тканях. Используйте дополнительный скрипт PCA_heatmap_plot. R размещен в репозитории:
>Rscript --vanilla ~/PCA. R ~/относительная концентрация.xls
ПРИМЕЧАНИЕ: Сценарий принимает файлы относительной концентрации .xls в качестве входных данных и генерирует два графика иерархической тепловой карты кластера и анализа главных компонент (PCA). - Чтобы изменить графики (оси тепловой карты, основные компоненты PCA) или параметры png (цвет, размер, маркировка), откройте скрипт R и следуйте инструкциям, приведенным в закомментированных разделах.
- В файле «Zuordnung.txt» присвойте имя каждому текстовому файлу, полученному в результате обнаружения штрих-кода, имени ткани/органа: имена текстовых файлов в первом столбце и соответствующие названия тканей/органов в назначении с разделителями-табуляциями.
Access restricted. Please log in or start a trial to view this content.
Результаты
Генерация библиотеки пептидных дисплеев AAV2. В качестве первого шага к выбору инженерных AAV описана генерация плазмидной библиотеки. Пептидная вставка производится с использованием вырожденных праймеров. Сокращение комбинации кодонов у лиц от 64 до 20 имеет преимущества в устр...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
В этом протоколе изложены этапы, необходимые для разработки капсида AAV на пептидных дисплеях и для скрининга библиотек AAV со штрих-кодом, а также для биоинформатического анализа состава библиотеки и характеристик капсида. Этот протокол фокусируется на шагах, которые облегчают биоинфор...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Д.Г. является соучредителем AaviGen GmbH. D.G. и K.R. являются изобретателями по находящейся на рассмотрении патентной заявке, связанной с генерацией вариантов капсида AAV, уклоняющихся от иммунитета. Остальным авторам раскрывать нечего.
Благодарности
D.G. высоко ценит поддержку со стороны Немецкого научно-исследовательского общества (DFG) через Центры совместных исследований DFG SFB1129 (Projektnummer 240245660) и TRR179 (Projektnummer 272983813), а также Немецкого центра инфекционных исследований (DZIF, BMBF; ТТУ-ВИЧ 04.819).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
Ссылки
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621(2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756(2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075(2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041(2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432(2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819(2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899(2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272(2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200(2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825(2021).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены