JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
아데노 관련 바이러스(AAV) 캡시드 변이체의 엔지니어링, 바코드 및 스크리닝을 통한 차세대 유전자 치료 벡터의 분리
요약
AAV 펩타이드 디스플레이 라이브러리 생성 및 차세대 AAV 생성을 위한 새로운 특성을 가진 후보 물질의 바코딩을 통한 후속 검증.
초록
아데노 관련 바이러스(AAV)에서 파생된 유전자 전달 벡터는 임상 데이터를 장려하고 여러 AAV 유전자 요법을 승인함으로써 입증된 유전 질환 치료를 위한 가장 유망한 도구 중 하나입니다. AAV 벡터의 성공에 대한 두 가지 주요 이유는 (i) 뚜렷한 특성을 가진 다양한 자연 발생 바이러스 혈청형의 사전 분리와 (ii) 분자 공학 및 높은 처리량의 용도 변경을 위한 강력한 기술의 후속 확립입니다. 이러한 기술의 잠재력을 더욱 강화하는 것은 최근 DNA 및 RNA 수준에서 선택된 AAV 캡시드를 바코딩하기 위한 전략으로 구현되어 단일 동물의 모든 주요 기관 및 세포 유형에서 포괄적이고 평행한 생체 내 계층화를 허용합니다. 여기에서는 사용 가능한 캡시드 엔지니어링 기술의 다양한 무기고를 나타내기 위해 AAV 펩타이드 디스플레이를 사용하여 이러한 보완 방법을 포함하는 기본 파이프라인을 제시합니다. 따라서, 우리는 먼저 원하는 특성을 가진 후보의 생체 내 선택을 위한 AAV 펩타이드 디스플레이 라이브러리의 생성을 위한 중추적인 단계를 설명한 다음, 2차 생체 내 스크리닝을 위해 가장 흥미로운 캡시드 변이체를 바코드화하는 방법에 대한 시연을 설명합니다. 다음으로, NGS 데이터 분석 중 가장 중요한 단계에 대한 개요로 마무리하기 전에 바코드 증폭 및 어댑터 결찰을 포함한 차세대 염기서열 분석(NGS)을 위한 라이브러리 생성 방법론을 예시합니다. 여기에 보고된 프로토콜은 다재다능하고 적응력이 뛰어나기 때문에 연구자들은 이를 쉽게 활용하여 선호하는 질병 모델 및 유전자 치료 응용 분야에서 최적의 AAV 캡시드 변이체를 풍부하게 할 수 있습니다.
서문
유전자 전달 요법은 질병을 예방, 치료, 치료 또는 개선하기 위해 세포 유전 물질을 복구, 교체 또는 변경하기 위해 세포에 유전 물질을 도입하는 것입니다. 생체 내 및 생체 외 모두에서 유전자 전달은 비바이러스 및 바이러스의 서로 다른 전달 시스템에 의존합니다. 바이러스는 표적 세포를 효율적으로 형질도입하기 위해 자연적으로 진화했으며 전달 벡터로 사용될 수 있습니다. 유전자 치료에 사용되는 다양한 유형의 바이러스 벡터 중에서, 아데노 관련 바이러스는 병원성, 안전성, 낮은 면역원성, 그리고 가장 중요하게는 장기간의 비통합 발현을 유지하는 능력의 부족으로 인해 점점 더 많이 사용되고 있다 1,2,3. AAV 유전자 치료는 지난 10년 동안 상당한 성과를 거두었습니다. 유럽 의약품 청 (European Medicines Agency)과 미국 식품의 약국 (Food and Drug Administration)은 3,4. 혈우병, 근육, 심장, 신경계 질환과 같은 다양한 질병을 치료하기 위한 여러 임상 시험도 진행 중이다3. 유전자 치료 분야는 수십 년에 걸친 발전에도 불구하고 최근 몇 년 동안 일련의 차질을 빚어왔다.4 가장 중요한 것은 임상 시험5에서 용량 제한 독성으로 인해 사망이 보류되었는데, 이는 특히 근육과 같이 거대하거나 뇌와 같이 접근하기 어려운 조직에서 발생했다6.
현재 임상 시험에 사용되고 있는 AAV 벡터는 몇 가지 예외를 제외하고 천연 혈청형에 속한다1. AAV 엔지니어링은 우수한 장기 또는 세포 특이성과 효율성을 가진 벡터를 개발할 수 있는 기회를 제공합니다. 지난 20년 동안 펩타이드 디스플레이, 루프 스왑, 캡시드 DNA 셔플링, 오류가 발생하기 쉬운 PCR 및 표적 설계와 같은 여러 접근 방식이 성공적으로 적용되어 다양한 특성을 가진 개별 AAV 변이체 또는 라이브러리를 생성했습니다7. 그런 다음 이들은 다른 곳에서 검토된 바와 같이 원하는 특성을 가진 변이체를 선택하기 위해 여러 차례의 지시된 진화를 거칩니다 1,3. 모든 캡시드 진화 전략 중에서 펩타이드 디스플레이 AAV 라이브러리는 몇 가지 고유한 특성으로 인해 가장 널리 사용되었습니다: 상대적으로 생성하기 쉽고 높은 다양성과 높은 처리량 시퀀싱을 달성할 수 있어 진화를 추적할 수 있습니다.
최초의 성공적인 펩타이드 삽입 AAV 라이브러리는 거의 20년 전에 설명되었습니다. 첫 번째 중 하나에서, Perabo et al.8 은 무작위로 생성된 올리고뉴클레오티드 풀이 캡시드로부터 돌출된 3중 축에서 VP1 캡시드 단백질의 아미노산 587에 해당하는 위치의 플라스미드에 삽입된 변형된 AAV2 캡시드의 라이브러리를 구성했습니다. 아데노바이러스 동시 감염을 사용하여 AAV 라이브러리는 여러 차례의 선택을 통해 진화되었으며, 최종 재표적 변이체는 부모 AAV2에 불응성인 세포주를 형질도입할 수 있는 것으로 나타났다8. 그 후 얼마 지나지 않아 Müller et al.9 는 라이브러리 제작을 위한 2단계 시스템을 도입하여 프로토콜을 크게 개선했습니다. 처음에는 플라스미드 라이브러리가 아데노바이러스 헬퍼 플라스미드와 함께 키메라 캡시드를 포함하는 AAV 라이브러리를 생성하는 데 사용됩니다. 이 AAV 셔틀 라이브러리는 세포당 하나의 바이러스 게놈을 도입하는 것을 목표로 낮은 감염 다중도(MOI)에서 세포를 감염시키는 데 사용됩니다. 아데노 바이러스와의 동시 감염은 일치하는 게놈과 캡시드9를 가진 AAV의 생산을 보장합니다. 약 10 년 후, Dalkara10 은 생체 내 진화를 사용하여 7m8 변종을 만들었습니다. 이 변이체는 10개의 아미노산 삽입(LALGETTRPA)을 가지며, 그 중 3개는 링커로 작용하고, 유리체강내 주사 후 외부 망막을 효율적으로 표적으로 한다10. 이 조작된 캡시드는 지금까지 클리닉에 도착한 몇 안 되는 조작된 캡시드 중 하나이기 때문에 탁월한 성공 사례입니다11.
이 분야는 차세대 염기서열 분석(NGS) 기술의 도입으로 두 번째 성장을 경험했습니다. 2014년 Adachi et al.12와 2015년 Marsic et al.13의 두 간행물은 바코드화된 AAV 캡시드 라이브러리의 분포를 높은 정확도로 추적하는 NGS의 힘을 보여주었습니다. 몇 년 후, 바코드 영역의 NGS는 캡시드 진화를 따르기 위해 펩티드 삽입 영역에 적응되었습니다. Körbelin et al.14는 폐 표적 AAV2 기반 캡시드를 식별하기 위해 NGS 유도 스크리닝을 수행했습니다. NGS 분석은 선택 라운드 간의 농축 점수, 조직 특이성을 결정하기 위한 일반 특이도 점수, 마지막으로 결합 점수14의 세 가지 평가 점수를 계산하는 데 도움이 되었습니다. Gradinaru 실험실15는 같은 해에 Cre-recombination-based AAV 표적 진화 (CREATE) 시스템을 발표하여 세포 유형별 선택을 용이하게했습니다. 이 시스템에서 캡시드 라이브러리는 polyA 신호가 두 개의 loxP 사이트 옆에 있기 때문에 Cre-반전 가능한 스위치를 전달합니다. 그런 다음 AAV 라이브러리를 Cre 마우스에 주입하여 polyA 신호가 Cre+ 세포에서만 반전되어 캡시드 유전자 내의 정방향 프라이머와 역방향 PCR 프라이머의 결합을 위한 주형을 제공합니다. 이 매우 특이적인 PCR 구조를 통해 AAV-PHP를 식별할 수 있었습니다. 혈액뇌장벽을 통과할 수 있는 B 변이체15. 이 시스템은 M-CREATE(Multiplexed-CREATE)로 더욱 발전했으며, 여기서 NGS와 합성 라이브러리 생성은 파이프라인(16)에 통합되었다.
맥과이어 연구실(Maguire lab)17의 개선된 RNA 기반 버전인 iTransduce는 세포를 기능적으로 형질도입하고 게놈을 발현하는 캡시드의 DNA 수준에서 선택할 수 있습니다. 펩티드 디스플레이 라이브러리의 바이러스 게놈은 유비쿼터스 프로모터의 제어 하에 Cre 유전자 및 p41 프로모터의 제어 하에 캡시드 유전자를 포함한다. 라이브러리는 tdTomato의 상류에 loxP-STOP-loxP 카세트를 갖는 마우스에 주입됩니다. 바이러스 게놈을 발현하는 AAV 변이체로 형질도입된 세포는 Cre가 tdTomato를 발현하고, 세포 마커와 결합하여 분류 및 선택될 수 있다17. 유사하게, Nonnenmacher et al.18 및 Tabebordbar et al.19는 캡시드 유전자 라이브러리를 조직 특이적 프로모터의 제어 하에 두었습니다. 상이한 동물 모델에 주사한 후, 바이러스 RNA를 사용하여 캡시드 변이체를 분리하였다.
또 다른 방법은 바코드를 사용하여 캡시드 라이브러리에 태그를 지정하는 것입니다. Björklund lab20 은 바코드 펩타이드 삽입 캡시드 라이브러리에 이 접근 방식을 사용하고 바코드 합리적 AAV 벡터 진화(BRAVE)를 개발했습니다. 한 플라스미드에서 Rep2Cap 카세트는 ITR(Inverted Terminal Repeats) 측면에 노란색 형광 단백질(YFP) 발현, 바코드 태그가 부착된 이식유전자 옆에 클로닝됩니다. 캡 끝과 바코드 시작 부분 사이의 loxP 부위를 사용하여 시험관 내 Cre 재조합은 NGS에 충분히 작은 단편을 생성함으로써 펩타이드 삽입과 고유한 바코드(룩업 테이블, LUT)의 결합을 허용합니다. AAV 생산은 플라스미드 라이브러리를 사용하여 수행되고 mRNA에서 발현된 바코드는 생체 내 적용 후 NGS20으로 다시 스크리닝됩니다. 캡시드 라이브러리가 전체 캡시드 유전자의 변이체(즉, 섞인 라이브러리)로 구성된 경우 긴 읽기 시퀀싱을 사용해야 합니다. 여러 그룹이 바코드를 사용하여 이러한 다양한 라이브러리에 태그를 지정했으며, 이를 통해 더 높은 판독 깊이의 NGS를 사용할 수 있습니다. Kay lab21 은 캡 polyA 신호의 다운스트림에 바코드가 있는 매우 다양한 캡 시드 셔플 라이브러리에 태그를 지정했습니다. 첫 번째 단계에서, 바코드화된 플라스미드 라이브러리가 생성되었고, 섞인 캡시드 유전자 라이브러리가 그 안에 클로닝되었다. 그런 다음 MiSeq(짧은 읽기, 더 높은 읽기 깊이) 및 PacBio(긴 읽기, 더 낮은 읽기 깊이) NGS와 Sanger 시퀀싱의 조합을 사용하여 LUT21을 생성했습니다. 2019년에 Church Lab22 의 Ogden과 동료들은 모든 위치에서 단일 지점 돌연변이, 삽입 및 결실이 있는 라이브러리를 사용하여 여러 기능에 대한 AAV2 캡시드 적합성을 설명했으며, 이는 궁극적으로 기계 유도 설계를 가능하게 했습니다. 라이브러리의 생성을 위해 캡시드 유전자의 더 작은 단편을 합성하고, 바코드로 태그를 지정하고, 차세대 시퀀싱한 다음, 전체 캡시드 유전자에 클로닝했습니다. NGS 데이터는 LUT를 생성하는 데 사용되었습니다. 그런 다음 라이브러리는 바코드와 짧은 판독 시퀀싱만을 사용하여 스크리닝되었으며, 이는 차례로 더 높은 판독 깊이(22)를 허용합니다.
바코드 라이브러리는 캡시드 라이브러리를 여러 차례 선택하거나 캡시드 진화 연구와 무관하게 알려진 자연적 및 조작된 변이체 풀을 스크리닝하는 데 주로 사용되었습니다. 이러한 라이브러리의 장점은 여러 캡시드를 스크리닝하는 동시에 동물 수를 줄이고 동물 간의 변동을 최소화할 수 있다는 것입니다. 이 기술을 AAV 분야에 도입한 첫 번째 연구는 거의 10년 전에 발표되었습니다. Nakai lab12 는 한 쌍의 12-뉴클레오티드 바코드를 사용하여 AAV9의 VP1에서 아미노산 356 내지 736을 포함하는 191개의 이중 알라닌 돌연변이체에 태그를 붙였습니다. NGS를 사용하여, 라이브러리는 갈락토오스 결합 및 기타 특성에 대해 생체 내에서 스크리닝되었다12. Marsic과 동료들은 1 년 후 이중 막대 분석을 사용하여 AAV 변이체의 생체 분포를 설명했다13. 인간이 아닌 영장류에 대한 보다 최근의 연구에서는 서로 다른 전달 경로를 사용하여 29개의 캡시드의 중추신경계 내 생체 분포를 비교했습니다23. 우리 연구실은 최근 천연 및 엔지니어링 AAV를 포함하는 183 가지 변형의 바코드 AAV 라이브러리 스크린을 발표했습니다. DNA 및 RNA 수준에 대한 이러한 스크리닝은 마우스에서 고도의 근친화성 AAV 변이체(24)를 식별하게 했으며, 마우스 뇌(25)에서 높은 세포 유형 특이성을 나타내는 다른 변이체를 식별했습니다.
여기에서는 이 작업에 사용된 방법론을 설명하고 AAV 펩타이드 디스플레이 라이브러리의 스크리닝을 포함하도록 확장합니다. 이것은 AAV2 펩타이드 디스플레이 라이브러리의 생성, 정량화를 위한 디지털 액적 PCR(dd-PCR) 방법, 그리고 마지막으로 부분적으로 Weinmann과 동료24의 작업을 기반으로 AAV 변이체를 분석하기 위한 NGS 파이프라인으로 구성됩니다. 마지막으로, 동일한 간행물에 사용된 바코드 AAV 라이브러리 및 NGS 파이프라인의 생성에 대한 설명이 제공됩니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. AAV2 무작위 7-mer 펩티드 디스플레이 라이브러리 준비
참고: AAV2 무작위 펩타이드 디스플레이 라이브러리의 준비를 위해 퇴행성 올리고뉴클레오티드를 단일 가닥 DNA로 합성하고, 이중 가닥 DNA로 변환하고, 소화하고, 수용체 플라스미드로 결찰하고, 전기포증합니다.
- 퇴행성 올리고뉴클레오타이드의 설계
- 퇴행성 올리고뉴클레오티드를 주문하고 코돈 편향을 피하십시오. 올리고뉴클레오티드 5' CAGTCGGCCAG AG W GGC(X01)7 GCCCAGGCGGCTGACGAG 3'에서, X01은 20개의 코돈에 해당하며, 각각은 20개의 아미노산 중 하나를 암호화한다. W는 A 또는 T일 수 있으며, 아미노산 아르기닌(R) 또는 세린(S)을 암호화하는 코돈 AGA 또는 AGT를 생성합니다.
- 증폭 프라이머 5' CTCGTCAGCCGCCTGG 3'을 주문합니다(자세한 내용은 그림 1 참조). 단백질 삽입물 R/S G X7을(를) 만들어냄. 이론적 다양성은 다음과 같이 계산됩니다 : 1 x 2 x 207 = 2.56 x 109 개의 고유 한 변형.
참고: 이러한 다양성은 변환 효율성에 의해 제한될 수 있다는 점에 유의해야 합니다.
- 두 번째 가닥 합성
- 두 올리고뉴클레오티드(퇴행성 올리고뉴클레오티드 및 증폭 프라이머)를 TE 완충액으로 100 μM 최종 농도로 재현탁합니다.
- PCR 반응의 경우 각 프라이머 1μL, 완충액 10μL, DMSO 1.5μL, dNTP(10mM) 0.5μL, Hi-fidelity Hot Start Polymerase II 0.5μL 및 뉴클레아제가 없는 물 35.5μL로 50μL 반응을 설정합니다.
- 반응물을 열순환기로 옮기고, 98°C에서 10초 동안 예비인큐베이션 단계를 실행한 다음, 98°C에서 10초, 59°C에서 30초, 72°C에서 10초의 3회 사이클을 실행한 다음, 72°C에서 5분 및 최종 냉각 단계를 실행한다.
- 뉴클레오티드 제거 키트를 사용하여 반응을 정제하고 뉴클레아제가 없는 물 100μL에서 용리합니다.
- Bioanalyzer에서 분석하여 두 번째 가닥 합성의 효율성을 확인합니다( 그림 2 참조). 제조업체의 지침에 따라 DNA 1000 Reagents 키트에서 미세유체 칩에 반응 1μL를 로딩하여 이중 가닥 삽입물의 크기와 순도를 분석합니다. 이 키트는 25-1,000bps의 이중 가닥 DNA 단편의 크기와 농도를 측정하도록 최적화되어 있습니다.
- 두 올리고뉴클레오티드(퇴행성 올리고뉴클레오티드 및 증폭 프라이머)를 TE 완충액으로 100 μM 최종 농도로 재현탁합니다.
- 삽입물 및 플라스미드 벡터의 분해
- 정제된 삽입물 85 μL를 10 μL의 10x 완충액과 5 μL의 BglI 효소와 함께 최종 100 μL 반응 부피로 분해합니다(자세한 내용은 그림 1 참조). 37°C에서 하룻밤 동안 배양합니다. 뉴클레오티드 제거 키트를 사용하여 정제하고, 뉴클레아제가 없는 물 50μL에서 용리하고, 분광광도계에서 "Oligo DNA" 유형을 사용하여 정량합니다.
- 10μg의 복제 적격 AAV 플라스미드(pRep2Cap2_PIS)26 (ITR 측면 바이러스 게놈)을 최종 200μL 반응 부피에서 20μL의 10x 완충액과 10μL의 SfiI 효소로 분해합니다(자세한 내용은 그림 1 참조). 50°C에서 하룻밤 동안 배양합니다. 겔 추출 키트를 사용하여 벡터를 1% 아가로스 겔 상에서 정제한 후, DNA 정제 키트를 사용하여 추가 정제 단계를 수행하였다. 분광 광도계에서 농도를 정량화합니다.
- 벡터에 삽입물 결찰
- 20μL 결찰 반응에서 2μL의 완충액과 2μL의 리가아제와 함께 45ng의 삽입물과 함께 955ng의 플라스미드 벡터를 Ligate합니다. 16°C에서 하룻밤 동안 인큐베이션한 다음, 70°C에서 10분 동안 열-불활성화시켰다.
- 변환, 복잡성 계산 및 플라스미드 라이브러리 준비
- 제조업체의 지침에 따라 DNA 정제 키트로 반응을 정제합니다. 뉴클레아제가 없는 물의 시작 부피의 약 80%에서 반응을 용리하고 후속 변형을 위해 얼음에 보관합니다.
- 전기 적격 셀 변환: 전기 적격 셀 바이알 하나를 얼음 위에서 10분 동안 해동합니다. 그런 다음 정제된 결찰 반응 1-2μL를 전기 적격 세포 30μL(바이알 1개)에 넣고 부드럽게 두드려 혼합합니다. 다음으로, 기포를 도입하지 않고 세포/DNA 혼합물을 미리 냉각된 1mm 간격의 전기천공 큐벳에 조심스럽게 피펫팅합니다.
- 1800 V, 600 Ω 및 10 μF 설정을 사용하여 전기 천공합니다. 전기천공 펄스의 10초 이내에 970μL의 예열된 회수 배지(전기 적격 셀과 함께 제공됨)를 큐벳에 추가하고 피펫팅으로 혼합합니다. 마지막으로 세포를 마이크로 원심분리 튜브로 옮기고 37°C에서 250rpm으로 1시간 동안 배양합니다. 원하는 다양성을 얻으려면 10-100 개의 반응을 수행하고 배양 후 모든 반응을 하나의 튜브에 모으십시오.
- PBS에서 10배, 100배 또는 1,000배의 풀링된 형질전환 10μL를 희석하고 적절한 항생제(암피실린 75mg/mL)가 들어 있는 영양 한천 플레이트에 100μL를 뿌려 다양성을 계산합니다. 한천 플레이트를 37°C에서 밤새 배양한 다음 한천 플레이트에서 콜로니를 계수합니다.
- 이론적 다양성을 다음과 같이 계산하십시오.
이론적 최대 다양성 = 10 x 희석 계수 x 콜로니 수 x 전기천공 반응 수.
참고: 라이브러리 품질을 확인하려면 Sanger 시퀀싱으로 최소 20개의 콜로니를 시퀀싱합니다. 대부분의 클론은 삽입물을 포함해야 하며 모두 고유해야 합니다. - 적절한 항생제가 들어 있는 LB 배지 400-1,000mL에 나머지 풀링된 형질전환을 접종하고 37°C, 180rpm에서 밤새 배양합니다.
- 플라스미드 라이브러리의 제조
- 하룻밤 배양에서 글리세롤 스톡(뉴클레아제가 없는 물에 동일한 부피의 박테리아 배양과 50% 글리세롤 용액을 혼합하고 -80°C에서 동결)을 준비하고 플라스미드 맥시 키트를 사용하여 플라스미드 라이브러리를 정제합니다.
- AAV 바이러스 라이브러리 제작
- 앞서 설명한 바와 같이 바이러스 라이브러리를 준비한다27. 플라스미드 라이브러리(pRep2Cap2_PI, 펩티드 삽입물)를 아데노헬퍼 플라스미드와 함께 폴리에틸렌이민(PEI)과 같은 형질주입 시약을 사용하여 HEK293T 세포에 형질주입한다.
- 3 일 후에 세포를 수집하고 3주기의 동결-해동을 실시합니다. 염화세슘 구배 초원심분리를 사용하여 바이러스 용해물을 정제한 다음 PBS로 완충액을 교환하고 마지막으로 바이러스 입자를 농축합니다.
- dd-PCR을 이용한 AAV 벡터 적정
- 198 μL의 뉴클레아제가 없는 물에 2 μL의 AAV 벡터 스톡을 연속적으로 희석하여 1:106 최종 희석을 산출합니다. 매번 200 μL 피펫을 사용하여 철저히 혼합합니다. 하나의 NTC(템플릿 없는 컨트롤)를 부정 컨트롤로 추가합니다.
참고: 추가로 더 낮거나 더 높은 희석액을 분석할 수 있습니다(1:105-1:107). - 20x 프라이머-프로브 혼합물을 준비합니다. 100μM 프라이머(정방향 및 역방향, Rep2 및 ITR) 각각 3.6μL, 100μM dd-PCR 프로브(Rep2 및 ITR) 각각 1μL 및 뉴클레아제가 없는 물 3.6μL를 1.5mL 원심분리기 튜브에 추가합니다.
참고: AAV 라이브러리는 FAM 표지 프로브로 검출된 이식유전자 표적 프라이머 프로브 세트(Rep2)와 HEX 표지 프로브로 검출된 ITR 표적 프라이머 프로브 세트를 사용하여 측정됩니다. - 5.5μL의 시료, 1.1μL의 20x 프라이머-프로브 혼합물, 11μL의 프로브용 dd-PCR 슈퍼믹스(dUTP 없음) 및 4.4μL의 뉴클레아제가 없는 물을 추가하여 22μL PCR 반응을 준비합니다. 이것은 프라이머와 프로브에 대해 각각 900 nM 및 250 nM의 농도를 산출합니다.
- 액적 발생기를 사용하여 액적을 생성하고, 반응을 96웰 플레이트로 옮기고, 플레이트를 열순환기에 넣고, 94°C에서 10분 동안 변성 단계를 실행한 다음, 94°C에서 30초, 58°C에서 1분 동안 40사이클을 실행합니다. 다음으로, 중합효소를 98°C에서 10분 동안 열불활성화시키고 최종 냉각 단계를 추가한다. 액적 판독기에서 반응을 읽고 분석28로 진행합니다.
- 분석 소프트웨어를 사용하여 저장된 dd-PCR 플레이트 파일을 엽니다. 1D 진폭 탭의 임계값 도구(형광 진폭 대 이벤트 번호)를 사용하여 NTC를 가이드로 사용하여 각 채널의 음수 및 양수 드롭렛을 분리하고 데이터를 csv 파일로 내보냅니다.
- 벡터 농도를 계산하려면 먼저 다음 공식을 사용하여 보정 계수 CF를 계산합니다.
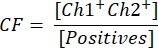
CF는 기능성 벡터 입자의 검출을 보장하기 위해 이식유전자 및 ITR[Ch1+ Ch2+] 모두에 대해 양성인 이식유전자[양성]에 대해 양성인 액적의 비율을 결정합니다. 최종 벡터 농도 c는 이제 다음 방정식을 사용하여 계산할 수 있습니다.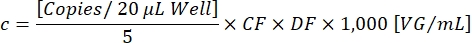
DF는 희석 계수입니다 (1 : 10,5-1 : 10,7 , 이전에 결정됨). 20 μL/웰 반응당 복사는 희석된 샘플의 5 μL에 해당합니다. 인자 1,000은 척도를 VG/mL(바이러스 게놈/mL)로 수정합니다. 예시적인 적정 결과는 표 1 및 도 3에 제시되어 있다.
- 198 μL의 뉴클레아제가 없는 물에 2 μL의 AAV 벡터 스톡을 연속적으로 희석하여 1:106 최종 희석을 산출합니다. 매번 200 μL 피펫을 사용하여 철저히 혼합합니다. 하나의 NTC(템플릿 없는 컨트롤)를 부정 컨트롤로 추가합니다.
- NGS에 의한 AAV 바이러스 라이브러리 분석
- 교정 판독 중합효소 키트(2x, 그림 4 참조)를 사용하여 20 μL PCR 반응을 설정하여 96-뉴클레오티드 펩타이드 삽입 단편을 증폭합니다. 1 x 108 vg , 0.5 μL의 100 μM 프라이머 (NGS_forward 및 NGS_reverse) 및 10 μL의 효소 혼합물을 포함하는 AAV 스톡 1 μL을 반응에 추가합니다. 뉴클레아제가 없는 물로 최종 부피를 20μL로 조정합니다.
- 반응물을 열순환기로 옮기고 98°C에서 3분 동안 변성 단계를 실행한 다음, 98°C에서 10초, 59°C에서 10초, 72°C에서 20초의 30-35 사이클을 실행한 다음, 72°C에서 5분 및 최종 냉각 단계를 실행합니다.
- PCR 정제 키트를 사용하여 샘플을 정제합니다. 분광 광도계에서 농도를 정량화하고 3% 아가로스 겔을 실행하여 순도와 단편 크기를 확인합니다.
- NGS 라이브러리의 제조를 위한 제조사의 지침에 따라 저복잡성 샘플용 라이브러리 시스템 키트를 사용하여 PCR 단편을 처리한다. 30ng의 PCR 단편으로 최종 복구 반응을 수행한 후 어댑터 결찰 및 10주기 동안 PCR 증폭을 수행합니다. 반응의 정제를 위해 PCR 정제 키트를 사용하십시오.
- Bioanalyzer에서 최종 제품을 처리하여 제조업체의 지침에 따라 DNA 시약 키트를 사용하여 크기와 순도를 확인합니다.
- 형광 측정기를 사용하여 앰플리콘을 정량화하고 풀링합니다. 형광측정기에서 최종 풀링된 NGS 라이브러리를 다시 정량화하고(제조업체의 지침에 따라) Bioanalyzer에서 품질을 확인합니다.
- 판독 길이가 84이고 인덱스 1이 8인 75사이클 고출력 키트를 사용하여 단일 종단(SE) 모드에서 NGS 라이브러리를 시퀀싱합니다.
참고: 이 기사의 예제 시퀀싱은 EMBL Heidelberg(http://www.genecore.embl.de/)의 GeneCore 시설에서 수행되었습니다. - Python 3 및 biopython을 사용하여 NGS 염기서열 분석 데이터를 분석합니다. 파일은 https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022(또는 https://doi.org/10.5281/zenodo.7032215)에서 찾을 수 있습니다. NGS 분석은 두 단계로 구성됩니다.
- 첫 번째 단계에서는 시퀀스 파일에서 특정 기준(삽입 부위 옆에 있는 인식 시퀀스의 존재)을 충족하는 시퀀스를 검색합니다( 그림 4, 단계 1.9.8.5 참조). 이 작업은 스크립트(Script#1)와 필요한 정보를 제공하는 구성 파일을 사용하여 수행됩니다. 올바른 시퀀스가 식별되면 프로그램은 시퀀스를 추출하여 시퀀스 파일과 동일한 이름의 txt 파일인 출력 파일에 저장합니다.
- 두 번째 단계는 출력 파일을 분석하는 것입니다. 라이브러리의 서열은 9개의 아미노산 삽입물에 있는 6개의 뉴클레오티드(AGWggc, W =A/T) 중 하나로 시작합니다. 이러한 개시 서열에 기초하여, 펩티드가 번역된다. 이렇게 하면 펩타이드 변이체(PV)가 포함된 출력 파일이 생성됩니다.
- 스크립트와 데이터라는 두 개의 폴더를 준비합니다. Data 폴더에 시퀀싱으로 생성된 gzip 압축 파일을 복사합니다. 스크립트 폴더에 Python 파일인 Script#1_DetectionExtraction_JoVE_Py3.py; 파이썬 파일: Script#2_PV_extraction_and_ranking_Py3.py; 구성 파일: Barcode_Script_JoVE.conf; 및 조회 테이블(LUT) 파일: Zuordnung.txt.
- 스크립트를 실행하기 전에 스크립트 폴더에서 다음 파일을 편집합니다. "Zuordnung.txt" 파일을 열고 탭으로 구분된 두 개의 열, gzip 파일 이름(열 1) 및 원하는 최종 이름(열 2, 탭으로 구분된 값)을 추가합니다.
참고: 샘플 txt 파일은 GitHub 폴더 "PV_analysis_script"에 있습니다. GitHub 폴더에 제공된 파일은 위 라이브러리의 세 가지 샘플 데이터(xaa.txt.gz, xab.txt.gz 및 xac.txt.gz)를 분석할 수 있도록 준비됩니다. 출력 파일도 제공됩니다. - 구성 파일 "Barcode_Script_JoVE.conf"에서 다음 변수를 변경합니다.
my_dir = "~/데이터/"
filename_sample_file = "~/스크립트/Zuordnung.txt"
서열별 변수: BCV_size = 27, BCV왼쪽 = TCCAGGGCCAG, BCV오른쪽 = GCCCAGG, BCVloc = 30, BCVmargin = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC 및 BCVloc_revcomp = 41입니다(자세한 내용은 그림 4 참조). - 다음 명령을 사용하여 변형 시퀀스 감지 및 추출을 호출합니다.
>python3 ~ / 스크립트 # 1_DetectionExtraction_JoVE_Py3.py ~ / Barcode_Script_JoVE.conf
참고: 출력은 추출된 DNA 서열과 판독 횟수가 포함된 txt 파일입니다. 이 파일의 헤더에는 통계 데이터(즉, 총 읽기 수 및 추출된 읽기)가 포함되어 있습니다. 이러한 데이터는 다음 파일로 전송됩니다. 이러한 txt 데이터는 DNA 서열이 번역, 순위 지정 및 분석되는 Script#2의 입력 파일입니다. - 다음 명령을 사용하여 PV 추출 및 분석을 수행합니다.
>python3 ~/스크립트#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - Script#2의 텍스트 출력 파일을 분석합니다. Script#2의 출력 파일은 분석 유형에 따라 확장자가 있는 "Zuordnung.txt"에서 LUT의 두 번째 열을 사용하여 이름이 지정됩니다.
참고: 세 개의 출력 파일에 첫 번째 행("# of Valid PV reads", "# of Invalid PV reads" 및 "# of unique PV reads"), 입력 txt 파일의 각 DNA 서열 인덱스가 있는 첫 번째 열(Script#1의 출력) 및 다음 열에 통계 데이터가 포함되어 있는지 확인합니다. analyzed_all.csv": "샘플:"(DNA 서열), "#"(판독 횟수), "Frw 또는 Rev"(순방향 또는 역방향 판독) 및 "PV"(번역된 펩타이드 서열). 유효하지 않은 시퀀스의 마지막 두 열에는 "NA" 및 "not valid"가 있습니다. (2) "... analyzed_validSeq.csv": 이전 파일과 동일하며 유효한 시퀀스에 대해 필터링됩니다. (3) "... analyzed_PV.csv": "PVs"(번역된 펩타이드 서열), "#"(판독 횟수) 및 "count"(이전 파일의 frw 및 rev 카운트가 병합되고 카운트가 1 또는 2로 지정됨). - 사용자의 필요에 따라 사용 가능한 소프트웨어를 사용하여 출력 파일을 시각화합니다.
2. AAV2 무작위 7-mer 펩타이드 디스플레이 라이브러리 선택
- 선택한 모델에서 방향성 진화를 위해 정량화 및 품질 관리(섹션 1) 후 AAV 라이브러리를 사용하여 원하는 특성을 가진 후보를 반복적으로 선택합니다(그림 5 참조)16,18,21.
참고: 이러한 후보는 아래 섹션 3에 설명된 대로 바코드 라이브러리를 생성하는 데 사용됩니다.
3. 바코드 AAV 캡시드 라이브러리 준비 및 분석
참고: 펩타이드 디스플레이 화면에서 잠재적으로 특이적이고 효율적인 AAV 캡시드 세트를 식별한 후 식별된 펩타이드 서열의 기능을 확인하고 일반적으로 사용되거나 잘 설명된 참조 AAV 캡시드 변이체 세트와 비교합니다. 이를 위해 캡시드 시퀀스는 ITR 없이 Rep/Cap 도우미 구문에 삽입됩니다.
- 바코드 AAV 라이브러리 제작
- 앞서 기술한 바와 같이, 3-플라스미드 시스템을 사용하여 각각의 캡시드 변이체에 대해 재조합 AAV 생산을 수행한다24.
참고: 서로 다른 캡시드 변이체를 구별하기 위해 ITR 측면 리포터 이식유전자 플라스미드에는 15개 뉴클레오티드 길이의 고유한 바코드가 있습니다. 바코드는 강화된 황색 형광 단백질(EYFP)과 polyA 신호 사이의 3' UTR(비번역 영역)에 위치한다( 도 6A 참조). EYFP 발현은 충분한 수준의 RNA 전사체를 제공하는 강력한 유비쿼터스 거대세포바이러스(CMV) 프로모터에 의해 구동됩니다. - 3개 미만의 뉴클레오티드, <65%29의 GC 함량, 4개 이상의 뉴클레오티드24의 해밍 거리로 15개 뉴클레오티드 길이의 바코드를 설계한다.
- 고유한 바코드를 운반하는 이식유전자 플라스미드와 함께 각 캡시드를 개별적으로 생산합니다. 이러한 방식으로 각 캡시드 변형에는 특정 추적을 가능하게 하는 고유한 바코드가 태그됩니다( 그림 6B 참조).
- 앞서 기술한 바와 같이, 3-플라스미드 시스템을 사용하여 각각의 캡시드 변이체에 대해 재조합 AAV 생산을 수행한다24.
- dd-PCR을 이용한 AAV 벡터 적정
- Rep2 프라이머 쌍을 YFP 프라이머 쌍으로 교체하여 섹션 1.8에서 이전에 설명한 대로 AAV 적정을 수행합니다.
- 개별 AAV 프로덕션을 정량화하고 각 프로덕션의 동일한 양을 모아 최종 바코드 라이브러리를 생성합니다.
- 최종 라이브러리를 다시 정량화하여 최종 농도와 품질을 확인합니다( 그림 7 참조).
- 바코드 AAV 라이브러리 in vivo 응용 프로그램
- 바코드화된 AAV 라이브러리를 선택한 모델 시스템에 체계적으로 적용합니다(예: 마우스에서 체계적으로24).
- 실험을 기반으로 온-/오프-타겟 조직(즉, 간, 폐, 심장, 횡격막, 평활근, 십이지장, 췌장, 결장, 이두박근, 난소, 위, 내이, 신장, 복부 대동맥, 흉부 대동맥, 뇌, 갈색 및 백색 지방, 비장) 또는 세포 유형을 수집합니다. -80°C에서 동결하고, DNA/RNA를 추출하고, 다음 섹션에 설명된 대로 NGS 정량 분석을 적용합니다.
- DNA/RNA 추출
- DNA/RNA Mini Kit를 사용하여 관심 조직에서 DNA와 RNA를 추출합니다.
- 관심있는 조직의 작은 조각 (1mm3, 약 5 mg)을 2 mL 반응 튜브에 넣는다.
- β-메르캅토에탄올(1%) 및 5mm 강철 비드와 혼합된 용해 완충액 350μL를 조직에 추가합니다(흄 후드 아래에서 β-메르캅토에탄올로 샘플 처리).
- 40Hz에서 45초 동안 tissueLyser에서 조직을 균질화합니다.
- 10μL의 프로테이나제 K(10mg/mL)를 넣고 400rpm으로 진탕하면서 55°C에서 15분 동안 배양합니다.
- 실온에서 20,000 x g 로 3분 동안 원심분리하여 상층액을 수집하고 제조업체의 DNA/RNA 키트 프로토콜에 따라 진행합니다.
- 각 단계에서 350 μL의 세척 완충액을 사용하여 세척 단계를 두 단계로 나눕니다. 이러한 세척 단계 사이에 RNase가 없는 DNase I로 컬럼에 남아 있는 DNA를 분해합니다. 제조업체의 지침에 따라 준비된 DNase I 용액 80μL를 컬럼에 추가하고 실온에서 15분 동안 배양합니다.
- 뉴클레아제가 없는 물로 컬럼의 RNA/DNA를 용리합니다. 분리된 RNA는 -80°C에서 보관하고 gDNA는 -20°C에서 보관합니다.
- cDNA 합성
- 역전사 반응 전에 RNA 샘플을 15-30분(RNA 샘플에서 오염된 DNA를 완전히 제거하기 위해)의 또 다른 DNase I 처리에 적용합니다. 1 μL의 DNase I 용액, 4 μL의 완충액(키트와 함께 제공됨) 및 뉴클레아제가 없는 물을 40 μL의 최종 부피에 212 ng의 RNA에 추가합니다. 실온에서 30분 동안 배양하고 70°C에서 10분 동안 열 비활성화합니다.
- 제조업체의 지침에 따라 키트를 사용하여 150ng의 RNA를 사용하여 cDNA를 합성합니다. 샘플에서 바이러스 DNA를 오염시키지 않도록 역전사효소가 없는 대조군을 포함합니다. cDNA는 -20°C에서 보관된다.
참고: 최적의 역전사를 위한 입력 RNA의 양은 조직 유형과 각 조직에서 예상되는 형질도입 효율에 따라 달라질 수 있습니다.
- AAV 바이러스 라이브러리 분석(in-vivo) 제작: NGS
- 저렴한 비용으로 높은 시퀀싱 깊이를 얻으려면 앞서 설명한 대로 Illumina 시퀀싱을 통해 NGS를 수행하십시오(섹션 1.9). 바코드 시퀀스를 증폭한 다음 염기서열 분석 어댑터를 앰플리콘에 연결합니다.
- 판독 길이가 짧고 앰플리콘 양쪽에 있는 염기서열 분석 어댑터의 결찰로 인해 설계 시 앰플리콘이 NGS 판독 내에 바코드 서열이 있는지 확인할 수 있을 만큼 충분히 작은지 확인하십시오. 바이러스 게놈 내 바코드 및 바이러스 전사체의 염기서열 분석을 위해 PCR 앰플리콘은 길이가 113bp가 되도록 설계되었습니다( 그림 8 참조).
- 프라이머 BC-seq를 정방향 및 BC-seq로 역방향으로 바코드화된 영역을 증폭합니다. 다음 PCR 반응을 준비합니다: Hi-fidelity DNA 중합효소 0.5μL, 5x 완충액 10μL, 각 100μM 프라이머(BC-seq fw/BC-seq rv) 0.25μL 및 10mM dNTP 1μl. 25ng의 cDNA 또는 DNA/반응을 주형으로 사용하고 뉴클레아제가 없는 물로 최종 부피를 50μL로 조정합니다.
- 오염을 방지하기 위해 깨끗한 PCR 후드 아래에서 PCR 마스터 믹스를 준비합니다. 98°C에서 30초, 98°C에서 10초, 72°C에서 20초 동안 40회 사이클, 72°C에서 최종 5분 스텝을 사용합니다.
- PCR 마스터 믹스에 오염된 DNA가 없는지 확인하기 위해 PCR 대조군을 포함합니다. cDNA 샘플의 경우, 역전사효소가 없는 대조군을 포함한다. 마지막으로 AAV 입력 라이브러리와 함께 샘플을 포함합니다. 이 정보는 분석에 사용되는 Normalization_Variant.txt 파일을 생성하는 데 사용됩니다.
- PCR 정제 전에 겔 전기영동으로 각 시료의 PCR 단편의 크기를 검증한다. 후자는 상업적으로 이용 가능한 마그네틱 비드 또는 컬럼 기반 DNA 정제 시스템을 사용하여 달성됩니다( 재료 표 참조).
- 섹션 1.9에서 이전에 설명한 바와 같이 제조업체의 지침에 따라 라이브러리 시스템을 사용하여 NGS 라이브러리를 준비합니다.
- dsDNA HS 키트를 통해 DNA 농도를 측정하고 앞서 설명한 대로(섹션 1.9.6) 라이브러리의 품질을 분석한 다음 풀링합니다. 형광측정기에서 풀링된 라이브러리를 정량화하고 Bioanalyzer에서 품질을 평가합니다.
- 섹션 1.9.7에서 설명한 대로 NGS 시퀀싱을 수행합니다.
- qPCR에 의해 이식유전자(바이러스 게놈) 및 하우스키핑 유전자의 복제 수를 정량화하여 DNA 상의 조직 또는 기관 사이의 풀링된 라이브러리의 분포를 평가합니다.
- EYFP(이식유전자) 및 GAPDH(글리세르알데히드 3-포스페이트 탈수소효소, 하우스키핑 유전자)의 복제 수를 결정하기 위해 다음과 같이 30 μL qPCR 반응을 설정합니다.
- EYFP(1.5μM YFP_fw, 1.5μM YFP_rv 및 0.6μM YFP_probe 재료 표 참조)에 대한 60x 프라이머/프로브 혼합물을 준비합니다. GAPDH 프라이머/프로브 혼합물( 재료 표 참조)을 사용하여 하우스키퍼 유전자의 복제 수를 결정합니다. 얼음 위에 반응을 설정하십시오.
- PCR 마스터 믹스(15 μL, 재료 표 참조)를 준비하고 모든 샘플 및 표준물질에 대해 60x 프라이머/프로브 믹스(0.5 μL)를 추가합니다(표준물질의 복제 수를 계산하려면 다음 링크 http://cels.uri.edu/gsc/cndna.html 사용). 얼음 위에 반응을 설정하십시오.
- 마스터 믹스 15.5μL를 96웰 플레이트에 옮기고 14.5μL의 샘플(총 DNA 농도 75ng) 또는 표준물질을 각 웰에 추가합니다. 96웰 플레이트를 호일로 밀봉하고 소용돌이를 일으키고 잠시 회전시킵니다.
- 각 샘플 10μL을 384웰 플레이트에 이중으로 옮깁니다. 플레이트를 호일로 밀봉하고 4°C에서 5분 동안 800 x g 로 회전합니다.
- 반응 혼합물을 50°C의 초기 온도를 사용하여 열순환기에서 2분 동안 인큐베이션한 다음, 95°C에서 10분의 초기 활성화 단계를 수행하였다. 95°C에서 15초 동안 변성의 40사이클을 수행하고 60°C에서 1분24초 동안 어닐링/연장을 수행합니다.
- 이배체 게놈(dg)의 수를 얻으려면 GAPDH 복제 수를 사용하고 2로 나눕니다. 그런 다음 EYFP 복제 수의 값을 dg 수로 나누어 이배체 게놈당 벡터 게놈(vg/dg)을 생성합니다. 이 값을 사용하여 생물정보학 분석을 위한 Normalization_Organ.txt 파일을 생성합니다.
- Weinmann et al.과 같은 NGS 시퀀싱 데이터 분석을 수행합니다.24, Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022)의 사용자 지정 코드를 사용합니다. 워크플로우는 측면 염기서열, 길이 및 위치(Script#1_BarcodeDetection.py)에 의해 안내되는 바코드 염기서열의 검출과 조직 세트에 대한 바코드 농축 및 분포 분석(Script#2_BarcodeAnalysis.py)으로 구성됩니다.
- 바코드를 감지하여 AAV 변형에 할당합니다. 시퀀싱 데이터를 하나의 디렉토리(예: "Data_to_analyze")에 보관된 fastq 파일로 배치합니다. 입력 라이브러리의 시퀀싱 데이터 파일은 이 디렉터리에 포함되며 입력 라이브러리의 캡시드 비율을 계산하는 데만 사용됩니다.
- 스크립트를 실행하기 전에 탭으로 구분된 두 개의 텍스트 파일, 즉 AAV 캡시드 변형 이름에 할당된 바코드 시퀀스가 있는 캡시드 변형 파일(예제 파일 "Variants.txt") 및 오염 가능성이 있는 바코드 시퀀스가 포함된 오염 파일("오염.txt" 참조)을 생성합니다(실험실에서 사용 가능한 다른 바코드, 오염에 기여함).
- 마지막으로 구성 파일 "Barcode_Script.conf"를 편집하여 시퀀싱 데이터가 있는 폴더 경로(예: "Data_to_analyze"), 바코드 측면 영역 순서, 바코드 위치 및 바코드 감지를 위한 창 크기(1.9.8.5와 유사, 그림 8 참조)를 포함합니다.
- 다음 명령을 사용하여 Script#1_BarcodeDetection.py 및 구성 파일에 제공된 경로로 바코드 감지를 호출합니다.
>python3 ~/스크립트#1_BarcodeDetection.py ~/Barcode_Script.conf
참고: Script#1_BarcodeDetection.py 실행의 출력은 캡시드 변형당 읽기 횟수와 원시 데이터에서 복구된 총 읽기 수가 포함된 텍스트 파일입니다. - 다음 txt 파일과 함께 Script#2_BarcodeAnalysis.py를 실행하여 조직 또는 기관 간에 바코드화된 AAV 캡시드의 분포를 평가합니다.
- "Zuordnung.txt" 파일에서 각 txt 파일에 이름을 할당하여 바코드 감지 실행에서 얻은 조직/장기 이름(첫 번째 열의 txt 파일 이름 및 탭으로 구분된 할당의 해당 조직/장기 이름)을 할당합니다.
참고: 예를 들어ample, "Example" 폴더(https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022)를 확인하십시오. 참고로, 조직/기관 이름에는 cDNA 또는 gDNA 측정 및 생물학적 복제 수(M1, M2 등)를 정의하는 문자가 포함될 수 있습니다. - "Zuordnung.txt" 할당 파일에 지정된 이름에 해당하는 ON 및 OFF 대상 장기의 이름 목록이 포함된 "장기.txt" 텍스트 파일을 만듭니다("Example" 폴더: https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 참조).
- 모든 캡시드 변이체 및 모든 장기/조직에 대해 정규화된 값을 사용하여 "Normalization_Organ.txt" 및 "Normalization_Variant.txt" 탭으로 구분된 텍스트 파일을 만듭니다. "Normalization_Organ.txt"파일의 첫 번째 열에는 각 장기에 대해 주어진 이름 (할당 파일 "Zuordnung.txt"에서와 같이)을 쓰고 두 번째 열에는 섹션 3.6.11에서 생성 된 해당 조직의 정규화 값을 씁니다.
- "Normalization_Variant.txt" 파일의 첫 번째 열을 캡시드 이름 목록으로 채우고 두 번째 열을 풀링된 라이브러리의 각 캡시드에 대한 읽기 횟수의 정규화된 값으로 채웁니다(정규화는 첫 번째 스크립트의 결과로 생성된 입력 라이브러리의 txt 출력 파일을 기반으로 계산할 수 있음).
- 위에서 언급한 모든 추가 파일의 전체 경로를 지정하여 구성 파일을 편집합니다. 스크립트 # 2_BarcodeAnalysis.py을 다음과 같이 실행하십시오.
>python3 /스크립트#2_BarcodeAnalysis.py ~/Barcode_Script.conf
참고: 바코드 분석 스크립트는 앞서 설명한 여러 정규화 단계를 기반으로 서로 다른 조직 내에서 캡시드 분포의 상대 농도(RC) 값이 있는 텍스트 파일과 텍스트 파일 데이터를 병합된 매트릭스 데이터로 결합하는 스프레드시트 파일 등 여러 파일을 출력합니다. 후자는 클러스터 분석 및 시각화에 사용할 수 있습니다. - 데이터를 시각화하고 매트릭스 데이터의 클러스터 분석을 수행하여 캡시드 특성을 구별하고 조직 전체의 RC 프로파일을 기반으로 유사성을 평가합니다. 추가 스크립트 PCA_heatmap_plot 사용합니다. R은 저장소에 배치됩니다.
>rscript --vanilla ~/PCA입니다. R ~/상대농도.xls
참고: 스크립트는 relativeconcentration.xls 파일을 입력으로 사용하고 계층적 클러스터 히트맵과 주성분 분석(PCA)의 두 플롯을 생성합니다. - 플롯(히트맵의 축, PCA의 주성분) 또는 png 매개변수(색상, 크기, 레이블 지정)를 수정하려면 R 스크립트를 열고 주석 처리된 섹션에 제공된 지침을 따릅니다.
- "Zuordnung.txt" 파일에서 각 txt 파일에 이름을 할당하여 바코드 감지 실행에서 얻은 조직/장기 이름(첫 번째 열의 txt 파일 이름 및 탭으로 구분된 할당의 해당 조직/장기 이름)을 할당합니다.
Access restricted. Please log in or start a trial to view this content.
결과
AAV2 펩타이드 디스플레이 라이브러리의 생성. 조작된 AAV를 선택하기 위한 첫 번째 단계로, 플라스미드 라이브러리의 생성에 대해 설명합니다. 펩티드 삽입물은 퇴화 프라이머를 사용하여 생산된다. 64에서 20으로 코돈의 조합을 줄이면 단백질 수준이 아닌 DNA의 라이브러리 다양성을 감소시킴으로써 정지 코돈을 제거하고 NGS 분석을 용이하게 하는 이점이 있습니다. 올리고뉴클레오티?...
Access restricted. Please log in or start a trial to view this content.
토론
이 프로토콜에서는 펩타이드 디스플레이 AAV 캡시드 엔지니어링 및 바코드 AAV 라이브러리 스크리닝, 라이브러리 구성 및 캡시드 성능의 생물정보학적 분석에 필요한 단계를 간략하게 설명합니다. 이 프로토콜은 대부분의 바이러스학 실험실이 분자 생물학 기술에 대한 숙련도에 맞게 프로그래밍 기술이 뒤쳐져 있기 때문에 이러한 유형의 라이브러리에 대한 생물정보학적 분석을 용이하게 하는 단...
Access restricted. Please log in or start a trial to view this content.
공개
D.G.는 AaviGen GmbH의 공동 설립자이며, D.G.와 K.R.은 면역 회피 AAV 캡시드 변이체의 생성과 관련하여 출원중인 특허 출원의 발명가입니다. 나머지 저자는 공개 할 것이 없습니다.
감사의 말
D.G.는 DFG 공동 연구 센터 SFB1129 (Projektnummer 240245660) 및 TRR179 (Projektnummer 272983813)와 독일 감염 연구 센터 (DZIF, BMBF; TTU-HIV 04.819).
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
참고문헌
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621(2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756(2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075(2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041(2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432(2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819(2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899(2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272(2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200(2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825(2021).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유