É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Isolamento de vetores de terapia gênica de próxima geração por meio de engenharia, código de barras e triagem de variantes do capsídeo do vírus adenoassociado (AAV)
Neste Artigo
Resumo
Geração de biblioteca de exibição de peptídeos AAV e posterior validação através do código de barras de candidatos com novas propriedades para a criação de AAVs de próxima geração.
Resumo
Os vetores de liberação gênica derivados do vírus adenoassociado (AAV) são uma das ferramentas mais promissoras para o tratamento de doenças genéticas, evidenciada por dados clínicos encorajadores e pela aprovação de várias terapias gênicas com AAV. Duas razões principais para o sucesso dos vetores AAV são (i) o isolamento prévio de vários sorotipos virais naturais com propriedades distintas, e (ii) o subsequente estabelecimento de tecnologias poderosas para sua engenharia molecular e reaproveitamento em alto rendimento. Aumentando ainda mais o potencial dessas técnicas são recentemente implementadas estratégias para a codificação de barras-capsídeos AAV selecionados no nível de DNA e RNA, permitindo sua estratificação in vivo abrangente e paralela em todos os principais órgãos e tipos celulares em um único animal. Aqui, apresentamos um pipeline básico que engloba esse conjunto de vias complementares, usando a exibição de peptídeos AAV para representar o arsenal diversificado de tecnologias de engenharia de capsídeos disponíveis. Assim, primeiro descrevemos as etapas fundamentais para a geração de uma biblioteca de exibição de peptídeos AAV para a seleção in vivo de candidatos com propriedades desejadas, seguido por uma demonstração de como codificar as variantes de capsídeos mais interessantes para triagem secundária in vivo. Em seguida, exemplificamos a metodologia para a criação de bibliotecas para sequenciamento de próxima geração (NGS), incluindo amplificação de código de barras e ligadura de adaptador, antes de concluir com uma visão geral das etapas mais críticas durante a análise de dados NGS. Como os protocolos relatados aqui são versáteis e adaptáveis, os pesquisadores podem facilmente aproveitá-los para enriquecer as variantes ideais do capsídeo AAV em seu modelo de doença favorito e para aplicações de terapia gênica.
Introdução
A terapia de transferência gênica é a introdução de material genético nas células para reparar, substituir ou alterar o material genético celular para prevenir, tratar, curar ou melhorar doenças. A transferência de genes, tanto in vivo quanto ex vivo, depende de diferentes sistemas de entrega, não virais e virais. Os vírus evoluíram naturalmente para transduzir eficientemente suas células-alvo e podem ser usados como vetores de entrega. Dentre os diferentes tipos de vetores virais empregados na terapia gênica, os vírus adenoassociados têm sido cada vez mais utilizados, devido à sua falta de patogenicidade, segurança, baixa imunogenicidade e, principalmente, sua capacidade de sustentar a expressão não integradora a longo prazo 1,2,3. A terapia gênica com AAV produziu conquistas consideráveis na última década; três terapias foram aprovadas pela Agência Europeia de Medicamentos e pela Food and Drug Administration dos Estados Unidos para uso em humanos 3,4. Vários ensaios clínicos também estão em andamento para tratar uma variedade de doenças, como hemofilia, doenças musculares, cardíacas e neurológicas, conforme revisado em outros estudos3. Apesar de décadas de avanço, o campo da terapia gênica tem experimentado uma série de contratempos nos últimos anos4 e, principalmente mortes em ensaios clínicos5 que foram suspensas devido a toxicidades limitantes da dose, particularmente para tecidos maciços, como o músculo, ou de difícil acesso, como océrebro6.
Os vetores do AAV atualmente utilizados em ensaios clínicos pertencem aos sorotipos naturais, com algumas exceções1. A engenharia AAV oferece a oportunidade de desenvolver vetores com especificidade e eficiência superior de órgãos ou células. Nas últimas duas décadas, várias abordagens têm sido aplicadas com sucesso, tais como exibição de peptídeos, loop-swap, embaralhamento de DNA do capsídeo, PCR propenso a erros e design direcionado, para gerar variantes individuais de AAV ou bibliotecas com propriedades diversas7. Estes são então submetidos a múltiplas rodadas de evolução dirigida para selecionar as variantes dentro deles com as propriedades desejadas, conforme revisado em outro artigo 1,3. De todas as estratégias de evolução do capsídeo, as bibliotecas AAV de exibição de peptídeos têm sido as mais utilizadas, devido a algumas propriedades únicas: são relativamente fáceis de gerar, e podem alcançar alta diversidade e sequenciamento de alto rendimento, o que permite acompanhar sua evolução.
As primeiras bibliotecas de AAV de inserção peptídica bem-sucedidas foram descritas há quase 20 anos. Em um dos primeiros, Perabo et al.8 construíram uma biblioteca de capsídeos AAV2 modificados, na qual um pool de oligonucleotídeos gerados aleatoriamente foi inserido em um plasmídeo em uma posição que corresponde ao aminoácido 587 da proteína VP1 capsídeo, no eixo de três vezes saliente do capsídeo. Usando a co-infecção de adenovírus, a biblioteca de AAV foi evoluída através de várias rodadas de seleção, e as variantes finais redirecionadas mostraram-se capazes de transduzir linhagens celulares refratárias ao AAV2 parental8. Pouco tempo depois, Müller et al.9 introduziram o sistema de duas etapas para a produção de bibliotecas, uma melhoria significativa do protocolo. Inicialmente, a biblioteca de plasmídeos, juntamente com um plasmídeo auxiliar adenoviral, são usados para produzir uma biblioteca de AAV que contém capsídeos quiméricos. Esta biblioteca de transporte AAV é usada para infectar células com baixa multiplicidade de infecção (MOI), com o objetivo de introduzir um genoma viral por célula. A co-infecção com adenovírus garante a produção de AAVs com genoma e capsídeo9 correspondentes. Cerca de uma década depois, Dalkara10 usou a evolução dirigida in vivo para criar a variante 7m8. Essa variante possui inserção de 10 aminoácidos (LALGETTRPA), três dos quais atuam como ligantes, e atingem eficientemente a retina externa após injeção intravítrea10. Este capsídeo projetado é uma história de sucesso excepcional, pois é um dos poucos capsídeos projetados a chegar à clínica até agora11.
O campo experimentou um segundo impulso com a introdução de técnicas de sequenciamento de próxima geração (NGS). Duas publicações de Adachi et al.12, em 2014, e de Marsic et al.13, em 2015, mostraram o poder da NGS para rastrear a distribuição de bibliotecas de capsídeos AAV com código de barras com alta precisão. Alguns anos depois, a NGS das regiões com código de barras foi adaptada à região de inserção do peptídeo para acompanhar a evolução do capsídeo. Körbelin et al.14 realizaram uma triagem guiada por NGS para identificar um capsídeo baseado em AAV2 direcionado para a pulmonar. A análise da NGS ajudou a calcular três escores de avaliação: o escore de enriquecimento entre as rodadas de seleção, o escore de especificidade geral para determinar a especificidade tecidual e, finalmente, o escore combinado14. O laboratório Gradinaru15 publicou o sistema de evolução direcionada AAV baseado em recombinação Cre (CREATE) no mesmo ano, o que facilita uma seleção específica do tipo de célula. Neste sistema, a biblioteca capsid carrega um switch Cre-invertible, já que o sinal polyA é flanqueado por dois sites loxP. A biblioteca AAV é então injetada em camundongos Cre, onde o sinal poliA é invertido apenas em células Cre+, fornecendo o molde para a ligação de um primer de PCR reverso com o primer forward dentro do gene do capsídeo. Este resgate por PCR altamente específico possibilitou a identificação do AAV-PHP. Variante B que pode atravessar a barreira hematoencefálica15. Este sistema evoluiu ainda para o M-CREATE (Multiplexed-CREATE), no qual NGS e geração de bibliotecas sintéticas foram integradas no pipeline16.
Uma versão melhorada desse sistema baseada em RNA do laboratório Maguire17, o iTransduce, permite a seleção no nível de DNA de capsídeos que transduzem funcionalmente as células e expressam seus genomas. O genoma viral da biblioteca de exibição de peptídeos compreende um gene Cre sob o controle de um promotor ubíquo e o gene do capsídeo sob o controle do promotor p41. A biblioteca é injetada em ratos que têm um loxP-STOP-loxP a montante de tdTomato. Células transduzidas com variantes do AAV que expressam o genoma viral e, portanto, Cre expressam tdTomato e, em combinação com marcadores celulares, podem ser classificadas e selecionadas17. Da mesma forma, Nonnenmacher et al.18 e Tabebordbar et al.19 colocaram a biblioteca gênica do capsídeo sob o controle de promotores tecido-específicos. Após injeção em diferentes modelos animais, o RNA viral foi usado para isolar as variantes do capsídeo.
Uma abordagem alternativa é usar código de barras para marcar bibliotecas de capsídeos. O laboratório de Björklund20 usou essa abordagem para bibliotecas de capsídeos de inserção de peptídeos de código de barras e desenvolveu a evolução vetorial AAV racional com código de barras (BRAVE). Em um plasmídeo, o Rep2Cap é clonado ao lado de um transgene marcado com código de barras e repetições terminais invertidas (ITR), com expressão de proteína fluorescente amarela (YFP). Usando sítios loxP entre o final da tampa e o início do código de barras, uma recombinação de Cre in vitro gera um fragmento pequeno o suficiente para NGS, permitindo assim a associação da inserção do peptídeo com o código de barras único (look-up table, LUT). A produção de AAV é realizada utilizando a biblioteca de plasmídeos e os códigos de barras expressos no RNAm são triados após aplicação in vivo , novamente com NGS20. Quando as bibliotecas do capsídeo compreendem variantes de todo o gene do capsídeo (ou seja, bibliotecas embaralhadas), o sequenciamento de leitura longa precisa ser usado. Vários grupos têm usado códigos de barras para marcar essas diversas bibliotecas, o que permite NGS com maior profundidade de leitura. O laboratório Kay21 marcou bibliotecas embaralhadas de capsídeos altamente diversos com códigos de barras a jusante do sinal poliA da tampa . Em uma primeira etapa, uma biblioteca de plasmídeos com código de barras foi gerada, e a biblioteca de genes do capsídeo embaralhada foi clonada nela. Em seguida, uma combinação de MiSeq (leitura curta, maior profundidade de leitura) e PacBio (leitura longa, menor profundidade de leitura), NGS e sequenciamento de Sanger foram usados para gerar sua LUT21. Em 2019, Ogden e colegas do laboratório da Igreja22 delinearam a aptidão do capsídeo AAV2 para múltiplas funções usando bibliotecas que tinham mutações de ponto único, inserções e exclusões em todas as posições, o que finalmente permitiu o design guiado por máquina. Para a geração da biblioteca, fragmentos menores do gene do capsídeo foram sintetizados, marcados com um código de barras, sequenciados de próxima geração e, em seguida, clonados no gene completo do capsídeo. Os dados de NGS foram usados para gerar uma LUT. A biblioteca foi então triada utilizando-se apenas os códigos de barras e o sequenciamento de leitura curta, o que, por sua vez, permite maior profundidade de leitura22.
As bibliotecas com código de barras têm sido predominantemente usadas para selecionar um conjunto de variantes conhecidas, naturais e projetadas após várias rodadas de seleção de bibliotecas de capsídeos ou independentes de um estudo de evolução de capsídeos. A vantagem de tais bibliotecas é a oportunidade de rastrear vários capsídeos, reduzindo o número de animais e minimizando a variação entre os animais. Os primeiros estudos que introduziram essa tecnologia no campo dos AAV foram publicados há quase uma década. O laboratório Nakai 12 marcou 191 mutantes duplos de alanina cobrindo aminoácidos 356 a 736 no VP1 de AAV9 com um par de códigos de barras de12 nucleotídeos. Usando NGS, a biblioteca foi triada in vivo para ligação de galactose e outras propriedades12. Marsic e colaboradores delinearam a biodistribuição de variantes do AAV usando também uma análise de barra dupla 1 ano depois13. Um estudo mais recente em primatas não humanos comparou a biodistribuição no sistema nervoso central de 29 capsídeos utilizando diferentes vias de parto23. Nosso laboratório publicou recentemente telas de biblioteca AAV com código de barras de 183 variantes que incluíam AAVs naturais e projetados. Essas telas no nível de DNA e RNA levaram à identificação de uma variante altamente miotrópica do AAV24 em camundongos, bem como outras exibindo uma alta especificidade do tipo celular no cérebro de camundongos25.
Aqui, descrevemos a metodologia usada neste trabalho e a expandimos para incluir a triagem de bibliotecas de exibição de peptídeos AAV. Isso compreende a geração de bibliotecas de exibição de peptídeos AAV2, um método de PCR digital em gotículas (dd-PCR) para quantificação e, finalmente, um pipeline NGS para analisar as variantes do AAV, baseado em parte no trabalho de Weinmann e colaboradores24. Finalmente, uma descrição da geração de bibliotecas AAV com código de barras e do pipeline NGS usado na mesma publicação é fornecida.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. AAV2 aleatório 7-mer peptídeo display biblioteca preparação
NOTA: Para a preparação de uma biblioteca de exibição de peptídeos aleatórios AAV2, sintetize os oligonucleotídeos degenerados como DNA de fita simples, converta-o em DNA de fita dupla, digerir, ligar-se ao plasmídeo aceitador e eletroporato.
- Projeto de oligonucleotídeos degenerados
- Ordene os oligonucleotídeos degenerados e evite o viés do códon. No oligonucleotídeo 5' CAGTCGGCCAG AG W GGC (X01)7 GCCCAGGCGGCTGACGAG 3', X01 corresponde a 20 códons, cada um codificando um dos 20 aminoácidos. O W pode ser A ou T, produzindo os códons AGA ou AGT, que codificam os aminoácidos arginina (R) ou serina (S).
- Solicite o primer de amplificação: 5' CTCGTCAGCCGCCTGG 3' (veja a Figura 1 para detalhes). Isso produz a seguinte inserção de proteína: R/S G X7. A diversidade teórica é calculada da seguinte forma: 1 x 2 x 207 = 2,56 x 109 variantes únicas.
NOTA: Deve-se notar que essa diversidade pode ser restringida pela eficiência da transformação.
- Síntese de segunda vertente
- Ressuspender ambos os oligonucleotídeos (oligonucleotídeos degenerados e primer de amplificação) para uma concentração final de 100 μM com tampão TE.
- Para a reação de PCR, estabelecer uma reação de 50 μL com 1 μL de cada primer, 10 μL de tampão, 1,5 μL de DMSO, 0,5 μL de dNTPs (10 mM), 0,5 μL de Polimerase de Início Quente de Alta Fidelidade II e 35,5 μL de água livre de nuclease.
- Transfira a reação para um termociclador e execute uma etapa de pré-incubação por 10 s a 98 °C, seguida por três ciclos de 10 s a 98 °C, 30 s a 59 °C e 10 s a 72 °C, depois 5 min a 72 °C e uma etapa final de resfriamento.
- Purificar a reação usando um kit de remoção de nucleotídeos e eluir em 100 μL de água livre de nucleases.
- Confirme a eficiência da síntese da segunda fita por análise em um Bioanalisador (ver Figura 2). Analise o tamanho e a pureza da pastilha de fita dupla carregando 1 μL da reação a um chip microfluídico de um kit de reagentes DNA 1000 de acordo com as instruções do fabricante. Este kit é otimizado para medir o tamanho e a concentração de fragmentos de DNA de fita dupla de 25-1.000 bps.
- Ressuspender ambos os oligonucleotídeos (oligonucleotídeos degenerados e primer de amplificação) para uma concentração final de 100 μM com tampão TE.
- Digestão de inserção e vetor plasmidial
- Digerir 85 μL da pastilha purificada com 10 μL de tampão 10x e 5 μL da enzima BglI num volume final de reacção de 100 μL (ver figura 1 para mais pormenores). Incubar a 37 °C durante a noite. Purificar usando um kit de remoção de nucleotídeos, eluir em 50 μL de água livre de nucleases e quantificar usando o tipo "Oligo DNA" em espectrofotômetro.
- Digerir 10 μg de um plasmídeo AAV competente em replicação (pRep2Cap2_PIS)26 (genoma viral flanqueado por ITR) com 20 μL de tampão 10x e 10 μL de enzima SfiI em um volume final de reação de 200 μL (ver Figura 1 para detalhes). Incubar a 50 °C durante a noite. Purificar o vetor em um gel de agarose a 1% usando o kit de extração em gel, seguido por uma etapa adicional de purificação usando um kit de purificação de DNA. Quantificar a concentração em espectrofotômetro.
- Ligadura do inserto ao vetor
- Ligate 955 ng de vetor plasmidial com 45 ng de inserção com 2 μL de tampão e 2 μL de ligase em uma reação de ligadura de 20 μL. Incubar a 16 °C durante a noite, seguido de 10 min a 70 °C para inativar a ligase pelo calor.
- Transformação, cálculo de complexidade e preparação da biblioteca de plasmídeos
- Purifice a reação com um kit purificador de DNA seguindo as instruções do fabricante. Eluir a reação em cerca de 80% do volume inicial de água livre de nucleases e armazenar no gelo para posterior transformação.
- Transforme células eletrocompetentes: descongele um frasco de células eletrocompetentes no gelo por 10 min. Em seguida, adicione 1-2 μL da reação de ligadura purificada a 30 μL (um frasco para injetáveis) de células eletrocompetentes e misture batendo suavemente. Em seguida, pipetar cuidadosamente a mistura célula/DNA para uma cubeta de eletroporação pré-refrigerada de 1 mm sem introduzir bolhas de ar.
- Eletroporato usando as seguintes configurações: 1800 V, 600 Ω e 10 μF. Dentro de 10 s do pulso de eletroporação, adicione 970 μL de meio de recuperação pré-aquecido (fornecido com as células eletrocompetentes) à cubeta e misture por pipetagem. Por fim, transfira as células para um tubo de microcentrífuga e incube por 1 h a 37 °C a 250 rpm. Para alcançar a diversidade desejada, realize de 10 a 100 reações e, após a incubação, agrupe todas as reações em um tubo.
- Calcular a diversidade diluindo 10 μL das transformações agrupadas 10, 100 ou 1.000 vezes em PBS e espalhar 100 μL em placas de ágar nutriente contendo o antibiótico apropriado (75 mg/mL de ampicilina). Incubar as placas de ágar durante a noite a 37 °C e, em seguida, contar as colónias nas placas de ágar.
- Calcule a diversidade teórica da seguinte forma:
Diversidade teórica máxima = 10 x fator de diluição x número de colônias x número de reações de eletroporação.
NOTA: Para confirmar a qualidade da biblioteca, sequencie pelo menos 20 colônias por sequenciamento de Sanger. A maioria dos clones deve conter uma inserção e todos devem ser exclusivos. - Inocular 400-1.000 mL de meio LB contendo o antibiótico apropriado com o restante das transformações agrupadas e incubar durante a noite a 37 °C, 180 rpm.
- Preparação da biblioteca de plasmídeos
- A partir da cultura noturna, preparar um estoque de glicerol (misturar volumes iguais de cultura bacteriana e solução de glicerol a 50% em água livre de nuclease e congelar a -80 °C) e purificar a biblioteca de plasmídeos usando um kit maxi de plasmídeo.
- Produção da biblioteca viral de AAV
- Preparar a biblioteca viral conforme descrito anteriormente27. Transfectar a biblioteca de plasmídeos (pRep2Cap2_PI, inserção de peptídeos) juntamente com um plasmídeo adeno-helper para células HEK293T usando um reagente de transfecção como a polietilenimina (PEI).
- Recolher as células após 3 dias e submetê-las a três ciclos de congelamento-descongelamento. Purificar o lisado viral usando ultracentrifugação com gradiente de cloreto de césio, seguido de troca tampão para PBS e, finalmente, concentrar as partículas virais.
- Titulação vetorial de AAV por dd-PCR
- Diluir serialmente 2 μL do estoque do vetor AAV em 198 μL de água livre de nuclease para obter uma diluição final de 1:106 . Misture bem cada vez usando uma pipeta de 200 μL. Adicione um controle sem modelo (NTC) como um controle negativo.
NOTA: Diluições adicionais inferiores ou superiores podem ser ensaiadas (1:105-1:107). - Prepare uma mistura de primer-sonda de 20x. Adicionar 3,6 μL de cada um dos primers de 100 μM (forward e reverse, Rep2 e ITR), 1 μL de cada uma das sondas de 100 μM dd-PCR (Rep2 e ITR) e 3,6 μL de água livre de nucleases a um tubo de centrífuga de 1,5 mL.
NOTA: A biblioteca AAV é medida usando um conjunto de sondas de primer direcionadas a transgenes (Rep2) detectadas com uma sonda marcada com FAM e um conjunto de sondas de primer direcionadas a ITR detectadas com uma sonda marcada com HEX. - Preparar uma reação de PCR de 22 μL adicionando 5,5 μL de amostra, 1,1 μL de mistura 20x primer-sonda, 11 μL de supermistura dd-PCR para sondas (sem dUTP) e 4,4 μL de água livre de nuclease. Isso produz concentrações de 900 nM e 250 nM para os primers e a sonda, respectivamente.
- Gere as gotículas usando um gerador de gotículas, transfira a reação para uma placa de 96 poços, coloque a placa em um termociclador e execute uma etapa de desnaturação por 10 min a 94 °C, seguida por 40 ciclos de 30 s a 94 °C e 1 min a 58 °C. Em seguida, inative a polimerase por 10 min a 98 °C e adicione uma etapa final de resfriamento. Leia as reações em um leitor de gotículas e prossiga para a análise28.
- Abra o arquivo de placa dd-PCR salvo usando o software de análise. Use a ferramenta de limite na guia Amplitude 1D (amplitude de fluorescência vs. número de evento) para separar as gotículas negativas e positivas para cada canal, usando o NTC como guia, e exportar os dados para um arquivo csv.
- Para calcular a concentração vetorial, primeiro calcule o fator de correção CF usando a fórmula:
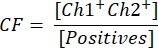
A FC determina a proporção de gotículas positivas para o transgene [Positivos] que são positivas para ambos, transgene e ITR [Ch1+ Ch2+], para garantir a detecção de partículas vetoriais funcionais. A concentração vetorial final c pode agora ser calculada usando a seguinte equação: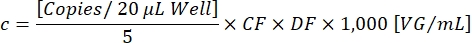
DF é o fator de diluição (1:105-1:107 conforme determinado anteriormente). As cópias por reação de 20 μL/poço correspondem a 5 μL da amostra diluída. O fator 1.000 corrige a escala para VG/mL (genoma viral/mL). Um resultado exemplar de titulação é demonstrado na Tabela 1 e na Figura 3.
- Diluir serialmente 2 μL do estoque do vetor AAV em 198 μL de água livre de nuclease para obter uma diluição final de 1:106 . Misture bem cada vez usando uma pipeta de 200 μL. Adicione um controle sem modelo (NTC) como um controle negativo.
- Análise da biblioteca viral de AAV por NGS
- Amplificar o fragmento de inserção do peptídeo de 96 nucleotídeos configurando uma reação de PCR de 20 μL usando um kit de revisão de polimerase (2x; ver Figura 4). Adicionar 1 μL de estoque de AAV contendo 1 x 108 vg, 0,5 μL de cada um dos 100 μM de primer (NGS_forward e NGS_reverse) e 10 μL da mistura enzimática à reação. Ajustar o volume final para 20 μL com água sem nuclease.
- Transfira a reação para um termociclador e execute uma etapa de desnaturação por 3 min a 98 °C, seguida por 30-35 ciclos de 10 s a 98 °C, 10 s a 59 °C e 20 s a 72 °C, seguido por 5 min a 72 °C e uma etapa final de resfriamento.
- Purificar as amostras usando um kit de purificação PCR. Quantifique a concentração em espectrofotômetro e execute um gel de agarose a 3% para verificar a pureza e o tamanho do fragmento.
- Processar os fragmentos de PCR usando o sistema de bibliotecas para amostras de baixa complexidade kit de acordo com as instruções do fabricante para a preparação de uma biblioteca NGS. Realizar a reação de reparo final com 30 ng de fragmento de PCR, seguido de ligadura do adaptador e amplificação por PCR por 10 ciclos. Use o kit de purificação PCR para a purificação das reações.
- Processar os produtos finais em um Bioanalisador para verificar o tamanho e pureza, usando um kit de reagentes de DNA de acordo com as instruções do fabricante.
- Quantifique os amplicons usando um fluorômetro e agrupe-os. Quantificar a biblioteca NGS agrupada final novamente em um fluorômetro (de acordo com as instruções do fabricante) e verificar a qualidade em um Bioanalisador.
- Sequencie as bibliotecas NGS em um modo de extremidade única (SE), usando um kit de alta saída de 75 ciclos, com um comprimento de leitura de 84 e um índice 1 de 8.
NOTA: O sequenciamento dos exemplos neste artigo foi realizado nas instalações GeneCore da EMBL Heidelberg (http://www.genecore.embl.de/). - Analise os dados de sequenciamento NGS com Python 3 e biopython. Os arquivos podem ser encontrados em https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (alternativamente em https://doi.org/10.5281/zenodo.7032215). A análise da NGS é composta por duas etapas.
- Na primeira etapa, pesquise nos arquivos de sequência sequências que satisfaçam determinados critérios (presença de sequências de reconhecimento flanqueando o local de inserção) (ver Figura 4, etapa 1.9.8.5.). Isso é feito usando um script (Script#1) e um arquivo de configuração que fornece as informações necessárias. Uma vez que a sequência correta é identificada, o programa extrai e armazena a sequência no arquivo de saída, que é um arquivo txt com o mesmo nome do arquivo de sequenciamento.
- O segundo passo é a análise dos arquivos de saída. As sequências na biblioteca começam com qualquer um dos seis nucleotídeos (AGWggc, W =A/T) na inserção de nove aminoácidos. Com base nessa sequência inicial, o peptídeo é traduzido. Isso gera os arquivos de saída que contêm as variantes de peptídeo (PVs).
- Prepare duas pastas: Script e Dados. Para a pasta Dados, copie os arquivos compactados com gzip resultantes do sequenciamento. Para a pasta Script, copie os seguintes arquivos, arquivo Python: Script#1_DetectionExtraction_JoVE_Py3.py; Arquivo Python: Script#2_PV_extraction_and_ranking_Py3.py; Arquivo de configuração: Barcode_Script_JoVE.conf; e arquivo de tabela de pesquisa (LUT): Zuordnung.txt.
- Antes de executar os scripts, edite os seguintes arquivos na pasta Script. Abra o arquivo "Zuordnung.txt" e adicione em duas colunas separadas por tabulações, os nomes dos arquivos gzip (coluna 1) e o nome final desejado (coluna 2; valores separados por tabulação).
Observação : arquivos txt de exemplo são encontrados na pasta GitHub "PV_analysis_script". Os arquivos fornecidos na pasta GitHub são preparados para a análise de três dados de exemplo da biblioteca acima: xaa.txt.gz, xab.txt.gz e xac.txt.gz. Os arquivos de saída também são fornecidos. - Altere as seguintes variáveis no arquivo de configuração "Barcode_Script_JoVE.conf":
my_dir = "~/Dados/"
filename_sample_file = "~/Script/Zuordnung.txt"
As variáveis específicas da sequência: BCV_size = 27, BCVesquerda = TCCAGGGCCAG, BCVdireita = GCCCAGG, BCVloc = 30,margem BCV = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC e BCVloc_revcomp = 41 (veja a Figura 4 para detalhes). - Use o seguinte comando para chamar a detecção e extração de sequência de variantes:
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
NOTA: A saída são arquivos txt com as sequências de DNA extraídas e seus números de leituras. O cabeçalho desse arquivo contém dados estatísticos (ou seja, o número total de leituras e as leituras extraídas). Esses dados são transferidos para os próximos arquivos. Esses dados txt são os arquivos de entrada para o Script#2, nos quais as sequências de DNA são traduzidas, classificadas e analisadas. - Execute a extração e análise PV usando o seguinte comando:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - Analise os arquivos de saída de texto do Script #2. Os arquivos de saída do Script#2 são nomeados usando a segunda coluna do LUT em "Zuordnung.txt" com extensões baseadas no tipo de análise.
NOTA: Certifique-se de que os três arquivos de saída contenham dados estatísticos nas primeiras linhas ("# de leituras PV válidas", "# de leituras PV inválidas" e "# de leituras PV exclusivas"), uma primeira coluna com o índice de cada sequência de DNA dos arquivos txt de entrada (saída do Script #1) e as seguintes colunas: (1) "... analyzed_all.csv": "Amostra:" (sequência de DNA), "#" (número de leituras), "Frw ou Rev" (leitura direta ou reversa) e "PVs" (sequência peptídica traduzida). As sequências inválidas têm "NA" e "not valid" nas duas últimas colunas. (2) "... analyzed_validSeq.csv": igual ao arquivo anterior, filtrado para sequências válidas. (3) "... analyzed_PV.csv": "PVs" (sequência peptídica traduzida), "#" (número de leituras) e "count" (as contagens frw e rev nos arquivos anteriores são mescladas e a contagem é dada 1 ou 2). - Visualize os arquivos de saída usando o software disponível com base nas necessidades do usuário.
2. AAV2 seleção aleatória da biblioteca de exibição de peptídeos de 7 meros
- Use a biblioteca AAV após quantificação e controle de qualidade (seção 1) para evolução dirigida em um modelo de escolha para selecionar iterativamente candidatos com propriedades desejadas (ver Figura 5)16,18,21.
NOTA: Esses candidatos são usados para a geração de uma biblioteca com código de barras, conforme descrito abaixo na seção 3.
3. Preparação e análise da biblioteca de capsídeos AAV com código de barras
NOTA: Após a identificação de um conjunto de capsídeos AAV potencialmente específicos e eficientes na tela de exibição de peptídeos, verifique a funcionalidade das sequências peptídicas identificadas e compare-as com um conjunto de variantes de capsídeos AAV de referência comumente usadas ou bem descritas. Para fazer isso, a sequência do capsídeo é inserida em uma construção auxiliar Rep/Cap sem ITRs.
- Produção de biblioteca AAV com código de barras
- Realizar a produção de AAV recombinante para cada variante do capsídeo utilizando o sistema de três plasmídeos, conforme descrito anteriormente24.
NOTA: Para distinguir as diferentes variantes do capsídeo, o plasmídeo transgênico do repórter do ITR abriga um código de barras único de 15 nucleotídeos de comprimento. O código de barras está localizado na UTR 3' (região não traduzida) entre a proteína fluorescente amarela reforçada (EYFP) e o sinal poliA (ver Figura 6A). A expressão de EYFP é impulsionada por um forte promotor de citomegalovírus (CMV) ubíquo que fornece níveis suficientes de transcritos de RNA. - Projetar códigos de barras de 15 nucleotídeos de comprimento com homopolímeros de menos de três nucleotídeos, conteúdo de CG de <65%29 e distância de Hamming maior que quatro nucleotídeos24.
- Produza cada capsídeo separadamente em combinação com um plasmídeo transgênico que carrega um código de barras único. Dessa forma, cada variante do capsídeo é marcada com um código de barras distinto que permite seu rastreamento específico (consulte a Figura 6B).
- Realizar a produção de AAV recombinante para cada variante do capsídeo utilizando o sistema de três plasmídeos, conforme descrito anteriormente24.
- Titulação vetorial de AAV por dd-PCR
- Realizar a titulação do AAV conforme descrito anteriormente na secção 1.8, substituindo o par de primers Rep2 pelo par de primers YFP.
- Quantifique as produções AAV individuais e agrupe quantidades iguais de cada produção para gerar a biblioteca final com código de barras.
- Quantificar novamente a biblioteca final para verificar a concentração e a qualidade finais (ver Figura 7).
- Biblioteca AAV com código de barras aplicação in vivo
- Aplique a biblioteca AAV com código de barras sistemicamente ao sistema modelo de escolha (por exemplo, sistemicamente em ratos24).
- Coletar tecidos alvo ON e OFF (ou seja, fígado, pulmão, coração, diafragma, músculo liso, duodeno, pâncreas, cólon, bíceps, ovários, estômago, orelha interna, rim, aorta abdominal, aorta torácica, cérebro, gordura marrom e branca e baço) ou tipos celulares com base no experimento. Congelá-los a -80 °C, extrair o DNA/RNA e aplicar a análise de quantificação de NGS, conforme descrito na próxima seção.
- Extração de DNA/RNA
- Extrair o DNA e RNA dos tecidos de interesse utilizando o Mini Kit DNA/RNA.
- Coloque um pequeno pedaço do tecido de interesse (1 mm3, cerca de 5 mg) em um tubo de reação de 2 mL.
- Adicionar 350 μL de tampão de lise misturado com β-mercaptoetanol (1%) e esferas de aço de 5 mm ao tecido (manusear amostras com β-mercaptoetanol sob uma capela de fumos).
- Homogeneizar o tecido em um tissueLyser por 45 s a 40 Hz.
- Adicionar 10 μL de proteinase K (10 mg/mL) e incubar por 15 min a 55 °C enquanto agita a 400 rpm.
- Centrifugar a 20.000 x g por 3 min à temperatura ambiente, coletar o sobrenadante e prosseguir com o protocolo do fabricante do Kit DNA/RNA.
- Divida a etapa de lavagem em duas etapas com 350 μL de tampão de lavagem em cada etapa. Entre estas etapas de lavagem, digerir o ADN remanescente na coluna com DNase I sem RNase. Adicionar 80 μL da solução de DNase I, preparada de acordo com as instruções do fabricante, na coluna e incubar à temperatura ambiente durante 15 minutos.
- Elute RNA/DNA da coluna com água livre de nucleases. Armazenar o RNA isolado a -80 °C e o gDNA a -20 °C.
- síntese de cDNA
- Submeter as amostras de RNA a outra rodada de tratamento com DNase I de 15-30 min (para remoção completa do DNA contaminante das amostras de RNA) antes da reação de transcrição reversa. Adicionar 1 μL da solução de DNase I, 4 μL de tampão (fornecido com o kit) e água livre de nuclease a um volume final de 40 μL a 212 ng de RNA. Incubar durante 30 min à temperatura ambiente e inactivar o calor a 70 °C durante 10 min.
- Sintetizar cDNA, usando 150 ng de RNA usando um kit de acordo com as instruções do fabricante. Incluir controles sem a transcriptase reversa, para garantir a ausência de contaminação do DNA viral da amostra. O cDNA é armazenado a -20 °C.
NOTA: A quantidade de RNA de entrada para transcrição reversa ideal pode variar dependendo do tipo de tecido e da eficiência de transdução esperada no respectivo tecido.
- Análise da biblioteca viral de AAV (in-vivo) por NGS
- Para alcançar alta profundidade de sequenciamento a baixo custo, execute NGS via sequenciamento Illumina, conforme descrito anteriormente (seção 1.9). Amplifique a sequência de código de barras e, em seguida, ligue os adaptadores de sequenciamento ao amplicon.
- Devido ao comprimento de leitura curto e à ligação dos adaptadores de sequenciamento em ambos os lados do amplicon, ao projetar, verifique se o amplicon é suficientemente pequeno para garantir a presença da sequência de código de barras dentro da leitura NGS. Para o sequenciamento dos códigos de barras dentro dos genomas virais e dos transcritos virais, o amplicon de PCR é projetado para ter 113 pb de comprimento (ver Figura 8).
- Amplificar a região do código de barras com os primers BC-seq forward e BC-seq reverse. Preparar a seguinte reação de PCR: 0,5 μL de DNA polimerase de alta fidelidade, 10 μL de tampão 5x, 0,25 μL de cada primer de 100 μM (BC-seq fw/BC-seq rv) e 1 μl de 10 mM dNTPs. Use 25 ng do cDNA ou DNA/reação como molde e ajuste o volume final para 50 μL com água livre de nucleases.
- Prepare a mistura mestra de PCR sob uma capa de PCR limpa para evitar contaminação. Use as seguintes condições de ciclo: 30 s a 98 °C, seguido por 40 ciclos a 98 °C por 10 s e 72 °C por 20 s, e um passo final de 5 minutos a 72 °C.
- Inclua controles de PCR para confirmar a ausência de DNA contaminante no master-mix de PCR. Para as amostras de cDNA, incluir os controles sem transcriptase reversa. Finalmente, inclua um exemplo com a biblioteca de entrada AAV. Essas informações serão usadas para gerar o arquivo Normalization_Variant.txt usado na análise.
- Verificar o tamanho do fragmento de PCR de cada amostra por eletroforese em gel antes da purificação da PCR. Este último é obtido usando esferas magnéticas comercialmente disponíveis ou sistemas de purificação de DNA baseados em colunas (ver Tabela de Materiais).
- Prepare a biblioteca NGS usando o sistema de bibliotecas para amostras de baixa complexidade de acordo com as instruções do fabricante, conforme descrito anteriormente na seção 1.9.
- Determinar a concentração de ADN através do dsDNA HS Kit e analisar a qualidade da biblioteca conforme descrito anteriormente (secção 1.9.6), seguido de agrupamento. Quantifique a biblioteca agrupada em um fluorômetro e avalie a qualidade em um Bioanalisador.
- Execute o sequenciamento NGS conforme discutido na seção 1.9.7.
- Quantificar por qPCR o número de cópias do transgene (genomas virais) e o gene housekeeping para avaliar a distribuição da biblioteca agrupada entre tecidos ou órgãos no DNA.
- Estabeleça uma reação de qPCR de 30 μL da seguinte forma, para determinar o número de cópias de EYFP (transgene) e GAPDH (gliceraldeído 3-fosfato desidrogenase, gene housekeeping):
- Preparar uma mistura de primer/sonda de 60x para EYFP (1,5 μM YFP_fw, 1,5 μM YFP_rv e 0,6 μM YFP_probe; ver Tabela de Materiais). Use a mistura de primer/sonda GAPDH (consulte Tabela de Materiais) para determinar o número de cópia do gene da governanta. Configure a reação no gelo.
- Prepare uma mistura mestre de PCR (15 μL, consulte Tabela de Materiais) e adicione 60x mistura de primer/sonda (0,5 μL) para todas as amostras e padrões (para calcular os números de cópia para os padrões, use o seguinte link: http://cels.uri.edu/gsc/cndna.html). Configure a reação no gelo.
- Transfira 15,5 μL da mistura mestre para uma placa de 96 poços e adicione 14,5 μL de amostra (75 ng de concentração total de DNA) ou padrão para o respectivo poço. Sele a placa de 96 poços com papel alumínio, vórtice e gire brevemente.
- Transfira 10 μL de cada amostra para uma placa de 384 poços em duplicatas. Sele a placa com papel alumínio e gire a 800 x g por 5 min a 4 °C.
- Incubar a mistura de reacção num termociclador utilizando uma temperatura inicial de 50 °C durante 2 minutos, seguida de uma fase de activação inicial de 10 minutos a 95 °C. Realizar 40 ciclos de desnaturação a 95 °C por 15 s e recozimento/extensão a 60 °C por 1 min24.
- Para obter o número de genomas diploides (dg), use o número de cópias do GAPDH e divida por dois. Em seguida, pegue o valor do número de cópias do EYFP e divida pelo número de dg, resultando em genomas vetoriais por genoma diploide (vg/dg). Use esse valor para gerar o arquivo Normalization_Organ.txt para a análise de bioinformática.
- Realizar a análise dos dados de sequenciamento de NGS como Weinmann et al.24, usando código personalizado em Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). O fluxo de trabalho compreende a detecção de sequências de código de barras guiadas por sequências de flanco, seu comprimento e localização (Script#1_BarcodeDetection.py), bem como a análise do enriquecimento e distribuição do código de barras sobre o conjunto de tecidos (Script#2_BarcodeAnalysis.py).
- Detecte códigos de barras e atribua-os a variantes AAV. Coloque os dados de sequenciamento como arquivos fastq arquivados em um diretório (por exemplo, "Data_to_analyze"). O arquivo de dados de sequenciamento para a biblioteca de entrada é incluído nesse diretório e usado apenas para calcular as proporções do capsid na biblioteca de entrada.
- Antes de executar o script, crie dois arquivos de texto delimitados por tabulações: o arquivo de variantes de capsid (veja o arquivo de exemplo "Variants.txt") com as sequências de código de barras atribuídas a nomes de variantes de capsid AAV e o arquivo de contaminação (consulte "Contaminações.txt") com sequências de código de barras provenientes de possível contaminação (outros códigos de barras disponíveis no laboratório, contribuindo para a contaminação).
- Finalmente, edite o arquivo de configuração "Barcode_Script.conf" para incluir as seguintes informações: caminho para a pasta com dados de sequenciamento (por exemplo, "Data_to_analyze"), sequência de regiões flanqueadoras dos códigos de barras, sua posição e tamanho da janela para detecção de código de barras (semelhante à 1.9.8.5, consulte a Figura 8).
- Use o seguinte comando para chamar a detecção de código de barras com caminhos fornecidos para Script#1_BarcodeDetection.py e arquivos de configuração:
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
Observação : A saída da execução de Script#1_BarcodeDetection.py é arquivos de texto com contagens de leitura por variante capsid, bem como o número total de leituras recuperadas dos dados brutos. - Avalie a distribuição de capsídeos AAV com código de barras entre tecidos ou órgãos, executando Script#2_BarcodeAnalysis.py juntamente com os seguintes arquivos txt:
- No arquivo "Zuordnung.txt", atribua o nome a cada arquivo txt obtido da execução de detecção de código de barras a um nome de tecido/órgão: nomes de arquivos txt na primeira coluna e nomes de tecidos/órgãos correspondentes em atribuição delimitada por tabulação.
Observação : para um exemplo, verifique a pasta "Exemplo" (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). É importante notar que o nome do tecido/órgão pode incluir caracteres que definem a medição de cDNA ou gDNA e o número de réplicas biológicas (M1, M2, etc.). - Crie um arquivo de texto "órgãos.txt" com a lista de nomes para órgãos de destino ON e OFF, que correspondem aos nomes fornecidos no arquivo de atribuição "Zuordnung.txt" (consulte a pasta "Exemplo": https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- Crie arquivos de texto delimitados por tabulações "Normalization_Organ.txt" e "Normalization_Variant.txt" com valores normalizados para todas as variantes do capsídeo e todos os órgãos/tecidos. Na primeira coluna do ficheiro "Normalization_Organ.txt", escreva os nomes indicados para cada órgão (como no ficheiro de atribuição "Zuordnung.txt") e na segunda coluna os valores de normalização para os tecidos correspondentes, gerados na secção 3.6.11.
- Preencha a primeira coluna do arquivo "Normalization_Variant.txt" com a lista de nomes de capsid e a segunda coluna com os valores normalizados das contagens de leitura para cada capsid na biblioteca em pool (a normalização pode ser calculada com base no arquivo de saída txt para a biblioteca de entrada resultante do primeiro script).
- Edite o arquivo de configuração especificando os caminhos completos para todos os arquivos adicionais mencionados acima. Execute Script#2_BarcodeAnalysis.py como:
>python3 /Script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
NOTA: O script de análise de código de barras produz vários arquivos: arquivos de texto com valores de concentração relativa (RC) de distribuição de capsídeo dentro de diferentes tecidos com base em várias etapas de normalização descritas anteriormente e o arquivo de planilha que combina dados de arquivo de texto em dados de matriz mesclados. Este último pode ser usado para análise e visualização de clusters. - Visualize os dados e realize a análise de agrupamento dos dados da matriz para distinguir as propriedades do capsídeo e avaliar suas semelhanças com base nos perfis de CR entre os tecidos. Use o script adicional PCA_heatmap_plot. R colocado no repositório:
>Rscript --baunilha ~/PCA. R ~/concentração relativa.xls
Observação : O script usa arquivos de concentração relativa.xls como entrada e gera dois gráficos de mapa de calor de cluster hierárquico e análise de componente principal (PCA). - Para modificar gráficos (eixos do mapa de calor, componentes principais da ACP) ou parâmetros png (cor, tamanho, rotulagem), abra o script R e siga as instruções fornecidas nas seções comentadas.
- No arquivo "Zuordnung.txt", atribua o nome a cada arquivo txt obtido da execução de detecção de código de barras a um nome de tecido/órgão: nomes de arquivos txt na primeira coluna e nomes de tecidos/órgãos correspondentes em atribuição delimitada por tabulação.
Access restricted. Please log in or start a trial to view this content.
Resultados
Geração de uma biblioteca de exibição de peptídeos AAV2. Como um primeiro passo para a seleção de AAVs projetados, a geração de uma biblioteca de plasmídeos é descrita. A inserção peptídica é produzida usando primers degenerados. Reduzir a combinação de códons naqueles de 64 para 20 tem as vantagens de eliminar os códons de parada e facilitar a análise de NGS, reduzindo a diversidade de bibliotecas no DNA, mas não o nível de proteínas. A inserção do oligonucleotídeo é adquirid...
Access restricted. Please log in or start a trial to view this content.
Discussão
Neste protocolo, são descritas as etapas necessárias para a engenharia do capsídeo AAV de exibição de peptídeos e para a triagem de bibliotecas de AAV com código de barras, bem como para a análise bioinformática da composição da biblioteca e do desempenho do capsídeo. Este protocolo se concentra nas etapas que facilitam a análise bioinformática desses tipos de bibliotecas, porque a maioria dos laboratórios de virologia tem atraso em habilidades de programação para corresponder à sua proficiência em té...
Access restricted. Please log in or start a trial to view this content.
Divulgações
D.G. é co-fundador da AaviGen GmbH. D.G. e K.R. são inventores de um pedido de patente pendente relacionado à geração de variantes do capsídeo AAV que evade a imunidade. Os demais autores não têm nada a revelar.
Agradecimentos
D.G. agradece muito o apoio da Fundação Alemã de Pesquisa (DFG) através dos Centros de Pesquisa Colaborativa DFG SFB1129 (Projektnummer 240245660) e TRR179 (Projektnummer 272983813), bem como do Centro Alemão de Pesquisa de Infecção (DZIF, BMBF; TTU-HIV 04.819).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
Referências
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621(2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756(2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075(2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041(2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432(2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819(2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899(2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272(2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200(2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825(2021).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados