Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Estudio multimodal de la remodelación cardiovascular murina: ecografía cuatridimensional y espectrometría de masas
* Estos autores han contribuido por igual
En este artículo
Resumen
En este artículo, describimos un protocolo para utilizar la ecografía cuatridimensional in vivo y la espectrometría de masas ex vivo para evaluar las alteraciones biomecánicas y biomoleculares en el sistema cardiovascular murino. Esta técnica se aplica para analizar el remodelado cardíaco en el infarto de miocardio inducido quirúrgicamente y los cambios vasculares en animales envejecidos.
Resumen
Las enfermedades cardiovasculares (ECV) son la principal causa de muerte en los Estados Unidos. El daño en el sistema cardiovascular puede deberse a la exposición ambiental, traumatismos, toxicidad de medicamentos o muchos otros factores. Como resultado, el tejido y la vasculatura cardíaca sufren cambios estructurales y muestran una función disminuida. El daño y la remodelación resultante se pueden detectar y cuantificar con imágenes de ultrasonido (US) a nivel de órgano e imágenes de espectrometría de masas (MSI) a nivel molecular. Este manuscrito describe una metodología innovadora para el estudio de la fisiopatología cardíaca murina, acoplando imágenes y análisis de ultrasonido cuatridimensional (4D) in vivo con desorción/ionización láser asistida por matriz ( MADLI) MSI del corazón. La ecografía 4D puede proporcionar mediciones volumétricas dinámicas, como el desplazamiento radial, la deformación de la superficie y la deformación longitudinal a lo largo de todo un ciclo cardíaco. En la vasculatura, la MSI y la ecografía se utilizan para evaluar la composición de la pared de los vasos, la hemodinámica y la dinámica de la pared de los vasos. La metodología se puede adaptar para estudiar una miríada de enfermedades CV mediante el ajuste de métricas funcionales de interés y/o la variación del protocolo MALDI MSI para dirigirse a moléculas específicas. MALDI MSI se puede utilizar para estudiar lípidos, metabolitos pequeños, péptidos y glicanos. Este protocolo describe el uso de MALDI MSI para el análisis lipidómico no dirigido y el uso de imágenes de ultrasonido para la hemodinámica y biomecánica cardiovascular.
Introducción
Las enfermedades cardiovasculares (ECV) son una de las principales causas de mortalidad en todo el mundo1. La prevención y el tratamiento de las ECV requieren una comprensión profunda de las adaptaciones moleculares a las fuerzas biomecánicas y los cambios resultantes en las propiedades mecánicas. A lo largo de todo el sistema cardiovascular, las fuerzas biomecánicas juegan un papel importante en la función y estructura del tejido2. Las propiedades mecánicas del tejido cardiovascular (CV) están influenciadas por estas fuerzas, convirtiéndolos en indicadores de salud y enfermedad 3,4,5,6. Para prevenir, diagnosticar y tratar las ECV, es crucial desarrollar métodos para comprender y observar los procesos de inicio y progresión de la enfermedad. Las imágenes biomédicas han sido clave en la generación de conocimientos fisiológicos y mecanicistas, y constantemente se desarrollan nuevas tecnologías de imagen y técnicas de análisis. Este protocolo demuestra una metodología para combinar dos técnicas de imagen y análisis cardiovascular para validar el potencial de estas modalidades de imagen en la enfermedad cardíaca isquémica y el envejecimiento vascular.
Los investigadores en el campo de la biomecánica a menudo abordan el estudio de la biomecánica a través de una combinación de métodos in vivo, ex vivo e in silico . La investigación previa en biomecánica molecular se ha centrado principalmente en las proteínas7 (en particular las proteínas de la matriz extracelular colágeno y elastina debido a su impacto en las propiedades biomecánicas), y el trabajo para combinar la biomecánica de imágenes in vivo con estudios moleculares se ha limitado a la histología y la inmunohistoquímica. Aunque estos enfoques pueden producir muchos indicadores moleculares y han producido mecanismos propuestos para la remodelación de la MEC y las células, generalmente se limitan a las tinciones o anticuerpos actualmente disponibles, respectivamente. En este campo de investigación faltan grandes clases de moléculas, por ejemplo, los lípidos. Si bien estas clases moleculares pueden o no estar involucradas mecánicamente, es importante comprender las adaptaciones moleculares resultantes porque estas moléculas podrían ser objetivos potenciales tanto para marcadores de diagnóstico como para terapias. Se pueden aplicar técnicas de química analítica, como la cromatografía líquida-espectrometría de masas (LC-MS); sin embargo, la orientación espacial de las moléculas en estas técnicas se pierde. Con las imágenes de espectrometría de masas (MSI), la distribución espacial de las moléculas permanece intacta y se pueden obtener imágenes de múltiples tipos de analitos (clases de moléculas) con secciones en serie. MSI es una poderosa herramienta analítica para investigar las distribuciones espaciales de casi todos los tipos de moléculas en el tejido biológico, incluidos metabolitos, lípidos, glicanos, péptidosy fármacos de pequeño peso molecular. La desorción/ionización láser asistida por matriz (MALDI) MSI es un tipo de MSI que es muy adecuado para el análisis basado en el descubrimiento de pesos moleculares en el rango de 50-8000 Da. MALDI-MSI es una técnica de ionización que aplica una matriz de absorción de energía láser a la muestra para ayudar en la ionización de los analitos de interés. Este enfoque evita limitarse a un objetivo molecular y puede utilizar herramientas bioinformáticas para determinar qué moléculas tienen un impacto en las propiedades biomecánicas y la remodelación.
La ecografía cuatridimensional (4DUS) es un método in vivo no invasivo útil para la caracterización temporal y espacial del corazón. 4DUS utiliza una serie de bucles de cine de alta velocidad de fotogramas de diferentes planos, compilándolos en un conjunto de datos 3D que incluye información temporal. Esto permite la visualización y cuantificación directas de los complejos cambios de forma en 3D de las cámaras cardíacas a lo largo del ciclo cardíaco sin depender de suposiciones geométricas como se requiere para la ecocardiografía 2D tradicional. 4DUS permite calcular métricas funcionales in vivo a partir de la compleja forma y movimiento del corazón 9,10, y MALDI MSI permite el estudio espacial de moléculas biológicas dentro del tejido cardíaco ex vivo11. Para comprender completamente las alteraciones en el corazón con ECV, es necesario investigar tanto los mecanismos mecánicos como los moleculares. Por lo tanto, se propone una metodología combinada para estudiar la fisiopatología del corazón murino, acoplando la imagen y el análisis de 4DUS con MALDI MSI de lípidos en el corazón. Esta metodología se demuestra en un modelo murino de infarto de miocardio.
La biomecánica vascular también desempeña un papel fundamental en la regulación de la función cardiovascular2. El endurecimiento vascular, que se asocia con el envejecimiento, es un factor de riesgo para la ECV12. Los cambios biomecánicos y hemodinámicos en los vasos se pueden visualizar mediante ecografía. Las composiciones moleculares de las paredes de los vasos son componentes importantes de la biomecánica y también son exquisitamente sensibles a las fuerzas hemodinámicas. Por ejemplo, el esfuerzo cortante oscilatorio de la pared se ha implicado en el desarrollo de placa aterosclerótica3. Posteriormente se presentarán los datos preliminares de mecánica de vasos y hemodinámica en animales envejecidos.
El equipo está interesado en la relación entre la biomecánica y la composición molecular en varios estados de enfermedad. Las imágenes preclínicas de ultrasonido y MSI se utilizan para determinar la distribución espacial de los cambios moleculares en un tejido y los cambios biomecánicos asociados que ocurren durante la progresión de la enfermedad. Este informe describe estas metodologías en detalle y presenta datos preliminares sobre el corazón y la vasculatura de la cabeza y el cuello.
Protocolo
Los experimentos con animales descritos se llevan a cabo con la aprobación del Comité Institucional de Cuidado y Uso de Animales de la Universidad de Tennessee, Knoxville.
1. Imágenes de ultrasonido13
- Montaje y preparación de los animales
- Antes de comenzar el experimento, compruebe el nivel de anestesia con isoflurano y vuelva a llenarlo si es necesario. Verifique los niveles del tanque de oxígeno antes de encender el gas. Pesa el carbón y anótalo en el recipiente.
- Configure el espacio de imágenes colocando los suministros cerca de la plataforma de imágenes. Los suministros incluyen cinta precortada, crema depilatoria, gasa, hisopos de algodón, agua esterilizada en autoclave, lubricante corneal, gel lubricante, sonda de temperatura rectal, cables de electrodos, gel de ultrasonido, gel de electrodos (si corresponde) y lámpara de calor (Figura 1A).
- Encienda la máquina de ultrasonido e inicialice el motor (el sistema también es capaz de recopilar datos de imágenes 4DSE). Configure un nuevo experimento en el sistema de ultrasonido: seleccione Nuevo estudio o Nueva serie y complete la información del mouse. Encienda la tableta para el control fisiológico.
- Verifique la llave de paso para asegurarse de que el flujo esté abierto a la caja de inducción y cerrado al cono de la nariz. Llene previamente la caja de inducción de anestesia encendiendo el oxígeno a 1 L/min en el vaporizador y girando el dial a 3%-5%.
- Retire el ratón de la jaula de la carcasa.
- Coloque el ratón en la caja de inducción hasta que el animal esté completamente anestesiado con isoflurano entre 3% y 5%.
- Cambie la llave de paso para cambiar el flujo al cono de la nariz en la placa de imágenes de ultrasonido. Gire el dial de isoflurano entre 1% y 2%.
- Retire el ratón de la caja de inducción y pese al animal. Anota el peso del animal.
- Coloque el ratón en posición supina sobre la placa de imagen.
- Aplique lubricante corneal en los ojos del animal.
- Si hay pelo, retírelo del área de imágenes con una crema depilatoria disponible en el mercado e hisopos de algodón. Limpie la crema de la piel con agua en esponjas para eliminar el exceso de crema depilatoria de la piel y prevenir quemaduras.
- Insertar los cables de electrocardiografía (ECG) por vía subcutánea a ambos lados del tórax y de la extremidad posterior, dependiendo de la posición del animal.
- Una vez insertados, sostenga los cables de ECG en su lugar con cinta adhesiva. Si no se utilizan derivaciones de ECG (es decir, no se insertan derivaciones de ECG), controle el ECG a través de la placa de imágenes para animales.
- Para la conducción eléctrica de la piel a la placa, coloque el gel conductor en la placa y mantenga las extremidades del animal en posición.
- Sostenga las extremidades del animal en su lugar con cinta adhesiva o una banda elástica colocada alrededor de la placa y sobre las extremidades (Figura 1B).
- Use gel lubricante para insertar la sonda de temperatura rectal y asegure la sonda con cinta adhesiva.
- Verifique las señales de ECG, respiración y temperatura en la tableta. Asegúrese de que la frecuencia cardíaca sea de ~350-600 lpm, la respiración de ~50-100 respiraciones/min y la temperatura rectal entre 35-37 °C. Ajuste los niveles de isoflurano según sea necesario.
- Si es necesario, encienda una lámpara de calor (por ejemplo, una bombilla calefactora infrarroja de 250 W)14 y ajuste la altura para mantener la temperatura central (Figura 1C).
- Coloque la lámpara de calor a no menos de 12 pulgadas del animal. Para los estudios iniciales para determinar la distancia óptima entre el bulbo y el animal, ajuste la altura y registre la temperatura de la piel con un termómetro infrarrojo, que no debe exceder los 42 °C15.
- Ecografía cardíaca 4D
- Coloque el transductor en el soporte en una posición semibloqueada.
- Oriente el punto en relieve en el transductor con el punto azul en la pantalla. La convención es colocar el punto hacia el lado derecho del animal.
- Gire el transductor para que quede orientado a lo largo del plano sagital del ratón con una muesca elevada apuntando en la dirección caudal.
- Utilice la palanca situada en la base de la placa de ultrasonido para inclinar al animal. Utilice la palanca en la base del transductor para ajustar el ángulo del transductor.
- Aplique una cantidad generosa de gel de ultrasonido en la superficie ventral de la cavidad torácica para el acoplamiento acústico entre la superficie de la piel y el transductor. Asegúrese de que no quede pelo antes del acoplamiento.
- Baje el transductor para que entre en contacto con el gel de ultrasonido.
- Realice microajustes con las perillas X/Y en la base de la placa, o mueva toda la placa para realizar ajustes grandes.
- Asegúrese de que una vista paraesternal de eje largo incluya el ápice, el tracto de salida del ventrículo izquierdo y la aorta alineados horizontalmente en la pantalla para obtener una imagen más precisa del eje corto (Figura 2A, B).
- Seleccione Nombre de imagen en la esquina inferior para guardar la imagen en la serie actual.
- Gire el transductor 90° en el sentido de las agujas del reloj para cambiar a una vista de eje corto paraesternal (Figura 2C).
NOTA: Para obtener la mejor calidad de imagen 4D, los usuarios deben intentar alinear el motor paso a paso a lo largo del eje largo paraesternal del ventrículo, que no siempre es directamente paralelo al esternón. El corazón/VI a menudo se encuentra en un ángulo hacia abajo a la izquierda. - Asegúrese de que el ventrículo izquierdo esté visible en el lado derecho de la pantalla y que el ventrículo derecho esté en el lado izquierdo de la pantalla (Figura 2D).
- Si el ratón tiene un ritmo respiratorio estable, pulse el cuadro de electrocardiograma en la esquina superior izquierda de la imagen para activar la activación respiratoria de la imagen 4D. Si no es estable, complete este paso durante el posprocesamiento.
- Seleccione el cubo en la esquina superior izquierda de la pantalla para configurar una imagen 4D.
- Restablezca el transductor antes de ajustar las posiciones de inicio y parada del transductor.
- Ajuste la posición de inicio justo debajo del ápice y la posición de parada al arco aórtico.
- Establece el Tamaño de paso en 0,08-0,13 mm y la Velocidad de fotogramas en 200-300 Hz.
NOTA: Los tamaños de paso más pequeños pueden proporcionar una mejor resolución de imagen para el análisis, pero aumentarán el tiempo de adquisición. Los tamaños de paso más grandes son aceptables si los datos se recopilan con una alineación ventricular izquierda casi perfecta y se necesita una reorientación mínima o nula durante el análisis. Considere la posibilidad de aumentar la velocidad de fotogramas cuando trabaje con animales con frecuencias cardíacas más altas (>450 lpm). - Asegúrese de que los signos vitales y la señal del electrocardiograma estén estables de forma constante (>350 lpm y respiración por encima de 50 resp/min) antes de realizar la exploración.
- Una vez que se completen el escaneo y el procesamiento, active Guardar datos EKV/4D para posprocesamiento y Respiration Gating .
- Seleccione Nombre de imagen en la parte inferior derecha e incluya el ID del mouse en el nombre.
- Para comprobar la calidad de la imagen, pulse Más controles y seleccione Cargar en 4D.
- Revise cada plano del corazón, asegurándose de que el centro del corazón no se mueva durante todo el ciclo cardíaco. El movimiento del centro indica variabilidad en el electrocardiograma y/o la activación respiratoria y, por lo tanto, complica el proceso de análisis. Vuelva a ajustar los monitores y repita el escaneo si es necesario.
- Análisis de ultrasonido cardíaco 4D
- Vaya al navegador del estudio. Exporte todas las imágenes 2D del modo B a Vevo Lab y exporte los datos 4D como otro tipo de archivo, seleccionando el formato de datos ".raw" a un disco duro.
NOTA: La caja de herramientas de la interfaz gráfica de usuario 4D utilizada en este trabajo no está actualmente disponible públicamente. Para solicitar acceso e instrucciones detalladas, comuníquese con el Dr. Craig Goergen, de la Universidad de Purdue. - Realice un análisis alternativo de datos 4D utilizando el software VevoLab. Para utilizar este marco, exporte los datos 4D de la máquina a los archivos de Vevo Lab.
- Vaya al navegador del estudio. Exporte todas las imágenes 2D del modo B a Vevo Lab y exporte los datos 4D como otro tipo de archivo, seleccionando el formato de datos ".raw" a un disco duro.
- Imágenes vasculares
- Para obtener imágenes en modo B de las arterias carótidas, coloque el transductor de ultrasonido paralelo a las arterias carótidas, cerca de la línea media.
- Una vez que la arteria carótida es visible, mueva el transductor hacia arriba para encontrar la bifurcación carotídea.
- Cuando se encuentre una imagen clara de la bifurcación carotídea, ajuste la configuración de ganancia a 35 dB y capture la imagen en modo B.
- Inclinar el transductor hacia la izquierda o hacia la derecha puede mejorar la imagen. Ajuste el ángulo del transductor o la etapa para que el recipiente de interés no esté directamente paralelo al transductor. Esto permite que el ángulo Doppler permanezca por debajo de 60°. De lo contrario, puede ser necesario un desplazamiento de +/- 15°.
- Cambie al modo Doppler de onda pulsada para obtener las mediciones de velocidad.
- Coloque el volumen de la muestra en el centro del recipiente. Ajuste el ángulo del cursor para que quede paralelo a la arteria carótida. A continuación, pulsa el botón de reproducción para iniciar las mediciones de velocidad y guardarlas para registrarlas.
- Para obtener la imagen en modo B de las venas yugulares, coloque el transductor de ultrasonido paralelo a las venas yugulares. Las venas yugulares son anteriores y laterales a las arterias carótidas.
- Una vez localizada la vena yugular, mueva el transductor al lugar donde se fusionan la yugular interna y externa. Cuando se encuentre una imagen nítida, ajuste la configuración de ganancia a 35 dB y capture la imagen en modo B.
- Cambie al modo Doppler de onda pulsada para obtener las mediciones de velocidad.
- Coloque el volumen de la muestra en el centro del recipiente. Ajuste el ángulo del cursor para que quede paralelo a la vena yugular. A continuación, pulsa el botón de reproducción para iniciar las mediciones de velocidad y guardarlas para registrarlas.
- Análisis ecográfico vascular
- Después de recopilar las imágenes del modo B y las mediciones Doppler de onda pulsada, mida los diámetros y las velocidades con el software.
- Una vez que las imágenes estén cargadas en el software, seleccione paquete vascular.
- Seleccione cada lado del recipiente para obtener el diámetro completo.
- En el caso de la arteria carótida, se realizan mediciones de diámetro en sístole y diástole utilizando la opción de diámetro del paquete vascular.
- Realice mediciones de velocidad utilizando la opción de velocidad en el paquete vascular.
- Para la arteria carótida, seleccione el pico más alto para la medición de la velocidad en sístole y el pico más bajo para la medición de la velocidad en la diástole.
- Utilice los valores de los diámetros para calcular el componente circunferencial del tensor de deformación de Green Lagrange utilizando la siguiente ecuación,
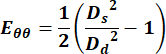 (1)
(1)
donde Ds representa el diámetro durante la sístole y Dd representa el diámetro durante la diástole. - Para la vena yugular, seleccione cualquier punto a lo largo del ciclo para obtener la medición de la velocidad.
2. Eutanasia y recolección de tejidos
- Prepare botes de papel de aluminio para la congelación instantánea de papel tisú (Figura 3).
- Al final de un estudio, se debe sacrificar al animal mediante una sobredosis de isoflurano al 5% de concentración y neumotórax bilateral o luxación cervical (solo estudio cardíaco).
- Usando pinzas para cubrir la piel, corte la piel con unas tijeras sobre el cuello para la vasculatura o justo debajo del esternón para el corazón.
- Cortar las capas de piel y músculo para exponer la vasculatura o cortar a través del hueso para exponer el corazón.
- Usando una disección roma con hisopos de algodón, aísle el corazón o la vasculatura de los tejidos circundantes, incluida la grasa. Asegúrese de separar el vaso carotídeo del nervio. Extirpar el corazón y los vasos sanguíneos con instrumentos quirúrgicos.
NOTA: Las suturas se pueden usar para eliminar la vasculatura suturando proximal y distal antes de extirpar los vasos. - Coloque el pañuelo de papel en un bote de papel de aluminio preetiquetado y coloque el bote en nitrógeno líquido (Figura 3).
- Almacene el tejido a -80 °C hasta el momento de la criosección. Transporta el tejido con hielo seco para mantener la temperatura.
3. Imágenes de espectrometría de masas
- Crioseccionamiento y montaje a temperatura óptima de corte (OCT) en portaobjetos de vidrio
- Ajuste la temperatura del criostato a -25 °C e inserte la cuchilla.
- Prepare un mandril de metal aplicando OCT y dejando que se congele en el criostato.
- Fije la base del corazón al mandril de metal preparado usando OCT (Figura 4A). No permita que la OCT toque la región de interés de la muestra porque la OCT contaminará los espectros de masas (contaminación por polietilenglicol [PEG]) y deprimirá las señales lipídicas de interés.
NOTA: Opción de realizar el montaje con agua para evitar la contaminación de la muestra. Consulte el protocolo adicional a continuación para obtener instrucciones. - Sobre la base de la importancia de la alineación espacial en esta metodología, utilice la representación digital a escala del corazón proporcionada por la GUI de MATLAB para guiar el seccionamiento (Figura 4B).
- Cuente cada revolución (10 μm) mientras secciona y anote la profundidad en milímetros de cada corte que se montará en la corredera.
NOTA: Es necesario realizar un seguimiento de ubicaciones específicas en el corazón o la vasculatura para obtener una coincidencia precisa con la representación digital de los datos de ultrasonido proporcionados por MATLAB. - Descongele las secciones de tejido de montaje (10 μm de grosor) en portaobjetos de microscopio.
NOTA: El tipo de portaobjetos dependerá del espectrómetro de masas utilizado. Cada portaobjetos incluye una sección de cada región del corazón. Se requiere un mínimo de tres portaobjetos (modo de iones positivos, modo de iones negativos, extra). - Para n = 1/grupo, seccione el corazón a través del eje largo para visualizar los cambios moleculares desde el ápice hasta la base.
- Para la vasculatura, incruste el tejido en gelatina y congele rápidamente antes de seccionar11.
- Guarde los portaobjetos en sobres de diapositivas a -80 °C hasta que se realicen experimentos de imágenes de espectrometría de masas.
- Si realiza cromatografía líquida-espectrometría de masas (LC-MS) para identificar y cuantificar aún más lípidos o metabolitos, seccione secciones de tejido en serie de ~ 60-100 μm y recójalas en tubos de 0,5-2 mL antes de congelar.
- Tejido de criosección y montaje en agua en portaobjetos de vidrio
- Llene un vaso de precipitados con agua de cromatografía líquida de alta resolución (HPLC) y déjelo a un lado con jeringas de 5 ml y 1 mL.
- Ajuste la temperatura del criostato a -25 °C e inserte la cuchilla.
- Coloque un par de pinzas en el criostato para que se enfríe antes del montaje.
- Extraiga 5 ml de agua HPLC en una jeringa y colóquela en el criostato.
- Justo antes de que la jeringa esté completamente congelada, vacíe el agua en el mandril de metal y deje que se congele por completo.
- Extraiga 1 ml de agua de HPLC y coloque la jeringa en el criostato.
- Después de aproximadamente 30-60 s, coloque un pequeño punto de agua parcialmente solidificada en el centro del mandril. Agarra inmediatamente el corazón con pinzas y colócalo en el punto antes de que se congele por completo. Sostenga el corazón en su lugar hasta que el agua circundante se haya congelado por completo.
NOTA: El corazón puede estar montado por el ápice o la base, dependiendo de la región de interés. Para este estudio, la base se montará con el vértice hacia afuera. - Realice el resto de los pasos de este método como se describe en el paso 3.1.
- Aplicación matricial
- Retire el portaobjetos del congelador y colóquelo en un desecador hasta que el portaobjetos esté seco.
- Encienda el pulverizador HTX M3+, abra la aplicación HTX en la computadora portátil y seleccione el método en el centro de la pantalla. Las concentraciones de matriz estándar se pueden encontrar en los métodos almacenados (los métodos en el lado izquierdo de la pantalla > OI_usermethods > matriz).
- Para este trabajo, ajuste la temperatura de la boquilla a 75 °C, el caudal a 100 μL/min y la presión a 10 psi.
- Escriba el nombre de la muestra, la polaridad, la matriz, el disolvente y la concentración en el cuaderno del laboratorio. Calcule la cantidad de matriz necesaria por concentración (por ejemplo, 5 mL a 10 mg/mL = 50 mg de matriz).
- Hacer solvente (por ejemplo, 70% de MeOH).
- Para este trabajo, prepare 40 mg/mL de solución de matriz de ácido 2,5-dihidroxibenzoico (DHB) para modo positivo y 10 mg/mL de solución de matriz de 9-aminoacridina (9AA) para modo negativo. Hacer ambas matrices con un disolvente MeOH al 70%. Para otras matrices y parámetros de pulverización comunes, siga los pasos 3.3.2.
- Pesar la matriz y añadirla a un tubo cónico de 15 mL. Asegúrese de que la cantidad esté cerca de la cantidad requerida calculada en el paso 3.3.3, pero no tiene que ser exacta. Calcule la cantidad de disolvente en función de la masa medida.
- Añada disolvente al tubo cónico con una pipeta.
NOTA: El volumen se basa en la masa medida en el paso 3.3.5. - Sonicar la mezcla durante 10 min. Mientras se sonica la matriz, retire la corredera del desecador.
- Abra la bandeja del rociador, coloque el portaobjetos en la esquina inferior izquierda y pegue los bordes con cinta adhesiva.
NOTA: Doble un extremo de la cinta para facilitar su extracción. - Seleccione la región de pulverización de la muestra . Cierre la bandeja.
- Use una jeringa y un filtro y vierta la matriz en la jeringa, luego filtre la matriz a través de la jeringa en el vial con la tapa negra en el lado izquierdo del rociador.
- Vuelva a colocar el vial en su lugar en el rociador e inserte el tubo de la línea D en el vial.
NOTA: Asegúrese de que el tubo no toque el fondo del vial y que esté completamente sumergido en el líquido. - Abra el gas inerte y asegúrese de que el manómetro del rociador marque 10 psi. Presione inicio. Una vez que el pulverizador esté a la temperatura deseada, seleccione el inicio intermitente.
- Una vez finalizada la pulverización, abra la bandeja, retire la muestra y colóquela en el portaobjetos MALDI o de nuevo en el desecador (Figura 4C).
- Escanee el soporte de diapositivas MALDI y deslícelo en el escáner, o tome una foto con un teléfono. Guarde la imagen en una unidad flash para usarla con MSI.
- Seleccione el lavado adecuado o la siguiente pulverización en el pulverizador. Mueva la línea D del vial de matriz al vaso de precipitados de residuos si se está lavando.
- Rocíe metanol en la bandeja y limpie para limpiar. Apague el nitrógeno.
- MALDI MSI
- En la computadora, navegue hasta Synapt, verifique que la polaridad se cambie al modo necesario y cambie si no es así.
- Cargue la diapositiva en el instrumento en la fuente MALDI y haga clic en el botón de carga en la computadora usando el software.
- Mueva la imagen (paso 1.2.17) a la carpeta Imágenes de la carpeta Proyecto .
- Vaya a HD Imaging y abra el proyecto anterior (uno que utilice la misma matriz).
- En un portátil de laboratorio, registre el rango de masa, atrape y transfiera la energía de colisión, la energía del láser, el modo neg/pos.
- En Imágenes HD, navegue hasta el icono de página blanca. A continuación, haga clic en la flecha desplegable y seleccione nueva placa.
- Busque la imagen en la carpeta Proyecto > Carpeta Imágenes y ábrala.
- Cambie el tipo de placa a MALDI Standard y defina los bordes de la placa (elija las 4 esquinas que se muestran a continuación con signos más rojos).
NOTA: El cuadro superior izquierdo es para navegar a cada esquina. El cuadro superior derecho es para seleccionar las esquinas. El cuadro inferior es solo para visualizar toda la diapositiva. - Navegue a la pestaña de patrón pequeño en la esquina superior izquierda y haga clic en lápiz u otras opciones de rectángulo o círculo para delinear el tejido. Delinee el tejido con los clics izquierdo y el clic derecho para terminar.
- El tamaño de píxel predeterminado es de 50 μm. Cámbielo si es necesario.
- Haga clic en Guardar como y vaya a Project Imaging AcqFolder y etiquételo como nombre de diapositiva (formato AAAAMMDD). A continuación, guarde.
- Haga clic en el botón Mass Lynx en la parte superior central para exportar a Mass Lynx.
- Vaya al software Mass Lynx.
- Haga clic en Archivo > abrir la carpeta del proyecto. Edite el nombre del archivo y el nombre del texto de la diapositiva (formato AAAAMMDD).
- Haga clic con el botón derecho en el archivo MS, busque y elija la diapositiva actual.
- Haga clic derecho nuevamente en el archivo MS y haga clic en Editar. Haga doble clic izquierdo en el cuadro azul.
- Vaya a las condiciones de escaneo. Busque la configuración de origen y cámbiela a definida por el usuario. A continuación, navegue hasta la carpeta de proyecto correcta > carpeta adquirida > diapositiva actual > ACEPTAR.
- Guarde haciendo clic en Guardar como e incluya el nombre de la diapositiva.
- Calibrar el instrumento utilizando un compuesto de referencia (por ejemplo, fósforo rojo)
- Vaya a Synapt (Calibrar). Haga clic en el botón de reproducción para comenzar a adquirir datos. Etiqueta como YYYYMMDD_cal1 (por ejemplo: 20230614_cal1).
- Asegúrese de que tenga el mismo rango de masa 50 - 2000. La energía del láser puede ser de aproximadamente 175.
- Pulsa el botón de reproducción láser de fuego y haz clic en el botón siguiente . Déjalo correr a unos 15 o 20. Haga clic en parada roja.
- Navega hasta W Console.
- Cree la calibración y haga clic en iniciar. En el editor de perfiles de calibración, haga clic en archivo > nuevo. Tipo YYYYMMDD_cal1.
- Edite el rango de masa al rango de masa deseado para el proyecto (por ejemplo, 50-2000).
- Elija la calibración manual. Elija el modo de resolución y haga clic en editar. Seleccione el fósforo rojo como compuesto de referencia.
- Haga clic en el archivo de datos sin procesar y desplácese hasta el final (debería ser lo que se acaba de adquirir).
- Vaya al historial en la parte inferior, seleccione Acc masa > está bien > está bien.
- Siga haciendo clic en siguiente y OK hasta que se vea el botón de inicio y presione iniciar.
- Navegue hasta la verificación verde y observe qué picos no se encontraron.
- Vaya a Mass Lynx (Inicie la ejecución real de las muestras experimentales).
- Pulsa el botón de reproducción y comprueba que el número de muestras es el correcto
- Vaya a Imágenes HD.
- Compruebe que el tamaño de píxel sigue siendo el que se estableció originalmente
- Vaya a Synapt para comprobar la polaridad.
- Haga clic en el botón de la cámara para asegurarse de que se esté moviendo y disparando el láser.
- Verifique y asegúrese de que esté recopilando datos. El espectro de masas adquirido debe mostrarse en la página Synapt.
- Análisis de EM
- Utilice el software HD Imaging y Mass Lynx para el análisis.
- Procese los datos de imágenes mediante la pestaña Proceso de HD Imaging.
- Visualice imágenes de iones moleculares, también llamadas imágenes MS, en Imágenes HD en la pestaña Análisis . Un mapa de calor de una imagen de iones moleculares muestra la intensidad de ese ion m/z en una región (Figura 5). Utilice las superposiciones de imágenes de MS para mostrar la distribución espacial de las moléculas (Figura 6 y Figura 7).
- Normalice los datos mediante TIC o la normalización adecuada para la creación de imágenes.
- Identifique los picos m/z que se correlacionan espacialmente con las regiones de interés. Ejecute una correlación espacial (R > 0,65) en HD Imaging para identificar picos colocalizados.
- Búsqueda de la identificación lipídica putativa de estos picos con LipidMaps16 y METASPACE17.
- Confirme las anotaciones de lípidos con los datos posteriores de LC-MS. Utilice Metaboanalyst para el análisis estadístico de datos LC-MS.
Resultados
Los protocolos de imagen descritos anteriormente se utilizaron para dos estudios preliminares: el remodelado del infarto de miocardio (IM) y el envejecimiento vascular. Para los experimentos cardíacos, se realizó una cirugía permanente de ligadura de arterias coronarias con el fin de inducir infarto agudo de miocardio18,19. La ecografía 4D y MALDI MSI se realizaron de forma progresiva en el mismo tejido, revelando cambios fis...
Discusión
Las imágenes de ultrasonido pueden depender del operador, pero el uso de puntos de referencia anatómicos y una capacitación adecuada pueden limitar el sesgo del usuario. La ecografía 2D es especialmente susceptible a la variabilidad entre usuarios porque las vistas dependen del ángulo, mientras que la 4DHE es menos susceptible ya que la adquisición abarca todo el volumen y es independiente del ángulo. También se determinó que la reproducibilidad de la imagen es más fácil de lo...
Divulgaciones
Craig J. Goergen es un consultor remunerado de FUJIFILM VisualSonics.
Agradecimientos
Allison Jones cuenta con el apoyo de la beca de posgrado del Departamento de Ingeniería Mecánica, Aeroespacial y Biomédica de la Universidad de Tennessee. La investigación reportada en esta publicación (Conner Earl) fue apoyada por el Instituto Nacional del Corazón, los Pulmones y la Sangre de los Institutos Nacionales de Salud F30HL162452. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente los puntos de vista oficiales de los Institutos Nacionales de Salud.
Materiales
| Name | Company | Catalog Number | Comments |
| 2,5-dihydroxybenzoic acid (DHB) | Supelco, >99.0% (HPLC) | 85707-10MG-F | DHB matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sigma/85707?cm_sp=Insite-_-wimsShippingEmailRecs_wims EmailAPI_wimsGruCrossEntropy-_-wimsEmailAPI10-3 |
| 9-aminoacridine (9AA) | Supelco, ≥99.5% (HPLC) | 92817-1G | 9-Aminoacridine matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sial/92817?srsltid=AfmBOooiQjQ4pWv_XxITkU 4Lkm0UnHXKekGS_ dFl7V40V9QLWoPpNLoc |
| Aquasonic Ultrasound Gel | Parker Laboratories | Parker 01-02 | Ultrasound Gel; https://www.parkerlabs.com/products/aquasonic-100-ultrasound-transmission-gel/ |
| Benchtop Dewar Flasks | ThermoScientific | 4150-2000 | Container for liquid nitrogen; https://www.thermofisher.com/order/catalog/product/4150-4000?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sQVc2f-NxN04Nb5Mv F6TZ7GLHWWDEeqDYmEvtKJSQ YHDeVgZ9qylvYaAs27EALw_wcB &source=google_shopping&ISO_ CODE=us&LANG_CODE=en&ef_id =Cj0KCQjwpvK4BhDUARIsADHt9 sQVc2f-NxN04Nb5MvF6TZ7GLHWWDE eqDYmEvtKJSQYHDeVgZ9qylvYa As27EALw_wcB:G:s&s_kwcid=AL!3652 !3!716188292869!!!g!2366243726129 !!21787513085!171591181194&ev_chn =shop&cid=0se_gaw_30092024_ PBYTXL&source=google_shopping &ISO_CODE=us&LANG_CODE= en&gad_source=1 |
| Cryostat | Leica Biosystems | CM Series | https://www.leicabiosystems.com/us/histology-equipment/cryostats/ |
| Dessicator | VWR | 89054-052 | https://us.vwr.com/store/product/9104882/desiccator-plastic-ace-glass-incorporated |
| Epredia MX35 Premier Disposable Low-profile Microtome Blades | Fisher Scientific | 3052835 | Cryostat blade; https://www.fishersci.com/shop/products/mx35-premier-disposable-low-profile-microtome-blades/3052835 |
| Falcon 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | Conical Tubes; https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/1495953A?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sSBcy5n-lhShligJUOX5KKVGn0bt87 8AB2_muOD2PPTue1phpZgeqwa AqgiEALw_wcB&ef_id=Cj0KCQjw pvK4BhDUARIsADHt9sSBcy5n-lhS hligJUOX5KKVGn0bt878AB2_muO D2PPTue1phpZgeqwaAqgiEALw_ wcB:G:s&ppc_id=PLA_goog_20861 45674_81843405034_1495953A__ 386247001345_165426395473886 37329&ev_chn=shop&s_kwcid=AL!4428!3 !386247001345!!!g!856907751004!& gad_source=1 |
| Flex-Tubes Microcentrifuge Tubes | Eppendorf | EP022364120 | Centrifuge tubes; https://www.sigmaaldrich.com/US/en/product/sigma/ep022364120?utm_source=google%2Cgoogle&utm _medium=organicshopping%2Ccpc&utm _campaign=21043330280&utm_ content=&gclid=Cj0KCQjwpv K4BhDUARIsADHt9sTHRD35k CHPtfI2A41axodnMVr6a1eBKk zM4bSUYQAyfEKo3UgTAEQa Ap7wEALw_wcB |
| Gas Nitrogen | Airgas | N/A | |
| Glass microscope slides | Electron Microscopy Sciences | 71873-02 | https://www.emsdiasum.com/positive-charge-microscope-slides |
| Liquid Nitrogen | Airgas | N/A | |
| Mass Spectrometer | Waters | Synapt G2-Si | https://www.waters.com/waters/en_US/SYNAPT-G2-Si-Mass-Spectrometry/nav.htm?locale=en_mkcid=1000251Foodety%3C/a%3E&cid=134740653&bcid= 134528734 |
| Matrix Sprayer | HTX Technologies | M3+ | https://www.htximaging.com/htx-m3-sprayer |
| Methanol (HPLC), >99.9% | Fisher Chemical | A4524 | Methanol; https://www.fishersci.com/shop/products/methanol-hplc-fisher-chemical-9/A4524?crossRef=A4524#?keyword=A4524 |
| Preclinical Ultrasound System | FUJIFILM VisualSonics | Vevo 3100 | https://www.visualsonics.com/product/imaging-systems/vevo-3100; Vevo F2 has replaced the Vevo 3100 in production. System includes isoflurane vaporizer and induction box. |
| Reynolds Wrap | N/A | N/A | Aluminum foil |
| Signagel Electrode Gel | Parker Laboratories | Parker 15-60 | Electrode Conducting Gel; https://www.parkerlabs.com/products/signagel-electrode-gel/ |
| Sterile Lubricating Jelly | Medline | MDS032273Z | Lubricating Gel; https://www.medline.com/ce/product/Sterile-Lubricating-Jelly/Lubricating-Jelly/Z05-PF03664?sku=MDPMDS032273H |
| Surgical instruments: scissors, forceps/tweezers, sutures | Fine Science Tools | 11252-00, 11050-10, 14016-14, 14084-08, 15000-08 | info@finescience.com |
| Surgical Sponges 200 Pack –Gauze Pads Non sterile -First Aid Wound Care Dressing Sponge –Woven Medical Nonstick, Non Adherent Mesh Scrubbing | Medpride | B08RZGQ5GW | Gauze; https://www.amazon.com/Medpride-Surgical-Sponges-200-Pack/dp/B08RZGQ5GW/ref=asc_df_B08RZGQ5GW/?tag=hyprod-20&linkCode=df0&h vadid=693270340506&hvpos= &hvnetw=g&hvrand=960915122 2290977669&hvpone=&hvptwo= &hvqmt=&hvdev=c&hvdvcmdl=& hvlocint=&hvlocphy=9192978&hv targid=pla-1245491514869&psc= 1&mcid=33f4d647c88630c79116 888d565a63b0 |
| Tissue-Plus O.C.T. Compound | Fisher Scientific | 23-730-571 | OCT; https://www.fishersci.com/shop/products/tissue-plus-o-c-t-compound-2/23730571 |
| Wood Handled Cotton Swabs and Applicators | Fisherbrand | 22-363-160 | Cotton swab; https://www.fishersci.com/shop/products/wood-handled-cotton-swabs-applicators-8/p-7146852 |
Referencias
- Tsao, C. W., et al. Heart disease and stroke statistics-2022 update: A report from the American Heart Association. Circulation. 145 (8), E153-E639 (2022).
- Kassab, G. S. Biomechanics of the cardiovascular system: The aorta as an illustratory example. J R Soc Interface. 3 (11), 719-740 (2006).
- Moore, J. E., Xu, C., Glagov, S., Zarins, C. K., Ku, D. N. Fluid wall shear stress measurements in a model of the human abdominal aorta: Oscillatory behavior and relationship to atherosclerosis. Atherosclerosis. 110 (2), 225-240 (1994).
- Nerem, R. M. Vascular fluid mechanics, the arterial wall, and atherosclerosis. J Biomech Eng. 114 (3), 274 (1992).
- Cecchi, E., et al. Role of hemodynamic shear stress in cardiovascular disease. Atherosclerosis. 214 (2), 249-256 (2011).
- Good, B. The influence of blood composition and loading conditions on the behavior of embolus analogs. J Mech Behav Biomed Mater. 140, 105738 (2023).
- Bao, G., et al. Molecular biomechanics: The molecular basis of how forces regulate cellular function NIH public access. Mol Cell Biomech. 3 (2), 91-105 (2010).
- Schwamborn, K., Caprioli, R. M. Molecular imaging by mass spectrometry-looking beyond classical histology. Nat Rev Cancer. 10 (9), 639-646 (2010).
- Soepriatna, A. H., Kevin Yeh, A., Clifford, A. D., Bezci, S. E., O'Connell, G. D., Goergen, C. J. Three-dimensional myocardial strain correlates with murine left ventricular remodelling severity post-infarction. J R Soc Interface. 16 (160), 20190570 (2019).
- Damen, F. W., et al. High-frequency 4-dimensional ultrasound (4DUS): A reliable method for assessing murine cardiac function. Tomography. 3 (4), 180-187 (2017).
- McDonald, R., Poulos, D., Gutzwiller, L., Sheth, R. A., Good, B., Crouch, A. C. A MALDI mass spectrometry imaging sample preparation method for venous thrombosis with initial lipid characterization of lab made and murine clots. J Am Soc Mass Spectrom. 34 (9), 1879-1889 (2023).
- Benetos, A., et al. Influence of age, risk factors, and cardiovascular and renal disease on arterial stiffness: Clinical applications. Am J Hypertens. 15 (12), 1101-1108 (2002).
- Pistner, A., Belmonte, S., Coulthard, T., Blaxall, B. C. Murine echocardiography and ultrasound imaging. J Vis Exp. (42), v2100 (2010).
- Bellantuono, I., et al. A toolbox for the longitudinal assessment of healthspan in aging mice. Nat Protoc. 15 (2), 540-574 (2020).
- Henriques, F. C., Moritz, A. R. Studies of thermal injury: I. The conduction of heat to and through skin and the temperatures attained therein. A theoretical and an experimental investigation. Am J Pathol. 23 (4), 530 (1947).
- Conroy, M. J., et al. LIPID MAPS: Update to databases and tools for the lipidomics community. Nucleic Acids Res. 52 (D1), D1677-D1682 (2024).
- Palmer, A., et al. FDR-controlled metabolite annotation for high-resolution imaging mass spectrometry. Nat Methods. 14 (1), 57-60 (2016).
- Michael, L. H., et al. Myocardial ischemia and reperfusion: A murine model. Am J Physiol. 269 (6 Pt 2), H2147-H2154 (1995).
- Clark, J. E., Flavell, R. A., Faircloth, M. E., Davis, R. J., Heads, R. J., Marber, M. S. Post-infarction remodeling is independent of mitogen-activated protein kinase kinase 3 (MKK3). Cardiovasc Res. 75 (3), 523-530 (2007).
- Hattori, Y., Konno, H., Abe, M., Miyoshi, H., Goto, T., Makabe, H. Synthesis, determination of the absolute configuration of tonkinelin, and inhibitory action with bovine heart mitochondrial complex I. Bioorg Med Chem. 15 (8), 3026-3031 (2007).
- Golam Mostofa, M., et al. CLIP and cohibin separate rDNA from nucleolar proteins destined for degradation by nucleophagy. J Cell Biol. 217 (8), 2675-2690 (2018).
- Chan, J. N. Y., Poon, B. P. K., Salvi, J., Olsen, J. B., Emili, A., Mekhail, K. Perinuclear cohibin complexes maintain replicative life span via roles at distinct silent chromatin domains. Dev Cell. 20 (6), 867-879 (2011).
- Dann, M. M., et al. Quantification of murine myocardial infarct size using 2-D and 4-D high-frequency ultrasound. Am J Physiol Heart Circ Physiol. 322 (3), H359-H372 (2022).
- Kaya, I., Sämfors, S., Levin, M., Borén, J., Fletcher, J. S. Multimodal MALDI imaging mass spectrometry reveals spatially correlated lipid and protein changes in mouse heart with acute myocardial infarction. J Am Soc Mass Spectrom. 31 (10), 2133-2142 (2020).
- Tressler, C., et al. Factorial design to optimize matrix spraying parameters for MALDI mass spectrometry imaging. J Am Soc Mass Spectrom. 32 (12), 2728 (2021).
- Zhang, L., et al. Vascular lipidomics analysis reveals increased levels of phosphocholine and lysophosphocholine in atherosclerotic mice. Nutr Metab. 20 (1), 1-16 (2023).
- Kohno, M., et al. Induction by lysophosphatidylcholine, a major phospholipid component of atherogenic lipoproteins, of human coronary artery smooth muscle cell migration. Circulation. 98 (4), 353-359 (1998).
- Crouch, A. C., Batra, A., Greve, J. M. Hemodynamic response to thermal stress varies with sex and age: A murine MRI study. Int J Hyperthermia. 39 (1), 69-80 (2022).
- Crouch, A. C., Scheven, U. M., Greve, J. M. Cross-sectional areas of deep/core veins are smaller at lower core body temperatures. Physiol Rep. 6 (16), e13839 (2018).
- Crouch, A. C., Castle, P. E., Fitzgerald, L., Scheven, U. M., Greve, J. M. Assessing structural and functional response of murine vasculature to acute β-adrenergic stimulation in vivo during hypothermic and hyperthermic conditions. Int J Hyperthermia. 36 (1), 1136-1145 (2019).
- Crouch, A. C., Manders, A. B., Cao, A. A., Scheven, U. M., Greve, J. M. Cross-sectional area of the murine aorta linearly increases with increasing core body temperature. Int J Hyperthermia. 34 (7), 1121-1133 (2018).
- Clift, C. L., Mehta, A., Drake, R. R., Angel, P. M. Multiplexed imaging mass spectrometry of histological staining, N-glycan and extracellular matrix from one tissue section: A tool for fibrosis research. Methods Mol Biol. 2350, 313-329 (2021).
- Salehi, H. S., et al. Coregistered photoacoustic and ultrasound imaging and classification of ovarian cancer: Ex vivo and in vivo studies. J Biomed Opt. 21 (4), 046006 (2016).
- Verbeeck, N., et al. Connecting imaging mass spectrometry and magnetic resonance imaging-based anatomical atlases for automated anatomical interpretation and differential analysis. Biochim Biophys Acta. 1865 (7), 967-977 (2017).
- Yang, E. Y., et al. Real-time co-registration using novel ultrasound technology: Ex vivo validation and in vivo applications. J Am Soc Echocardiogr. 24 (7), 720 (2011).
- Maris, L., et al. Method for co-registration of high-resolution specimen PET-CT with histopathology to improve insight into radiotracer distributions. EJNMMI Phys. 11 (1), 1-20 (2024).
- Lin, B. J., et al. MSIr: Automatic registration service for mass spectrometry imaging and histology. Anal Chem. 95 (6), 3317-3324 (2023).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados