Method Article
Imágenes y cuantificación de la morfología mitocondrial en C. elegans durante el envejecimiento
En este artículo
Resumen
Este protocolo proporciona un enfoque estandarizado para obtener imágenes de la morfología mitocondrial en múltiples tejidos de C. elegans durante el envejecimiento.
Resumen
Las mitocondrias, orgánulos celulares importantes que se encuentran en la mayoría de las células eucariotas, son sitios importantes de producción de energía a través de la respiración aeróbica. Más allá de este papel bien conocido como la "fuente de energía celular", las mitocondrias también están involucradas en muchos otros procesos celulares esenciales, incluida la regulación del metabolismo celular, la proliferación, la señalización inmune y la señalización hormonal. El deterioro de la función mitocondrial durante el envejecimiento o bajo estrés mitocondrial a menudo se caracteriza por cambios distintivos en la morfología y el volumen mitocondrial. El nematodo C. elegans es un modelo ideal para estudiar estos cambios debido a su cuerpo transparente y su corta vida útil, que facilitan la microscopía en vivo durante toda su vida. Sin embargo, incluso dentro del campo de C. elegans , se dispone de numerosas construcciones y métodos transgénicos para la obtención de imágenes mitocondriales, cada uno con sus propias limitaciones. Aquí, se presentan construcciones de GFP localizadas en matriz de una sola copia como un método robusto y confiable para obtener imágenes de la morfología mitocondrial en C. elegans. Este estudio se centra específicamente en los factores controlables experimentalmente para minimizar los errores y reducir la variabilidad entre las réplicas y entre los estudios al realizar imágenes mitocondriales durante el proceso de envejecimiento. Además, se recomienda mitoMAPR como un método robusto para cuantificar los cambios en la morfología mitocondrial en todos los tipos de tejidos durante el envejecimiento.
Introducción
Las mitocondrias son orgánulos celulares encerrados por una membrana de fosfolípidos dobles y desempeñan un papel importante en el mantenimiento de la bioenergética celular como sitio clave para el metabolismo yla producción de energía. Un gran número de mitocondrias generan continuamente energía en forma de ATP para satisfacer las demandas celulares2. Además de estas importantes funciones, las mitocondrias también participan en procesos celulares complejos como la transducción de señales, la autofagia, la inmunidad innata, el ciclo celular y las vías de muerte celular 2,3. Las mitocondrias exhiben diversas morfologías, que van desde pequeños orgánulos individuales hasta extensas redes tubulares interconectadas, dependiendo de los diferentes requisitos energéticos y la salud mitocondrial4.
Una característica clave de las mitocondrias es su naturaleza dinámica, mediante la cual pueden alternar entre estas diversas formas a través de una secuencia estrechamente coordinada y continua de eventos de fisión y fusión4. El equilibrio preciso entre estos procesos opuestos regula la morfología mitocondrial, el número, el tamaño y la posición dentro del citoplasma5. Además, estos procesos de fusión y fisión son importantes para mantener el control de calidad de las mitocondrias5. Por ejemplo, la fisión mitocondrial es un componente esencial para la eliminación de mitocondrias dañadas a través de la mitofagia y la eliminación selectiva de mitocondrias a través de la autofagia2. La dinámica mitocondrial también juega un papel importante en la división celular, el desarrollo, la resistencia a diversos factores estresantes y el mantenimiento del metabolismo celular6.
La alteración de la dinámica mitocondrial está implicada en numerosas enfermedades, incluidos los trastornos neurodegenerativos, las enfermedades metabólicas, las enfermedades cardiovasculares y los cánceres4. Además, el envejecimiento se asocia con una remodelación dramática de las mitocondrias, en gran parte debido a cambios en estas dinámicas de fusión-fisión7. Por lo tanto, visualizar y monitorear los cambios en la morfología mitocondrial bajo diversas condiciones de estrés o enfermedad y durante todo el proceso de envejecimiento ofrece información valiosa para comprender la función celular, el mecanismo de la enfermedad y las posibles estrategias terapéuticas.
Al igual que muchas vías moleculares, la dinámica y la función mitocondrial también están muy conservadas en todos los eucariotas, incluidos los organismos modelo como el nematodo Caenorhabditis elegans. Al igual que en las células humanas, la fusión mitocondrial en C. elegans se logra mediante la función de las proteínas guanina trifosfatasa (GTPasa) relacionadas con la dinamina, incluidas FZO-1 (ortólogo de Mfn1/2 de mamíferos) y EAT-3 (ortólogo de Opa1 de mamíferos) que controlan la fusión de la membrana mitocondrial externa (OMM) y la membrana mitocondrial interna (IMM), respectivamente8. La fisión mitocondrial está regulada por la proteína relacionada con la dinamina (DRP-1, ortólogo de Drp1 humano), que promueve la fisión mitocondrial mediante la formación de complejos en forma de anillo alrededor de la membrana externa mitocondrial que constriñen y finalmente separan las membranas mitocondriales9. La fusión mitocondrial desempeña un papel fundamental en el control de calidad mitocondrial al permitir la mezcla de los contenidos mitocondriales, incluidos el ADN mitocondrial, las proteínas y los lípidos, lo que permite la complementación de las mitocondrias parcialmente dañadas por los contenidos de las mitocondrias sanas9. Por otro lado, la fisión mitocondrial permite que las mitocondrias se dividan, creando nuevas mitocondrias y facilitando su distribución no solo dentro del citoplasma sino también a las células hijas durante la división celular, asegurando una adecuada herencia y función mitocondrial9. Este evento también es esencial para segregar segmentos mitocondriales dañados o disfuncionales, que luego pueden ser objeto de degradación a través de la mitofagia9.
C. elegans ha sido considerado durante mucho tiempo como uno de los sistemas de modelos genéticos más poderosos debido al genoma completo y la disponibilidad de una variedad de herramientas genéticas, incluido CRISPR/Cas9 que facilita las modificaciones genéticas10, numerosos métodos para la sobreexpresión de genes y un método bacteriano basado en alimentos para la interferencia de ARN (ARNi)11. Además, su anatomía transparente permite la obtención de imágenes microscópicas en organismos vivos12. Finalmente, su vida relativamente corta, bajos costos de mantenimiento y facilidad para producir un gran número de animales de la misma edad los convierten en un excelente sistema modelo para la biología del envejecimiento. Estos beneficios, mezclados con la conocida conservación de las principales vías reguladoras mitocondriales, hacen de C. elegans un sistema modelo muy atractivo para estudiar la dinámica mitocondrial durante el envejecimiento.
Los marcadores fluorescentes se utilizan ampliamente en la investigación biológica para visualizar y estudiar los componentes celulares, incluidas las mitocondrias. Existen colorantes específicos permeables a las células, como el MitoTracker y el éster etílico de tetrametilrodamina (TMRE), que se utilizan habitualmente14. El primero se utiliza para teñir la matriz mitocondrial15 cargada negativamente en general, mientras que el segundo se utiliza para evaluar el potencial relativo de la membrana mitocondrial a través de su ion trifenilfosfonio16 cargado positivamente. Si bien se benefician de no requerir transgénicos, la cutícula gruesa de los gusanos que cambia en estructura y permeabilidad durante el envejecimiento y la variabilidad de la infiltración de tintes a través de diferentes tejidos hacen que los enfoques basados en tintes sean desafiantes en C. elegans17. Además, la evaluación de la morfología mitocondrial se ve confundida por los posibles efectos fuera del objetivo de los colorantes, como la agregación de colorantes17. En cambio, los métodos genéticos para expresar fluoróforos localizados en mitocondrias se utilizan comúnmente en el modelo de gusano.
En este caso, este estudio se centra en destacar las cepas que expresan una GFP localizada en la matriz mitocondrial (en adelante, MLS::GFP) bajo promotores específicos del tipo celular. Es importante destacar que estas líneas transgénicas se elaboraron utilizando el método MosSCI para garantizar la expresión de una sola copia del reportero en un locus genómico conocido, lo que evita problemas con otras cepas disponibles. Por ejemplo, la alta expresión de algunas proteínas dirigidas a las mitocondrias conduce a una variabilidad en los niveles de expresión debido a la integración de matrices de plásmidos extracromosómicos en un locus aleatorio con un número de copia desconocido18. Además, se ha demostrado previamente que inducen daño a las mitocondrias, ya que es una alta carga para las células localizar adecuadamente las proteínas mitocondriales sobreexpresadas19. Por lo tanto, la expresión génica precisa, estable y baja y la facilidad de cruzamiento con loci conocidos hacen que estos transgénicos MosSCI sean el método preferido. Usando estas cepas, este estudio estandariza los métodos para obtener imágenes de las mitocondrias en el músculo, el intestino y la hipodermis de C. elegans. Además, destaca las importantes aplicaciones técnicas y los métodos de resolución de problemas que es importante tener en cuenta y garantizar un análisis experimental reproducible de las mitocondrias durante el envejecimiento en C. elegans.
Protocolo
Los detalles sobre los reactivos y el equipo utilizado en este estudio se enumeran en la Tabla de Materiales.
1. Crecimiento y mantenimiento de C. elegans
- Preparación de placas de medios de crecimiento de nematodos (NGM)
- Para el cultivo de C. elegans, utilice placas de agar estándar al 2% con NGM que contengan 1 mM de CaCl2, 12,93 μM (5 μg/mL) de colesterol, 25 mM de KPO4 (pH 6,0), 1 mM de MgSO4, 2,5 mM (0,25% p/v) de peptona y 51,3 mM de NaCl. Véase el método detallado de vertido de placas NGM descrito en Castro Torres et al.20.
NOTA: Para experimentos de ARN de interferencia (ARNi), agregue 1 mL de IPTG de 1 M y 1 mL de 100 mg/mL de carbenicilina para placas de agar NGM de 1 L. - Después de que las placas de NGM se hayan solidificado, cultivar un cultivo de OP50 en caldo de lisogenia (LB) durante 24-48 h a temperatura ambiente o cultivar un cultivo de HT115 portador de un plásmido vectorial vacío pL4440 en LB con antibióticos (ampicilina/carbenicilina 100 μg/mL + tetraciclina 5 μg/mL) con agitación a 37 °C durante 12-16 h.
NOTA: Este plásmido está disponible en fuentes comerciales (ver Tabla de Materiales). - Siembre 200 μL de cultivo OP50 o HT115 en placas de 60 mm, o 1 mL en placas de 100 mm.
- Seque las placas hasta que ya no estén mojadas y guárdelas en recipientes sellados a 4 °C durante un máximo de 2 meses.
- Opcional: Agregue 100 μL de 10 mg/mL de 5-fluoro-2'-desoxiuridina (FUDR) directamente sobre las placas de agar NGM que se siembran con bacterias para esterilizar químicamente los gusanos L4/adultos y prevenir el desarrollo de gusanos más jóvenes.
NOTA: Las cepas desarrolladas y utilizadas en este estudio son:
(1) RHS191 - uthSi17[myo3p::MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] I
(2) RHS192 - uthSi83[col19p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)]I
(3) RHS193 - uthSi80[vha-6p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] IV
- Para el cultivo de C. elegans, utilice placas de agar estándar al 2% con NGM que contengan 1 mM de CaCl2, 12,93 μM (5 μg/mL) de colesterol, 25 mM de KPO4 (pH 6,0), 1 mM de MgSO4, 2,5 mM (0,25% p/v) de peptona y 51,3 mM de NaCl. Véase el método detallado de vertido de placas NGM descrito en Castro Torres et al.20.
- Sincronizar C. elegans por blanqueo
- Para un ensayo a gran escala que requiera un gran número de nematodos, corte una placa de agar NGM de 60 mm llena de animales en trozos pequeños y córtelos en placas de 100 mm que se siembran con bacterias para su expansión. Espere de 2 a 3 días para que las placas de 100 mm estén llenas de gusanos cuando crezcan a 20 °C después de cortarlas. Para recomendaciones más detalladas sobre animales en expansión, consultar Castro Torres et al.20.
- Para recoger los gusanos para la sincronización, vierta 5-10 mL de solución M9 (22 mM de KH2PO4 monobásico, 42,3 mM de Na2HPO4, 85,6 mM de NaCl, 1 mM de MgSO4) en placas de agar NGM de 100 mm con gusanos, y agite suavemente la solución M9 para aflojar los gusanos del césped bacteriano.
- Extraiga los gusanos con una pipeta serológica y transfiéralos a tubos cónicos de 15 ml.
NOTA: Se recomiendan pipetas serológicas de vidrio, ya que las pipetas serológicas de plástico tienden a hacer que los gusanos se adhieran a la pared interna de las pipetas de plástico. - Centrifugar durante 30 s a 1.100 x g a temperatura ambiente para granular los gusanos y aspirar el sobrenadante mediante una bomba de vacío.
- Prepare 5 mL de solución blanqueadora que contenga 1,5 mL de hipoclorito de sodio al 6%, 0,75 mL de 5 M de NaOH o KOH y 2,75 mL de dH2O. Vierta los 5 mL de solución blanqueadora en la pelletza de gusano.
PRECAUCIÓN: Se recomienda usar guantes y una bata de laboratorio en este paso, ya que las soluciones de hipoclorito e hidróxido de sodio son corrosivas. - Incube la mezcla de lombrices + solución blanqueadora durante ~ 4-6 minutos hasta que todos los cuerpos de los animales se disuelvan por completo y solo queden los huevos. Agite la mezcla vigorosamente para ayudar al proceso de disolución.
NOTA: Revise los gusanos cada minuto bajo un microscopio de disección. Acorte o extienda el tiempo de blanqueo en función de cuánto se disuelvan los cuerpos de los gusanos adultos. Dejar los huevos dentro de una solución blanqueadora durante un período prolongado dañará los huevos y afectará la viabilidad de los nematodos. - Centrifugar la mezcla de huevos/solución blanqueadora durante 30 s a 1.100 x g a temperatura ambiente para granular los huevos y, a continuación, aspirar cuidadosamente el sobrenadante con una bomba de vacío, asegurándose de no aspirar el pellet de huevo.
NOTA: Se recomienda realizar lavados rápidos para evitar la exposición prolongada de los huevos a la solución blanqueadora. - Lavar los huevos con 5-10 mL de solución M9 4 veces.
- Vuelva a suspender los huevos en un pequeño volumen de solución M9 y colóquelos en placas de NGM sembradas con bacterias pipeteando hasta 50 μL. Calcule una estimación aproximada del número de huevos pipeteando 3-5 μL de la mezcla de huevo en un plato y contando el número de huevos en este volumen. Agregue M9 para diluir la mezcla de huevo a un número contable de huevos en este volumen.
NOTA: Se recomienda colocar menos de 100 lombrices por placa, ya que las lombrices pueden morir de hambre antes del día 1 de la edad adulta cuando hay demasiados animales en placas. Para recomendaciones más detalladas sobre el número de animales, véase Castro Torres et al.20. - Alternativamente, L1 detiene los nematodos para una sincronización de edad más estricta. Para la detención de L1, agregue el volumen adecuado de solución M9 a la peleta de huevo en un tubo cónico de 15 mL hasta un volumen de 10-12 mL. Deje que los gusanos giren en un rotador durante 16-24 h a 20 °C. Centrifugar (como en el paso 1.2.7) y aspirar el sobrenadante. Calcule la concentración de animales L1 y póngalos en placas NGM como se describe para los huevos en el paso 1.2.9.
- Envejezca la población de C. elegans a la edad deseada: Transfiera los animales L4/adultos a placas que contengan 5-fluoro-2'-desoxiuridina (FUDR) como se describe en el paso 1.1.6 para prevenir la progenie y eliminar cualquier descendencia ya producida. Para métodos alternativos para envejecer gusanos sin FUDR, consulte Castro Torres et al.20.
NOTA: Se deben considerar algunos factores importantes antes de elegir el blanqueo como método para sincronizar los gusanos en su primera etapa larvaria. Como se destacó anteriormente, la sobreexposición a la lejía puede dañar los embriones, reducir las tasas de eclosión y crear presión de selección para las cepas resistentes a la lejía, lo que podría alterar la genética de la población. Además, si los embriones se dejan sin alimentar durante largos períodos de tiempo, el agotamiento de los nutrientes puede afectar el metabolismo y el desarrollo. Por lo tanto, los métodos de puesta de huevos o NemaSync se pueden utilizar como alternativa a los métodos de blanqueo. El método para el procedimiento de puesta de huevos se describe de manera escalonada en el estudio referido de Castro Torres et al.20. NemaSync es un método comercial más nuevo y más caro, en el que se utiliza un sincronizador manual de gusanos para sincronizar gusanos sin el uso de productos químicos21.
2. Imagen de las mitocondrias en C. elegans
- Preparación de portaobjetos para la obtención de imágenes de C. elegans
- Agregue 10-20 μL de solución M9 en portaobjetos de vidrio y transfiera un número deseado de gusanos adultos a las edades específicas a la solución en el portaobjetos. Se recomiendan aproximadamente 25 gusanos.
NOTA: El uso de métodos tradicionales para paralizar gusanos (por ejemplo, tetramisol o azida de sodio) puede causar fragmentación mitocondrial bajo ciertas condiciones (ver más abajo). En cambio, se recomienda el uso de portaobjetos HistoBond, que tienen una superficie con una carga superficial positiva permanente, reducirá el movimiento de los gusanos sin usar medicamentos que puedan introducir artefactos. Es importante poner una proporción adecuada de gusanos a la solución M9, ya que los volúmenes bajos pueden hacer que los animales se aplasten entre el portaobjetos y el cubreobjetos, mientras que los volúmenes altos harán que los animales se muevan durante la adquisición de la imagen. Las recomendaciones se pueden encontrar en la Tabla 1. - Alternativamente, para el uso de medicamentos que paralizan a los gusanos, consulte los resultados representativos de este manuscrito para conocer un rango de concentración y puntos de tiempo que se pueden usar con una fragmentación limitada de las mitocondrias.
NOTA: Aunque no hubo una fragmentación importante en animales sanos de tipo salvaje, es importante recordar que ciertos mutantes o condiciones pueden experimentar fragmentación mitocondrial con la misma concentración de estos medicamentos, por lo que se requieren algunas pruebas cuando se usan estos medicamentos. - Cubra los gusanos con la solución M9 en el portaobjetos con un cubreobjetos y use esmalte de uñas para sellar los lados y evitar la evaporación de la solución M9.
- Opcional: Para obtener imágenes musculares, haga rodar los gusanos empujando suavemente el cubreobjetos. Esto permite que los músculos de la pared del cuerpo en los lados de los gusanos estén mejor alineados con el plano focal para obtener imágenes de mayor calidad.
- Agregue 10-20 μL de solución M9 en portaobjetos de vidrio y transfiera un número deseado de gusanos adultos a las edades específicas a la solución en el portaobjetos. Se recomiendan aproximadamente 25 gusanos.
- Obtención de imágenes de las mitocondrias con un microscopio compuesto
- Utilice un microscopio de campo amplio estándar para mitocondrias musculares e hipodérmicas (myo-3p::MLS::GFP y col-19p::MLS::GFP).
NOTA: Se utilizó un generador de imágenes disponible en el mercado equipado con un objetivo Aprocromático Plan de 63x/1.4, filtro GFP (11525314), cámara DFC9000 GT y fuente de luz LED5, y se utilizó software de microscopio compatible. - Optimice la configuración de imágenes para cada microscopio y configuración experimental.
NOTA: Sin embargo, como punto de partida, los parámetros para la obtención de imágenes con el generador de imágenes utilizado en este manuscrito son los siguientes: Las dimensiones son 2048 (x) * 2048 (y) píxeles a un tamaño de paso de 0,50 μm para las mitocondrias hipodérmicas y "System Optimized" calculado automáticamente por el software para las mitocondrias musculares. El tiempo de exposición es de 50 ms para las mitocondrias musculares y de 50 ms para las mitocondrias hipodérmicas. El LED de 475 nm se configuró al 50% de potencia y el filtro de densidad neutra se configuró al 30% para el músculo y al 17% para las mitocondrias hipodérmicas. El rango Z comienza desde donde la señal GFP es visible hasta donde termina. Para minimizar el ruido en una proyección máxima, se puede disminuir el número de planos z utilizados para la proyección máxima. - Para obtener imágenes de las mitocondrias intestinales (vha-6p::MLS::GFP), use un microscopio confocal.
NOTA: Las imágenes se realizaron en un microscopio confocal disponible en el mercado equipado con un objetivo Plan ApoChromat de 63x/1.4, láser de luz blanca (WLL), divisor de haz acusto-óptico, detectores HyD S y se ejecutó en software de microscopio. Como se mencionó anteriormente, se debe optimizar cada configuración experimental individual, pero como punto de partida, los parámetros para la obtención de imágenes con los Stellaris utilizados en este estudio son los siguientes: El WLL se estableció en 85.00 % de potencia máxima y con una línea láser de 485 nm a una intensidad de 3.00 %. El detector HyD S se configuró a 490-590 nm con una ganancia de 25 en la configuración analógica. Escaneamos unidireccionalmente un área de 1024 (x) * 1024 (y) o 82,01 μm (x) * 82,01 μm (y) con 5 (z) píxeles a un tamaño de paso de 0,495 μm (la dimensión z varía según el tamaño de los gusanos o tejidos) con una velocidad de escaneo de 1.000 Hz, zoom de 2,25, promedio de línea de 2 y estenopeico de 1 UA (95,5 μm). El rango Z comienza desde donde la señal GFP es visible hasta donde termina. Para minimizar el ruido en una proyección máxima, se puede disminuir el número de planos z utilizados para la proyección máxima.
- Utilice un microscopio de campo amplio estándar para mitocondrias musculares e hipodérmicas (myo-3p::MLS::GFP y col-19p::MLS::GFP).
- Cuantificación de la morfología mitocondrial
NOTA: Para más detalles, ver Schindelin et al.22 y ver Figura Suplementaria 1A-D.- Descargue e instale el software FIJI (Fiji es solo ImageJ, https://fiji.sc/). A continuación, abra FIJI.
- Para descargar e instalar la macro de MitoMAPR, descargue el código fuente 1 de https://doi.org/10.7554/eLife.49158.033. A continuación, copie todo el código mencionado en "A.). Código IJM para MitoMAPR-1.0". A continuación, abra Fiji, vaya a Plugins > New > Macro y pegue el código copiado anteriormente en la ventana de macros. Vaya a la opción Archivo > Guardar como y guarde la macro para usarla en el futuro.
- Abra una imagen microscópica 3D con pilas Z en FIJI arrastrando y soltando archivos en la barra de herramientas de Fiji o yendo a Archivo > Abrir.
- Para crear una proyección máxima de la imagen, vaya a Pilas de > de imágenes > Proyecto Z, seleccione el rango de cortes con imágenes enfocadas que se proyectarán al máximo y seleccione Intensidad máxima como tipo de proyección.
- Guarde la imagen como TIFF yendo a Archivo > Guardar como > Tiff.
- Recorte regiones de interés (ROI) de imágenes completas utilizando la herramienta "Rectángulo", dibujando un ROI y luego yendo a Imagen > Recortar.
- Guarde la imagen como TIFF yendo a Archivo > Guardar como > Tiff.
- Ejecute la macro de MitoMAPR previamente guardada arrastrando y soltando el archivo de macro en la barra de herramientas de Fiji, luego haga clic en Ejecutar. Una nueva ventana le pedirá que seleccione la carpeta donde se guardó el archivo Tiff en 2.3.6.
- Una nueva ventana le pedirá que seleccione un área; cree un rectángulo en la imagen usando la herramienta rectángulo como se mencionó anteriormente en el paso 2.3.4 y presione Aceptar.
NOTA: Se puede seleccionar toda la imagen si se recortó previamente como se describe en el paso 2.3.6. Guarde la ventana "Datos" con todos los valores relacionados con la morfología mitocondrial como un archivo de Excel yendo a Archivo > Guardar como > Guardar.
- Procesamiento por lotes de muestras mediante macros FIJI
- Cree una macro para especificar la región de las imágenes que se van a analizar. Abra Fiji y vaya a Plugins > New > Macro. Pegue el siguiente código en el cuadro de texto y guarde la macro como se describe en 2.3.2 (Figura complementaria 1E-H).
NOTA: Haga un rectángulo (100, 100, 200, 200); run (Especificar..., ancho = 100, alto = 100, x = 100, y =100 escalado). - Cree una macro para guardar todas las imágenes abiertas como archivos TIFF. Dentro de la ventana de macros, vaya a Archivo > nuevo y pegue el siguiente código en el cuadro de texto. Guarde la macro como se describe en 2.3.2.
NOTA: dir = getDirectory(" Seleccionar una carpeta"); ids=newArray(nImágenes); para (i=0; i - Cree una macro para el lote MitoMAPR. Vaya a https://doi.org/10.7554/eLife.49158.033 código fuente 1 y copie todos los códigos mencionados en B.). Código IJM para MitoMAPR-1.0_Batch". Dentro de la ventana de macros, vaya a Archivo > nuevo, pegue el código del código fuente 1 en el cuadro de texto y guarde la macro como se describe en el paso 2.3.2.
- Cree una macro para el recorte iterativo. Vaya a https://doi.org/10.7554/eLife.49158.033 código fuente 1 y copie todos los códigos mencionados en C.) Código IJM para CropR". Dentro de la ventana de macros, vaya a Archivo > nuevo, pegue el código del código fuente 1 en el cuadro de texto y guarde la macro como se describe en el paso 2.3.2.
- Para comenzar el procesamiento por lotes, primero guarde todas las imágenes que se van a procesar como TIFF abriendo todas las imágenes y ejecutando la macro guardada en el paso 2.4.2. Todas las imágenes se guardarán como TIFF en la carpeta especificada. Cierre todas las imágenes una vez guardadas.
- Si las imágenes guardadas en el paso 2.4.5 son imágenes 3D (imágenes que contienen pilas Z), conviértalas en imágenes 2D convirtiéndolas en proyecciones Z. Para procesar las imágenes en un proyecto Z por lotes, abra la ventana de procesamiento por lotes yendo a Procesar > lote > macro en la ventana principal de FIJI.
- En "Entrada", seleccione la ubicación de la carpeta que contiene todas las imágenes 3D a procesar de la versión 2.4.5. En "Salida", seleccione la ubicación deseada para las imágenes guardadas después del procesamiento. Seleccione el formato de salida como TIFF. Pegue el siguiente código en el cuadro de texto grande de la ventana y presione el botón de proceso : run("Z Project...", "projection=[Max Intensity]").
- Recorte ROI con dimensiones idénticas de todas las imágenes 2D que se van a procesar utilizando la macro de recorte iterativa del paso 2.4.4 en combinación con la macro de región de especificación del paso 2.4.3. Ejecute la macro de recorte iterativa del paso 2.4.4. Aparecerá una ventana solicitando un directorio.
- Seleccione la carpeta que contiene solo imágenes 2D del paso 2.4.5 o las imágenes proyectadas en Z 2D del paso 2.4.6 y pulse el botón Seleccionar . La macro abrirá una de las imágenes de la carpeta seleccionada y aparecerá una ventana con la etiqueta "hacer una selección" con dos botones.
- Ejecute la macro de selección especificada desde el paso 2.4.3. Modifique los valores de anchura y altura de ese cuadro de texto de macro según sea necesario para cambiar la dimensión de selección y pulse Ejecutar para observar la nueva selección. Al cambiar los valores X e Y en la macro, se modificará la posición de la esquina superior izquierda de la selección.
- Una vez que las dimensiones de la selección sean satisfactorias, arrastre el rectángulo al área deseada y luego presione el botón OK en la ventana "hacer una selección". La región recortada se guardará en la carpeta seleccionada en el paso 2.4.7.
- La macro iterará a través de todas las imágenes restantes en la carpeta. Para cada imagen, ejecute la macro desde el paso 2.4.8 sin cambiar los valores de dimensión para asegurarse de que se recortan las mismas dimensiones para todas las imágenes. Para cada imagen, arrastre el rectángulo de selección a la ubicación deseada y presione el botón OK en la ventana "hacer una selección".
- Abra la carpeta que contiene las imágenes recortadas de los pasos 2.4.8-2.4.9 y mueva todas las imágenes recortadas a una nueva carpeta.
- Analice todas las imágenes recortadas ejecutando la macro MitoMAPR por lotes del paso 2.4.3. Una nueva ventana le pedirá que seleccione un directorio. Seleccione la carpeta con todas las imágenes recortadas del paso 2.4.10. Una vez completado, aparecerá una ventana llamada "Datos" con todos los valores relacionados con la morfología mitocondrial. Guarde todos los datos como un archivo de Excel como se describe en el paso 2.3.9.
- Cree una macro para especificar la región de las imágenes que se van a analizar. Abra Fiji y vaya a Plugins > New > Macro. Pegue el siguiente código en el cuadro de texto y guarde la macro como se describe en 2.3.2 (Figura complementaria 1E-H).
Resultados
C. elegans es un gran modelo para la obtención de imágenes mitocondriales debido a su cuerpo transparente, que permite obtener fácilmente imágenes de animales vivos y gusanos enteros sin una preparación excesiva de la muestra. Además, la morfología mitocondrial en diferentes tejidos se puede visualizar fácilmente mediante el uso de promotores específicos de tejido para expresar proteínas fluorescentes dirigidas a las mitocondrias. Aquí, se utilizaron myo-3p (para las mitocondrias musculares), vha-6p (para las mitocondrias intestinales) y col-19p (para las mitocondrias hipodérmicas) para impulsar la expresión de una GFP (secuencia de localización mitocondrial de la proteína ATP-1) dirigida a las mitocondrias. Las mitocondrias de los músculos de la pared corporal de C. elegans exhiben una morfología tubular, alineándose a lo largo de las fibras musculares (miofibrillas) (Figura 1A); las mitocondrias intestinales mostraron estructuras altamente interconectadas, en forma de telaraña, con una alineación menos uniforme (Figura 1B); y la hipodermis tiene mitocondrias tubulares que parecen más redondeadas u ovaladas en comparación con el intestino o el músculo (Figura 1C). La morfología mitocondrial es generalmente consistente a lo largo del gusano en el músculo y el intestino, pero la hipodermis muestra algunas diferencias menores en la interconexión de las mitocondrias entre los extremos proximal y distal del gusano. Por lo tanto, para lograr resultados experimentales reproducibles, se recomienda centrarse en una región específica de sus cuerpos. Aquí, el área entre la faringe y la vulva ~ 100-200 μm por debajo de la faringe se muestra consistentemente.
En el caso de las mitocondrias musculares e hipodérmicas, la obtención de imágenes bajo un microscopio compuesto proporciona una resolución suficiente debido a la planitud de las células y a los bajos niveles de luz desenfocada. Sin embargo, las mitocondrias intestinales son difíciles de observar con un microscopio compuesto, ya que el gran volumen del intestino limita la resolución debido a la gran cantidad de luz desenfocada de otras secciones del intestino (Figura 2). Por lo tanto, se recomienda obtener imágenes de las mitocondrias intestinales bajo un microscopio confocal para reducir la luz desenfocada y visualizar la morfología mitocondrial adecuada.
La forma de las mitocondrias es muy dinámica y cambia en función del entorno metabólico de los animales23 o incluso debido a la exposición a rigideces variables del sustrato24. Por lo tanto, dejar a los animales en portaobjetos de microscopio durante largos períodos de tiempo en ausencia de una fuente de alimento y sobre el sustrato rígido del vidrio puede afectar potencialmente la morfología mitocondrial. Aquí, este estudio encontró que las mitocondrias de los gusanos se fragmentan después de aproximadamente 30 minutos en un portaobjetos en solución M9, siendo las mitocondrias hipodérmicas las que muestran la mayor fragmentación (Figura 3). Por lo tanto, las imágenes de las mitocondrias deben realizarse rápidamente después de la preparación de la muestra.
Los productos químicos que restringen la movilidad de los gusanos, como la azida de sodio y la tetramisola, se utilizan comúnmente para obtener imágenes en vivo de C. elegans, ya que se requieren gusanos estacionarios para capturar múltiples secciones z de animales para obtener imágenes en 3D. Estudios previos han demostrado que la exposición a la azida sódica o tetramisol puede resultar en la fragmentación mitocondrial25. Sorprendentemente, se encontró que la azida de sodio, incluso en altas concentraciones, tenía un impacto limitado en la morfología mitocondrial en el músculo o el intestino. Sin embargo, la hipodermis mostró una fragmentación mitocondrial más temprana que los controles M9 (Figura 3). Es importante destacar que las altas concentraciones de tetramisol (100 mM) dieron lugar a una fragmentación mitocondrial significativa inmediatamente después de la exposición en todos los tipos de células. En comparación, las concentraciones medias (10 mM) resultaron en una fragmentación más rápida en comparación con un control M9. Las bajas concentraciones (1 mM) tuvieron un impacto limitado en la morfología mitocondrial. Estos datos sugieren que la utilización de azida sódica puede ser una opción viable para la obtención rápida de imágenes de las mitocondrias, mientras que el tetramisol debe evitarse en general.
Como se esperaba, las mitocondrias de todos los tejidos de C. elegans muestran fragmentación durante el proceso natural de envejecimiento (Figura 4A). La fragmentación se puede visualizar como mitocondrias que se presentan como estructuras más truncadas y esféricas, que son dramáticamente diferentes de las mitocondrias lineales y tubulares que se muestran en animales jóvenes. Es importante tener en cuenta que el intestino aumenta en estructuras esféricas autofluorescentes a una edad más avanzada, por lo que se debe tener cuidado de no confundir la autofluorescencia con las estructuras mitocondriales reales. Debido a que puede haber variabilidad de cambios en la estructura mitocondrial entre los gusanos, es importante realizar la cuantificación de la morfología mitocondrial en un tamaño de muestra significativo en lugar de solo obtener imágenes de unos pocos gusanos. En este caso, se utilizó mitoMAPR, que permite la cuantificación automatizada de la morfología mitocondrial utilizando un conjunto diverso de métricas, incluidos objetos, redes, uniones por red, puntos de unión, longitud del objeto, huella mitocondrial, cobertura mitocondrial y área del objeto (Tabla Suplementaria 1 y Tabla Suplementaria 2). La automatización elimina el sesgo subjetivo del usuario. Aquí, informamos que la métrica de longitud del objeto es óptima para medir cuantitativamente los cambios en la morfología mitocondrial muscular e intestinal durante el envejecimiento, y las métricas de puntos de unión para medir los cambios en la morfología mitocondrial hipodérmica durante el envejecimiento (Figura 4B).
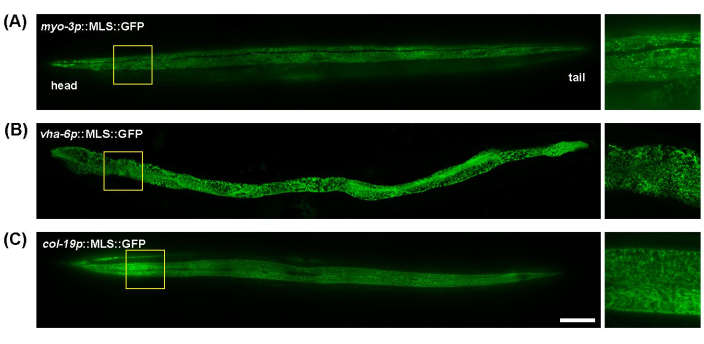
Figura 1: Imágenes de las mitocondrias en todo el cuerpo de C. elegans. Se realizaron imágenes mitocondriales durante todo el día 5 en animales adultos cultivados con EV desde la etapa L1 en animales transgénicos con expresión de MLS::GFP en el músculo (A), el intestino (B) y la hipodermis (C). Barra de escala: 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
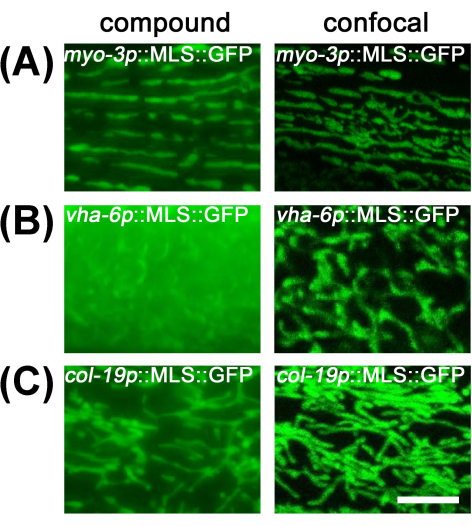
Figura 2: Comparación de las imágenes mitocondriales entre el microscopio compuesto y el microscopio confocal. Las imágenes mitocondriales se realizaron en animales adultos de día 1 cultivados en EV desde la etapa L1 en animales transgénicos con expresión de MLS::GFP en el músculo (A), el intestino (B) y la hipodermis (C). Barra de escala: 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
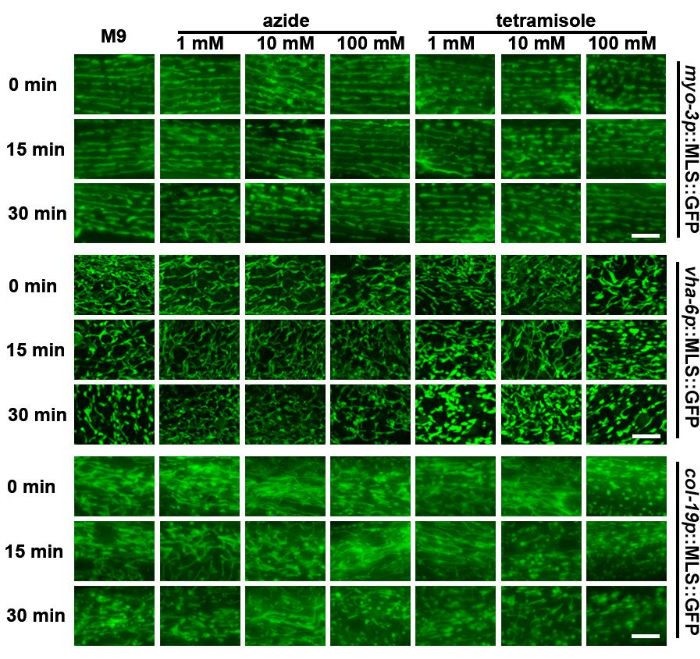
Figura 3: Valoración de la fragmentación mitocondrial de diferentes tejidos tras el tratamiento con M9, azida sódica y tetramisole. Las imágenes mitocondriales se realizaron en animales adultos de día 1 con expresión de MLS::GFP en el músculo, el intestino y la hipodermis. Los animales se cultivaron en EV desde la etapa L1. Los animales se colocaron en portaobjetos que contenían M9, azida sódica (1 mM, 10 mM y 100 mM) o tetramisol (1 mM, 10 mM y 100 mM) y las imágenes se realizaron inmediatamente después de la preparación del portaobjetos (0 min) o 15 min o 30 min después de la preparación del portaobjetos. Se muestran imágenes representativas para n > 5 animales por cepa para 2 réplicas biológicas. Barras de escala: 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
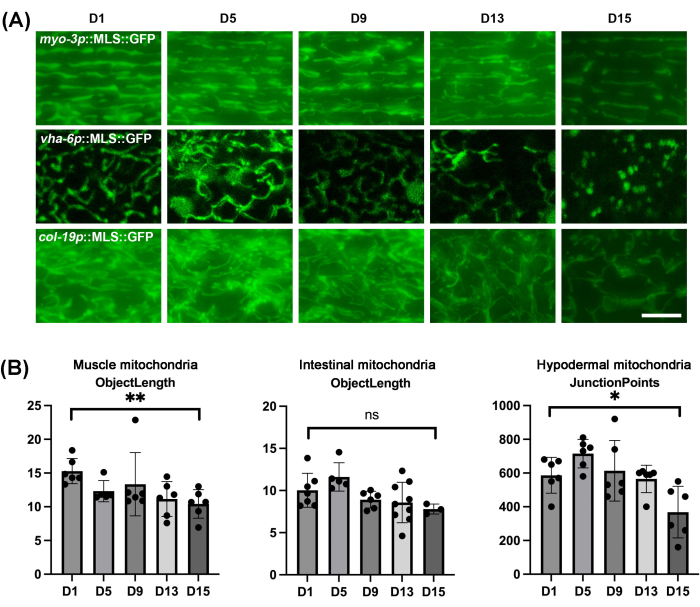
Figura 4: Imágenes mitocondriales de C. elegans durante el envejecimiento en diferentes tejidos y cuantificación de la morfología mitocondrial mediante MitoMAPR. (A) Se realizaron imágenes mitocondriales en animales adultos en los días 1, 5, 9, 13 y 15 de la edad adulta con expresión de MLS::GFP en el músculo, el intestino y la hipodermis. Los animales se cultivaron en EV desde la etapa L1 y se trasladaron a placas EV que contenían FUDR desde la etapa adulta del día 1. Las imágenes son representativas de n ≥ 5 animales por cepa para ≥ 3 réplicas biológicas. Barra de escala: 5 μm. (B) La cuantificación de la longitud del objeto de las mitocondrias musculares y las mitocondrias intestinales de los gusanos en los días 1, 5, 9, 13 y 15, y la cuantificación de los puntos de unión de las mitocondrias hipodérmicas de los gusanos en los días 1, 5, 9, 13 y 15. Todos los puntos de datos individuales combinados con la media ± DE. Los gráficos se trazaron y analizaron estadísticamente mediante una prueba t de Student. ns = no significativo, *p < 0,03; **p < 0,002; p < 0,0002; p < 0,0001. Haga clic aquí para ver una versión más grande de esta figura.
| Días de lombrices | Número de gusanos | Volumen de tampón (mL) |
| 1 | 25 | 13 |
| 5 | 25 | 14 |
| 9 | 20 | 15 |
| 13 | 20 | 15 |
Tabla 1: Números recomendados de gusanos y volúmenes de tampón al preparar portaobjetos.
Figura complementaria 1: Flujos de trabajo de la aplicación de una cuantificación de una sola imagen utilizando MitoMAPR y procesamiento por lotes para múltiples imágenes. (A) Flujo de trabajo de cuantificación de la morfología mitocondrial a partir de una sola imagen utilizando la macro MitoMAPR. (B) Proyección Z de un archivo de imagen 3D sin procesar usando Fiji (proyección máxima). (C) Recortar la región de interés de la imagen proyectada Z. (D) Imágenes de Skeletor adquiridas de la macro MitoMAPR. Barras de escala: 5 μm. (E) Flujo de trabajo de cuantificación de la morfología mitocondrial a partir de múltiples imágenes utilizando un proceso por lotes utilizando la macro MitoMAPR. (F) Captura de pantalla de la macro de recorte. (G) Captura de pantalla de un proceso por lotes para la proyección Z. (H) Captura de pantalla de la ventana de confirmación de selección de la macro de recorte por lotes. Haga clic aquí para descargar este archivo.
Tabla suplementaria 1: Análisis cuantitativo de las métricas de morfología mitocondrial en tejido muscular de C. elegans . Las métricas registradas incluyen objetos, redes, cruces por red, puntos de unión, longitud del objeto, huella mitocondrial, área del objeto y cobertura mitocondrial, medidos a lo largo de diferentes días. Los datos proporcionan una base para evaluar los cambios en la morfología mitocondrial con el envejecimiento. Haga clic aquí para descargar este archivo.
Tabla suplementaria 2: Efectos de los productos químicos inmovilizadores sobre la morfología mitocondrial en C. elegans. Los datos incluyen mediciones como objetos, redes, uniones por red, puntos de unión, longitud del objeto, huella mitocondrial, área del objeto y cobertura mitocondrial en diferentes concentraciones y puntos de tiempo después de la preparación de la muestra con tampón M9 o diferentes concentraciones de tetramisol y azida de sodio. Haga clic aquí para descargar este archivo.
Discusión
Las imágenes fluorescentes de la morfología mitocondrial son la forma más común de determinar los cambios en las mitocondrias. Si bien las técnicas de microscopía avanzadas, como la microscopía electrónica de transmisión (TEM), la microscopía de fuerza atómica y la criomicroscopía electrónica, ofrecen una mayor resolución, la microscopía de fluorescencia sigue siendo más asequible y accesible para la mayoría de los investigadores. Además, la microscopía fluorescente se puede realizar en células vivas, y en organismos modelo claros como C. elegans, las imágenes se pueden realizar en animales enteros26,27. Las imágenes de C. elegans transgénicos son bastante simples, y los marcadores fluorescentes codificados genéticamente permiten obtener imágenes más confiables y robustas de las mitocondrias, ya que no requieren un procesamiento complejo de muestras ni sufren de tinción variable en todos los tipos de células o condiciones de los tintes mitocondriales convencionales como MitoTracker o TMRE 28,29,30. Los marcadores fluorescentes codificados genéticamente generalmente implican el marcaje directo de proteínas mitocondriales con fluoróforos o la conjugación de fluoróforos con una secuencia de localización mitocondrial mínima. Estas construcciones a menudo son impulsadas por promotores específicos de tejidos, lo que permite la visualización de mitocondrias en diferentes tejidos como el músculo, el intestino o los tejidos de la hipodermis31. Generalmente, estas proteínas marcadas con fluorescencia están sobreexpresadas, lo que puede tener efectos fisiológicos potencialmente no deseados si las proteínas mitocondriales de longitud completa se sobreexpresan; por lo tanto, las secuencias mínimas de MLS son una mejor opción32. Sin embargo, incluso con la sobreexpresión de fusiones mínimas de proteínas fluorescentes MLS, se debe evitar una alta sobreexpresión, ya que la importación de una gran cantidad de proteína a las mitocondrias puede colapsar el potencial de la membrana mitocondrial e impactar en la salud animal33. Si bien una caracterización exhaustiva de todas las cepas de C. elegans actualmente disponibles está más allá del alcance de este manuscrito, aquí se puede encontrar un análisis comparativo detallado de numerosos reporteros mitocondriales y los pros y contras de cada uno31.
Para la obtención de imágenes de células vivas de las mitocondrias en C. elegans, la microscopía compuesta estándar o de campo amplio puede ser una opción preferida debido a la alta velocidad y facilidad de estos métodos en comparación con la microscopía confocal. En este estudio, se demuestra que las células planas como el músculo y la hipodermis se benefician mínimamente de la microscopía confocal, y la microscopía compuesta permite la adquisición con suficiente resolución para visualizar la morfología mitocondrial adecuada. Las células más grandes, como el intestino, dificultan la microscopía compuesta debido a la luz desenfocada. Por lo tanto, se requiere microscopía confocal para obtener imágenes confiables de la morfología mitocondrial intestinal.
Una consideración importante para la obtención de imágenes 3D en todo el grosor del tejido en animales vivos es evitar el movimiento de los gusanos durante la adquisición de imágenes. Los investigadores a menudo utilizan métodos para paralizar gusanos, como el tetramisol o la azida de sodio34. La azida sódica inhibe la citocromo c oxidasa (complejo IV), una enzima crítica en la cadena de transporte de electrones de las mitocondrias, lo que conduce a la parálisis general debido a la falta de ATP necesario para las contracciones musculares y otras funciones celulares34,35. El tetramisol actúa imitando a la acetilcolina en las uniones neuromusculares, causando una despolarización persistente y contracción muscular36. Sin embargo, la exposición a estos fármacos puede inducir la fragmentación mitocondrial al causar estrés oxidativo23. En este estudio, se encontró que el tetramisol indujo muy rápidamente la fragmentación mitocondrial, pero la azida sódica tuvo un efecto mucho más limitado.
C. elegans ofrece una manera muy simple y fácil de estudiar el impacto del envejecimiento en la morfología mitocondrial debido a su corta vida útil y la facilidad para envejecer a los animales. En este caso, se optó por utilizar la exposición al FUDR, que es un método robusto para esterilizar químicamente a los animales evitando la replicación del ADN 20,37,38. Sin embargo, el FUDR puede tener efectos no deseados en parámetros específicos del envejecimiento, y para aquellos preocupados por los efectos fuera del objetivo del FUDR, se pueden utilizar otras estrategias para eliminar la progenie39. Por ejemplo, hay mutantes estériles, como el mutante de la línea germinal glp-4, sensible a la temperatura, o mutantes deficientes en espermatozoides, como CF512 40,41,42. Como alternativa, los animales también pueden envejecer de forma natural seleccionando manualmente a los adultos de su progenie a diario.
La cuantificación de los cambios en la morfología mitocondrial también es una consideración muy importante, ya que puede haber una variabilidad significativa en la morfología mitocondrial entre los animales. Por lo tanto, es esencial realizar análisis cuantitativos y estadísticas en un tamaño de muestra suficiente para sacar conclusiones significativas sobre los cambios en la morfología mitocondrial. Sin embargo, el análisis y la cuantificación de imágenes pueden sufrir en gran medida el sesgo subjetivo y los desafíos de la obtención de imágenes de estructuras muy complejas como las mitocondrias interconectadas. Con este fin, aquí hay un resumen de un método automatizado para cuantificar la morfología mitocondrial utilizando numerosas métricas desarrolladas por el laboratorio de Mair. MitoMAPR permite la cuantificación objetiva y automatizada de la morfología mitocondrial, midiendo varios aspectos de las mitocondrias, incluida la red mitocondrial, la longitud del objeto, la distribución, la cobertura de la red y la huella mitocondrial. MitoMAPR es una macro gratuita para ImageJ y, por lo tanto, está disponible para todos los laboratorios con una computadora funcional. Un aspecto importante del uso de MitoMAPR es realizar la cuantificación en una muestra grande para determinar qué métrica de la morfología mitocondrial es más robusta para determinar los cambios en las condiciones experimentales que se están probando43,44. Aquí, se encuentra que la longitud del objeto y los puntos de unión son las mejores métricas para determinar los cambios durante el envejecimiento en el músculo, el intestino y la hipodermis. Un enfoque alternativo para analizar los cambios en la morfología mitocondrial es la generación de representaciones 3D de las mitocondrias a partir de imágenes z-stack, seguidas de análisis 3D16,45. Esto se puede lograr utilizando software disponible comercialmente, como Image-Pro Plus con los módulos SharpStack Total Deconvolution y 3D Constructor. Se ha demostrado que la cuantificación de las representaciones mitocondriales en 3D proporciona información más precisa sobre la forma mitocondrial y las propiedades de la red46. Sin embargo, este método es técnicamente más complejo y costoso en comparación con el enfoque semi-3D, que implica colapsar múltiples secciones de imagen de una pila z confocal o de campo amplio en una sola proyección 2D y analizarlas con herramientas como mitoMAPR. Este estudio resume el uso de la expresión de una sola copia de GFP dirigida a la matriz mitocondrial y detalla algunos escollos que se deben evitar durante la obtención de imágenes.
Limitaciones y consideraciones de tiempo
Aunque la microscopía confocal se recomienda para tejidos grandes y gruesos, como el tejido intestinal, las técnicas estándar de microscopía confocal de barrido lineal pueden ser demasiado lentas para realizar imágenes rápidas de las mitocondrias. Esto es especialmente cierto teniendo en cuenta nuestros datos que muestran que mantener los gusanos en los portaobjetos durante largos períodos de tiempo puede resultar en la fragmentación de las mitocondrias. Aunque la tecnología de microscopía confocal ha avanzado significativamente, los microscopios que pueden realizar imágenes rápidas, como el disco giratorio, Airyscan o el utilizado en este estudio, pueden tener un costo prohibitivo para algunos laboratorios. En estos casos, la microscopía compuesta puede combinarse con métodos de aclaramiento computacional para eliminar la luz desenfocada, como la deconvolución47.
Además, como se describe, MitoMAPR es un potente macro automatizado para medir cuantitativamente la longitud de las mitocondrias y la interconexión de la red mitocondrial. Sin embargo, debe usarse con cuidado teniendo en cuenta las limitaciones enumeradas aquí. En primer lugar, el uso de una GFP dirigida a la matriz solo observa la fragmentación mitocondrial de la membrana interna, que puede no recapitular completamente la morfología mitocondrial de la membrana externa, ya que los eventos de fisión de la membrana interna pueden ocurrir en ausencia de fisión de la membrana externa. Por lo tanto, para obtener imágenes más refinadas de ambas membranas, se debe utilizar un fluoróforo dirigido a la matriz y a la membrana externa mitocondrial. Dado que los marcadores externos de la membrana mitocondrial pueden sufrir las mismas consecuencias que las proteínas localizadas en la matriz si están muy sobreexpresados, se recomienda el uso de marcadores de copia única, especialmente aquellos que utilizan secuencias de localización mitocondrial mínimas en lugar de proteínas completas como las utilizadas aquí31.
Como se describe en la Figura 3, las mitocondrias pueden fragmentarse rápidamente bajo el microscopio, y los diferentes métodos para preparar portaobjetos de muestra pueden afectar en gran medida la morfología dependiendo del tampón que se utilice. Los datos representativos descritos en este manuscrito muestran que las mitocondrias de los gusanos experimentan una fragmentación mínima bajo M9 y bajas concentraciones de azida sódica. El tetramisol, otro químico ampliamente utilizado para paralizar gusanos, fragmenta rápidamente la mitocondria, lo que indica que su uso debe evitarse en general. Aunque M9 y azida de sodio no mostraron una fragmentación significativa de las mitocondrias, es importante tener en cuenta que las diferentes cepas podrían responder de manera diferente a los resultados que se muestran aquí. Si bien otros estudios han demostrado que la azida de sodio puede fragmentar las mitocondrias, es posible que nuestras cepas no muestren una fragmentación mitocondrial significativa con la exposición a la azida de sodio debido al bajo nivel de expresión de nuestras construcciones. La alta expresión de proteínas localizadas en las mitocondrias puede colapsar el potencial de membrana y, por lo tanto, las diferentes cepas utilizadas en otros estudios pueden hacer que las mitocondrias sean más susceptibles a la fragmentación de las mismas concentraciones de azida de sodio utilizadas en este estudio. En cualquier caso, se debe tener cuidado para garantizar que los productos químicos que paralizan los gusanos no causen fragmentación mitocondrial en condiciones específicas se prueben antes de su uso en todos los estudios, ya que los mutantes o condiciones específicas pueden ser más susceptibles a la fragmentación mitocondrial inducida por fármacos. Además, incluso mantener los gusanos en M9 puede resultar en cambios en su morfología y actividad mitocondrial natural, ya que se ha demostrado previamente que su actividad de natación en el tampón M9 afecta la fisión mitocondrial y la dinámica de fusión. Además, dejar gusanos en M9 durante largos períodos de tiempo puede activar respuestas de hipoxia que afectan significativamente a las mitocondrias al causar interrupciones de la proteostasis mitocondrial48,49.
Finalmente, otra consideración importante es que, si bien los cambios en la morfología mitocondrial a menudo se correlacionan con cambios en la función mitocondrial, no siempre existe una correlación directa entre los dos. Por lo tanto, se recomienda un análisis más exhaustivo de la función mitocondrial. Por ejemplo, la tasa de consumo de oxígeno se puede medir usando un instrumento Seahorse50, el potencial de membrana mitocondrial se puede medir usando colorantes de potencial de membrana como JC9 o TMRE51, el estado oxidativo mitocondrial se puede medir usando colorantes sensibles a redox como roGFP52, y la resistencia al estrés mitocondrial se puede medir usando la sensibilidad a factores estresantes como la rotenona53. Debido a que las imágenes de las mitocondrias pueden ser bastante rápidas, ofrecemos los métodos aquí como un primer paso fácil para determinar si las condiciones experimentales afectan la morfología mitocondrial. Estos métodos son susceptibles incluso para el cribado a gran escala de fármacos o genes, y se recomienda el seguimiento de un análisis mitocondrial más exhaustivo utilizando métricas adicionales. En conjunto, aquí revisamos lo que se cree que son los métodos más simples para obtener imágenes de la morfología mitocondrial en C. elegans con errores experimentales mínimos .
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
J.K. cuenta con el apoyo de la USC Provost Fellowship; M.A. y G.G. cuentan con el apoyo de T32AG052374; M.V. es compatible con 1R25AG076400; y R.H.S. cuenta con el apoyo de R01AG079806 del Instituto Nacional sobre el Envejecimiento y 2022-A-010-SUP de la Fundación Larry L. Hillblom. Algunas cepas fueron proporcionadas por el CGC, que está financiado por la subvención P40 OD010440 de la Oficina de Programas de Infraestructura de Investigación de los NIH. Algunos análisis genéticos se realizaron utilizando Wormbase, que está financiado por una subvención U41 HG002223.
Materiales
| Name | Company | Catalog Number | Comments |
| 5-fluoro-2'-deoxyuridine (FUDR) | Spectrum Chemical | F2026-10GMBL | for proliferation inhibition |
| APEX IPTG | Genesee | 18-242 | for RNAi |
| Bacto Agar | VWR | 90000-764 | for NGM plates |
| Bacto Peptone | VWR | 97064-330 | for NGM plates |
| Calcium chloride dihydrate | VWR | 97061-904 | for NGM plates |
| Carbenicillin | VWR | 76345-522 | for RNAi |
| Cholesterol | VWR | 80057-932 | for NGM plates |
| Confocal microscope | Stellaris 5 | ||
| DMSO | VWR | BDH1115-1LP | solvent for drugs |
| HistoBond microscope slides | VWR | 16005-110 | for slides preparation |
| LB Broth | VWR | 95020-778 | for LB |
| LEICA S7E Dissecting Scope | Leica | 10450840 | Standard dissecting microscope |
| LEICA STELLARIS 5 | Leica | 158101100 | Confocal Microscope |
| LEICA THUNDER | Leica | 11525679 | Compound Microscope |
| Magnesium sulfate heptahydrate | VWR | 97062-132 | for NGM plates, M9 |
| pL4440 empty vector plasmid | addgene | plasmid #1654 | for empty vector plasmid |
| Potassium Chloride | VWR | 97061-566 | for bleach soluton |
| Potassium phosphate dibasic | VWR | EM-PX1570-2 | for NGM plates |
| Potassium phosphate monobasic | VWR | EM-PX1565-5 | for M9 |
| Sodium azide | VWR | 97064-646 | for paralyzing worms |
| Sodium Chloride | VWR | EM-SX0420-5 | for NGM plates, M9 |
| Sodium hypochlorite | VWR | RC7495.7-32 | for bleach solution |
| Sodium phosphate dibasic | VWR | 71003-472 | for M9 |
| Tetracycline hydrochloride | VWR | 97061-638 | for RNAi |
| WOB-L® Dry Vacuum Pumps, Standard-Duty, Welch® | VWR | 80077-612 | for aspiration |
Referencias
- Protasoni, M., Zeviani, M. Mitochondrial structure and bioenergetics in normal and disease conditions. Int J Mol Sci. 22 (2), 586 (2021).
- Wang, Y., et al. The role of mitochondrial dynamics in disease. MedComm. 4 (6), e462 (2023).
- Kamer, K. J., Mootha, V. K. The molecular era of the mitochondrial calcium uniporter. Nat Rev Mol Cell Biol. 16 (9), 545-553 (2015).
- Chen, W., Zhao, H., Li, Y. Mitochondrial dynamics in health and disease: mechanisms and potential targets. Signal Transduct Target Ther. 8 (1), 333 (2023).
- Adebayo, M., Singh, S., Singh, A. P., Dasgupta, S. Mitochondrial fusion and fission: The fine-tune balance for cellular homeostasis. FASEB J. 35 (6), e21620 (2021).
- Madan, S., Uttekar, B., Chowdhary, S., Rikhy, R. Mitochondria lead the way: mitochondrial dynamics and function in cellular movements in development and disease. Front Cell Dev Biol. 9, 781933 (2021).
- Sharma, A., Smith, H. J., Yao, P., Mair, W. B. Causal roles of mitochondrial dynamics in longevity and healthy aging. EMBO Rep. 20 (12), e48395 (2019).
- Rolland, S. G., Lu, Y., David, C. N., Conradt, B. The BCL-2-like protein CED-9 of C. elegans promotes FZO-1/Mfn1,2- and EAT-3/Opa1-dependent mitochondrial fusion. J Cell Biol. 186 (4), 525-540 (2009).
- Labrousse, A. M., Zappaterra, M. D., Rube, D. A., van der Bliek, A. M. C. elegans dynamin-related protein DRP-1 controls severing of the mitochondrial outer membrane. Mol Cell. 4 (5), 815-826 (1999).
- Dickinson, D. J., Pani, A. M., Heppert, J. K., Higgins, C. D., Goldstein, B. Streamlined genome engineering with a self-excising drug selection cassette. Genetics. 200 (4), 1035-1049 (2015).
- Bosher, J. M., Labouesse, M. RNA interference: Genetic wand and genetic watchdog. Nat Cell Biol. 2 (2), E31-E36 (2000).
- Yu, C. C. J., et al. Expansion microscopy of C. elegans. eLife. 9, e46249 (2020).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Front Endocrinol. 11, 554994 (2020).
- Dingley, S., et al. Mitochondrial respiratory chain dysfunction variably increases oxidant stress in Caenorhabditis elegans. Mitochondrion. 10 (2), 125-136 (2010).
- Presley, A. D., Fuller, K. M., Arriaga, E. A. MitoTracker Green labeling of mitochondrial proteins and their subsequent analysis by capillary electrophoresis with laser-induced fluorescence detection. J Chromatogr B Anal Technol Biomed Life Sci. 793 (1), 141-150 (2003).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Curr Protoc Cell Biol. Chapter 4, Unit 4.25.1-Unit 4.25.21 (2010).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. Novel imaging tools to study mitochondrial morphology in Caenorhabditis elegans. Life Sci Alliance. 7 (11), e202402918 (2024).
- Benedetti, C., Haynes, C. M., Yang, Y., Harding, H. P., Ron, D. Ubiquitin-like protein 5 positively regulates chaperone gene expression in the mitochondrial unfolded protein response. Genetics. 174 (1), 229-239 (2006).
- Begelman, D. V., et al. An aco-2::gfp knock-in enables the monitoring of mitochondrial morphology throughout C. elegans lifespan. microPublication Biol. 2022, (2022).
- Castro Torres, T., et al. Surveying low-cost methods to measure lifespan and healthspan in Caenorhabditis elegans. J Vis Exp. 183, e64091 (2022).
- Rasmussen, N. R., Reiner, D. J. Nuclear translocation of the tagged endogenous MAPK MPK-1 denotes a subset of activation events in C. elegans development. J Cell Sci. 134 (17), jcs258456 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Luz, A. L., et al. Mitochondrial morphology and fundamental parameters of the mitochondrial respiratory chain are altered in Caenorhabditis elegans strains deficient in mitochondrial dynamics and homeostasis processes. PLoS One. 10 (6), e0130940 (2015).
- Oorloff, M., et al. Mechanical stress through growth on stiffer substrates impacts animal health and longevity in C. elegans. bioRxiv. , (2024).
- Chu, X., Wu, S., Raju, R. NLRX1 regulation following acute mitochondrial injury. Front Immunol. 10, 2431 (2019).
- Shah, P., Bao, Z., Zaidel-Bar, R. Visualizing and quantifying molecular and cellular processes in Caenorhabditis elegans using light microscopy. Genetics. 221 (4), iyac068 (2022).
- Wang, Y., Wang, P., Li, C. Fluorescence microscopic platforms imaging mitochondrial abnormalities in neurodegenerative diseases. Adv Drug Deliv Rev. 197, 114841 (2023).
- Ding, J., et al. An expanded GCaMP reporter toolkit for functional imaging in Caenorhabditis elegans. G3 (Bethesda, Md). 13 (10), jkad183 (2023).
- Sarasija, S., Norman, K. R. Analysis of mitochondrial structure in the body wall muscle of Caenorhabditis elegans. Bio-Protocol. 8 (7), e2801 (2018).
- Chaweeborisuit, P., Suriyonplengsaeng, C., Suphamungmee, W., Sobhon, P., Meemon, K. Nematicidal effect of plumbagin on Caenorhabditis elegans: A model for testing a nematicidal drug. Z Naturforsch C J Biosci. 71 (5-6), 121-131 (2016).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. . Novel imaging tools to study mitochondrial dynamics in Caenorhabditis elegans. 7, 11 (2024).
- Bolognesi, B., Lehner, B. Reaching the limit. eLife. 7, e39804 (2018).
- Jishi, A., Qi, X. Altered mitochondrial protein homeostasis and proteinopathies. Front Mol Neurosci. 15, 867935 (2022).
- Morton, K. S., Wahl, A. K., Meyer, J. N. The effect of common paralytic agents used for fluorescence imaging on redox tone and ATP levels in Caenorhabditis elegans. PLoS One. 19 (4), e0292415 (2024).
- Noumi, T., Maeda, M., Futai, M. Mode of inhibition of sodium azide on H+-ATPase of Escherichia coli. FEBS Lett. 213 (2), 381-384 (1987).
- Siete, C., Xiong, R., Khalid, A., Hsieh, Y. -. W., Chuang, C. -. F. Immobilization of C. elegans with different concentrations of an anesthetic for time-lapse imaging of dynamic protein trafficking in neurons. microPublication Biology. 2024, (2024).
- Mitchell, D. H., Stiles, J. W., Santelli, J., Sanadi, D. R. Synchronous growth and aging of Caenorhabditis elegans in the presence of fluorodeoxyuridine. J Gerontol. 34 (1), 28-36 (1979).
- Santi, D. V., McHenry, C. S. 5-Fluoro-2'-deoxyuridylate: covalent complex with thymidylate synthetase. Proc Natl Acad Sci U S A. 69 (7), 1855-1857 (1972).
- Wang, H., Zhao, Y., Zhang, Z. Age-dependent effects of floxuridine (FUdR) on senescent pathology and mortality in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun. 509 (3), 694-699 (2019).
- Beanan, M. J., Strome, S. Characterization of a germ-line proliferation mutation in C. elegans. Development (Cambridge, England). 116 (3), 755-766 (1992).
- Austin, J., Kimble, J. glp-1 is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans. Cell. 51 (4), 589-599 (1987).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Zhang, Y., et al. Neuronal TORC1 modulates longevity via AMPK and cell nonautonomous regulation of mitochondrial dynamics in C. elegans. eLife. 8, e49158 (2019).
- Dutta, N., et al. Investigating impacts of the mycothiazole chemotype as a chemical probe for the study of mitochondrial function and aging. GeroScience. 46 (6), 6009-6028 (2024).
- Tronstad, K., et al. Regulation and quantification of cellular mitochondrial morphology and content. Curr Pharm Des. 20 (35), 5634-5652 (2014).
- Nikolaisen, J., et al. Automated quantification and integrative analysis of 2D and 3D mitochondrial shape and network properties. PLoS One. 9 (7), e101365 (2014).
- Lee, J. S., Wee, T. L. E., Brown, C. M. Calibration of wide-field deconvolution microscopy for quantitative fluorescence imaging. J Biomol Tech. 25 (1), 31-40 (2014).
- Hong, M., Kwon, J. Y., Shim, J., Lee, J. Differential hypoxia response of hsp-16 genes in the nematode. J Mol Biol. 344 (2), 369-381 (2004).
- Yan, J., Sun, C. L., Shin, S., Van Gilst, M., Crowder, C. M. Effect of the mitochondrial unfolded protein response on hypoxic death and mitochondrial protein aggregation. Cell Death Dis. 12 (7), 711 (2021).
- Koopman, M., et al. A screening-based platform for the assessment of cellular respiration in Caenorhabditis elegans. Nat Protoc. 11 (10), 1798-1816 (2016).
- Shpilka, T., et al. UPRmt scales mitochondrial network expansion with protein synthesis via mitochondrial import in Caenorhabditis elegans. Nat Comm. 12 (1), 479 (2021).
- Braeckman, B. P., Smolders, A., Back, P., De Henau, S. In vivo detection of reactive oxygen species and redox status in Caenorhabditis elegans. antioxidants & redox signaling. 25 (10), 577-592 (2016).
- Bar-Ziv, R., et al. Measurements of physiological stress responses in C. elegans. J Vis Exp. 159, e61001 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados