Method Article
Imagerie et quantification de la morphologie mitochondriale chez C. elegans au cours du vieillissement
Dans cet article
Résumé
Ce protocole fournit une approche standardisée pour l’imagerie de la morphologie mitochondriale dans plusieurs tissus de C. elegans au cours du vieillissement.
Résumé
Les mitochondries, organites cellulaires importants présents dans la plupart des cellules eucaryotes, sont des sites majeurs de production d’énergie par la respiration aérobie. Au-delà de ce rôle bien connu de « centrale cellulaire », les mitochondries sont également impliquées dans de nombreux autres processus cellulaires essentiels, notamment la régulation du métabolisme cellulaire, la prolifération, la signalisation immunitaire et la signalisation hormonale. La détérioration de la fonction mitochondriale au cours du vieillissement ou sous stress mitochondrial est souvent caractérisée par des changements distincts de la morphologie et du volume mitochondriaux. Le nématode C. elegans est un modèle idéal pour étudier ces changements en raison de son corps transparent et de sa courte durée de vie, qui facilitent la microscopie en direct tout au long de sa vie. Cependant, même dans le domaine de C. elegans , de nombreuses constructions et méthodes transgéniques pour l’imagerie mitochondriale sont disponibles, chacune avec ses propres limites. Ici, les constructions GFP à copie unique, localisées dans la matrice, sont présentées comme une méthode robuste et fiable pour l’imagerie de la morphologie mitochondriale chez C. elegans. Cette étude se concentre spécifiquement sur les facteurs contrôlables expérimentalement afin de minimiser les erreurs et de réduire la variabilité entre les répétitions et entre les études lors de la réalisation d’une imagerie mitochondriale pendant le processus de vieillissement. De plus, mitoMAPR est recommandé comme méthode robuste pour quantifier les changements de morphologie mitochondriale à travers les types de tissus au cours du vieillissement.
Introduction
Les mitochondries sont des organites cellulaires entourés d’une double membrane phospholipidique et jouent un rôle important dans le maintien de la bioénergétique cellulaire en tant que site clé du métabolisme et de la production d’énergie1. Un grand nombre de mitochondries génèrent en permanence de l’énergie sous forme d’ATP pour répondre aux demandes cellulaires2. En plus de ces rôles importants, les mitochondries participent également à des processus cellulaires complexes tels que la transduction du signal, l’autophagie, l’immunité innée, le cycle cellulaire et les voies de mort cellulaire 2,3. Les mitochondries présentent des morphologies diverses, allant de petits organites individuels à de vastes réseaux tubulaires interconnectés, en fonction de divers besoins énergétiques et de la santé mitochondriale4.
Une caractéristique clé des mitochondries est leur nature dynamique qui leur permet d’alterner entre ces différentes formes à travers une séquence étroitement coordonnée et continue d’événements de fission et de fusion4. L’équilibre précis entre ces processus opposés régule la morphologie, le nombre, la taille et le positionnement des mitochondries dans le cytoplasme5. De plus, ces processus de fusion et de fission sont importants pour maintenir le contrôle de la qualité des mitochondries5. Par exemple, la fission mitochondriale est un composant essentiel de l’élimination des mitochondries endommagées par mitophagie et de l’élimination sélective des mitochondries par autophagie2. La dynamique mitochondriale joue également un rôle important dans la division cellulaire, le développement, la résistance à divers facteurs de stress et le maintien du métabolisme cellulaire6.
La perturbation de la dynamique mitochondriale est impliquée dans de nombreuses maladies, notamment les troubles neurodégénératifs, les maladies métaboliques, les maladies cardiovasculaires et les cancers4. De plus, le vieillissement est associé à un remodelage spectaculaire des mitochondries, en grande partie dû à des changements dans ces dynamiques fusion-fission7. Par conséquent, la visualisation et le suivi des changements dans la morphologie mitochondriale dans diverses conditions de stress ou de maladie et tout au long du processus de vieillissement offrent des informations précieuses pour comprendre la fonction cellulaire, le mécanisme de la maladie et les stratégies thérapeutiques potentielles.
Comme de nombreuses voies moléculaires, la dynamique et la fonction mitochondriales sont également hautement conservées chez les eucaryotes, y compris les organismes modèles tels que le nématode Caenorhabditis elegans. À l’instar des cellules humaines, la fusion mitochondriale chez C. elegans est réalisée par la fonction des protéines de la guanine triphosphatase apparentée à la dynamine (GTPase), y compris FZO-1 (orthologue du mammifère Mfn1/2) et EAT-3 (orthologue du mammifère Opa1) qui contrôlent la fusion de la membrane mitochondriale externe (OMM) et de la membrane mitochondriale interne (IMM), respectivement8. La fission mitochondriale est régulée par la protéine liée à la dynamine (DRP-1, orthologue de Drp1 humain), qui favorise la fission mitochondriale en formant des complexes en forme d’anneau autour de la membrane externe mitochondriale qui resserrent et finissent par séparer les membranes mitochondriales9. La fusion mitochondriale joue un rôle central dans le contrôle de la qualité mitochondriale en permettant le mélange du contenu mitochondrial, y compris l’ADN mitochondrial, les protéines et les lipides, permettant la complémentarité des mitochondries partiellement endommagées par le contenu de mitochondries saines9. D’autre part, la fission mitochondriale permet aux mitochondries de se diviser, créant de nouvelles mitochondries et facilitant leur distribution non seulement dans le cytoplasme mais aussi vers les cellules filles pendant la division cellulaire, assurant ainsi une bonne hérédité et fonction mitochondriale9. Cet événement est également essentiel pour séparer les segments mitochondriaux endommagés ou dysfonctionnels, qui peuvent ensuite être ciblés pour la dégradation par mitophagie9.
C. elegans a longtemps été considéré comme l’un des systèmes de modèles génétiques les plus puissants en raison du génome complet et de la disponibilité d’une variété d’outils génétiques, y compris CRISPR/Cas9 qui facilite les modifications génétiques10, de nombreuses méthodes de surexpression des gènes et une méthode d’interférence ARN (ARNi) basée sur l’alimentation bactérienne11. De plus, leur anatomie transparente permet l’imagerie microscopique chez les organismes vivants12. Enfin, leur durée de vie relativement courte, leurs faibles coûts d’entretien et leur facilité à produire un grand nombre d’animaux du même âge en font un excellent système modèle pour la biologie du vieillissement13. Ces avantages, combinés à la conservation connue des principales voies de régulation mitochondriale, font de C. elegans un système modèle très attrayant pour l’étude de la dynamique mitochondriale au cours du vieillissement.
Les marqueurs fluorescents sont largement utilisés dans la recherche biologique pour visualiser et étudier les composants cellulaires, y compris les mitochondries. Il existe des colorants spécifiques perméables aux cellules tels que le MitoTracker et l’ester éthylique de tétraméthylrhodamine (TMRE) qui sont couramment utilisés14. Le premier est utilisé pour colorer la matrice mitochondriale15 chargée négativement, tandis que le second est utilisé pour évaluer le potentiel relatif de la membrane mitochondriale grâce à son ion triphénylphosphonium16 chargé positivement. Bien qu’ils ne nécessitent pas de transgéniques, l’épaisse cuticule des vers qui change de structure et de perméabilité au cours du vieillissement et la variabilité de l’infiltration de colorant à travers différents tissus rendent difficiles les approches basées sur les colorants chez C. elegans17. De plus, l’évaluation de la morphologie mitochondriale est faussée par les effets potentiels hors cible des colorants, tels que l’agrégation de colorants17. Au lieu de cela, des méthodes génétiques pour exprimer des fluorophores localisés dans les mitochondries sont couramment utilisées dans le modèle de ver.
Ici, cette étude se concentre sur la mise en évidence de souches exprimant une matrice mitochondriale localisée GFP (ci-après appelée MLS ::GFP) sous des promoteurs spécifiques de type cellulaire. Il est important de noter que ces lignées transgéniques ont été fabriquées à l’aide de la méthode MosSCI pour assurer l’expression en une seule copie du rapporteur dans un locus génomique connu, ce qui évite les problèmes avec d’autres souches disponibles. Par exemple, l’expression à haute dose de certaines protéines ciblées par les mitochondries entraîne une variabilité des niveaux d’expression en raison de l’intégration de réseaux de plasmides extrachromosomiques dans un locus aléatoire avec un nombre de copies inconnu18. De plus, il a déjà été démontré qu’ils induisent des dommages aux mitochondries, car il est difficile pour les cellules de localiser correctement les protéines mitochondriales surexprimées19. Par conséquent, la précision, la stabilité et la faible expression des gènes et la facilité de croisement avec des loci connus font de ces transgéniques MosSCI la méthode préférée. À l’aide de ces souches, cette étude normalise les méthodes d’imagerie des mitochondries dans le muscle, l’intestin et l’hypoderme de C. elegans. En outre, il met en évidence les applications techniques importantes et les méthodes de dépannage qu’il est important de prendre en compte et d’assurer une analyse expérimentale reproductible des mitochondries au cours du vieillissement chez C. elegans.
Protocole
Des détails sur les réactifs et l’équipement utilisés dans cette étude sont énumérés dans la table des matériaux.
1. Croissance et maintien de C. elegans
- Préparation des plaques de milieu de croissance des nématodes (NGM)
- Pour la culture de C. elegans, utiliser des plaques de gélose étalon à 2 % avec NGM contenant 1 mM de CaCl2, 12,93 μM (5 μg/mL) de cholestérol, 25 mM de KPO4 (pH 6,0), 1 mM de MgSO4, 2,5 mM (0,25 % p/v) de peptone et 51,3 mM de NaCl. Voir la méthode détaillée de coulée des plaques NGM décrite dans Castro Torres et al.20.
REMARQUE : Pour les expériences d’interférence ARN (ARNi), ajouter 1 mL de 1 M d’IPTG et 1 mL de 100 mg/mL de carbenicilline pour 1 L de plaques de gélose NGM. - Une fois que les plaques NGM se sont solidifiées, cultiver une culture d’OP50 dans un bouillon de lysogénie (LB) pendant 24 à 48 h à température ambiante ou cultiver une culture de HT115 portant un plasmide vecteur vide pL4440 dans LB avec des antibiotiques (ampicilline/carbénicilline 100 μg/mL + tétracycline 5 μg/mL) en agitant à 37 °C pendant 12 à 16 h.
REMARQUE : Ce plasmide est disponible auprès de sources commerciales (voir le tableau des matériaux). - Semez 200 μL de culture OP50 ou HT115 sur des plaques de 60 mm, ou 1 mL sur des plaques de 100 mm.
- Séchez les plaques jusqu’à ce qu’elles ne soient plus mouillées et conservez-les dans des récipients hermétiques à 4 °C jusqu’à 2 mois.
- Facultatif : Ajouter 100 μL de 5-fluoro-2'-désoxyuridine (FUDR) de 10 mg/mL directement sur des plaques de gélose NGM ensemencées de bactéries pour stériliser chimiquement les vers L4/adultes et prévenir le développement de vers plus jeunes.
REMARQUE : Les souches développées et utilisées dans cette étude sont les suivantes :
(1) RHS191 - uthSi17[myo3p ::MLS ::GFP ::unc-54 3'UTR ::cb-unc-119(+)] I
(2) RHS192 - uthSi83[col19p :: MLS ::GFP ::unc-54 3'UTR ::cb-unc-119(+)]I
(3) RHS193 - uthSi80[vha-6p :: MLS ::GFP ::unc-54 3'UTR ::cb-unc-119(+)] IV
- Pour la culture de C. elegans, utiliser des plaques de gélose étalon à 2 % avec NGM contenant 1 mM de CaCl2, 12,93 μM (5 μg/mL) de cholestérol, 25 mM de KPO4 (pH 6,0), 1 mM de MgSO4, 2,5 mM (0,25 % p/v) de peptone et 51,3 mM de NaCl. Voir la méthode détaillée de coulée des plaques NGM décrite dans Castro Torres et al.20.
- Synchronisation C. elegans par blanchiment
- Pour un essai à grande échelle qui nécessite un grand nombre de nématodes, coupez une plaque de gélose NGM de 60 mm remplie d’animaux en petits morceaux et coupez-les en morceaux sur des plaques de 100 mm qui sont ensemencées de bactéries pour l’expansion. Attendez 2-3 jours pour que les plaques de 100 mm soient pleines de vers lorsqu’elles sont cultivées à 20 °C après le morceau. Pour des recommandations plus détaillées sur l’expansion des animaux, veuillez consulter Castro Torres et al.20.
- Pour recueillir les vers en vue de la synchronisation, verser 5 à 10 mL de solution M9 (22 mM de KH2PO4 monobasique, 42,3 mM de Na2HPO4, 85,6 mM de NaCl, 1 mM de MgSO4) sur des plaques de gélose NGM de 100 mm avec des vers, et agiter doucement la solution M9 pour détacher les vers des pelouses bactériennes.
- Prélevez les vers à l’aide d’une pipette sérologique et transférez-les dans des tubes coniques de 15 ml.
REMARQUE : Les pipettes sérologiques en verre sont recommandées car les pipettes sérologiques en plastique ont tendance à faire adhérer les vers à la paroi interne des pipettes en plastique. - Centrifugeuse pendant 30 s à 1 100 x g à température ambiante pour réduire les vers et aspirer le surnageant à l’aide d’une pompe à vide.
- Préparez 5 ml de solution de blanchiment contenant 1,5 ml d’hypochlorite de sodium à 6 %, 0,75 ml de NaOH ou de KOH 5 M et 2,75 ml de dH2O. Versez les 5 ml de solution de blanchiment dans la pastille de ver.
ATTENTION : Le port de gants et d’une blouse de laboratoire est recommandé à cette étape, car les solutions d’hypochlorite et d’hydroxyde de sodium sont corrosives. - Incuber le mélange ver + solution de blanchiment pendant ~4-6 min jusqu’à ce que tous les corps des animaux soient complètement dissous et qu’il ne reste que les œufs. Secouez vigoureusement le mélange pour faciliter le processus de dissolution.
REMARQUE : Vérifiez les vers toutes les minutes sous un microscope de dissection. Raccourcissez ou prolongez le temps de blanchissement en fonction de la quantité de dissolution du corps des vers adultes. Laisser les œufs à l’intérieur d’une solution de blanchiment pendant une période prolongée endommagera les œufs et affectera la viabilité des nématodes. - Centrifuger le mélange œufs/solution de blanchiment pendant 30 s à 1 100 x g à température ambiante pour granuler les œufs, puis aspirer soigneusement le surnageant à l’aide d’une pompe à vide, en veillant à ne pas aspirer la pastille d’œuf.
REMARQUE : Il est recommandé d’effectuer les lavages rapidement pour éviter une exposition prolongée des œufs à la solution de blanchiment. - Lavez les œufs avec 5 à 10 ml de solution M9 4 fois.
- Remettre les œufs en suspension dans un petit volume de solution M9 et les déposer sur des plaques NGM ensemencées de bactéries en pipetant jusqu’à 50 μL. Calculer une estimation approximative du nombre d’œufs en pipetant 3 à 5 μL du mélange d’œufs sur une plaque et en comptant le nombre d’œufs dans ce volume. Ajoutez M9 pour diluer le mélange d’œufs en un nombre dénombrable d’œufs dans ce volume.
REMARQUE : Il est recommandé de plaquer moins de 100 vers par plaque car les vers peuvent mourir de faim avant le premier jour de l’âge adulte lorsqu’il y a trop d’animaux plaqués. Pour des recommandations plus détaillées sur le nombre d’animaux, voir Castro Torres et al.20. - Alternativement, L1-arrêter les nématodes pour une synchronisation d’âge plus serrée. Pour l’arrêt de L1, ajouter le volume approprié de solution M9 à la pastille d’œuf dans un tube conique de 15 mL à un volume de 10 à 12 mL. Laissez les vers tourner dans un rotateur pendant 16 à 24 h à 20 °C. Centrifuger (comme à l’étape 1.2.7) et aspirer le surnageant. Calculer la concentration d’animaux L1 et les déposer sur des plaques NGM comme décrit pour les œufs à l’étape 1.2.9.
- Déterminer l’âge de la population de C. elegans jusqu’à l’âge souhaité : Transférer les animaux L4/adultes sur des plaques contenant de la 5-fluoro-2'-désoxyuridine (FUDR) comme décrit à l’étape 1.1.6 pour prévenir la progéniture et éliminer toute progéniture déjà produite. Pour d’autres méthodes pour éliminer les vers sans FUDR, reportez-vous à Castro Torres et al.20.
REMARQUE : Certains facteurs importants doivent être pris en compte avant de choisir le blanchiment comme méthode de synchronisation des vers à leur premier stade larvaire. Comme nous l’avons souligné précédemment, la surexposition à l’eau de Javel peut endommager les embryons, réduire les taux d’éclosion et créer une pression de sélection pour les souches résistantes à l’eau de Javel, ce qui peut altérer la génétique de la population. De plus, si les embryons ne sont pas nourris pendant de longues périodes, l’épuisement des nutriments peut nuire au métabolisme et au développement. Par conséquent, les méthodes de ponte ou NemaSync peuvent être utilisées comme alternative aux méthodes de blanchiment. La méthode de ponte des œufs est décrite par étapes dans l’étude de Castro Torres et al.20. NemaSync est une méthode commerciale plus récente et plus coûteuse, où un synchroniseur de vers manuel est utilisé pour synchroniser les vers sans utiliser de produits chimiques21.
2. Imagerie des mitochondries chez C. elegans
- Préparation de lames pour l’imagerie de C. elegans
- Ajouter 10 à 20 μL de solution M9 sur des lames de verre et transférer le nombre désiré de vers adultes aux âges spécifiques dans la solution sur la lame. Environ 25 vers sont recommandés.
REMARQUE : L’utilisation de méthodes traditionnelles pour paralyser les vers (par exemple, le tétramisole ou l’azoture de sodium) peut provoquer une fragmentation mitochondriale dans certaines conditions (voir ci-dessous). Au lieu de cela, l’utilisation de lames HistoBond est recommandée ici, qui ont une surface avec une charge de surface positive permanente, réduira le mouvement des vers sans utiliser de médicaments susceptibles d’introduire des artefacts. Il est important de mettre un rapport approprié entre les vers et la solution M9, car de faibles volumes peuvent provoquer l’écrasement des animaux entre la lame et la lamelle, tandis que des volumes élevés entraîneront le déplacement des animaux pendant l’acquisition de l’image. Les recommandations se trouvent dans le tableau 1. - Alternativement, pour l’utilisation de médicaments qui paralysent les vers, reportez-vous aux résultats représentatifs de ce manuscrit pour une gamme de concentrations et de points temporels qui peuvent être utilisés avec une fragmentation limitée des mitochondries.
REMARQUE : Bien qu’il n’y ait pas eu de fragmentation majeure chez les animaux en bonne santé de type sauvage, il est important de se rappeler que certains mutants ou conditions peuvent subir une fragmentation mitochondriale avec la même concentration de ces médicaments, de sorte que certains tests sont nécessaires lors de l’utilisation de ces médicaments. - Couvrez les vers dans la solution M9 sur la lame avec un verre de couverture et utilisez du vernis à ongles pour sceller les côtés et empêcher l’évaporation de la solution M9.
- Facultatif : Pour l’imagerie musculaire, roulez les vers en poussant doucement la lamelle. Cela permet aux muscles de la paroi corporelle sur les côtés des vers d’être mieux alignés avec le plan focal pour des images de meilleure qualité.
- Ajouter 10 à 20 μL de solution M9 sur des lames de verre et transférer le nombre désiré de vers adultes aux âges spécifiques dans la solution sur la lame. Environ 25 vers sont recommandés.
- Imagerie des mitochondries à l’aide d’un microscope composé
- Utilisez un microscope standard à grand champ pour les mitochondries musculaires et hypodermiques (myo-3p ::MLS ::GFP et col-19p ::MLS ::GFP).
REMARQUE : Un imageur disponible dans le commerce équipé d’un objectif Plan Aprochromat 63x/1.4, d’un filtre GFP (11525314), d’une caméra GT DFC9000 et d’une source lumineuse LED5 a été utilisé, et un logiciel de microscope compatible a été utilisé. - Optimisez les paramètres d’imagerie pour chaque microscope et configuration expérimentale.
REMARQUE : Cependant, comme point de départ, les paramètres d’imagerie avec l’imageur utilisé dans ce manuscrit sont les suivants : Les dimensions sont de 2048 (x) * 2048 (y) pixels à une taille de pas de 0,50 μm pour les mitochondries hypodermiques et « System Optimized » calculé automatiquement par le logiciel pour les mitochondries musculaires. Le temps d’exposition est de 50 ms pour les mitochondries musculaires et de 50 ms pour les mitochondries hypodermiques. La LED de 475 nm a été réglée sur une puissance de 50 % et le filtre à densité neutre a été réglé sur 30 % pour les mitochondries musculaires et 17 % pour les mitochondries hypodermiques. La plage Z commence à partir de l’endroit où le signal GFP est visible jusqu’à l’endroit où il se termine. Pour minimiser le bruit dans une projection max, on peut diminuer le nombre de plans z utilisés pour la projection max. - Pour l’imagerie des mitochondries intestinales (vha-6p ::MLS ::GFP), utilisez un microscope confocal.
REMARQUE : L’imagerie a été réalisée sur un microscope confocal disponible dans le commerce équipé d’un objectif Plan ApoChromat 63x/1,4, d’un laser à lumière blanche (WLL), d’un séparateur de faisceau acousto-optique, de détecteurs HyD S et d’un logiciel de microscope. Comme mentionné précédemment, il convient d’optimiser chaque configuration expérimentale individuelle, mais comme point de départ, les paramètres d’imagerie avec le Stellaris utilisé dans cette étude sont les suivants : La CMU a été réglée à 85,00 % de puissance maximale et avec une ligne laser de 485 nm à une intensité de 3,00 %. Le détecteur HyD S a été réglé sur 490-590 nm avec un gain de 25 en mode analogique. Nous avons balayé de manière unidirectionnelle une zone de 1024 (x) * 1024 (y) ou 82,01 μm (x) * 82,01 μm (y) avec 5 (z) pixels à une taille de pas de 0,495 μm (la dimension z est variable en fonction de la taille des vers ou des tissus) avec une vitesse de balayage de 1 000 Hz, un zoom de 2,25, une moyenne de ligne de 2 et un sténopé de 1 UA (95,5 μm). La plage Z commence à partir de l’endroit où le signal GFP est visible jusqu’à l’endroit où il se termine. Pour minimiser le bruit dans une projection max, on peut diminuer le nombre de plans z utilisés pour la projection max.
- Utilisez un microscope standard à grand champ pour les mitochondries musculaires et hypodermiques (myo-3p ::MLS ::GFP et col-19p ::MLS ::GFP).
- Quantification de la morphologie mitochondriale
REMARQUE : Pour plus de détails, reportez-vous à Schindelin et al.22 et à la figure supplémentaire 1A-D.- Téléchargez et installez le logiciel FIJI (Fiji n’est que ImageJ, https://fiji.sc/). Ouvrez ensuite FIJI.
- Pour télécharger et installer la macro MitoMAPR, téléchargez le code source 1 à partir de https://doi.org/10.7554/eLife.49158.033. Copiez ensuite tout le code mentionné sous "A.). Code IJM pour MitoMAPR-1.0. Ensuite, ouvrez Fidji, allez dans Plugins > New > Macro, et collez le code précédemment copié dans la fenêtre de macro. Allez dans l’option Fichier > Enregistrer sous et enregistrez la macro pour une utilisation ultérieure.
- Ouvrez une image microscopique 3D avec des piles Z dans FIJI en glissant et en déposant des fichiers dans la barre d’outils Fiji ou en allant dans Fichier > Ouvrir.
- Créez une projection maximale de l’image en accédant à Piles d’images > > Projet Z, puis en sélectionnant la plage de tranches avec des images nettes à projeter au maximum et en sélectionnant Intensité maximale comme type de projection.
- Enregistrez l’image au format TIFF en accédant à Fichier > Enregistrer sous > Tiff.
- Recadrez les zones d’intérêt (ROI) à partir d’images complètes à l’aide de l’outil « Rectangle », en dessinant un retour sur investissement, puis en accédant à Image > Recadrage.
- Enregistrez l’image au format TIFF en accédant à Fichier > Enregistrer sous > Tiff.
- Exécutez la macro MitoMAPR précédemment enregistrée en glissant et déposant le fichier de macro dans la barre d’outils Fidji, puis cliquez sur Exécuter. Une nouvelle fenêtre vous demandera de sélectionner le dossier dans lequel le fichier Tiff a été enregistré dans la version 2.3.6.
- Une nouvelle fenêtre vous demandera de sélectionner une zone ; créez un rectangle dans l’image à l’aide de l’outil Rectangle comme mentionné précédemment à l’étape 2.3.4 et appuyez sur Ok.
REMARQUE : L’image entière peut être sélectionnée si elle a été recadrée précédemment comme décrit à l’étape 2.3.6. Enregistrez la fenêtre « Données » avec toutes les valeurs liées à la morphologie mitochondriale sous forme de fichier Excel en allant dans Fichier > Enregistrer sous > Enregistrer.
- Traitement par lots d’échantillons à l’aide de macros FIJI
- Créez une macro pour spécifier la région des images à analyser. Ouvrez Fiji et allez dans Plugins > New > Macro. Collez le code suivant dans la zone de texte et enregistrez la macro comme décrit à la section 2.3.2 (Figure supplémentaire 1E-H).
REMARQUE : Faites un rectangle (100, 100, 200, 200) ; run (Spécifier..., largeur = 100, hauteur = 100, x = 100, y = 100 à l’échelle). - Créez une macro pour enregistrer toutes les images ouvertes sous forme de fichiers TIFF. Dans la fenêtre de macro, accédez à Fichier > Nouveau et collez le code suivant dans la zone de texte. Enregistrez la macro comme décrit à la section 2.3.2.
REMARQUE : dir = getDirectory(« Sélectionner un dossier ») ; ids=newArray(nImages) ; pour (i=0 ; i - Créez une macro pour le lot MitoMAPR. Allez à https://doi.org/10.7554/eLife.49158.033 code source 1 et copiez tous les codes mentionnés sous B.). Code IJM pour MitoMAPR-1.0_Batch". Dans la fenêtre de macro, accédez à Fichier > Nouveau, collez le code du code source 1 dans la zone de texte et enregistrez la macro comme décrit à l’étape 2.3.2.
- Créez une macro pour le recadrage itératif. Allez à https://doi.org/10.7554/eLife.49158.033 code source 1 et copiez tous les codes mentionnés sous C.) Code IJM pour CropR. Dans la fenêtre de macro, allez dans Fichier > Nouveau, collez le code du code source 1 dans la zone de texte et enregistrez la macro comme décrit à l’étape 2.3.2.
- Pour commencer le traitement par lots, enregistrez d’abord toutes les images à traiter au format TIFF en ouvrant toutes les images et en exécutant la macro enregistrée à l’étape 2.4.2. Toutes les images seront enregistrées au format TIFF dans le dossier spécifié. Fermez toutes les images une fois enregistrées.
- Si les images enregistrées à l’étape 2.4.5 sont des images 3D (images contenant des piles z), convertissez-les en images 2D en les transformant en projections Z. Pour projeter les images en Z par lots, ouvrez la fenêtre de traitement par lots en accédant à Traiter > Lot > Macro dans la fenêtre principale de FIJI.
- Pour « Entrée », sélectionnez l’emplacement du dossier contenant toutes les images 3D à traiter à partir de la version 2.4.5. Pour « Sortie », sélectionnez l’emplacement souhaité pour les images enregistrées après le traitement. Sélectionnez le format de sortie TIFF. Collez le code suivant dans la grande zone de texte de la fenêtre et appuyez sur le bouton de processus : run(« Z Project... », « projection=[Intensité maximale] »).
- Recadrez les zones d’intérêt avec des dimensions identiques à partir de toutes les images 2D à traiter à l’aide de la macro de recadrage itérative de l’étape 2.4.4 en combinaison avec la macro de région de spécification de l’étape 2.4.3. Exécutez la macro de recadrage itératif à partir de l’étape 2.4.4. Une fenêtre s’affiche pour vous demander de lire un répertoire.
- Sélectionnez le dossier contenant uniquement les images 2D de l’étape 2.4.5 ou les images 2D projetées en Z de l’étape 2.4.6 et appuyez sur le bouton Sélectionner . La macro ouvrira l’une des images du dossier sélectionné, et une fenêtre intitulée « faire une sélection » avec deux boutons apparaîtra.
- Exécutez la macro de sélection spécifiée à partir de l’étape 2.4.3. Modifiez les valeurs de largeur et de hauteur dans cette zone de texte de macro selon vos besoins pour modifier la dimension de la sélection et appuyez sur Exécuter pour observer la nouvelle sélection. La modification des valeurs X et Y dans la macro modifiera la position du coin supérieur gauche de la sélection.
- Une fois que les dimensions de sélection sont satisfaisantes, faites glisser le rectangle jusqu’à la zone souhaitée, puis appuyez sur le bouton OK de la fenêtre « faire une sélection ». La région recadrée sera enregistrée dans le dossier sélectionné à l’étape 2.4.7.
- La macro parcourra toutes les images restantes du dossier. Pour chaque image, exécutez la macro à partir de l’étape 2.4.8 sans modifier les valeurs de dimension pour vous assurer que les mêmes dimensions sont recadrées pour toutes les images. Pour chaque image, faites glisser le rectangle de sélection à l’endroit souhaité et appuyez sur le bouton OK dans la fenêtre « faire une sélection ».
- Ouvrez le dossier contenant les images recadrées des étapes 2.4.8-2.4.9 et déplacez toutes les images recadrées dans un nouveau dossier.
- Analysez toutes les images recadrées en exécutant la macro MitoMAPR par lots à partir de l’étape 2.4.3. Une nouvelle fenêtre vous demandera de sélectionner un répertoire. Sélectionnez le dossier contenant toutes les images recadrées à l’étape 2.4.10. Une fois terminé, une fenêtre nommée « Données » apparaîtra avec toutes les valeurs liées à la morphologie mitochondriale. Enregistrez toutes les données sous forme de fichier Excel comme décrit à l’étape 2.3.9.
- Créez une macro pour spécifier la région des images à analyser. Ouvrez Fiji et allez dans Plugins > New > Macro. Collez le code suivant dans la zone de texte et enregistrez la macro comme décrit à la section 2.3.2 (Figure supplémentaire 1E-H).
Résultats
C. elegans est un excellent modèle pour l’imagerie mitochondriale en raison de son corps transparent, qui permet d’imager facilement les vers entiers et les animaux vivants sans préparation excessive des échantillons. De plus, la morphologie mitochondriale dans différents tissus peut être facilement visualisée en utilisant des promoteurs spécifiques aux tissus pour exprimer des protéines fluorescentes ciblées sur les mitochondries. Ici, myo-3p (pour les mitochondries musculaires), vha-6p (pour les mitochondries intestinales) et col-19p (pour les mitochondries hypodermiques) ont été utilisés pour stimuler l’expression d’une GFP (séquence de localisation mitochondriale de la protéine ATP-1) ciblée sur les mitochondries. Les mitochondries des muscles de la paroi corporelle de C. elegans présentent une morphologie tubulaire, s’alignant le long des fibres musculaires (myofibrilles) (Figure 1A) ; les mitochondries intestinales présentaient des structures fortement interconnectées, en forme de toile, avec un alignement moins uniforme (Figure 1B) ; et l’hypoderme a des mitochondries tubulaires qui semblent plus arrondies ou de forme ovale par rapport à l’intestin ou au muscle (Figure 1C). La morphologie mitochondriale est généralement cohérente sur toute la longueur du ver dans le muscle et l’intestin, mais l’hypoderme présente quelques différences mineures dans l’interconnexion des mitochondries entre les extrémités proximale et distale du ver. Ainsi, pour obtenir des résultats expérimentaux reproductibles, il est recommandé de se concentrer sur une région spécifique de leur corps. Ici, la zone entre le pharynx et la vulve ~100-200 μm sous le pharynx est systématiquement imagée.
Pour les mitochondries musculaires et hypodermiques, leur imagerie au microscope composé offre une résolution suffisante en raison de la planéité des cellules et des faibles niveaux de lumière floue. Cependant, les mitochondries intestinales sont difficiles à observer avec un microscope composé, car le grand volume de l’intestin limite la résolution en raison d’une grande quantité de lumière floue provenant d’autres sections de l’intestin (Figure 2). Par conséquent, il est recommandé d’imager les mitochondries intestinales sous un microscope confocal pour réduire la lumière floue et visualiser la morphologie mitochondriale appropriée.
La forme des mitochondries est très dynamique et change en fonction de l’environnement métabolique des animaux23 ou même en raison de l’exposition à des rigidités variables du substrat24. Par conséquent, laisser des animaux sur des lames de microscope pendant de longues périodes en l’absence d’une source de nourriture et sur le substrat rigide du verre peut potentiellement avoir un impact sur la morphologie mitochondriale. Ici, cette étude a révélé que les mitochondries des vers se fragmentent après environ 30 minutes sur une lame dans une solution M9, les mitochondries hypodermiques présentant le plus de fragmentation (Figure 3). Par conséquent, l’imagerie des mitochondries doit être effectuée rapidement après la préparation de l’échantillon.
Les produits chimiques qui limitent la mobilité des vers, notamment l’azoture de sodium et le tétramisole, sont couramment utilisés pour l’imagerie en direct de C. elegans, car les vers stationnaires sont nécessaires pour capturer plusieurs sections en Z d’animaux pour l’imagerie 3D. Des études antérieures ont montré que l’exposition à l’azoture de sodium ou au tétramisole peut entraîner une fragmentation mitochondriale25. Étonnamment, il a été constaté que l’azoture de sodium - même à des concentrations élevées - avait un impact limité sur la morphologie mitochondriale dans le muscle ou l’intestin. Cependant, l’hypoderme présentait une fragmentation mitochondriale plus précoce que les témoins M9 (Figure 3). Il est important de noter que des concentrations élevées de tétramisole (100 mM) ont entraîné une fragmentation mitochondriale significative immédiatement après l’exposition dans tous les types de cellules. En comparaison, des concentrations moyennes (10 mM) ont entraîné une fragmentation plus rapide par rapport à un témoin M9. De faibles concentrations (1 mM) ont eu un impact limité sur la morphologie mitochondriale. Ces données suggèrent que l’utilisation de l’azoture de sodium peut être une option viable pour l’imagerie rapide des mitochondries, tandis que le tétramisole doit généralement être évité.
Comme prévu, les mitochondries de tous les tissus de C. elegans présentent une fragmentation au cours du processus de vieillissement naturel (Figure 4A). La fragmentation peut être visualisée comme des mitochondries se présentant sous la forme de structures plus tronquées et sphériques, qui sont radicalement différentes des mitochondries linéaires et tubulaires observées chez les jeunes animaux. Il est important de noter que l’intestin augmente dans les structures autofluorescentes sphériques à un âge plus avancé, il faut donc veiller à ne pas confondre l’autofluorescence avec les structures mitochondriales réelles. Étant donné qu’il peut y avoir une variabilité des changements dans la structure mitochondriale d’un vers à l’autre, il est important d’effectuer une quantification de la morphologie mitochondriale sur un échantillon de taille significative plutôt que de simplement imager quelques vers. Ici, mitoMAPR a été utilisé, qui permet une quantification automatisée de la morphologie mitochondriale à l’aide d’un ensemble diversifié de mesures, y compris les objets, les réseaux, les jonctions par réseau, les points de jonction, la longueur de l’objet, l’empreinte mitochondriale, la couverture mitochondriale et la surface de l’objet (tableau supplémentaire 1 et tableau supplémentaire 2). L’automatisation élimine les biais subjectifs de l’utilisateur. Ici, nous rapportons que la métrique de la longueur de l’objet est optimale pour mesurer quantitativement les changements dans la morphologie mitochondriale musculaire et intestinale au cours du vieillissement, et les mesures des points de jonction pour mesurer les changements dans la morphologie mitochondriale hypodermique au cours du vieillissement (Figure 4B).
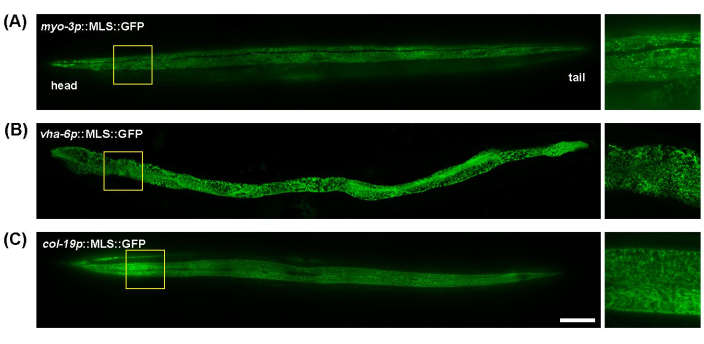
Figure 1 : Images de mitochondries dans tout le corps de C. elegans. L’imagerie mitochondriale a été réalisée pendant toute la durée du jour 5 chez des animaux adultes élevés sous EV à partir du stade L1 chez des animaux transgéniques avec une expression MLS ::GFP dans le muscle (A), l’intestin (B) et l’hypoderme (C). Barre d’échelle : 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
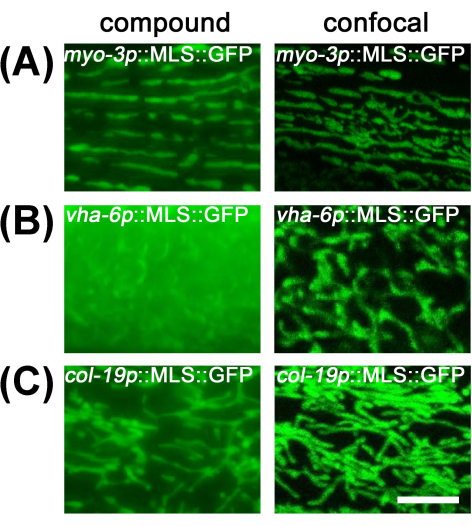
Figure 2 : Comparaison de l’imagerie mitochondriale entre le microscope composé et le microscope confocal. L’imagerie mitochondriale a été réalisée chez des animaux adultes du jour 1 cultivés sous EV à partir du stade L1 chez des animaux transgéniques avec une expression de MLS ::GFP dans le muscle (A), l’intestin (B) et l’hypoderme (C). Barre d’échelle : 5 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
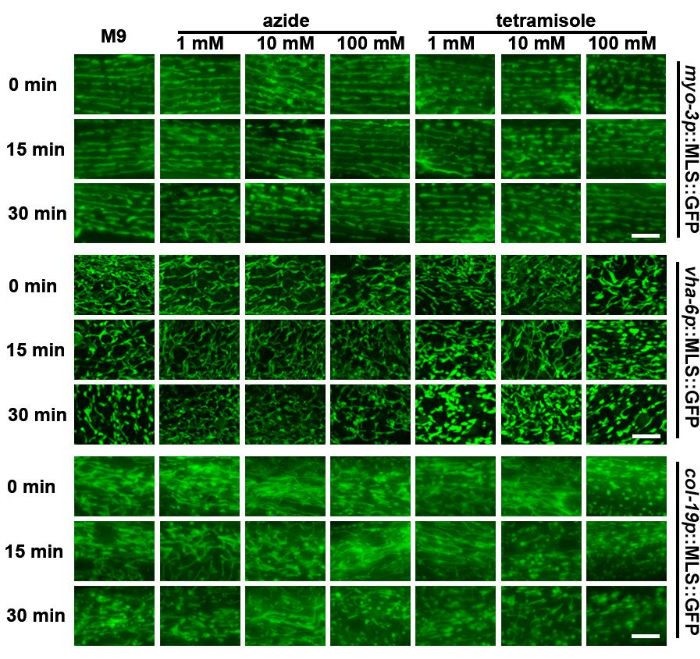
Figure 3 : Évaluation de la fragmentation mitochondriale de différents tissus après traitement avec M9, azoture de sodium et tétramisole. L’imagerie mitochondriale a été réalisée chez des animaux adultes du jour 1 avec expression de MLS ::GFP dans le muscle, l’intestin et l’hypoderme. Les animaux ont été élevés sur EV à partir du stade L1. Les animaux ont été placés sur des lames contenant du M9, de l’azoture de sodium (1 mM, 10 mM et 100 mM) ou du tétramisole (1 mM, 10 mM et 100 mM) et l’imagerie a été réalisée immédiatement après la préparation de la lame (0 min) ou 15 min ou 30 min après la préparation de la lame. Des images représentatives sont montrées pour n > 5 animaux par souche pour 2 répétitions biologiques. Barres d’échelle : 5 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
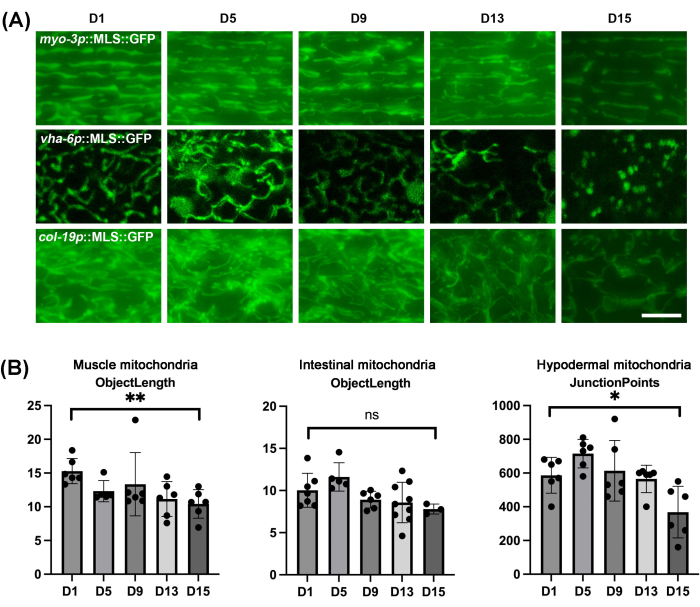
Figure 4 : Imagerie mitochondriale de C. elegans au cours du vieillissement dans différents tissus et quantification de la morphologie mitochondriale à l’aide de MitoMAPR. (A) L’imagerie mitochondriale a été réalisée chez des animaux adultes aux jours 1, 5, 9, 13 et 15 de l’âge adulte avec expression de MLS ::GFP dans le muscle, l’intestin et l’hypoderme. Les animaux ont été élevés sur EV à partir du stade L1 et déplacés sur des plaques EV contenant du FUDR à partir du stade adulte du jour 1. Les images sont représentatives de n ≥ 5 animaux par souche pour ≥ 3 répétitions biologiques. Barre d’échelle : 5 μm. (B) La quantification de la longueur de l’objet des mitochondries musculaires et intestinales des vers les jours 1, 5, 9, 13 et 15, et la quantification des points de jonction des mitochondries hypodermiques des vers les jours 1, 5, 9, 13 et 15. Tous les points de données individuels combinés à la moyenne ± à l’écart-type. Les graphiques ont été tracés et analysés statistiquement à l’aide d’un test t d’étudiant. ns = non significatif, *p < 0,03 ; **p < 0,002 ; p < 0,0002 ; p < 0,0001. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Jours de vers | Nombre de vers | Volume de tampon (mL) |
| 1 | 25 | 13 |
| 5 | 25 | 14 |
| 9 | 20 | 15 |
| 13 | 20 | 15 |
Tableau 1 : Nombre recommandé de vers et volumes de mémoire tampon lors de la préparation des lames.
Figure supplémentaire 1 : Flux de travail de l’application d’une quantification d’image unique à l’aide de MitoMAPR et du traitement par lots pour plusieurs images. (A) Flux de travail de quantification de la morphologie mitochondriale à partir d’une seule image à l’aide de la macro MitoMAPR. (B) Projection Z d’un fichier d’image 3D brut à l’aide de Fidji (projection Max). (C) Recadrage de la région d’intérêt à partir de l’image projetée Z. (D) Images de Skeletor acquises à partir de la macro MitoMAPR. Barres d’échelle : 5 μm. (E) Flux de travail de quantification de la morphologie mitochondriale à partir de plusieurs images à l’aide du traitement par lots à l’aide de la macro MitoMAPR. (F) Capture d’écran de la macro de recadrage. (G) Capture d’écran d’un processus par lots pour la projection Z. (H) Capture d’écran de la fenêtre de confirmation de sélection de la macro de recadrage par lots. Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire 1 : Analyse quantitative des paramètres morphologiques mitochondriaux dans le tissu musculaire de C. elegans . Les mesures enregistrées incluent les objets, les réseaux, les jonctions par réseau, les points de jonction, la longueur de l’objet, l’empreinte mitochondriale, la surface de l’objet et la couverture mitochondriale, mesurées sur différents jours. Les données fournissent une base pour évaluer les changements de la morphologie mitochondriale avec le vieillissement. Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire 2 : Effets des produits chimiques immobilisants sur la morphologie mitochondriale chez C. elegans. Les données comprennent des mesures telles que les objets, les réseaux, les jonctions par réseau, les points de jonction, la longueur de l’objet, l’empreinte mitochondriale, la surface de l’objet et la couverture mitochondriale à des concentrations et des points temporels variables après la préparation de l’échantillon avec un tampon M9 ou différentes concentrations de tétramisole et d’azoture de sodium. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
L’imagerie fluorescente de la morphologie mitochondriale est le moyen le plus courant de déterminer les changements dans les mitochondries. Alors que les techniques de microscopie avancées telles que la microscopie électronique à transmission (MET), la microscopie à force atomique et la cryo-microscopie électronique offrent une résolution plus élevée, la microscopie à fluorescence reste plus abordable et accessible à la plupart des chercheurs. De plus, la microscopie fluorescente peut être réalisée dans des cellules vivantes, et dans des organismes modèles clairs comme C. elegans, l’imagerie peut être réalisée sur des animaux entiers26,27. L’imagerie de C. elegans transgéniques est assez simple, et les marqueurs fluorescents génétiquement codés permettent une imagerie plus fiable et plus robuste des mitochondries car elle ne nécessite pas de traitement complexe d’échantillons ou ne souffre pas de coloration variable entre les types de cellules ou les conditions des colorants mitochondriaux conventionnels comme MitoTracker ou TMRE 28,29,30. Les marqueurs fluorescents génétiquement codés impliquent généralement le marquage direct des protéines mitochondriales avec des fluorophores ou la conjugaison de fluorophores avec une séquence de localisation mitochondriale minimale. Ces constructions sont souvent pilotées par des promoteurs spécifiques aux tissus, permettant de visualiser les mitochondries dans différents tissus comme les muscles, l’intestin ou les tissus de l’hypoderme31. En général, ces protéines marquées par fluorescence sont surexprimées, ce qui peut avoir des effets physiologiques potentiellement indésirables si les protéines mitochondriales de pleine longueur sont surexprimées ; ainsi, les séquences MLS minimales sont une meilleure option32. Cependant, même avec la surexpression de fusions minimes de protéines fluorescentes MLS, une surexpression élevée doit être évitée, car l’importation d’une grande quantité de protéines dans les mitochondries peut réduire le potentiel de la membrane mitochondriale et avoir un impact sur la santé animale33. Bien qu’une caractérisation exhaustive de toutes les souches de C. elegans actuellement disponibles dépasse le cadre de ce manuscrit, une analyse comparative détaillée de nombreux rapporteurs mitochondriaux et des avantages et inconvénients de chacun peut être trouvée ici31.
Pour l’imagerie de cellules vivantes de mitochondries chez C. elegans, la microscopie composée standard ou à champ large peut être une option privilégiée en raison de la vitesse élevée et de la facilité de ces méthodes par rapport à la microscopie confocale. Dans cette étude, il est montré que les cellules plates comme le muscle et l’hypoderme bénéficient de manière minimale de la microscopie confocale, et que la microscopie composée permet une acquisition avec une résolution suffisante pour visualiser la morphologie mitochondriale appropriée. Les cellules plus grandes comme l’intestin rendent la microscopie composée difficile en raison de la lumière floue. Par conséquent, la microscopie confocale est nécessaire pour une imagerie fiable de la morphologie mitochondriale intestinale.
Une considération importante pour l’imagerie 3D sur toute l’épaisseur des tissus chez les animaux vivants est d’empêcher le mouvement des vers lors de l’acquisition d’images. Les chercheurs utilisent souvent des méthodes pour paralyser les vers, telles que le tétramisole ou l’azoture de sodium34. L’azoture de sodium inhibe la cytochrome c oxydase (complexe IV), une enzyme essentielle dans la chaîne de transport d’électrons des mitochondries, entraînant une paralysie générale due au manque d’ATP nécessaire aux contractions musculaires et à d’autres fonctions cellulaires34,35. Le tétramisole agit en imitant l’acétylcholine au niveau des jonctions neuromusculaires, provoquant une dépolarisation persistante et une contraction musculaire36. Cependant, l’exposition à ces médicaments peut induire une fragmentation mitochondriale en provoquant un stress oxydatif23. Dans cette étude, il a été constaté que le tétramisole induisait très rapidement la fragmentation mitochondriale, mais que l’azoture de sodium avait un effet beaucoup plus limité.
C. elegans offre un moyen très simple et facile d’étudier l’impact du vieillissement sur la morphologie mitochondriale en raison de sa courte durée de vie et de la facilité avec laquelle les animaux vieillissent. Ici, nous avons choisi d’utiliser l’exposition au FUDR, qui est une méthode robuste pour stériliser chimiquement les animaux en empêchant la réplication de l’ADN 20,37,38. Cependant, le FUDR peut avoir des effets indésirables sur des paramètres de vieillissement spécifiques, et pour ceux qui s’inquiètent des effets hors cible du FUDR, d’autres stratégies pour éliminer la descendance peuvent être utilisées39. Par exemple, il existe des mutants stériles, y compris le mutant germinal glp-4 sensible à la température, ou des mutants déficients en spermatozoïdes, tels que CF512 40,41,42. Comme alternative, les animaux peuvent également être naturellement vieillis en choisissant manuellement des adultes de leur progéniture quotidiennement.
La quantification des changements dans la morphologie mitochondriale est également une considération très importante, car il peut y avoir une variabilité significative de la morphologie mitochondriale entre les animaux. Par conséquent, il est essentiel d’effectuer des analyses quantitatives et des statistiques sur un échantillon de taille suffisante pour tirer des conclusions significatives sur les changements de la morphologie mitochondriale. Cependant, l’analyse et la quantification d’images peuvent grandement souffrir des biais subjectifs et des défis liés à l’imagerie de structures très complexes comme les mitochondries interconnectées. À cette fin, voici un résumé d’une méthode automatisée de quantification de la morphologie mitochondriale à l’aide de nombreuses métriques développées par le laboratoire Mair. MitoMAPR permet une quantification objective et automatisée de la morphologie mitochondriale, en mesurant divers aspects des mitochondries, notamment le réseau mitochondrial, la longueur de l’objet, la distribution, la couverture du réseau et l’empreinte mitochondriale. MitoMAPR est une macro gratuite pour ImageJ et est donc disponible pour tous les laboratoires disposant d’un ordinateur fonctionnel. Un aspect important de l’utilisation de MitoMAPR est d’effectuer une quantification sur un échantillon de grande taille afin de déterminer quelle métrique de la morphologie mitochondriale est la plus robuste pour déterminer les changements dans les conditions expérimentales testées43,44. Ici, on constate que la longueur de l’objet et les points de jonction sont les meilleures mesures pour déterminer les changements au cours du vieillissement dans les muscles, l’intestin et l’hypoderme. Une autre approche pour analyser les changements dans la morphologie mitochondriale est la génération de représentations 3D des mitochondries à partir d’images z-stack, suivie d’une analyse 3D16,45. Cela peut être réalisé à l’aide de logiciels disponibles dans le commerce, tels que Image-Pro Plus avec les modules SharpStack Total Deconvolution et 3D Constructor. Il a été démontré que la quantification des représentations mitochondriales 3D fournit des informations plus précises sur la forme mitochondriale et les propriétés du réseau46. Cependant, cette méthode est techniquement plus complexe et plus coûteuse que l’approche semi-3D, qui consiste à réduire plusieurs sections d’image d’un empilement z à grand champ ou confocaux en une seule projection 2D et à les analyser avec des outils tels que mitoMAPR. Cette étude résume l’utilisation de l’expression en copie unique de la GFP ciblée sur la matrice mitochondriale et détaille quelques pièges à éviter lors de l’imagerie.
Limites et considérations temporelles
Bien que la microscopie confocale soit recommandée pour les tissus larges et épais, tels que les tissus intestinaux, les techniques standard de microscopie confocale à balayage linéaire peuvent être trop lentes pour effectuer une imagerie rapide des mitochondries. C’est particulièrement vrai compte tenu de nos données qui montrent que le fait de garder des vers sur des lames pendant de longues périodes peut entraîner la fragmentation des mitochondries. Bien que la technologie de microscopie confocale ait considérablement progressé, les microscopes capables d’effectuer une imagerie rapide, tels que le disque rotatif, Airyscan ou celui utilisé dans cette étude, peuvent être prohibitifs pour certains laboratoires. Dans ces cas, la microscopie composée peut être combinée à des méthodes de clairance computationnelle pour supprimer la lumière floue, telles que la déconvolution47.
De plus, comme décrit, MitoMAPR est une puissante macro automatisée permettant de mesurer quantitativement la longueur des mitochondries et l’interconnexion du réseau mitochondrial. Cependant, il doit être utilisé avec prudence en tenant compte des limitations énumérées ici. Tout d’abord, l’utilisation d’une GFP ciblée sur la matrice n’observe que la fragmentation mitochondriale de la membrane interne, qui peut ne pas récapituler complètement la morphologie mitochondriale de la membrane externe, car des événements de fission de la membrane interne peuvent se produire en l’absence de fission de la membrane externe. Ainsi, pour une imagerie plus fine des deux membranes, un fluorophore ciblé sur la matrice et un fluorophore ciblé sur la membrane externe mitochondriale doivent être utilisés. Étant donné que les marqueurs de la membrane mitochondriale externe peuvent subir les mêmes conséquences que les protéines localisées dans la matrice s’ils sont fortement surexprimés, l’utilisation de marqueurs à copie unique est recommandée, en particulier ceux utilisant des séquences de localisation mitochondriale minimales au lieu de protéines complètes comme celles utilisées ici31.
Comme décrit dans la figure 3, les mitochondries peuvent se fragmenter rapidement au microscope, et différentes méthodes de préparation des lames d’échantillon peuvent largement affecter la morphologie en fonction du tampon utilisé. Les données représentatives décrites dans ce manuscrit montrent que les mitochondries des vers subissent une fragmentation minimale sous M9 et de faibles concentrations d’azoture de sodium. Le tétramisole, un autre produit chimique largement utilisé pour paralyser les vers, fragmente rapidement les mitochondries, indiquant que son utilisation doit être généralement évitée. Bien que M9 et l’azoture de sodium n’aient pas montré de fragmentation significative des mitochondries, il est important de noter que différentes souches pourraient réagir différemment des résultats présentés ici. Bien que d’autres études aient montré que l’azoture de sodium peut fragmenter les mitochondries, il est possible que nos souches ne présentent pas de fragmentation mitochondriale significative lors de l’exposition à l’azoture de sodium en raison du faible niveau d’expression de nos constructions. Une expression élevée de protéines localisées dans les mitochondries peut réduire le potentiel membranaire et, par conséquent, différentes souches utilisées dans d’autres études peuvent rendre les mitochondries plus sensibles à la fragmentation à partir des mêmes concentrations d’azoture de sodium utilisées dans cette étude. Quoi qu’il en soit, il faut veiller à ce que les produits chimiques qui paralysent les vers ne provoquent pas de fragmentation mitochondriale dans des conditions spécifiques à tester avant d’être utilisées dans toutes les études, car des mutants ou des conditions spécifiques peuvent être plus sensibles à la fragmentation mitochondriale induite par le médicament. De plus, même le fait de garder les vers dans M9 peut entraîner des modifications de leur morphologie et de leur activité mitochondriales naturelles, car il a déjà été démontré que leur activité de nage dans le tampon M9 affecte la fission mitochondriale et la dynamique de fusion. De plus, laisser les vers dans M9 pendant de longues périodes peut activer des réponses d’hypoxie qui affectent considérablement les mitochondries en provoquant des perturbations de la protéostasie mitochondriale48,49.
Enfin, une autre considération importante est que, bien que les modifications de la morphologie mitochondriale soient souvent corrélées aux modifications de la fonction mitochondriale, il n’y a pas toujours de corrélation directe entre les deux. Ainsi, une analyse plus approfondie de la fonction mitochondriale est recommandée. Par exemple, le taux de consommation d’oxygène peut être mesuré à l’aide d’un instrument Seahorse50, le potentiel de la membrane mitochondriale peut être mesuré à l’aide de colorants de potentiel membranaire tels que JC9 ou TMRE51, l’état oxydatif mitochondrial peut être mesuré à l’aide de colorants sensibles à l’oxydoréduction tels que roGFP52, et la résilience au stress mitochondrial peut être mesurée à l’aide de la sensibilité aux facteurs de stress tels que la roténone53. Parce que l’imagerie des mitochondries peut être assez rapide, nous proposons ici les méthodes comme un premier passage facile pour déterminer si les conditions expérimentales affectent la morphologie mitochondriale. Ces méthodes se prêtent même au criblage à grande échelle de médicaments ou de gènes, avec un suivi d’une analyse mitochondriale plus approfondie à l’aide de mesures supplémentaires recommandées. Dans l’ensemble, nous passons en revue ici ce que l’on pense être les méthodes les plus simples pour imager la morphologie mitochondriale chez C. elegans avec un minimum d’erreurs expérimentales.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
J.K. est soutenu par la bourse du doyen de l’USC ; M.A. et G.G. sont soutenus par T32AG052374 ; M.V. est pris en charge par 1R25AG076400 ; et R.H.S. est soutenu par R01AG079806 du National Institute on Aging et 2022-A-010-SUP de la Fondation Larry L. Hillblom. Certaines souches ont été fournies par la CGC, qui est financée par la subvention P40 du Bureau des programmes d’infrastructure de recherche des NIH OD010440. Une analyse génétique a été effectuée à l’aide de Wormbase, qui est financé par une subvention U41 HG002223.
matériels
| Name | Company | Catalog Number | Comments |
| 5-fluoro-2'-deoxyuridine (FUDR) | Spectrum Chemical | F2026-10GMBL | for proliferation inhibition |
| APEX IPTG | Genesee | 18-242 | for RNAi |
| Bacto Agar | VWR | 90000-764 | for NGM plates |
| Bacto Peptone | VWR | 97064-330 | for NGM plates |
| Calcium chloride dihydrate | VWR | 97061-904 | for NGM plates |
| Carbenicillin | VWR | 76345-522 | for RNAi |
| Cholesterol | VWR | 80057-932 | for NGM plates |
| Confocal microscope | Stellaris 5 | ||
| DMSO | VWR | BDH1115-1LP | solvent for drugs |
| HistoBond microscope slides | VWR | 16005-110 | for slides preparation |
| LB Broth | VWR | 95020-778 | for LB |
| LEICA S7E Dissecting Scope | Leica | 10450840 | Standard dissecting microscope |
| LEICA STELLARIS 5 | Leica | 158101100 | Confocal Microscope |
| LEICA THUNDER | Leica | 11525679 | Compound Microscope |
| Magnesium sulfate heptahydrate | VWR | 97062-132 | for NGM plates, M9 |
| pL4440 empty vector plasmid | addgene | plasmid #1654 | for empty vector plasmid |
| Potassium Chloride | VWR | 97061-566 | for bleach soluton |
| Potassium phosphate dibasic | VWR | EM-PX1570-2 | for NGM plates |
| Potassium phosphate monobasic | VWR | EM-PX1565-5 | for M9 |
| Sodium azide | VWR | 97064-646 | for paralyzing worms |
| Sodium Chloride | VWR | EM-SX0420-5 | for NGM plates, M9 |
| Sodium hypochlorite | VWR | RC7495.7-32 | for bleach solution |
| Sodium phosphate dibasic | VWR | 71003-472 | for M9 |
| Tetracycline hydrochloride | VWR | 97061-638 | for RNAi |
| WOB-L® Dry Vacuum Pumps, Standard-Duty, Welch® | VWR | 80077-612 | for aspiration |
Références
- Protasoni, M., Zeviani, M. Mitochondrial structure and bioenergetics in normal and disease conditions. Int J Mol Sci. 22 (2), 586 (2021).
- Wang, Y., et al. The role of mitochondrial dynamics in disease. MedComm. 4 (6), e462 (2023).
- Kamer, K. J., Mootha, V. K. The molecular era of the mitochondrial calcium uniporter. Nat Rev Mol Cell Biol. 16 (9), 545-553 (2015).
- Chen, W., Zhao, H., Li, Y. Mitochondrial dynamics in health and disease: mechanisms and potential targets. Signal Transduct Target Ther. 8 (1), 333 (2023).
- Adebayo, M., Singh, S., Singh, A. P., Dasgupta, S. Mitochondrial fusion and fission: The fine-tune balance for cellular homeostasis. FASEB J. 35 (6), e21620 (2021).
- Madan, S., Uttekar, B., Chowdhary, S., Rikhy, R. Mitochondria lead the way: mitochondrial dynamics and function in cellular movements in development and disease. Front Cell Dev Biol. 9, 781933 (2021).
- Sharma, A., Smith, H. J., Yao, P., Mair, W. B. Causal roles of mitochondrial dynamics in longevity and healthy aging. EMBO Rep. 20 (12), e48395 (2019).
- Rolland, S. G., Lu, Y., David, C. N., Conradt, B. The BCL-2-like protein CED-9 of C. elegans promotes FZO-1/Mfn1,2- and EAT-3/Opa1-dependent mitochondrial fusion. J Cell Biol. 186 (4), 525-540 (2009).
- Labrousse, A. M., Zappaterra, M. D., Rube, D. A., van der Bliek, A. M. C. elegans dynamin-related protein DRP-1 controls severing of the mitochondrial outer membrane. Mol Cell. 4 (5), 815-826 (1999).
- Dickinson, D. J., Pani, A. M., Heppert, J. K., Higgins, C. D., Goldstein, B. Streamlined genome engineering with a self-excising drug selection cassette. Genetics. 200 (4), 1035-1049 (2015).
- Bosher, J. M., Labouesse, M. RNA interference: Genetic wand and genetic watchdog. Nat Cell Biol. 2 (2), E31-E36 (2000).
- Yu, C. C. J., et al. Expansion microscopy of C. elegans. eLife. 9, e46249 (2020).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Front Endocrinol. 11, 554994 (2020).
- Dingley, S., et al. Mitochondrial respiratory chain dysfunction variably increases oxidant stress in Caenorhabditis elegans. Mitochondrion. 10 (2), 125-136 (2010).
- Presley, A. D., Fuller, K. M., Arriaga, E. A. MitoTracker Green labeling of mitochondrial proteins and their subsequent analysis by capillary electrophoresis with laser-induced fluorescence detection. J Chromatogr B Anal Technol Biomed Life Sci. 793 (1), 141-150 (2003).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Curr Protoc Cell Biol. Chapter 4, Unit 4.25.1-Unit 4.25.21 (2010).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. Novel imaging tools to study mitochondrial morphology in Caenorhabditis elegans. Life Sci Alliance. 7 (11), e202402918 (2024).
- Benedetti, C., Haynes, C. M., Yang, Y., Harding, H. P., Ron, D. Ubiquitin-like protein 5 positively regulates chaperone gene expression in the mitochondrial unfolded protein response. Genetics. 174 (1), 229-239 (2006).
- Begelman, D. V., et al. An aco-2::gfp knock-in enables the monitoring of mitochondrial morphology throughout C. elegans lifespan. microPublication Biol. 2022, (2022).
- Castro Torres, T., et al. Surveying low-cost methods to measure lifespan and healthspan in Caenorhabditis elegans. J Vis Exp. 183, e64091 (2022).
- Rasmussen, N. R., Reiner, D. J. Nuclear translocation of the tagged endogenous MAPK MPK-1 denotes a subset of activation events in C. elegans development. J Cell Sci. 134 (17), jcs258456 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Luz, A. L., et al. Mitochondrial morphology and fundamental parameters of the mitochondrial respiratory chain are altered in Caenorhabditis elegans strains deficient in mitochondrial dynamics and homeostasis processes. PLoS One. 10 (6), e0130940 (2015).
- Oorloff, M., et al. Mechanical stress through growth on stiffer substrates impacts animal health and longevity in C. elegans. bioRxiv. , (2024).
- Chu, X., Wu, S., Raju, R. NLRX1 regulation following acute mitochondrial injury. Front Immunol. 10, 2431 (2019).
- Shah, P., Bao, Z., Zaidel-Bar, R. Visualizing and quantifying molecular and cellular processes in Caenorhabditis elegans using light microscopy. Genetics. 221 (4), iyac068 (2022).
- Wang, Y., Wang, P., Li, C. Fluorescence microscopic platforms imaging mitochondrial abnormalities in neurodegenerative diseases. Adv Drug Deliv Rev. 197, 114841 (2023).
- Ding, J., et al. An expanded GCaMP reporter toolkit for functional imaging in Caenorhabditis elegans. G3 (Bethesda, Md). 13 (10), jkad183 (2023).
- Sarasija, S., Norman, K. R. Analysis of mitochondrial structure in the body wall muscle of Caenorhabditis elegans. Bio-Protocol. 8 (7), e2801 (2018).
- Chaweeborisuit, P., Suriyonplengsaeng, C., Suphamungmee, W., Sobhon, P., Meemon, K. Nematicidal effect of plumbagin on Caenorhabditis elegans: A model for testing a nematicidal drug. Z Naturforsch C J Biosci. 71 (5-6), 121-131 (2016).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. . Novel imaging tools to study mitochondrial dynamics in Caenorhabditis elegans. 7, 11 (2024).
- Bolognesi, B., Lehner, B. Reaching the limit. eLife. 7, e39804 (2018).
- Jishi, A., Qi, X. Altered mitochondrial protein homeostasis and proteinopathies. Front Mol Neurosci. 15, 867935 (2022).
- Morton, K. S., Wahl, A. K., Meyer, J. N. The effect of common paralytic agents used for fluorescence imaging on redox tone and ATP levels in Caenorhabditis elegans. PLoS One. 19 (4), e0292415 (2024).
- Noumi, T., Maeda, M., Futai, M. Mode of inhibition of sodium azide on H+-ATPase of Escherichia coli. FEBS Lett. 213 (2), 381-384 (1987).
- Siete, C., Xiong, R., Khalid, A., Hsieh, Y. -. W., Chuang, C. -. F. Immobilization of C. elegans with different concentrations of an anesthetic for time-lapse imaging of dynamic protein trafficking in neurons. microPublication Biology. 2024, (2024).
- Mitchell, D. H., Stiles, J. W., Santelli, J., Sanadi, D. R. Synchronous growth and aging of Caenorhabditis elegans in the presence of fluorodeoxyuridine. J Gerontol. 34 (1), 28-36 (1979).
- Santi, D. V., McHenry, C. S. 5-Fluoro-2'-deoxyuridylate: covalent complex with thymidylate synthetase. Proc Natl Acad Sci U S A. 69 (7), 1855-1857 (1972).
- Wang, H., Zhao, Y., Zhang, Z. Age-dependent effects of floxuridine (FUdR) on senescent pathology and mortality in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun. 509 (3), 694-699 (2019).
- Beanan, M. J., Strome, S. Characterization of a germ-line proliferation mutation in C. elegans. Development (Cambridge, England). 116 (3), 755-766 (1992).
- Austin, J., Kimble, J. glp-1 is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans. Cell. 51 (4), 589-599 (1987).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Zhang, Y., et al. Neuronal TORC1 modulates longevity via AMPK and cell nonautonomous regulation of mitochondrial dynamics in C. elegans. eLife. 8, e49158 (2019).
- Dutta, N., et al. Investigating impacts of the mycothiazole chemotype as a chemical probe for the study of mitochondrial function and aging. GeroScience. 46 (6), 6009-6028 (2024).
- Tronstad, K., et al. Regulation and quantification of cellular mitochondrial morphology and content. Curr Pharm Des. 20 (35), 5634-5652 (2014).
- Nikolaisen, J., et al. Automated quantification and integrative analysis of 2D and 3D mitochondrial shape and network properties. PLoS One. 9 (7), e101365 (2014).
- Lee, J. S., Wee, T. L. E., Brown, C. M. Calibration of wide-field deconvolution microscopy for quantitative fluorescence imaging. J Biomol Tech. 25 (1), 31-40 (2014).
- Hong, M., Kwon, J. Y., Shim, J., Lee, J. Differential hypoxia response of hsp-16 genes in the nematode. J Mol Biol. 344 (2), 369-381 (2004).
- Yan, J., Sun, C. L., Shin, S., Van Gilst, M., Crowder, C. M. Effect of the mitochondrial unfolded protein response on hypoxic death and mitochondrial protein aggregation. Cell Death Dis. 12 (7), 711 (2021).
- Koopman, M., et al. A screening-based platform for the assessment of cellular respiration in Caenorhabditis elegans. Nat Protoc. 11 (10), 1798-1816 (2016).
- Shpilka, T., et al. UPRmt scales mitochondrial network expansion with protein synthesis via mitochondrial import in Caenorhabditis elegans. Nat Comm. 12 (1), 479 (2021).
- Braeckman, B. P., Smolders, A., Back, P., De Henau, S. In vivo detection of reactive oxygen species and redox status in Caenorhabditis elegans. antioxidants & redox signaling. 25 (10), 577-592 (2016).
- Bar-Ziv, R., et al. Measurements of physiological stress responses in C. elegans. J Vis Exp. 159, e61001 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon