Method Article
Bildgebung und Quantifizierung der mitochondrialen Morphologie bei C. elegans während des Alterns
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet einen standardisierten Ansatz für die Abbildung der mitochondrialen Morphologie in mehreren Geweben von C. elegans während des Alterns.
Zusammenfassung
Mitochondrien, wichtige zelluläre Organellen, die in den meisten eukaryotischen Zellen vorkommen, sind wichtige Orte der Energieerzeugung durch aerobe Atmung. Neben dieser bekannten Rolle als "zelluläres Kraftwerk" sind Mitochondrien auch an vielen anderen essentiellen zellulären Prozessen beteiligt, darunter die Regulierung des Zellstoffwechsels, der Proliferation, der Immunsignalisierung und der hormonellen Signalübertragung. Eine Verschlechterung der mitochondrialen Funktion während des Alterns oder unter mitochondrialem Stress ist oft durch deutliche Veränderungen der mitochondrialen Morphologie und des Volumens gekennzeichnet. Der Fadenwurm C. elegans ist aufgrund seines transparenten Körpers und seiner kurzen Lebensdauer, die die Live-Mikroskopie während seiner gesamten Lebensdauer erleichtern, ein ideales Modell für die Untersuchung dieser Veränderungen. Aber auch innerhalb des C . elegans-Feldes stehen zahlreiche transgene Konstrukte und Methoden für die mitochondriale Bildgebung zur Verfügung, von denen jedes seine eigenen Grenzen hat. In dieser Arbeit werden matrixlokalisierte GFP-Konstrukte mit einer einzigen Kopie als robuste und zuverlässige Methode zur Abbildung der mitochondrialen Morphologie in C. elegans vorgestellt. Diese Studie konzentriert sich speziell auf experimentell kontrollierbare Faktoren, um Fehler zu minimieren und die Variabilität zwischen Replikaten und zwischen Studien zu reduzieren, wenn mitochondriale Bildgebung während des Alterungsprozesses durchgeführt wird. Darüber hinaus wird mitoMAPR als robuste Methode zur Quantifizierung von Veränderungen der mitochondrialen Morphologie über Gewebetypen hinweg während des Alterns empfohlen.
Einleitung
Mitochondrien sind zelluläre Organellen, die von einer doppelten Phospholipidmembran umschlossen sind und eine wichtige Rolle bei der Aufrechterhaltung der zellulären Bioenergetik als Schlüsselstelle für den Stoffwechsel und die Energieproduktion spielen1. Eine große Anzahl von Mitochondrien erzeugt kontinuierlich Energie in Form von ATP, um den zellulären Bedarf zu decken2. Zusätzlich zu diesen wichtigen Rollen sind Mitochondrien auch an komplexen zellulären Prozessen wie Signaltransduktion, Autophagie, angeborener Immunität, Zellzyklus und Zelltodwegen beteiligt 2,3. Mitochondrien weisen unterschiedliche Morphologien auf, die von kleinen einzelnen Organellen bis hin zu ausgedehnten, miteinander verbundenen röhrenförmigen Netzwerken reichen, abhängig von unterschiedlichen energetischen Anforderungen und der Gesundheit der Mitochondrien4.
Ein wesentliches Merkmal der Mitochondrien ist ihre dynamische Natur, durch die sie durch eine eng koordinierte und kontinuierliche Abfolge von Spaltungs- und Fusionsereignissen zwischen diesen verschiedenen Formen wechseln können4. Das genaue Gleichgewicht zwischen diesen gegensätzlichen Prozessen reguliert die Morphologie, Anzahl, Größe und Positionierung der Mitochondrien innerhalb des Zytoplasmas5. Darüber hinaus sind diese Fusions- und Spaltungsprozesse wichtig für die Aufrechterhaltung der Qualitätskontrolle der Mitochondrien5. Zum Beispiel ist die mitochondriale Spaltung ein wesentlicher Bestandteil der Entfernung geschädigter Mitochondrien durch Mitophagie und der selektiven Entfernung von Mitochondrien durch Autophagie2. Die mitochondriale Dynamik spielt auch eine wichtige Rolle bei der Zellteilung, der Entwicklung, der Resistenz gegen verschiedene Stressoren und der Aufrechterhaltung des Zellstoffwechsels6.
Eine Störung der mitochondrialen Dynamik ist an zahlreichen Krankheiten beteiligt, darunter neurodegenerative Erkrankungen, Stoffwechselerkrankungen, Herz-Kreislauf-Erkrankungen und Krebs4. Darüber hinaus ist das Altern mit einem dramatischen Umbau der Mitochondrien verbunden, der hauptsächlich auf Veränderungen in dieser Fusions-Spaltungsdynamik zurückzuführenist 7. Daher bietet die Visualisierung und Überwachung der Veränderungen der mitochondrialen Morphologie unter verschiedenen Stress- oder Krankheitsbedingungen und während des gesamten Alterungsprozesses wertvolle Einblicke in das Verständnis der zellulären Funktion, des Krankheitsmechanismus und potenzieller therapeutischer Strategien.
Wie viele molekulare Signalwege sind auch die mitochondriale Dynamik und Funktion in Eukaryoten, einschließlich Modellorganismen wie dem Fadenwurm Caenorhabditis elegans, stark konserviert. Ähnlich wie bei menschlichen Zellen wird die mitochondriale Fusion in C. elegans durch die Funktion von Dynamin-verwandten Guanintriphosphatase (GTPase)-Proteinen erreicht, einschließlich FZO-1 (Ortholog des Säugetieres Mfn1/2) und EAT-3 (Ortholog des Säugetieres Opa1), die die Fusion der äußeren Mitochondrienmembran (OMM) bzw. der inneren Mitochondrienmembran (IMM) kontrollieren8. Die mitochondriale Spaltung wird durch das Dynamin-verwandte Protein (DRP-1, Ortholog des menschlichen Drp1) reguliert, das die mitochondriale Spaltung fördert, indem es ringförmige Komplexe um die mitochondriale Außenmembran bildet, die die mitochondrialen Membranen verengen und schließlich trennen9. Die mitochondriale Fusion spielt eine zentrale Rolle bei der mitochondrialen Qualitätskontrolle, indem sie das Mischen von mitochondrialen Inhalten, einschließlich mitochondrialer DNA, Proteine und Lipide, ermöglicht und die Komplementierung von teilweise geschädigten Mitochondrien durch Inhalte aus gesunden Mitochondrien ermöglicht9. Auf der anderen Seite ermöglicht die mitochondriale Spaltung den Mitochondrien, sich zu teilen, wodurch neue Mitochondrien entstehen und ihre Verteilung nicht nur innerhalb des Zytoplasmas, sondern auch zu den Tochterzellen während der Zellteilung erleichtert wird, wodurch eine ordnungsgemäße mitochondriale Vererbung und Funktion gewährleistetwird 9. Dieses Ereignis ist auch wichtig, um beschädigte oder dysfunktionale mitochondriale Segmente zu trennen, die dann durch Mitophagie abgebaut werden können9.
C. elegans gilt seit langem als eines der leistungsfähigsten genetischen Modellsysteme, da das Genom vollständig ist und eine Vielzahl genetischer Werkzeuge zur Verfügung steht, darunter CRISPR/Cas9, das genetische Veränderungen ermöglicht10, zahlreiche Methoden zur Überexpression von Genen und eine bakterielle Lebensmittelmethode zur RNA-Interferenz (RNAi)11. Darüber hinaus ermöglicht ihre transparente Anatomie mikroskopische Bildgebung in lebenden Organismen12. Schließlich machen sie ihre relativ kurze Lebensdauer, ihre geringen Wartungskosten und die einfache Herstellung einer großen Anzahl von Tieren mit gleichaltrigem Alter zu einem hervorragenden Modellsystem für die Alternsbiologie13. Diese Vorteile, kombiniert mit der bekannten Konservierung der wichtigsten mitochondrialen Regulationswege, machen C. elegans zu einem äußerst attraktiven Modellsystem für die Untersuchung der mitochondrialen Dynamik während des Alterns.
Fluoreszierende Marker werden in der biologischen Forschung häufig verwendet, um zelluläre Komponenten, einschließlich Mitochondrien, sichtbar zu machen und zu untersuchen. Es gibt spezifische zellpermeable Farbstoffe wie MitoTracker und Tetramethylrhodaminethylester (TMRE), die häufig verwendet werden14. Ersteres wird verwendet, um die gesamte negativ geladene mitochondriale Matrix15 zu färben, während letzteres verwendet wird, um das relative mitochondriale Membranpotential durch sein positiv geladenes Triphenylphosphonium-Ion16 zu bewerten. Sie profitieren zwar davon, dass sie keine Transgene benötigen, aber die dicke Kutikula der Würmer, die sich während des Alterns in ihrer Struktur und Durchlässigkeit verändert, und die Variabilität der Farbstoffinfiltration in verschiedenen Geweben machen farbstoffbasierte Ansätze bei C. elegans schwierig 17. Darüber hinaus wird die Bewertung der mitochondrialen Morphologie durch mögliche Off-Target-Effekte der Farbstoffe, wie z. B. die Farbstoffaggregation17, verwirrt. Stattdessen werden im Wurmmodell häufig genetische Methoden zur Expression von mitochondrienlokalisierten Fluorophoren verwendet.
Hier konzentriert sich diese Studie auf die Hervorhebung von Stämmen, die eine mitochondriale Matrix lokalisiertes GFP (im Folgenden als MLS::GFP bezeichnet) unter zelltypspezifischen Promotoren exprimieren. Wichtig ist, dass diese transgenen Linien mit der MosSCI-Methode hergestellt wurden, um eine Einzelkopienexpression des Reporters in einem bekannten genomischen Locus zu gewährleisten, wodurch Probleme mit anderen verfügbaren Stämmen vermieden werden. Zum Beispiel führt eine hohe Kopienexpression einiger auf Mitochondrien abzielender Proteine zu einer Variabilität der Expressionsniveaus aufgrund der Integration extrachromosomaler Plasmidarrays in einen zufälligen Locus mit einer unbekannten Kopiennummer18. Darüber hinaus wurde bereits gezeigt, dass sie die Mitochondrien schädigen, da es für die Zellen eine hohe Belastung darstellt, die überexprimierten mitochondrialen Proteine richtig zu lokalisieren19. Daher machen die präzise, stabile und geringe Genexpression und die einfache Kreuzung mit bekannten Loci diese MosSCI-Transgene zur bevorzugten Methode. Unter Verwendung dieser Stämme standardisiert diese Studie Methoden zur Abbildung von Mitochondrien in den Muskeln, im Darm und in der Unterhaut von C. elegans. Darüber hinaus werden die wichtigen technischen Anwendungen und Methoden zur Fehlerbehebung hervorgehoben, die wichtig sind, um eine reproduzierbare experimentelle Analyse der Mitochondrien während des Alterns bei C. elegans zu berücksichtigen und sicherzustellen.
Protokoll
Einzelheiten zu den in dieser Studie verwendeten Reagenzien und Geräten sind in der Materialtabelle aufgeführt.
1. Wachstum und Erhaltung von C. elegans
- Vorbereitung von Nematoden-Wachstumsmedien (NGM)-Platten
- Verwenden Sie für die Kultur von C. elegans Standard-Agarplatten mit 2 % Agarplatten mit NGM, die 1 mM CaCl2, 12,93 μM (5 μg/ml) Cholesterin, 25 mM KPO4 (pH 6,0), 1 mM MgSO4, 2,5 mM (0,25 % w/v) Pepton und 51,3 mM NaCl enthalten. Siehe die detaillierte Methode zum Gießen von NGM-Platten, die in Castro Torres et al.20 beschrieben ist.
HINWEIS: Für RNA-Interferenz-Experimente (RNAi) fügen Sie 1 ml 1 M IPTG und 1 ml 100 mg/ml Carbenicillin für 1 l NGM-Agar-Platten hinzu. - Nachdem die NGM-Platten erstarrt sind, züchten Sie eine Kultur von OP50 in Lysogeny-Bouillon (LB) für 24-48 h bei Raumtemperatur oder züchten Sie eine Kultur von HT115 mit einem leeren Vektorplasmid pL4440 in LB mit Antibiotika (Ampicillin/Carbenicillin 100 μg/ml + Tetracyclin 5 μg/ml) unter Schütteln bei 37 °C für 12-16 h.
HINWEIS: Dieses Plasmid ist aus kommerziellen Quellen erhältlich (siehe Materialtabelle). - 200 μl OP50- oder HT115-Kultur auf 60-mm-Platten oder 1 ml auf 100-mm-Platten säen.
- Trocknen Sie die Platten, bis sie nicht mehr nass sind, und lagern Sie die Platten in verschlossenen Behältern bei 4 °C bis zu 2 Monate.
- Optional: Geben Sie 100 μl 10 mg/ml 5-Fluor-2'-Desoxyuridin (FUDR) direkt auf NGM-Agarplatten, die mit Bakterien besiedelt sind, um L4/adulte Würmer chemisch zu sterilisieren und die Entwicklung jüngerer Würmer zu verhindern.
HINWEIS: Die in dieser Studie entwickelten und verwendeten Stämme sind:
(1) RHS191 - uthSi17[myo3p::MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] I
(2) RHS192 - uthSi83[col19p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)]I
(3) RHS193 - uthSi80[vha-6p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] IV
- Verwenden Sie für die Kultur von C. elegans Standard-Agarplatten mit 2 % Agarplatten mit NGM, die 1 mM CaCl2, 12,93 μM (5 μg/ml) Cholesterin, 25 mM KPO4 (pH 6,0), 1 mM MgSO4, 2,5 mM (0,25 % w/v) Pepton und 51,3 mM NaCl enthalten. Siehe die detaillierte Methode zum Gießen von NGM-Platten, die in Castro Torres et al.20 beschrieben ist.
- Synchronisierend C. elegans durch Bleichen
- Für einen groß angelegten Assay, der eine hohe Anzahl von Nematoden erfordert, schneiden Sie eine 60 mm große NGM-Agarplatte voller Tiere in kleine Stücke und stücken Sie sie auf 100 mm große Platten, die zur Expansion mit Bakterien besiedelt sind. Warten Sie 2-3 Tage, bis die 100-mm-Platten voller Würmer sind, wenn sie nach dem Chunking bei 20 °C wachsen. Für detailliertere Empfehlungen für expandierende Tiere siehe Castro Torres et al.20.
- Um die Würmer für die Synchronisation zu sammeln, gießen Sie 5-10 mL M9-Lösung (22 mM KH2PO4 monobasisch, 42,3 mM Na2HPO4, 85,6 mM NaCl, 1 mM MgSO4) auf 100 mm NGM-Agarplatten mit Würmern und schwenken Sie die M9-Lösung vorsichtig, um die Würmer von den Bakterienrasen zu lösen.
- Ziehen Sie die Würmer mit einer serologischen Pipette auf und überführen Sie sie in konische 15-ml-Röhrchen.
HINWEIS: Serologische Glaspipetten werden empfohlen, da serologische Kunststoffpipetten dazu neigen, dass die Würmer an der Innenwand der Kunststoffpipetten haften bleiben. - 30 s bei 1.100 x g bei Raumtemperatur zentrifugieren, um die Würmer zu pelletieren und den Überstand mit einer Vakuumpumpe abzusaugen.
- Bereiten Sie 5 ml Bleichlösung vor, die 1,5 ml 6 % Natriumhypochlorit, 0,75 ml 5 M NaOH oder KOH und 2,75 ml dH2O enthält. Gießen Sie die 5 ml Bleichlösung in das Wurmpellet.
ACHTUNG: In diesem Schritt wird das Tragen von Handschuhen und einem Laborkittel empfohlen, da Natriumhypochlorit und Hydroxidlösungen ätzend sind. - Inkubieren Sie den Wurm + die Mischung aus Bleichlösung für ~4-6 Minuten, bis alle Körper der Tiere vollständig aufgelöst sind und nur noch die Eier übrig sind. Schütteln Sie die Mischung kräftig, um den Auflösungsprozess zu unterstützen.
HINWEIS: Kontrollieren Sie die Würmer jede Minute unter einem Präpariermikroskop. Verkürzen oder verlängern Sie die Bleichzeit, je nachdem, wie stark die Körper der erwachsenen Würmer aufgelöst werden. Wenn die Eier über einen längeren Zeitraum in einer Bleichlösung belassen werden, führt dies zu einer Beschädigung der Eier und beeinträchtigt die Lebensfähigkeit der Nematoden. - Die Mischung aus Eiern und Bleichlösung 30 s lang bei 1.100 x g bei Raumtemperatur zentrifugieren, um die Eier zu pelletieren, und dann den Überstand vorsichtig mit einer Vakuumpumpe ansaugen, wobei darauf zu achten ist, dass das Eipellet nicht abgesaugt wird.
HINWEIS: Es wird empfohlen, die Wäschen schnell durchzuführen, um eine längere Exposition der Eier gegenüber der Bleichlösung zu vermeiden. - Waschen Sie die Eier 4 Mal mit 5-10 ml M9-Lösung.
- Resuspendieren Sie die Eier in einem kleinen Volumen M9-Lösung und verteilen Sie sie auf NGM-Platten, die mit Bakterien besiedelt sind, indem Sie bis zu 50 μl pipettieren. Berechnen Sie eine grobe Schätzung der Anzahl der Eier, indem Sie 3-5 μl der Eimischung auf eine Platte pipettieren und die Anzahl der Eier in diesem Volumen zählen. Fügen Sie M9 hinzu, um die Eimischung auf eine zählbare Anzahl von Eiern in diesem Volumen zu verdünnen.
HINWEIS: Es wird empfohlen, weniger als 100 Würmer pro Platte zu panzern, da die Würmer vor Tag 1 des Erwachsenenalters verhungern können, wenn zu viele Tiere plattiert sind. Für detailliertere Empfehlungen für die Plattierung von Tierzahlen siehe Castro Torres et al.20. - Alternativ können die Nematoden mit L1 fixiert werden, um eine engere Alterssynchronisation zu ermöglichen. Für die L1-Absperrung geben Sie das entsprechende Volumen M9-Lösung in einem konischen 15-ml-Röhrchen auf ein Volumen von 10-12 mL. Lassen Sie die Würmer 16-24 h bei 20 °C in einem Rotator rotieren. Zentrifugieren (wie in Schritt 1.2.7) und den Überstand ansaugen. Berechnen Sie die Konzentration der L1-Tiere und plattieren Sie sie auf NGM-Platten, wie für Eier in Schritt 1.2.9 beschrieben.
- Altern der C. elegans-Population auf das gewünschte Alter: Die L4/adulten Tiere werden auf Platten mit 5-Fluor-2'-Desoxyuridin (FUDR) übertragen, wie in Schritt 1.1.6 beschrieben, um Nachkommen zu verhindern und bereits produzierte Nachkommen zu eliminieren. Für alternative Methoden, um Würmer ohne FUDR altern zu lassen, siehe Castro Torres et al.20.
HINWEIS: Einige wichtige Faktoren müssen berücksichtigt werden, bevor Sie sich für das Bleichen als Methode zur Synchronisierung von Würmern in ihrem ersten Larvenstadium entscheiden. Wie bereits erwähnt, kann eine übermäßige Exposition gegenüber Bleichmitteln Embryonen schädigen, die Schlupfraten verringern und einen Selektionsdruck für bleichmittelresistente Stämme erzeugen, was möglicherweise die Genetik der Population verändert. Wenn Embryonen über einen längeren Zeitraum nicht gefüttert werden, kann ein Nährstoffmangel den Stoffwechsel und die Entwicklung beeinträchtigen. Daher können Eiablage- oder NemaSync-Methoden als Alternative zu Bleaching-Methoden eingesetzt werden. Die Methode für das Eiablageverfahren wird in der referenzierten Studie von Castro Torres et al.20 schrittweise beschrieben. NemaSync ist eine neuere und teurere kommerzielle Methode, bei der ein manueller Wurmsynchronisierer verwendet wird, um Würmer ohne den Einsatz von Chemikalien zu synchronisieren21.
2. Bildgebung der Mitochondrien bei C. elegans
- Vorbereitung von Objektträgern für die Bildgebung von C. elegans
- Geben Sie 10-20 μl M9-Lösung auf Objektträger und übertragen Sie eine gewünschte Anzahl adulter Würmer in einem bestimmten Alter auf die Lösung auf dem Objektträger. Empfohlen werden ca. 25 Würmer.
HINWEIS: Die Verwendung herkömmlicher Methoden zur Lähmung von Würmern (z. B. Tetramisol oder Natriumazid) kann unter bestimmten Bedingungen zu einer mitochondrialen Fragmentierung führen (siehe unten). Stattdessen empfiehlt sich hier die Verwendung von HistoBond-Objektträgern, die eine Oberfläche mit einer dauerhaften positiven Oberflächenladung aufweisen, um die Bewegung der Würmer zu reduzieren, ohne Medikamente zu verwenden, die Artefakte einführen können. Es ist wichtig, ein angemessenes Verhältnis von Würmern zu M9-Lösung zu verwenden, da geringe Volumina dazu führen können, dass Tiere zwischen Objektträger und Deckglas zerquetscht werden, während hohe Volumina dazu führen, dass sich die Tiere während der Bildaufnahme bewegen. Empfehlungen sind in Tabelle 1 zu finden. - Alternativ zur Verwendung von Medikamenten, die Würmer lähmen, beziehen Sie sich auf die repräsentativen Ergebnisse dieses Manuskripts für eine Reihe von Konzentrations- und Zeitpunkten, die mit begrenzter Fragmentierung der Mitochondrien verwendet werden können.
HINWEIS: Obwohl es bei gesunden Wildtyp-Tieren keine größere Fragmentierung gab, ist es wichtig, sich daran zu erinnern, dass bestimmte Mutanten oder Erkrankungen bei der gleichen Konzentration dieser Arzneimittel eine mitochondriale Fragmentierung erfahren können, so dass bei der Verwendung dieser Arzneimittel einige Tests erforderlich sind. - Decken Sie die Würmer mit M9-Lösung auf dem Objektträger mit einem Deckglas ab und verwenden Sie Nagellack, um die Seiten abzudichten und das Verdampfen der M9-Lösung zu verhindern.
- Optional: Für die Muskelbildgebung rollen Sie Würmer, indem Sie das Deckglas vorsichtig anstoßen. Dadurch können die Körperwandmuskeln an den Seiten der Würmer besser an der Fokusebene ausgerichtet werden, um Bilder in höherer Qualität zu erhalten.
- Geben Sie 10-20 μl M9-Lösung auf Objektträger und übertragen Sie eine gewünschte Anzahl adulter Würmer in einem bestimmten Alter auf die Lösung auf dem Objektträger. Empfohlen werden ca. 25 Würmer.
- Bildgebung von Mitochondrien mit einem Verbundmikroskop
- Verwenden Sie ein Standard-Weitfeldmikroskop für Muskel- und hypodermale Mitochondrien (myo-3p::MLS::GFP und col-19p::MLS::GFP).
HINWEIS: Es wurde ein kommerziell erhältlicher Imager verwendet, der mit einem 63x/1,4 Plan Aprochromat-Objektiv, einem GFP-Filter (11525314), einer DFC9000 GT-Kamera und einer LED5-Lichtquelle ausgestattet war, und es wurde eine kompatible Mikroskopsoftware verwendet. - Optimieren Sie die Bildgebungseinstellungen für jedes Mikroskop und jeden Versuchsaufbau.
HINWEIS: Als Ausgangspunkt sind die Parameter für die Bildgebung mit dem in diesem Manuskript verwendeten Imager jedoch wie folgt: Die Abmessungen betragen 2048 (x) * 2048(y) Pixel bei einer Schrittweite von 0,50 μm für hypodermale Mitochondrien und "System Optimized", das von der Software automatisch für Muskelmitochondrien berechnet wird. Die Expositionszeit beträgt 50 ms für Muskelmitochondrien und 50 ms für hypodermale Mitochondrien. Die 475-nm-LED wurde auf 50 % Leistung eingestellt, und der Neutraldichtefilter wurde auf 30 % für Muskel- und 17 % für hypodermale Mitochondrien eingestellt. Der Z-Bereich beginnt dort, wo das GFP-Signal sichtbar ist, bis zu seinem Ende. Um das Rauschen in einer Max-Projektion zu minimieren, kann man die Anzahl der für die Max-Projektion verwendeten Z-Ebenen verringern. - Für die Bildgebung der intestinalen Mitochondrien (vha-6p::MLS::GFP) wird ein konfokales Mikroskop verwendet.
HINWEIS: Die Bildgebung wurde an einem kommerziell erhältlichen konfokalen Mikroskop durchgeführt, das mit einem 63x/1,4 Plan ApoChromat-Objektiv, einem Weißlichtlaser (WLL), einem akusto-optischen Strahlteiler und HyD S-Detektoren ausgestattet war und mit Mikroskopsoftware ausgeführt wurde. Wie bereits erwähnt, sollte man jeden einzelnen Versuchsaufbau optimieren, aber als Ausgangspunkt sind die Parameter für die Bildgebung mit dem Stellaris, die in dieser Studie verwendet werden, wie folgt: Die Tragfähigkeit wurde auf 85,00 % maximale Leistung und mit einer 485 nm Laserlinie bei einer Intensität von 3,00 % eingestellt. Der HyD S-Detektor wurde auf 490-590 nm mit einer Verstärkung von 25 in der analogen Einstellung eingestellt. Wir haben unidirektional einen Bereich von 1024 (x) * 1024(y) oder 82,01 μm (x) * 82,01 μm (y) mit 5 (z) Pixeln bei einer Schrittweite von 0,495 μm (Dimension z ist abhängig von der Größe der Würmer oder Gewebe) mit einer Scangeschwindigkeit von 1.000 Hz, einem Zoom von 2,25, einem Liniendurchschnitt von 2 und einer Lochblende von 1 AU (95,5 μm) gescannt. Der Z-Bereich beginnt dort, wo das GFP-Signal sichtbar ist, bis zu seinem Ende. Um das Rauschen in einer Max-Projektion zu minimieren, kann man die Anzahl der für die Max-Projektion verwendeten Z-Ebenen verringern.
- Verwenden Sie ein Standard-Weitfeldmikroskop für Muskel- und hypodermale Mitochondrien (myo-3p::MLS::GFP und col-19p::MLS::GFP).
- Quantifizierung der mitochondrialen Morphologie
HINWEIS: Für Details siehe Schindelin et al.22 und siehe Ergänzende Abbildung 1A-D.- Laden Sie die Software FIJI (Fiji ist nur ImageJ, https://fiji.sc/) herunter und installieren Sie sie. Öffnen Sie dann FIJI.
- Um das MitoMAPR-Makro herunterzuladen und zu installieren, laden Sie den Quellcode 1 von https://doi.org/10.7554/eLife.49158.033 herunter. Kopieren Sie dann den gesamten Code, der unter "A." erwähnt wird. IJM-Code für MitoMAPR-1.0." Öffnen Sie als Nächstes Fiji, gehen Sie zu Plugins > Neues > Makro und fügen Sie den zuvor kopierten Code in das Makrofenster ein. Gehen Sie zur Option Datei > Speichern unter und speichern Sie das Makro für die zukünftige Verwendung.
- Öffnen Sie ein mikroskopisches 3D-Bild mit Z-Stapeln in FIJI, indem Sie Dateien per Drag & Drop in die Fiji-Symbolleiste ziehen oder zu Datei > Öffnen gehen.
- Erstellen Sie eine maximale Projektion des Bildes, indem Sie zu Bild > Stapel > Z-Projekt wechseln, dann den Bereich der Slices mit fokussierten Bildern auswählen, die maximal projiziert werden sollen, und dann Max. Intensität als Projektionstyp auswählen.
- Speichern Sie das Bild als TIFF, indem Sie zu Datei > Speichern als > Tiff gehen.
- Schneiden Sie interessante Regionen (ROIs) aus vollständigen Bildern zu, indem Sie das Werkzeug "Rechteck" verwenden, einen ROI zeichnen und dann zu Bild > Zuschneiden gehen.
- Speichern Sie das Bild als TIFF, indem Sie zu Datei > Speichern als > TIFF gehen.
- Führen Sie das zuvor gespeicherte MitoMAPR-Makro aus, indem Sie die Makrodatei per Drag & Drop auf die Fiji-Symbolleiste ziehen und dann auf Ausführen klicken. In einem neuen Fenster werden Sie aufgefordert, den Ordner auszuwählen, in dem die Tiff-Datei in 2.3.6 gespeichert wurde.
- In einem neuen Fenster werden Sie aufgefordert, einen Bereich auszuwählen. Erstellen Sie ein Rechteck im Bild mit dem Rechteck-Werkzeug, wie bereits in Schritt 2.3.4 erwähnt, und drücken Sie OK.
HINWEIS: Das gesamte Bild kann ausgewählt werden, wenn es zuvor wie in Schritt 2.3.6 beschrieben zugeschnitten wurde. Speichern Sie das Fenster "Daten" mit allen Werten, die sich auf die mitochondriale Morphologie beziehen, als Excel-Datei, indem Sie auf Datei gehen > Speichern unter > Speichern.
- Stapelverarbeitung von Proben mit FIJI-Makros
- Erstellen Sie ein Makro, um den Bereich der zu analysierenden Bilder anzugeben. Öffnen Sie Fiji und gehen Sie zu Plugins > New > Macro. Fügen Sie den folgenden Code in das Textfeld ein und speichern Sie das Makro wie in 2.3.2 beschrieben (Ergänzende Abbildung 1E-H).
HINWEIS: Rechteck erstellen (100, 100, 200, 200); run (Angeben..., Breite = 100, Höhe = 100, x = 100, y = 100 skaliert). - Erstellen Sie ein Makro zum Speichern aller geöffneten Bilder als TIFF-Dateien. Wechseln Sie im Makrofenster zu Datei > Neu , und fügen Sie den folgenden Code in das Textfeld ein. Speichern Sie das Makro wie in 2.3.2 beschrieben.
HINWEIS: dir = getDirectory(" Einen Ordner auswählen"); ids=newArray(nBilder); für (i=0; i - Erstellen Sie ein Makro für den Batch MitoMAPR. Gehen Sie zu https://doi.org/10.7554/eLife.49158.033 Quellcode 1 und kopieren Sie alle unter B.) genannten Codes. IJM-Code für MitoMAPR-1.0_Batch". Gehen Sie im Makrofenster zu Datei > Neu, fügen Sie den Code aus dem Quellcode 1 in das Textfeld ein und speichern Sie das Makro wie in Schritt 2.3.2 beschrieben.
- Erstellen Sie ein Makro für das iterative Zuschneiden. Gehen Sie zu https://doi.org/10.7554/eLife.49158.033 Quellcode 1 und kopieren Sie alle unter C.) IJM-Code für CropR" genannten Codes. Gehen Sie im Makrofenster zu Datei > Neu, fügen Sie den Code aus dem Quellcode 1 in das Textfeld ein und speichern Sie das Makro wie in Schritt 2.3.2 beschrieben.
- Um die Stapelverarbeitung zu starten, speichern Sie zunächst alle Bilder, die verarbeitet werden sollen, als TIFF, indem Sie alle Bilder öffnen und das in Schritt 2.4.2 gespeicherte Makro ausführen. Alle Bilder werden als TIFF im angegebenen Ordner gespeichert. Schließen Sie alle Bilder, sobald sie gespeichert sind.
- Wenn es sich bei den in Schritt 2.4.5 gespeicherten Bildern um 3D-Bilder (Bilder mit Z-Stapeln) handelt, konvertieren Sie sie in 2D-Bilder, indem Sie sie zu Z-Projektionen machen. Um die Bilder im Stapel zu projizieren, öffnen Sie das Fenster für die Stapelverarbeitung, indem Sie im Hauptfenster von FIJI auf > Batch > Makro verarbeiten gehen.
- Wählen Sie bei "Eingabe" den Speicherort des Ordners aus, der alle 3D-Bilder enthält, die ab Version 2.4.5 verarbeitet werden sollen. Wählen Sie bei "Ausgabe" den gewünschten Speicherort für die gespeicherten Bilder nach der Verarbeitung aus. Wählen Sie als Ausgabeformat TIFF aus. Fügen Sie den folgenden Code in das große Textfeld im Fenster ein und drücken Sie die Prozesstaste: run("Z Project...", "projection=[Max Intensity]").
- Zuschneiden ROIs mit identischen Abmessungen aus allen zu verarbeitenden 2D-Bildern, indem Sie das iterative Zuschneidemakro aus Schritt 2.4.4 in Kombination mit dem Makro zum Festlegen des Bereichs aus Schritt 2.4.3 verwenden. Führen Sie das iterative Zuschneidemakro aus Schritt 2.4.4 aus. Es erscheint ein Fenster, in dem Sie nach einem Verzeichnis gefragt werden.
- Wählen Sie den Ordner aus, der nur 2D-Bilder aus Schritt 2.4.5 oder die 2D-Z-projizierten Bilder aus Schritt 2.4.6 enthält, und klicken Sie auf die Schaltfläche Auswählen . Das Makro öffnet eines der Bilder im ausgewählten Ordner, und es erscheint ein Fenster mit der Bezeichnung "Auswahl treffen" mit zwei Schaltflächen.
- Führen Sie das angegebene Auswahlmakro aus Schritt 2.4.3 aus. Ändern Sie die Werte für Breite und Höhe in diesem Makrotextfeld nach Bedarf, um die Auswahlbemaßung zu ändern, und drücken Sie Ausführen , um die neue Auswahl zu beobachten. Wenn Sie die X- und Y-Werte im Makro ändern, wird die Position der Auswahl in der oberen linken Ecke geändert.
- Sobald die Auswahlabmessungen zufriedenstellend sind, ziehen Sie das Rechteck in den gewünschten Bereich und drücken Sie dann die OK-Taste im Fenster "Auswahl treffen". Der beschnittene Bereich wird in dem Ordner gespeichert, den Sie in Schritt 2.4.7 ausgewählt haben.
- Das Makro durchläuft alle verbleibenden Bilder im Ordner. Führen Sie für jedes Bild das Makro aus Schritt 2.4.8 aus, ohne die Bemaßungswerte zu ändern, um sicherzustellen, dass für alle Bilder dieselben Abmessungen beschnitten werden. Ziehen Sie für jedes Bild das Auswahlrechteck an die gewünschte Stelle und drücken Sie die OK-Taste im Fenster "Auswahl treffen".
- Öffnen Sie den Ordner mit den zugeschnittenen Bildern aus den Schritten 2.4.8-2.4.9 und verschieben Sie alle zugeschnittenen Bilder in einen neuen Ordner.
- Analysieren Sie alle zugeschnittenen Bilder, indem Sie das Batch-Makro MitoMAPR aus Schritt 2.4.3 ausführen. In einem neuen Fenster werden Sie aufgefordert, ein Verzeichnis auszuwählen. Wählen Sie den Ordner mit allen zugeschnittenen Bildern aus Schritt 2.4.10 aus. Sobald der Vorgang abgeschlossen ist, öffnet sich ein Fenster mit dem Namen "Daten" mit allen Werten, die sich auf die mitochondriale Morphologie beziehen. Speichern Sie alle Daten als Excel-Datei, wie in Schritt 2.3.9 beschrieben.
- Erstellen Sie ein Makro, um den Bereich der zu analysierenden Bilder anzugeben. Öffnen Sie Fiji und gehen Sie zu Plugins > New > Macro. Fügen Sie den folgenden Code in das Textfeld ein und speichern Sie das Makro wie in 2.3.2 beschrieben (Ergänzende Abbildung 1E-H).
Ergebnisse
C. elegans ist aufgrund seines transparenten Körpers, der eine einfache Bildgebung von ganzen Würmern und lebenden Tieren ohne übermäßige Probenvorbereitung ermöglicht, ein großartiges Modell für die mitochondriale Bildgebung. Darüber hinaus kann die mitochondriale Morphologie in verschiedenen Geweben leicht visualisiert werden, indem gewebespezifische Promotoren verwendet werden, um auf Mitochondrien ausgerichtete fluoreszierende Proteine zu exprimieren. Hier wurden myo-3p (für Muskelmitochondrien), vha-6p (für intestinale Mitochondrien) und col-19p (für subdermale Mitochondrien) verwendet, um die Expression eines auf Mitochondrien ausgerichteten GFP (mitochondriale Lokalisationssequenz des ATP-1-Proteins) zu steuern. Die Mitochondrien der Körperwandmuskulatur von C. elegans weisen eine röhrenförmige Morphologie auf, die sich entlang der Muskelfasern (Myofibrillen) ausrichtet (Abbildung 1A); Die intestinalen Mitochondrien zeigten stark miteinander verbundene, netzartige Strukturen mit weniger gleichmäßiger Ausrichtung (Abbildung 1B); und die Hypodermis hat röhrenförmige Mitochondrien, die im Vergleich zum Darm oder Muskel runder oder ovaler erscheinen (Abbildung 1C). Die mitochondriale Morphologie ist im Allgemeinen über die gesamte Länge des Wurms in den Muskeln und im Darm konsistent, aber die Hypodermis weist einige geringfügige Unterschiede in der Vernetzung der Mitochondrien zwischen den proximalen und distalen Enden des Wurms auf. Um reproduzierbare experimentelle Ergebnisse zu erzielen, empfiehlt es sich daher, sich auf eine bestimmte Region ihres Körpers zu konzentrieren. Dabei wird der Bereich zwischen dem Pharynx und der Vulva ~100-200 μm unterhalb des Pharynx konsistent abgebildet.
Bei Muskel- und hypodermalen Mitochondrien bietet die Bildgebung unter einem Verbundmikroskop aufgrund der Flachheit der Zellen und des geringen unscharfen Lichts eine ausreichende Auflösung. Darmmitochondrien sind jedoch mit einem Verbundmikroskop schwer zu beobachten, da das große Volumen des Darms die Auflösung aufgrund einer großen Menge an unscharfem Licht aus anderen Bereichen des Darms einschränkt (Abbildung 2). Daher wird empfohlen, die Darmmitochondrien unter einem konfokalen Mikroskop abzubilden, um unscharfes Licht zu reduzieren und die richtige mitochondriale Morphologie sichtbar zu machen.
Die Form der Mitochondrien ist hochdynamisch und ändert sich je nach dem metabolischen Milieu der Tiere23 oder sogar aufgrund der Exposition gegenüber variablen Substratsteifigkeiten24. Daher kann das Belassen von Tieren auf Objektträgern über einen längeren Zeitraum ohne Nahrungsquelle und auf dem steifen Substrat aus Glas möglicherweise die mitochondriale Morphologie beeinflussen. Hier ergab diese Studie, dass die Mitochondrien von Würmern nach etwa 30 Minuten auf einem Objektträger in M9-Lösung fragmentieren, wobei die subdermalen Mitochondrien die stärkste Fragmentierung aufwiesen (Abbildung 3). Daher sollte die Bildgebung der Mitochondrien schnell nach der Probenvorbereitung durchgeführt werden.
Chemikalien, die die Beweglichkeit der Würmer einschränken, einschließlich Natriumazid und Tetramisol, werden häufig für die Live-Bildgebung von C. elegans verwendet, da stationäre Würmer erforderlich sind, um mehrere Z-Schnitte von Tieren für die 3D-Bildgebung zu erfassen. Frühere Studien haben gezeigt, dass die Exposition gegenüber Natriumazid oder Tetramisol zu einer mitochondrialen Fragmentierung führen kann25. Überraschenderweise zeigte sich, dass Natriumazid - selbst in hohen Konzentrationen - nur einen begrenzten Einfluss auf die mitochondriale Morphologie im Muskel oder Darm hatte. Die Hypodermis zeigte jedoch eine frühere mitochondriale Fragmentierung als die M9-Kontrollen (Abbildung 3). Wichtig ist, dass hohe Konzentrationen von Tetramisol (100 mM) in allen Zelltypen unmittelbar nach der Exposition zu einer signifikanten mitochondrialen Fragmentierung führten. Im Vergleich dazu führten mittlere Konzentrationen (10 mM) zu einer schnelleren Fragmentierung im Vergleich zu einer M9-Kontrolle. Niedrige Konzentrationen (1 mM) hatten einen begrenzten Einfluss auf die mitochondriale Morphologie. Diese Daten deuten darauf hin, dass die Verwendung von Natriumazid eine praktikable Option für die schnelle Bildgebung von Mitochondrien sein kann, während Tetramisol im Allgemeinen vermieden werden sollte.
Erwartungsgemäß zeigen die Mitochondrien aller Gewebe von C. elegans während des natürlichen Alterungsprozesses eine Fragmentierung (Abbildung 4A). Die Fragmentierung kann als Mitochondrien visualisiert werden, die sich als stumpfe und kugelförmige Strukturen präsentieren, die sich dramatisch von den linearen, röhrenförmigen Mitochondrien unterscheiden, die bei Jungtieren zu sehen sind. Es ist wichtig zu beachten, dass der Darm in einem späteren Alter an sphärischen autofluoreszierenden Strukturen zunimmt, so dass darauf geachtet werden muss, dass die Autofluoreszenz nicht mit tatsächlichen mitochondrialen Strukturen verwechselt wird. Da es eine Variabilität der Veränderungen in der mitochondrialen Struktur bei Würmern geben kann, ist es wichtig, die Quantifizierung der mitochondrialen Morphologie an einer signifikanten Stichprobengröße durchzuführen, anstatt nur einige wenige Würmer abzubilden. Hier wurde mitoMAPR verwendet, das eine automatisierte Quantifizierung der mitochondrialen Morphologie anhand einer Vielzahl von Metriken ermöglicht, darunter Objekte, Netzwerke, Verbindungen pro Netzwerk, Verbindungspunkte, Objektlänge, mitochondrialer Fußabdruck, mitochondriale Abdeckung und Objektfläche (Ergänzende Tabelle 1 und Ergänzende Tabelle 2). Die Automatisierung beseitigt subjektive Vorurteile beim Benutzer. Hier berichten wir, dass die Objektlängenmetrik optimal ist, um die Veränderungen der mitochondrialen Morphologie von Muskeln und Darm während des Alterns quantitativ zu messen, und die Verbindungspunktmetrik, um die Veränderungen der hypodermalen mitochondrialen Morphologie während des Alterns zu messen (Abbildung 4B).
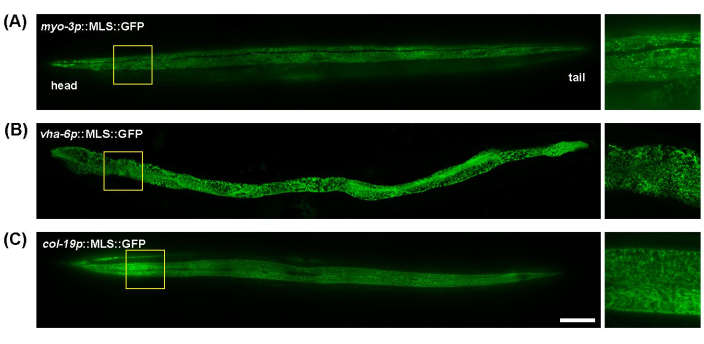
Abbildung 1: Bilder von Mitochondrien im gesamten Körper von C. elegans. Die mitochondriale Bildgebung wurde über die gesamte Länge von adulten Tieren an Tag 5 durchgeführt, die auf EV aus dem L1-Stadium gezüchtet wurden, bei transgenen Tieren mit MLS::GFP-Expression im Muskel (A), Darm (B) und in der Hypodermis (C). Maßstab: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
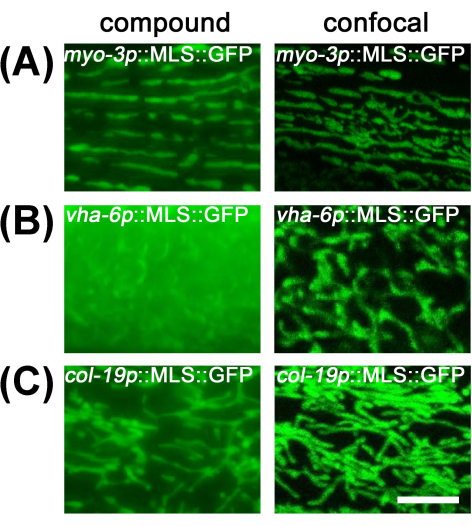
Abbildung 2: Vergleich der mitochondrialen Bildgebung zwischen Verbundmikroskop und Konfokalmikroskop. Die mitochondriale Bildgebung wurde an adulten Tieren am Tag 1 durchgeführt, die auf EV aus dem L1-Stadium gezüchtet wurden, in transgenen Tieren mit MLS::GFP-Expression im Muskel (A), Darm (B) und in der Hypodermis (C). Maßstab: 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
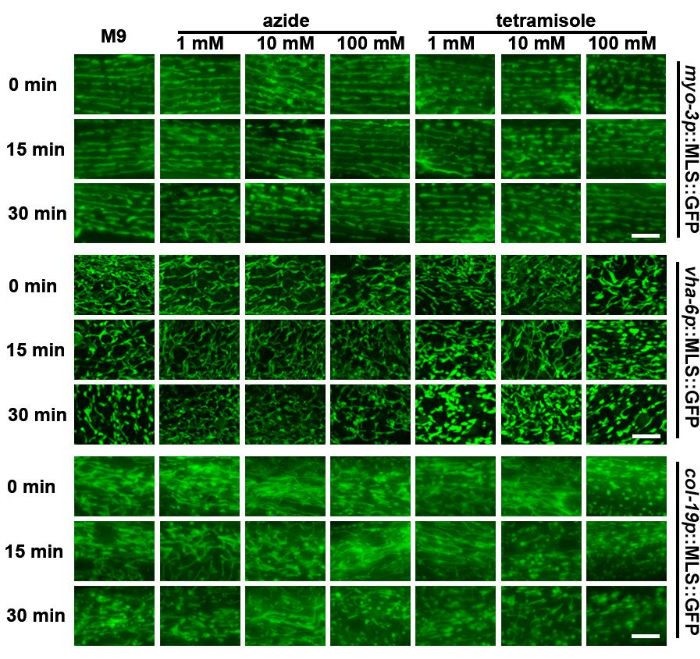
Abbildung 3: Beurteilung der mitochondrialen Fragmentierung verschiedener Gewebe nach Behandlung mit M9, Natriumazid und Tetramisol. Mitochondriale Bildgebung wurde an adulten Tieren am Tag 1 mit Expression von MLS::GFP in Muskeln, Darm und Hypodermis durchgeführt. Die Tiere wurden ab der L1-Stufe auf EV gezüchtet. Die Tiere wurden auf Objektträger gelegt, die M9, Natriumazid (1 mM, 10 mM und 100 mM) oder Tetramisol (1 mM, 10 mM und 100 mM) enthielten, und die Bildgebung wurde unmittelbar nach der Objektträgervorbereitung (0 min) oder 15 min oder 30 min nach der Objektträgervorbereitung durchgeführt. Repräsentative Bilder sind für n > 5 Tiere pro Stamm für 2 biologische Replikate zu sehen. Maßstabsleisten: 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
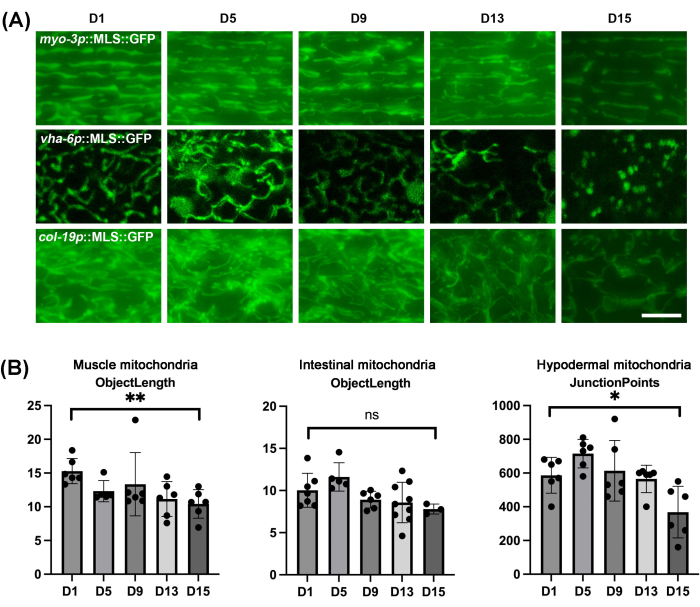
Abbildung 4: Mitochondriale Bildgebung von C. elegans während des Alterns in verschiedenen Geweben und Quantifizierung der mitochondrialen Morphologie mittels MitoMAPR. (A) Die mitochondriale Bildgebung wurde an erwachsenen Tieren an den Tagen 1, 5, 9, 13 und 15 des Erwachsenenalters mit Expression von MLS::GFP in den Muskeln, im Darm und in der Hypodermis durchgeführt. Die Tiere wurden auf EV aus dem L1-Stadium gezüchtet und auf EV-Platten mit FUDR aus dem adulten Tagesstadium 1 gebracht. Die Bilder sind repräsentativ für n ≥ 5 Tiere pro Stamm für ≥ 3 biologische Replikate. Maßstab: 5 μm. (B) Die Quantifizierung der Objektlänge von Muskelmitochondrien und Darmmitochondrien von Würmern an den Tagen 1, 5, 9, 13 und 15 und die Quantifizierung der Verbindungspunkte der hypodermalen Mitochondrien von Würmern an den Tagen 1, 5, 9, 13 und 15. Alle einzelnen Datenpunkte in Kombination mit dem Mittelwert ± SD. Graphen wurden mit Hilfe eines studentischen t-Tests grafisch dargestellt und statistisch analysiert. ns = nicht signifikant, *p < 0,03; **p < 0,002; p < 0,0002; p < 0,0001. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Tage der Würmer | Anzahl der Würmer | Volumen des Puffers (ml) |
| 1 | 25 | 13 |
| 5 | 25 | 14 |
| 9 | 20 | 15 |
| 13 | 20 | 15 |
Tabelle 1: Empfohlene Anzahl von Würmern und Puffervolumen bei der Vorbereitung von Objektträgern.
Ergänzende Abbildung 1: Arbeitsabläufe bei der Anwendung einer Einzelbildquantifizierung mit MitoMAPR und der Stapelverarbeitung für mehrere Bilder. (A) Arbeitsablauf zur Quantifizierung der mitochondrialen Morphologie aus einem einzigen Bild mit Hilfe des MitoMAPR-Makros. (B) Z-Projektion einer rohen 3D-Bilddatei mit Fidschi (Max-Projektion). (C) Zuschneiden des interessierenden Bereichs aus dem projizierten Z-Bild. (D) Skelettbilder, die mit dem MitoMAPR-Makro aufgenommen wurden. Maßstabsleisten: 5 μm. (E) Arbeitsablauf zur Quantifizierung der mitochondrialen Morphologie aus mehreren Bildern mittels Batch-Verfahren unter Verwendung des MitoMAPR-Makros. (F) Screenshot des Zuschneidemakros. (G) Screenshot eines Batch-Prozesses für die Z-Projektion. (H) Screenshot des Auswahlbestätigungsfensters des Batch-Cropping-Makros. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 1: Quantitative Analyse der mitochondrialen Morphologiemetriken im Muskelgewebe von C. elegans . Zu den aufgezeichneten Metriken gehören Objekte, Netzwerke, Kreuzungen pro Netzwerk, Verbindungspunkte, Objektlänge, mitochondrialer Fußabdruck, Objektfläche und mitochondriale Abdeckung, gemessen über verschiedene Tage. Die Daten bilden eine Grundlage für die Beurteilung von Veränderungen der mitochondrialen Morphologie mit zunehmendem Alter. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 2: Auswirkungen immobilisierender Chemikalien auf die mitochondriale Morphologie bei C. elegans. Zu den Daten gehören Messungen wie Objekte, Netzwerke, Übergänge pro Netzwerk, Verbindungspunkte, Objektlänge, mitochondrialer Fußabdruck, Objektfläche und mitochondriale Abdeckung über unterschiedliche Konzentrationen und Zeitpunkte nach Probenvorbereitung mit M9-Puffer oder unterschiedlichen Konzentrationen von Tetramisol und Natriumazid. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die Fluoreszenzbildgebung der mitochondrialen Morphologie ist die gebräuchlichste Methode, um Veränderungen in den Mitochondrien zu bestimmen. Während fortschrittliche Mikroskopietechniken wie die Transmissionselektronenmikroskopie (TEM), die Rasterkraftmikroskopie und die Kryo-Elektronenmikroskopie eine höhere Auflösung bieten, bleibt die Fluoreszenzmikroskopie erschwinglicher und für die meisten Forscher zugänglich. Darüber hinaus kann die Fluoreszenzmikroskopie in lebenden Zellen durchgeführt werden, und in klaren Modellorganismen wie C. elegans kann die Bildgebung an ganzen Tieren durchgeführt werden26,27. Die Bildgebung von transgenen C. elegans ist recht einfach, und genetisch kodierte Fluoreszenzmarker ermöglichen eine zuverlässigere und robustere Bildgebung von Mitochondrien, da sie keine komplexe Probenverarbeitung erfordert oder unter unterschiedlichen Färbungen über Zelltypen oder Bedingungen hinweg durch herkömmliche mitochondriale Farbstoffe wie MitoTracker oder TMRE leidet 28,29,30. Genetisch kodierte Fluoreszenzmarker beinhalten in der Regel die direkte Markierung von mitochondrialen Proteinen mit Fluorophoren oder die Konjugation von Fluorophoren mit einer minimalen mitochondrialen Lokalisierungssequenz. Diese Konstrukte werden oft von gewebespezifischen Promotoren angetrieben, die die Visualisierung von Mitochondrien in verschiedenen Geweben wie Muskel-, Darm- oder Unterhautgewebe ermöglichen31. Im Allgemeinen werden diese fluoreszenzmarkierten Proteine überexprimiert, was potenziell unerwünschte physiologische Auswirkungen haben kann, wenn mitochondriale Proteine in voller Länge überexprimiert werden. daher sind minimale MLS-Sequenzen eine bessere Option32. Aber selbst bei der Überexpression minimaler MLS-fluoreszierender Proteinfusionen sollte eine hohe Überexpression vermieden werden, da der Import einer großen Menge an Protein in die Mitochondrien das Potenzial der Mitochondrienmembran verringern und die Tiergesundheit beeinträchtigen kann33. Während eine erschöpfende Charakterisierung aller derzeit verfügbaren C. elegans-Stämme den Rahmen dieses Manuskripts sprengen würde, findet sich hier eine detaillierte vergleichende Analyse zahlreicher mitochondrialer Reporter und deren Vor- und Nachteile31.
Für die Bildgebung von Mitochondrien in C. elegans kann die Standard-Compound- oder Weitfeldmikroskopie aufgrund der hohen Geschwindigkeit und Einfachheit dieser Methoden gegenüber der konfokalen Mikroskopie eine bevorzugte Option sein. In dieser Studie wurde gezeigt, dass flache Zellen wie der Muskel und die Unterhaut nur minimal von der konfokalen Mikroskopie profitieren, und die Verbindungsmikroskopie ermöglicht eine Erfassung mit ausreichender Auflösung, um die richtige mitochondriale Morphologie sichtbar zu machen. Größere Zellen wie der Darm machen die Verbindungsmikroskopie aufgrund des unscharfen Lichts zu einer Herausforderung. Daher ist die konfokale Mikroskopie für eine zuverlässige Darstellung der mitochondrialen Morphologie des Darms erforderlich.
Ein wichtiger Aspekt bei der 3D-Bildgebung über die gesamte Gewebedicke bei lebenden Tieren besteht darin, die Bewegung der Würmer während der Bildaufnahme zu verhindern. Forscher verwenden häufig Methoden, um Würmer zu lähmen, wie Tetramisol oder Natriumazid34. Natriumazid hemmt die Cytochrom-c-Oxidase (Komplex IV), ein kritisches Enzym in der Elektronentransportkette der Mitochondrien, was aufgrund des Mangels an ATP, das für Muskelkontraktionen und andere zelluläre Funktionen benötigt wird, zu einer allgemeinen Lähmung führt34,35. Tetramisol wirkt, indem es Acetylcholin an den neuromuskulären Verbindungen nachahmt, was zu anhaltender Depolarisation und Muskelkontraktion führt36. Die Exposition gegenüber diesen Arzneimitteln kann jedoch eine mitochondriale Fragmentierung induzieren, indem sie oxidativen Stress verursacht23. In dieser Studie wurde festgestellt, dass Tetramisol sehr schnell eine mitochondriale Fragmentierung induzierte, Natriumazid jedoch eine viel begrenztere Wirkung hatte.
C. elegans bietet eine sehr einfache und unkomplizierte Möglichkeit, die Auswirkungen des Alterns auf die mitochondriale Morphologie zu untersuchen, da es eine kurze Lebensdauer hat und Tiere leicht altern lässt. Hier haben wir uns für die Exposition gegenüber FUDR entschieden, einer robusten Methode zur chemischen Sterilisation von Tieren, die die DNA-Replikation verhindert 20,37,38. FUDR kann jedoch unerwünschte Auswirkungen auf bestimmte Alterungsparameter haben, und für diejenigen, die über Off-Target-Effekte von FUDR besorgt sind, können andere Strategien zur Entfernung von Nachkommen angewendet werden39. Zum Beispiel gibt es sterile Mutanten, einschließlich der temperaturempfindlichen Keimbahnmutante glp-4, oder spermiendefiziente Mutanten, wie CF512 40,41,42. Alternativ können Tiere auch auf natürliche Weise gealtert werden, indem täglich von Hand erwachsene Tiere aus ihren Nachkommen entnommen werden.
Die Quantifizierung der Veränderungen in der mitochondrialen Morphologie ist ebenfalls ein sehr wichtiger Aspekt, da es bei den Tieren eine signifikante Variabilität in der mitochondrialen Morphologie geben kann. Daher ist die Durchführung quantitativer Analysen und Statistiken über eine ausreichende Stichprobengröße unerlässlich, um aussagekräftige Schlussfolgerungen über Veränderungen der mitochondrialen Morphologie zu ziehen. Die Bildanalyse und -quantifizierung kann jedoch stark unter subjektiven Verzerrungen und Herausforderungen bei der Abbildung hochkomplexer Strukturen wie miteinander verbundener Mitochondrien leiden. Zu diesem Zweck finden Sie hier eine Zusammenfassung einer automatisierten Methode zur Quantifizierung der mitochondrialen Morphologie unter Verwendung zahlreicher Metriken, die vom Mair-Labor entwickelt wurden. MitoMAPR ermöglicht eine objektive, automatisierte Quantifizierung der mitochondrialen Morphologie, indem verschiedene Aspekte der Mitochondrien gemessen werden, einschließlich des mitochondrialen Netzwerks, der Objektlänge, der Verteilung, der Netzwerkabdeckung und des mitochondrialen Fußabdrucks. MitoMAPR ist ein kostenloses Makro für ImageJ und steht somit allen Laboren mit einem funktionsfähigen Rechner zur Verfügung. Ein wichtiger Aspekt bei der Verwendung von MitoMAPR ist die Quantifizierung über eine große Stichprobengröße, um zu bestimmen, welche Metrik der mitochondrialen Morphologie am robustesten ist, um Veränderungen der getesteten Versuchsbedingungen zu bestimmen43,44. Hier zeigt sich, dass die Objektlänge und die Verbindungspunkte die besten Metriken sind, um Veränderungen während des Alterns in Muskeln, Darm und Hypodermis zu bestimmen. Ein alternativer Ansatz zur Analyse von Veränderungen in der mitochondrialen Morphologie ist die Generierung von 3D-Darstellungen von Mitochondrien aus Z-Stack-Bildern, gefolgt von einer 3D-Analyse16,45. Dies kann mit kommerziell erhältlicher Software wie Image-Pro Plus mit den Modulen SharpStack Total Deconvolution und 3D Constructor erreicht werden. Es hat sich gezeigt, dass die Quantifizierung von mitochondrialen 3D-Darstellungen genauere Einblicke in die Form und die Netzwerkeigenschaften der Mitochondrien liefert46. Allerdings ist diese Methode technisch komplexer und kostspieliger im Vergleich zum Semi-3D-Ansatz, bei dem mehrere Bildausschnitte aus einem Weitfeld- oder konfokalen Z-Stack zu einer einzigen 2D-Projektion zusammengeklappt und mit Tools wie mitoMAPR analysiert werden. Diese Studie fasst die Verwendung der Einzelkopienexpression von mitochondrialer Matrix-gesteuertem GFP zusammen und beschreibt einige Fallstricke, die während der Bildgebung vermieden werden sollten.
Einschränkungen und Zeitüberlegungen
Obwohl die konfokale Mikroskopie für große und dicke Gewebe wie Darmgewebe empfohlen wird, können die standardmäßigen konfokalen Zeilenmikroskopietechniken zu langsam sein, um eine schnelle Bildgebung der Mitochondrien durchzuführen. Dies gilt insbesondere angesichts unserer Daten, die zeigen, dass das Halten von Würmern auf Objektträgern über einen längeren Zeitraum zu einer Fragmentierung der Mitochondrien führen kann. Obwohl sich die konfokale Mikroskopietechnologie erheblich weiterentwickelt hat, können Mikroskope, die eine schnelle Bildgebung durchführen können, wie z. B. Spinning Disk, Airyscan oder das in dieser Studie verwendete Mikroskop, für einige Labore unerschwinglich sein. In diesen Fällen kann die Verbindungsmikroskopie mit computergestützten Clearance-Methoden kombiniert werden, um unscharfes Licht zu entfernen, wie z. B. Dekonvolution47.
Wie beschrieben, ist MitoMAPR ein leistungsstarkes automatisiertes Makro zur quantitativen Messung der Länge der Mitochondrien und der Verbindung des mitochondrialen Netzwerks. Es sollte jedoch unter Berücksichtigung der hier aufgeführten Einschränkungen mit Vorsicht verwendet werden. Erstens beobachtet die Verwendung einer matrixgerichteten GFP nur die mitochondriale Fragmentierung der inneren Membran, was die mitochondriale Morphologie der äußeren Membran möglicherweise nicht vollständig rekapituliert, da die innere Membranspaltung auch ohne äußere Membranspaltung auftreten kann. Daher sollte für eine verfeinerte Bildgebung beider Membranen sowohl ein matrixgesteuertes als auch ein mitochondriales äußeres membrangerichtetes Fluorophor verwendet werden. Da äußere mitochondriale Membranmarker bei starker Überexpression die gleichen Folgen erleiden können wie matrixlokalisierte Proteine, wird die Verwendung von Einzelkopienmarkern empfohlen, insbesondere solche, die minimale mitochondriale Lokalisationssequenzen anstelle von vollständigen Proteinen wie den hier verwendeten verwenden31.
Wie in Abbildung 3 beschrieben, können Mitochondrien unter dem Mikroskop schnell fragmentieren, und unterschiedliche Methoden zur Vorbereitung von Objektträgern können die Morphologie je nach verwendetem Puffer stark beeinflussen. Die repräsentativen Daten, die in diesem Manuskript beschrieben werden, zeigen, dass die Mitochondrien der Würmer unter M9 eine minimale Fragmentierung und geringe Konzentrationen von Natriumazid aufweisen. Tetramisol, eine weitere weit verbreitete Chemikalie zur Lähmung von Würmern, fragmentiert die Mitochondrien schnell, was darauf hindeutet, dass ihre Verwendung generell vermieden werden sollte. Obwohl M9 und Natriumazid keine signifikante Fragmentierung der Mitochondrien zeigten, ist es wichtig zu beachten, dass verschiedene Stämme unterschiedlich reagieren können als die hier gezeigten Ergebnisse. Während andere Studien gezeigt haben, dass Natriumazid Mitochondrien fragmentieren kann, ist es möglich, dass unsere Stämme aufgrund des geringen Expressionsniveaus unserer Konstrukte keine signifikante mitochondriale Fragmentierung zeigen, wenn sie Natriumazid ausgesetzt sind. Eine hohe Expression von Mitochondrien-lokalisierten Proteinen kann das Membranpotenzial kollabieren, und daher können verschiedene Stämme, die in anderen Studien verwendet wurden, Mitochondrien anfälliger für die Fragmentierung durch die gleichen Konzentrationen von Natriumazid machen, die in dieser Studie verwendet wurden. Unabhängig davon sollte darauf geachtet werden, dass Chemikalien, die Würmer lähmen, unter bestimmten Bedingungen keine mitochondriale Fragmentierung verursachen, die vor der Anwendung in allen Studien getestet werden müssen, da bestimmte Mutanten oder Bedingungen anfälliger für eine arzneimittelinduzierte mitochondriale Fragmentierung sein können. Darüber hinaus kann selbst die Verhaltung der Würmer in M9 zu Veränderungen ihrer natürlichen mitochondrialen Morphologie und Aktivität führen, da zuvor gezeigt wurde, dass ihre Schwimmaktivität im M9-Puffer die mitochondriale Spaltung und Fusionsdynamik beeinflusst. Darüber hinaus kann das Verbleiben von Würmern über einen längeren Zeitraum in M9 Hypoxiereaktionen aktivieren, die die Mitochondrien signifikant beeinflussen, indem sie Störungen der mitochondrialen Proteostase verursachen48,49.
Eine weitere wichtige Überlegung ist schließlich, dass Veränderungen der mitochondrialen Morphologie zwar oft mit Veränderungen der mitochondrialen Funktion korrelieren, es aber nicht immer eine direkte Korrelation zwischen den beiden gibt. Daher wird eine gründlichere Analyse der mitochondrialen Funktion empfohlen. Zum Beispiel kann die Sauerstoffverbrauchsrate mit einem Seahorse-Instrument50 gemessen werden, das mitochondriale Membranpotential kann mit Membranpotentialfarbstoffen wie JC9 oder TMRE51 gemessen werden, der mitochondriale oxidative Zustand kann mit redoxsensitiven Farbstoffen wie roGFP52 gemessen werden, und die mitochondriale Stressresilienz kann mit Hilfe der Empfindlichkeit gegenüber Stressoren wie Rotenon53 gemessen werden. Da die Bildgebung von Mitochondrien recht schnell erfolgen kann, bieten wir die Methoden hier als einfachen ersten Versuch an, um festzustellen, ob experimentelle Bedingungen die mitochondriale Morphologie beeinflussen. Diese Methoden sind sogar für groß angelegte Screenings von Medikamenten oder Genen geeignet, wobei eine weiterführende mitochondriale Analyse mit zusätzlichen Metriken empfohlen wird. Insgesamt geben wir hier einen Überblick über die vermutlich einfachsten Methoden, um die mitochondriale Morphologie von C. elegans mit minimalen experimentellen Fehlern abzubilden.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
J.K. wird unterstützt durch das USC Provost Fellowship; M.A. und G.G. werden unterstützt von T32AG052374; M.V. wird unterstützt von 1R25AG076400; und R.H.S. wird unterstützt durch R01AG079806 des National Institute on Aging und 2022-A-010-SUP der Larry L. Hillblom Foundation. Einige Stämme wurden vom CGC zur Verfügung gestellt, das durch den Zuschuss P40 OD010440 des NIH Office of Research Infrastructure Programs finanziert wird. Einige Genanalysen wurden mit Wormbase durchgeführt, das im Rahmen eines U41-Stipendiums HG002223 finanziert wird.
Materialien
| Name | Company | Catalog Number | Comments |
| 5-fluoro-2'-deoxyuridine (FUDR) | Spectrum Chemical | F2026-10GMBL | for proliferation inhibition |
| APEX IPTG | Genesee | 18-242 | for RNAi |
| Bacto Agar | VWR | 90000-764 | for NGM plates |
| Bacto Peptone | VWR | 97064-330 | for NGM plates |
| Calcium chloride dihydrate | VWR | 97061-904 | for NGM plates |
| Carbenicillin | VWR | 76345-522 | for RNAi |
| Cholesterol | VWR | 80057-932 | for NGM plates |
| Confocal microscope | Stellaris 5 | ||
| DMSO | VWR | BDH1115-1LP | solvent for drugs |
| HistoBond microscope slides | VWR | 16005-110 | for slides preparation |
| LB Broth | VWR | 95020-778 | for LB |
| LEICA S7E Dissecting Scope | Leica | 10450840 | Standard dissecting microscope |
| LEICA STELLARIS 5 | Leica | 158101100 | Confocal Microscope |
| LEICA THUNDER | Leica | 11525679 | Compound Microscope |
| Magnesium sulfate heptahydrate | VWR | 97062-132 | for NGM plates, M9 |
| pL4440 empty vector plasmid | addgene | plasmid #1654 | for empty vector plasmid |
| Potassium Chloride | VWR | 97061-566 | for bleach soluton |
| Potassium phosphate dibasic | VWR | EM-PX1570-2 | for NGM plates |
| Potassium phosphate monobasic | VWR | EM-PX1565-5 | for M9 |
| Sodium azide | VWR | 97064-646 | for paralyzing worms |
| Sodium Chloride | VWR | EM-SX0420-5 | for NGM plates, M9 |
| Sodium hypochlorite | VWR | RC7495.7-32 | for bleach solution |
| Sodium phosphate dibasic | VWR | 71003-472 | for M9 |
| Tetracycline hydrochloride | VWR | 97061-638 | for RNAi |
| WOB-L® Dry Vacuum Pumps, Standard-Duty, Welch® | VWR | 80077-612 | for aspiration |
Referenzen
- Protasoni, M., Zeviani, M. Mitochondrial structure and bioenergetics in normal and disease conditions. Int J Mol Sci. 22 (2), 586 (2021).
- Wang, Y., et al. The role of mitochondrial dynamics in disease. MedComm. 4 (6), e462 (2023).
- Kamer, K. J., Mootha, V. K. The molecular era of the mitochondrial calcium uniporter. Nat Rev Mol Cell Biol. 16 (9), 545-553 (2015).
- Chen, W., Zhao, H., Li, Y. Mitochondrial dynamics in health and disease: mechanisms and potential targets. Signal Transduct Target Ther. 8 (1), 333 (2023).
- Adebayo, M., Singh, S., Singh, A. P., Dasgupta, S. Mitochondrial fusion and fission: The fine-tune balance for cellular homeostasis. FASEB J. 35 (6), e21620 (2021).
- Madan, S., Uttekar, B., Chowdhary, S., Rikhy, R. Mitochondria lead the way: mitochondrial dynamics and function in cellular movements in development and disease. Front Cell Dev Biol. 9, 781933 (2021).
- Sharma, A., Smith, H. J., Yao, P., Mair, W. B. Causal roles of mitochondrial dynamics in longevity and healthy aging. EMBO Rep. 20 (12), e48395 (2019).
- Rolland, S. G., Lu, Y., David, C. N., Conradt, B. The BCL-2-like protein CED-9 of C. elegans promotes FZO-1/Mfn1,2- and EAT-3/Opa1-dependent mitochondrial fusion. J Cell Biol. 186 (4), 525-540 (2009).
- Labrousse, A. M., Zappaterra, M. D., Rube, D. A., van der Bliek, A. M. C. elegans dynamin-related protein DRP-1 controls severing of the mitochondrial outer membrane. Mol Cell. 4 (5), 815-826 (1999).
- Dickinson, D. J., Pani, A. M., Heppert, J. K., Higgins, C. D., Goldstein, B. Streamlined genome engineering with a self-excising drug selection cassette. Genetics. 200 (4), 1035-1049 (2015).
- Bosher, J. M., Labouesse, M. RNA interference: Genetic wand and genetic watchdog. Nat Cell Biol. 2 (2), E31-E36 (2000).
- Yu, C. C. J., et al. Expansion microscopy of C. elegans. eLife. 9, e46249 (2020).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Front Endocrinol. 11, 554994 (2020).
- Dingley, S., et al. Mitochondrial respiratory chain dysfunction variably increases oxidant stress in Caenorhabditis elegans. Mitochondrion. 10 (2), 125-136 (2010).
- Presley, A. D., Fuller, K. M., Arriaga, E. A. MitoTracker Green labeling of mitochondrial proteins and their subsequent analysis by capillary electrophoresis with laser-induced fluorescence detection. J Chromatogr B Anal Technol Biomed Life Sci. 793 (1), 141-150 (2003).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Curr Protoc Cell Biol. Chapter 4, Unit 4.25.1-Unit 4.25.21 (2010).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. Novel imaging tools to study mitochondrial morphology in Caenorhabditis elegans. Life Sci Alliance. 7 (11), e202402918 (2024).
- Benedetti, C., Haynes, C. M., Yang, Y., Harding, H. P., Ron, D. Ubiquitin-like protein 5 positively regulates chaperone gene expression in the mitochondrial unfolded protein response. Genetics. 174 (1), 229-239 (2006).
- Begelman, D. V., et al. An aco-2::gfp knock-in enables the monitoring of mitochondrial morphology throughout C. elegans lifespan. microPublication Biol. 2022, (2022).
- Castro Torres, T., et al. Surveying low-cost methods to measure lifespan and healthspan in Caenorhabditis elegans. J Vis Exp. 183, e64091 (2022).
- Rasmussen, N. R., Reiner, D. J. Nuclear translocation of the tagged endogenous MAPK MPK-1 denotes a subset of activation events in C. elegans development. J Cell Sci. 134 (17), jcs258456 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Luz, A. L., et al. Mitochondrial morphology and fundamental parameters of the mitochondrial respiratory chain are altered in Caenorhabditis elegans strains deficient in mitochondrial dynamics and homeostasis processes. PLoS One. 10 (6), e0130940 (2015).
- Oorloff, M., et al. Mechanical stress through growth on stiffer substrates impacts animal health and longevity in C. elegans. bioRxiv. , (2024).
- Chu, X., Wu, S., Raju, R. NLRX1 regulation following acute mitochondrial injury. Front Immunol. 10, 2431 (2019).
- Shah, P., Bao, Z., Zaidel-Bar, R. Visualizing and quantifying molecular and cellular processes in Caenorhabditis elegans using light microscopy. Genetics. 221 (4), iyac068 (2022).
- Wang, Y., Wang, P., Li, C. Fluorescence microscopic platforms imaging mitochondrial abnormalities in neurodegenerative diseases. Adv Drug Deliv Rev. 197, 114841 (2023).
- Ding, J., et al. An expanded GCaMP reporter toolkit for functional imaging in Caenorhabditis elegans. G3 (Bethesda, Md). 13 (10), jkad183 (2023).
- Sarasija, S., Norman, K. R. Analysis of mitochondrial structure in the body wall muscle of Caenorhabditis elegans. Bio-Protocol. 8 (7), e2801 (2018).
- Chaweeborisuit, P., Suriyonplengsaeng, C., Suphamungmee, W., Sobhon, P., Meemon, K. Nematicidal effect of plumbagin on Caenorhabditis elegans: A model for testing a nematicidal drug. Z Naturforsch C J Biosci. 71 (5-6), 121-131 (2016).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. . Novel imaging tools to study mitochondrial dynamics in Caenorhabditis elegans. 7, 11 (2024).
- Bolognesi, B., Lehner, B. Reaching the limit. eLife. 7, e39804 (2018).
- Jishi, A., Qi, X. Altered mitochondrial protein homeostasis and proteinopathies. Front Mol Neurosci. 15, 867935 (2022).
- Morton, K. S., Wahl, A. K., Meyer, J. N. The effect of common paralytic agents used for fluorescence imaging on redox tone and ATP levels in Caenorhabditis elegans. PLoS One. 19 (4), e0292415 (2024).
- Noumi, T., Maeda, M., Futai, M. Mode of inhibition of sodium azide on H+-ATPase of Escherichia coli. FEBS Lett. 213 (2), 381-384 (1987).
- Siete, C., Xiong, R., Khalid, A., Hsieh, Y. -. W., Chuang, C. -. F. Immobilization of C. elegans with different concentrations of an anesthetic for time-lapse imaging of dynamic protein trafficking in neurons. microPublication Biology. 2024, (2024).
- Mitchell, D. H., Stiles, J. W., Santelli, J., Sanadi, D. R. Synchronous growth and aging of Caenorhabditis elegans in the presence of fluorodeoxyuridine. J Gerontol. 34 (1), 28-36 (1979).
- Santi, D. V., McHenry, C. S. 5-Fluoro-2'-deoxyuridylate: covalent complex with thymidylate synthetase. Proc Natl Acad Sci U S A. 69 (7), 1855-1857 (1972).
- Wang, H., Zhao, Y., Zhang, Z. Age-dependent effects of floxuridine (FUdR) on senescent pathology and mortality in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun. 509 (3), 694-699 (2019).
- Beanan, M. J., Strome, S. Characterization of a germ-line proliferation mutation in C. elegans. Development (Cambridge, England). 116 (3), 755-766 (1992).
- Austin, J., Kimble, J. glp-1 is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans. Cell. 51 (4), 589-599 (1987).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Zhang, Y., et al. Neuronal TORC1 modulates longevity via AMPK and cell nonautonomous regulation of mitochondrial dynamics in C. elegans. eLife. 8, e49158 (2019).
- Dutta, N., et al. Investigating impacts of the mycothiazole chemotype as a chemical probe for the study of mitochondrial function and aging. GeroScience. 46 (6), 6009-6028 (2024).
- Tronstad, K., et al. Regulation and quantification of cellular mitochondrial morphology and content. Curr Pharm Des. 20 (35), 5634-5652 (2014).
- Nikolaisen, J., et al. Automated quantification and integrative analysis of 2D and 3D mitochondrial shape and network properties. PLoS One. 9 (7), e101365 (2014).
- Lee, J. S., Wee, T. L. E., Brown, C. M. Calibration of wide-field deconvolution microscopy for quantitative fluorescence imaging. J Biomol Tech. 25 (1), 31-40 (2014).
- Hong, M., Kwon, J. Y., Shim, J., Lee, J. Differential hypoxia response of hsp-16 genes in the nematode. J Mol Biol. 344 (2), 369-381 (2004).
- Yan, J., Sun, C. L., Shin, S., Van Gilst, M., Crowder, C. M. Effect of the mitochondrial unfolded protein response on hypoxic death and mitochondrial protein aggregation. Cell Death Dis. 12 (7), 711 (2021).
- Koopman, M., et al. A screening-based platform for the assessment of cellular respiration in Caenorhabditis elegans. Nat Protoc. 11 (10), 1798-1816 (2016).
- Shpilka, T., et al. UPRmt scales mitochondrial network expansion with protein synthesis via mitochondrial import in Caenorhabditis elegans. Nat Comm. 12 (1), 479 (2021).
- Braeckman, B. P., Smolders, A., Back, P., De Henau, S. In vivo detection of reactive oxygen species and redox status in Caenorhabditis elegans. antioxidants & redox signaling. 25 (10), 577-592 (2016).
- Bar-Ziv, R., et al. Measurements of physiological stress responses in C. elegans. J Vis Exp. 159, e61001 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten