Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Assistée par ordinateur à grande échelle de visualisation et quantification de la messe des îlots pancréatiques, distribution de taille et d'architecture
Dans cet article
Résumé
Méthodes assistées par ordinateur du roman achats à grande échelle et d'analyse des échantillons pancréatiques immunohistochimiquement colorées sont décrites: (1) la capture Slice virtuelle de toute la section, l'analyse de masse (2) de données à grande échelle; (3) la reconstruction de la 2D tranches virtuelles (4); cartographie des îlots 3D, et (5) L'analyse mathématique.
Résumé
Les îlots pancréatiques est un unique micro-organe composé de cellules sécrétant l'hormone de plusieurs endocriniens tels que les bêta-cellules (l'insuline), les cellules alpha (glucagon), et delta-cellules (somatostatine) qui sont incorporés dans les tissus exocrines et comprennent 1 - 2% de tout le pancréas. Il ya une corrélation étroite entre le corps et le poids du pancréas. Total des masse des cellules bêta augmente aussi proportionnellement pour compenser la demande d'insuline dans le corps. Ce qui échappe à cette expansion proportionnelle est la distribution en taille des îlots. Les grands animaux tels que les humains partagent les mêmes distributions de taille des îlots avec des souris, suggérant que cet organe de micro-a une limite certaine taille pour être fonctionnel. L'incapacité du pancréas de produire de gros animaux îlots proportionnellement plus est compensée par une augmentation du nombre d'îlots et par une augmentation de la proportion de grands îlots dans leur distribution îlot taille globale. Par ailleurs, les îlots présentent une plasticité frappant dans la composition cellulaire et de l'architecture parmi les différentes espèces et aussi au sein de la même espèce dans différentes conditions physiopathologiques. Dans la présente étude, nous décrivons de nouvelles approches pour l'analyse de données d'images biologiques afin de faciliter l'automatisation des processus analytiques, qui permettent l'analyse de grandes collections de données hétérogènes et dans l'étude de ces dynamiques des processus biologiques et les structures complexes. Ces études ont été entravés en raison de difficultés techniques d'échantillonnage impartiale et générant grande échelle des ensembles de données avec précision saisir la complexité des processus biologiques de la biologie de l'îlot. Ici, nous montrons des méthodes pour recueillir impartiale "représentative" des données au sein de la disponibilité limitée des échantillons (ou de minimiser le prélèvement de l'échantillon) et les paramètres standard expérimentale, et d'analyser précisément le complexe structure tridimensionnelle de l'îlot. Assistée par ordinateur permet l'automatisation de la collecte et l'analyse des ensembles de données à grande échelle et assure également l'interprétation impartiale des données. Par ailleurs, la quantification précise de la distribution granulométrique des îlots et les coordonnées spatiales (ie X, Y, Z-positions) ne conduit pas seulement à une visualisation précise de la structure et la composition des îlots pancréatiques, mais nous permet également de repérer les tendances au cours du développement et l'adaptation aux conditions de modification grâce à la modélisation mathématique. Les méthodes développées dans cette étude sont applicables aux études d'un grand nombre d'autres systèmes et organismes ainsi.
Protocole
1. Création de tranches virtuel du immunohistochimique Images Vitrail
- StereoInvestigator Ouvrir. Placez la lame propre maintenir l'échantillon dans le support de microscope et de le visualiser en stéréo chercheur en cliquant sur "Acquisition", puis "Image Live" (Acquisition → Photo Live). Déterminer les niveaux d'exposition pour chaque canal; dans notre exemple précis, le canal 2 est utilisé pour DAPI, Channel 3 pour la GFP, Channel 4 pour les DP, Channel 5 pour Cy5, et Channel 6 pour Cy7. Utilisez la fonction «histogramme Vidéo" fenêtre, qui affiche l'intensité de la fluorescence, pour déterminer le niveau d'exposition. Appropriées intensités de fluorescence sont atteintes lorsque les queues d'intensité à l'arrêt à l'extrémité droite de l'histogramme vidéo. Les niveaux d'exposition peuvent être changés dans les "Réglages de l'appareil" fenêtre. L'histogramme vidéo peut être porté avec la molette de mise microscope.
- Avant de créer une tranche virtuel, le contour de l'échantillon en cliquant sur l'écran à un point éloigné de l'échantillon, qui marquera un point de référence, puis en cliquant sur le contour de l'échantillon. Lorsque le contour est terminé, faites un clic droit et sélectionnez "Contour Fermer" pour relier le début et la fin.
- Une fois le contour est fermé, la capture une tranche virtuelle pour l'échantillon (→ Acquisition Acquérir Slice virtuel). Lorsque les options Virtual fenêtre Slice ouverte, sélectionnez "High Speed Acquérir" ainsi que mise au point automatique en décochant la case à côté de "Manuel". Enregistrez le fichier.
- La tranche virtuelle mise au point préalable doit être calibré en sélectionnant (clic droit → Ajouter à la liste de discussion du site) et mise au point manuelle de plusieurs sections aléatoires, c'est à dire les sites, dans l'aperçu Slice virtuel. Lorsque plusieurs sites ont été ciblés, démarrer le Slice virtuelle (clic droit sur Démarrer → Slice virtuel avec Prefocus). Après la tranche virtuelle pour le premier échantillon est terminée, passer au canal suivant et régler l'exposition en conséquence. Répétez les étapes 4 et 5 pour chaque canal.
2. Assistée par ordinateur à deux dimensions d'analyse
Quantification des îlots
- Les images sont ensuite traitées à l'aide d'une macro ImageJ nommé "IHCVS», qui prépare d'abord les images chargées pour l'analyse et quantifie les caractéristiques des îlots tels que la composition cellulaire (c'est à dire chaque domaine de la bêta-, alpha-et delta-populations de cellules, et l'îlot région à la valeur de somme). La pile est divisée en ses canaux séparés dans ImageJ, où chaque canal montre une image distincte sur la base de coloration immunohistochimique. Chaque image est alors automatiquement seuillée, après quoi une image 8-bits en noir et blanc, ou un masque, de l'image seuillée est produite. Les images de canal distinctes sont ensuite additionnées arithmétiquement dans un masque composite, et l'analyse de particules ImageJ intégré est effectué sur l'image composite.
- ROIs sont ensuite identifiés, tout en excluant les particules plus petites que d'une cellule beta unique (<170 um 2; surface calculée à l'aide d'un diamètre de ~ 15 um; Hara et al, 2003).. Le ROI résultant sont analysés et quantifiés à l'aide d'ImageJ, la quantification peut inclure le périmètre (une distance entourant une zone), la circularité (un degré de rondeur, où le nombre 1.0 représente un cercle parfait), le diamètre Féret (la plus longue distance au sein d'une région) et le centre coordonnées de chaque région analysée afin que la distribution des îlots peuvent être mathématiquement analysées à l'aide de diverses combinaisons de ces paramètres et tracés dans un graphique en 3D, les données qui seront utilisées pour la modélisation mathématique. Une image seule tranche virtuelle est analysé en 30 secondes. Plusieurs images peuvent être analysés en intégrant le traitement des images et le script d'analyse dans une syntaxe de boucle soutenue par ImageJ macro langage. La boucle s'ouvre tous les fichiers dans un répertoire donné, analyse les images, et les sorties des résultats dans un nouvel emplacement dans un fichier Excel.
- Les résultats agrégés sont enregistrées dans un tableur Excel, stocker des données telles que la surface, le périmètre, la circularité, diamètre Féret, et le centre de l'îlot pour chaque îlot, avec les numéros correspondants étiquetés dans l'image.
Analyse computationnelle et configuration d'histogramme
- Scripts Mathematica, qui s'exécute automatiquement une fois commencé, sont utilisés pour traiter les données recueillies tableur, s'élevant à des centaines de milliers d'entrées. L'un des processus essentiels qui sont exécutés est une analyse de fréquence, qui construit un histogramme utilisé pour étudier la distribution de taille et aussi pour déterminer les valeurs aberrantes dans les données. Sous le script, la zone totale des îlots est mesurée et comparée à d'autres paramètres, en particulier aux points de temps ou de l'âge du pancréas, et ensuite en conséquence affichés dans un graphique.
3. Reconstruction tridimensionnelle des deux dimensions immunohistochimique Stained Images Slice virtuel
Reconstruction 3D de coupes virtuelles
- Copiez le script ("im_jp2_2_tiff") dans le répertoire (par exemple, dossier) quecontient les images JP2. Exécuter le script avec l'interpréteur de commandes Linux en tapant "./im_jp2_2_tiff" dans la console puis appuyez sur ENTRÉE. L'exécution de ce script va convertir automatiquement toutes les images JP2 dans le répertoire dans des fichiers TIFF, qui est le format utilisé dans la construction et la quantification des trois dimensions des piles de l'tranches virtuelles. (Remarque:. Tapez "chmod + x im_jp2_2_tiff" si le script ne fonctionne pas, qui va convertir le fichier texte dans un script afin qu'il puisse fonctionner correctement)
- Lorsque toutes les images capturées ont été convertis de JP2 pour les fichiers TIFF, ils sont prêts à être utilisés pour l'analyse dans ImageJ.
- Avec ImageJ, ouvrez toutes les images pour le premier échantillon (cinq images dans cette affaire: DAPI, GFP, RFP, Cy5, Cy7) et de les fusionner en une seule image (Image → Couleur → Fusionner les couches). Répétez cette procédure pour chacun des échantillons. Lorsque tous les échantillons ont été combinés sous forme d'images simples, nettoyer les images en sélectionnant les régions non désirés avec le «Polygone Sélections" outil et les remplir (Édition → Remplir). (Redimensionner les images d'une taille plus petite, si nécessaire). Puis convertir chaque image en une image couleur RVB (Image → → Type de couleur RVB). Enfin, créer une pile à des images (Image → → Image à Stacks Stacks), après quoi ils peuvent encore être aligné avec le "Stack Reg" plug-in, si nécessaire. Finalement, la fusion des canaux, puis en combinant les images 3D crée un montage de l'ensemble des îlots dans le pancréas entier. Pour afficher l'image, cliquez sur "Plugins" puis "visionneuse 3D."
4. Cartographie Islet
Collecte des piles d'images
- Situer les îlots pancréatiques toute monter sous le microscope.
- Ouvrez le logiciel Slidebook. Accéder à la «capture et Focus Control" en appuyant sur "Ctrl + Maj + E." Dans le "Contrôle Focus" fenêtre, fixé comme objectif de 20x ou 40x, fonction de la taille de l'îlot. Appliquer de l'eau si vous utilisez un objectif immersion dans l'eau.
- Afin d'utiliser l'oculaire du microscope pour repérer les îlots, réglez "Bin" à 2X et mis le jeu de filtres à User1 dans le "contrôle le focus" fenêtre. "Sélection d'émission" doit être réglé sur Yeux 100% et de densité neutre à 1. Cliquez sur "GFP ey", puis "Ouvrir Fluor".
- Pour visualiser l'échantillon dans Slidebook, retour à la Focus fenêtre des contrôles et l'interrupteur "Définir le filtre" à "Live" et cliquez sur "GFP DS." Utilisez le joystick au centre de l'îlot dans la fenêtre de la caméra 1 aussi bien que possible. Focus pour une profondeur, quelque part près du centre de l'îlot, où les cellules peuvent être discernées facilement.
- Dans le «concentrer les contrôles de fenêtre", sélectionnez l'onglet "Z". Cliquez sur Configurer dans le coin supérieur gauche de la fenêtre afin de définir la profondeur actuelle comme point de référence. Utilisez le contrôle de la portée pour faire défiler à la profondeur la plus élevée de l'îlot dans lequel les caractéristiques peuvent être discernés clairement et cliquez sur «Set Top». Faites défiler vers le bas de l'îlot et cliquez sur «Set Bas." Cliquez sur "Go" pour revenir au point de référence.
- Dans la fenêtre de capture, réglez "Facteur Bin" à "2X" et réglez "Filter Set» à «en direct». Régler le "type de capture" à "3D" (et seulement en 3D). Sur le côté droit de la fenêtre, cliquez sur "Utiliser Positions Haut et Bas." Cliquez sur "Retour au point de référence après la capture." Réglez le "Step Size" à "3".
- Sous la "Box Set Filtre", sélectionnez DAPI Mémorandum d'accord, la GFP Mémorandum d'accord, DP Mémorandum d'accord, et Cy5 Mémorandum d'accord. Pour chacun d'eux, cliquez sur «Trouver meilleur" sous "ajuster l'exposition," puis cliquez sur "Test" pour s'assurer que l'exposition choisi est adapté. Cliquez sur "Démarrer" pour lancer la capture.
- Lorsque la capture est terminée, ajuster les niveaux que nécessaire et changer la couleur affichée pour DP au blanc. Enregistrer la diapositive et allez dans Affichage → Exporter → TIFF série. Tapez le nom de la diapositive avec un tiret à la fin et l'enregistrer. Des dizaines de fichiers TIFF individuels sont créés, de sorte qu'il peut être utile de garder la pile d'images chaque îlot dans un dossier séparé.
Cartographie des piles d'images
- Ouvrez chercheur stéréo. Visualisez l'image en ouvrant l'image de piles (Fichier → Image Stack Stack Image → Ouvrir). Dans le "menu Image Scaling," ensemble "distance entre les images" à 3,00 um. Pour corriger binning 2X en Slidebook, sélectionnez "Ignorer la mise à l'échelle X et Y» et «utilisateur spécifié pour la source de X et Y" Scaling, et entrez 0,65 pour X et Y.
- Centre l'image en cliquant dans le milieu de l'îlot d'intérêt dans la "fenêtre Macro." Marquer au moins une cellule en utilisant le marqueur approprié. Les bêta-cellules apparaissent en vert, les cellules alpha apparaissent en rouge, et les cellules delta apparaissent en blanc; Marker utiliser 2 (un cercle vide) pour marquer les cellules bêta, Marker 5 (un triangle creux) pour marquer les cellules alpha, et Marker 6 ( un triangle creux) pour les cellules delta. Les cellules doivent être marqués dans le centre de leur noyau, qui apparaissent en bleu si l'échantillon a été coloré avec du DAPI.
- Une fois au moins une cellule a été marquée, d'afficher la "vue orthogonale" à l'aide de l'icône dans le menu du haut. Cochez la case "Z-filtre» et «symétrique». La gamme de sHould être réglé à 15,00. Démarrage au niveau 0,0 Z, faire défiler l'îlot à l'aide de la molette de la souris et marquer chaque cellule avec le marqueur approprié. Chaque cellule doit être marqué qu'une seule fois au centre de sa profondeur. Après avoir terminé marquant chacun des Z-niveaux, le «Z-filtre" case peut être cochée. Cela montrera tous les marqueurs de tous les Z-niveaux.
- Lorsque chaque cellule a été marquée, enregistrez le fichier comme un "DAT", puis allez dans Fichier → Exporter Tracing. Enregistrer le tracé comme un "txt". Cliquez sur "Nouveau fichier de données" pour effacer l'espace de travail avant de tenter de cartographier de nouveaux îlots.
5. Les résultats représentatifs:
La préparation des tranches virtuelles à partir d'un échantillon de pancréas immunohistochimiquement teinté permet d'examiner l'ensemble des cellules endocrines (alpha, bêta et delta-cellules) dans le pancréas entier, les deux ensemble comme des îlots (figure 1A) et individuellement dans des canaux séparés ( Figure 1B). Avec l'aide de programmes informatiques et des scripts, une analyse de masse de données à grande échelle peuvent être réalisées sur ces tranches virtuelles. Plus précisément, une analyse des particules composites de masques (figure 1C) est sortie comme une table de statistiques contenant des paramètres tels que la superficie des îlots, de périmètre (la distance entourant une zone), la circularité (un degré de rondeur, où 1,0 représente un cercle parfait), et Diamètre Féret (la plus longue distance dans une zone) pour chaque îlot détectée (figure 1D). L'analyse à grande échelle de ces résultats d'images dans la production de nombre d'îlots totale et histogrammes de distribution de taille ainsi que d'une comparaison détaillée des alpha, bêta, delta et cellules domaines. De plus, chaque tranche virtuel est pris à une profondeur d'environ 5 microns, et l'ensemble de l'individu 2D tranches virtuelles sont encore empilés pour créer une reconstruction en 3D de l'échantillon du pancréas entier. Cartographie Islet démontre une autre instance non seulement de capturer des îlots en 3D, mais aussi des détails analyse assistée par ordinateur. Cartographie Islet se compose de la capture des îlots distincts (figure 2A) et le marquage des ultérieure alpha, bêta, delta-et les cellules à différents Z-plans (figure 2B) pour visualiser un îlot en 3D (figure 2C, D). Automatisé analyse mathématique des îlots cartographiés affiche leur composition cellulaire et l'architecture, y compris cellule-cellule distances (figure 2E) et les probabilités cumulées de distributions de distance cellule-cellule (figure 2F).
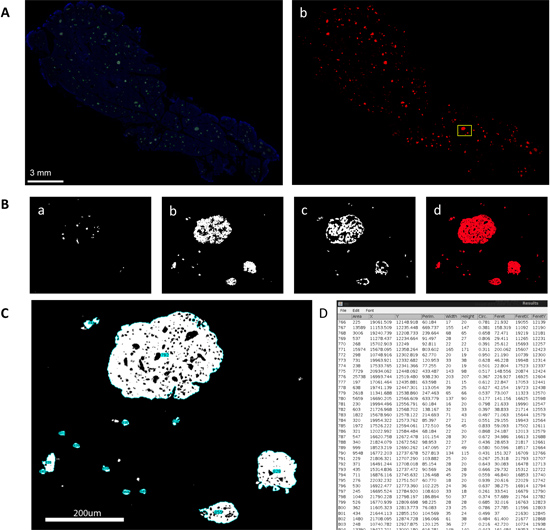
Figure 1. Grande échelle la capture et l'analyse de la distribution de l'îlot à l'aide Slice virtuel. Vue A. tranche virtuelle d'une section du pancréas humain. a. La coloration immunohistochimique de l'insuline (vert), le glucagon (rouge), la somatostatine (blanc) et le DAPI (bleu). b. Converti 8-bit masque après seuillage automatique. Une zone encadrée est amplifié dans les vues BB de chaque canal. a. delta-cellules, b. cellules bêta, c. les cellules alpha, et d. fusionnées image composite. L'analyse des particules C. effectuée sur le masque composite. Notez que chaque structure des îlots dont les grappes petite cellule est numérotée (surbrillance bleue). Tableau D. Statistiques des différents paramètres mesurés pour les structures individuelles, qui ont des références qui correspondent à des balises indiquées en C.

Figure 2. Analyse de Slice virtuel immunohistochimique. Parcelle A. dispersent 3D de la figure 1 montrant la distribution de taille et la forme de chaque îlot par des paramètres comme la superficie, la circularité et le diamètre de Féret. Parcelle B. dispersent 3D de la figure 1 montrant la composition des îlots cellulaires et la taille. La distribution de taille C Islet de l'analyse de la section humain tout entier de la figure 1 équipé d'une distribution de densité de probabilité lognormale. D. L'analyse mathématique des ratios de composition cellulaire (cellules bêta dans le vert, les cellules alpha en rouge et delta-cellules en bleu) pour chaque bac diamètre effectif îlot de la figure 1. E. a. Distribution de la taille de l'analyse Islet échantillonnage aléatoire immunohistochimiques (à gauche). Distribution de la taille de l'analyse Islet tranche virtuelle (à droite). b. Log-normale de comparaison parcelle de l'analyse immunohistochimique échantillonnage aléatoire (rouge) et la tranche virtuel (bleu).
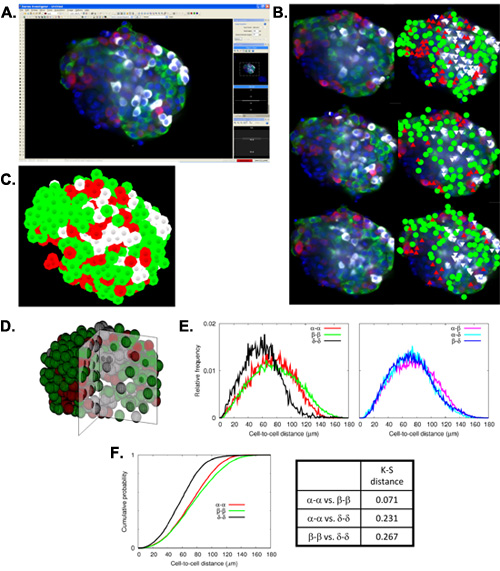
Figure 3. Cartographie Islet et l'analyse mathématique de la composition cellulaire et l'architecture. A: capture d'écran montrant un seul plan focal d'un empilement 3D reconstruite d'images d'un îlot humaines téléchargés dans Stéréo-chercheur (bêta-cellules en vert, les cellules alpha en rouge, le delta-cellules en blanc, et des noyaux en bleu) . B: les images de fluorescence (à gauche) et de données correspondant mappé (à droite) dans trois différents plans focaux montré à un intervalle de 10 um. C: vue 3D reconstruite Représentant de données de cartographie îlot. D: reconstruction 3D du quart de l'îlot en tranches sur la base des coordonnées obtenues par la cartographie des îlots. E: L'analyse mathématique de la composition cellulaire et l'architecture. Lauche. Fréquence relative des cellule-cellule distances entre deux cellules dans une population cellulaire unique. Droite. Fréquence relative de la cellule-cellule distances entre deux populations de cellules différentes. F: de Kolmogorov-Smirnov (KS) de test. Gauche. Probabilités cumulées de cellule à cellule distributions de distance pour l'alpha-à-alpha, bêta-à-bêta et delta-à-delta cellules. Droite. Distances KS pour les trois probabilités cumulées correspondantes.
Discussion
Le assistée par ordinateur à grande échelle de visualisation et de quantification présentée ici se permettre quatre points clés dans les études des îlots pancréatiques: (1) Une analyse à grande échelle d'échantillons pancréatiques fournit une vue complète de la distribution des îlots de taille globale et l'architecture des îlots. (2) La reconstruction 3D et l'analyse mathématique de la composition cellulaire et l'architecture de faciliter davantage l'examen de l'arrangement spati...
Déclarations de divulgation
Remerciements
L'étude est soutenue par l'US Public Health Service Grant DK-081527, 072473 et DK-DK-20595 à l'Université de Chicago recherche sur le diabète et le Centre de Formation (Animal Core Model), et un don de la Fondation de la Famille Kovler.
matériels
| Name | Company | Catalog Number | Comments | |
| Fluorescent microscope | Microscope | Olympus Corporation | IX-81 | |
| Stereo Investigator | Program | MBF Bioscience | ||
| MIP-GFP mice | Mice | Jackson Laboratory | ||
| Mathematica | Program | Wolfram | ||
| Image J | Program | National Institutes of Health | ||
| Slidebook | Program | Olympus |
Références
- Steiner, D. J., Kim, A., Miller, K., Hara, M. Pancreatic islet plasticity - Interspecies comparison of islet architecture and composition. ISLETS. 2, 135-145 (2010).
- Kim, A., Miller, K., Jo, J., Wojcik, P. l., Kilimnik, G., Hara, M. Islet architecture - a comparative study. ISLETS. 1, 129-136 (2009).
- Kilimnik, G., Kim, A., Jo, J., Miller, K., Hara, M. Quantification of pancreatic islet distribution in situ in mice. Am J Physiol Endocrinol Metab. 297, E1331-E1338 (2009).
- Hara, M., Dizon, R. F., Glick, B. S., Lee, C. S., Kaestner, K. H., Piston, D. W., Bindokas, V. P. Imaging pancreatic beta-cells in the intact pancreas. Am J Physiol Endocrinol Metab. 290, E1041-E1047 (2006).
- Hara, M., Wang, X., Kawamura, T., Bindokas, V. P., Dizon, R. F., Alcoser, S. Y., Magnuson, M. A., Bell, G. I. Transgenic mice with green fluorescent protein-labeled pancreatic beta -cells. Am J Physiol Endocrinol Metab. 284, E177-E183 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon