Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Test in vitro de l'adhésion bactérienne sur des mammifères cellules épithéliales
Dans cet article
Résumé
Ce protocole est un test simple adhésion bactérienne consistant à compter le nombre de colonies bactériennes formant des unités qui sont collées sur des cellules cultivées. Le test est robuste, indépendante de l'adhésine étudié, et de nombreuses variations sont utilisés dans la plupart des laboratoires travaillant sur la pathogenèse bactérienne.
Résumé
Pour provoquer des infections, les bactéries colonisent doit leur hôte. Les bactéries pathogènes d'exprimer différentes molécules ou de structures capables de favoriser l'attachement aux cellules hôtes 1. Ces adhésines s'appuyer sur les interactions avec les récepteurs de l'hôte à la surface cellulaire ou des protéines solubles agissant comme un pont entre les bactéries et l'hôte. L'adhésion est une première étape essentielle avant l'invasion et / ou la sécrétion de toxines, il est donc un événement clé pour être étudiés dans la pathogénie bactérienne. Par ailleurs, les bactéries induisent souvent respectées délicieusement affiné les réponses cellulaires, les études qui ont donné naissance au domaine de la 2 'microbiologie cellulaire. Dosages robuste pour l'adhérence des bactéries sur les cellules hôtes et leur invasion par conséquent jouer un rôle clé dans la pathogenèse bactérienne et les études ont longtemps été utilisés dans de nombreux laboratoires pionniers 3,4. Ces tests sont maintenant pratiquée par la plupart des laboratoires travaillant sur la pathogenèse bactérienne.
Ici, nous décrivons un test standard de l'adhésion illustrant la contribution d'une adhésine spécifique. Nous utilisons la souche d'Escherichia coli 2787 5, une souche pathogène humaine exprimant la Autotransporter impliquées dans l'adhérence diffuse (ACRA) Adhesin. Comme contrôle, on utilise une souche mutante dépourvue du gène aida, 2787Δ aida (F. Berthiaume et M. Mourez, non publié), et une souche de laboratoire commercial de E. coli, C600 (New England Biolabs). Les bactéries sont de gauche à adhérer aux cellules de l'couramment utilisés HEp-2 lignées cellulaires épithéliales humaines. Ce test a été moins largement décrite avant 6.
Protocole
1. Préliminaire: Les souches bactériennes et les cellules épithéliales.
Les manipulations des cellules et des bactéries sont réalisées de façon aseptique, sous une hotte à flux laminaire.
- Fraîchement isoler l'E. coli 2787, 2787Δ Aida et C600 à partir de stocks de glycérol sur bouillon lysogénie (LB) gélose (1% de tryptone, le chlorure de sodium à 0,5%, extrait de levure 0,5%, 1,5% de gélose) et de croître à 37 ° C. Afin de minimiser la variabilité dans le dosage, il est conseillé de toujours utiliser des souches fraîchement plaquée et de garder les souches à 4 ° C sur des plaques de Pétri scellées seulement pour un maximum de deux semaines.
- Culture cellules HEp-2 (ATCC CCL-23) au moyen de modification de glucose élevé Dulbecco Eagle (DMEM) supplémenté avec 10% inactivé par la chaleur du sérum bovin (inactivation thermique est réalisé à 56 ° C pendant 30 min.) Pendant la culture de routine, nous aussi ajouter 10 U / ml de pénicilline et 10 pg / ml de streptomycine.
- Cultivez cellules Hep-2 à 37 ° C dans une cellule avec une atmosphère contenant 5% de CO 2 incubateur. Utiliser les procédures standard de culture cellulaire pour maintenir les cellules, qui sont cultivés en fioles de 75 cm2 et repiquées chaque fois qu'ils atteignent la confluence. Nous utilisons nos cellules en culture jusqu'à ce qu'ils atteignent 30 à 40 passages et puis les jeter.
- Préparer un essai lorsque les cellules HEp-2 dans un flacon de 75 cm2 sont presque atteindre la confluence et sont donc prêts pour un test d'adhérence.
2. JOUR 1: Préparation de l'inoculum et les cellules épithéliales
- Laver les-deux cellules Hep dans le ballon une fois avec du phosphate chaude Dulbecco saline tamponnée (DPBS: 8 g / L de NaCl, 0,2 g / L de KCl, 0,2 g / L KH 2 PO 4, 0,21 g / L de Na 2 HPO 4: 7H 2 O)
- Les cellules sont incubées avec 0,05% de trypsine - EDTA pendant 5 min avant d'ajouter fraîcheur milieu chaud complet. Après un milieu frais est ajouté, les cellules sont remises en suspension par aspiration et refoulement.
- Centrifuger la suspension cellulaire à 2000 rpm pendant 5 min et remettre les boulettes dans du DPBS et centrifuger à nouveau.
- Resuspendre les cellules dans un milieu frais supplémenté avec du sérum de 10% mais ne contenant pas d'antibiotiques, à une concentration de 2x10 5 cellules / ml.
- Graines un millilitre de suspension cellulaire dans trois ensembles de deux puits (un pour chaque souche) dans le centre d'une plaque de 24 puits et incuber la plaque pendant la nuit dans un incubateur de culture cellulaire.
Remarque: Il est important que le sérum décomplémenté être soigneusement et que les antibiotiques sont omis après cette étape. Ne pas le faire pourrait tuer les bactéries infectant. - Inoculer une colonie isolée de chaque souche bactérienne (2787, 2787Δ aida et C600) dans 5 ml de bouillon LB (1% de tryptone, le chlorure de sodium à 0,5%, extrait de levure 0,5%) et pousser une nuit à 37 ° C avec une agitation vigoureuse (180 tours par minute ).
3. JOUR 2: L'infection de cellules
- Inspectez les cellules HEp-2, sous un microscope inversé pour s'assurer qu'ils sont au moins 90% de confluence et non contaminés.
- Lavé les cellules avec du DPBS chaud et, dans chaque puits, ajouter 1 ml de milieu frais supplémenté avec du sérum de 10% mais ne contenant pas d'antibiotiques. Ajoutez également un milieu frais à trois puits sans cellules. Il sera utilisé pour déterminer le nombre total de bactéries présentes dans l'inoculum pour chaque souche.
- Mesurer la densité optique à 600 nm (DO 0,600 nm) des cultures bactériennes. Ajouter une aliquote de chaque culture bactérienne à un ensemble de deux puits contenant les cellules Hep-2 et à un bien ne contenant pas de cellules. Habituellement, nous utilisons un volume de culture de nuit correspondant à 10 6 unités formant colonies (UFC). Cela représente une multiplicité d'infection (MOI) de 5h01 (bactéries: les cellules). Bien que nous utilisons directement nos cultures de la nuit, il est parfois conseillé de centrifuger les bactéries et les remettre en suspension dans du DPBS pour éviter les effets délétères des molécules sécrétées présents dans les cultures de la nuit (comme cytotoxines, par exemple)
Remarque: Le MOI peut varier entre 100:1 et 01:10. MOI plus élevé des rendements plus élevés et de la variabilité de fond et les bactéries ont tendance à coller au plastique de la plaque. Basse MOI donne aussi une forte variabilité. Une fois une MOI est choisi, il est impératif d'être cohérent et de garder cette MOI. - Incuber les cellules infectées dans la culture cellulaire incubateur pendant 3 heures à 37 ° C avec 5% de CO 2.
Note: Il est également possible de centrifuger brièvement la plaque à basse vitesse (par exemple 1000 xg pendant 1-2 min), afin d'apporter toutes les bactéries directement en contact avec les cellules. Cela a l'avantage supplémentaire de synchronisation de l'infection, et permettant des temps d'incubation plus courte (aussi peu que 15-30 min). - Retirez le support à partir des cellules infectées et laver les cellules 3 fois avec du DPBS chaud. À cette étape, les bactéries adhérentes peuvent généralement être vu avec un microscope standard et nous avons l'habitude d'effectuer cette vérification.
- Pour lyser les cellules et détacher le bac respectéestères, ajouter 100 l de 1% de Triton X-100 dans chaque puits contenant les cellules. Autres détergents peuvent être utilisés (tel que la saponine par exemple).
- Incuber les cellules pendant 10 min à température ambiante, puis ajouter 900 ul de milieu LB.
- Doucement homogénéiser les suspensions par répétés de haut en bas de pipetage. Le puits sans cellules contenant l'inoculum sont également homogénéisé doucement.
Remarque: Certaines bactéries pourraient être trop sensibles aux détergents, dans ce cas nous détacher les cellules épithéliales par incubation avec la trypsine 0,05% - EDTA pendant 20 min à 37 ° C. Les cellules avec des bactéries peuvent également être respectées raclées des puits avec une pipette. - Préparer série 10 des dilutions des suspensions de bactéries respectées et l'inoculum à l'aide de bouillon LB et la plaque 100 ul de 3 dilutions (généralement les dilutions 1:1000, 1:10.000 et 1:100.000) sur gélose LB et incuber une nuit à 37 ° C .
4. JOUR 3: comptage ufc et la présentation des données.
- Le comptage des colonies sur les plaques et de calculer le nombre d'UFC de bactéries adhéré et de l'inoculum en faisant la moyenne de chaque série de dilutions. Seules les plaques avec entre 10 à 300 colonies doivent être comptées.
5. Les résultats représentatifs:
Le tableau suivant montre des résultats typiques d'ufc comptage des bactéries respectées et l'inoculum à partir de 3 expériences réalisées à des jours différents:

Les résultats peuvent être rapportés directement comme respectées ufc, comme le montre la figure 1A. Depuis les tailles d'inoculum peut varier entre les souches en raison de différences dans les taux de croissance ou des erreurs de pipetage, il est souvent conseillé de rapporter les résultats en pourcentage des bactéries collées. Le pourcentage de bactéries respectées peut être calculé en divisant le nombre d'UFC de bactéries respectées par le nombre de cfu de l'inoculum, comme montré dans la figure 1B. En effectuant une mesure répétée ANOVA et post-tests de Bonferroni pour comparer toutes les colonnes de la figure 1B, on peut voir qu'il ya des différences significatives (p <0,05) entre 2787 et 2787Δ aida, ainsi qu'entre 2787 et C600, mais aucune différence significative entre 2787Δ aida et C600.
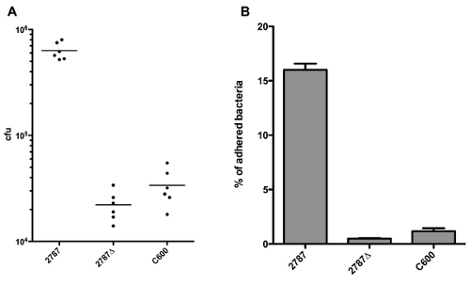
Figure 1. Représentation des résultats en UFC de bactéries respectées (A) ou le pourcentage de bactéries respectées (B)
Le pourcentage d'adhésion observée est souvent très dépendante de l'expérimental (et, dans une moindre mesure, sur l'expérimentateur). Particulièrement importantes sont la MOI et le nombre de lavages, de sorte que le pourcentage ne doit pas être interprétée littéralement.
Les bactéries présentes dans l'inoculum peut parfois se développent beaucoup plus vite que les bactéries dans la présence de cellules, biaisant la taille de l'inoculum par comparaison avec le nombre réel total de bactéries dans les puits avec des cellules. Pour remédier à ce problème, il est recommandé de: (i) l'utilisation des puits avec des cellules afin de déterminer l'inoculum: cellules HEp-2 sont ensemencées dans un puits supplémentaire et infectées. A la fin de l'essai, au lieu de jeter le surnageant et laver, 100 ul de 10% de Triton X-100 est ajoutée à la bien. Les cellules seront Lyse et le lysat contenant des bactéries respectées et non respecté est doucement homogénéisé par répétés de haut en bas de pipetage;. Ou (ii) de recueillir les surnageants de cellules infectées à la fin de l'essai, ainsi que les surnageants de les lavages DPBS et déterminer le nombre d'UFC dans les surnageants mis en commun. Cela donnera le nombre de cfu de bactéries non respectées. Le pourcentage de bactéries respectées peut alors être calculé en divisant le nombre d'UFC de bactéries respectées par l'ajout du nombre d'UFC de bactéries respectées et non-respectées.
Pour illustrer la robustesse de l'analyse, nous pouvons comparer ce protocole avec le test effectué avec S4074, une souche porcine adhésive pathogènes d'Actinobacillus pleuropneumoniae 7, incubées avec des cellules d'une lignée récemment créé porcelets trachéale cellulaire, nptr 8. Mis à part les différences dans les médias nécessaires à la croissance des bactéries et des cellules, la seule différence est que les bactéries utilisées pour l'infection d'une culture sont en croissance exponentielle, et non d'une culture en phase stationnaire pendant la nuit. Avec A. pleuropneumoniae, il est également très important de respecter une MOI de 10h01 et à ne pas dépasser 3 heures de l'infection, sinon la sécrétion de toxines peut entraîner la mort cellulaire et biaiser les résultats. De plus, cette souche de A. pleuropneumoniae peut facilement adhérer au plastique de sorte qu'il est important d'utiliser des cellules confluentes et vérifier visuellement que les cellules ne sont pas desquamation.
Discussion
Ce protocole décrit un dosage standard de l'adhérence des bactéries qui peuvent être modifiés pour étudier l'invasion (par exemple en utilisant la gentamicine protection de test 3). Le comptage des unités formant colonie permet la quantification, par comparaison avec les approches s'appuyant sur des techniques de visualisation standard sous un microscope, comme la coloration de Giemsa. Ce dernier ne donne qu'une vue qualitative de l'adhérence, mais est souvent un complément utile ...
Déclarations de divulgation
Remerciements
Le travail dans les laboratoires des auteurs est soutenu par des subventions de recherches en sciences naturelles et en génie du Canada, les Instituts de recherche en santé du Canada d', et le Programme des chaires de recherche du Canada. JL est soutenu par une bourse de la Groupe d'Etude des Protéines Membranaires (GEPROM) grâce au financement du Fonds de Recherche en santé du Québec.
matériels
| Name | Company | Catalog Number | Comments |
| Réactifs | Société | Numéro de catalogue | |
|---|---|---|---|
| De glucose à haute DMEM | GIBCO-Invitrogen | 12430-054 | |
| DDPBS | GIBCO-Invitrogen | 14190-144 | |
| Sérum de croissance bovine | Hyclone | SH3054 | |
| Pénicilline / streptomycine | GIBCO-Invitrogen | 15140-148 | |
| Plaques de 24 puits | Corning | 3337 | |
| Triton X-100 | Fisher Scientific | BP151-100 |
Références
- Kline, K. A. Bacterial adhesins in host-microbe interactions. Cell Host Microbe. 5, 580-580 (2009).
- Cossart, P., Boquet, P., Normark, S., Rappuoli, R. Cellular microbiology emerging. Science. 271, 315-315 (1996).
- Elsinghorst, E. A. Measurement of invasion by gentamicin resistance. Methods Enzymol. 236, 405-405 (1994).
- Pizarro-Cerda, J., Lecuit, M., Cossart, P., Sansonetti, P., Zychlinsky, A. . Molecular Cellular Microbiology. , 161-161 (2002).
- Srivastava, A., Isberg, R. R., Sansonetti, P., Zychlinsky, A. . Molecular Cellular Microbiology. , 179-179 (2002).
- Benz, I., Schmidt, M. A. Cloning and expression of an adhesin (AIDA-I) involved in diffuse adherence of enteropathogenic Escherichia coli. Infect Immun. 57, 1506-1506 (1989).
- Charbonneau, M. E., Berthiaume, F., Mourez, M. Proteolytic processing is not essential for multiple functions of the Escherichia coli autotransporter adhesin involved in diffuse adherence (AIDA-I). J Bacteriol. 188, 8504-8504 (2006).
- Jacques, M., Paradis, S. E. Adhesin-receptor interactions in Pasteurellaceae. FEMS Microbiol Rev. 22, 45-45 (1998).
- Auger, E. Host-pathogen interactions of Actinobacillus pleuropneumoniae with porcine lung and tracheal epithelial cells. Infect Immun. 77, 1426-1426 (2009).
- Jacques, M. Role of lipo-oligosaccharides and lipopolysaccharides in bacterial adherence. Trends Microbiol. 4, 408-408 (1996).
- Nataro, J. P., Kaper, J. B. Diarrheagenic Escherichia coli. Clin Microbiol Rev. 11, 142-142 (1998).
- Jallat, C., Darfeuille-Michaud, A., Rich, C., Joly, B. Survey of clinical isolates of diarrhoeogenic Escherichia coli: diffusely adhering E. coli strains with multiple adhesive factors. Res Microbiol. 145, 621-621 (1994).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon